
 , 王红1
, 王红1

1 中国科学院大学,北京 100049;
2 中国科学院遗传与发育生物学研究所,北京 100101
收稿日期:2017-07-21;接收日期:2017-09-12 基金项目:国家自然科学基金(Nos. 61672489, 61379081)资助
摘要:植物Ⅲ型聚酮合酶(Polyketide synthases,PKSs)催化形成一系列结构迥异、生理活性不同的聚酮类化合物的基本骨架结构,是聚酮类化合物生物合成途径的关键酶。目前已从植物中克隆和鉴定了多种功能不同的Ⅲ型PKSs。定点突变技术是研究蛋白质结构与功能之间复杂关系的重要方法。文中综述了近年来基于定点突变的植物Ⅲ型PKSs结构与功能关系的研究进展,包括利用定点突变技术修饰各种可能影响植物Ⅲ型PKSs结构的氨基酸残基,来研究其对功能的影响(如控制起始底物的特异性、缩合反应次数以及中间产物环化方式),以期为植物Ⅲ型PKSs结构与功能关系的研究提供参考。
关键词:聚酮类化合物 植物Ⅲ型聚酮合酶 结构与功能 定点突变
Advances in structure-function relation of plant type Ⅲ polyketide synthases by site-directed mutagenesis
Li Xing1,2, Chen Mo1, Chai Tuanyao1

 , Wang Hong1
, Wang Hong1

1 University of Chinese Academy of Sciences, Beijing 100049, China;
2 Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101, China
Received: July 21, 2017; Accepted: September 12, 2017
Supported by: National Natural Science Foundation of China (Nos. 61672489, 61379081)
Corresponding author:Tuanyao Chai. Tel: +86-10-88256343; E-mail: tychai@ucas.ac.cn
Hong Wang. Tel: +86-10-69672628; E-mail: hwang@ucas.ac.cn
Abstract: Plant type Ⅲ polyketide synthases (PKSs), the pivotal enzymes in the biosynthesis of polyketides, produce backbones of many structurally diverse and functionally different polyketides. So far, a variety of functionally diverse plant type Ⅲ PKSs have been cloned and identified from plant origin. Site-directed mutagenesis is a useful technique to study the complex relationship between protein structure and function. This review summarized advances in the structure-function relation of plant type Ⅲ polyketide synthases by site-directed mutagenesis in recent years, including the modification of the amino acid residues influencing enzyme architectures (such as controlling the specificity of starter substrates, the number of condensation reactions, and the cyclization reactions of the intermediate product). This review provides information to study the structure-function relation of plant type Ⅲ polyketide synthases.
Key words: polyketides plant type Ⅲ polyketide synthases structure and function site-directed mutagenesis
聚酮类化合物(Polyketides)是一大类在植物、微生物中广泛存在的结构复杂、功能多样的天然产物,具有抗菌、消炎、抗氧化、抗病毒、抗肿瘤和免疫抑制等药理活性。聚酮类化合物虽然种类繁多、结构复杂,但其在生物体内具有共同的合成机制,聚酮合酶(Polyketide synthases,PKSs)是催化聚酮类化合物合成的关键酶。根据PKSs酶系的结构,可将其分为Ⅰ、Ⅱ和Ⅲ型PKSs。其中,Ⅰ型PKSs多存在于细菌和真菌中,是一类以模块形式存在的多功能酶,每一模块含有一套独特的、非重复使用的催化功能域[1]。Ⅱ型PKSs存在于土壤微生物中,是一个多功能酶复合体,只包含一套可重复使用的结构域。Ⅲ型PKSs即查尔酮合酶超家族(Chalcone synthase superfamily, CHS superfamily),主要分布在植物界,近年来在细菌和真菌中也鉴定了多种Ⅲ型PKSs[2]。植物Ⅲ型PKSs与Ⅰ型和Ⅱ型PKSs相比结构简单,由40?45 kDa的同源二聚体组成,每个亚基的活性中心具有多种活性,能够完成底物加载、聚酮链延伸、特定的环化和产物的释放。
植物Ⅲ型PKSs催化形成一系列结构迥异、生理活性不同的聚酮类化合物的骨架结构,包括查尔酮(Chalcone)、二苯乙烯(Stilbene)、间苯三酚(Phloroglucinol)、间苯二酚(Resorcinol)、萘(Naphthalene)、吖啶酮(Acridone)、吡喃酮(Pyrone)、联苄(Bibenzyl)、苯亚甲基丙酮(Benzalacetone)、二苯甲酮(Benzophenone)、色原酮(Chromone)、联苯(Biphenyl)、喹诺酮(Quinolinone)、姜黄素(Curcuminoid)、异香豆素(Isocoumarin)、芪-羧酸酯(Stilbenecarboxylate)等[3-4]。植物Ⅲ型PKSs的功能多样性主要体现在对起始底物的选择性、缩合反应次数(聚酮链延伸的长度)以及产物的不同环化方式等[5]。植物Ⅲ型PKSs可以利用多种酰基辅酶A (Coenzyme A, CoA)作为起始底物,包括脂肪族酰基CoA和芳香族酰基CoA,从较小的乙酰CoA到较大的N-甲基邻氨基苯甲酰-CoA (N-methylanthraniloyl-CoA),从极性的丙二酰CoA到非极性的N-己酰CoA均可以作为其底物。尽管多数植物Ⅲ型PKSs催化3次缩合反应,但不同的植物Ⅲ型PKSs催化的缩合反应次数可以从1次到8次不等。植物Ⅲ型PKSs催化产物环化反应的方式主要包括:C6→C1 Claisen型环化(Claisen-type cyclization),如查尔酮合酶(Chalcone synthase, CHS)、吖啶酮合酶(Acridone synthase, ACS)、苯戊酮合酶(Valerophenone synthase, VPS)、二苯甲酮合酶(Benzophenone synthase, BPS);C2→C7 Aldol型环化(Aldol-type cyclization),如芪合酶(Stilbene synthase, STS)、二羟基戊苯合酶(Olivetol synthase, OLS);内酯化(Lactonization),如2-吡喃酮合酶(2-Pyrone synthase, 2-PS)。另外,还有一些植物Ⅲ型PKSs产物不经过环化,如苯亚甲基丙酮合酶(Benzalacetone synthase, BAS)和姜黄素合酶(Curcuminoid synthase, CUS)[3] (图 1)。正是由于植物Ⅲ型PKSs起始底物的特异性、缩合反应次数以及产物环化方式三个方面的不同决定了植物聚酮类化合物的数量、结构以及生理活性的多样性[3, 5]。
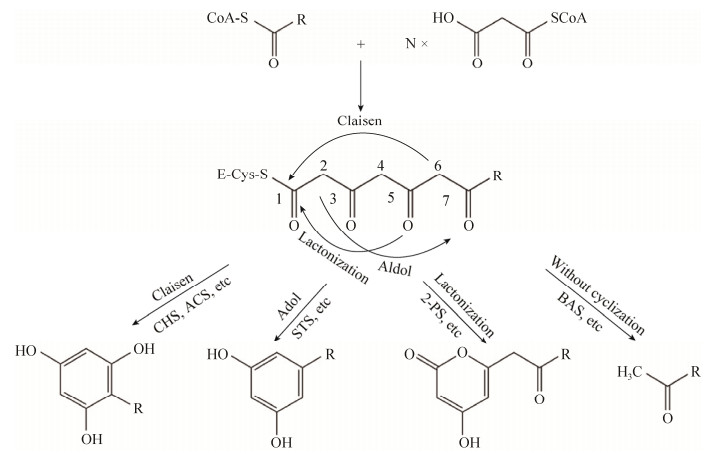 |
| 图 1 植物Ⅲ型PKSs所催化的反应及产物(环化反应类型及位置在图中标出) Figure 1 Typical enzyme reactions catalyzed by plant type Ⅲ PKSs (The position and type of cyclization reaction of the presumed linear polyketide intermediate are depicted). ACS: acridone synthase; BAS: benzalacetone synthase; CHS: chalcone synthase; 2-PS: 2-pyrone synthase; STS: stilbene synthase. |
| 图选项 |
紫花苜蓿CHS (Medicago sativa CHS, MsCHS)等植物Ⅲ型PKSs晶体结构的解析和定点突变研究表明,植物Ⅲ型PKSs家族成员具有相似的三维结构和催化机制[6-8]。Cys164、His303和Asn336 (以MsCHS氨基酸序列编号,以下未特别说明均按此编号) 3个位点的氨基酸残基在已发现的植物Ⅲ型PKSs中高度保守,它们组成酶活性中心催化三联体(Catalytic triad)。其中Cys164是聚酮链形成过程中的亲核活性位点,通过共价键与起始底物分子和反应中间体结合,而His303和Asn336则起着稳定或活化起始底物和延伸底物的作用。植物Ⅲ型PKSs的催化机制为:1)起始底物-CoA加载到酶活性中心Cys164的SH基团;2)延伸底物丙二酰CoA脱羧形成乙酰CoA;3)聚酮链开始延伸;4)聚酮中间体-酶复合物完成环化和芳构化[7]。MsCHS晶体结构的解析鉴定了位于活性腔内对起始底物的选择性和聚酮链的延伸长度具有决定作用的多个氨基酸残基,包括Thr132、Ser133、Thr194、Thr197、Gly256、Phe265和Ser338,这些氨基酸残基在CHS中保守,但在其他Ⅲ型PKSs中被相应的氨基酸替代,正是由于特定位置上这些重要氨基酸的替换决定了不同的植物Ⅲ型PKSs对起始底物的选择性和聚酮链延伸长度的不同,从而形成不同的产物[7-8]。
定点突变技术是研究蛋白质结构与功能之间复杂关系的重要工具,近年来随着不同的植物Ⅲ型PKSs基因的克隆和鉴定,人们通过定点突变技术对各种可能影响Ⅲ型PKSs所催化反应(如控制聚酮链长、催化腔大小和环化机制)的氨基酸残基进行了定点突变研究以揭示这类酶的结构与功能的关系。本文将综述近年来基于定点突变技术的植物Ⅲ型PKSs结构与功能关系的研究进展。
1 已鉴定的植物Ⅲ型PKSs植物Ⅲ型PKSs家族中,发现最早、分布最广泛、研究最为透彻的是查尔酮合酶CHS。1983年,Reimold等从欧芹Petroselinum hortense的培养细胞中克隆了第一个植物Ⅲ型PKSs家族成员—查尔酮合酶基因[9],目前已从苔藓、蕨类、裸子植物和被子植物中克隆到大量的CHS基因。该酶是类黄酮类化合物生物合成途径的关键酶,它催化来自丙二酰辅酶A (Ⅲ型PKSs的延伸底物)的3个乙酰基团通过连续3步缩合反应连接到4-香豆酰辅酶A (Ⅲ型PKSs的起始底物)分子上,形成四酮(Tetraketide)中间体,之后通过克莱森型(Claisen type)环化反应生成芳香族聚酮化合物柚皮素查尔酮(Naringenin chalcone)—类黄酮类化合物生物合成的前体。芪合酶STS是另一个被广泛研究的植物Ⅲ型PKSs家族成员,系统发育分析表明,该基因是植物在进化过程中由CHS进化来的,不同植物中二者之间具有65%以上的氨基酸序列同源性。1988年,Schr?der等首次从花生Arachis hypogaea培养细胞中克隆了STS基因[10],目前已从被子植物、裸子植物、蕨类植物中克隆到多个STS基因[11]。STS利用与CHS相同的起始底物4-香豆酰CoA,催化3分子丙二酰CoA缩合形成与CHS相同的四酮中间体,但随后二者的环化方式不同,STS催化四酮中间体通过分子内C2→C7 Aldol型缩合形成白藜芦醇,其中C1以CO2形式被脱去。与植物中普遍存在的CHS不同,STS只存在于能够产生白藜芦醇的植物中。
除了CHS和STS,植物中已鉴定的Ⅲ型PKSs还包括联苄合酶(Bibenzyl synthase, BBS)[12]、2-PS[13]、C-methylchalcone synthase (PstrCHS2)[14]、VPS[15]、p-Coumaroyltriacetic acid synthase, CTAS)[16]、ACS[17]、BAS[18]、BPS[19]、Stilbenecarboxylate synthase (STCS)[20]、芦荟松合酶(Aloesone synthase, ALS)[21]、聚五酮色原酮合酶(Pentaketide chromone synthase, PCS)[22]、聚八酮合酶(Octaketide synthase, OKS)[23]、联苯合酶(Biphenyl synthase, BIS)[24]、CUS[25]、聚六酮合酶(Hexaketide synthase, HKS)[26-27]、Isobutyrophenone synthase (BUS)[28]、OLS[29]、喹诺酮合酶(Quinolone synthase, QNS)[30]、5-甲基间苯二酚合酶(Orcinol synthase, ORS)[31]、烷基乙酮CoA合酶(Alkyldiketide-CoA synthase, ADS)和烷基喹诺酮合酶(Alkylquinolone synthase, AQS)[32]等。不同的植物Ⅲ型PKSs之间的差别主要体现在起始底物的特异性、缩合反应次数(聚酮链延伸的长度)以及产物合成所采取的不同环化方式等[5]。现在一般认为植物PKSs起源于脂肪酸合酶(Fatty acid synthases, FASs)系统,由具有类似结构的KS Ⅲ (Ketoacyl synthase Ⅲ)进化而来,后者与现在的PKSs具有相似的三级结构和底物结合位点[5]。
2 植物Ⅲ型PKSs序列分析随着越来越多的植物Ⅲ型PKSs被鉴定,序列分析表明,植物Ⅲ型PKSs之间氨基酸序列相似性在60%?75%以上,都编码约400个氨基酸的蛋白质,在体内通常以同源二聚体的形式存在和发挥功能[3]。查尔酮合酶基因在裸子植物和被子植物中大多以多个拷贝存在,同时,由于查尔酮合酶基因和其他植物Ⅲ型聚酮合酶基因的核苷酸序列具有很高的一致性和相似性,这意味着很可能现在的植物Ⅲ型聚酮合酶家族基因是由查尔酮合酶基因经过基因的复制和变异进化而来的[33]。
本文所涉及的植物Ⅲ型PKSs的氨基酸序列比对结果见图 2。从图中可以看出,组成酶活性中心催化三联体的3个氨基酸残基Cys164、His303和Asn336在所有已发现的Ⅲ型PKSs中绝对保守;起到“Gatekeeper”作用的2个氨基酸残基Phe215和Phe265,具有调节活性腔与CoA结合通道之间的空间结构的功能,这2个氨基酸残基在大多数PKSs中高度保守,但在有些PKSs中被其他氨基酸所替换,如掌叶大黄Rheum palmatum的BAS中Phe215被Leu替代[18],虎杖Polygonum cuspidatum的PcPKS2 (功能上是1个BAS)中,Phe215和Phe265分别被Leu和Cys取代[34] (图 2)。另外,位于活性中心腔内的多个氨基酸残基,包括Thr132、Ser133、Thr194、Thr197、Gly256、Phe265和Ser338,被认为对聚酮链的延伸长度以及对起始底物和产物的特异性具有决定作用。这些氨基酸残基在CHS中保守,而在其他不同功能Ⅲ型PKSs中则被相应的氨基酸替代(图 2)。因此,这些氨基酸残基通常是研究Ⅲ型PKSs结构与功能关系、基于结构的Ⅲ型PKSs分子定向改造的重要靶点。
 |
| 图 2 植物Ⅲ型PKSs家族成员氨基酸序列比较 Figure 2 Comparison of the amino acid sequences of plant type Ⅲ PKSs. The catalytic triad (Cys164, His303, Asn336) are marked with dots. The two gatekeepers (Phe 215 and Phe 265) are marked with triangles. The critical residues lining the active-site cavity (Thr132, Ser133, Thr197, Gly256 and Ser 338) are marked with squares (numbering in MsCHS). PsSTS: Pinus sylvestris stilbene synthase; MsCHS: Medicago sativa chalcone synthase; GH2PS: Gerbera hybrid 2-pyrone synthase; AaOKS: Aloe arborescens octaketide synthase; AaPCS: A. arborescens pentaketide chromone synthase; HaBPS: Hypericum androsaemum benzophenone synthase; RpALS: Rheum palmatum loesone synthase; RpBAS: R. palmatum bezalacetone synthase; RgACS: Ruta graveolens acridone synthase; AmQNS: Aegle marmelos quinolone synthase; ErADS: Evodia rutaecarpa alkyldiketide-CoA synthase; ErAQS: E. rutaecarpa alkylquinolone synthase. |
| 图选项 |
3 利用定点突变研究植物Ⅲ型PKSs结构与功能的关系3.1 2-吡喃酮合酶2-吡喃酮合酶2-PS催化起始底物乙酰CoA和2分子丙二酰CoA缩合形成吡喃酮。2000年,Jez等解析了非洲菊Gerbera hybrid 2-PS-乙酰乙酰CoA复合物2.05 ?的晶体结构。结果表明,非洲菊2-PS与MsCHS具有相似的三维折叠、相同的催化残基和高度保守的CoA结合位点[8]。进一步的结构解析显示,MsCHS中位于活性腔的Thr197、Ile254、Gly256和Ser338在非洲菊2-PS中分别被Leu、Met、Leu和Ile替代,这4个氨基酸的替代使得非洲菊2-PS催化腔的体积只有MsCHS的1/3,因此只能利用比4-香豆酰CoA侧链基团更小的乙酰CoA作为起始底物,催化2个乙酰基团通过连续的缩合反应连接到乙酰CoA上,之后由内酯化反应生成6-甲基-4-羟基-2-吡喃酮(6-Methyl-4-hydroxy-2-pyrone)。作者随后进行的定点突变研究表明,将MsCHS的197位、256位和338位3个位点的氨基酸同时突变为非洲菊2-PS相对应的氨基酸后,由于T197L/G256L/S338I突变后各氨基酸残基的侧链基团较野生型变大,使得突变体不能接受4-香豆酰CoA为起始底物,而是以乙酰CoA为起始底物与2分子丙二酰CoA缩合形成较查尔酮分子量更小的吡喃酮,在功能上已完全转变为1个2-PS (表 1),这也是首次实现对植物Ⅲ型PKS的定向改造[8]。他们结合晶体结构信息进一步证明,MsCHS这3个氨基酸残基的突变使其催化腔体积变小,从而影响了酶对起始底物分子的选择和聚合反应的次数,从而最终决定了产物分子的大小。基于上述结果,Jez等提出了空间模型理论(Steric modulation)来解释Ⅲ型PKS家族蛋白底物和反应类型的多样性。该模型认为,Ⅲ型PKSs活性口袋空间的大小决定了它们起始底物的大小和聚酮产物延伸的长度。非洲菊2-PS的活性口袋大小只有MsCHS的1/3,所以更适合接受乙酰辅酶A这种侧链基团较小的底物[8]。此外,Abe等(2005)同源建模的结果表明,能够催化乙酰CoA与丙二酰CoA进行8次聚合反应的木立芦荟Aloe arborescens的OKS,其催化口袋的体积确实要比仅能催化3次聚合反应的MsCHS更大,这也是空间模型理论的证据之一[23]。
表 1 植物Ⅲ型PKSs定点突变后底物、产物及功能的比较Table 1 Comparison of plant type Ⅲ PKSs in substrates, products and functions after site-directed mutagenesis
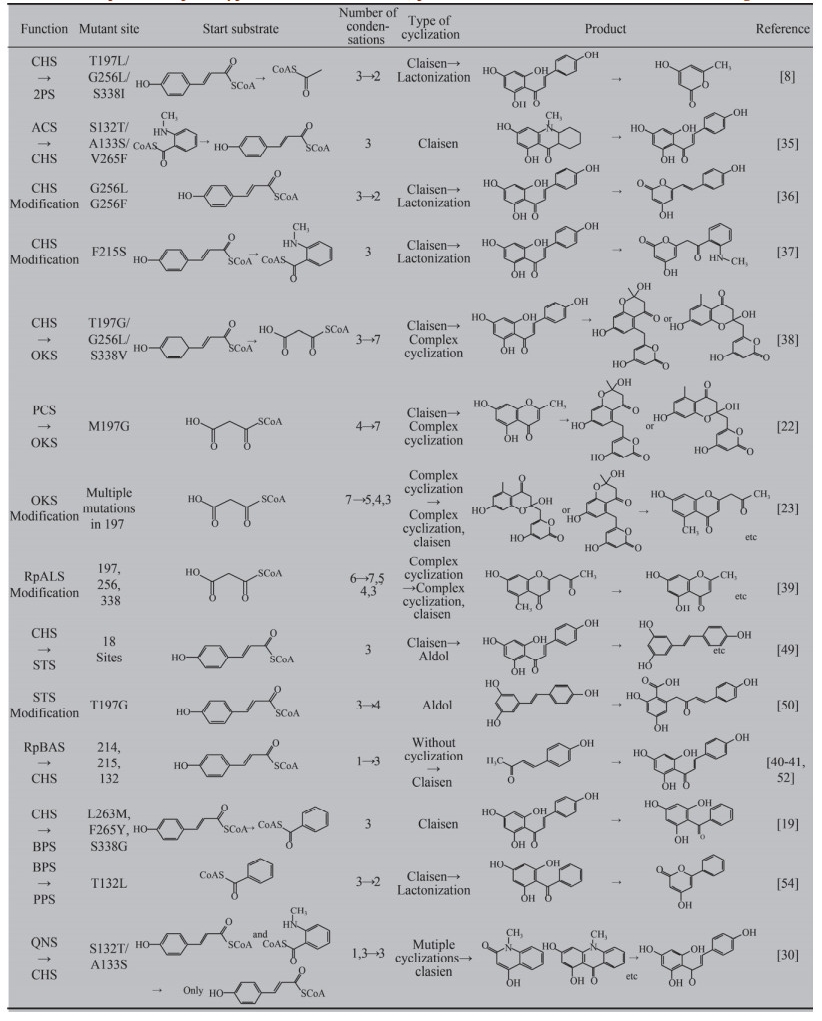 |
表选项
3.2 吖啶酮合酶吖啶酮合酶ACS催化起始底物N-甲基邻氨基苯甲酰-CoA和3分子丙二酰CoA缩合形成吖啶酮。根据芸香Ruta graveolens的2个ACS (ACS1、ACS2)与MsCHS序列比对和同源建模分析结果,Luka?in等推测芸香ACS2的Ser132、Ala133和Val265三个氨基酸残基(MsCHS中分别为Thr132、Ser133和Phe265,以MsCHS氨基酸序列编号)可能对其底物选择特异性具有重要作用。随后芸香ACS2定点突变结果表明,V265F突变体的活性只有野生型的75%,而且突变体明显地优先选择4-香豆酰CoA为其底物,而ACS2 (S132T/A133S/V265F)三突变体在功能上已完全转变为CHS (仍具有较弱的ACS活性) (表 1)。该研究结果也表明,ACS是由CHS通过少数几个氨基酸的替换进化而来的[35]。
3.3 查尔酮合酶Gly256位于CHS催化腔的表面,与延伸的聚酮链直接接触,因此该位点被认为是研究催化腔体积与Ⅲ型PKSs聚酮链长度相关性的理想靶点。Jez等对MsCHS2的Gly256进行了一系列定点突变(G256A、G256V、G256L、G256F),其结果表明,当以4-香豆酰CoA为底物时,G256A和G256V突变体中四酮内酯(Tetraketide lactone)的合成量较野生型明显增加;G256L和G256F突变体中催化腔的体积进一步受到限制,此时苯乙烯基吡喃酮产物BNY (bis-Noryangonin)的合成量明显增加(表 1)。进一步的X-衍射晶体结构分析表明,第256位氨基酸侧链的大小既影响聚合反应的次数,也影响聚酮中间体的构象。据此他们认为在Ⅲ型PKS家族中,第256位氨基酸的特异性进化可能是在不破坏酶的催化活力的前提下快速实现产物多样性的一个重要因素[36]。
2002年,Jez等推测MsCHS活性位点入口处的2个被称为“Gatekeepers”的苯丙氨酸残基(Phe215与Phe265)很可能与酶对起始底物特异性选择有关。他们对这2个位点进行了一系列的定点突变,随后体外添加不同性质和大小的CoA硫酯作为测试底物,在检测了突变蛋白对不同底物的利用情况后发现,F215S突变蛋白能利用一种野生型CHS蛋白原本并不能接受的N-甲基邻氨基苯甲酰-CoA (N-methylanthraniloyl-CoA)作为其底物,催化产生一种新的生物碱类化合物(N-methylanthraniloyltriacetic acid lactone) (表 1)[37]。这一研究结果表明,单一位点的氨基酸残基突变后可以改变酶对底物的选择性,这也是一个通过体外改造植物Ⅲ型PKS使之合成新的次生代谢物的成功实例。
2006年,Abe实验室根据黄芩Scutellaria baicalensis SbCHS与木立芦荟OKS活性位点(197、256、338)氨基酸序列的差异,对SbCHS的Thr197、Gly256、Ser338进行了一系列的单突变、双突变和三突变研究,发现在以丙二酰CoA为底物时(缺乏4-香豆酰CoA),SbCHS及其突变体均能催化丙二酰CoA脱羧缩合,但聚酮链延伸在三酮阶段终止,形成三乙酸内酯(Triacetic acid lactone,TAL)为主产物,这与MsCHS的T197L、G256L、S338I单突变体形成TAL的报道一致[8, 38]。另外,通过对SbCHS S338V酶促反应产物的仔细检测发现,该突变体除了形成TAL外,还产生微量的八酮产物SEK4和SEK4b,而且在SbCHS (T197G/G256L/S338V)突变体(该突变体在功能上类似OKS)中形成SEK4/SEK4b的活性明显增加(表 1)。作者推测SbCHS S338V催化的机制是由于S338V突变为聚酮中间体延伸至埋藏于野生型CHS中的1个额外的口袋提供了空间引导,进而导致形成较长的聚酮链[38]。该研究表明,植物中普遍存在的CHS可以通过对其单一活性位点的修饰(S338V)而使其产生八酮化合物SEK4/SEK4b,该位点的重要性也被其他植物Ⅲ型PKSs的定点突变研究所证实。如掌叶大黄ALS T338S突变体丧失了ALS活性[39],而该植物的BAS S338V突变体的BAS活性增加了2倍[40-41]。
2007年,Wanibuchi等从苔藓植物蛇足石杉Huperzia serrata中鉴定了一个多功能的Ⅲ型聚酮合酶HsPKS1,在体外酶促反应中,该酶可以催化形成多种芳香类四酮产物,包括柚皮素查尔酮、二苯甲酮、间苯三酚和吖啶酮。有趣的是,HsPKS1还可以利用一些CHS不能利用的较大的起始底物,如4-甲氧基肉桂酰-CoA (4-methoxycinnamoyl-CoA)和N-甲基邻氨基苯甲酰-CoA,与3分子丙二酰CoA缩合形成4-methoxy-2’, 4’, 6’-trihydroxychalcone和1, 3-dihydroxy-N-methylacridone,表明该酶具有1个较大的底物结合腔[42]。进一步的定点突变研究表明,HsPKS1 S348G突变(对应MsCHS Ser338)使突变体中活性腔的体积扩展,不仅使聚酮链延伸的长度增加,而且环化机制也发生了改变,从而可以催化起始底物2-氨甲酰基苯甲酰CoA (2-Carbamoylbenzoyl-CoA)与3分子丙二酰CoA缩合形成一种新颖的聚酮-生物碱类骨架Dibenzoazepine,表明通过对Ⅲ型PKSs的定向改造,可以合成自然界不存在的具有活性的复杂化合物[43]。
近年来,本实验室从药用植物虎杖中克隆到多个植物Ⅲ型PKS基因,包括PcPKS1[44]、PcPKS2[34]、PcCHS、PcSTS[45]和PcCHS1[46]等。其中PcCHS1是1个典型的查尔酮合酶,在pH 7?8时能高效地催化合成查尔酮作为单一产物,在pH 9时,除合成查尔酮之外,还能合成一定量的苯亚甲基丙酮[46]。对PcCHS1进行了一系列定点突变研究后发现,一些位于非活性位点甚至处在酶蛋白表面的氨基酸残基不仅具有维持蛋白结构的作用,还直接参与对酶活性的调控。例如,当PcCHS1第82位的Gln突变为Pro后,Q82P突变蛋白的查尔酮合成活性较野生型降低,且在pH 9时不能合成BA;当第198位的Cys突变为Phe后,C198F突变蛋白几乎完全失去活性;但是,Q82P/C198F双突变酶蛋白却能在pH 8–9时恢复较高的BA合成活性,而且在pH 9时,该双突变蛋白还能较大量地催化合成许多CHS常见的2种脱轨产物CTAL (4-Coumaroyltriacetic acid lactone)和BNY;此外,当PcCHS1第105位的Arg突变为Gln后,R105Q突变蛋白改变了野生型蛋白BA合成活性对pH值的依赖性。这些结果表明,远离活性中心甚至是处在酶蛋白表面的氨基酸残基也可能对Ⅲ型PKS的结构与催化活性产生直接的影响[47]。
Fukuma等通过对葛根Pueraria lobata CHS位于酶蛋白表面保守的精氨酸残基(Arg68、Arg172、Arg199、Arg328和Arg350)的系列定点突变研究表明,Arg199和Arg350对维持酶蛋白结构的完整性和酶蛋白的可折叠性具有重要作用,而Arg68、Arg172和Arg326则通过与其他保守氨基酸残基的互作协助其催化腔的精确定位及维持活性位点的正确拓扑结构,该研究也表明远离活性中心的一些氨基酸残基对酶蛋白的催化活性具有重要影响[48]。
3.4 聚五酮色酮合酶和聚八酮合酶木立芦荟的聚五酮色酮合酶PCS催化5分子丙二酰CoA通过分子间连续4步缩合反应形成五酮产物5, 7-Dihydroxy-2-methylchromone[22],而来自同一植物的聚八酮合酶OKS则催化8分子丙二酰CoA通过分子间连续7步缩合反应形成八酮产物SEK4和SEK4b[23]。序列比对结果显示,二者的氨基酸序列具有91%的一致性,MsCHS活性腔保守的3个氨基酸Thr197、Gly256和Ser338在木立芦荟PCS中分别被Met、Leu、Val替换,而在OKS则分别被Gly、Leu、Thr替换[22-23]。定点突变研究显示,将木立芦荟PCS中207位的Met突变为OKS中对应的Gly后(对应MsCHS Thr197),PCS M207G突变体在功能上转变为OKS,即催化8分子丙二酰CoA缩合形成八酮产物SEK4和SEK4b (表 1)[22]。而OKS中G207由小到大的系列突变(G207A、G207T、G207M、G207L、G207F和G207W)结果显示,虽然所有突变体均丧失了OKS活性,但突变体获得了形成较短的聚酮链产物的活性(根据侧链大小不同,可以形成从三酮到七酮大小不同的聚酮化合物),其中OKS G207M突变体(将OKS中207位的Gly突变为PCS中对应的Met)在功能上转变PCS (表 1)[23]。上述结果也被RpALS A197T和A197G突变体分别催化形成五酮产物和八酮产物的报道所证实[39]。这些研究结果清楚地表明,207位氨基酸残基(对应MsCHS Thr197)控制着聚酮链延伸的长度及产物的特异性,暗示Ⅲ型PKSs的功能多样性可以通过单一位点氨基酸残基的替换进化而来。
3.5 芦荟松合酶掌叶大黄的芦荟松合酶RpALS催化乙酰CoA为起始底物与6分子丙二酰CoA脱羧缩合形成七酮化合物芦荟松(Aloesone)。序列比对显示,MsCHS活性腔中保守的3个氨基酸残基Thr197、Gly256、Ser338在RpALS中分别被Ala197、Leu256、Thr338替换[21]。同源建模预测,RpALS中这3个氨基酸残基的替换使得其活性腔的结构更适合利用较4-香豆酰CoA更小的乙酰CoA作为起始底物,从而为合成较CHS更长的聚酮链提供了足够的空间,使得最终形成七酮产物芦荟松[39]。定点突变研究表明,将RpALS上述3个位点的氨基酸残基分别突变为MsCHS对应的氨基酸后,3个突变体(A197T、L256G、T338S)均完全丧失了原来的七酮合成活性。其中,A197T突变体形成五酮产物2, 7-dihydroxy-5-methylchromone,而L256G和T338S仅能形成三酮产物TAL (表 1)。进一步定点突变的结果表明,L256G突变体能够接受原来野生型RpALS不能接受的4-香豆酰CoA为起始底物,表明ALS中第256位氨基酸残基控制着酶对底物的选择性。A197T突变体能够高效催化乙酰CoA与4分子丙二酰CoA缩合形成五酮产物2, 7-dihydroxy-5-methylchromone,而A197G突变体则形成八酮产物SEK4和SEK4b,表明ALS中第197位氨基酸残基控制着聚酮链延伸的长度,与木立芦荟PCS和OKS中197位氨基酸残基控制聚酮链长度的结果相吻合[22-23]。RpALS中338位Thr靠近催化位点Cys164,对聚酮中间体进入活性腔提供了空间引导,进而导致七酮的形成。另外,RpALS (A197T/L256G/T338S)三突变体在功能上已转变为1个四酮合酶,该酶利用4-香豆酰CoA为起始底物,形成四酮产物CTAL[39]。
3.6 芪合酶芪合酶STS利用与CHS相同的底物(4-香豆酰CoA和丙二酰CoA)形成相同的四酮中间体。随后,二者通过不同的机制进行环化,STS催化四酮中间体通过分子内C2→C7 Aldol型缩合,同时以CO2形式脱去C1形成白藜芦醇;而CHS则催化四酮中间体通过分子内C6→C1 Claisen型缩合形成柚皮素查尔酮。
2004年,Austin等报道了欧洲赤松Pinus sylvestris STS 2.1 ?的晶体结构。结构解析显示,欧洲赤松STS与MsCHS活性腔的拓扑结构仅存在细微的差异,但这些细微的差异不足以导致这两种酶催化相同的四酮中间体进行不同的环化反应。通过对二者晶体结构的仔细比对发现,STS活性位点132–136残基之间一个环的构象明显改变,使得该处的Thr132残基发生了细微的位移,从而使其侧链羟基能够与紧邻催化活性腔Cys的Ser338-stabilized水分子形成氢键。由于Thr132在Ⅲ型PKSs中高度保守,STS中其位置的移动明显地影响催化活性腔Cys164周围的氢键网络,形成了所谓的“Aldol-switch”硫酯酶样(Thioesterase-like)氢键网络,从而使STS的环化机制与CHS不同,所以Thr132残基在STS和CHS不同的环化机制中起着至关重要的作用[49]。
随后Austin等将MsCHS中与欧洲赤松STS构象不同的4个区域内共18个位点突变为STS中对应的氨基酸而构建18×CHS突变体,结果发现该突变体能够合成白藜芦醇,在功能上与STS类似(表 1)。对18×CHS突变体Apo和白藜芦醇复合物晶体结构的解析发现,突变区域显示STS-like的构象变化,证实了硫酯酶样构象变化是导致STS Aldol型环化反应的原因。进一步在18×CHS突变体的基础上(保留18×CHS中已经引入的STS-like构象变化的突变),再引入扰乱其硫酯酶样氢键网络的点突变而构建多个18×(+1) CHS突变体(T132A、S131A、E192Q),结果发现这些突变体中白藜芦醇的合成量减少而查尔酮的合成量增加,再次确认了STS活性腔Cys164附近由Ser338-H2O-Thr132-Glu192形成的氢键网络对其特异的Aldol型环化反应具有至关重要的作用[49]。
基于对葡萄Vitis vinifera STS同源建模的结果,Bhan等预测,葡萄STS T197G突变体中底物结合口袋及环化口袋的体积将增大。体外酶促反应结果显示,当以4-香豆酰CoA和丙二酰CoA为底物时,STS T197G突变体产生了一种新的由香豆酰衍生的五酮产物Resorcylic acid (表 1)。进一步研究表明,T197G替代使得突变体中底物结合及环化口袋的整体体积由1 029.2 ?3增大至1 134.3 ?3,从而使突变体较野生型能够多接受1个乙酰基而形成五酮中间体,后者再经过C2→C7 Aldol型缩合形成五酮产物Resorcylic acid[50]。该研究表明,通过对STS-like酶的理性改造,可以通过C2→C7 Aldol型缩合而产生自然界不存在的新化合物。
3.7 苯亚甲基丙酮合酶2001年,Abe等在掌叶大黄R. palmatum中首次克隆到苯亚甲基丙酮合酶(Bezalacetone synthase, BAS)基因[18]。RpBAS与CHS的不同之处在于它仅催化起始底物4-香豆酰CoA与1分子丙二酰CoA通过一步脱羧缩合形成二酮产物—苯亚甲基丙酮。序列比对结果表明,在其他植物和细菌的Ⅲ型PKSs家族成员中均高度保守的Phe215残基在RpBAS中被Leu替代。CHS中疏水性的Phe215位于活性腔的入口处,推测该位点控制着聚酮链延伸反应的次数[37, 51]。
2003年,Abe等将RpBAS的214IL突变为MsCHS对应的氨基酸残基,结果发现突变后的RpBAS I214L/L215F在pH 6.5时表现出CHS酶活性,该结果证实了214IL残基确实与RpBAS二酮产物苯亚甲基丙酮的形成有关,RpBAS中活性位点Phe215的缺失导致聚酮链的延伸在二酮中间体阶段即被打断(表 1)[52]。随后,他们又将黄芩Scutellaria baicalensis CHS的214LF突变为IL,结果黄芩CHS L214I/F215L突变体的CHS活性明显下降,三酮化合物BNY成为主要产物,但无论是野生型还是突变体中均未检测到二酮化合物苯亚甲基丙酮。进一步对黄芩CHS F215残基进行的一系列突变(F215W、F215Y、F215S、F215A、F215H和F215C)研究结果显示,所有突变后的酶蛋白体外酶促反应产物中均未检测到BA。该结果暗示,RpBAS可能在进化上晚于CHS,前者由后者进化而来。RpBAS蛋白215位置上的Leu对于该酶的BAS活性是必需的,对应于CHS同一位置上的Phe的缺失导致RpBAS中聚酮链的延伸在二酮中间体阶段即被打断[52]。该研究也是通过修饰活性位点周围的单个氨基酸残基而改变Ⅲ型PKSs功能多样性的有趣实例。
2007年,Abe等对RpBAS C197T、C197G、G256L、S338V定点突变的研究结果表明,这4个突变体的产物模式均未改变,只是S338V突变导致酶活性较野生型增加2倍,而G256L突变则使酶活性下降2倍,但C197T和C197G这2个突变体的功能几乎与野生型相同,该结果排除了Cys197可能作为RpBAS第2个活性位点的推测[40]。
2010年,Morita等报道了野生型RpBAS和合成查尔酮的RpBAS I207L/L208F (RpBAS氨基酸编号)突变体1.8 ?的晶体结构[41]。RpBAS与MsCHS活性位点的结构对比显示,RpBAS中Leu208残基的主链构象与MsCHS相似,但其侧链伸入RpBAS活性腔内,所以导致其活性腔体积缩小。另外,RpBAS中紧邻活性位点Cys的Ser331 (对应MsCHS的Ser338)对于催化活性的调控至关重要,如前文所述,RpBAS S338V突变使其苯亚甲基丙酮合成活性增加2倍,也佐证了该位点对于二酮化合物的形成具有重要作用[40]。晶体结构解析显示,RpBAS中Ser331的羟基旋转了近120°,堵塞了MsCHS中香豆酰结合口袋的入口[7]。上述RpBAS晶体结构中Leu208和Ser331构象改变的结果导致MsCHS中的香豆酰结合口袋在RpBAS活性腔中消失,使其活性腔体积(350 ?3)缩小到仅为MsCHS (750 ?3)的一半,活性腔体积的缩小导致RpBAS中聚酮链的延伸在二酮中间体阶段即被打断,从而合成二酮化合物。RpBAS I207L/L208F突变体晶体结构的解析显示,突变体中第208位氨基酸残基的替换为野生型中原本埋藏的香豆酰结合口袋打开了一个入口,使得聚酮链延伸反应次数增加到3次,因而突变体恢复了查尔酮形成活性[41]。
RpBAS晶体结构解析还显示,MsCHS中保守的Thr132 (对应MsCHS氨基酸编号)在RpBAS中被Leu所替换,从而导致RpBAS活性腔体积缩小,使RpBAS只能形成二酮产物而不能形成四酮产物[41]。2010年,Shimokawa等对Thr132进行了一系列的定点突变研究(L132G,L132A,L132S,L132C,L132T,L132F,L132Y,L132W,L132P),结果显示,L132T突变恢复了RpBAS的查尔酮合酶活性,而L132A、L132S、L132C突变则增加产物的链长而形成CTAL (表 1),同源建模揭示可能是由于这些突变恢复了RpBAS活性腔的香豆酰结合口袋[53]。
3.8 二苯甲酮合酶金丝桃Hypericum androsaemum的二苯甲酮合酶BPS催化起始底物苯甲酰CoA与3分子丙二酰CoA通过脱羧缩合形成2, 4, 6-三羟基二苯甲酮(2, 4, 6-trihydroxybenzophenone),该酶还可以利用3-羟基苯甲酰-CoA (3-hydroxybenzoyl-CoA)和N-甲基邻氨基苯甲酰-CoA为起始底物,活性分别是苯甲酰CoA为起始底物时的18.8%和10.9%,但不能利用肉桂酰CoA为起始底物。但该植物的CHS则可以利用肉桂酰CoA为起始底物(活性为香豆酰CoA为起始底物时的87.5%),同时还可以利用苯甲酰CoA和3-羟基苯甲酰-CoA为起始底物。氨基酸序列比对显示,位于CHS活性腔的Gly256、Leu263、Phe265和Ser338在BPS中分别被Ala260、Met267、Tyr269和Gly342所替代(BPS氨基酸编号)。定点突变结果表明,BPS突变体或者与野生型功能类似(如G342S,A260G/ M267L/Y269F),或者丧失二苯甲酮和查尔酮形成活性(如A260G,A260G/G342S,M267L/Y269F/ G342S,A260G/M267L/Y269L/G342S);而CHS L263M/F265Y/S338G突变体却转变为1个对苯甲酰CoA较香豆酰CoA具有更高活性的酶,即突变后的CHS在功能上已转变为1个BPS (表 1)[19]。该研究结果也表明,BPS可以由CHS通过少数几个氨基酸的替换进化而来,而反过来即由BPS转变成CHS则很难,这与芸香ACS可以通过3个氨基酸的突变(S132T/A133S/V265F)转变成CHS而CHS则很难转变为ACS的报道相一致[35]。
2009年,Klundt等对金丝桃BPS活性口袋内的T135 (对应MsCHS中T132)突变为Leu后发现,原本催化苯甲酰CoA与3分子丙二酰CoA聚合形成二苯甲酮的BPS在功能上转变为1个苯基吡喃酮合酶(Phenylpyrone synthase, PPS)。该位点的突变改变了野生型BPS的底物与产物特异性,突变后的BPS催化苯甲酰CoA与2分子丙二酰CoA合成6-苯基-4-羟基-吡喃酮(6-phenyl-4-hydroxy-pyrone),而且与野生型不同的是,突变后的BPS不再接受3-羟基苯甲酰-CoA为其底物(表 1)。同源建模后发现,金丝桃BPS T135L突变体中Leu的侧链可能伸进延伸口袋内阻断了聚酮链的延伸路径;同时,T135L替换可能在突变体中打开了一个新的口袋(该口袋入口在野生型中被堵塞),通过Leu侧链与生长中的聚酮链苯基之间的互作,活性口袋中的三酮中间体被重新定向进入突变体中新形成的口袋中。同源建模显示新口袋的体积为168 ?3,足以容纳1个苯基(144 ?3),但是却不能容纳生长中的聚酮链再添加第3个乙酰基,最终三酮中间体经过杂环内酯化形成苯基吡喃酮(Phenylpyrone)[54]。同源建模的结果合理解释了T135L突变体底物与产物特异性显著改变的机理,该研究也充分证明,单一活性位点氨基酸残基的改变完全可以彻底改变一种Ⅲ型PKS原有的活性而产生另一种全新的酶。
3.9 喹诺酮合酶木橘Aegle marmelos的喹诺酮合酶QNS催化喹诺酮类生物碱的合成。2013年,Resmi等鉴定了木橘的QNS并进行了定点突变研究。木橘QNS具有广泛的底物特异性,可以接受多种芳香类及脂肪类CoA作为起始底物,在以N-methylanthraniloyl-CoA为主要底物时,QNS在体外酶促反应中可以形成喹诺酮和吖啶酮,可能是1个双功能酶。基于同源性的结构建模发现,Ser132和Ala133是位于QNS活性口袋的2个关键氨基酸残基(MsCHS中对应Thr132和Ser133),为了研究其功能,通过定点突变构建了QNS S132T/A133S双突变体和QNS S132T/A133S/V265F三突变体。同源建模显示,这2个突变体的活性腔体积较野生型明显缩小,丧失了结合较大起始底物的机会。随后的体外酶促反应证实QNS S132T/A133S双突变体能够以香豆酰CoA为起始底物催化3分子乙酰CoA缩合形成柚皮素查尔酮,即QNS在功能上已完全转变为1个CHS,而QNS S132T/A133S/V265F三突变体以香豆酰CoA为起始底物时未能检测到产物(表 1)。同时,这2个突变体以N-methylanthraniloyl-CoA为底物时未形成产物,也进一步验证了同源建模的结果[30]。该研究也与芸香ACS可以通过3个氨基酸的突变(S132T/A133S/V265F)转变成CHS的结果一致[35]。该研究也表明,木橘QNS蛋白可能是由CHS超家族中一个进化上相关的成员仅仅通过2个氨基酸残基的替换进化而来,同时该研究也是通过基因操作产生新型生物碱骨架的成功范例。
3.10 烷基乙酮CoA合酶和烷基喹诺酮合酶最近,Matsui等从药用植物吴茱萸Evodia rutaecarpa中鉴定了2个新的植物Ⅲ型聚酮合酶ADS和AQS,并对其功能进行了研究[32]。体外功能分析表明,ADS和AQS能够协同催化N-methylanthraniloyl-CoA和丙二酰CoA缩合形成生物碱类物质2-烷基喹诺酮(2-alkylquinolone,2AQ)。其中ADS能够高效催化癸酰基CoA (Decanoyl-CoA)与1分子丙二酰CoA脱羧缩合形成癸酰基乙酮CoA (Decanoyldiketide-CoA),而AQS则特异性催化ADS的羟化产物Decanoyldiketide acid与N-methylanthraniloyl-CoA脱羧缩合形成2AQ骨架。随后作者分别解析了ADS与CoA-SH复合物1.80 ?和AQS 2.20 ?的晶体结构,并构建了一系列的ADS和AQS Y215V突变体。ADS系列突变体(W332Q、W332I、W332V、W332A、W332G以及C191G、C191A)的体外功能检测并结合晶体结构解析结果表明,Trp332和Cys191对于ADS产物的形成具有至关重要的作用,这两个位点决定了该酶蛋白活性腔的结构,从而调控其底物和产物的特异性。而对AQS Y215V突变体的体外功能检测并结合晶体结构解析结果显示,AQS的Tyr215残基主要通过控制该酶蛋白的CoA结合通道的构象而调控其底物和产物的特异性[32]。
4 总结与展望不同类型的Ⅲ型PKSs晶体结构解析以及基于定点突变的结构与功能分析表明,活性位点周围的单个或是少数氨基酸残基的性质可以决定活性口袋空间的大小和催化反应的特异性。同时,不同种类的植物Ⅲ型PKSs催化口袋的体积和活性位点氨基酸残基的性质可以直接影响PKSs底物的特异性和产物类型,从而产生了不同的催化反应和结构迥异的聚酮化合物分子。
近年来,越来越多的具有不同功能的植物Ⅲ型PKSs基因被克隆和鉴定功能,极大地拓宽了人们对Ⅲ型PKSs催化反应类型的认识。当不同的Ⅲ型PKSs基因被克隆以后,对各种可能影响所催化的反应(如控制聚酮链长、成环机制和催化口袋大小)的氨基酸残基进行定点突变就成为鉴定这类酶的活性残基的首选方法。通常,突变某个Ⅲ型PKS的少数甚至是单个处于活性位点或其附近的氨基酸残基,有可能会使酶蛋白获得新的活性,从而催化产生新的聚酮化合物。通过定点突变对植物Ⅲ型PKSs进行定向改造,不仅可以研究酶蛋白的结构与功能的关系,而且可以丰富聚酮化合物的种类,为一些复杂的、具有重要药用价值的天然或非天然聚酮化合物组合生物合成提供便利。同时,突变以后的Ⅲ型PKSs很可能可以获得新的催化活性,这也可以为研究和阐明植物Ⅲ型PKSs超家族酶的催化机制与进化关系提供新的视野和证据。
参考文献
| [1] | Shen B. Polyketide biosynthesis beyond the type Ⅰ, Ⅱ and Ⅲ polyketide synthase paradigms.Curr Opin Chem Biol, 2003, 7(2): 285–295.DOI: 10.1016/S1367-5931(03)00020-6 |
| [2] | Lim YP, Go MK, Yew WS. Exploiting the biosynthetic potential of type Ⅲ polyketide synthases.Molecules, 2016, 21(6): 806.DOI: 10.3390/molecules21060806 |
| [3] | Abe I, Morita H. Structure and function of the chalcone synthase superfamily of plant type Ⅲ polyketide synthases.Nat Prod Rep, 2010, 27(6): 809–838.DOI: 10.1039/b909988n |
| [4] | Ma LQ, Shi GL, Ye HC, et al. Plant-specific type Ⅲ polyketide synthase superfamily: gene structure, function and metabolistes.Chin J Biotech, 2010, 26(11): 1482–1492.(in Chinese). 马兰青, 师光禄, 叶和春, 等. 植物类型Ⅲ聚酮合酶超家族基因结构、功能及代谢产物.生物工程学报, 2010, 26(11): 1482-1492. |
| [5] | Austin MB, Noel JP. The chalcone synthase superfamily of type Ⅲ polyketide synthases.Nat Prod Rep, 2003, 20(1): 79–110.DOI: 10.1039/b100917f |
| [6] | Lü HS, Liu CM, Lu P, et al. Plant-specific type Ⅲ polyketide synthases superfamily: crystal structures and functions.Chin J Biotech, 2011, 28(1): 1–14.(in Chinese). 吕鹤书, 柳春梅, 路平, 等. 植物类型Ⅲ聚酮合酶超家族晶体结构与功能.生物工程学报, 2011, 28(1): 1-14. |
| [7] | Ferrer JL, Jez JM, Bowman ME, et al. Structure of chalcone synthase and the molecular basis of plant polyketide biosynthesis.Nat Struct Biol, 1999, 6(8): 775–784.DOI: 10.1038/11553 |
| [8] | Jez JM, Austin MB, Ferrer JL, et al. Structural control of polyketide formation in plant-specific polyketide synthases.Chem Biol, 2000, 7(12): 919–930.DOI: 10.1016/S1074-5521(00)00041-7 |
| [9] | Reimold U, Kr?ger M, Kreuzaler F, et al. Coding and 3′ non-coding nucleotide sequence of chalcone synthase mRNA and assignment of amino acid sequence of the enzyme.EMBO J, 1983, 2(10): 1801–1805. |
| [10] | Schr?der G, Brown JWS, Schr?der J. Molecular analysis of resveratrol synthase: cDNA, genomic clones and relationship with chalcone synthase.Eur J Biochem, 1988, 172(1): 161–169.DOI: 10.1111/ejb.1988.172.issue-1 |
| [11] | Chong JL, Poutaraud A, Hugueney P. Metabolism and roles of stilbenes in plants.Plant Sci, 2009, 177(3): 143–155.DOI: 10.1016/j.plantsci.2009.05.012 |
| [12] | Preisiq-Müller R, Gnau P, Kindl H. The inducible 9, 10-dihydrophenanthrene pathway: characterization and expression of bibenzyl synthase and S-adenosylhomocysteine hydrolase.Arch Biochem Biophys, 1995, 317(1): 201–207.DOI: 10.1006/abbi.1995.1154 |
| [13] | Eckermann S, Schr?der G, Schmidt J, et al. New pathway to polyketides in plants.Nature, 1998, 396(6709): 387–390.DOI: 10.1038/24652 |
| [14] | Schr?der J, Raiber S, Berger T, et al. Plant polyketide synthases: a chalcone synthase-type enzyme which performs a condensation reaction with methylmalonyl-CoA in the biosynthesis of C-methylated chalcones.Biochemistry, 1998, 37(23): 8417–8425.DOI: 10.1021/bi980204g |
| [15] | Zuurbier KWM, Leser J, Berger T, et al. 4-Hydroxy-2-pyrone formation by chalcone and stilbene synthase with nonphysiological substrates.Phytochemistry, 1998, 49(7): 1945–1951.DOI: 10.1016/S0031-9422(98)00346-X |
| [16] | Akiyama T, Shibuya M, Liu HM, et al. p-Coumaroyltriacetic acid synthase, a new homologue of chalcone synthase, from Hydrangea macrophylla var. thunbergii.Eur J Biochem, 1999, 263(3): 834–839.DOI: 10.1046/j.1432-1327.1999.00562.x |
| [17] | Springob K, Luka?in R, Ernwein C, et al. Specificities of functionally expressed chalcone and acridone synthases from Ruta graveolens.Eur J Biochem, 2000, 267(22): 6552–6559.DOI: 10.1046/j.1432-1327.2000.01746.x |
| [18] | Abe I, Takahashi Y, Morita H, et al. Benzalacetone synthase: a novel polyketide synthase that plays a crucial role in the biosynthesis of phenylbutanones in Rheum palmatum.Eur J Biochem, 2001, 268(11): 3354–3359.DOI: 10.1046/j.1432-1327.2001.02255.x |
| [19] | Liu BY, Falkenstein-Paul H, Schmidt W, et al. Benzophenone synthase and chalcone synthase from Hypericum androsaemum cell cultures: cDNA cloning, functional expression, and site-directed mutagenesis of two polyketide synthases.Plant J, 2003, 34(6): 847–855.DOI: 10.1046/j.1365-313X.2003.01771.x |
| [20] | Eckermann C, Schr?der G, Eckermann S, et al. Stilbenecarboxylate biosynthesis: a new function in the family of chalcone synthase-related proteins.Phytochemistry, 2003, 62(3): 271–286.DOI: 10.1016/S0031-9422(02)00554-X |
| [21] | Abe I, Utsumi Y, Oguro S, et al. The first plant type Ⅲ polyketide synthase that catalyzes formation of aromatic heptaketide.FEBS Lett, 2004, 562(1-3): 171–176.DOI: 10.1016/S0014-5793(04)00230-3 |
| [22] | Abe I, Utsumi Y, Oguro S, et al. A plant Type Ⅲ polyketide synthase that produces pentaketide chromone.J Am Chem Soc, 2005, 127(5): 1362–1363.DOI: 10.1021/ja0431206 |
| [23] | Abe I, Oguro S, Utsumi Y, et al. Engineered biosynthesis of plant polyketides: chain length control in an octaketide-producing plant type Ⅲ polyketide synthase.J Am Chem Soc, 2005, 127(36): 12709–12716.DOI: 10.1021/ja053945v |
| [24] | Liu B, Raeth T, Beuerle T, et al. Biphenyl synthase, a novel type Ⅲ polyketide synthase.Planta, 2007, 225(6): 1495–1503.DOI: 10.1007/s00425-006-0435-5 |
| [25] | Katsuyama Y, Matsuzawa M, Funa N, et al. In vitro synthesis of curcuminoids by type Ⅲ polyketide synthase from Oryza sativa.J Biol Chem, 2007, 282(52): 37702–37709.DOI: 10.1074/jbc.M707569200 |
| [26] | Springob K, Samappito S, Jindaprasert A, et al. A polyketide synthase of Plumbago indica that catalyzes the formation of hexaketide pyrones.FEBS J, 2007, 274(2): 406–417.DOI: 10.1111/j.1742-4658.2006.05588.x |
| [27] | Jindaprasert A, Springob K, Schmidt J, et al. Pyrone polyketides synthesized by a type Ⅲ polyketide synthase from Drosophyllum lusitanicum.Phytochemistry, 2008, 69(18): 3043–3053.DOI: 10.1016/j.phytochem.2008.03.013 |
| [28] | Klingauf P, Beuerle T, Mellenthin A, et al. Biosynthesis of the hyperforin skeleton in Hypericum calycinum cell cultures.Phytochemistry, 2005, 66(2): 139–145.DOI: 10.1016/j.phytochem.2004.11.003 |
| [29] | Taura F, Tanaka S, Taguchi C, et al. Characterization of olivetol synthase, a polyketide synthase putatively involved in cannabinoid biosynthetic pathway.FEBS Lett, 2009, 583(12): 2061–2066.DOI: 10.1016/j.febslet.2009.05.024 |
| [30] | Resmi MS, Verma P, Gokhale RS, et al. Identification and characterization of a type Ⅲ polyketide synthase involved in quinolone alkaloid biosynthesis from Aegle marmelos Correa.J Biol Chem, 2013, 288(10): 7271–7281.DOI: 10.1074/jbc.M112.429886 |
| [31] | Taura F, Iijima M, Yamanaka E, et al. A novel class of plant type Ⅲ polyketide synthase involved in orsellinic acid biosynthesis from Rhododendron dauricum.Front Plant Sci, 2016, 7: 1452. |
| [32] | Matsui T, Kodama T, Mori T, et al. 2-Alkylquinolone alkaloid biosynthesis in the medicinal plant Evodia rutaecarpa involves collaboration of two novel type Ⅲ polyketide synthases.J Biol Chem, 2017, 292(22): 9117–9135.DOI: 10.1074/jbc.M117.778977 |
| [33] | Yang J, Gu HY. Duplication and divergent evolution of the CHS and CHS-like genes in the chalcone synthase (CHS) superfamily.Chin Sci Bull, 2006, 51(5): 505–509.(in Chinese). 杨继, 顾红雅. 查尔酮合酶超家族(chalcone synthase superfamily)基因重复和分化的式样.科学通报, 2006, 51(5): 505-509. |
| [34] | Ma LQ, Guo YW, Gao DY, et al. Identification of a Polygonum cuspidatum three-intron gene encoding a type Ⅲ polyketide synthase producing both naringenin and p-hydroxybenzalacetone.Planta, 2009, 229(5): 1077–1086.DOI: 10.1007/s00425-009-0899-1 |
| [35] | Luka?in R, Schreiner S, Matern U. Transformation of acridone synthase to chalcone synthase.FEBS Lett, 2001, 508(3): 413–417.DOI: 10.1016/S0014-5793(01)03061-7 |
| [36] | Jez JM, Bowman ME, Noel JP. Structure-guided programming of polyketide chain-length determination in chalcone synthase.Biochemistry, 2001, 40(49): 14829–14838.DOI: 10.1021/bi015621z |
| [37] | Jez JM, Bowman ME, Noel JP. Expanding the biosynthetic repertoire of plant type Ⅲ polyketide synthases by altering starter molecule specificity.Proc Natl Acad Sci USA, 2002, 99(8): 5319–5324.DOI: 10.1073/pnas.082590499 |
| [38] | Abe I, Watanabe T, Morita H, et al. Engineered biosynthesis of plant polyketides: manipulation of chalcone synthase.Org Lett, 2006, 8(3): 499–502.DOI: 10.1021/ol052912h |
| [39] | Abe I, Watanabe T, Lou WW, et al. Active site residues governing substrate selectivity and polyketide chain length in aloesone synthase.FEBS J, 2006, 273(1): 208–218.DOI: 10.1111/ejb.2006.273.issue-1 |
| [40] | Abe T, Morita H, Noma H, et al. Structure function analysis of benzalacetone synthase from Rheum palmatum.Bioorg Med Chem Lett, 2007, 17(11): 3161–3166.DOI: 10.1016/j.bmcl.2007.03.029 |
| [41] | Morita H, Shimokawa Y, Tanio M, et al. A structure-based mechanism for benzalacetone synthase from Rheum palmatum.Proc Natl Acad Sci USA, 2010, 107(2): 669–773.DOI: 10.1073/pnas.0909982107 |
| [42] | Wanibuchi K, Zhang P, Abe T, et al. An acridone-producing novel multifunctional type Ⅲ polyketide synthase from Huperzia serrata.FEBS J, 2007, 274(4): 1073–1082.DOI: 10.1111/j.1742-4658.2007.05656.x |
| [43] | Morita H, Yamashita K, Shi SP, et al. Synthesis of unnatural alkaloid scaffolds by exploiting plant polyketide synthase.Proc Natl Acad Sci USA, 2011, 108(33): 13504–13509.DOI: 10.1073/pnas.1107782108 |
| [44] | Ma LQ, Pang XB, Shen HY, et al. A novel type Ⅲ polyketide synthase encoded by a three-intron gene from Polygonum cuspidatum.Planta, 2009, 229(3): 457–469.DOI: 10.1007/s00425-008-0845-7 |
| [45] | Guo YW, Guo HL, Li X, et al. Two type Ⅲ polyketide synthases from Polygonum cuspidatum: gene structure, evolutionary route and metabolites.Plant Biotechnol Rep, 2013, 7(3): 371–381.DOI: 10.1007/s11816-012-0271-y |
| [46] | Li X, Wang H. Cloning and characterization of PcCHS1 from Polygonum cuspidatum.J Graduate Univ Chin Acad Sci, 2013, 30(2): 206–212.(in Chinese). 李星, 王红. 虎杖查尔酮合酶PcCHS1基因的克隆与功能分析.中国科学院研究生院学报, 2013, 30(2): 206-212. |
| [47] | Shen YL, Li X, Chai TY, et al. Outer-sphere residues influence the catalytic activity of a chalcone synthase from Polygonum cuspidatum.FEBS Open Bio, 2016, 6(6): 610–618.DOI: 10.1002/2211-5463.12072 |
| [48] | Fukuma K, Neuls ED, Ryberg JM, et al. Mutational analysis of conserved outer sphere arginine residues of chalcone synthase.J Biochem, 2007, 142(6): 731–739.DOI: 10.1093/jb/mvm188 |
| [49] | Austin MB, Bowman ME, Ferrer JL, et al. An aldol switch discovered in stilbene synthases mediates cyclization specificity of type Ⅲ polyketide synthases.Chem Biol, 2004, 11(9): 1179–1194.DOI: 10.1016/j.chembiol.2004.05.024 |
| [50] | Bhan N, Li LY, Cai C, et al. Enzymatic formation of a resorcylic acid by creating a structure-guided single-point mutation in stilbene synthase.Protein Sci, 2015, 24(2): 167–173.DOI: 10.1002/pro.2600 |
| [51] | Jez JM, Ferrer JL, Bowman ME, et al. Dissection of malonyl-coenzyme a decarboxylation from polyketide formation in the reaction mechanism of a plant polyketide synthase.Biochemistry, 2000, 39(5): 890–902.DOI: 10.1021/bi991489f |
| [52] | Abe I, Sano Y, Takahashi Y, et al. Site-directed mutagenesis of benzalacetone synthase: the role of Phe215 in plant type Ⅲ polyketide synthases.J Biol Chem, 2003, 278(27): 25218–25226.DOI: 10.1074/jbc.M303276200 |
| [53] | Shimokawa Y, Morita H, Abe I. Structure-based engineering of benzalacetone synthase.Bioorg Med Chem Lett, 2010, 20(17): 5099–5103.DOI: 10.1016/j.bmcl.2010.07.022 |
| [54] | Klundt T, Bocola M, Lütge M, et al. A single amino acid substitution converts benzophenone synthase into phenylpyrone synthase.J Biol Chem, 2009, 284(45): 30957–30964.DOI: 10.1074/jbc.M109.038927 |
