
 , 杨瑞馥2
, 杨瑞馥21 河北北方学院 动物科技学院,河北 张家口 075131;
2 军事医学科学院微生物流行病研究所 病原微生物生物安全国家重点实验室,北京 100071
收稿日期:2017-10-08;接收日期:2017-12-26 基金项目:国家重点基础研究发展计划(973计划) (No. 2014CB744405),河北省科技计划项目(No. 16236605D-1(2017))资助
摘要:基因敲除技术是了解基因功能的重要技术手段。以大肠杆菌K-12 MG1655基因组hfq (309 bp)和rne-710 (1056 bp)基因为模型,首先构建Δhfq∷Spe和Δrne-710∷Spe菌株,通过融合PCR方法分别构建缺失hfq (309 bp)和rne-710 (1056 bp)的融合片段并连接至辅助质粒,缺失hfq和rne-710的片段经重组分别替换壮观霉素抗性盒,得到无痕敲除株Δhfq和Δrne-710。双质粒无痕敲除和融合PCR方法相结合为大片段基因缺失开辟了新的途径。
关键词:大肠杆菌 hfq rne-710 双质粒无痕敲除 融合PCR Ⅰ-Sce Ⅰ内切酶
Two-plasmid scarless genetic modification in Escherichia coli hfq and rne-710
Jing Wang1,2, Ruichen Lv2, Yanping Han2

 , Ruifu Yang2
, Ruifu Yang2 1 College of Animal Science and Technology, Hebei North University, Zhangjiakou 075131, Hebei, China;
2 State Key Laboratory of Pathogen and Biosecurity, Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Beijing 100071, China
Received: October 8, 2017; Accepted: December 26, 2017
Supported by: National Basic Research Program of China (973 Program) (No. 2014CB744405), Science and Technology Program of Hebei Province, China (No. 16236605D-1(2017))
Corresponding author:Yanping Han. Tel: +86-10-66948562; E-mail: hypiota@hotmail.com
Abstract: Gene modification is an important technique to understand gene function. We firstly constructed Δhfq::Spe and Δrne-710::Spe mutant strains of Escherichia coli MG1655. The fragment lacking of hfq and rne-710 was ligated to the auxiliary plasmid and separately replace the spectinomycin box by homologous recombinase system to obtain the Δhfq and Δrne-710 mutant strains. The combination of two-plasmid scarless genetic modification and fusion PCR led to a new way for the long DNA fragment gene deletions.
Key words: Escherichia coli hfq rne-710 two-plasmid scarless genetic modification fusion PCR endonuclease Ⅰ-Sce Ⅰ
基因敲除技术是20世纪80年代末发展起来的新型分子生物学技术,常用的Red系统重组方法是通过外源DNA与染色体DNA之间的同源重组[1-4],即用设计的同源片段替代靶基因片段,使机体特定的基因缺失,从而达到基因敲除的目的[5-6],这种方法具有专一性强、染色体DNA与目的片段共同稳定遗传等特点[7-8]。通过抗性筛选完成的基因敲除,基因组留存一定的抗性基因片段,影响了后续研究工作。抗性基因两侧含有2个同向的FRT位点,研究者通常利用FLP/FRT位点特异性重组系统删除抗性基因,该系统的FLP重组酶(Flippase recombination enzyme)可以介导FRT位点(FLP recombination target)之间的基因发生删除重组。即使这样,在重组分子上仍残留2个48 bp的FRT序列,达不到无痕敲除的目的[9]。
目前,无痕敲除技术主要有单质粒敲除法[10]、两步同源重组正负筛选法[11]、Tn5靶向的Cre/loxP切除系统法[12]等,这些方法操作复杂、转化效率低、实验周期长。双质粒无痕敲除法将λ-Red系统与Ⅰ-Sce Ⅰ核酸内切酶相结合[13-14],通过两步Red基因重组即可实现[15],首先进行第一次同源重组,将两端带有Ⅰ-Sce Ⅰ核酸内切酶的抗性基因片段替换目的基因[16],然后在辅助质粒的参与下,通过诱导激活Ⅰ-Sce Ⅰ内切酶,暴露替换基因,具有同源的基因片段发生第二次重组,从而删除抗性基因[17],辅助质粒被Ⅰ-Sce Ⅰ内切酶切成片段而失活,最后经抗性筛选得到重组菌株[18-19]。第一步重组技术非常成熟,已经广泛应用于各领域[20-24],第二步操作简单易行,转化效率高达90%。双质粒无痕敲除法是无痕敲除方法中的最优选择,以其快速的优点将无痕敲除技术推到了更高的层次。
双质粒无痕敲除法多用于基因定点插入、点突变的研究,对于长片段的基因敲除未见报道。本研究采用双质粒无痕敲除法,以大肠杆菌K-12 MG1655基因组hfq (309 bp)和rne-710 (1 056 bp)基因为模型,通过融合PCR方法分别构建缺失hfq (309 bp)和rne-710 (1 056 bp)的融合片段并连接至辅助质粒,通过同源重组完成高效无痕敲除技术,为大片段的无痕敲除技术提供理论依据和方法借鉴。
1 材料与方法1.1 菌株和质粒pREDTKI、pKSI-1、pMDISI三种质粒(图 1)由中国科学院上海生命科学研究院植物生理生态研究所合成生物学重点实验室杨晟研究员赠送,大肠杆菌Escherichia coli K-12 MG1655为军事医学科学院生物工程研究所王恒樑老师赠送。
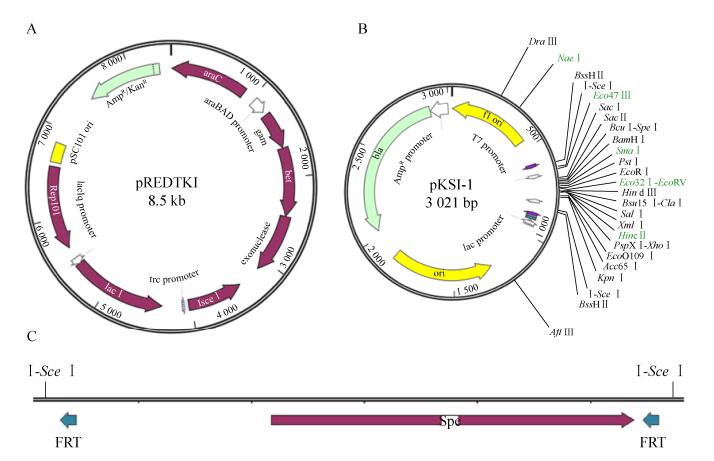 |
| 图 1 质粒图谱[18] (A:pREDTKI质粒具有araB promoter,当加入L-阿拉伯糖时可诱导λ-Red重组功能,pREDTKI质粒同时具有trc promoter,当加入IPTG时可诱导Ⅰ-Sce Ⅰ酶表达;B:pKSI-1载体具有一个多克隆位点,含有两个Ⅰ-Sce Ⅰ识别位点;C:pMDISI质粒具有壮观抗性基因,两侧有FRT和Ⅰ-Sce Ⅰ识别位点) Figure 1 Plasmids profiles[18]. (A) pREDTKI with arabinose-inducible (araB promoter) λ-Red recombinase functions and IPTG-inducible (trc promoter)Ⅰ-Sce Ⅰ expression. (B) pKSI-1 vector with an MCS and two Ⅰ-Sce Ⅰ recognition sites. (C) Part of plasmid pMDISI with the spectinomycin resistance gene flanked by FRT sites and Ⅰ-Sce Ⅰ recognition sites. |
| 图选项 |
1.2 引物设计结合双质粒无痕敲除法(图 2)和融合PCR方法,根据构建无痕敲除基因的原理,设计hfq和rne-710基因扩增引物和鉴定引物(表 1)。
 |
| 图 2 双质粒无痕敲除法原理图[18] Figure 2 Diagram of the two-plasmid scarless genetic modification method[18]. |
| 图选项 |
表 1 本研究中使用的寡核苷酸引物Table 1 Oligonucleotide primers used in this study
| Primer name | Primer sequence (5'–3') |
| mdHfq-F | GCAATTTTTTCAGAATCGAAAGGTTCAAAGTACAAATAAGCATATAAGGAAAAGAGAGACCCTTCCCGGCGATCCTCTGG |
| mdHfq-R | AATAGATCAGCGGGGAACGCAGGATCGCTGGCTCCCCGTGTAAAAAAACAGCCCGAAACCCCCGCATGACGGCAAGTGGACG |
| Hfq-I-F | CGGACTTGCCTTCCATTC |
| Hfq-Spe-I-R | GCAGGATAGGTGAAGTAGG |
| Hfq-Spe-I-F | CTCCAGCCTACACATTACC |
| Hfq-I-R | CGGAAGAGACCAGAGATTC |
| Hfq-F | TTACAAGATCCGTTCCTGAAC |
| Hfq-R | GATGGTAGTTACTGCTGGTAC |
| Hfq-F-1 (Sac Ⅰ) | TCCGAGCTCAGGTAGATCCGGTTGCGGCAGCAAGGATTCATC |
| Hfq-R-1 | CCCGAAACCTCTCTCTTTTCCTTATATGCTTATTTGTACTTTG |
| Hfq-F-2 | AAAGAGAGAGGTTTCGGGCTGTTTTTTTACACGGGGAGCCAG |
| Hfq-R-2 (Hind Ⅲ) | ACTAAGCTTCCAGGCGCGACTGTATCTGCACGATGCGATTACG |
| Hfq-QC-F | GCCGAACGCTGAAGAGTTAC |
| Hfq-QC-R | CCGACATCTGCAACGTCAATAC |
| mdRne-F | CAACAAGAAGCGAAGGCGCTGAATGTTGAAGAGCAATCTGTTCAGGAAACCGAACAGGAACCCTTCCCGGCGATCCTCTGG |
| mdRne-R | TCAAAGATGAAATAAAAAAGCCCTGGCAGTTACCAGGGCTTGATTACTTTGAGCTAATTACCCGCATGACGGCAAGTGGACG |
| Rne-I-F | AGTGAACGTACTGAAGGCAGCGATAATCGCG |
| Rne-Spe-I-R | TAGACTTTCCTTGGTGTATCCAACGGCGTCAG |
| Rne-Spe-I-F | ATGTCGCTGCCGACTGGGCAATGGAGCGCC |
| Rne-I-R | ATGCAACTATTTCATCTTACATCATGCAGC |
| Rne-F | GCCAGTTGTTGAAGAAGTG |
| Rne-R | ATGATGTGTTGCCGTATGAC |
| Rne-F-1 (Sac Ⅰ) | TCTGAGCTCCGTCTGAAGAAGAGTTCGCTGAACGTAAGC |
| Rne-R-1 | ACTTTGAGCTAATTATTCCTGTTCGGTTTCCTGAACAGATTGCTC |
| Rne-F-2 | TAATTAGCTCAAAGTAATCAAGCCCTGGTAACTGC |
| Rne-R-2 (Hind Ⅲ) | GCGAAGCTTCTGAAACCAATCTTTTTGCCATGCTGGATAGTGCC |
| Rne-QC-F | CTGTGCCAATCGCTTCTTACC |
| Rne-QC-R | CGGAACCAGACGGTAGCG |
| TKI-Kan-F | TGGGTATAAATGGGCTCG |
| TKI-Kan-R | ATCCTGGTATCGGTCTGC |
| Homologous arms sequences are underlined. Enzyme restriction sites are in italic font. | |
表选项
1.3 质粒提取按照QIAprep Spin Miniprep Kit (QIAGEN)操作步骤提取pREDTKI、pKSI-1和pMDISI质粒。
1.4 pREDTKI质粒电转至E. coli K-12 MG1655感受态细胞取过夜活化的E. coli K-12 MG1655菌液转接LB培养基中,培养至OD600为1.0左右离心收集菌体;预冷的10%甘油洗涤菌体并以100 μL重悬,加入1 μL pREDTKI进行质粒电击,菌液30 ℃摇床复苏1 h,取50 μL涂布含有卡那霉素的LB固体平板培养,12–16 h后挑取单个菌落扩大培养并保种,用TKI-Kan-F/TKI-Kan-R引物鉴定pREDTKI质粒。
1.5 λ-Red重组1.5.1 PCR扩增壮观霉素抗性盒NanoDrop 2000测定提取的pMDISI质粒浓度,再用去离子水稀释成5 ng/μL作为模板。50 μL PCR体系:10×Ex Taq缓冲液5 μL,dNTP Mixture (2.5 mmol/L) 4 μL,Forward primer (10 μmol/L) 1 μL,Reverse primer (10 μmol/L) 1 μL,Ex Taq聚合酶0.4 μL,Pfu聚合酶0.1 μL,pMDISI质粒(5 ng/μL) 8 μL,灭菌双蒸水30.5 μL,共扩增10个体系。PCR扩增条件:95 ℃ 5 min;95 ℃ 40 s,58 ℃ 40 s,72 ℃ 2 min,共30个循环;72 ℃ 10 min。使用QIAquick PCR Purification Kit (QIAGEN)纯化回收PCR产物。
1.5.2 Dpn Ⅰ酶消化甲基化的模板质粒100 μL酶切体系:PCR产物30 μL,Dpn Ⅰ Enzyme 5 μL,10×NE缓冲液10 μL,灭菌双蒸水55 μL。37℃酶切4 h。酶切产物使用QIAquick Gel Extraction Kit (QIAGEN)胶回收壮观霉素抗性盒。
1.5.3 感受态细胞的制备及电转化取活化菌K12 MG1655/pREDTKI扩大培养至OD600为0.8左右,培养时加入L-阿拉伯糖至终浓度10 mmol/L、卡那霉素至终浓度50 μg/mL,离心收集菌体并用冰冷的10%甘油洗涤,100 μL 10%甘油重悬后加入10 μL高浓度的壮观霉素抗性盒,预冷20 min后进行电击,加入液体培养基复苏,取离心后重悬菌体200 μL涂含卡那霉素、壮观霉素的LB固体平板,挑取单个菌落扩大培养并保种,将壮观抗性盒敲入的菌株记为Δhfq∷Spe (壮观霉素Spectinomycin)和Δrne∷Spe;分别用Hfq-I-F/Hfq-Spe-I-R、Hfq-Spe-I-F/ Hfq-I-R、Hfq-F/Hfq-R、Hfq-I-F/Hfq-I-R引物和Rne-I-F/Rne-Spe-I-R、Rne-Spe-I-F/Rne-I-R、Rne-F/ Rne-R、Rne-I-F/Rne-I-R引物鉴定重组菌。
1.6 融合PCR在hfq上下游和rne-710上下游600 bp左右设计引物对,待融合片段上下游引入Sac Ⅰ和HindⅢ酶切位点。分别以Hfq-F-1/Hfq-R-1、Hfq-F-2/Hfq-R-2、Rne-F-1/Rne-R-1、Rne-F-2/Rne-R-2共4对引物,k-12 MG1655菌株基因组为模板进行PCR扩增,再以上述PCR产物为模板,分别用引物对Hfq-F-1/Hfq-R-2和Rne-F-1/Rne-R-2进行扩增,得到缺失hfq和rne-710基因的上下游融合片段。PCR体系和扩增条件同1.5.1,将Ex Taq替换为高保真LA Taq聚合酶,胶回收目的片段。按照常规操作分别酶切融合片段和pKSI-1质粒,PCR回收试剂盒纯化酶切产物,再将回收的片段与载体分别连接,连接产物转化DH5α感受态细胞,引物对Hfq-F-1/Hfq-R-2和Rne-F-1/Rne-R-2进行PCR鉴定,将鉴定正确的细菌培养12 h后提取质粒。pKSI-1-Δhfq和pKSI-1-Δrne-710质粒分别电转至Δhfq∷Spe和Δrne-710∷Spe,方法同上,引物对Hfq-F-1/Hfq-R-2和Rne-F-1/Rne-R-2进行菌落PCR鉴定。
1.7 诱导重组过夜培养K-12 MG1655/pKSI-1-Δhfq/pREDTKI和K-12 MG1655/pKSI-1-Δrne/pREDTKI两种菌液,添加卡那霉素、氨苄青霉素和壮观霉素至常规浓度,5%葡萄糖至终浓度0.5%。接种50 μL上述菌液至5 mL LB液体培养基,加入卡那霉素、L-阿拉伯糖和5%葡萄糖,30 ℃振荡培养6 h至OD600达到0.5左右,加入终浓度为20 mmol/L的IPTG培养过夜;取过夜菌转接至新鲜培养基,同时加入卡那霉素、L-阿拉伯糖、5%葡萄糖和IPTG,30 ℃振荡培养6 h至菌液OD600为0.5左右。
取50 μL菌液用PBS进行10倍梯度稀释,取100、10–1、10–2稀释液各100 μL分别涂平板A (Kanr、10 mmol/L阿拉伯糖、20 mmol/L IPTG)和平板B (Kanr、10 mmol/L阿拉伯糖、20 mmol/L IPTG、Sper),培养后挑取平板A单个菌落分别涂平板A和平板B,选择平板A生长而平板B不生长的菌落,引物对Hfq-QC-F/Hfq-QC-R、Hfq-I-F/ Hfq-I-R、Hfq-F/Hfq-R和Rne-QC-F/Rne-QC-R、Rne-I-F/Rne-I-R、Rne-F/Rne-R分别进行PCR鉴定。
1.8 pREDTKI质粒的消除将鉴定正确的菌液转接入LB液体培养基,42 ℃、80 r/min培养2 h;取少量菌液10倍梯度稀释后分别涂LB板(Kanr)和无抗性LB板,37 ℃温箱过夜培养;选择阴性板生长而抗性板不生长的单克隆,分纯三代并进行PCR鉴定。
2 结果与分析2.1 构建Δhfq∷Spe敲除株pREDTKI质粒电转至E. coli K-12 MG1655感受态细胞,然后使用设计的引物扩增壮观霉素抗性盒,PCR产物纯化回收后经Dpn Ⅰ酶消化质粒模板,再通过胶回收得到壮观霉素抗性盒,使用L-阿拉伯糖诱导Red重组酶,制备E. coli K-12 MG1655/pREDTKI感受态细胞,将纯化的壮观霉素抗性盒电转化至具有Red重组系统的感受态细胞中,染色体上的hfq基因经同源重组被壮观霉素抗性盒替代,从而实现λ-Red重组。通过hfq内引物、外引物以及外引物同壮观霉素抗性盒鉴定引物交叉配对共4对引物鉴定敲除株的正确性。结果可见,内部引物野生株有235 bp大小的条带,敲除株没有条带;外部引物敲除株有2 002 bp大小的条带;第一对交叉引物敲除株有242 bp大小的条带;第二对交叉引物敲除株有537 bp大小的条带,以上结果均与预期一致(图 3)。内部引物鉴定说明敲除株基因组已经不含有hfq基因,外部引物显示分子量较大的条带表明基因组已经含有壮观霉素抗性盒,交叉引物的鉴定说明敲除株同源替换部位准确。以上结果表明Δhfq∷Spe敲除株构建成功。
 |
| 图 3 Δhfq∷Spe敲除株的鉴定 Figure 3 Identification of Δhfq∷Spe. M: DL 5000 DNA marker. 1–3: Hfq-F/Hfq-R. 1: WT; 2: Δhfq; 3: negative control. 4, 5: Hfq-I-F/Hfq-I-R. 4: Δhfq; 5: negative control. 6, 7: Hfq-Spe-I-F/Hfq-I-R. 6: Δhfq; 7: negative control. 8, 9: Hfq-I-F/Hfq-Spe-I-R. 8: Δhfq; 9: negative control. |
| 图选项 |
2.2 构建Δrne-710∷Spe敲除株经过λ-Red重组,壮观霉素抗性盒取代rne-710基因。生长的单克隆中大部分为杂菌,只有少量是敲除株,需要使用引物进行PCR鉴定,使用外引物同壮观霉素抗性盒引物交叉配对、内引物、外引物全面鉴定敲除株。第一对交叉引物敲除株有510 bp大小的条带,第二对交叉引物敲除株有620 bp大小的条带,交叉引物的鉴定说明壮观霉素抗性盒有小部分存在基因组中,敲除株同源替换部位准确;外部引物敲除株有2 054 bp大小的条带,比rne-710分子量大,包含了壮观霉素抗性盒的全部碱基;内部引物敲除株没有条带,野生株有490 bp大小的条带,表明敲除株基因组不含rne-710基因。所有条带的大小与预期结果一致(图 4),表明Δrne-710∷Spe敲除株构建成功。
 |
| 图 4 Δrne∷Spe敲除株的鉴定 Figure 4 Identification of Δrne-710∷Spe. M: DL 5 000 DNA marker. 1, 2: Rne-I-F/Rne-spe-I-R. 1: negative control; 2: Δrne-710. 3, 4: Rne-spe-I-F/Rne-I-R. 3: negative control; 4: Δrne-710. 5, 6: Rne-I-F/Rne-I-R. 5: negative control; 6: Δrne-710. 7–9: Rne-F/Rne-R. 7: negative control; 8: Δrne-710; 9: WT. |
| 图选项 |
2.3 辅助质粒pKSI-1-Δhfq的构建通过融合PCR的方法缺失目的基因片段,设计引物时保证上游的下游引物和下游的上游引物有15–20个重复碱基。首先PCR扩增分体片段594 bp和619 bp,再用上游的上游引物和下游的下游引物扩增融合片段,胶回收后同载体分别双酶切并进行连接转化构建pKSI-1-Δhfq,PCR鉴定可见与预期一致的1 195 bp大小的条带(图 5)。
 |
| 图 5 pKSI-1-Δhfq的鉴定 Figure 5 Identification of pKSI-1-Δhfq. M: DL 2000 DNA marker; 1: negative control; 2: pKSI-1-Δhfq. |
| 图选项 |
2.4 辅助质粒pKSI-1-Δrne-710的构建首先采用融合PCR的方法扩增缺失rne-710片段,再与pKSI-1连接。在缺失rne-710片段的上下游设计引物扩增593 bp和563 bp大小的片段,再进行第二轮扩增融合片段,胶回收纯化PCR产物,同载体分别双酶切,连接并转化DH5α感受态细胞,挑取单克隆进行PCR鉴定,条带的大小为1 156 bp (图 6),表明pKSI-1-Δrne-710构建成功。
 |
| 图 6 pKSI-1-Δrne-710的鉴定 Figure 6 Identification of pKSI-1-Δrne-710. M: DL 2000 DNA marker; 1: negative control; 2: pKSI-1-Δrne-710. |
| 图选项 |
2.5 无痕敲除Δhfq的构建Δhfq∷Spe菌株含有pKSI-1-Δhfq和pREDTKI质粒。加入IPTG进行诱导,使pREDTKI分泌Ⅰ-Sce Ⅰ酶,将含有Ⅰ-Sce Ⅰ酶切位点的壮观霉素和pKSI-1载体酶切,L-阿拉伯糖诱导条件下,pREDTKI质粒发挥λ-Red重组功能,使得酶切后的线性融合片段替代壮观霉素抗性盒,再通过卡那霉素抗性筛选,得到含有pREDTKI质粒的无痕敲除菌株Δhfq。通过鉴定引物进行验证敲除株,外部引物敲除株有499 bp大小的条带,野生株有808 bp大小的条带,敲除株条带小于野生株,证明基因有缺失;内部引物敲除株没有条带,野生株有235 bp大小的条带,说明该片段在敲除株中已经不存在;在融合基因外部设计全长引物进行鉴定,敲除株有1 678 bp大小的条带,野生株有1 987 bp大小的条带,敲除株与野生株大小不同。以上结果与预期的大小一致(图 7),表明无痕敲除Δhfq构建成功。
 |
| 图 7 Δhfq敲除株的鉴定 Figure 7 Identification of Δhfq. M: DL 2000 DNA marker. 1–3: Hfq-I-F/Hfq-I-R. 1: negative control; 2: Δhfq; 3: WT. 4–6: Hfq-F/ Hfq-R. 4: negative control; 5: Δhfq; 6: WT. 7–9: Hfq-QC-F/Hfq-QC-R. 7: negative control; 8:Δhfq; 9: WT. |
| 图选项 |
2.6 无痕敲除Δrne-710的构建Δrne-710∷Spe菌株在保证足够糖供给的情况下,经L-阿拉伯糖和IPTG诱导发生重组。pREDTKI分泌Ⅰ-Sce Ⅰ内切酶,将连接到载体的融合片段酶切成线性游离状态;pREDTKI分泌Red重组酶,促进融合片段与壮观霉素抗性盒发生同源重组,从而得到敲除株Δrne-710。为了准确验证敲除株是否正确,需要3对引物进行PCR扩增鉴定。全长引物敲除株有1 384 bp大小的条带,野生株有2 440 bp大小的条带,敲除株比野生株片段小;外部引物敲除株有551 bp大小的条带,野生株有1 607 bp大小的条带,敲除株条带明显小于野生株,证明基因缺失了大片段;内部引物敲除株没有条带,野生株有490 bp大小的条带,对比野生株说明敲除株rne-710基因缺失。图示结果与预期条带大小一致(图 8),表明无痕敲除Δrne-710构建成功。
 |
| 图 8 Δrne-710敲除株的鉴定 Figure 8 Identification of Δrne-710. M: DL 2000 DNA marker. 1–3: Rne-QC-F/Rne-QC-R. 1: negative control; 2: Δrne-710; 3: WT. 4–6: Rne-I-F/Rne-I-R. 4: negative control; 5: Δrne-710; 6: WT. 7–9: Rne-F/Rne-R. 7: negative control; 8: Δrne-710; 9: WT. |
| 图选项 |
2.7 Δhfq敲除株pREDTKI质粒消除鉴定无痕敲除的Δhfq菌株含有pREDTKI质粒,pREDTKI是温敏型质粒,通过高温培养即可将其消除。此质粒存在的适合温度是30 ℃,本实验将温度提高到42 ℃,低速培养2 h,菌液微浑浊即进行稀释涂板,选择在阴性板生长而在卡那霉素抗性板不生长的菌株,再用无痕敲除鉴定引物以及卡那盒鉴定引物进行PCR鉴定。结果可见,Δhfq内引物、外引物和全长引物均扩增出目的条带,pREDTKI质粒鉴定引物扩增的敲除株没有条带,野生株有613 bp大小的条带,与预期结果一致(图 9),表明敲除株pREDTKI质粒消除成功。
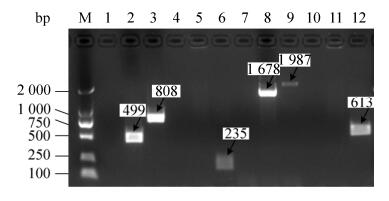 |
| 图 9 Δhfq敲除株pREDTKI质粒消除鉴定 Figure 9 Identification of Δhfq pREDTKI-cured. M: DL 2000 DNA marker. 1–3: Hfq-I-F/Hfq-I-R. 1: negative control; 2: Δhfq; 3: WT. 4–6: Hfq-F/Hfq-R. 4: negative control; 5: Δhfq; 6: WT. 7–9: Hfq-QC-F/Hfq-QC-R. 7: negative control; 8: Δhfq; 9: WT. 10–12: TKI-Kan-F/ TKI-Kan-R. 10: negative control; 11: Δhfq; 12: WT (pREDTKI). |
| 图选项 |
2.8 Δrne-710敲除株pREDTKI质粒消除鉴定无痕敲除的Δrne-710菌株含有pREDTKI质粒,该质粒的存在可能影响对菌株的后续操作。由于pREDTKI是温敏型质粒,提高培养温度至42 ℃即可将其消除,培养后的菌液稀释涂板,通过无抗性筛选即可得到消除菌株。经PCR扩增鉴定,Δrne-710内引物、外引物和全长引物均扩增出目的条带,使用pREDTKI质粒含有的卡那盒鉴定引物扩增的敲除株没有条带,野生株有613 bp大小的条带,与预期结果一致(图 10),表明敲除株pREDTKI质粒消除成功。
 |
| 图 10 Δrne-710敲除株pREDTKI质粒消除鉴定 Figure 10 Identification of Δrne-710 pREDTKI-cured. M: DL 2000 DNA marker. 1–3: Rne-QC-F/Rne-QC-R. 1: negative control; 2: Δrne-710; 3: WT. 4–6: Rne-I-F/Rne-I-R. 4: negative control; 5: Δrne-710; 6: WT. 7–9: Rne-F/Rne-R. 7: negative control; 8: Δrne-710; 9: WT. 10–12: TKI-Kan-F/TKI-Kan-R. 10: negative control; 11: Δrne-710; 12: WT (pREDTKI). |
| 图选项 |
3 讨论大肠杆菌RNase E由Rne基因编码,全长1 061 aa,分为N端催化区(1–529 aa)和支架区(430–1 061 aa)。Hfq与Rne结合位点在711–750 aa。751–1 061 aa对介导sRNA与靶mRNA降解是必需的。因此,本研究在不改变RNase E活性的情况下,保留N端催化区,在支架区从710 aa开始敲除rne基因构建Δrne-710。
常见的基因敲除方法一般选择30–50 bp同源臂,对于一些特殊的基因,这种短同源臂利用一步法很难敲除,必须采用二步法才可实现,同源臂加长到500 bp左右,采取融合PCR方法构建含有同源臂与抗性基因的片段,再同源重组并筛选菌株达到敲除的目的。
本研究无痕敲除过程中,第一步的Red重组将同源臂设计为60 bp,按照常规一步法操作难以实现壮观霉素抗性盒替换目的基因,后来改进了试验方案。由于电转时高强度的电击会损伤菌的活性,因此,在制备感受态细胞时菌液量由5 mL增加到20 mL,菌液的OD600由0.8增加到1.0以上,足够多的菌量接受电击,目的是保证转化的活菌量。加入L-阿拉伯糖的时间由培养终止前1–2 h提前到接菌时同时加入L-阿拉伯糖,增加诱导时间。转化时电击后复苏时间一般为1 h,本研究适当延长时间为1–2 h,更容易获得重组菌。
辅助质粒的参与使得无痕敲除变得简单易行,pKSI-1载体多克隆位点上下游分别含有Ⅰ-Sce Ⅰ酶切位点,两个Ⅰ-Sce Ⅰ位点范围内可以插入靶基因。本研究通过融合PCR构建目的基因敲除片段作为靶基因,第一步分体PCR扩增后不需要纯化回收,直接用产物40倍稀释作模板进行第二轮PCR扩增即可得到融合片段。为了获得较高的扩增产量,对比LA taq酶+pfu酶和EX taq酶+pfu酶扩增效果,发现使用LA taq酶+pfu酶扩增得到的条带更亮,扩增效率更高。用于融合片段的长度在600 bp左右即可,太长不利于融合,并且连接载体的效率也会降低。融合片段连接至pKSI-1载体后需要鉴定,PCR扩增鉴定由于特异性较差,还需要测序相结合,关于测序鉴定需要注意的是:1)测序时不能使用PCR产物,否则测序结果发生的碱基突变,分不清是基因突变还是扩增过程造成的;2)在pKSI-1载体上接近融合片段的两端设计引物,使用特异引物进行测序才能得到正确的结果。
得到的Δhfq∷Spe和Δrne-710∷Spe菌株分别含有pKSI-1-Δhfq和pKSI-1-Δrne-710质粒,同时含有pREDTKI质粒,抗性为卡那霉素、氨苄青霉素和壮观霉素。如果想获得无痕敲除的菌株,必须使pKSI-1载体上的缺失hfq和rne-710片段代替壮观霉素盒,分别重组到Δhfq和Δrne-710敲除株中。在IPTG诱导条件下,pREDTKI分泌Ⅰ-Sce Ⅰ酶,一方面,将pKSI-1载体的Ⅰ-Sce Ⅰ酶切位点部位进行酶切,此区域包含的融合片段即成线性,为进一步的基因重组作好了准备,在L-阿拉伯糖诱导条件下,pREDTKI质粒发挥λ-Red重组功能,可以帮助实现替换过程;另一方面,含有Ⅰ-Sce Ⅰ酶切位点的壮观霉素和pKSI-1载体在Ⅰ-Sce Ⅰ酶切作用下失活。通过卡那霉素抗性筛选,可以得到含有pREDTKI质粒的无痕敲除菌株,pREDTKI为pKD46改造的质粒,温敏型的特点经42 ℃培养即可将其消除。
本研究通过融合PCR方法构建目的基因缺失片段,采用双质粒无痕敲除法,利用λ-Red系统与Ⅰ-Sce Ⅰ核酸内切酶相结合的技术,构建了敲除株Δhfq和Δrne-710,为进一步研究其功能奠定了基础。
参考文献
| [1] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products.Proc Natl Acad Sci USA, 2000, 97(12): 6640–6645.DOI: 10.1073/pnas.120163297 |
| [2] | Muyrers JPP, Zhang YM, Testa G, et al. Rapid modification of bacterial artificial chromosomes by ET-recombination.Nucleic Acids Res, 1999, 27(6): 1555–1557.DOI: 10.1093/nar/27.6.1555 |
| [3] | Muyrers JPP, Zhang YM, Stewart AF. ET-cloning: think recombination first.Genet Eng, 2000, 22: 77–98. |
| [4] | Muyrers JPP, Zhang YM, Stewart AF. Techniques: recombinogenic engineering-new options for cloning and manipulating DNA.Trends Biochem Sci, 2001, 26(5): 325–331.DOI: 10.1016/S0968-0004(00)01757-6 |
| [5] | Court DL, Sawitzke JA, Thomason LC. Genetic engineering using homologous recombination.Annu Rev Genet, 2002, 36: 361–388.DOI: 10.1146/annurev.genet.36.061102.093104 |
| [6] | Murphy KC, Campellone KG, Poteete AR. PCR-mediated gene replacement in Escherichia coli.Gene, 2000, 246(1/2): 321–330. |
| [7] | Takahashi N, Kobayashi I. Evidence for the double-strand break repair model of bacteriophage lambda recombination.Proc Natl Acad Sci USA, 1990, 87(7): 2790–2794.DOI: 10.1073/pnas.87.7.2790 |
| [8] | Ellis HM, Yu DG, DiTizio T, et al. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides.Proc Natl Acad Sci USA, 2001, 98(12): 6742–6746.DOI: 10.1073/pnas.121164898 |
| [9] | Tischer BK, von Einem J, Kaufer B, et al. Two-step red-mediated recombination for versatile high-efficiency markerless DNA manipulation in Escherichia coli.Biotechniques, 2006, 40(2): 191–197.DOI: 10.2144/000112096 |
| [10] | Yu BJ, Kang KH, Lee JH, et al. Rapid and efficient construction of markerless deletions in the Escherichia coli genome.Nucleic Acids Res, 2008, 36(14): e84.DOI: 10.1093/nar/gkn359 |
| [11] | Hashimoto M, Ichimura T, Mizoguchi H, et al. Cell size and nucleoid organization of engineered Escherichia coli cells with a reduced genome.Mol Microbiol, 2005, 55(1): 137–149. |
| [12] | Yu BJ, Sung BH, Koob MD, et al. Minimization of the Escherichia coli genome using a Tn5-targeted Cre/Loxp excision system.Nat Biotechnol, 2002, 20(10): 1018–1023.DOI: 10.1038/nbt740 |
| [13] | Kolisnychenko V, Plunkett III G, Herring CD, et al. Engineering a reduced Escherichia coli genome.Genome Res, 2002, 12(4): 640–647.DOI: 10.1101/gr.217202 |
| [14] | Pósfai G, Kolisnychenko V, Bereczki Z, et al. Markerless gene replacement in Escherichia coli stimulated by a double-strand break in the chromosome.Nucleic Acids Res, 1999, 27(22): 4409–4415.DOI: 10.1093/nar/27.22.4409 |
| [15] | Doublet B, Douard G, Targant H, et al. Antibiotic marker modifications of λ red and FLP helper plasmids, pKD46 and pCP20, for inactivation of chromosomal genes using PCR products in multidrug-resistant strains.J Microbiol Methods, 2008, 75(2): 359–361.DOI: 10.1016/j.mimet.2008.06.010 |
| [16] | Friedman DI, Court DL. Bacteriophage lambda: alive and well and still doing its thing.Curr Opin Microbiol, 2001, 4(2): 201–207.DOI: 10.1016/S1369-5274(00)00189-2 |
| [17] | Murphy KC. Use of bacteriophage λ recombination functions to promote gene replacement in Escherichia coli.J Bacteriol, 1998, 180(8): 2063–2071. |
| [18] | Yang JJ, Sun BB, Huang H, et al. High-efficiency scarless genetic modification in Escherichia coli by using lambda red recombination and Ⅰ-Sce Ⅰ cleavage.Appl Environ Microbiol, 2014, 80(13): 3826–3834.DOI: 10.1128/AEM.00313-14 |
| [19] | Murphy KC, Campellone KG. Lambda red-mediated recombinogenic engineering of enterohemorrhagic and enteropathogenic E. coli.BMC Mol Biol, 2003, 4: 11.DOI: 10.1186/1471-2199-4-11 |
| [20] | Poteete AR. What makes the bacteriophage λ red system useful for genetic engineering: molecular mechanism and biological function.FEMS Microbiol Lett, 2001, 201(1): 9–14. |
| [21] | Dabert P, Smith GR. Gene replacement with linear DNA fragments in wild-type Escherichia coli: enhancement by Chi sites.Genetics, 1997, 145(4): 877–889. |
| [22] | El Karoui M, Amundsen SK, Dabert P, et al. Gene replacement with linear DNA in electroporated wild-type Escherichia coli.Nucleic Acids Res, 1999, 27(5): 1296–1299.DOI: 10.1093/nar/27.5.1296 |
| [23] | Figueroa-Bossi N, Uzzau S, Maloriol D, et al. Variable assortment of prophages provides a transferable repertoire of pathogenic determinants in Salmonella.Mol Microbiol, 2001, 39(2): 260–272.DOI: 10.1046/j.1365-2958.2001.02234.x |
| [24] | Russell CB, Thaler DS, Dahlquist FW. Chromosomal transformation of Escherichia coli recD strains with linearized plasmids.J Bacteriol, 1989, 171(5): 2609–2613.DOI: 10.1128/jb.171.5.2609-2613.1989 |
