

1 天津大学 化工学院 教育部系统生物工程重点实验室,天津 300072
2 天津化学化工协同创新中心,天津 300072
网络出版时间:2016-05-06
基金项目:国家自然科学基金(No. NSFC-21390201),国家重点基础研究发展计划(973计划) (No. 2012CB725203)资助
摘要: 苹果酸是一种重要的C4二羧酸,在食品、医药、化工等领域有广泛的应用。本文主要研究羧化途径强化及苹果酸酶失活对大肠杆菌好氧发酵生产苹果酸的影响。首先在大肠杆菌E2中过表达了磷酸烯醇式丙酮酸羧化酶基因ppc,得到菌株E21,苹果酸积累量从0.57 g/L提高到3.83 g/L。随后,分别过表达来自谷氨酸棒杆菌的丙酮酸羧化酶基因pyc和来自琥珀酸放线杆菌的磷酸烯醇式丙酮酸激酶pck基因,相应的工程菌株E21 (pTrcpyc)和E21 (pTrc-A-pck)分别产6.04和5.01 g/L苹果酸,得率分别达到0.79和0.65 mol/mol葡萄糖。敲除E21中的苹果酸酶基因maeA和maeB,苹果酸产量也显著提高了36%,达到5.21 g/L,得率为0.62 mol/mol。然而,在过表达pyc的基础上敲除苹果酸酶基因并不能进一步提高苹果酸的产量。经过摇瓶发酵条件的初步优化,菌株E21 (pTrcpyc)生产12.45 g/L苹果酸,得率为0.84 mol/mol,达到理论得率的63.2%。
关键词: 大肠杆菌 代谢工程 苹果酸 苹果酸酶 羧化途径
Effects of overexpression of carboxylation pathway genes and inactivation of malic enzymes on malic acid production in Escherichia coli
Fei Lou1,2, Ning Li1,2, Yujiao Zhao1,2, Shiting Guo1,2, Zhiwen Wang1,2, Tao Chen1,2


1 Key Laboratory of Systems Bioengineering, Ministry of Education, School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072, China;
2 Collaborative Innovation Center of Chemical Science and Engineering, Tianjin 300072, China
Received: March 13, 2016; Accepted: April 18, 2016
Supported by:National Natural Science Foundation of China (No. NSFC-21390201), National Basic Research Program of China (973 Program) (No. 2012CB725203)
Corresponding authors:Tao Chen. Tel: +86-22-27406770; E-mail: chentao@tju.edu.cn
Abstract: Malic acid is a dicarboxylic acid that is widely used in food, pharmaceutical and chemical industries. We studied the effects of overexpression of carboxylation pathway genes and inactivation of malic enzymes on the aerobic production of malic acid. Over expression of phosphoenolpyruvate (PEP) carboxylase (ppc) generated strain E21, which increased malic acid production from 0.57 g/L to 3.83 g/L. Then pyc gene from Coryenbacterium glutamicus and pck gene from Actinobacillus succinogenes were overexpressed in E21 separately. The resulting strains E21 (pTrcpyc) and E21 (pTrc-A-pck) produced 6.04 and 5.01 g/L malate with a yield of 0.79 and 0.65 mol/mol glucose, respectively. Deleting two malic enzymes (encoded by maeA and maeB) also led to an increase of 36% in malic acid production with a production of 5.21 g/L. However, the combination of malic enzymes deletion and pyc overexpression could not further increase the yield of malic acid. After optimization of fermentation conditions, strain E21 (pTrcpyc) produced 12.45 g/L malic acid with a yield of 0.84 mol/mol which is 63.2% of the theoretical yield.
Key words: Escherichia coli metabolic engineering malic acid malic enzyme carboxylation pathway
苹果酸,又名2-羟基丁二酸,是生物体内三羧酸循环的重要一员,其酸味持久柔和,是优良的酸味剂,苹果酸在医药、食品等多个行业中都有广泛用途[1-2]。苹果酸的生产方法包括从植物中提取、酶转化法、化学合成法及微生物发酵法[1],由于能源和环境的日益恶化,微生物发酵法越来越受重视。
曲霉属是已知的最好的苹果酸发酵生产菌株,可以高效发酵葡萄糖生产苹果酸。Battat等[3]使用黄曲霉菌株Aspergillus flavus Kyowa A-114 (ATCC 13697)通过优化发酵条件和发酵培养基,在16 L罐上发酵,最终苹果酸产量高达113 g/L,产率为0.59 g/(L?h),得率为1.28 mol/mol。但是由于发酵过程有黄曲霉毒素产生,因此不适于工业化生产。除黄曲霉外,曲霉属中的黑曲霉和米曲霉都能以葡萄糖为碳源生产苹果酸[4-7]。其中,黑曲霉Aspergillus niger ATCC 9124以酒糟为底物可以生产苹果酸17 g/L,葡萄糖得率为0.8 g/g[4]。Brown等[5]在米曲霉Aspergillus nigerNRRL 3488中通过过表达细胞质中的丙酮酸羧化酶、苹果酸脱氢酶及C4-二羧酸转运蛋白,发酵164 h,生产苹果酸154 g/L,生产速率0.94 g/(L?h),得率为1.38 mol/mol,达到最大理论得率的69%,这是已报道的苹果酸生产速率最高的菌株。另外,酿酒酵母也可用于生产苹果酸[8-12]。Pines等[10]通过过表达细胞质中的苹果酸脱氢酶MDH2使得苹果酸产量提高了3.7倍。Zelle等[11]通过表达内源丙酮酸羧化酶、苹果酸脱氢酶,来自裂殖酵母的异源苹果酸运输蛋白构建的工程菌株,在好氧分批培养中产生59 g/L苹果酸,得率为0.42 mol/mol。大肠杆菌作为应用最广泛的模式微生物之一,已被成功用于发酵生产多种重要的有机酸如琥珀酸、富马酸等[13-15],基于大肠杆菌构建生产苹果酸的代谢工程菌株也日益受到重视[13, 16-17]。Moon等[17]在pta突变菌株中,通过表达外源丙酮酸羧激酶基因pckA来增强PEP羧化通量,好氧发酵12 h,生产9.25 g/L苹果酸。Zhang等[13]在生产琥珀酸的大肠杆菌的基础上敲除了富马酸还原酶基因frdBC、苹果酸酶基因sfcA和maeB以及富马酸酶基因fumB和fumAC,构建了工程菌XZ658。该菌株采用双阶段厌氧发酵,72 h后L-苹果酸产量可以达到253 mmol/L,得率为1.42 mol/mol,产率0.47 g/(L?h),是目前大肠杆菌产苹果酸中得率最高的菌株。
本课题组在前期研究中通过进化工程得到1株有效激活乙醛酸循环的大肠杆菌E2,该菌株经过进一步的代谢工程改造,可在好氧条件下高效利用甘油合成琥珀酸和富马酸[14-15]。本文将进一步研究E2菌株在好氧条件下发酵生产苹果酸的潜力,尝试通过强化羧化途径相关基因的表达及敲除苹果酸酶基因来提高苹果酸的产量和得率。
1 材料与方法1.1 菌株、质粒和引物本研究所用菌株和质粒列于表 1中,所用引物列于表 2中,E2作为产苹果酸工程菌的出发菌株。
表 1 本研究所用菌株和质粒Table 1 Escherichia colistrains and plasmids used in this study
| Strains/plasmids | Relevant characteristics | References/sources |
| strains | ||
| E2 | BL21 (DE3), Δppc, aceBAK:trc | [14] |
| E21 | E2, ppc:trc | This study |
| E21 (pTrcpyc) | E21 harboring pTrcpyc | This study |
| E21 (pTrc-A-pck) | E21 harboring pTrc-A-pck | This study |
| E22 | E21, ΔmaeA | This study |
| E23 | E21, ΔmaeA, ΔmaeB | This study |
| E23 (pTrcpyc) | E23 harboring pTrcpyc | This study |
| Plasmids | ||
| pTKRED | pSC101 replication, ParaBAD-driven I-SceI gene, λ-Red, SpR | [18] |
| pTKS/CS | p15A replication, LP regions, I-SceI restriction sites, CmR, TetR | [18] |
| pKMtrc | Bla, kan, kan from pKD4 cloned into pUCtrc | Lab station |
| pKD46 | Bla, λ-Red recombinase under araBAD promoter, temperature-conditional replicon | [19] |
| pCP20 | Bla, cat, yeast Flp recombinase | [20] |
| pTrcpyc | pyc gene from Corynebacterium glutamicum cloned in plasmid pTrc99a | Lab station |
| pTrc-A-pck | A-pckgene from Actinobacillus succinogenes cloned in plasmid pTrc99a | This study |
表选项
表 2 本研究所用引物Table 2 Primers used in this study
| Primers | DNA sequence (5'-3') |
| T1 | GCGTGAAGTGGTTCGGTTG |
| T2 | CTGTAATGCAGGTAAAGCGATC |
| maeA U-F | CAGTGATATCTACCAGCAAACGA |
| maeA U-R | CGTCTTTGCTTTACCGCCAG |
| maeA T-F | CTTGAGGCCGACGCCCTGGCGGTAAAGCAAAGACGTAGGGATAACAGGGTAATGTACCA |
| maeA T-R | CGTCTTTGCTTTACCGCCAGGGCGTCGGCCTCAAGATTACCCTGTTATCCCTACTAAG |
| maeA L-F | CTTGAGGCCGACGCCCTGGCGGTAAAGCAAAGACGTCTCATTTGCCTCACCTGCTA |
| maeA L-R | ATGCACTCTTCCGCCATCCC |
| maeB U-F | CAGCTTTGAGTAACAGCGAA |
| maeB U-R | TTGGTTGTTCCTTTCACGTA |
| maeB T-F | CTTTATTTGTGAACGTTACGTGAAAGGAACAACCAATAGGGATAACAGGGTAATGTACC |
| maeB T-R | TTGGTTGTTCCTTTCACGTAACGTTCACAAATAAAGATTACCCTGTTATCCCTACTAAG |
| maeB L-F | CTTTATTTGTGAACGTTACGTGAAAGGAACAACCAATTAACTCTCACGCTTATCCTG |
| maeB L-R | AGTTAGCGTCATAATGCCAA |
| ppc-F | GGCGATTTTTTAACATTTCCATAAGTTACGCTTATTTAAAGCGTCGTGAGTGTAGGCTGGAGCT GCTTC |
| ppc-R | TTGCCGAGCATACTGACATTACTACGCAATGCGGAATATTGTTCGTTCATGGTCTGTTTCCTGT GTGAA |
表选项
1.2 目的基因的敲除及ppc启动子的置换用于基因敲除的方法是基于λ-Red介导的重组系统[21],片段的构建需要3轮PCR,第一轮PCR的引物是U-F/U-R和L-R/L-F,以大肠杆菌基因组为模板,扩增获得5'和3'同源臂。分别以T2/T-F和T1/T-R为引物从质粒pTKS/CS上扩增得到筛选标记片段tetA-U和tetA-L。其中引物U-R和L-F的5'延伸区包含Ⅰ-SceⅠ和30 bp的重叠区域分别与引物T-F,T-R的引物序列互补。在第二轮PCR中,用引物U-F/T和T1/U-R,将5'同源臂和tetA-U,及3'同源臂和tetA-L融合成2个片段。最后这2个片段进一步融合,用U-F/L-R作引物。最终获得的片段通过电转导入到含有pTKRED的感受态细胞中,过夜培养,通过PCR挑选阳性转化子。将验证正确的细菌转入LB中培养,加入0.2% (W/V)的阿拉伯糖诱导Ⅰ-SceⅠ内切酶的表达,过夜培养,稀释适当浓度,涂布含有阿拉伯糖的平板,PCR验证tetA基因有没有敲掉。
基因启动子的置换原理是用包含有抗生素抗性基因和trc启动子的DNA片段通过λ-Red同源重组的方式置换该基因本身的启动子区,并需前后各50 bp序列作为同源臂。选择P基因编码区上游50 bp序列为上游同源臂,P基因编码区从ATG开始前50 bp为下游同源臂,并将上下游同源臂设计到引物P-F/R中,以质粒pKMtrc为模板,扩增卡那霉素抗性基因和trc启动子。将扩增得到的DNA片段电转入含有质粒pKD46的感受态细胞中,30℃过夜培养,通过卡那霉素进行筛选,并用PCR验证启动子是否替换成功。将目的转化子转入液体LB中37℃过夜培养消除质粒pKD46。将质粒pCP20转入pKD46消除的菌株中,在含氯霉素和氨苄青霉素的平板上30℃过夜培养,得到阳性转化子,随后,将转化子在不加抗生素的固体LB平板上划线,43℃培养过夜。选取若干个单克隆点在含有卡那霉素、氨苄青霉素和无抗LB固体平板上。若在氨苄青霉素平板上和卡那霉素平板上均没有生长,而在无抗生素平板上生长,说明质粒pCP20和卡那抗性均已消除。挑选阳性克隆进行PCR验证,并保存抗性基因弹出菌株。PCR中所用引物列于表 2。
1.3 培养基及培养条件1.3.1 培养基LB培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10。配置固体培养基时加入20 g/L琼脂粉。
M9培养基(g/L):NaH2PO4 6.78,KH2PO4 3,NaCl 0.5,NH4Cl 1,使用时按体积分数1%的量补加MgSO4、CaCl2和微量元素,并添加适量葡萄糖作为碳源。
微量元素(g/L):FeSO4·7H2O 2.8×10-4,Thiamine·HCl 1.0×10-7,CoCl2·6H2O 7.1×10-6,(NH4)6Mo7O24·4H2O 3.7×10-6,MnCl2·4H2O 1.6×10-5,CuSO42.41×10-6,ZnSO4·7H2O 2.8×10-6,H3BO32.5×10-5,MOPS 20.9。
抗生素:使用终浓度为氨苄青霉素100 μg/mL,奇霉素100 μg/mL,四环素20 μg/mL,氯霉素5 μg/mL,卡那霉素10 μg/mL。
1.3.2 培养条件发酵实验所用的培养基为添加有相应抗生素和葡萄糖的M9培养基。若需诱导基因的表达,则添加0.2 mmol/L IPTG。含有丙酮酸羧化酶活性的菌株需要添加1 mg/L的生物素。
有氧摇瓶发酵:将-80℃保存的菌株在加有相应抗生素的LB平板上划线,挑取单菌落接种到5 mL LB试管中,37℃、220 r/min过夜培养,1%的转接量转接到装有25 mL M9培养基、10 g/L葡萄糖的250 mL锥形瓶中,37℃、220 r/min培养至对数中期,转接到装有50 mL M9培养基,10 g/L葡萄糖的500 mL锥形瓶中,初始OD600为0.05,37℃、220 r/min发酵,发酵周期48 h。
1.4 代谢产物分析细胞生长量用紫外分光光度计检测600 nm处的吸光值;葡萄糖浓度使用生物传感分析仪(山东省科学院生物研究所)检测;有机酸检测由高效液相色谱法(HPLC)检测,色谱柱为Aminex 87-H;检测器为紫外检测器(Agilent,G1315D)和示差折光检测器(Agilent,HP1047A);紫外检测波长为210 nm;流动相:2.5 mmol/L H2SO4;流速:0.4 mL/min;进样量:20 μL;柱温:65℃。
1.5 苹果酸最大理论得率计算在好氧条件下以葡萄糖为底物经乙醛酸循环和羧化途径合成苹果酸具有最大的理论得率,其反应式为:
3 C6H12O6+10 NAD++2 FAD+6 H2O→
4 C4H6O5+10 NADH+2 FADH2+2 CO2+10 H+
从上式可知,得率为1.33 mol/mol。
2 结果与分析2.1 羧化途径强化对苹果酸积累的影响本研究的出发菌株E2是1株通过适应性进化激活乙醛酸途径的大肠杆菌[14],该菌株在好氧条件下可以积累少量的苹果酸(0.57 g/L,表 3)。这说明乙醛酸循环途径的增强可以提高苹果酸的积累。以葡萄糖为底物生产苹果酸的代谢途径可以分为非氧化途径、氧化途径和乙醛酸循环途径。有报道表明,乙醛酸循环途径中苹果酸合酶,还原途径中丙酮酸羧化酶及细胞质中苹果酸脱氢酶都能促进苹果酸的生产[11, 22-23]。根据文献报道,增强羧化途径有利于苹果酸的积累[17, 24-25]。因此,我们首选研究了过表达羧化途径相关基因对苹果酸积累的影响。
表 3 重组菌株摇瓶发酵结果Table 3 Fermentation results of recombinant strains in shake flask
| Strains | Glucose (g/L) | OD600 | Malate (g/L) | Mal yielda | Fermentation product concentration (g/L) | |||||
| Pyr | Suc | Fum | Ace | AKG | OAA | |||||
| E2 | 10.1±0.1 | 3.8±0.2 | 0.57±0.02 | 0.08 | 0.42±0.02 | 0.11±0.01 | - | 2.31±0.18 | 0.45±0.02 | - |
| E21 | 10.1±0.1 | 4.4±0.2 | 3.83±0.04 | 0.51 | 0.31±0.03 | 0.11±0.02 | 0.11±0.01 | 1.52±0.15 | 0.51±0.05 | 0.12±0.01 |
| E21 (pTrcpyc) | 10.2±0.2 | 4.5±0.2 | 6.04±0.06 | 0.79 | - | 0.31±0.09 | 0.05±0.01 | 0.16±0.01 | - | 0.21±0.02 |
| E21 (pTrc-A-pck) | 10.3±0.2 | 4.4±0.2 | 5.01±0.1 | 0.65 | 0.12±0.01 | 0.31±0.02 | - | 0.52±0.01 | - | 0.11±0.01 |
| E22 | 10.3±0.1 | 4.5±0.2 | 4.09±0.1 | 0.53 | 0.25±0.03 | 0.12±0.01 | - | 1.51±0.14 | 0.18±0.02 | 0.12±0.01 |
| E23 | 11.3±0.3 | 4.3±0.2 | 5.21±0.13 | 0.62 | 0.15±0.01 | 0.21±0.02 | - | 1.32±0.08 | 0.21±0.01 | 0.58±0.03 |
| E23(pTrcpyc) | 11.5±0.1 | 4.0±0.2 | 3.84±0.05 | 0.44 | - | 0.18±0.03 | - | 1.71±0.13 | 0.10±0.01 | 0.76±0.02 |
| a: yield was calculated as mol of malate produced per mol of glucose consumed; Mal: malate; Pyr: pyruvate; Suc: succinate; Ace: acetate; AKG: α-ketoglutarate; OAA: oxaloacetate. | ||||||||||
表选项
首先,将大肠杆菌自身ppc基因的启动子置换为trc强启动子,构建菌株E21。在好氧发酵条件下,菌株E21可以积累苹果酸3.83 g/L,同E2相比,菌株E21的乙酸产量由2.3 g/L降低到1.52 g/L,丙酮酸产量也减少到0.31 g/L,同时a-酮戊二酸的积累量增加到0.51 g/L。实验结果表明强化羧化途径可以提高前体物草酰乙酸的供给,有利于苹果酸的积累。由于菌株E21仍然积累1.52 g/L的乙酸以及0.31 g/L的丙酮酸,这表明可以进一步强化从丙酮酸或PEP到草酰乙酸的通量以减少上述副产物的积累量。因此,我们进一步在菌株E21中分别过表达了来自谷氨酸棒杆菌的丙酮酸羧化酶基因pyc和产琥珀酸杆菌的磷酸烯醇式丙酮酸激酶pck基因,以考察其对苹果酸积累量和得率的影响。
我们以pTrc为载体,分别构建了pyc和pck的表达质粒pTrcpyc和pTrc-A-pck,将上述质粒导入菌株E21中,得到菌株E21 (pTrcpyc)和E21 (pTrc-A-pck)。在好氧摇瓶发酵中,2株工程菌的苹果酸积累量分别达到6.04和5.01 g/L,比对照菌株E21提高了56%和29%;得率分别达到0.79和0.65 mol/mol葡萄糖。同E21相比,菌株E21 (pTrcpyc)的乙酸产量降低到0.16 g/L,草酰乙酸的积累量增加到0.21 g/L,同时未检测到丙酮酸的积累,这表明pyc基因的过表达有效加强了从丙酮酸到草酰乙酸的通量,并进一步导致苹果酸积累量的增加。菌株E21 (pTrc-A-pck)的乙酸产量降低到0.52 g/L,丙酮酸的积累降低到0.12 g/L,而草酰乙酸的积累量未发生明显变化,这表明pck基因的表达效果差于pyc基因,我们推测可能E21中ppc的表达造成胞内PEP浓度显著降低,丙酮酸的浓度由于葡萄糖通过PTS系统运输而保持较高的水平,从而导致上述两种基因表达效果的不同。另外,虽然pck基因涉及的羧化反应比PYC催化的反应多生成1个ATP,但在好氧发酵条件下,我们并没有观察到pck基因比pyc基因有任何优势。
2.2 苹果酸酶的敲除对苹果酸积累的影响苹果酸酶催化苹果酸和丙酮酸之间的相互转化,但这两个酶对丙酮酸的亲和力远远小于苹果酸,它们主要催化苹果酸向丙酮酸转化[26]。因此可以敲除苹果酸酶基因来降低苹果酸的消耗,从而提高苹果酸的积累量。我们依次在E21菌种中敲除了2个苹果酸酶编码基因maeA和maeB,构建了菌株E22 (E21,ΔmaeA)和E23 (E21,ΔmaeA,ΔmaeB),摇瓶好氧发酵结果见表 3。同对照菌株E21相比,菌株E22的苹果酸积累量有少量增加,达到4.09 g/L,得率为0.53 mol/mol,a-酮戊二酸的产量显著降低,但丙酮酸、乙酸等副产物的积累量几乎未发生明显改变。maeA和maeB双敲除菌株E23的苹果酸产量进一步增加到5.21 g/L,比E21提高了36%。除了a-酮戊二酸的产量显著降低外,副产物丙酮酸的产量也降低到0.15 g/L,但草酰乙酸的积累量增加至0.58 g/L。上列实验结果表明敲除maeA和maeB后可降低丙酮酸的积累量,有效阻断苹果酸的消耗途径,提高苹果酸的产量和得率。
2.3 苹果酸酶敲除和羧化途径增强的组合对苹果酸积累的影响上述实验结果显示,增强羧化途径或者敲除2个苹果酸酶都可以显著提高苹果酸的积累。因此,我们尝试将苹果酸酶敲除和羧化途径强化的基因修饰组合起来,以进一步提高苹果酸产量及得率。将质粒pTrcpyc导入菌株E23后得到E23 (pTrcpyc),发酵结果见表 3。工程菌E23 (pTrcpyc)的苹果酸产量没有继续增加,反而降低到3.84 g/L,这与我们预期的结果完全不同。与对照菌株E21 (pTrcpyc)和E23相比,菌株E23 (pTrcpyc)的发酵副产物草酰乙酸增加到0.76 g/L,这表明在敲除苹果酸酶基因的基础上继续增强羧化途径使得草酰乙酸进一步得到积累,但其并没有完全通过TCA循环生成苹果酸,反而促进了副产物乙酸的生成。有文献报道,草酰乙酸的浓度一定程度上会影响柠檬酸合成酶的催化速率[27]。因此草酰乙酸的积累可能影响TCA循环的通量,从而造成乙酸的生成及苹果酸产量的下降,造成上述现象的原因还有待进一步深入研究。
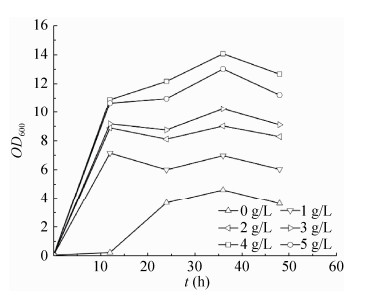
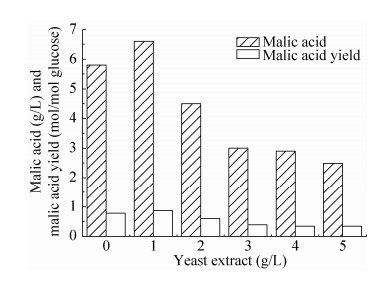
2.4 菌株E21 (pTrcpyc)的发酵优化实验基于以上实验结果,选择苹果酸得率最高的工程菌E21 (pTrcpyc)进行后续发酵优化实验。虽然在好氧条件下,菌株E21 (pTrcpyc)在基本盐培养基中的苹果酸得率可以达到0.79 mol/mol,但菌体生长及耗糖速度较慢。因此我们研究了不同浓度的酵母抽提物对苹果酸积累的影响。菌株E21 (pTrcpyc)按1.3所描述的培养条件,分别添加终浓度为0、1、2、3、4、5 g/L的酵母抽提物,菌体生长及发酵结果分别如图 1和图 2所示。当添加的酵母抽提物浓度范围在0?4 g/L时,随酵母抽提物浓度的升高,菌体生长的最大OD逐渐增加;当浓度达到5 g/L时,菌体最大OD不再增加。当酵母抽提物浓度为1 g/L时,菌株E21 (pTrcpyc)的苹果酸产量和得率均为最高,分别达到6.61 g/L和0.87 mol/mol。
 |
| 图 1 不同酵母抽提物浓度对菌体生长的影响 Figure 1 Effects of different concentration of yeast extract on cell growth. |
| 图选项 |
 |
| 图 2 不同酵母抽提物浓度对苹果酸得率的影响 Figure 2 Effects of different concentration of yeast extract on malic acid yield. |
| 图选项 |
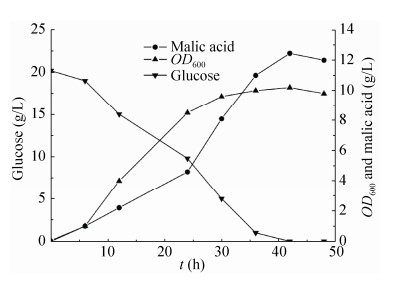
随后,我们进一步将葡萄糖浓度提高到20 g/L,摇瓶发酵结果如图 3所示。发酵周期约42 h,耗糖约20 g/L,产苹果酸12.45 g/L,得率为0.84 mol/mol,达到最大理论得率的63.2%。本研究构建的菌株在初步优化后的摇瓶发酵条件下,拥有较好的生长性能,并显著缩短了发酵周期,但其苹果酸产量和产率较低,因此在后续的研究中需要进一步开发该菌株的流加发酵工艺,以显著改进苹果酸产量及产率。
 |
| 图 3 好氧条件下菌株E21 (pTrcpyc)的发酵曲线图 Figure 3 The fermentation curve of strain E21 (pTrcpyc) under aerobic conditions. |
| 图选项 |
3 结论进化菌株E2被改造用于好氧条件下苹果酸的生产,增强羧化途径或者敲除苹果酸酶都能显著提高苹果酸的积累。其中过表达ppc和pyc的菌株E21 (pTrcpyc)在好氧发酵条件下产苹果酸6.04 g/L,得率为0.79 mol/mol。然而,强化羧化途径和敲除苹果酸酶基因的组合修饰却不能进一步增加苹果酸产量。对菌株E21 (pTrcpyc)进行发酵培养基的初步优化,好氧发酵42 h,可生产苹果酸12.45 g/L,得率为0.84 mol/mol,达到最大理论得率的63.2%。
参考文献
| [1] | Tsao GT, Cao NJ, Du J, et al. Production of multifunctional organic acids from renewable resources.Adv Biochem Eng Biot,1999, 65(65): 243–280. |
| [2] | Bressler E, Pines O, Goldberg I, et al. Conversion of fumaric acid to L-malic by sol-gel immobilized Saccharomyces cerevisiae in a supported liquid membrane bioreactor.Biotechnol Progr,2002, 18(18): 445–450. |
| [3] | Battat E, Peleg Y, Bercovitz A, et al. Optimization of L-malic acid production by Aspergillus flavus in a stirred fermentor.Biotechnol Bioeng,1991, 37(11): 1108–1116.DOI: 10.1002/(ISSN)1097-0290 |
| [4] | West TP. Malic acid production from thin stillage by Aspergillus species.Biotechnol Lett,2011, 33(12): 2463–2467.DOI: 10.1007/s10529-011-0720-7 |
| [5] | Brown SH, Bashkirova L, Berka R, et al. Metabolic engineering of Aspergillus oryzae NRRL 3488 for increased production of L-malic acid.Appl Microbiol Biot,2013, 97(20): 8903–8912.DOI: 10.1007/s00253-013-5132-2 |
| [6] | Knuf C, Nookaew I, Brown SH, et al. Investigation of malic acid production in Aspergillus oryzae under nitrogen starvation conditions.Appl Environ Microb,2013, 79(19): 6050–6058.DOI: 10.1128/AEM.01445-13 |
| [7] | Knuf C, Nookaew I, Remmers I, et al. Physiological characterization of the high malic acid-producing Aspergillus oryzae strain 2103a-68.Appl Microbiol Biot,2014, 98(8): 3517–3527.DOI: 10.1007/s00253-013-5465-x |
| [8] | Chen X, Xu G, Xu N, et al. Metabolic engineering of Torulopsis glabrata for malate production.Metab Eng,2013, 19(5): 10–16. |
| [9] | Pines O, Even-Ram S, Elnathan N, et al. The cytosolic pathway of L-malic acid synthesis in Saccharomyces cerevisiae: the role of fumarase.Appl Microbiol Biot,1996, 46(4): 393–399. |
| [10] | Pines O, Shemesh S, Battat E, et al. Overexpression of cytosolic malate dehydrogenase (MDH2) causes overproduction of specific organic acids in Saccharomyces cerevisiae.Appl Microbiol Biot,1997, 48(2): 248–255.DOI: 10.1007/s002530051046 |
| [11] | Zelle RM, De Hulster E, Van Winden WA, et al. Malic acid production by Saccharomyces cerevisiae: engineering of pyruvate carboxylation, oxaloacetate reduction, and malate export.Appl Environ Microb,2008, 74(9): 2766–2777.DOI: 10.1128/AEM.02591-07 |
| [12] | Nakayama S, Tabata K, Oba T, et al. Characteristics of the high malic acid production mechanism in Saccharomyces cerevisiae sake yeast strain No. 28.J Biosci Bioeng,2012, 114(3): 281–285.DOI: 10.1016/j.jbiosc.2012.04.003 |
| [13] | Zhang X, Wang X, Shanmugam KT, et al. L-malate production by metabolically engineered Escherichia coli.Appl Environ Microb,2011, 77(2): 427–434.DOI: 10.1128/AEM.01971-10 |
| [14] | Li N, Zhang B, Chen T, et al. Directed pathway evolution of the glyoxylate shunt in Escherichia coli for improved aerobic succinate production from glycerol.J Ind Microbiol Biot,2013, 40(12): 1461–1475.DOI: 10.1007/s10295-013-1342-y |
| [15] | Li N, Zhang B, Wang ZW, et al. Engineering Escherichia coli for fumaric acid production from glycerol.Bioresource Technol,2014, 174: 81–87.DOI: 10.1016/j.biortech.2014.09.147 |
| [16] | Wu YB, Zhang L, Shi GY. Construction of recombinant Escherichia coli for L-malic acid production.Chin J Bioproc Eng,2014, 12(3): 12–18.(in Chinese). 吴亚斌, 张梁, 石贵阳. 产L-苹果酸重组大肠杆菌的构建.生物加工过程, 2014, 12(3): 12-18. |
| [17] | Moon SY, Hong SH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of malic acid.Biochem Eng J,2008, 40(2): 312–320.DOI: 10.1016/j.bej.2008.01.001 |
| [18] | Kuhlman TE, Cox EC. Site-specific chromosomal integration of large synthetic constructs.Nucleic Acids Res,2010, 38(6): e92.DOI: 10.1093/nar/gkp1193 |
| [19] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products.Proc Nati Acad Sci USA,2000, 97(12): 6640–6645.DOI: 10.1073/pnas.120163297 |
| [20] | Cherepanov PP, Wackernagel W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant.Gene,1995, 158(1): 9–14.DOI: 10.1016/0378-1119(95)00193-A |
| [21] | Lin ZQ, Xu ZB, Li YF, et al. Metabolic engineering of Escherichia coli for the production of riboflavin.Microb Cell Fact,2014, 13: 104. |
| [22] | Chi Z, Wang ZP, Wang GY, et al. Microbial biosynthesis and secretion of L-malic acid and its applications.Crit Rev Biotechnol,2014: 1–9. |
| [23] | Koganemaru K, Ohura Y, Kanda K, et al. Mechanism of malic acid production by a cycloheximide-resistant sake yeast.J Soc Brew Jpn,2001, 96(4): 275–281.DOI: 10.6013/jbrewsocjapan1988.96.275 |
| [24] | Zhou L, Cui WJ, Liu ZM, et al. Advance in L-malate production based on metabolic engineering strategies.Sci Technol Food Ind,2015, 36(10): 383–388.(in Chinese). 周丽, 崔文璟, 刘中美, 等. 基于代谢工程策略合成L-苹果酸研究进展.食品工业科技, 2015, 36(10): 383-388. |
| [25] | Wu JL, Wu QP, Zhang JM, et al. Progress in L-malic acid biosynthesis.Food Sci,2014, 35(3): 238–242.(in Chinese). 吴军林, 吴清平, 张菊梅, 等. L-苹果酸生物合成研究进展.食品科学, 2014, 35(3): 238-242. |
| [26] | Yamaguchi M. Studies on regulatory functions of malic enzymes. IV. Effects of sulfhydryl group modification on the catalytic function of NAD-linked malic enzyme from Escherichia coli.J Biochem,1979, 86(2): 325–333. |
| [27] | Robinson MS, Easom RA, Danson MJ, et al. Citrate synthase of Escherichia coli. Characterisation of the enzyme from a plasmid-cloned gene and amplification of the intracellular levels.FEBS Lett,1983, 154(154): 51–54. |
