
 , 吴胜2
, 吴胜2 

1. 沈阳药科大学生命科学和生物制药学院,辽宁 沈阳 110016
2. 中国科学院微生物研究所 微生物资源前期开发国家重点实验室,北京 100101
摘要:从嗜高温放线菌Thermobifida fusca中分离得到的苯基丙酮单加氧酶主要催化芳香族化合物的Baeyer-Villiger氧化反应。对该酶的结构和功能进行研究时,发现位于底物结合口袋的Met446位点突变可以赋予突变酶催化C-H键活化的新功能,氧化吲哚合成靛蓝和靛玉红,但产量仅为1.89 mg/L。为了获得合成靛蓝和靛玉红的全细胞催化剂,直接补加吲哚并不能提高细胞合成效率,补加吲哚的前体物质L-色氨酸可以使细胞合成靛蓝和靛玉红的能力提高4.5倍,达到8.43 mg/L。为了进一步提高细胞的生物合成效率,通过代谢工程改造大肠杆菌的糖代谢途径,阻断葡萄糖异构酶基因pgi,使磷酸戊糖途径代替糖酵解途径成为葡萄糖的主要代谢通路,从而为细胞提供更多氧化吲哚所需的辅因子NADPH,导致细胞合成靛蓝和靛玉红的效率进一步提高3倍,达到25 mg/L。通过组合蛋白质工程和代谢工程设计全细胞催化剂不仅可以高效地合成靛蓝和靛玉红,而且设计理念为相关全细胞催化剂的开发提供了一种新的策略。
关键词: 靛蓝靛玉红苯基丙酮单加氧酶蛋白质工程代谢工程
Biosynthesis of indigo and indirubin by whole-cell catalyst designed by combination of protein engineering and metabolic engineering
Yang Li1, 2, Junge Zhu1, Jianjun Wang2, Huanzhang Xia1

 , and Sheng Wu2
, and Sheng Wu2

1. School of Life Science and Biopharmaceutics, Shenyang Pharmaceutical University, Shenyang 110016, Liaoning, China
2 State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101,China
Received: March 30, 2015; Accepted: May 15, 2015
Supported by: National Natural Science Foundation of China (No. 31070718), Knowledge Innovation Program of the ChineseAcademyof Sciences (No. KSCX2-EW-J-6).
Corresponding authors: Huanzhang Xia. Tel/Fax: +86-24-23986438; E-mail: xiahz612@sina.comSheng Wu. Tel: +86-10-62628482; Fax: +86-10-64807429; E-mail: shengwu@im.ac.cn
Abstract: The phenylacetone monooxygenase, isolated from Thermobifida fusca, mainly catalyzes Baeyer-Villiger oxidation reaction towards aromatic compounds. Met446 plays a vital role in catalytic promiscuity, based on the structure and function of phenylacetone monooxygenase. Mutation in Met446 locus can offer enzyme new catalytic feature to activate C—H bond, oxidizing indole to finally generate indigo and indirubin, but the yield was only 1.89 mg/L. In order to further improve the biosynthesis efficiency of the whole-cell catalyst, metabolic engineering was applied to change glucose metabolism pathway of Escherichia coli. Blocking glucose isomerase gene pgi led to pentose phosphate pathway instead of the glycolytic pathway to become the major metabolic pathways of glucose, which provided more cofactor NADPH needed in enzymatic oxidation of indole. Engineering the host E. coli led to synthesis of indigo and indirubin efficiency further increased to 25 mg/L. Combination of protein and metabolic engineering to design efficient whole-cell catalysts not only improves the synthesis of indigo and indirubin, but also provides a novel strategy for whole-cell catalyst development.
Keywords: indigo,indirubinphenylacetone monooxygenaseprotein engineeringmetabolic engineering
靛蓝 (Indigo),是最早发现的天然染料之一,距今已有3 000多年的历史。《荀子·劝学》中有“青,取之于蓝而青于蓝”,此“青”即靛蓝。靛蓝广泛应用于食品、医药和日用化妆品的着色等。靛玉红 (Indirubin),靛蓝的同分异构体,是传统中药当归龙荟丸的有效成分[1],具有抗癌活性[2, 3, 4]。
靛蓝和靛玉红的生产主要有3种方法:植物提取、化学合成及微生物转化[5]。植物提取工艺繁琐,产量低;化学合成法具有效率高、产品纯度高、过程简单等优点,是目前靛蓝和靛玉红制备的主要方法。但是,化学合成使用的催化剂、物料以及副产物具有一定的毒性对人和环境带来危害。自1928年首次报道靛蓝的微生物合成现象以来,绿色环保的微生物法合成靛蓝引发越来越多的关注[6]。研究发现许多降解芳香族碳氢化合物的微生物具有产生靛蓝的能力。具有产生此类异生物质的细菌通常含有双加氧酶或单加氧酶,例如萘双加氧酶[7, 8, 9, 10]、甲苯双加氧酶[11]、苯乙烯单加氧酶[12, 13]、黄素蛋白单加氧酶[14]和P450单加氧酶[15, 16]等。一般认为吲哚经过酶促氧化生成羟基吲哚,后者自发缩合生成靛蓝或靛玉红等产物 (图1)。通过蛋白质工程改造野生酶获得催化效率大幅提高的突变酶以及通过优化培养条件提高生产菌株合成靛蓝能力作为有效的策略应用于靛蓝生物合成的实践中[17]。
 |
| 图1 生物合成靛蓝和靛玉红 Fig.1 Biosynthesis of indigo and indirubin by indole oxidization pathway. |
| 图选项 |
Baeyer-Villiger单加氧酶 (Baeyer-Villiger monooxygenase,BVMO) 是一类含有黄素FAD、NADPH辅因子的重要生物酶催化剂,主要催化羰基类化合物氧化,在羰基和一个邻近烃基之间引入一个氧原子,得到相应的酯或内酯[18]。BVMO具有催化的不专一性,已报道这类酶的典型代表环己酮单加氧酶 (Cyclohexanone monooxygenase,CHMO) 还具有氧化硫、氮等杂原子的能力以及催化碳碳双键环氧化的能力等[19]。最新的研究发现苯丙酮单加氧单个氨基酸的改变导致突变酶拥有氧化酶活力[20]。NADPH是一种昂贵的辅因子,在以制备催化产物为目的的研究中,选择辅酶再生系统或以全细胞而非纯酶作为催化剂是一种经济、实用的策略[20]。
本研究旨在探索利用蛋白质工程改造PAMO,赋予突变酶氧化吲哚合成靛蓝及其衍生物的能力,在此基础上,通过代谢工程改造大肠杆菌的糖代谢途径,增加细胞内NADPH浓度,从而设计高效的全细胞催化剂用于合成靛蓝和靛玉红。
1 材料与方法 1.1 试剂 葡萄糖、氯化钠购自国药集团试剂有限公司。磷酸氢二钾、磷酸二氢钾购自北京化工厂。靛蓝标准品、吲哚标准品购自北京百灵威科技有限公司。质粒提取试剂盒购自天根生化科技 (北京) 有限公司。靛玉红标准品购自Aladdin。L(-)-Tryptophan购自Acros Organics。胰蛋白胨、酵母提取物购自Oxoid Company Ltd。KOD Taq DNA聚合酶购自日本Toyobo公司。DNA marker,Protein marker,DpnⅠ限制酶购自Fermentas。甲醇 (色谱纯) 购自Thermo Fisher Scientific。二氯甲烷购自Dikma Technologies Inc。二甲基亚砜 (DMSO) 购自Merck公司。薄层层析 (TLC) 板购自烟台江友硅胶开发有限公司。
TB培养基:用于培养大肠杆菌。胰蛋白胨12 g,酵母提取物24 g,甘油4 mL溶解于900 mL去离子水中,高压灭菌后冷却到60 ℃,再加入100 mL灭菌的磷酸缓冲液 (0.17 mol/L KH2PO4和0.72 mol/L K2HPO4)。
1.2 菌种与质粒 大肠杆菌Escherichia coli TOP10、E. coli BW25113 (Δpgi),以及质粒pBAD均为本实验室保存;E. coli TOP10 (Δpgi) 菌株利用P1噬菌体转导E. coli BW25113 (Δpgi) 筛选获得。 E. coli TOP10、E. coli TOP10 (Δpgi) 用于基因克隆和PAMO的表达,pBAD用于pamo基因 (No. AAZ55526) 的表达质粒构建。
1.3 方法 1.3.1 同源重组制备E. coli TOP10 (Δpgi) 菌株 1) 制备P1噬菌体溶菌产物[21, 22]:取E. coli BW25113 (Δpgi) 作为供体菌,在供体菌葡萄糖异构酶基因pgi中插入了卡那霉素抗性基因,从而导致pgi失活。接种E. coli BW25113 (Δpgi) 于含25 µg/mL卡那霉素抗性的LB液体培养基中,37 ℃、220 r/min培养过夜。取50 µL过夜培养物加入到5 mL补加有20%葡萄糖,25 µL浓度为1 mol/L的CaCl2的LB液体培养基中,然后加入100 µL 109−1010 PFU/mL P1噬菌体,继续培养直到细胞溶解,加入氯仿继续振荡几分钟,10 000 r/min、4 ℃离心10 min,取上清备用。
2) P1转导[21, 22]:取E. coli TOP10作为受体菌,接种于LB液体培养基中37 ℃、220 r/min培养过夜。取1.5 mL于室温下离心,弃上清。加入750 µL上述制备的P1溶菌产物重悬细胞,室温下噬菌体吸附30 min。加入终浓度为0.2 mol/L的柠檬酸钠1 mL。37 ℃培养1 h。取1.5 mL菌液离心,弃上清。用100 µL Tris-HCl缓冲液 (pH 8.0) 重悬细胞,涂布到含有25 µg/mL卡那霉素抗性平板上,培养过夜。次日挑取单菌落划线培养,连续5次传代后随机挑取克隆,备份并提取其基因组为模板,用pgi引物扩增,PCR产物经测序后证明其中含有完整的卡那抗性基因,从而获得E. coli TOP10 (Δpgi) 菌株。
1.3.2 M446位点饱和突变体的构建 引物设计如表1所示,用于构建Met446位点的饱和突变体,模板为pPAMO,PCR体系和程序见参考文献[18]。
所得PCR产物经DpnⅠ消化2.5 h,去除模板DNA。然后采用PCR产物纯化试剂盒纯化,纯化后的PCR产物用化学转化法分别转化入E. coli TOP10和E. coli TOP10 (Δpgi) 中,转化产物涂布于含有相应抗生素的LB固体培养基上 (转入TOP10表达体系时涂布于100 µg/mL氨苄青霉素抗性平板上;转入TOP10 (Δpgi) 表达体系时涂布于100 µg/mL氨苄青霉素和25 µg/mL卡那霉素抗性平板上),37 ℃过夜培养得到突变文库,随机挑取克隆,测序获得446位点的其他19种突变体,甘油保存备用。
1.3.3 细胞培养 10 µL甘油保存的菌体接到5 mL含100 µg/mL氨苄青霉素的LB液体培养基中培养过夜,1%转至200 mL TB培养基 (根据宿主菌不同加入相应抗生素),同时加入0.01%的L-阿拉伯糖作为诱导剂,37 ℃、220 r/min培养24 h。
1.3.4 细胞及产物处理 由于靛蓝为细胞内生产物,水溶性差,发酵上清液中含量甚微,随着培养时间的延长,产物的积累会在摇甁底部析出。因此培养24 h后,培养物经5 000 r/min、10 ℃离心7 min,弃上清,用15 mL Tris-HCl缓冲液 (50 mmol/L,pH 8.0) 重悬,然后在冰浴中用超声波破碎细胞60 min,工作条件:功率300 W,工作2 s,间歇2 s。经破碎后的悬液用二氯甲烷萃取3遍,合并萃取液,旋转蒸发仪浓缩后,用二甲基亚砜 (DMSO) 溶解制备样品备用。
表1 引物名称及序列Table 1 Primer name and primer sequence
| Primer name | Primer sequence (5¢-3¢) |
| M1 | CTCAGCAACNNKCTGGTCTCT |
| M2 | AGAGACCAGNNKGTTGCTGAG |
表选项
1.3.5 薄层层析分析方法 将上述处理好的样品稀释后用毛细管点样于硅胶薄层板上,展开剂为苯∶氯仿∶丙酮=5∶4∶1[23],溶剂前沿到达距薄层板顶端2 cm左右时取出,置通风橱里吹干,254 nm紫外灯下检测。
1.3.6 最大紫外吸收法定量 将靛蓝和靛玉红标准品置于分光光度仪(Spectrophotometer Beckman Counter DU800) 下进行200-800 nm全波长扫描,测得靛玉红的最大吸收在549 nm,靛蓝的最大吸收在618 nm。取靛蓝和靛玉红标准品配制成不同浓度梯度,绘制标准曲线。通过标准曲线法获得靛玉红和靛蓝在各自最大吸收波长下的消光系数分别为27.2和8.96 L/(mmol·cm)。根据朗伯比尔定律 (A=εcl) 对其产量进行定量。
1.3.7 高效液相法定性定量分析 采用Waters超高效液相色谱仪Acouity UPLC进行分析,反相色谱柱C18 (Symmetry,5 µm,4.6 mm×250 mm),流动相为甲醇/水,线性梯度60/40-70/30,流速为0.8 mL/min。取靛蓝和靛玉红标准品配制成不同浓度梯度,绘制标准曲线,将测得的样品数值代入回归方程,确定其浓度。
2 结果与讨论 2.1 靛蓝和靛玉红的鉴定 2.1.1 薄层层析初步鉴定化合物 在前期的研究中,我们发现非保守的Hinge结构对PAMO的催化活性和立体选择性有非常大的影响[24]。在进一步研究PAMO 底物结合口袋周围氨基酸残基对酶催化行为影响时,通过构建丙氨酸扫描突变体 (L153A、Q152A、S196A、W501A、M446A、D66A、M515A等),测试突变对酶催化活力、对映选择性等的影响时,意外发现M446A突变体在大肠杆菌TOP10细胞表达体系培养过程中发酵液变成了浅蓝色,萃取后通过薄层层析的方法初步鉴定该化合物。发现在蓝色斑点的下方出现了玫红色的斑点,结合文献报道[25, 26],以靛蓝、靛玉红标准品作为对照,薄层板上Rf值基本一致,初步确定这种物质是靛蓝和靛玉红的混合物 (图2)。
 |
| 图2 薄层层析分析靛蓝和靛玉红 Fig.2 Analysis of indigo and indirubin by thin-layer chromatography. Lane 1 is the sample,M1 and M2 are indigo standard and indirubin standard,respectively. |
| 图选项 |
2.1.2 高效液相色谱法和质谱分析产物 结合标准品,HPLC和MS分析发现浅蓝色的培养物中含有吲哚、靛蓝、靛玉红和靛红 (表2)。靛红是靛蓝和靛玉红的降解产物。体外稳定性分析表明靛蓝在室温下放置非常不稳定,很容易分解,靛玉红相对比较稳定。
2.1.3 靛蓝和靛玉红的产量随时间变化趋势 靛蓝和靛玉红互为同分异构体,靛蓝是由两分子3-羟基吲哚聚合而成,靛玉红是由一分子2-羟基吲哚和一分子3-羟基吲哚聚合而成。HPLC分析未检测到2-羟基吲哚和3-羟基吲哚,可能是含量非常低的原因。为了研究E. coli TOP10 (pPAMO) 突变体细胞M446A生长过程与靛蓝和靛玉红生物合成之间的相互关系,取同批次发酵6、12、18、24、36、42 h发酵液,萃取处理后,测定靛蓝和靛玉红的生成量。据图3曲线所示,靛蓝在发酵6 h左右开始出现,随着发酵时间延长,靛蓝的产量逐渐增加,当发酵24 h左右产量达到最大 (1.3 mg/L),继续培养靛蓝的产量反而下降。靛玉红的出现比靛蓝晚大约6 h,在发酵12 h后开始出现,随着培养时间的延长,靛玉红的产量增加,但当发酵36 h时,产量最高 (0.3 mg/L),继续培养产量也出现下降的现象,相对靛蓝,靛玉红的分解趋势明显偏低,表明其相对靛蓝较为稳定,这与前面体外实验结果一致。
 |
| 图3 靛蓝和靛玉红的生成量随时间变化的曲线 Fig.3 The production profile of indigo and indirubin during the growth process of E. coli TOP10 (pPAMO) muant M446A. |
| 图选项 |
表2 M446A培养物中的产物分析Table 2 Major compounds in the culture of M446A mutant
| Compounds | Structure | Retention time (min) | m/z |
| Indole | 16.4 | 118 | |
| Isatin | 6.6 | 148 | |
| Indigo | 24.3 | 263 | |
| Indirubin | 42.8 | 263 |
表选项
2.2 构建饱和突变体 以前的文献报道中提到Met446Gly突变酶具有合成靛蓝的能力[19]。我们的实验结果表明446位点Met突变为Ala不仅赋予了PAMO氧化吲哚合成靛蓝的能力,同时也具有合成靛玉红的能力。为了探索该位点其他氨基酸的存在如何影响酶催化氧化吲哚的效力以及形成最终缩合产物的差异,通过饱和突变的方法构建了该位点的其他19种氨基酸突变体,转化大肠杆菌Top10,培养24 h后,测定生成的靛蓝和靛玉红产量。靛蓝和靛玉红的最大吸收波长分别为618 nm和549 nm,在最大吸收波长下,二者没有干扰。紫外-可见光分光光度法较HPLC用于定量分析更为简便快捷。利用标准品测试结果表明,两种方法所检测的结果是一致的。所以在实际的定量检测中,选择紫外-可见光分光光度法测定靛蓝和靛玉红的含量。定量实验结果显示M446A、M446G活性较高。M446C和M446S也有微弱的活性,其他突变体活性很低或者基本没有活性,各突变体产生靛蓝和靛玉红的活性对比汇总见图4。Met446是底物结合口袋的组成单元,但并不直接与底物发生相互作用。甲硫氨酸变成分子较小的丙氨酸或甘氨酸等后,使得底物结合口袋构象发生变化,能够使吲哚以一种新的方式进入到底物口袋,其吡咯环中C2-C3之间的双键更容易受到FAD C4α部位形成的过氧中间体攻击,从而发生反应,最终形成2-位和3-位发生氧化的产物。2-羟基吲哚和3-羟基吲哚通过分子间缩合反应聚合形成靛玉红,两分子3-羟基吲哚聚合形成靛蓝,目前普遍认为此缩合反应是自发进行的。
 |
| 图4 PAMO野生型与M446位点突变体生成靛蓝和靛玉红活性对比 Fig.4 Comparison of the productivity of indigo and indirubin producing by PAMO-WT and M446 mutants. |
| 图选项 |
2.3 补加前体物质提高靛蓝和靛玉红的产量 尽管PAMO的突变体可以合成靛蓝和靛玉红,但产量并不高,每升培养液可以生成总量约2.0 mg的靛蓝和靛玉红,效率很低。因为吲哚是合成靛蓝和靛玉红的前体物质,为了提高生成效率,尝试在培养基中直接添加不同浓度的吲哚,发现没有明显地促进靛蓝和靛玉红产量增加的作用,实际上过量的吲哚加入导致细胞生长受到抑制。这可能与吲哚的低溶解度和对细胞的毒性有关[26]。靛蓝和靛玉红合成的前体物质是吲哚,细胞中吲哚主要来自于色氨酸的分解代谢。色氨酸酶在色氨酸分解代谢中扮演着主要的角色,大肠杆菌的色氨酸酶的转录非常活跃,这与TB培养基中色氨酸的含量比较丰富有关。既然直接添加吲哚无益于靛蓝和靛玉红的生物合成,因此尝试加入吲哚的前体物质色氨酸作为补料物质。在细胞培养过程中直接添加色氨酸,并设置不同的浓度梯度,实验结果表明色氨酸的最佳添加量4 mmol/L。由表3可见,在一定范围内随着色氨酸浓度的提高,细胞产生靛蓝和靛玉红的能力也加强,当色氨酸的终浓度达到4 mmol/L,产量最高。进一步增加色氨酸终浓度时,可能是由于吲哚的积累,抑制了细胞的活性[26]。实验结果表明补加4 mmol/L色氨酸,可以使细胞合成靛蓝和靛玉红的能力提高4.5倍,达到8.43 mg/L。
表3 色氨酸对生物合成靛蓝和靛玉红产量的影响Table 3 Effect of tryptophan on the production of indigo and indirubin
| L-trytophan (mmol/L) | Indigo (mg/L) | Indirubin (mg/L) | Yield of indigo and indirubin (mg/L) |
| 0 | 1.51 | 0.38 | 1.89 |
| 1 | 4.31 | 1.22 | 5.54 |
| 2 | 5.16 | 1.47 | 6.62 |
| 3 | 5.78 | 1.87 | 7.66 |
| 4 | 6.28 | 2.15 | 8.43 |
| 5 | 5.58 | 1.57 | 7.16 |
| 6 | 4.47 | 1.49 | 5.97 |
表选项
2.4 利用代谢工程改造宿主菌提高靛蓝和靛玉红的产量 PAMO是一种依赖NADPH的单加氧酶,经NADPH还原后的FAD能够与氧气分子发生反应,生成过氧化的FAD中间体,该中间体具有非常高的氧化活性[27]。NADPH即是一种辅因子,也是BVMO氧化反应的一种底物,因此推测如果能够提高细胞内NADPH的水平则有可能提高细胞氧化吲哚的能力,从而提高生物合成靛蓝和靛玉红的产量。细胞内NADPH的主要来源是葡萄糖代谢,它不仅能提供能源,参与某些酶促反应,还能提供物质合成所必需的还原能力。因此我们想到了通过改变代谢途径来积累细胞内NADPH含量的方法。
糖酵解途径是绝大多数生物所共有的一条主流代谢途径。一分子葡萄糖为底物,经耗能和产能两阶段产生两分子丙酮酸、两分子NADH和两分子ATP的生化反应过程。磷酸戊糖途径是葡萄糖氧化分解的另一种方式。由于此途径是由6-磷酸葡萄糖开始,最终生成的 3-磷酸甘油醛和6-磷酸果糖重新进入糖酵解途径,故也称为己糖磷酸旁路,该途径是细胞内NADPH的主要来源 (图5)。
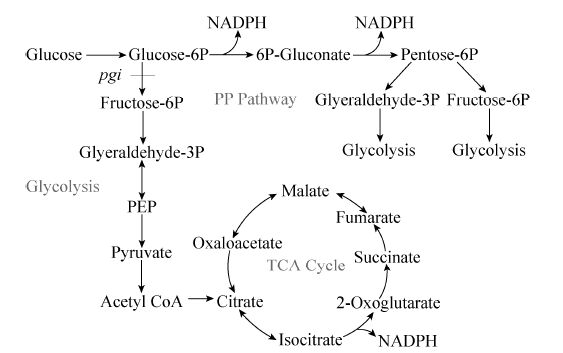 |
| 图5 大肠杆菌代谢葡萄糖途径简图 Fig.5 Glucose metabolism pathway in E. coli. E. coli utilizes glucose to produce cofactor NADH through glycolysis pathway,and produce NADPH by pentose phosphate pathway (PP) pathway. Metabolizing one molecule of glucose by PP pathway yields two molecule of NADPH. |
| 图选项 |
通过同源重组的方法阻断糖酵解途径的葡萄糖异构酶基因pgi,阻断糖酵解途径使细胞内糖的代谢以磷酸戊糖途径为主流代谢途径,后者是细胞合成NADPH的主要来源。结果表明经过代谢工程改造后大肠杆菌TOP10 (Δpgi) 作为宿主菌,同时在发酵培养基中添加终浓度为 4 mmol/L的色氨酸,合成靛蓝和靛玉红的能力得到进一步的提升,达到25 mg/L。对宿主菌的代谢工程改造,使得全细胞合成靛蓝和靛玉红的能力进一步提高3倍。
综上所述,蛋白质工程改造赋予PAMO全新的催化活力,氧化吲哚合成靛蓝和靛玉红。在此基础上,在培养过程中补加前体物质色氨酸以及通过代谢工程改造宿主菌都大幅度地提高了全细胞生物合成靛蓝和靛玉红的能力。图6展示了PAMO突变体M446A发酵瓶 (6A) 与补加4 mmol/L色氨酸发酵瓶 (6B) 以及补加4 mmol/L色氨酸同时使用代谢工程改造后宿主菌发酵瓶 (6C) 效果对比图,发酵液颜色逐渐变深,直观地表明靛蓝和靛玉红的产量变化趋势。
 |
| 图6 M446A在不同条件下;发酵培养基颜色对比 Fig.6 Color comparison of the mutant M446A fermentation medium under different conditions. (A) General M446A fermentation bottle. (B) Fermentation bottle adding 4 mmol/L L-tryptophan. (C) Fermentation bottle adding 4 mmol/L L-tryptophan as well as using genetically modified E. coli host. |
| 图选项 |
3 结论 蛋白质工程是一种用于研究蛋白质构效关系非常有效的策略,如改变酶已有的各种催化行为,赋予酶新的催化活力等。本研究所用的苯基丙酮单加氧酶PAMO主要功能是催化羰基化合物氧化,活化C-C键,在此我们展示了通过工程化改造其底物结合口袋,发现Met446位点氨基酸残基的改变赋予酶C-H键氧化活力,催化吲哚生成靛蓝和靛玉红。为了进一步提高全细胞合成靛蓝和靛玉红的生成效率,在蛋白质工程改造获得新酶活的基础上,组合使用了前体补加以及对宿主菌进行代谢工程改造的研究策略,设计出一种全细胞催化剂可以高效地合成靛蓝和靛玉红,每升发酵液可以生成25 mg产物。这种多策略组合使用设计高效全细胞催化剂的理念为全细胞催化剂的开发研究提供了一种新的思路。有关苯基丙酮单加氧酶催化氧化C—H键活化的分子机制研究正在进行中。
参考文献
| [1] | Hoessel R, Leclerc S, Endicott JA, et al. Indirubin, the active constituent of a Chinese antileukaemia medicine, inhibits cyclin-dependent kinases. Nat Cell Biol, 1999, 1(1): 60-67. |
| [2] | Leclerc S, Garnier M, Hoessel R, et al. Indirubins inhibit glycogen synthasekinase-3β and CDK5/P25, two protein kinases involved in abnormal tau phosphorylation in Alzheimer's disease. J Biol Chem, 2001, 276(1): 251-260. |
| [3] | Xiao Z, Hao Y, Liu B, et al. Indirubin and meisoindigo in the treatment of chronic myelogenous leukemia in China. Leu Lymphoma, 2002, 43(1): 1763-1768. |
| [4] | Kunikata T, Tatefuji T, Aga H, et al. Indirubin inhibits inflammatory reactions in delayed-type hypersensitivity. Eur J Pharmacol, 2000, 410(1): 93-100. |
| [5] | Ma Q, Qu YY, Zhang XW, et al. Recent advances in microbial synthesis of indigo. Chin J Appl Environ Biol, 2012, 18(2): 344-350 in Chinese). 马桥, 曲媛媛, 张旭旺, 等. 靛蓝的微生物合成研究新进展. 应用与环境生物学报, 2012, 18(2): 344-350. |
| [6] | Gray PHH. The formation of indigotin from indole by soil bacteria. Proc R Soc Lond B Biol Sci, 1928, 102: 263-280. |
| [7] | Ensley BD, Ratzkin BJ, Osslund TD, et al. Expression of naphthalene oxidation genes in Escherichia coli results in the biosynthesis of indigo. Science, 1983, 222(4620): 167-169. |
| [8] | Bhushan B, Samanta SK, Jain RK. Indigo production by naphthalene-degrading bacteria. Lett Appl Microbiol, 2000, 31: 5-9. |
| [9] | Pathak H, Madamwar D. Biosythesis of indigo dye by newly isolated naphthalene-degrading strain Pseudomonas sp. HOB1 and its application in dyeing cotton fabric. Appl Biochem Biotechnol, 2010, 160(6): 1616-1626. |
| [10] | Mercadal JPR, Isaac P, Sineriz F, et al. Indigo production by Pseudomonas sp. J26, a marine naphthalene-degrading strain. J Basic Microbiol, 2010, 50: 290-293. |
| [11] | O’Connor KE, Hartmans S. Indigo formation by aromatic hydrocarbon-degarding bacteria. Biotechnol Lett, 1998, 20(3): 219-223. |
| [12] | O’Connor KE, Dobson ADW, Hartmans S. Indigo formation by microorganisms expressing styrene monooxygenase activity. Appl Environ Microbiol, 1997, 63(11): 4287-4291. |
| [13] | Marconi AM, Beltrametti F, Bestetti G. et al. Cloning and characterization of styrene catabolism genes from Pseudomonas fluorescens ST. Appl Environ Microbiol, 1996, 62(1): 121-127. |
| [14] | Choi HS, Kim JK, Cho EH. A novel flain- containing monooxygenase from Methylophaga sp. strain SK1 and its indigo synthesis in Escherichia coli. Biochem Biophys Res Commun, 2003, 306: 930-936. |
| [15] | Gillam EMJ, Aguinaldo AMA, Notley LM. Formation of indigo by recombinant mammalian cytochrome P450. Biochem Biophys Res Commun, 1999, 265: 469-472. |
| [16] | Zhang PP, Hu S, Mei LH, et al. A triple mutant improving activity of cytochrome P450 BM-3 to catalyze indole hydroxylation: D168L/E435T/ V445A CIESC J, 2014, 68(4): 1374-1380 in Chinese). 张彭湃, 胡升, 梅乐和, 等. 一个有利于细胞色素P450 BM-3催化吲哚合成靛蓝的三位点突变酶: D168L/E435T/V445A. 化工学报, 2014, 68(4): 1374-1380. |
| [17] | Rui L, Reardon KF, Wood TK. Protein engineering of toluene ortho-monooxygenase of Burkholderia cepacia G4 for regiospecific hydroxylation of indole to form various indigoid compounds. Appl Microbiol Biotechnol, 2005, 66(4): 422-429. |
| [18] | Reetz MT, Wu S. Laboratory evolution of robust and enantioselective Baeyer-Villiger monooxygenases for asymmetric catalysis. J Am Chem Soc, 2009, 131(42): 15424-15432. |
| [19] | Pazmiño DET, Snajdrova R, Rial DV, et al. Altering the substrate specificity and enantioselectivity of phenylacetone monooxygenase by structure-inspired enzyme redesign. Adv Synth Catal, 2007, 349(8/9): 1361-1368. |
| [20] | Brondani PB, Dudek HM, Martinoli C, et al. Finding the switch: turning a Baeyer-Villiger monooxygenase into a NADPH oxidase. J Am Chem Soc, 2014, 136: 16966-16969. |
| [21] | E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology.03]. http://www.ncbi.nlm.nih.gov/pubmed/ [22] Kim S, Lee CH, Nam SW, et al. Alteration of reducing powers in an isogenic phosphoglucose isomerase(pgi) disrupted Escherichia coli expressing NADP-dependent malic enzymes and NADP-dependent glyceraldehyde-3-phosphate dehydrogenase. Lett Appl Microbiol, 2011, 52(5): 433-440. [ |
| [23] | Mercadal JPR, Isaac P, Siñeriz F, et al. Indigo production by Pseudomonas sp. J26, a marine naphthalene-degrading strain. J Basic Microbiol, 2010, 50(3): 290-293. |
| [24] | Liang QL, Wu S. Nonconserved hinge in Baeyer-Villiger monooxygenase affects catalytic activity and stereoselectivity. Chin J Biotech, 2015, 31(3): 361-374 in Chinese). 梁秋玲, 吴胜. Baeyer-Villiger单加氧酶非保守Hinge影响酶的催化活性和立体选择性. 生物工程学报, 2015, 31(3): 361-374. |
| [25] | Riebel A, Dudek HM, de Gonzalo G, et al. Expanding the set of rhodococcal Baeyer-Villiger monooxygenases by high-throughput cloning, expression and substrate screening. Appl Microbiol Biotechnol, 2012, 95: 1479-1489. |
| [26] | Doukyu N, Aono R. Biodegradation of indole at high concentration by persolvent fermentation with Pseudomonas sp. ST-200. Extremophiles, 1997, 1(2): 100-105. |
| [27] | Torres Pazmiño DE, Dudek HM, Fraaije MW. Baeyer-Villiger monooxygenases: recent advances and future challenges. Curr Opin Chem Biol, 2010, 14(2): 138-144. |
