刘婉1, 严兴2

 , 沈潇2, 张国庆1, 周志华2
, 沈潇2, 张国庆1, 周志华2 1. 中国科学院计算生物学重点实验室, 生物医学大数据中心, 中国科学院上海营养与健康研究所, 上海 200031;
2. 中国科学院分子植物科学卓越创新中心, 合成生物学重点实验室, 上海 200032
收稿日期:2021-02-13;修回日期:2021-04-15;网络出版日期:2021-09-28
基金项目:中国科学院战略生物资源服务网络计划生物资源衍生库建设项目(KFJ-BRP-009);国家科技部科技基础资源调查专项(2019FY100100)
作者简介:刘婉,女,博士,中国科学院上海营养与健康研究所生物医学大数据中心中国科学院计算生物学重点实验室工程师,主要研究方向为合成生物学数据库与数据仓库、微生物相关数据库与数据仓库。参与了国家重点研发计划、中国科学院国际合作局对外合作重点项目、中国科学院科技服务网络计划、上海张江国家自主创新示范区专项发展资金重大项目计划和中国科学院重点部署项目等多个项目,在国际学术期刊和国内核心学报期刊发表相关论文7篇,其中SCI期刊论文5篇.
*通信作者:严兴, Tel: +86-21-54924049;Fax: +86-21-54924050;E-mail: yanxing@cemps.ac.cn.
摘要:https://www.biosino.org/rdbsb/or https://www.biosino.org/npbiosys/)。在建设过程中,我们首先建立了与SBOL语言兼容的《催化元件数据标准》、多种重要元件的标准功能测试方法以及安全高效的元件提交系统,并在此基础上完成了元件数量最多且信息最为全面的催化元件数据库的构建。目前,收集了30多万个催化元件,包括7万多个具有文献支持功能表征信息的催化元件,并保藏了1万个以上的实物元件和5000个底盘菌株,通过网站实现了数据和实物的公开与共享。上述元件库已经对国内合成生物学的研究和应用产生了积极地推动作用,访问量超过100万次/年,提供元件和底盘共享服务大于100次/年,并为多家企业和研究单位提供了元件和底盘的定制服务。虽然我们的元件库建设取得了巨大的进步,但是仍需在调控元件的收集整理和共享机制创新等方面取得更多的突破,在数据有源、多层审核、资源共享、信息公开、信息安全和授权访问的基础上,加速生物元件数据、实物和设计工具的汇聚,并进一步服务合成生物学研究。]]>
关键词:元件库合成生物学共享机制催化元件调控元件
Research progress of biopart databases
Wan Liu1, Xing Yan2

 , Xiao Shen2, Guoqing Zhang1, Zhihua Zhou2
, Xiao Shen2, Guoqing Zhang1, Zhihua Zhou2 1. CAS Key Laboratory of Computational Biology, Bio-Med Big Data Centre, Shanghai Institute of Nutrition and Health, Chinese Academy of Sciences, Shanghai 200031, China;
2. CAS Key Laboratory of Synthetic Biology, CAS Centre for Excellence in Molecular Plant Sciences, Chinese Academy of Sciences, Shanghai 200032, China
Received: 13 February 2021; Revised: 15 April 2021; Published online: 28 September 2021
*Corresponding author: Xing Yan, Tel: +86-21-54924049; Fax: +86-21-54924050; E-mail: yanxing@cemps.ac.cn.
Foundation item: Supported by the Biological Resources Programme, Chinese Academy of Sciences (KFJ-BRP-009) and by the National Science Resource Investigation Program of China (2019FY100100)
Abstract: https://www.biosino.org/rdbsb/or https://www.biosino.org/npbiosys/) by working together with other research institutes. This biopart dabase has been established based on a "data standard for catalytic bioparts" system compatible with SBOL language, and a series of developed standard functional test methods for different sorts of bioparts as well as a set up safe and efficient submission system for bioparts. Until now, RDBSB has collected more than 300000 catalytic bioparts, of which more than 70000 ones have been described with their functional characterization supported by Scientific literatures preserved more than 10000 physical bioparts and 5000 chassis strains, and set up an efficient sharing system to distribute the data and physical bioparts through its website. In the past two years, RDBSB has attracted one million visits per year, and provided more than 100 Services per year to build customized bioparts or chassis strains to researchers or the customized bio-technologic services of build bioparts or chassis for companies and scientific research units per year, which demonstrated its facilitation in the research and application of synthetic biology. However, more breakthroughs are still required in the collection and investigation of regulatory bioparts and innovation of mechanisms for sharing data, metadata physical bioparts and related tools. We are looking forward to building a database with convergence of data, metadata physical objects and design tools to better serve the synthetic biology research basing on a basic regulation including biopart data sourcing, multi-layer reviewing, resource sharing, biopart data disclosure, secured biopart data and authorized access.]]>
Keywords: biopart databasessynthetic biologysharing mechanismcatalytic partsregulatory parts
1 建设生物元件库的重大意义 生物元件是合成生物学的基石,生物元件的设计组装及生物元件库的建设对于合成生物学的工程化非常必要和重要。Brothers等在2010年阐述合成生物学所面临的五大挑战时,明确指出生物元件种类匮乏、大部分生物元件的表征描述不准确以及人工系统中元件之间或元件与人工系统间的不适配性是发展合成生物学必须扫除的障碍[1]。
由于生命体系的高度动态、灵活调控、非线性且难以预测的特点,作为其最基本单元的生物元件的序列结构与功能也呈现高度的多样性。如何获得功能表征明确的最适生物元件来构建目标人工生命系统,是合成生物学的核心科学问题之一。通过收集、整理和保藏各类生物元件的实物及定性与定量功能表征信息构建生物元件库,可以实现元件信息和实物的充分共享,从而大幅度提高研究者利用合适的生物元件对生命系统或过程进行重设计和工程化构建的效率。
“十三五”期间科技部启动“合成生物学”和“绿色生物制造”等专项的许多项目都涉及生物元件的挖掘、设计与构建,但这些生物元件数据、元数据和实物都保存在各自的项目或单位,通过加强生物元件资源平台的建设,对生物元件相关的研究成果进行汇聚和分享,将会进一步推动我国合成生物学的发展。本文将对国内外常见的生物元件库(表 1)的研究进展进行综述,总结国内外元件库研究和建设的现状,为国内生物元件库的建设和发展提供可借鉴的经验,促进国内元件库的发展建设,更好地服务于合成生物学研究。
表 1. 常见的生物元件库 Table 1. Overview of biopart databases
| Database and access URL | Creation time | Country | Number of biopart | Functional characterization | Data download format | Data submission | Security | Online analysis | Tools for online visualization | Data format for biopart exchange |
| Inventory of Composable Elements (https://public-registry.jbei.org/f) | 2007 | USA | 5145a | N/A | csv, csv+sequence, custom | Yes | Public, registration required | Yes | Yes | GenBank, Fasta, GFF3, SBOL1, SBOL2 |
| Registry of Standard Biological Parts (http://parts.igem.org/Catalog) | 2003 | USA | 22019b | Fulltext description | Genbank, SBOL | Yes | Public | Yes | Yes | GenBank, SBOL |
| BioBricks Foundation (https://biobricks.org/bpa/find-a-part/https://stanford.freegenes.org/) | 2006 | USA | 2315 | Fulltext description in Freegenes | api, csv | Yes | Public | No | No | N/A |
| SynbioML (http://www.synbioml.org/) | 2009 | China | 5848 | Structured qualitative functional characterization | Yes | Public | No | No | N/A | |
| Registry and database of bioparts for synthetic biology(https://www.biosino.org/rdbsb/) | 2016 | China | 366041 | Structured reaction, qualitative and quantitative functional characterization | N/A | Yes | Public | Yes | Yes | json |
| Data collection upto December, 2020; a: including biopart and plasmid collected from ACS Registry, JBEI Registry, JGI Registry, Synberc Registry and Agile BioFoundry Registry; b: including both biopart and device. | ||||||||||
表选项
2 国外生物元件库研究进展 2.1 标准生物元件登记库 美国麻省理工学院2003年创建了国际遗传工程机器大赛(iGEM),建立了供参赛者进行作品提交的“标准生物元件登记库” (http://parts.igem.org/Main_Page),这是合成生物学领域最为著名的元件库。目前标准生物元件登记库收集了超过20000种生物元件(http://parts.igem.org/Collections),其包含有功能表征信息的元件15000个左右,既包括启动子、转录单元、质粒骨架、接合转移元件、转座子、蛋白质编码区等DNA序列,也包括核糖体结合位点、终止子以及一些蛋白质结构域。iGEM不仅将iGEM参赛者和iGEM加盟实验室提交的所有生物元件数据和元数据进行共享,全球的学术研究机构也可以通过申请加入iGEM实验室而获得标准生物元件登记库的元件实物(https://igem.org/Labs_Program)。
2.2 生物积块基金会 2006年创建的“生物积块基金会”(BioBricks Foundation,http://www.biobricks.org/)是由许多参与iGEM基金会的人员创立。相对于iGEM,其受众更广泛、操作更规范、愿景更宏大,致力于推动合成生物学在生物医药、生物材料、环境保护和农业等技术领域的发展[2]。生物积块基金会致力于元件的公开共享与免费获取,降低元件的分发成本,在生物元件共享机制和相关协议制定方面做了大量的工作。首先主导制定了生物积块公共协议(The BioBrickTM Public Agreement,BPA),为元件的共享提供了一个简便易行的法律框架,研究者一旦签署BPA,就可以免费从生物积块基金会获得生物元件。随后,生物积块基金会又主导制定了开放生物材料转移协定(OpenMTA)[3],允许元件的获取者在所签署的OpenMTA协议框架内,对生物元件进行再次分发并对元件进行修饰、改造甚至商业化,从而进一步促进生物元件的开发、共享和利用。生物积块基金会还与Twist Bioscience合作开展Free genes的1万个基因元件合成项目由研究人员提出基因元件合成申请,然在OpenMTA协议下免费获得这些元件实物。目前,通过该项目已经实现了2000多个基因元件的免费共享。生物积块基金会还尝试通过bionet项目实现元件实物、数据和元数据的去中心化管理,使研究人员可以通过bionet系统在全球分享自己开发的实物元件及相关信息并在本地管理这些信息,决定哪些元件实物、数据和元数据可以共享,同时研究人员也可以通过bionet系统查找自己感兴趣的元件,通过签署OpenMTA协议实现实物元件的分发与获取。
2.3 Inventory of Composable Elements 联合生物能源研究所(JBEI,Joint BioEnergy Institute)是美国能源部(DOE,U.S. Department of Energy)的生物能源研究中心,致力于开发先进的生物燃料,从植物生物质储存的太阳能中提取液体燃料替代汽油、柴油和喷气式燃料。其开发的ICE (Inventory of Composable Elements)平台,是一个开放源码的生物部件信息管理平台。ICE建立在网络注册的理念之上,为生物元件的分发、互联式使用提供了强有力的支持,并且提供序列注释、操作和分析的图形应用程序,各个实验室也可以用它来跟踪和搜索其构建的生物元件。该元件库的元件来源包括ACS Registry、JBEI Registry、JGI Registry、Synberc Registry和Agile BioFoundry Registry等多个项目的数据汇交。除了元件之外,联合生物能源研究所ICE平台也收集微生物菌株、质粒、拟南芥种子和蛋白等其他生物部件。
2.4 国际合成生物学开放语言 合成生物学开放语言(SBOL,Synthetic Biology Open Language,https://sbolstandard.org/)用于描述生物元件的结构和基本的定性功能表征信息,是目前合成生物学领域进行元件设计、数据交换的标准语言[4-6]。SBOL是根据合成生物学的需求设计的、社区驱动的、标准化的,可以用于软件工具、研究小组和商业服务提供商之间元件设计的交流语言。SBOL开发者不仅提供了XML/RDF数据格式,而且提供软件库和规范文档帮助开发者在自己的软件中使用SBOL[7]。SBOL还可以对系统生物学标记语言(SBML,systems biology markup language)进行注释,也可以将数据从SBML格式转成SBOL的格式,将生物系统设计和元件序列结构信息进行关联[8-9]。目前,包括联合生物能源研究所ICE和标准生物元件登记库等都提供了SBOL数据格式供数据标准化共享[10]。
3 国内生物元件库研究进展 3.1 合成生物学元件与数据库 2016年以来,由中国科学院合成生物学重点实验室牵头,联合中国科学院计算生物学重点实验室生物医学大数据中心、中国科学院微生物所和中国科学院天津工业生物技术研究所,共同建设国内第一个合成生物学元件库(合成生物学元件与数据库,Registry and database of bioparts for synthetic biology,以下简称“元件库”),包括相应的数据库和实物库。
3.1.1 数据整合及审编: 在数据库建设方面,元件库对国际上主流的序列功能公共数据库的(MetaCyc、Brenda、KEGG、Uniprot和Rhea)进行了充分调研,并结合催化元件自身的特点制定了《催化元件数据标准》。根据这一数据标准对上述公共数据库的序列功能数据进行筛选整合获得了30多万个催化元件信息,再通过NLP识别和人工审编处理,针对包括pH、温度、元件与底盘相容性等信息进行审编以进一步提高元件数据质量,获得7万多个具有表征信息且有文献/实验支持的催化元件,所有元件信息均已在元件库网站上(Registry and database of bioparts for synthetic biology,https://www.biosino.org/rdbsb/)公开共享,并可以进行元件信息的交互查询,以便用于合成途径的初步设计。目前,元件库的催化元件数量已经远远超过标准生物元件登记库等国外元件库,即使与传统的酶学数据库Brenda相比, 元件库催化元件数量要也比Brenda数据库中酶的数量多1倍。元件库网站每年的访问量已经达到100万次以上并还在不断上升,服务用户已经达到2万名以上。到目前为止,为国内10个省市以及美国、英国、日本和法国等4个国家和地区的100多家研究单位分发元件和底盘200以上,其中单染色体酵母系列底盘已经向5个国家的35个实验室进行分发。为云南、浙江、上海、辽宁的多家企业定制元件与细胞工厂15次以上。在元件库平台支持下,利用元件库的底盘细胞和元件资源,实现了包括人参皂苷Rh2、黄芪素和5α-羟基紫杉二烯等多种天然产物的高效合成[11-13]。通过天然产物细胞工厂定制,向多个企业和研究单位提供了包括原人参二醇、原人参三醇、人参皂苷Rh2和Rg1等克级以上的通过合成生物学方法制备的天然产物。元件库已经作为国家重点研发计划合成生物学专项“生物元件标准化设计组装与应用研究”项目以及“大科学计划培育专项项目-面向天然产物生物合成的人工细胞”等项目的元件汇交与共享平台,为元件数据、元数据和实物提交以及设计工具集成提供服务。
3.1.2 数据汇交: 建立安全高效的生物元件数据提交系统,不仅可以改变元件数据被动收集的模式,而且还可以解决元件数据收录迟缓的问题。标准生物元件登记库等国外元件数据库均有自己独立的元件提交系统,元件库基于《催化元件数据标准》以及前期在调控元件收集方面的工作积累,完成了元件数据提交系统的设计。本提交系统采用线上(https://www.biosino.org/npbiosys/submit)和线下两种方式进行(图 1),并对数据进行分级管理。截至2020年12月,已经汇交了上百个重要元件数据,并服务于Nature Protocols、Scientific Reports和Science Bulletin等杂志的SCI论文的元件和底盘数据、元数据及实物的提交工作。
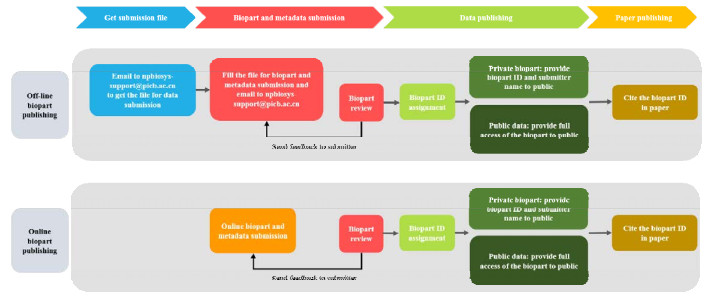 |
| 图 1 合成生物学元件与数据库数据提交流程 Figure 1 Workflow of the data submission to Registry and database of bioparts for synthetic biology. |
| 图选项 |
3.1.3 元件实物收集: 在元件实物收集方面,元件库首先对中国科学院合成生物学重点实验室在过去几十年研究中积累的元件和底盘进行了收集和整理,目前已经收集了1万个实物元件以及5000个底盘细胞的实物资源。元件实物资源,包括糖基转移酶、糖基水解酶、甲基转移酶、异戊烯基转移酶、P450和萜合酶等多种类型催化元件实物资源,以及用于大肠杆菌和放线菌等细菌、酵母和丝状真菌进行基因组编辑的多套工具元件。此外元件库还构建了品种较为齐全的底盘细胞库。例如,在染色体功能研究方面具有非常广泛用途的酵母单染色体底盘,包括线性单染色体酵母底盘和环形染色体酵母底盘。此外,还有多种天然产物合成相关的底盘细胞,例如:萜类合成底盘细胞,包括三萜、二萜和倍半萜底盘细胞。其中,三萜类底盘包括通用型的三萜底盘细胞以及特定三萜骨架的物底盘细胞,其中原人参二醇和原人参三醇酵母底盘细胞的合成效率都居于国际领先水平[11]。黄酮类化合物底盘细胞,包括柚皮素和山萘酚等较为通用的黄酮类化合物底盘细胞。生物碱底盘细胞,包括托品烷生物碱N-甲基吡咯啉底盘细胞[14-15]。而在实物元件和底盘分发方面,元件库暂时没有采用OpenMTA协议,而是参照Addgene的模式,制定了元件提供者、元件接收者以及元件库三方参与的材料转移协议(MTA),其中,元件提供者拥有元件的知识产权,并由元件提供者和元件接受者协商彼此的权利义务并对MTA模板进行修改,元件库仅起到元件存储和分发的作用,元件接受者需要向元件库缴纳一定合理的费用用于元件的存储和分发。与Addgene不同的是,元件库不仅分发质粒形式的元件而且也分发底盘菌株。
3.2 糖基转移酶数据库 糖基转移酶数据库(Glycosyltransferases Database,https://www.biosino.org/gtdb/,以下简称“GTDB”)是元件库的特色子库,收录了来自21647个分类的520179种糖基转移酶和394种酶学反应,包含了蛋白质分类家族、催化反应和代谢途径等信息,是糖基转移酶注释的综合资源。GTDB的数据是通过CAZy、NCBI Protein、NCBI Nucleotide、NCBI Gene、NCBI PubMed、NCBI Taxonomy、RCSB PDB、UniProt、Metacyc、KEGG等10个数据库的数据整合及eggNOG-mapper序列注释获得的。GTDB还提供了强大的搜索功能、BLAST工具、交互式浏览器和GTdock等工具,便于用户对糖基转移酶进行检索、注释、数据可视化和酶反应对接模拟[16]。
3.3 SynbioML 国内天津大学建立的SynbioML (http://www.synbioml.org/)也存储了近6000个催化元件和模块以及400多个底盘数据和实物并提供公开共享服务(表 1)。该数据库的元件模块数据具有结构化的元件定性表征信息,但是缺少相应的定量信息以及元件与底盘之间的适配信息。该元件库从功能上将元件分为初级代谢模块、产物模块和其他模块三大模块,但是没有搭建元件的检索工具。该元件库收录的底盘数据包括酿酒酵母、大肠杆菌、枯草芽胞杆菌、耶氏解脂酵母和蓝藻等多种合成生物学研究中常见的底盘。
4 我国生物元件库发展亟待解决的问题 虽然“十二五”和“十三五”期间,在973、863及中科院项目的资助下,国内已经开始元件库的建设,在催化元件的数量和质量上已经优于国外元件库,但是与“标准生物元件登记库”等国外著名元件库相比,国内元件的中库元件种类和元件共享机制建设等方面还存在明显的差距,需要加以完善。
4.1 元件库在调控元件的资源收集和整理方面进展缓慢 调控元件可以在时间或者空间上控制目标元件的精确表达,因此在合成生物学线路设计方面具有重要的应用价值[17],但是目前元件库在元件收集方面还偏重于催化元件的收集,而在调控元件的收集整理进展非常缓慢。近年来国内外在调控元件设计和构建等方面取得了长足进展[18-20],例如中国科学院娄春波研究员通过从头设计和优化在大肠杆菌中构建了包括10个模块化、正交化的细胞间通讯系统的工具箱[19],同时Smolke等提出了基于模型的人工酵母启动子设计方法[20]。元件库将密切跟踪这些新的研究进展,尽快完成调控元件数据标准的制定,并尽早开展调控元件数据、元数据实物以及设计工具的收集与整合工作。
4.2 元件库在共享机制建设方面亟待创新 为去中心化管理可以加速元件底盘数据和实物的共享与分发,生物积块基金会等国外元件库都积极地进行尝试。生物积块基金会等国外元件库采用的OpenMTA协议允许元件接收者对所接受的元件进行二次分发,实际上就属于元件实物存储去中心化管理的具体措施。同时,生物积块基金会还通过bionet项目尝试采用更为深入的中心化方式管理元件和底盘,以加速元件数据和实物的共享与分发。国内元件库在去中心化管理的研究和实施方面均远落后于国外元件库,目前主要的元件数据元数据和实物还是集中存储在各自的项目或单位,只有部分的元件分布存储在合作研究单位。元件数据、元数据和实物的去中心化管理需要对相应的支撑技术进行深入研究和探索。目前,区块链技术作为新的去中心化数据共享技术,正逐步在各个领域开展应用[21-24]。对于高价值的元件信息,可以采用区块链技术推动生物元件库的有效共享和安全利用但还需实现若干关键技术的突破,包括刻画元件信息的新型区块链数据结构和共享算法,基于区块链的元件质量控制评估与共享机制,利用区块链智能合约实现去中心化的联合学习方法等。综合利用区块链技术的优势,通过元件、底盘的数据、元数据和实物的去中心化管理,可以大幅提高元件和底盘汇聚和分享的速度,降低运输成本,对于元件库的建设具有重要意义。
除了元件和底盘之外,合成生物学软件和工具的共享方面存在共享难、无统一共享机制等问题。对于合成生物学软件的分享和使用,可以借鉴计算机行业包括BSD开源协议、MIT许可证、Apache Licence 2.0协议(https://aws.amazon.com/cn/apache-2-0/)、GNU通用公共许可协议(GPL,GNU general public license)和程序库通用许可证(LGPL,library general public license)等开源软件协议。元件库需要对这些已有的软件共享机制进行消化、吸收和创新,制定合理的共享协议使得更多的合成生物学软件和工具能够汇聚到元件库平台。
5 展望 虽然国内元件库建设取得了巨大进步,但仍需进一步完善生物元件数据标准和测试标准,以提高元件的数量和质量,同时在元件共享机制建设方面借鉴国外元件库的先进经验,进一步提高元件共享效率。在“合成生物学”和“绿色生物制造”等专项的支持下,可以预见国内合成生物学研究成果将加速涌现,生物元件库将会在数据有源、多层审核、资源共享、信息公开、信息安全和授权访问的基础上,加速合成生物学数据、元数据、实物和设计工具的汇聚与分享,并进一步服务合成生物学研究。
References
| [1] | Kwok R. Five hard truths for synthetic biology. Nature, 2010, 463(7279): 288-290. DOI:10.1038/463288a |
| [2] | Smolke CD. Building outside of the box: iGEM and the BioBricks Foundation. Nature Biotechnology, 2009, 27(12): 1099-1102. DOI:10.1038/nbt1209-1099 |
| [3] | Kahl L, Molloy J, Patron N, Matthewman C, Haseloff J, Grewal D, Johnson R, Endy D. Opening options for material transfer. Nature Biotechnology, 2018, 36(10): 923-927. DOI:10.1038/nbt.4263 |
| [4] | Baig H, Fontanarrosa P, Kulkarni V, McLaughlin JA, Vaidyanathan P, Bartley B, Beal J, Crowther M, Gorochowski TE, Grünberg R, Misirli G, Scott-Brown J, Oberortner E, Wipat A, Myers CJ. Synthetic biology open language (SBOL) version 3.0.0. Journal of Integrative Bioinformatics, 2020, 17(2/3): e0017. DOI:10.1515/jib-2020-0017 |
| [5] | Bartley B, Beal J, Clancy K, Misirli G, Roehner N, Oberortner E, Pocock M, Bissell M, Madsen C, Nguyen T, Zhang Z, Gennari JH, Myers C, Wipat A, Sauro H. Synthetic biology open language (SBOL) version 2.0.0. Journal of Integrative Bioinformatics, 2015, 12(2): 902-991. DOI:10.1515/jib-2015-272 |
| [6] | Quinn JY, Cox RS, Adler A, Beal J, Bhatia S, Cai YZ, Chen J, Clancy K, Galdzicki M, Hillson NJ, Le Novère N, Maheshwari AJ, McLaughlin JA, Myers CJ, P U, Pocock M, Rodriguez C, Soldatova L, Stan GBV, Swainston N, Wipat A, Sauro HM. SBOL visual: a graphical language for genetic designs. PLoS Biology, 2015, 13(12): e1002310. DOI:10.1371/journal.pbio.1002310 |
| [7] | Galdzicki M, Clancy KP, Oberortner E, Pocock M, Quinn JY, Rodriguez CA, Roehner N, Wilson ML, Adam L, Anderson JC, Bartley BA, Beal J, Chandran D, Chen J, Densmore D, Endy D, Grünberg R, Hallinan J, Hillson NJ, Johnson JD, Kuchinsky A, Lux M, Misirli G, Peccoud J, Plahar HA, Sirin E, Stan GB, Villalobos A, Wipat A, Gennari JH, Myers CJ, Sauro HM. The Synthetic Biology Open Language (SBOL) provides a community standard for communicating designs in synthetic biology. Nature Biotechnology, 2014, 32(6): 545-550. DOI:10.1038/nbt.2891 |
| [8] | Nguyen T, Roehner N, Zundel Z, Myers CJ. A converter from the systems biology markup language to the synthetic biology open language. ACS Synthetic Biology, 2016, 5(6): 479-486. DOI:10.1021/acssynbio.5b00212 |
| [9] | Roehner N, Myers CJ. A methodology to annotate systems biology markup language models with the synthetic biology open language. ACS Synthetic Biology, 2014, 3(2): 57-66. DOI:10.1021/sb400066m |
| [10] | Hillson NJ, Plahar HA, Beal J, Prithviraj R. Improving synthetic biology communication: recommended practices for visual depiction and digital submission of genetic designs. ACS Synthetic Biology, 2016, 5(6): 449-451. DOI:10.1021/acssynbio.6b00146 |
| [11] | Wang PP, Wei W, Ye W, Li XD, Zhao WF, Yang CS, Li CJ, Yan X, Zhou ZH. Synthesizing ginsenoside Rh2 in Saccharomyces cerevisiae cell factory at high-efficiency. Cell Discovery, 2019, 5: 5. DOI:10.1038/s41421-018-0075-5 |
| [12] | Li JH, Mutanda I, Wang KB, Yang L, Wang JW, Wang Y. Chloroplastic metabolic engineering coupled with isoprenoid pool enhancement for committed taxanes biosynthesis in Nicotiana benthamiana. Nature Communications, 2019, 10(1): 4850. DOI:10.1038/s41467-019-12879-y |
| [13] | Li JH, Tian CF, Xia YH, Mutanda I, Wang KB, Wang Y. Production of plant-specific flavones baicalein and scutellarein in an engineered E. coli from available phenylalanine and tyrosine. Metabolic Engineering, 2019, 52: 124-133. DOI:10.1016/j.ymben.2018.11.008 |
| [14] | Ping Y, Li XD, Xu BF, Wei W, Wei WP, Kai GY, Zhou ZH, Xiao YL. Building microbial hosts for heterologous production of N-methylpyrrolinium. ACS Synthetic Biology, 2019, 8(2): 257-263. DOI:10.1021/acssynbio.8b00483 |
| [15] | Ping Y, Li XD, You WJ, Li GQ, Yang MQ, Wei WP, Zhou ZH, Xiao YL. De novo production of the plant-derived tropine and pseudotropine in yeast. ACS Synthetic Biology, 2019, 8(6): 1257-1262. DOI:10.1021/acssynbio.9b00152 |
| [16] | Zhou CF, Xu QW, He S, Ye W, Cao RF, Wang PY, Ling YC, Yan X, Wang QZ, Zhang GQ. GTDB: an integrated resource for glycosyltransferase sequences and annotations. Database, 2020, 2020: 1-9. |
| [17] | Tang HT, Wu YL, Deng JL, Chen NZ, Zheng ZH, Wei YJ, Luo XZ, Keasling JD. Promoter architecture and promoter engineering in Saccharomyces cerevisiae. Metabolites, 2020, 10(8): 320. DOI:10.3390/metabo10080320 |
| [18] | Zheng Y, Meng FK, Zhu ZH, Wei WJ, Sun Z, Chen JC, Yu B, Lou CB, Chen GQ. A tight cold-inducible switch built by coupling thermosensitive transcriptional and proteolytic regulatory parts. Nucleic Acids Research, 2019, 47(21): e137. DOI:10.1093/nar/gkz785 |
| [19] | Du P, Zhao HW, Zhang HQ, Wang RS, Huang JY, Tian Y, Luo XD, Luo XX, Wang M, Xiang YH, Qian L, Chen YH, Tao Y, Lou CB. De novo design of an intercellular signaling toolbox for multi-channel cell-cell communication and biological computation. Nature Communications, 2020, 11: 4226. DOI:10.1038/s41467-020-17993-w |
| [20] | Kotopka BJ, Smolke CD. Model-driven generation of artificial yeast promoters. Nature Communications, 2020, 11: 2113. DOI:10.1038/s41467-020-15977-4 |
| [21] | Kuo TT, Kim HE, Ohno-Machado L. Blockchain distributed ledger technologies for biomedical and health care applications. Journal of the American Medical Informatics Association, 2017, 24(6): 1211-1220. DOI:10.1093/jamia/ocx068 |
| [22] | Hasselgren A, Kralevska K, Gligoroski D, Pedersen SA, Faxvaag A. Blockchain in healthcare and health sciences-A scoping review. International Journal of Medical Informatics, 2020, 134: 104040. DOI:10.1016/j.ijmedinf.2019.104040 |
| [23] | Mashamba-Thompson TP, Crayton ED. Blockchain and artificial intelligence technology for novel coronavirus disease-19 self-testing. Diagnostics, 2020, 10(4): 198. DOI:10.3390/diagnostics10040198 |
| [24] | Patelli N, Mandrioli M. Blockchain technology and traceability in the agrifood industry. Journal of Food Science, 2020, 85(11): 3670-3678. DOI:10.1111/1750-3841.15477 |
