徐娜1,2, 龚云霞1,2, 邵彦春1,2, 高书山3, 陈守文4, 陈福生1,2


1. 华中农业大学, 湖北省传统发酵食品国际科技合作基地, 湖北 武汉 430070;
2. 华中农业大学食品科学与技术学院, 湖北 武汉 430070;
3. 中国科学院微生物研究所微生物资源国家重点实验室, 北京 100101;
4. 湖北大学生命科学学院, 湖北 武汉 430062
收稿日期:2020-08-14;修回日期:2021-01-25;网络出版日期:2021-02-10
基金项目:国家自然科学基金(31730068, 31330059);中央高校基本科研业务费专项(2662019Y015)
*通信作者:陈福生, Fax: +86-27-87282111;E-mail: chenfs@mail.hzau.edu.cn.
摘要:CRISPR-Cas(clustered regularly interspaced short palindromic repeats(CRISP)and CRISPR associated proteins)系统是细菌用来防御病毒、质粒等外源核酸入侵的一种获得性免疫防御系统。随着研究的深入,CRISPR-Cas系统已发展为一种重要的基因编辑工具,并成功应用于动物、植物和微生物的基因改造中。但该基因编辑方法有时存在基因脱靶效应,从而限制了其推广应用。最近,通过将1种新发现的抗CRISPR蛋白(Anti-CRISPR protein,ACP)与CRISPR-Cas系统相结合,已成功开发出可控制基因脱靶效率的CRISPR-Cas基因编辑工具。本文首先对CRISPR-Cas系统及ACP进行了简要介绍,然后就CRISPR-Cas基因编辑工具及ACP在微生物基因改造的应用现状进行了综述,并对ACP介导的CRISPR-Cas基因编辑方法(ACP-CRISPR-Cas)在微生物基因编辑中的应用前景进行了讨论。
关键词:CRISPR-Casanti-CRISPR基因编辑微生物
Research progress of CRISPR-Cas system and anti-CRISPR protein in microorganisms
Na Xu1,2, Yunxia Gong1,2, Yanchun Shao1,2, Shushan Gao3, Shouwen Chen4, Fusheng Chen1,2


1. Hubei International Scientific and Technological Cooperation Base of Tranditional Fermented Foods, Wuhan 430070, Hubei Province, China;
2. College of Food Science and Technology, Huazhong Agricultural University, Wuhan 430070, Hubei Province, China;
3. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
4. College of Life Science, Hubei University, Wuhan 430062, Hubei Province, China
Received: 14 August 2020; Revised: 25 January 2021; Published online: 10 February 2021
*Corresponding author: Fusheng Chen, Fax: +86-27-87282111; E-mail: chenfs@mail.hzau.edu.cn.
Foundation item: Supported by the Major Programs of the National Natural Science Foundation of China (31730068, 31330059) and by the Fundamental Research Funds for the Central Universities (2662019Y015)
Abstract: The CRISPR-Cas (clustered regularly interspaced short palindromic repeats (CRISPR) and CRISPR associated proteins) system is a bacterial acquired immune defense system, which can be used by bacteria to prevent the invasion of viruses, plasmids and other exogenous nucleic acids. With the further research, the CRISPR-Cas system has been successfully developed into an important tool to edit genes, and applied into gene modification in animals, plants and microorganisms. However, the CRISPR-Cas gene-editing tool sometimes exists in gene off-target effect, which limits its wide application. Recently, by combining a newly discovered anti-CRISPR protein (ACP) with CRISPR-Cas system, a gene-editing tool that can control the efficiency of gene off-target has been successfully developed. In this review, the CRISPR-Cas system and ACP were firstly introduced in brief, then the application of CRISPR-Cas gene editing tool and ACP in microbial gene modification was summarized, and the prospect of ACP-CRISPR-Cas applied in microbial gene editing was discussed.
Keywords: CRISPR-Casanti-CRISPRgene-editing toolmicroorganisms
CRISPR-Cas (clustered regularly interspaced short palindromic repeats (CRISPR) and CRISPR associated proteins)系统,即规律成簇间隔短回文重复序列及其相关蛋白,是广泛存在于细菌和古菌中的一种获得性免疫防御系统。该系统由CRISPR核酸阵列和Cas蛋白(核酸酶)两部分组成[1],前者用于储存和记录入侵外源核酸片段,即当噬菌体(病毒)入侵时,细菌或古菌细胞可将噬菌体核酸(外源核酸)的特异性片段捕捉并整合到自身基因组的CRISPR序列中;后者则可切割再次入侵噬菌体的核酸,以实现免疫防御功能,即当同一种噬菌体再次入侵时,首次被捕捉并整合到细菌CRISPR序列中的特异性核酸片段被转录加工成CRISPR RNA (crRNA),并与一种或多种Cas核酸酶结合后被引导到再次入侵噬菌体中的CRISPR同源序列(靶序列),从而酶切和降解噬菌体核酸,以实现对噬菌体入侵的监控和清除[2-6]。随着对CRISPR-Cas系统组成及其相关机理的深入研究,该系统已被成功改造并发展为一种基因编辑工具,在动物、植物和微生物的基因编辑(改造)中得到了很好的应用,但是采用该基因编辑工具进行基因编辑时,有时存在基因脱靶效应(gene off target effect,即未能准确改造目标基因反而可能使非目标基因产生核酸突变的现象),从而在很大程度上限制了其推广应用。最近,在噬菌体中发现了1种可抑制CRISPR-Cas作用的抗CRISPR蛋白(Anti-CRISPR protein,ACP),通过对ACP作用机制的研究,表明其可有效控制CRISPR-Cas基因编辑中的脱靶效应,并被成功地应用于提高CRISPR-Cas基因编辑的精准性[7-8]。
尽管基于CRISPR-Cas系统开发的基因编辑方法及其相关应用已有文献报道[9-14],但是关于CRISPR-Cas基因编辑工具和ACP在微生物基因改造方面的研究应用的综述不多,特别是相关的中文文献很少[15]。本综述将首先对CRISPR-Cas系统和ACP进行简介,然后就CRISPR-Cas基因编辑工具及ACP在微生物基因改造的应用现状进行了综述,并对ACP介导的CRISPR-Cas基因编辑工具(ACP-CRISPR-Cas)在微生物基因编辑中的应用前景进行了讨论。
1 CRISPR-Cas系统与ACP简介 1.1 CRISPR-Cas系统 CRISPR-Cas系统的研究始于1987年,Ishino等[16]在研究大肠杆菌(Escherichia coli)中负责碱性磷酸酶同工酶转化的iap基因及其侧翼区DNA片段的核苷酸序列时,发现了一段位于iap 3′末端侧翼区域的29 pb重复序列。与大多数重复序列通常以串联重复的存在形式不同,这种重复序列被5个32 bp的非重复序列间隔。随后的研究发现这种重复序列广泛存在于细菌和古菌的基因组中。2000年,Mojica等[17]发现,这种间隔重复序列(spaced repeat sequence)存在于当时已测序的40%细菌和90%古菌的基因组中,并将其作为一个“家族”,命名为短规律间隔重复序列(short regularly spaced repeats,SRSRs)家族。
以上发现激发了人们对这种微生物间隔重复元件的研究兴趣。2002年,Jansen等[18]发现,这种间隔重复序列仅存在于细菌和古菌等原核生物的核酸中,并将此类序列正式命名为规律成簇间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR),并且发现了与CRISPR相关的高度保守的cas基因。2005年,进一步研究表明,CRISPR是来源于细菌或古菌染色质以外的与噬菌体核酸同源的间隔核酸序列,即间隔子(spacer)序列,并且发现噬菌体无法再次感染已含有同一种噬菌体间隔子序列的古菌[19-21]。因此,推测此类间隔子序列可作为一种免疫记忆,防御噬菌体感染,但具体机制当时仍不清楚。2007年,Barrangou等[2]首次通过实验证明了CRISPR-Cas系统的适应性免疫作用,该系统中的间隔子序列可特异性地与细菌核酸中的靶序列结合,而Cas核酸酶则可获取间隔子序列并防御噬菌体再次入侵。2008年,Brouns等[22]证明了CRISPR序列可被转录并加工成熟为包含单个间隔子序列的RNA,即CRISPR RNA (crRNA),以引导Cas核酸酶对噬菌体特异性核酸序列进行识别和切割。同年,发现Cas核酸酶特异性切割的靶标为DNA而不是RNA[23]。但随后研究也发现了可特异性识别并切割单链RNA的Cas核酸酶[24]。至2011年,CRISPR-Cas系统作为细菌和古菌获得性免疫系统的相关机理已研究得相当清楚,CRISPR-Cas系统在执行功能时包括免疫捕获、表达和免疫干扰3个阶段[25-27]。在免疫捕获阶段,Cas核酸酶通过识别入侵噬菌体的核酸序列获取间隔子序列(spacers),并将其整合到细菌和古菌的CRISPR序列中,形成免疫记忆;在表达阶段,CRISPR序列转录并加工为成熟的crRNA后,与Cas核酸酶结合形成crRNA-Cas复合体;在免疫干扰阶段,crRNA引导Cas核酸酶靶向并切割入侵噬菌体的核酸。图 1是参考相关文献[28]以Ⅰ型CRISPR-Cas系统为例绘制的作用机理示意图。
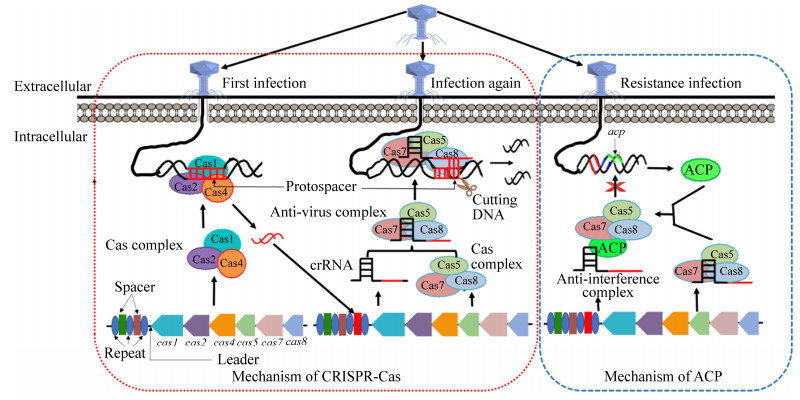 |
| 图 1 CRISPR-Cas系统(Ⅰ型)与ACP的作用机理示意图 Figure 1 The schematic diagram of action mechanisms of CRISPR-Cas system (Type Ⅰ) and ACP. |
| 图选项 |
随着对CRISPR-Cas系统的深入研究,各种Cas核酸酶相继被发现,它们的功能也相继被明确(表 1),并基于它们的功能与组合对CRISPR-Cas系统进行了分类(图 2)。尽管不同类型CRISPR-Cas系统存在一定差异,但作用机理基本相同。2005年,Haft等[29]通过对40个细菌和古菌基因组分析,根据cas基因操纵子(operon)的组成和结构以及Cas核酸酶蛋白进化树的拓扑结构将CRISPR-Cas系统划分为8个亚型。2011年,Makarova等[30]根据Cas核酸酶核心元件序列的差异,将CRISPR-Cas系统划分为Ⅰ、Ⅱ和Ⅲ型,其核心Cas分别为Cas3、Cas9和Cas10,并结合cas基因操纵子的组成和结构,将这3种类型划分为10个亚类。2015年,Makarova等[31]根据CRISPR-Cas系统中Cas复合体的组成与cas的结构特征,提出了2类-5型-18亚类的CRISPR-Cas分类方法,目前,该方法已被普遍接受并不断完善,图 2就是基于此分类方法绘制的。依据该方法可将大多数CRISPR-Cas系统明确划分为不同的类型和亚型。第1类包括Ⅰ、Ⅲ和Ⅳ共3型,12亚型,它们的核酸酶复合体由多个Cas组成。而Ⅰ型又分为7个亚型,即Ⅰ-A至Ⅰ-F及Ⅰ-U。在Ⅰ型CRISPR-Cas系统中均包含可降解目标核酸序列的Cas3,其与Cas1及Cas2组成CRISPR相关抗病毒复合物(CRISPR associated complex for antiviral defense,简称抗病毒复合物)参与加工形成crRNA;Ⅲ型CRISPR-Cas系统均包含Cas10,且此型CRISPR-Cas系统不仅可靶向DNA也可靶向RNA;Ⅳ型系统中的抗病毒复合物由Cas5、Cas7和Csf1组成,此系统通常无CRISPR序列,也缺少Cas1和Cas2。第2类包括Ⅱ和Ⅴ共2个型,6个亚型,其抗病毒复合物不是由不同的Cas组成,而是一个具有多个结构域,分子量较大的蛋白。Ⅱ型系统包括Ⅱ-A、Ⅱ-B和Ⅱ-C共3个亚型,在Ⅱ型系统中Cas均为Cas9,它具有切割靶标DNA及加工crRNA的功能;Ⅴ型系统的Cas蛋白主要为Cas12 (Cpf1),也包含Cas1和Cas2。此外,2016年张峰等[24]在沙氏纤毛菌(Leptotrichia shahii)中还发现了一种新型CRISPR-Cas系统Ⅵ,它包括A、B、C共3个亚型,其Cas主要为Cas13。图 2是目前已报道的CRISPR-Cas系统。
表 1. Cas的分类与功能 Table 1. Classification and function of Cas
| Protein names | Functions | System types | References |
| Cas1 | Participate in spacer insertion | Ⅰ, Ⅱ, Ⅲ-A, Ⅴ | [18] |
| Cas2 | Participate in spacer insertion | Ⅰ, Ⅱ, Ⅲ-A, Ⅴ-A, Ⅴ-B, Ⅴ-E | [18] |
| Cas3 | Participate in DNA cleavage | Ⅰ | [18] |
| Cas4 | Participate in spacer insertion | Ⅰ-A, Ⅰ-B, Ⅰ-C, Ⅰ-D, Ⅱ-B, Ⅴ-A, Ⅴ-B, Ⅴ-E | [18] |
| Cas5 | Participate in crRNA and target DNA binding | Ⅰ, Ⅲ, Ⅳ | [29] |
| Cas6 | Participate in crRNA maturation | Ⅰ-A, Ⅰ-B, Ⅰ-D, Ⅲ-A, Ⅲ-B | [29] |
| Cas7 | Participate in crRNA and target DNA binding | Ⅰ, Ⅲ, Ⅳ | [30] |
| Cas8 | Participate in crRNA and target DNA binding | Ⅰ | [30] |
| Cas9 | Participate in target DNA cleavage | Ⅱ | [23] |
| Cas10 | Participate in single-stranded DNA cleavage | Ⅲ | [3] |
| Cas12 | Participate in target DNA binding and cleavage | Ⅴ | [57] |
| Cas13 | Participate in target DNA binding and cleavage | Ⅵ | [24] |
表选项
 |
| 图 2 CRISPR-Cas系统的分类(改编自[30]) Figure 2 The classification of CRISPR-Cas systems. The oval represents the repeated sequence and the colorful rectangular box represents the spacer sequence. |
| 图选项 |
1.2 ACP ACP是存在于噬菌体中可抑制CRISPR-Cas系统的抗CRISPR-Cas蛋白复合体。首个ACP是2013年在铜绿假单胞菌UCBPP-PA14 (Pseudomonas aeruginosa,UCBPP-PA14)的噬菌体中发现的,并证明其可使噬菌体核酸避免被细菌CRISPR-Cas系统破坏[7]。随后,还发现ACP也存在于其他铜绿假单胞菌[32-33]、脑膜炎奈瑟菌(Neisseria meningitides)[33]、单增李斯特菌(Listeria monocytogenes)[34]和厦门希瓦氏菌(Shewanella xiamenensis)[33]等细菌细胞中。
ACP蛋白复合体包括由多种可移动遗传元件(mobile genetic elements,MGEs)编码的蛋白,可在不同阶段抑制CRISPR-Cas系统功能[35-36]。目前,ACP主要以其被发现的顺序与所抑制的CRISPR-Cas系统进行命名[37]。例如,目前广泛使用的AcrIIA4是指第4个被发现的可抑制Ⅱ-A型CRISPR-Cas系统的ACP。截止到2019年,已经发现了45种非同源的ACP,其中24种为1类CRISPR-Cas系统的抑制蛋白,21种为2类CRISPR-Cas系统的抑制蛋白,不同的ACP具有不同的作用机制和结构,且彼此之间无显著的序列相似性[38-40]。图 1显示的是抑制Ⅰ型CRISPR-Cas系统的ACP通过与抗病毒复合物结合从而阻止抗病毒复合物行使功能的示意图。另外,抗Ⅰ型CRISPR-Cas系统的ACP可通过阻止Cas3被募集而抑制靶DNA的降解[41];而抗Ⅱ型CRISPR-Cas系统的ACP可通过与Cas9结合而阻止其与靶DNA结合[41-44]。
图 3为CRISPR-Cas系统与ACP发现和研究的主要历程。
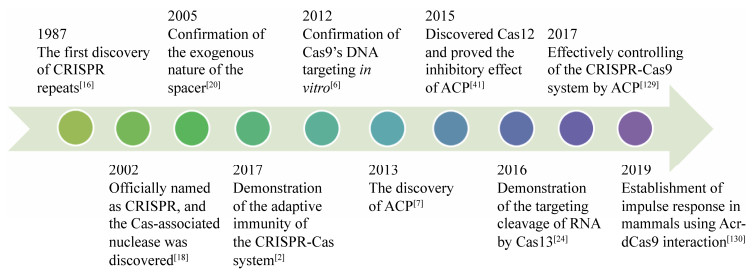 |
| 图 3 CRISPR-Cas系统与ACP的主要研究历程 Figure 3 The main research history of CRISPR-Cas systems and anti-CRISPR proteins. |
| 图选项 |
2 CRISPR-Cas基因编辑系统在微生物中的应用 2012年,Jinek等首次在体外证明了CRISPR- Cas9系统特异性切割靶DNA的功能,并将crRNA和反式激活(trans-activating) CRISPR RNA (crRNA: tracrRNA)改造为sgRNA (single guide RNA),从而使CRISPR-Cas9系统应用于基因编辑成为可能[6]。由此揭开了将CRISPR-Cas系统改造为基因编辑工具的序幕。近年来,CRISPR-Cas基因组编辑系统,凭借其低成本、易操作和高效率等优势被广泛应用于动物、植物和微生物的基因编辑,特别是在动植物的基因编辑中得到了广泛应用,相关的综述也较多[9-11],所以在此不再赘述。而在微生物基因编辑方面,由于过去成熟的基因改造方法很多,所以CRISPR-Cas基因组编辑系统在微生物基因编辑中的研究相对较少,相关综述也不多,特别是中文综述很少[12-14],以下将就CRISPR-Cas基因编辑工具在大肠杆菌、蓝细菌(Cyanobacteria)、放线菌(Actinomycetes)等原核微生物,以及酵母菌与黑曲霉(Aspergillus niger)、米曲霉(A. oryzae)、烟曲霉(A. fumigatus)、构巢曲霉(A. nidulans)、土曲霉(A. terreus)、产黄青霉(Penicillium chrysogenum)、里氏木霉(Trichoderma reesei)和红曲菌(Monascus spp.)等真核微生物基因编辑中的应用进行概述。
2.1 在原核微生物中的应用
2.1.1 在大肠杆菌中的应用: 由于大肠杆菌培养容易且遗传背景清晰,所以常被用作基因编辑(改造)的出发菌株[45]。2013年,张峰团队利用CRISPR-Cas9基因编辑技术在大肠杆菌中成功实现了基因改造[46],他们首先将一段没有特殊标记的DNA突变序列引入大肠杆菌中,然后采用CRISPR-Cas基因编辑技术成功使65%大肠杆菌中被引入的DNA获得目标突变。2015年,利用CRISPR-Cas9基因编辑技术对大肠杆菌实现了包括基因敲除、插入等多种精确的基因修饰,且采用该技术可同时对cadA、maeA、maeB等3个基因进行基因敲除[47]。2016年,Abudayyeh等[24]通过构建含单拷贝LshCas13a (Cas13 from Leptotrichia shahii)基因座的载体,并在大肠杆菌中异源表达,从而使大肠杆菌免遭MS2噬菌体(一种裂解性单链RNA噬菌体)的侵入,并首次在大肠杆菌中实现了RNA的敲除。夏军等[48]利用CRISPR-Cas偶联λ-Red重组酶(λ-Red重组酶由Exo、Beta和Gam 3种蛋白质组成,这3种蛋白对dsDNA (double-strand DNA)重组是必需的),以一个二元载体pTargetF/pCas快速敲除了大肠杆菌MG1655中与脂肪酸代谢相关基因ppc和fadD,并成功获得了单敲除及双敲除菌株。2017年,利用CRISPR-Cas9将长度为7 kb的外源片段整合到大肠杆菌染色质中,实现了DNA大片段的高效同源重组[49]。
对大肠杆菌中相关产物代谢途径的多基因进行调控是实现生物质燃料、化学药品与药物等高效转化的重要途径之一。为此,基于CRISPR-Cas系统已开发出可特异性地调控基因表达的CRISPR干扰(CRISPR interference,CRISPRi)方法,该方法可在不破坏靶基因的情况下瞬时或组成型控制相关基因的表达量,以减少或阻止代谢途径中的中间产物流向副产物通路,增加其流向目标产物通路的量,以提高目标产物的产率[50]。采用CRISPRi通过微调碳中心代谢途径也可提高大肠杆菌基因工程菌中类黄酮的产量[51-52]。Cai等[52]通过CRISPRi系统抑制银松素(pinosylvin)合成途径中fabD (编码malonyl-CoA- ACP转酰基酶)基因的表达量,使大肠杆菌工程菌的银松素产量提高了1.9倍。Kim等[53]采用CRISPRi在大肠杆菌中对与醋酸盐、乳酸盐、琥珀酸盐及乙醇等代谢副产物合成相关的4个内源基因(pta、frdA、ldhA和adhE)的表达水平实现了有效抑制。
2.1.2 在蓝细菌中的应用: 蓝细菌作为一种光合自养的原核生物,已被成功设计改造成可用于烷烃、萜类化合物等生物质燃料与生物活性物质的产生菌株[54]。但是,过去蓝细菌的基因改良方法较为单一,主要是采用转化率很低的同源重组方法,且由于蓝细菌的基因多具有较高的拷贝数,所以通常很难获得稳定的改良菌株[55]。为此,2016年,Pakrasi研究组将CRISPR-Cas9基因编辑技术应用于蓝细菌模式菌株细长聚球藻(Synechococcus elongatus) PCC 7942和UTEX 2973的基因改造中[56],但发现Cas9对PCC 7942和UTEX 2973均存在不同程度的细胞毒性,从而限制了该系统在蓝细菌中的应用。
为克服这一问题,该研究组进一步利用CRISPR-Cas12对蓝细菌进行了基因编辑[57-58]。通过将FnCas12(Cas12 from Francisella novicida)基因及靶向目标基因的crRNA克隆至广谱宿主质粒RSF1010中,成功对细长聚球藻UTEX2973、集胞藻(Synechocystis sp.) PCC6803和鱼腥藻(Anabaena sp.) 7120 3种不同类型蓝细菌中的基因组实现了敲除、插入和点突变,并发现,FnCas12对蓝细菌的细胞毒性比Cas9低得多。因此,作者认为对蓝细菌的基因编辑而言,CRISPR-Cas12的潜力更大。
此外,通过使用CRISPR-dCas9 (dead Cas9)或CRISPR-dCas12 (dead Cas12)系统,成功对集胞藻PCC 6803[59]、细长聚球藻PCC 7942[60]、鱼腥藻PCC 7120[61-62]和聚球藻UTEX 2973等菌株中的相关基因实现了动态调控,提高了它们的脂肪酸、氨基酸、琥珀酸和乳酸等的产率。
2.1.3 在放线菌中的应用: 放线菌作为一类可产生不同天然活性物质的微生物一直倍受关注[63-64]。2014年,采用CRISPR-Cas9基因编辑技术成功对放线菌中的3种链霉菌——变铅青链霉菌(Streptomyces lividans)、白色链霉菌(S. albus)及绿产色链霉菌(S. viridochromogenes)实现了20-30 kb DNA片段的敲除,敲除率为70%-100%[65]。通过插入双sgRNA策略,在变铅青链霉菌中成功对与合成十一烷基灵菌红素(undecylprodigiosin)的相关基因red N,以及与合成放线紫红素(actinorhodin)的相关基因actVA-OPF5同时进行了编辑[65]。将CRISPR-Cas9基因编辑工具与Gibson克隆(能高效、无痕的组装多个DNA片段的无缝克隆)组装技术联合使用,可实现无需使用抗性筛选标志基因的基因同框缺失和启动子更换、目的基因簇的构建以及对链霉菌次级代谢基因簇进行重构和异源表达[66-68]。以CRISPR-Cas基因编辑技术将异源启动子精确引入链霉菌的聚酮化合物(polyketide)和磷酸酯等的合成基因簇中,成功激活了5种链霉菌中沉默的蓝色素(indigoidine)、放线紫红素与十一烷基灵菌红素等基因簇[69]。
除链霉菌外,CRISPR-Cas基因编辑技术也被用于其他放线菌的基因改造,并实现了相关化合物的高效生产。例如,采用CRISPR-Cas基因编辑技术敲除糖多孢红霉菌(Saccharopolyspora erythraea)中与合成红霉素A (erythromycin A)相关的基因SACE-1765后,使红霉素A产量提高了12.7%,进一步在SACE-0712基因中插入启动子PermE,则可使红霉素A的产量提高80.3%[70]。
2.1.4 在其他细菌中的应用: CRISPR-Cas基因编辑工具也成功应用于包括结核分枝杆菌(Mycobacterium tuberculosis)、金黄色葡萄球菌(Staphylococcus aureus)、铜绿假单胞菌、肺炎克雷伯菌(Klebsiella Pneumoniae)和鼠疫耶尔森氏菌(Yersinia pestis)等病原菌的基因编辑中。采用基于CRISPR-Cas9的pCasSA系统[71-72]并将Cas9切口酶与胞苷脱氨酶融合开发的高效便捷的pnCasSA-BEC碱基编辑方法[73],成功实现了对金黄色葡萄球菌中相关基因的高效失活和点突变。对于结核分枝杆菌这种很难进行基因改造的病原菌,采用Cas9-CRISPRi技术通过抑制相关基因表达,促进了相关基因功能的分析[74-76]。Cas12a介导的基因编辑方法在耻垢分枝杆菌(Mycobacterium smegmatis)的基因改造中也得到了成功应用[77],由CRISPR-FnCpf1辅助的NHEJ系统(CRISPR-FnCpf1-assisted NHEJ system)可使基因编辑效率提高70%。另外,CRISPR-Cas9/ Cas12a基因编辑工具已成功应用于恶臭假单胞菌(P. putida)的基因编辑中[78-79],从而为其提供了更加高效、快速的基因编辑工具;利用CRISPRi系统成功抑制了绿脓假单胞菌(P. pyocyaneum)、荧光假单胞菌(P. fluorescens)和恶臭假单胞菌中荧光铁载体(pyoverdine)合成基因pvdH的表达[80]。此外,在肺炎克雷伯菌中,已开发了基于CRISPR-Cas9的基因组编辑[81]和干扰方法[82],并对其代谢副产物乳酸合成相关基因(pmd、aldA、ldhA、mgsA)实现了单个或多个基因的同时抑制[82],而在鼠疫耶尔森氏菌中,利用CRISPR-Cas12a辅助的重组系统,实现了在不需要引入抗生素抗性基因的情况下对hmst及y4098基因的无痕点突变[83]。
最近,陈守文课题组将Cas9n (Cas9 nickase)用于地衣芽孢杆菌(Bacillus licheiforiis)中的基因改造中,成功实现了单基因敲除、多基因破坏、DNA大片段缺失与单基因整合[84],并将dCas9 (nuclease-deactivated Cas9)介导的CRISPRi应用于地衣芽孢杆菌中,成功抑制了单个和多个基因的表达[85]。
2.2 在真核微生物中的应用
2.2.1 在酵母中的应用: 酵母菌是一类单细胞的真核微生物,具有易培养、繁殖快等特点。酵母菌,特别是酿酒酵母(Saccharomyces cerevisiae)作为人类最早利用的微生物之一,广泛应用于食品酿造和发酵中[86]。此外,酿酒酵母也是研究真核细胞各种生命过程的重要模式生物[87],还是其他真核生物基因异源表达的主要宿主之一[88]。由于CRISPR-Cas基因编辑的优越性,运用其对酿酒酵母的基因进行编辑可实现更加快捷地改良酿酒酵母,并丰富酿酒酵母的基因改造方法。
CRISPR-Cas基因编辑工具在酿酒酵母基因改造中的应用主要包括基因整合(gene integration)、敲除或突变。
在基因整合方面,通过引入外源合成酶基因或基因簇[89],或更换启动子、核糖体结合位点(ribosomal binding site)等方法[90],可调控酿酒酵母细胞中的物质代谢流(metabolic flux),从而提高目标产物的产率。但传统的酿酒酵母基因整合常需要以抗生素进行多轮抗性压力筛选,而运用CRISPR-Cas基因编辑技术可以减少,甚至避免进行抗生素筛选,从而加速酿酒酵母的基因整合过程。2013年,Dicarlo等首次将CRISPR-Cas9基因编辑技术应用于酿酒酵母细胞,通过将1.4 kb的KanMX基因整合到酿酒酵母中,实现了近100%的重组率[91],但表达载体的制备过程复杂,效率较低。2015年,Horwitz等[88]采用线性质粒与多个sgRNA和供体DNA的共转化,成功构建了一次转化可整合6个基因片段的酿酒酵母CRISPR-Cas基因编辑技术,从而大大提高了效率。Ronda等[92]结合EasyClone基因工具(可将外源基因导入受体细胞的载体工具),开发了CrEdit (CRISPR/Cas9 mediated genome editing)基因编辑方法,并成功将3个外源β-胡萝卜素合成基因片段整合到酿酒酵母细胞不同染色体中,也大大提高了整合效率。2017年,Li等[93]以FnCas12作为酿酒酵母CRISPR-Cas基因编辑中的Cas核酸酶,分别以95%、52%和43%的效率实现了单基因、双基因和三基因的整合。Verwaal等[94]也证明LbCas12 (Cas12 from Lachnospiraceae bacterium)可以在酿酒酵母中实现单和多基因组整合,整合效率分别达到100%和91.5%。
CRISPR-Cas基因编辑工具在酿酒酵母的基因敲除方面也得到了较为广泛研究和应用。2014年,Xu等[95]以stCas9 (Cas9 from Streptococcus thermophilus)及优化的sgRNA在酿酒酵母细胞中实现了12%靶向效率的基因敲除,为开发基于CRISPR-Cas系统,构建酿酒酵母的基因敲除方法奠定了基础。同年,Ryan等[96]以肝炎病毒(hepatitis virus)核酶修饰gRNA,构建了高效的CRISPRm (multiple CRISPR)基因编辑工具,以近乎100%的效率对11个基因实现了敲除,并首次尝试了一个CRISPR序列同时对2-3个基因进行敲除。此后,许多研究者采用CRISPR-Cas基因编辑工具对酿酒酵母中相关基因进行了高效敲除。Bao等[97]构建基于iCas9 (野生Cas9变体)的CRISPR-Cas同源整合系统(HⅠ-CRISPR),对酿酒酵母细胞中与氢化可的松(hydrocotisone)合成相关的ATF2、GCY1和YPR1共3个基因实现了同时敲除,敲除效率几乎达到100%。2017年,?wiat等[98]采用FnCas12在酵母细胞中实现了4个基因的同时敲除。2019年,Liu等[99]采用CRISPR-Cas9基因编辑工具同时敲除了酿酒酵母YS-7中的与乙醇产生相关的3个基因,使其乙醇产量提高了40.63%。
针对工业中使用的酿酒酵母菌常为多倍体,难以进行有效基因编辑的问题[100],2014年,Zhang等[101]采用CRISPR-Cas9基因编辑方法以15%-60%的效率对酿酒酵母多倍体工程菌ATCC4124中的4个基因依次进行了完全敲除,并获得了4种营养缺陷型突变体;2018年,Lian等[102]以高拷贝质粒表达gRNA,分别对二倍体和三倍体酿酒酵母菌株中影响木糖利用的ALD6、PHO13、LEU2和URA3共4个基因实现了100%完全敲除。
此外,2013年,Farzadfard等[103]通过将crisprTFs (CRISPR-based transcription factor)靶向至酿酒酵母内源性启动子的不同位置,实现了转录因子的激活或抑制。Gilbert等[104]将dCas9融合到具有调控功能的效应域上,成功实现了基因的稳定转录激活或抑制,且发现CRISPRi介导的转录抑制具有高度特异性。2015年,Lenstra等[105]将dCas9靶向至酿酒酵母菌非编码RNA (non-coding RNA),发现CRISPR-dCas9基因编辑技术可在不影响参与半乳糖代谢基因GAL10转录的情况下,实现对其ncRNA的NT链(non-template strand)的特异性抑制。
除了酿酒酵母菌外,CRISPR-Cas基因编辑工具也成功应用于解脂耶氏酵母(Yarrowialipolytica)[106-107]和毕赤酵母(Pichia pastoris)[108-110]等的基因编辑中。2016年,Schwartz等[106]在解脂耶氏酵母中建立了CRISPR-Cas9基因编辑技术,其单基因打靶效率高达92%。Gao等[107]采用CRISPR-Cas基因编辑技术在解脂耶氏酵母细胞中成功实现了多基因敲除。此外,毕赤酵母作为广泛使用的外源蛋白的表达宿主,其同源重组效率低下是工程菌株改造的重要障碍之一,而CRISPR-Cas为其提供了一种新的更加高效的基因组修饰和改造方法。2016年,Weninger等[108]构建了超过90个包含不同优化密码子的Cas9核酸酶DNA序列以及不同RNA聚合酶启动子的gRNA表达载体,靶向率达到100%。2019年,Gu等[110]从乳酸克鲁维酵母(Kluyveromyces lactis)中分离出包含自主复制序列(autonomously replicating sequences,ARS)的附加型质粒,并开发了基于CRISPR-Cas系统的高效基因编辑方法,与之前报道的使用内源性ARS的方法相比,CRISPR-Cas基因编辑效率提高了10倍。
2.2.2 在丝状真菌中的应用: 丝状真菌产生的丰富次生代谢产物,一方面是人类和动植物的重要致病因素,另一方面又是抗生素等化合物的重要来源[111]。黑曲霉、米曲霉、产黄青霉和里氏木霉等丝状真菌,已经被用于生产多种酶或代谢产物[112]。为提高这些产物的产量,对丝状真菌进行基因改造是非常重要的,为此已建立了多种适合于丝状真菌的基因工程改造方法,但现有的方法存在效率偏低,以及需要引入抗生素或荧光蛋白基因等以实现改良菌株筛选的不足[113],而CRISPR-Cas基因编辑方法,除高效与便捷外,还可在不引入抗生素或荧光标记基因情况下实现单或多基因编辑,因此,近年来CRISPR-Cas基因编辑方法在丝状真菌基因编辑改造中的应用已成为研究热点之一,并成功应用于里氏木霉、烟曲霉、构巢曲霉、黑曲霉、米曲霉、土曲霉、产黄青霉和红曲菌等丝状真菌的基因编辑中。
2015年,CRISPR-Cas基因编辑工具率先被应用于里氏木霉中,成功将外源基因通过同源重组的方式导入目标位点,并实现了多基因的同时插入[114]。另外,在曲霉属(Aspergillus)的丝状真菌中,也有很多采用CRISPR-Cas基因编辑技术进行基因敲除的成功案例。2015年,Fuller等[115]采用CRISPR-Cas基因编辑技术成功敲除了烟曲霉中黑色素(melanin)聚酮合酶基因pksP。同年,采用该技术对构巢曲霉中控制分生孢子颜色的一个基因yA进行破坏,成功使分生孢子的颜色由绿色变成黄色[116]。2016年,Zhang等[117]以MMEJ-CRISPR (microhomology-mediated end joining)方法成功使烟曲霉中的pksP和CnaA基因实现了双突变。Kuivanen等[118]以CRISPR-Cas9基因编辑技术,通过敲除黑曲霉中与半乳糖酸分解代谢相关的基因而使由D-半乳糖醛酸氧化生成的半乳糖酸大量累积。Katayama等[119]将CRISPR-Cas9基因编辑技术成功应用于米曲霉,使参与孢子色素沉着和嘧啶代谢的wA、yA和pyrG基因成功突变。2017年,Sarkari等[120]将土曲霉中编码线粒体载体蛋白的Mtta基因整合到黑曲霉中的pyrG基因特定位点,从而使突变株将顺式乌头酸由线粒体运输到细胞质中。此外,采用CRISPR-Cas9基因编辑工具对产黄青霉的黑曲霉进行无标记基因编辑,成功调控了次级代谢产物的生物合成途径[121]。2020年,高书山课题组构建了一种基于Cas9的双质粒基因编辑方法,即密码子优化后的Cas9质粒和含目的基因上下游同源臂的同源重组质粒,成功实现了红曲菌(Monascus spp.)中桔霉素(citrinin)合成基因簇(> 15 kb)的无痕敲除[122]。
3 ACP在微生物基因编辑中的应用 针对CRISPR-Cas基因编辑过程中的脱靶效应,过去主要是采用注射核糖核蛋白(ribonucleo- proteins,RNP)复合体[123]、引入调控活性结构域[124]或将点突变引入Cas核酸酶[125-126]等方法,通过控制Cas核酸酶的作用以减少基因脱靶。但这些方法存在设计困难,需大量时间进行筛选及优化等不足。ACP的发现为降低CRISPR-Cas基因编辑的脱靶效应提供了一种更加灵活、可调的方法。已知ACP可通过干扰Cas核酸酶活性,阻碍Cas复合物与DNA结合,以及影响crRNA的装载等方式提高CRISPR-Cas基因编辑的准确性和效率,并在动植物细胞的基因编辑中已得到了很好的应用[127-128]。例如,2017年,Shin等[129]证明AcrIIA4可在人类细胞中有效抑制CRISPR-Cas9发挥作用。此外,新开发的基于AcrIIA4并由特征性MicroRNAs控制的Cas蛋白活性开关,能够将SpyCas9 (Cas9 from Streptococcus pyogenes)的活性限定于肝细胞与心肌细胞中[127]。由于ACP对Cas9-sgRNA复合物具有很高的亲和力,从而表现出较强的控制CRISPR-Cas9活性的能力。2019年,Lee等[128]研究发现在成年小鼠中通过腺病毒相关载体(adenovirus associated vector,AAV)与NmeCas9 (Cas9 from Neisseria meningitides)共同递送的AcrIIC3因带有肝脏特异性miR-122结合位点,因此只允许NmeCas9在肝脏细胞中进行基因编辑,而能抑制其在心肌细胞中的编辑。
目前,基于ACP可阻止Cas核酸酶与靶标结合的特性,以酿酒酵母为宿主,已研究开发出生物传感器和合成基因电路(gene synthesis circuit)[130-131],构建了由ACP介导的抗CRISPR表达及其活性诱导方法,且研发出与荧光表达增强耦合的生物传感器[131]。
4 ACP-CRISPR-Cas在微生物基因编辑中的应用前景 目前,虽然CRISPR-Cas基因编辑技术已成功用于包括大多数模式和工业微生物在内的许多微生物的基因改造中,且相比于以前的基因改造方法,该技术表现出高效、便捷、无痕和可同时改造多个基因的优越性,特别是对于同源重组效率低的丝状真菌的基因改造表现出巨大的应用潜力。而且采用该基因编辑工具对微生物中相关基因进行编辑时,基因的编辑效率通常都较高,有的甚至达到100%,也有些基因的编辑效率仅为15%[101]。我们以CRISPR-Cas基因编辑方法对红曲菌中桔霉素合成基因簇进行无痕敲除时[122],敲除效率也仅约为10%。而采用ACP介导的CRISPR-Cas基因编辑方法(ACP-CRISPR-Cas)有望大大提高相关微生物的基因编辑效率。
目前已知通过ACP识别并与被基因编辑靶细菌中的内源性CRISPR-Cas系统作用,可抑制该系统的活性,从而使基于靶细菌内源CRISPR-Cas系统发展而来的基因编辑工具失效或部分失效,以控制基因脱靶,提高其基因编辑效率;也可通过增强CRISPR-Cas基因编辑工具的时序控制,从而编辑以前不能实现基因编辑的细菌和噬菌体[132]。ACP可作为CRISPR-Cas系统的关闭开关,可根据需要在基因编辑过程中,于最适合的时间点阻断CRISPR-Cas的基因编辑功能,以实现基因编辑的调控[129]。另外,研究发现同一个ACP (AcrIIA5、AcrIIC1或AcrVA1)可同时抑制多个直系同源的Cas核酸酶[133],因此,一个ACP有可能同时抑制多个直系同源的Cas核酸酶,甚至有可能构建一种通用的可抑制不同Cas核酸酶活性的ACP。还有,上文提到的应用CRISPR-Cas9基因编辑工具对蓝细菌进行基因编辑时,因Cas9核酸酶高水平表达导致蓝细菌死亡而无法采用该基因编辑方法实现基因编辑的问题[54],也有望通过采用相应ACP调控Cas9蛋白的表达而克服。
为了更加快速和灵活地实现ACP对CRISPR-Cas基因编辑能力的控制,目前已经开发出针对acp转录、转录后修饰和光调控等手段以调控ACP对CRISPR-Cas基因编辑活性和编辑时间点[133-136]。在控制acp转录方面,当Anti-CRISPR相关蛋白(anti-CRISPR-associated protein,Aca)与天然acp基因启动子结合时,可抑制acp转录[132]。由于aca通常与acp在同一个操纵子中,且Aca可下调acp转录水平,因此可通过诱导型启动子调控acp转录从而调节ACP-CRISPR-Cas9基因编辑工具中Cas9核酸酶的产量[131, 133]。在acp转录后修饰方面,采用组织特异性的小RNA (microRNA),通过修饰acrIIA4和acrIIC3等acp转录本3′端的非翻译区,可实现acp转录后调控,从而控制Cas9核酸酶活性[135]。在光调控方面,Bubeck等[135]通过改造ACP,可实现对CRISPR-Cas9基因编辑的光调控。在该系统中,光、氧、电压结构域2 (light-oxygen- voltage sensing domain,LOV2)作为一种光开关蛋白域可插入AcrIIA4中,形成AcrIIA4-LOV杂合体;在没有光激发情况下,该杂合体可抑制SpyCas9活性;在光激活下,LOV2发生构象变化,并使ACP丧失对Cas9的抑制作用。
另外,由于ACP在细菌中普遍存在,例如,已知超过30%的含有CRISPR-Cas系统的铜绿假单胞菌株中包含一个或多个acp或其同源基因[137],大于50%编码Cas9核酸酶的单增李斯特菌中包含抑制Cas9的ACP[138],所以如果细菌中存在编码针对内源CRISPR-Cas系统的acp,那么采用来自外源的CRISPR-Cas基因编辑工具对相关基因进行编辑,可避免ACP对CRISPR-Cas编辑效率的影响[6]。此外,随着耐药性细菌在全球范围内的传播,噬菌体疗法(phage therapy)可能是对抗细菌感染的一种有效方法[139],但病原细菌中CRISPR-Cas系统的存在,大大影响了噬菌体疗法的效果,而通过改造ACP有望克服这种影响。
还有,目前已发现的ACP分子量均较小(约50-200个氨基酸),所以有利于以腺病毒相关载体对其进行递送,实现ACP对CRISPR-Cas基因编辑过程的调控[132]。
总之,尽管目前采用ACP-CRISPR-Cas基因编辑方法对微生物进行基因编辑的研究刚刚起步,但是这种方法在提高微生物基因编辑的效率方面已经表现出了巨大的应用前景。针对采用CRISPR-Cas基因编辑方法对红曲菌中桔霉素合成基因簇进行无痕敲除时[122],其敲除效率仅约为10%的问题,目前我们正研究采用ACP-CRISPR- Cas基因编辑方法提高其基因编辑效率。
References
| [1] | Yosef I, Goren MG, Qimron U. Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli. Nucleic Acids Research, 2012, 40(12): 5569-5576. DOI:10.1093/nar/gks216 |
| [2] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [3] | Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964. DOI:10.1126/science.1159689 |
| [4] | Garneau JE, Dupuis Mè, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature, 2010, 468(7320): 67-71. DOI:10.1038/nature09523 |
| [5] | Gasiunas G, Barrangou R, Horvath P, Siksnys V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): E2579-E2586. DOI:10.1073/pnas.1208507109 |
| [6] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [7] | Bondy-Denomy J, Pawluk A, Maxwell KL, Davidson AR. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system. Nature, 2013, 493(7432): 429-432. DOI:10.1038/nature11723 |
| [8] | Pawluk A, Shah M, Mejdani M, Calmettes C, F.Moraes T, Maxwell K, Davidon A. Disabling a Type I-E CRISPR-Cas nuclease with a bacteriophage-encoded anti-CRISPR protein. Mbio, 2017, 8(6): e01751-17. |
| [9] | Wang C, Wang KJ. Advances in CRISPR-Cas-mediated genome editing system in plants. Chinese Journal of Biotechnology, 2017, 33(10): 1712-1722. (in Chinese) 王春, 王克剑. CRISPR-Cas系统在植物基因组编辑中的研究进展. 生物工程学报, 2017, 33(10): 1712-1722. |
| [10] | Vats S, Kumawat S, Kumar V, Patil GB, Joshi T, Sonah H, Sharma TR, Deshmukh R. Genome editing in plants: exploration of technological advancements and challenges. Cell, 2019, 8(11): 1386. DOI:10.3390/cells8111386 |
| [11] | Zhu HC, Li C, Gao CX. Applications of CRISPR-Cas in agriculture and plant biotechnology. Nature Reviews Molecular Cell Biology, 2020, 21(11): 661-677. DOI:10.1038/s41580-020-00288-9 |
| [12] | Shapiro RS, Chavez A, Collins JJ. CRISPR-based genomic tools for the manipulation of genetically intractable microorganisms. Nature Reviews Microbiology, 2018, 16(6): 333-339. DOI:10.1038/s41579-018-0002-7 |
| [13] | Yin CM, Fan X, Shi D, Gao H. Research progress of CRISPR/Cas9 system in filamentous fungi. Biotechnology Bulletin, 2017, 33(03): 58-65. (in Chinese) 殷朝敏, 范秀芝, 史徳芳, 高虹. CRISPR/Cas基因编辑技术及其在真菌中的应用. 生物技术通报, 2017, 33(03): 58-65. |
| [14] | Meng J, Liu D, Huang C, Han B, Chen J. Development and application of CRISPR/Cas genome editing system in the construction of prokaryotic microbial cell factories. Microbiology China, 2019, 46(10): 2730-2742. (in Chinese) 孟娇, 刘丁玉, 黄灿, 韩北忠, 陈晶瑜. CRISPR/Cas基因编辑系统在原核微生物细胞工厂构建中的开发与应用. 微生物学通报, 2019, 46(10): 2730-2742. |
| [15] | Wang Q, Yang H, Wang Z, Chen C. High specificity CRISPR/Cas system and anti-CRISPR proteins. Chemistry of Life, 2020, 40(10): 1760-1770. (in Chinese) 王钱繁, 杨海涛, 王泽方, 陈成. 高特异性CRISPR-Cas系统及其Acrs. 生命的化学, 2020, 40(10): 1760-1770. |
| [16] | Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [17] | Mojica FJ, Díez-Villase?or C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria. Molecular Microbiology, 2000, 36(1): 244-246. DOI:10.1046/j.1365-2958.2000.01838.x |
| [18] | Jansen R, Embden JDAV, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [19] | Mojica FJM, Díez-Villase?or C, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [20] | Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology: Reading, England, 2005, 151(Pt 3): 653-663. |
| [21] | Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8): 2551-2561. DOI:10.1099/mic.0.28048-0 |
| [22] | Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964. DOI:10.1126/science.1159689 |
| [23] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. DOI:10.1126/science.1165771 |
| [24] | Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DBT, Shmakov S, Makarova KS, Semenova E, Minakhin L, Severinov K, Regev A, Lander ES, Koonin EV, Zhang F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [25] | Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao YJ, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [26] | Sapranauskas R, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Research, 2011, 39(21): 9275-9282. DOI:10.1093/nar/gkr606 |
| [27] | Heler R, Samai P, Modell JW, Weiner C, Goldberg GW, Bikard D, Marraffini LA. Cas9 specifies functional viral targets during CRISPR-Cas adaptation. Nature, 2015, 519(7542): 199-202. DOI:10.1038/nature14245 |
| [28] | Ishino Y, Krupovic M, Forterre P. History of CRISPR-Cas from encounter with a mysterious repeated sequence to genome editing technology. Journal of Bacteriology, 2018, 200(7): e00580-17. |
| [29] | Haft DH, Selengut J, Mongodin EF, Nelson KE. A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. PLoS Computational Biology, 2005, 1(6): e60. DOI:10.1371/journal.pcbi.0010060 |
| [30] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [31] | Makarova KS, Wolf YI, Alkhnbashi OS, Costa F, Shah SA, Saunders SJ, Barrangou R, Brouns SJJ, Charpentier E, Haft DH, Horvath P, Moineau S, Mojica FJM, Terns RM, Terns MP, White MF, Yakunin AF, Garrett RA, van der Oost J, Backofen R, Koonin EV. An updated evolutionary classification of CRISPR-Cas systems. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [32] | Pawluk A, Bondy-Denomy J, Cheung VHW, Maxwell KL, Davidson AR. A new group of phage anti-CRISPR genes inhibits the type I-E CRISPR-Cas system of Pseudomonas aeruginosa. mBio, 2014, 5(2): e00896. |
| [33] | Pawluk A, Staals RHJ, Taylor C, Watson BNJ, Saha S, Fineran PC, Maxwell KL, Davidson AR. Inactivation of CRISPR-Cas systems by anti-CRISPR proteins in diverse bacterial species. Nature Microbiology, 2016, 1(8): 16085. DOI:10.1038/nmicrobiol.2016.85 |
| [34] | Rauch BJ, Silvis MR, Hultquist JF, Waters CS, McGregor MJ, Krogan NJ, Bondy-Denomy J. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell, 2017, 168(1/2): 150-158. |
| [35] | Borges AL, Davidson AR, Bondy-Denomy J. The discovery, mechanisms, and evolutionary impact of anti-CRISPRs. Annual Review of Virology, 2017, 4(1): 37-59. DOI:10.1146/annurev-virology-101416-041616 |
| [36] | Stanley SY, Maxwell KL. Phage-encoded anti-CRISPR defenses. Annual Review of Genetics, 2018, 52: 445-464. DOI:10.1146/annurev-genet-120417-031321 |
| [37] | Bondy-Denomy J, Davidson AR, Doudna JA, Fineran PC, Maxwell KL, Moineau S, Peng X, Sontheimer EJ, Wiedenheft B. A unified resource for tracking anti-CRISPR names. The CRISPR Journal, 2018, 1: 304-305. DOI:10.1089/crispr.2018.0043 |
| [38] | Trasanidou D, Gerós AS, Mohanraju P, Nobrega FL, Staals RHJ. Keeping CRISPR in check: diverse mechanisms of phage-encoded anti-CRISPRs. FEMS Microbiology Letters, 2019, 366: 1709. |
| [39] | Knott GJ, Thornton BW, Lobba MJ, Liu JJ, Al-Shayeb B, Watters KE, Doudna JA. Broad-spectrum enzymatic inhibition of CRISPR-Cas12a. Nature Structural & Molecular Biology, 2019, 26(4): 315-321. |
| [40] | Dong LY, Guan XY, Li NN, Zhang F, Zhu YW, Ren K, Yu L, Zhou FX, Han ZF, Gao N, Huang ZW. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology, 2019, 26(4): 308-314. |
| [41] | Bondy-Denomy J, Garcia B, Strum S, Du MJ, Rollins MF, Hidalgo-Reyes Y, Wiedenheft B, Maxwell KL, Davidson AR. Multiple mechanisms for CRISPR-Cas inhibition by anti-CRISPR proteins. Nature, 2015, 526(7571): 136-139. DOI:10.1038/nature15254 |
| [42] | Dong D, Guo M, Wang S, Zhu Y, Wang S, Xiong Z, Yang J, Xu Z, Huang Z. Structural basis of CRISPR-SpyCas9 inhibition by an anti-CRISPR protein. Nature, 2017, 546(7658): 436-439. DOI:10.1038/nature22377 |
| [43] | Jiang FG, Liu JJ, Osuna BA, Xu M, Berry JD, Rauch BJ, Nogales E, Bondy-Denomy J, Doudna JA. Temperature-responsive competitive inhibition of CRISPR-Cas9. Molecular Cell, 2019, 73(3): 601-610. DOI:10.1016/j.molcel.2018.11.016 |
| [44] | Harrington LB, Doxzen KW, Ma EB, Liu JJ, Knott GJ, Edraki A, Garcia B, Amrani N, Chen JS, Cofsky JC, Kranzusch PJ, Sontheimer EJ, Davidson AR, Maxwell KL, Doudna JA. A broad-spectrum inhibitor of CRISPR-cas9. Cell, 2017, 170(6): 1224-1233. DOI:10.1016/j.cell.2017.07.037 |
| [45] | Liu H, Yu C, Feng DX, Cheng T, Meng X, Liu W, Zou HB, Xian M. Production of extracellular fatty acid using engineered Escherichia coli. Microbial Cell Factories, 2012, 11: 41. DOI:10.1186/1475-2859-11-41 |
| [46] | Jiang WY, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [47] | Jiang Y, Chen B, Duan CL, Sun BB, Yang JJ, Yang S. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [48] | Xia J, Zheng MG, Wang L, Sun CJ, Zheng L, Zhu JB. Knocking out phosphoenolpyruvate carboxylase gene by CRISPR/Cas and its influence on fatty acid metabolism in Escherichia coli. Microbiology, 2016, 43(8): 1864-1871. (in Chinese) 夏军, 郑明刚, 王玲, 孙承君, 郑立, 祝建波. 运用CRISPR/Cas系统敲除大肠杆菌磷酸烯醇式丙酮酸羧化酶基因及其对脂肪酸代谢的影响. 微生物学通报, 2016, 43(8): 1864-1871. |
| [49] | Chung ME, Yeh IH, Sung LY, Wu MY, Chao YP, Ng IS, Hu YC. Enhanced integration of large DNA into E. coli chromosome by CRISPR/Cas9. Biotechnology and Bioengineering, 2017, 114(11): 172-183. |
| [50] | Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [51] | Wu JJ, Du GC, Chen J, Zhou JW. Enhancing flavonoid production by systematically tuning the central metabolic pathways based on a CRISPR interference system in Escherichia coli. Scientific Reports, 2015, 5: 13477. DOI:10.1038/srep13477 |
| [52] | Liang JL, Guo LQ, Lin JF, He ZQ, Cai FJ, Chen JF. A novel process for obtaining pinosylvin using combinatorial bioengineering in Escherichia coli. World Journal of Microbiology and Biotechnology, 2016, 32(6): 1-10. |
| [53] | Kim SK, Seong W, Han GH, Lee DH, Lee SG. CRISPR interference-guided multiplex repression of endogenous competing pathway genes for redirecting metabolic flux in Escherichia coli. Microbial Cell Factories, 2017, 16(1): 188. DOI:10.1186/s12934-017-0802-x |
| [54] | McNeely K, Xu Y, Bennette N, Bryant DA, Dismukes GC. Redirecting reductant flux into hydrogen production via metabolic engineering of fermentative carbon metabolism in a cyanobacterium. Applied and Environmental Microbiology, 2010, 76(15): 5032-5038. DOI:10.1128/AEM.00862-10 |
| [55] | Yao L, Qi FX, Tan XM, Lu XF. Improved production of fatty alcohols in cyanobacteria by metabolic engineering. Biotechnology for Biofuels, 2014, 7(1): 1-9. DOI:10.1186/1754-6834-7-1 |
| [56] | Wendt KE, Ungerer J, Cobb RE, Zhao HM, Pakrasi HB. CRISPR/Cas9 mediated targeted mutagenesis of the fast growing cyanobacterium Synechococcus elongatus UTEX 2973. Microbial Cell Factories, 2016, 15(1): 1-8. DOI:10.1186/s12934-015-0402-6 |
| [57] | Ungerer J, Pakrasi HB. Cpf1 is A versatile tool for CRISPR genome editing across diverse species of cyanobacteria. Scientific Reports, 2016, 6: 39681. DOI:10.1038/srep39681 |
| [58] | Knoot CJ, Biswas S, Pakrasi HB. Tunable repression of key photosynthetic processes using Cas12a CRISPR interference in the fast-growing cyanobacterium Synechococcus sp. UTEX 2973. ACS Synthetic Biology, 2020, 9(1): 132-143. DOI:10.1021/acssynbio.9b00417 |
| [59] | Shabestary K, Anfelt J, Ljungqvist E, Jahn M, Yao L, Hudson EP. Targeted repression of essential genes to arrest growth and increase carbon partitioning and biofuel titers in cyanobacteria. ACS Synthetic Biology, 2018, 7(7): 1669-1675. DOI:10.1021/acssynbio.8b00056 |
| [60] | Choi SY, Woo HM. CRISPRi-dCas12a: A dCas12a-mediated CRISPR interference for repression of multiple genes and metabolic engineering in cyanobacteria. ACS Synthetic Biology, 2020, 18, 9(9): 2351-2361. |
| [61] | Higo A, Ehira S. Spatiotemporal gene repression system in the heterocyst-forming multicellular cyanobacterium Anabaena sp. PCC 7120. ACS Synthetic Biology, 2019, 8(4): 641-646. DOI:10.1021/acssynbio.8b00496 |
| [62] | Higo A, Isu A, Fukaya Y, Ehira S, Hisabori T. Application of CRISPR interference for metabolic engineering of the heterocyst-forming multicellular cyanobacterium Anabaena sp. PCC 7120. Plant & Cell Physiology, 2018, 59(1): 119-127. |
| [63] | Liu G, Chater KF, Chandra G, Niu G, Tan H. Molecular regulation of antibiotic biosynthesis in Streptomyces. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-143. DOI:10.1128/MMBR.00054-12 |
| [64] | Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology, 2011, 4(2): 207-215. DOI:10.1111/j.1751-7915.2010.00219.x |
| [65] | Cobb RE, Wang YJ, Zhao HM. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system. ACS Synthetic Biology, 2015, 4(6): 723-728. DOI:10.1021/sb500351f |
| [66] | Jiang WJ, Zhu TF. Targeted isolation and cloning of 100-kb microbial genomic sequences by Cas9-assisted targeting of chromosome segments. Nature Protocols, 2016, 11(5): 960-975. DOI:10.1038/nprot.2016.055 |
| [67] | Jiang WJ, Zhao XJ, Gabrieli T, Lou CB, Ebenstein Y, Zhu TF. Cas9-assisted targeting of chromosome segments CATCH enables one-step targeted cloning of large gene clusters. Nature Communications, 2015, 6: 8101. DOI:10.1038/ncomms9101 |
| [68] | Li L, Zheng GS, Chen J, Ge M, Jiang WH, Lu YH. Multiplexed site-specific genome engineering for overproducing bioactive secondary metabolites in actinomycetes. Metabolic Engineering, 2017, 40: 80-92. DOI:10.1016/j.ymben.2017.01.004 |
| [69] | Zhang M, Wong F, Wang Y, Luo S, Heng E, Cobb RE, Ang EL. CRISPR-Cas9 strategy for activation of silent Streptomyces biosynthetic gene clusters. Nature Chemical Biology, 2017, 13(6): 607-609. DOI:10.1038/nchembio.2341 |
| [70] | Liu Y, Wei WP, Ye BC. High GC content Cas9-mediated genome-editing and biosynthetic gene cluster activation in Saccharopolyspora erythraea. ACS Synthetic Biology, 2018, 7(5): 1338-1348. DOI:10.1021/acssynbio.7b00448 |
| [71] | Petchiappan A, Chatterji D. Antibiotic resistance: current perspectives. ACS Omega, 2017, 2(10): 7400-7409. DOI:10.1021/acsomega.7b01368 |
| [72] | Chen WZ, Zhang YF, Yeo WS, Bae T, Ji QJ. Rapid and efficient genome editing in Staphylococcus aureus by using an engineered CRISPR/Cas9 system. Journal of the American Chemical Society, 2017, 139(10): 3790-3795. DOI:10.1021/jacs.6b13317 |
| [73] | Gu TN, Zhao SQ, Pi YS, Chen WZ, Chen CY, Liu Q, Li M, Han DL, Ji QJ. Highly efficient base editing in Staphylococcus aureus using an engineered CRISPR RNA-guided cytidine deaminase. Chemical Science, 2018, 9(12): 3248-3253. DOI:10.1039/C8SC00637G |
| [74] | Choudhary E, Thakur P, Pareek M, Agarwal N. Gene silencing by CRISPR interference in Mycobacteria. Nature Communications, 2015, 6: 6267. DOI:10.1038/ncomms7267 |
| [75] | Singh AK, Carette X, Potluri LP, Sharp JD, Xu RF, Prisic S, Husson RN. Investigating essential gene function in Mycobacterium tuberculosis using an efficient CRISPR interference system. Nucleic Acids Research, 2016, 44(18): e143. DOI:10.1093/nar/gkw625 |
| [76] | Rock JM, Hopkins FF, Chavez A, Diallo M, Chase MR, Gerrick ER, Pritchard JR, Church GM, Rubin EJ, Sassetti CM, Schnappinger D, Fortune SM. Programmable transcriptional repression in Mycobacteria using an orthogonal CRISPR interference platform. Nature Microbiology, 2017, 2: 16274. DOI:10.1038/nmicrobiol.2016.274 |
| [77] | Sun BB, Yang JJ, Yang S, Ye RD, Chen DJ, Jiang Y. A CRISPR-Cpf1-assisted non-homologous end joining genome editing system of Mycobacterium smegmatis. Biotechnology Journal, 2018, 13(9): e1700588. DOI:10.1002/biot.201700588 |
| [78] | Aparicio T, de Lorenzo V, Martínez-García E. CRISPR/Cas9-based counterselection boosts recombineering efficiency in Pseudomonas putida. Biotechnology Journal, 2018, 13(5): e1700161. DOI:10.1002/biot.201700161 |
| [79] | Sun J, Wang QZ, Jiang Y, Wen ZQ, Yang LR, Wu JP, Yang S. Genome editing and transcriptional repression in Pseudomonas putida KT2440 via the type Ⅱ CRISPR system. Microbial Cell Factories, 2018, 17(1): 41. DOI:10.1186/s12934-018-0887-x |
| [80] | Tan S, Reisch CR, Prather KLJ. A robust CRISPR interference gene repression system in Pseudomonas. Journal of Bacteriology, 2018, 200(7): e00575-17. |
| [81] | Shen J, Zhou JT, Chen GQ, Xiu ZL. Efficient genome engineering of a virulent Klebsiella bacteriophage using CRISPR-Cas9. Journal of Virology, 2018, 92(17): e00534-18. |
| [82] | Wang JX, Zhao P, Li Y, Xu LD, Tian PF. Engineering CRISPR interference system in Klebsiella pneumoniae for attenuating lactic acid synthesis. Microbial Cell Factories, 2018, 17(1): 1-12. DOI:10.1186/s12934-017-0850-2 |
| [83] | Yan M, Yan H, Ren G, Zhao J, Guo X, Sun Y. CRISPR-Cas12a-assisted recombineering in bacteria. Applied and Environmental Microbiology, 2017, 83(17): e00947-17. |
| [84] | Li K, Cai D, Wang Z, He Z, Chen S. Development of an efficient genome editing tool in Bacillus licheniformis using CRISPR-Cas9 nickase. Applied and Environmental Microbiology, 2018, 84(6): e02608-17. |
| [85] | Zhan YY, Xu Y, Zheng PL, He M, Sun SH, Wang D, Cai DB, Ma X, Chen SW. Establishment and application of multiplexed CRISPR interference system in Bacillus licheniformis. Applied Microbiology and Biotechnology, 2020, 104(1): 391-403. DOI:10.1007/s00253-019-10230-5 |
| [86] | Song H, Li M. Progress and application in molecular biology of yeast. Journal of Northwest Institute of Light Industry, 2001(4): 78-83. (in Chinese) 宋宏新, 李敏康. 酵母分子生物学理论及应用研究进展. 西北轻工业学院学报, 2001(4): 78-83. DOI:10.3969/j.issn.1000-5811.2001.04.021 |
| [87] | Liu Q, Yu L. Yeast: a model organism. Chemistry of Life, 2000, 20(2): 61-65. (in Chinese) 刘擎, 余龙. 酵母: 一种模式生物. 生命的化学, 2000, 20(2): 61-65. DOI:10.3969/j.issn.1000-1336.2000.02.006 |
| [88] | He L, Zhao X, Bai F. Engineering industrial Saccharomyces cerevisiae strain with the FLO1-derivative gene isolated from the flocculating yeast SPSC01 for constitutive flocculation and fuel ethanol production. Applied Energy, 2012, 100: 33-40. DOI:10.1016/j.apenergy.2012.03.052 |
| [89] | Horwitz AA, Walter JM, Schubert MG, Hawkins K, Platt DM, Hernday AD, Mahatdejkul-Meadows T, Chandran SS, Newman JD. Efficient multiplexed integration of synergistic alleles and metabolic pathways in yeasts via CRISPR-Cas. Cell Systems, 2015, 1(1): 88-96. DOI:10.1016/j.cels.2015.02.001 |
| [90] | Horwitz AA, Walter JM, Schubert MG, Kung SH, Hawkins K, Platt DM, Hernday AD, Mahatdejkul-Meadows T, Szeto W, Chandran SS, Newman JD. Efficient multiplexed integration of synergistic alleles and metabolic pathways in yeasts via CRISPR-cas. Cell Systems, 2015, 1(1): 88-96. DOI:10.1016/j.cels.2015.02.001 |
| [91] | Jako?iūnas T, Bonde I, Herrg?rd M, Harrison SJ, Kristensen M, Pedersen LE, Jensen MK, Keasling JD. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae. Metabolic Engineering, 2015, 28: 213-222. DOI:10.1016/j.ymben.2015.01.008 |
| [92] | DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research, 2013, 41(7): 4336-4343. DOI:10.1093/nar/gkt135 |
| [93] | Ronda C, Maury J, Jako?iunas T, Baallal Jacobsen SA, Germann SM, Harrison SJ, Borodina I, Keasling JD, Jensen MK, Nielsen AT. CrEdit: CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae. Microbial Cell Factories, 2015, 14(1): 1-11. DOI:10.1186/s12934-014-0183-3 |
| [94] | Li ZH, Liu M, Wang FQ, Wei DZ. Cpf1-assisted efficient genomic integration of in vivo assembled DNA parts in Saccharomyces cerevisiae. Biotechnology Letters, 2018, 40(8): 1253-1261. DOI:10.1007/s10529-018-2574-8 |
| [95] | Verwaal R, Buiting-Wiessenhaan N, Dalhuijsen S, Roubos JA. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast: Chichester, England, 2018, 35(2): 201-211. DOI:10.1002/yea.3278 |
| [96] | Xu K, Ren CH, Liu ZT, Zhang T, Zhang TT, Li D, Wang L, Yan Q, Guo LJ, Shen JC, Zhang ZY. Efficient genome engineering in eukaryotes using Cas9 from Streptococcus thermophilus. Cellular and Molecular Life Sciences, 2015, 72(2): 383-399. DOI:10.1007/s00018-014-1679-z |
| [97] | Ryan OW, Skerker JM, Maurer MJ, Li X, Tsai JC, Poddar S, Lee ME, DeLoache W, Dueber JE, Arkin AP, Cate JHD. Selection of chromosomal DNA libraries using a multiplex CRISPR system in Saccharomyces cerevisiae. bioRxiv, 2014. DOI:10.1101/007310 |
| [98] | Bao ZH, Xiao H, Liang J, Zhang L, Xiong X, Sun N, Si T, Zhao HM. Homology-integrated CRISPR-Cas (HI-CRISPR) system for one-step multigene disruption in Saccharomyces cerevisiae. ACS Synthetic Biology, 2015, 4(5): 585-594. DOI:10.1021/sb500255k |
| [99] | ?wiat MA, Dashko S, Wijsman M, Marc Daran JM, Lapujade PD. FnCpf1:a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research, 2017, 45(21): 12585-12598. DOI:10.1093/nar/gkx1007 |
| [100] | Liu K, Yuan X, Liang L, Fang J. Using CRISPR/Cas9 for multiplex genome engineering to optimize the ethanol metabolic pathway in Saccharomyces cerevisiae. Biochemical Engineering Journal, 2019, 145: 120-126. DOI:10.1016/j.bej.2019.02.017 |
| [101] | Stovicek V, Borodina I, Forster J. CRISPR-Cas system enables fast and simple genome editing of industrial Saccharomyces cerevisiae strains. Metabolic Engineering Communications, 2015, 2: 13-22. DOI:10.1016/j.meteno.2015.03.001 |
| [102] | Zhang G, Kong I, Kim H, Liu J, Jamie H, Jin Y. Construction of a quadruple auxotrophic mutant of an industrial polyploid Saccharomyces cerevisiae strain by using RNA-guided Cas9 nuclease. Applied & Environmental Microbiology, 2014, 80(24): 7694-7701. |
| [103] | Lian JZ, Bao ZH, Hu SM, Zhao HM. Engineered CRISPR/Cas9 system for multiplex genome engineering of polyploid industrial yeast strains. Biotechnology and Bioengineering, 2018, 115(6): 1630-1635. DOI:10.1002/bit.26569 |
| [104] | Farzadfard F, Perli SD, Lu TK. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology, 2013, 2(10): 604-613. DOI:10.1021/sb400081r |
| [105] | Gilbert LA, Larson MH, Morsut L, Liu ZR, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451. DOI:10.1016/j.cell.2013.06.044 |
| [106] | Lenstra TL, Coulon A, Chow CC, Larson DR. Single-molecule imaging reveals a switch between spurious and functional ncRNA transcription. Molecular Cell, 2015, 60(4): 597-610. DOI:10.1016/j.molcel.2015.09.028 |
| [107] | Schwartz CM, Hussain MS, Blenner M, Wheeldon I. Synthetic RNA polymerase Ⅲ promoters facilitate high-efficiency CRISPR-Cas9-mediated genome editing in Yarrowia lipolytica. ACS Synthetic Biology, 2016, 5(4): 356-359. DOI:10.1021/acssynbio.5b00162 |
| [108] | Gao SL, Tong YY, Wen ZQ, Zhu L, Ge M, Chen DJ, Jiang Y, Yang S. Multiplex gene editing of the Yarrowia lipolytica genome using the CRISPR-Cas9 system. Journal of Industrial Microbiology & Biotechnology, 2016, 43(8): 1085-1093. DOI:10.1007/s10295-016-1789-8 |
| [109] | Weninger A, Hatzl AM, Schmid C, Vogl T, Glieder A. Combinatorial optimization of CRISPR/Cas9 expression enables precision genome engineering in the methylotrophic yeast Pichia pastoris. Journal of Biotechnology, 2016, 235: 139-149. DOI:10.1016/j.jbiotec.2016.03.027 |
| [110] | Hou CL, Yang YK, Xing Y, Zhan CJ, Liu GQ, Liu XX, Liu CL, Zhan JL, Xu DH, Bai ZH. Targeted editing of transcriptional activator MXR1 on the Pichia pastoris genome using CRISPR/Cas9 technology. Yeast: Chichester, England, 2020, 37(4): 305-312. DOI:10.1002/yea.3462 |
| [111] | Gu Y, Gao JC, Cao MF, Dong C, Lian JZ, Huang L, Cai J, Xu ZN. Construction of a series of episomal plasmids and their application in the development of an efficient CRISPR/Cas9 system in Pichia pastoris. World Journal of Microbiology and Biotechnology, 2019, 35(6): 1-10. DOI:10.1007/s11274-019-2654-5 |
| [112] | Evidente A, Kornienko A, Cimmino A, Andolfi A, Lefranc F, Mathieu V, Kiss R. Fungal metabolites with anticancer activity. Natural Product Reports, 2014, 31(5): 617-627. DOI:10.1039/C3NP70078J |
| [113] | Punt PJ, van Biezen N, Conesa A, Albers A, Mangnus J, van den Hondel C. Filamentous fungi as cell factories for heterologous protein production. Trends in Biotechnology, 2002, 20(5): 200-206. DOI:10.1016/S0167-7799(02)01933-9 |
| [114] | Jiang DW, Zhu W, Wang YC, Sun C, Zhang KQ, Yang JK. Molecular tools for functional genomics in filamentous fungi: recent advances and new strategies. Biotechnology Advances, 2013, 31(8): 1562-1574. DOI:10.1016/j.biotechadv.2013.08.005 |
| [115] | Liu R, Chen L, Jiang YP, Zhou ZH, Zou G. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR/Cas9 system. Cell Discovery, 2015, 1: 15007. |
| [116] | Fuller KK, Chen S, Loros JJ, Dunlap JC. Development of the CRISPR/Cas9 system for targeted gene disruption in Aspergillus fumigatus. Eukaryotic Cell, 2015, 14(11): 1073-1080. DOI:10.1128/EC.00107-15 |
| [117] | N?dvig CS, Nielsen JB, Kogle ME, Mortensen UH. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS ONE, 2015, 10(7): e0133085. DOI:10.1371/journal.pone.0133085 |
| [118] | Zhang C, Meng XH, Wei XL, Lu L. Highly efficient CRISPR mutagenesis by microhomology-mediated end joining in Aspergillus fumigatus. Fungal Genetics and Biology, 2016, 86: 47-57. DOI:10.1016/j.fgb.2015.12.007 |
| [119] | Kuivanen J, Wang YMJ, Richard P. Engineering Aspergillus niger for galactaric acid production: elimination of galactaric acid catabolism by using RNA sequencing and CRISPR/Cas9. Microbial Cell Factories, 2016, 15(1): 1-9. DOI:10.1186/s12934-015-0402-6 |
| [120] | Katayama T, Tanaka Y, Okabe T, Nakamura H, Fujii W, Kitamoto K, Maruyama JI. Development of a genome editing technique using the CRISPR/Cas9 system in the industrial filamentous fungus Aspergillus oryzae. Biotechnology Letters, 2016, 38(4): 637-642. DOI:10.1007/s10529-015-2015-x |
| [121] | Sarkari P, Marx H, Blumhoff ML, Mattanovich D, Sauer M, Steiger MG. An efficient tool for metabolic pathway construction and gene integration for Aspergillus niger. Bioresource Technology, 2017, 245(Pt B): 1327-1333. |
| [122] | Pohl C, Kiel JAKW, Driessen AJM, Bovenberg RAL, Nygard Y. CRISPR/Cas9 based genome editing of Penicillium chrysogenum. ACS Synthetic Biology, 2016, 5(7): 754-764. DOI:10.1021/acssynbio.6b00082 |
| [123] | Liu WW, An CY, Shu X, Meng XX, Yao YP, Zhang J, Chen FS, Xiang H, Yang SY, Gao X, Gao SS. A dual-plasmid CRISPR/cas system for mycotoxin elimination in polykaryotic industrial fungi. ACS Synthetic Biology, 2020, 9(8): 2087-2095. DOI:10.1021/acssynbio.0c00178 |
| [124] | Kim S, Kim D, Cho SW, Kim J, Kim JS. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research, 2014, 24(6): 1012-1019. DOI:10.1101/gr.171322.113 |
| [125] | Senturk S, Shirole NH, Nowak DG, Corbo V, Pal D, Vaughan A, Tuveson DA, Trotman LC, Kinney JB, Sordella R. Rapid and tunable method to temporally control gene editing based on conditional Cas9 stabilization. Nature Communications, 2017, 8: 14370. DOI:10.1038/ncomms14370 |
| [126] | Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen N, Joung JK, Zheng Z. High-fidelity CRISPR-Cas9 variants with undetectable genome-wide off-targets. Nature, 2016, 529(7587): 490-495. DOI:10.1038/nature16526 |
| [127] | Slaymaker IM, Gao LY, Zetsche B, Scott DA, Yan WX, Zhang F. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351(6268): 84-88. DOI:10.1126/science.aad5227 |
| [128] | Hirosawa M, Fujita Y, Saito H. Cell-type-specific CRISPR activation with MicroRNA-responsive AcrllA4 switch. ACS Synthetic Biology, 2019, 8(7): 1575-1582. DOI:10.1021/acssynbio.9b00073 |
| [129] | Lee J, Mou HW, Ibraheim R, Liang SQ, Liu PP, Xue W, Sontheimer EJ. Tissue-restricted genome editing in vivo specified by microRNA-repressible anti-CRISPR proteins. RNA, 2019, 25(11): 1421-1431. DOI:10.1261/rna.071704.119 |
| [130] | Shin J, Jiang FG, Liu JJ, Bray NL, Rauch BJ, Baik SH, Nogales E, Bondy-Denomy J, Corn JE, Doudna JA. Disabling Cas9 by an anti-CRISPR DNA mimic. Science Advances, 2017, 3(7): e1701620. DOI:10.1126/sciadv.1701620 |
| [131] | Nakamura M, Srinivasan P, Chavez M, Carter MA, Dominguez AA, La Russa M, Lau MB, Abbott TR, Xu XS, Zhao DH, Gao YC, Kipniss NH, Smolke CD, Bondy-Denomy J, Qi LS. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications, 2019, 10(1): 194. DOI:10.1038/s41467-018-08158-x |
| [132] | Jing L, Zeng L, Aleksandr C, Mario AM. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering, 2018, 12(1): 1-14. DOI:10.1186/s13036-017-0092-1 |
| [133] | Marino ND, Pinilla-Redondo R, Cs?rg? B, Bondy-Denomy J. Anti-CRISPR protein applications: natural brakes for CRISPR-Cas technologies. Nature Methods, 2020, 17(5): 471-479. DOI:10.1038/s41592-020-0771-6 |
| [134] | Marino ND, Zhang JY, Borges AL, Sousa AA, Leon LM, Rauch BJ, Walton RT, Berry JD, Joung JK, Kleinstiver BP, Bondy-Denomy J. Discovery of widespread type I and type V CRISPR-Cas inhibitors. Science, 2018, 362(6411): 240-242. DOI:10.1126/science.aau5174 |
| [135] | Hoffmann MD, Aschenbrenner S, Grosse S, Rapti K, Domenger C, Fakhiri J, Mastel M, B?rner K, Eils R, Grimm D, Niopek D. Cell-specific CRISPR-Cas9 activation by microRNA-dependent expression of anti-CRISPR proteins. Nucleic Acids Research, 2019, 47(13): e75. DOI:10.1093/nar/gkz271 |
| [136] | Bubeck F, Hoffmann MD, Harteveld Z, Aschenbrenner S, Bietz A, Waldhauer MC, B?rner K, Fakhiri J, Schmelas C, Dietz L, Grimm D, Correia BE, Eils R, Niopek D. Engineered anti-CRISPR proteins for optogenetic control of CRISPR-Cas9. Nature Methods, 2018, 15(11): 924-927. DOI:10.1038/s41592-018-0178-9 |
| [137] | Stanley SY, Borges AL, Chen KH, Swaney DL. Anti-CRISPR-associated proteins are crucial repressors of anti-CRISPR transcription. Cell, 2019, 178(6): 1452-1464. DOI:10.1016/j.cell.2019.07.046 |
| [138] | van Belkum A, Soriaga LB, LaFave MC, Akella S, Veyrieras JB, Barbu EM, Shortridge D, Blanc B, Hannum G, Zambardi G, Miller K, Enright MC, Mugnier N, Brami D, Schicklin S, Felderman M, Schwartz AS, Richardson TH, Peterson TC, Hubby B, Cady KC. Phylogenetic distribution of CRISPR-cas systems in antibiotic-resistant Pseudomonas aeruginosa. mBio, 2015, 6(6): e01796-e01715. |
| [139] | Rauch BJ, Silvis MR, Hultquist JF, McGregor MJ, Krogan NJ, Bondy-Denomy J. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell, 2017, 168(1/2): 150-158. |
| [140] | Nobrega FL, Costa AR, Kluskens LD, Azeredo J. Revisiting phage therapy: new applications for old resources. Trends in Microbiology, 2015, 23(4): 185-191. DOI:10.1016/j.tim.2015.01.006 |
