刘潇1,2, 张正2,3, 王丽芳2, 于永翔2, 王印庚2,3

 , 廖梅杰2,3, 谢国驷2, 邢婧1, 张浩2, 王凯2, 许悦2
, 廖梅杰2,3, 谢国驷2, 邢婧1, 张浩2, 王凯2, 许悦2 1. 中国海洋大学水产学院, 水产动物病害与免疫学实验室, 山东 青岛 266003;
2. 中国水产科学研究院黄海水产研究所, 农业农村部海水养殖病害防治重点实验室, 山东 青岛 266071;
3. 海洋渔业科学与食品产出过程功能实验室, 青岛海洋科学与技术试点国家实验室, 山东 青岛 266237
收稿日期:2020-09-18;修回日期:2021-01-21;网络出版日期:2021-01-29
基金项目:国家重点研发计划课题(2019YFD0900102);山东省重大科技创新工程项目(2018YFJH0703);山东省泰山产业领军人才项目(LJNY201802)
*通信作者:王印庚, Tel: +86-532-85817991; E-mail: wangyg@ysfri.a.cn.
摘要:美人鱼发光杆菌美人鱼亚种(Photobacterium damselae subsp.damselae,PDD)是一种广泛分布于海洋环境内的重要病原菌,可导致多种海洋生物患病死亡,近年来在我国不同养殖区域内均有发现和报道。[目的] 通过生理代谢表型、毒力基因分布和分子遗传分析,系统比较我国海南地区和环渤海湾不同株系PDD的表型与遗传多样性特征。[方法] 使用选择性培养基测定PDD的蔗糖发酵能力、运动性、溶血性和磷脂酶活性;通过PCR检测质粒pPHDD1复制起始点基因pPHDD1 replication origin和毒力基因dly,hlyApl,hlyAch,plpV;基于toxR基因序列构建系统发育树,分析16株PDD的遗传关系。[结果] 4株PDD蔗糖发酵测试呈阳性,12株PDD蔗糖发酵测试呈阴性。不同株系PDD运动能力存在显著差异,其中环渤海湾分离株Pdd1608、Pdd2009和Pdd1704的运动能力较强。仅有环渤海湾分离株Pdd1608表现为强溶血性、强磷脂酶活性;海南地区分离株Pdd1612和Pdd0912表现为弱溶血性、无磷脂酶活性;其余菌株表现为弱溶血性、弱磷脂酶活性。菌株Pdd1608的毒力基因谱为pPHDD1 replication origin-dly-hlyAp-hlyAch-plpV,其余菌株的毒力基因谱为hlyAch-plpV。系统发育分析揭示不同株系PDD具有高度的遗传多样性。[结论] 流行于海南地区和环渤海湾的PDD可能都由多克隆种群组成,它们具有复杂的表型特征、相似的毒力基因分布和高度的遗传多样性。
关键词:水产养殖美人鱼发光杆菌美人鱼亚种毒力表型毒力基因遗传多样性
Phenotypic and genetic diversity of Photobacterium damsela subsp. damsela isolated from aquatic animals in Hainan Province and Bohai Sea region
Liu Xiao1,2, Zhang Zheng2,3, Wang Lifang2, Yu Yongxiang2, Wang Yingeng2,3

 , Liao Meijie2,3, Xie Guosi2, Xing Jing1, Zhang Hao2, Wang Kai2, Xu Yue2
, Liao Meijie2,3, Xie Guosi2, Xing Jing1, Zhang Hao2, Wang Kai2, Xu Yue2 1. Laboratory of Pathology and Immunology of Aquatic Animals, Fisheries College, Ocean University of China, Qingdao 266003, Shandong Province, China;
2. Key Laboratory of Mariculture Organism Disease Control, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, Shandong Province, China;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, Shandong Province, China
Received: 18 September 2020; Revised: 21 January 2021; Published online: 29 January 2021
*Corresponding author: Yingeng Wang, Tel: +86-532-85817991; E-mail: wangyg@ysfri.a.cn.
Foundation item: Supported by the National Key Research and Development Program of China (2019YFD0900102), by the Key Projects of Science and Technology Innovation of Shandong Province (2018YFJH0703) and by the Project of Taishan Industry Leading Talent Project of Shandong Province (LJNY201802)
Abstract: Photobacterium damselae subsp. damselae (PDD) is an important pathogen distributed widely in global marine environment, which could infect multiple marine organisms, and reported in various mariculture regions of China in recent years. [Objective] This study aimed to systematically compare the phenotypic and genetic diversity of PDD isolated from Hainan province and Bohai Sea region base on physiological and metabolic phenotype, virulence gene distribute and molecular genetic analysis. [Methods] The physiological and metabolic phenotypes of sucrose fermentation, moveability, hemolysis, and phospholipase activity were assayed. The profiles of pPHDD1 replication origin, dly, hlyApl, hlyAch and plpV were investigated by PCR detection. Genetic diversity of PDD was analyzed based on the toxR sequences. [Results] Four PDD strains were positive for sucrose fermentation while other twelve strains were negative. There are significant differences among the motility of PDD, in which strain Pdd1608, Pdd2009, and Pdd1704 showed strong motility. Pdd1608 isolated from Bohai Sea region exhibited strong hemolysis and phospholipase activities, whlie strain Pdd1612 and Pdd0912 isolated from Hainan Province showed weak hemolysis and none phospholipase activities, and 13 strains showed weak hemolysis and phospholipase activities. The virulence gene profile of Pdd1608 was pPHDD1 replication origin-dly-hlyApl-hlyAch-plpV, while other strains were hlyAch-plpV. Phylogenetic analysis suggested remarkable genetic diversity among different kinds of PDD. [Conclusion] PDD isolated from Hainan Province and Bohai Sea region were probably composed of a polyclonal bacterial population with complex phenotypic characterizations, similar virulence gene profiles, and high genetic diversity.
Keywords: aquaculturePhotobacterium damselae subsp. damselaevirulent phenotypevirulence genegenetic diversity
美人鱼发光杆菌美人鱼亚种(Photobacterium damselae subsp. Damselae,PDD)是广泛分布于全球海洋环境中的一种革兰氏阴性致病菌,对多种海洋动物如软体动物、甲壳类、鱼类和鲸豚类以及人类都具有致病性[1-2]。PDD最早由Milton等于1981年从患病斑鳍光鳃鱼(Chromis punctipinnis)的皮肤溃疡分离、鉴定为Vibrio damselae[3];随后Smith等将其重新命名为美人鱼发光杆菌美人鱼亚种,与美人鱼发光杆菌杀鱼亚种(Photobacterium damselae subsp. Piscicida,PDP)联合构成美人鱼发光杆菌的2个亚种[4]。近年来,该菌感染海水养殖动物发病的案例越来越多,如金头鲷(Sparus aurata)[5-6]、鲈鱼(Dicentrarchus labrax)[5-6]和军曹鱼(Rachycentron canadum)[7]等。目前,PDD已被国内外****认为是威胁海水养殖发展的一种重要的病原菌。
现有研究结果表明,PDD具有高度遗传多样性。Chiu等利用脉冲场凝胶电泳技术(PFGE)分析了50株台湾海产品的PDD分离株,发现42种不同的PFGE谱型[8]。Pedersen等对16株丹麦虹鳟鱼(Oncorhynchus mykiss)的PDD分离株进行分析,发现每一株菌株都具有一个独特的PFGE谱型[9]。Terceti等通过基于toxR基因序列的遗传分析,发现流行于土耳其黑海养殖环境中的PDD和引起丹麦虹鳟鱼养殖场疾病暴发的PDD都存在多克隆种群现象[10-11]。在毒力机制研究方面,质粒pPHDD1是介导PDD高致病力的重要因子,该质粒大小为153 kb,含有与菌株溶血性相关的毒力基因dly和hlyApl[12]。此外,PDD染色体上含有分别与菌株溶血性和磷脂酶活性相关的毒力基因hlyAch和plpV[13]。这些毒力因子构成了PDD的主要毒力系统,与菌株的致病力密切相关。
海南省和环渤海湾分别是我国南、北方具有典型养殖环境迥异和养殖种类差异的海水养殖区域。本研究以分离于海南地区和环渤海湾海水养殖动物的PDD为对象,从生理代谢表型、毒力基因分布以及遗传特性进行分析,相关结果为比较我国南、北地区PDD的致病力差异,精准构建PDD检测与防控技术体系提供了理论基础。
1 材料和方法 1.1 实验菌株 实验菌株共16株,由本实验室收集、保存。其中8株分离自海南地区发病的养殖石斑鱼(Epinephelus spp.)[14]和凡纳滨对虾(Penaeus vannamei)[15],取样点位于万宁市、昌江县、三亚市和东方市;8株分离自环渤海湾发病的养殖许氏平鲉(Sebastes schlegelii)[16]、石斑鱼(Epinephelus spp.)、大泷六线鱼(Hexagrammos otakii)、大菱鲆(Scophthalmus maximus)和凡纳滨对虾(Penaeus vannamei),取样点位于烟台市、黄骅市和天津市。所有菌株均经过16S rDNA鉴定和PDD特异性基因ureC [17]检测分析。菌株信息见表 1。
表 1. 实验用PDD菌株信息表 Table 1. Details of PDD used in this study
| Strains number | Sites | Hosts | Time |
| Pdd1614 | Wanning, Hainan Province | Epinephelus fuscoguttatus | December 2016 |
| Pdd1612 | Changjiang, Hainan Province | Epinephelus Lanceolatus | November 2016 |
| Pdd1613 | Changjiang, Hainan Province | Epinephelus Lanceolatus | November 2016 |
| Pdd1615 | Changjiang, Hainan Province | Epinephelus Lanceolatus | December 2016 |
| Pdd1616 | Sanya, Hainan Province | Epinephelus fuscoguttatus | November 2016 |
| Pdd0911 | Dongfang, Hainan Province | Penaeus vannamei | June 2019 |
| Pdd0912 | Dongfang, Hainan Province | Penaeus vannamei | June 2019 |
| Pdd0913 | Dongfang, Hainan Province | Penaeus vannamei | June 2019 |
| Pdd1608 | Changdao, Yantai, Shangdong Province | Sebastes schlegelii | August 2016 |
| Pdd1611 | Changdao, Yantai, Shangdong Province | Sebastes schlegelii | November 2016 |
| Pdd1701 | Changdao, Yantai, Shangdong Province | Sebastes schlegelii | January 2017 |
| Pdd1809 | Changdao, Yantai, Shangdong Province | Hexagrammos otakii | July 2018 |
| Pdd2009 | Laizhou, Yantai, Shangdong Province | Epinephelus Lanceolatus | August 2020 |
| Pdd2010 | Laizhou, Yantai, Shangdong Province | Epinephelus akaara | August 2020 |
| Pdd0908 | Huanghua, Hebei Province | Scophthalmus maximus | June 2009 |
| Pdd1704 | Hangu, Tianjing | Penaeus vannamei | May 2017 |
表选项
1.2 表型分析 将实验室低温保存的菌株接种于添加1.5% NaCl的胰蛋白胨大豆肉汤(TSB)琼脂培养基,28 ℃活化培养24 h后,挑取单菌落进行转接,转接培养2次后,得到PDD纯培养。挑取纯化培养得到的单菌落,使用无菌PBS制备成浓度约为1×108 CFU/mL的菌悬液(OD600≈0.5)。
(1) 蔗糖发酵测试:使用接种环挑取单菌落,划线接种于硫代硫酸盐柠檬酸盐胆盐蔗糖(TCBS)琼脂培养基,28 ℃恒温培养24 h后,记录菌落形态和菌株蔗糖发酵能力。同时使用蔗糖生化管(青岛海博生物技术有限公司)进行验证分析。
(2) 运动性测试:使用TSB培养基添加0.25%琼脂制成半固体培养基。使用穿刺针挑取单菌落,穿刺接种于半固体培养基,28 ℃恒温培养24 h后,依据菌株在培养基中的扩散直径记录菌株运动性。
(3) 溶血性测试:使用TSB琼脂培养基添加5%无菌脱纤维绵羊血制成血平板。吸取10 μL菌悬液转接于血平板,28 ℃恒温培养36 h之后,拍照记录菌株的溶血能力。
(4) 磷脂酶活性测试:使用TSB琼脂培养基添加3%卵黄乳液制成卵黄平板。吸取5 μL菌悬液转接于卵黄平板,28 ℃恒温培养48 h之后,拍照记录菌株的卵磷脂分解能力。
1.3 毒力基因检测 挑取PDD单菌落,使用细菌DNA提取试剂盒(诺唯赞生物科技股份有限公司)提取基因组DNA。通过PCR检测PDD毒性质粒复制起点基因pPHDD1 replication origin[10],溶血性相关基因dly[12]、hlyApl[12]和hlyAch[13]以及磷脂酶活性相关基因plpV[13]在不同菌株中的分布情况。引物合成由生工生物工程(上海)股份有限公司完成(表 2)。PCR反应体系(50 μL)为:2×TSINGKE Master Mix 25 μL,上下游引物(10 μmol/L)各1 μL,模板DNA 1 μL,双蒸水23 μL。PCR扩增条件为:94 ℃ 5 min;94 ℃ 30 s,52.5 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 10 min,4 ℃保存。PCR产物通过1%琼脂糖凝胶电泳(95 V,45 min)检测。
表 2. 毒力相关基因的引物信息 Table 2. The primers of virulence related genes
| Target genes | Primer Sequnces (5′→3′) | Product size/bp |
| toxR | GGGATTTTATGGTACACAAA | 985 |
| ATCATAACCAGAGAGATGCT | ||
| dly | CCTATGGGACATGAATGG | 549 |
| GCTCTAGGCTAAATGAATC | ||
| hlyApl | GCTATAAATGAATAAGAAAA | 767 |
| TTGAAGCTAACTCAAAAA | ||
| hlyAch | AATGTTTCTTTCCGTTGGGC | 340 |
| CCGGAGTTCCACCAGTAAAT | ||
| plpV | TCTCATAATAGCAGTAATCT | 1618 |
| TTACTAAGCAGAATCCAGCC | ||
| pPHDD1 replication origin | TGGAATAACTATGAGTAACA | 3052 |
| CATTACCAAAACATCTACAT |
表选项
1.4 toxR基因序列测定 通过PCR扩增菌株管家基因toxR[18],产物经1%琼脂糖凝胶电泳(95 V,45 min)检测后测序。基因测序由生工生物工程(上海)股份有限公司完成。引物信息见表 2。细菌基因组DNA提取,PCR反应体系和PCR扩增条件见1.3。
1.5 系统发育分析 参考Terceti等的研究结果,选取toxR基因序列进行系统发育分析[10-11]。使用软件MEGA 7.0[19],通过Clustal W对齐基因序列[20],以邻接法(Neighbor-Joining)为统计方法[21],最大合成似然法(maximum composite likelhood)为核苷酸替代模型[22],Bootstrap (1000次重复)为检验方法[23],构建基于toxR基因序列的系统发育树。
2 结果和分析 2.1 不同株系PDD菌株表型差异性分析 (1) 蔗糖发酵能力:海南地区分离株Pdd1615、Pdd1616和环渤海湾分离株Pdd2009、Pdd2010在TCBS琼脂培养基上生长形成黄色菌落,同时菌株蔗糖生化测试呈阳性,表明这4株PDD具有蔗糖发酵能力;海南地区分离株Pdd1614、Pdd1612、Pdd1613、Pdd0911、Pdd0912、Pdd0913和环渤海湾分离株Pdd1608、Pdd1611、Pdd1701、Pdd1809、Pdd0908、Pdd1704在TCBS琼脂培养基上生长形成绿色菌落,同时菌株蔗糖生化测试呈阴性,表明这12株PDD不具有蔗糖发酵能力。
(2) 运动性:环渤海湾分离株Pdd1608、Pdd2009和Pdd1704表现出较强运动能力;海南地区分离株Pdd1612、Pdd1615、Pdd1616、Pdd0913和环渤海湾分离株Pdd1809、Pdd2010表现出较弱的运动能力;海南地区分离株Pdd1612、Pdd1615、Pdd1616、Pdd0913和环渤海湾分离株Pdd1809、Pdd2010表现出较弱的运动能力;海南地区分离株Pdd1614、Pdd1613、Pdd0911、Pdd0912和环渤海湾分离株Pdd1611、Pdd1701、Pdd0908表现出几乎不具有运动能力。
(3) 溶血性:环渤海湾分离株Pdd1608表现为强溶血性;海南地区分离株Pdd1614、Pdd1612、Pdd1613、Pdd1615、Pdd1616、Pdd0911、Pdd0912、Pdd0913和环渤海湾分离株Pdd1611、Pdd1701、Pdd1809、Pdd2009、Pdd2010、Pdd0908、Pdd1704表现为弱溶血性。
(4) 磷脂酶活性:环渤海湾分离株Pdd1608表现为强磷脂酶活性;海南地区分离株Pdd1614、Pdd1613、Pdd1615、Pdd1616、Pdd0911、Pdd0913和环渤海湾分离株Pdd1611、Pdd1701、Pdd1809、Pdd2009、Pdd2010、Pdd0908、Pdd1704表现为弱磷脂酶活性;海南地区分离株Pdd1612和Pdd0912表现为无磷脂酶活性。
PDD的不同溶血性和磷脂酶活性表型特征见图 1。菌株表型详细信息见图 2。
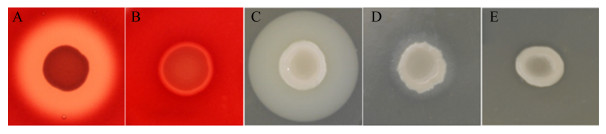 |
| 图 1 PDD的不同溶血性表型特征与磷脂酶活性表型特征 Figure 1 Different hemolysis and phospholipase activity phenotype of PDD. A: strong hemolysis: Pdd1608; B: weak hemolysis: Pdd1614, Pdd1612, Pdd1613, Pdd1615, Pdd1616, Pdd0911, Pdd0912, Pdd0913, Pdd1611, Pdd1701, Pdd1809, Pdd2009, Pdd2010, Pdd0908, Pdd1704; C: strong phospholipase activity: Pdd1608; D: weak phospholipase activity: Pdd1614, Pdd1613, Pdd1615, Pdd1616, Pdd0911, Pdd0913, Pdd1611, Pdd1701, Pdd1809, Pdd2009, Pdd2010, Pdd0908, Pdd1704; E: no phospholipase activity: Pdd1612, Pdd0912. |
| 图选项 |
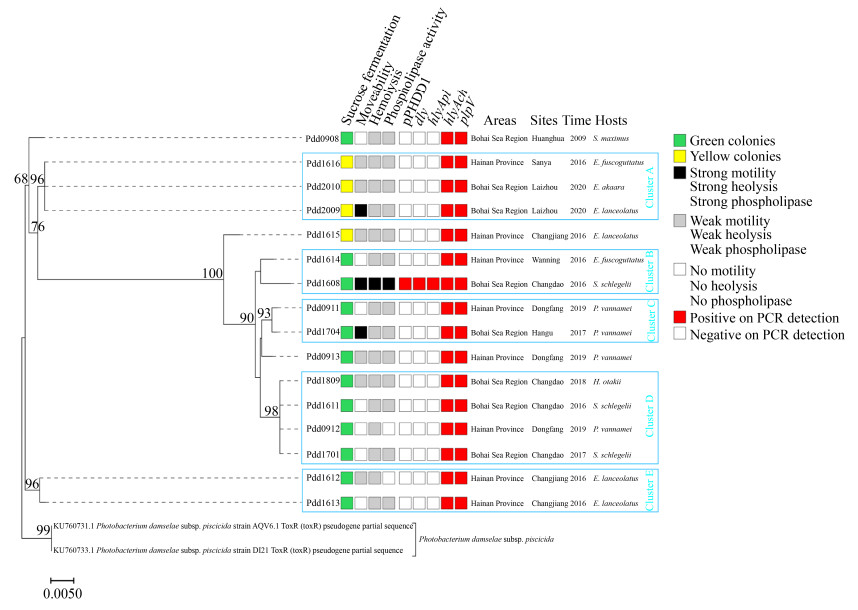 |
| 图 2 不同株系PDD的表型特征、毒力基因分布以及系统发育分析 Figure 2 Phenotypic characteristics, virulence gene distribution, and phylogenetic analysis of different PDD strians. |
| 图选项 |
2.2 不同株系PDD毒力基因携带差异性分析 通过对不同株系PDD的5种毒力基因进行PCR检测,发现仅有环渤海湾分离株Pdd1608的pPHDD1 replication origin呈阳性,其余17株PDD的pPHDD1 replication origin呈阴性,表明Pdd1608携带毒性质粒pPHDD1,其余17株PDD不携带毒性质粒pPHDD1。环渤海湾分离株Pdd1608的溶血素相关基因dly呈阳性,其余17株PDD溶血素相关基因dly呈阴性。环渤海湾分离株Pdd1608的溶血素相关基因hlyApl呈阳性,其余17株PDD溶血素相关基因hlyApl呈阴性。所有PDD的溶血素相关基因hlyAch和磷脂酶活性相关基因plpV都呈阳性。即环渤海湾分离株Pdd1608同时携带毒性质粒pPHDD1、毒力基因dly、hlyApl、hlyAch和plpV,海南地区分离株Pdd1614、Pdd1612、Pdd1613、Pdd1615、Pdd1616、Pdd0911、Pdd0912、Pdd0913和环渤海湾分离株Pdd1611、Pdd1701、Pdd1809、Pdd2009、Pdd2010、Pdd0908、Pdd1704携带毒力基因hlyAch和plpV。不同株系PDD的毒力基因分布信息见图 2。
2.3 基于toxR的不同株系PDD遗传差异性分析 通过基于toxR基因序列的系统发育分析,发现PDD海南地区分离株和环渤海湾分离株在系统发育树中分化为8个分支。分离于2016年的海南三亚株Pdd1616和分离于2020年的环渤海湾莱州株Pdd2009、Pdd2010聚簇形成Cluster A;分离于2016年的海南万宁株Pdd1614和分离于2016年的环渤海湾长岛株Pdd1608聚簇形成Cluster B;分离于2019年的海南东方株Pdd0911和分离于2017年的环渤海湾汉沽株Pdd1704聚簇形成Cluster C;分离于2019年的海南东方株Pdd0912和分别分离于2016–2018年的环渤海湾长岛株Pdd1809、Pdd1611、Pdd1701聚簇形成Cluster D;分离于2016年的海南昌江株Pdd1612和Pdd1613聚簇形成Cluster E;分离于2009年的环渤海湾黄骅株Pdd0908、分离于2016年的海南昌江株Pdd1615和分离于2019年的海南东方株Pdd0913则单独成为一支。其中,构成Cluster B、Cluster C和Cluster D的菌株、海南昌江株Pdd1615和海南东方株Pdd0913共同构成一个大分支。
8株海南地区分离株分布于系统发育树的7个分支,其中三亚株Pdd1616位于Cluster A,万宁株Pdd1614位于Cluster B,东方株Pdd0911位于Cluster C,东方株Pdd0912位于Cluster D,昌江株Pdd1612和Pdd1613位于Cluster E,昌江株Pdd1615和东方株Pdd0913单独成为一支。8株环渤海湾分离株分布于系统发育树的5个分支。莱州株Pdd2009、Pdd2010位于Cluster A,长岛株Pdd1608位于Cluster B,长岛株Pdd1611、Pdd1701、Pdd1809位于Cluster D,汉沽株Pdd1704位于Cluster C,黄骅株Pdd0908单独成为一支。系统发育分析具体结果见图 2。
3 讨论 PDD是广泛分布于海洋环境中的一种致病菌,全球范围内有很多PDD感染不同海洋动物发病的案例报道[24-25]。近年来,我国国内不断有PDD感染海水养殖动物的报道,如银鲳(Pampus argenteus)[26]、许氏平鲉(Sebastes schlegelii)[27]、半滑舌鳎(Cynoglossus semilaevis)[28]以及凡纳滨对虾(Penaeus vannamei)[15]等。PDD已逐渐成为威胁我国海水养殖产业发展的一种重要新发病原菌。虽然,有关PDD感染海水养殖动物的报道越来越多,但在国内仍缺乏对PDD毒力表型和遗传特征的研究报道。
本研究通过生理代谢表型测试,发现海南地区和环渤海湾的PDD分离株都具有表型多样性。现有研究表明,大多数PDD在TCBS培养基上产生绿色菌落,但仍有少部分菌株产生黄色菌落[10]。本研究通过蔗糖发酵测试,发现12株PDD不能发酵蔗糖,在TCBS培养基上产生绿色菌落;4株PDD能发酵蔗糖,在TCBS培养基上产生黄色菌落。蔗糖发酵测试结果表明,PDD在TCBS琼脂培养基上的菌落颜色可为绿色,也可为黄色。Chaban等的研究表明,鞭毛赋予了细菌运动能力,同时与菌株致病性密切相关[29]。本研究通过运动测试,发现16株PDD的运动能力存在显著差异,其中环渤海湾分离株Pdd1608、Pdd2009和Pdd1704的运动能力明显强于其他菌株。运动测试结果提示不同株系PDD的致病力可能具有差异,这一推测有待于未来进行人工感染试验加以验证。
PDD的致病性主要由4种不同毒力因子决定,分别是位于质粒pPHDD1的溶血性相关基因dly和hlyApl,以及位于染色体的溶血性相关基因hlyAch和磷脂酶活性相关基因plpV[30]。此外,邓益琴等发现PDD的致病性还可能与hlyA和vvh等非典型毒力基因相关[31]。本研究通过对不同株系PDD的毒力基因检测,发现环渤海湾分离株Pdd1608的毒力基因谱为pPHDD1 replication origin-dly-hlyApl-hlyAch-plpV,其余菌株的毒力基因谱为hlyAch-plpV,表明所有菌株都持有毒力基因hlyAch和plpV,但只有Pdd1608持有质粒pPHDD1以及毒力基因dly和hlyApl。这与现有研究结果相符,即持有质粒pPHDD1的PDD可含有毒力基因dly、hlyApl、hlyAch和plpV;不持有质粒pPHDD1的PDD可含有毒力基因hlyAch和plpV[12-13]。Osorio等的研究表明持有质粒pPHDD1的PDD表现为强溶血性,仅持有hlyAch的PDD表现为弱溶血性;持有质粒pPHDD1的PDD表现为强磷脂酶活性,仅持有plpV的PDD表现为弱磷脂酶活性[31]。本研究的表型测试发现仅有持有质粒pPHDD1的菌株Pdd1608表现为强溶血性、强磷脂酶活性,不持有质粒pPHDD1的菌株中,除了海南地区分离株Pdd1612和Pdd0912表现为弱溶血性、无磷脂酶活性,其余菌株都表现为弱溶血性、弱磷脂酶活性。菌株Pdd1612和Pdd0912持有plpV却表现为无磷脂酶活性,说明其plpV基因没有正常表达,具体原因有待探究。
Pedersen等发现,质粒pPHDD1和强溶血性是PDD高致病力菌株的标志,弱溶血性菌株相对强溶血性菌株致病力极低[9],随后Terceti等发现表现为弱磷脂酶活性的无溶血性菌株同样具有一定程度的致病能力[11]。本研究结果表明,持有质粒pPHDD1的强溶血性菌株Pdd1608可能为强致病力毒株,其余菌株可能为弱/无致病力毒株(数据待发表)。本研究使用的PDD宿主包括大菱鲆(Scophthalmus maximus)、石斑鱼(Epinephelus spp.)、许氏平鲉(Sebastes schlegeli)、大泷六线鱼(Hexagrammos otakii)和凡纳滨对虾(Penaeus vannamei)。另据报道,PDD还能感染章鱼(Octopus joubini)[32]、海豚(Tursiops truncatus)[33]和鲸鱼(Balaenoptera edeni)[33],表明PDD可感染广泛的水生动物。Isenberg发现许多病原微生物对宿主具有特异性,此外对宿主的不同组织的影响也并不相同[34]。由此,推测本研究所获PDD对不同宿主的致病力,以及对宿主的不同组织的影响可能存在差异,该假设需要后续研究加以证实。
Osorio和Klose发现,弧菌科细菌的管家基因toxR具有高度的变异性[17]。Terceti等的研究证实toxR的序列多态性允许对密切相关的PDD菌株进行鉴别,并由此揭示PDD多克隆种群的存在[10-11]。本研究基于toxR基因序列对不同株系PDD进行了系统发育分析,发现PDD海南地区分离株和环渤海湾分离株都具有极高的遗传多样性。系统发育树中,不同地点来源的PDD分布在不同的Cluster中,甚至同一地点的PDD也分布在不同的Cluster中。海南地区分离株PDD0911、Pdd0913和Pdd0912都分离自东方市,但它们分别分布于系统发育树的Cluster C、Cluster D和单独一支;Pdd1612、Pdd1613和菌株Pdd1615都分离自昌江县,但它们分别分布于系统发育树的Cluster A和单独一支;环渤海湾分离株Pdd1608和Pdd1611、Pdd1701、Pdd1809都分离自长岛县,但它们分别分布于Cluster B和Cluster D。这表明流行于海南地区和环渤海湾不同地点的PDD可能由不同遗传株系的菌株组成,甚至同一地点的PDD也存在株系分化的现象。值得注意的是,部分不同区域养殖动物的PDD发生了聚簇现象。海南地区的石斑鱼分离株Pdd1616和环渤海湾的石斑鱼分离株Pdd2009、Pdd2010聚簇形成Cluster A,它们的toxR基因相似性为100%;海南地区的凡纳滨对虾分离株Pdd0912,环渤海湾的许氏平鲉分离株Pdd1611、Pdd1701,以及环渤海湾的大泷六线鱼分离株Pdd1809聚簇形成Cluster D,它们的toxR基因相似性为100%。这表明同一株系的PDD能在不同地区、不同养殖动物之间传播。推测之所以发生这种现象,是因为在我国养殖实践过程中存在南鱼北养、北鱼南养,导致海水养殖动物携带的病原菌在不同区域发生交叉传播。此外,Cluster D含有的3株环渤海湾长岛株Pdd1611、Pdd1701和Pdd1809分别分离于2016年、2017年和2018年,表明该簇的菌株于2016–2018年3年间一直存在于长岛县深水网箱养殖环境中,它们可能是PDD环渤海湾长岛流行株的主要种群。Matanza和Osorio发现PDD的致病力与水温等环境因子密切相关[35],由此推测某些株系的PDD可能长期存在于海水养殖环境中,只有在环境条件如水温出现显著变化时才会感染养殖动物发病。
综上所述,流行于海南地区和环渤海湾的PDD具有复杂的表型特征与相似的毒力基因分布,它们由不同遗传株系的菌株构成,表明流行于我国海南和环渤海湾养殖区的PDD可能都是多克隆种群,且不同株系的菌株对宿主的致病力也许存在差异。上述研究有利于全面了解我国PDD的表型、遗传和分布特征,对实现PDD监测、疾病防控及水产养殖产业绿色发展具有重要指导意义和支撑作用。未来,我们将广泛收集我国沿海不同区域的PDD,深入分析不同株系PDD的病原时空变化等区系特征;同时,对不同表型、基因型的PDD进行深度解析,特别在疫苗方面对强毒株和弱毒株进行研发和产业化应用。
References
| [1] | Takahashi H, Miya S, Kimura B, Yamane K, Arakawa Y, Fujii T. Difference of genotypic and phenotypic characteristics and pathogenicity potential of Photobacterium damselae subsp. damselae between clinical and environmental isolates from Japan. Microbial Pathogenesis, 2008, 45(2): 150-158. DOI:10.1016/j.micpath.2008.04.008 |
| [2] | Rivas AJ, Lemos ML, Osorio CR. Photobacterium damselae subsp. damselae, a bacterium pathogenic for marine animals and humans. Frontiers in Microbiology, 2013: 283-283. |
| [3] | Love M, Teebken-Fisher D, Hose JE, Farmer JJ, Hickman FW, Fanning GR. Vibrio damsela, a marine bacterium, causes skin ulcers on the damselfish Chromis punctipinnis. Science, 1981, 214(4525): 1139-1140. DOI:10.1126/science.214.4525.1139 |
| [4] | Smith SK, Sutton DC, Fuerst JA, Reichelt JL. Evaluation of the genus Listonella and reassignment of Listonella damsela (Love et al.) MacDonell and Colwell to the genus Photobacterium as Photobacterium damsela comb. nov. with an emended description. International Journal of Systematic Bacteriology, 1991, 41(4): 529-534. DOI:10.1099/00207713-41-4-529 |
| [5] | Abdel-Aziz M, Eissa AE, Hanna M, Okada MA. Identifying some pathogenic Vibrio/Photobacterium species during mass mortalities of cultured Gilthead seabream (Sparus aurata) and European seabass (Dicentrarchus labrax) from some Egyptian coastal provinces. International Journal of Veterinary Science and Medicine, 2013, 1(2): 87-95. DOI:10.1016/j.ijvsm.2013.10.004 |
| [6] | Sadok K, Faouzi L, Amina B, Kamel G. Virulence properties, biofilm formation and random amplified polymorphic DNA analysis of Photobacterium damselae subsp. damselae isolates from cultured sea bream (Sparus aurata) and seabass (Dicentrarchus labrax). Microbial Pathogenesis, 2014, 69/70: 13-19. DOI:10.1016/j.micpath.2014.03.007 |
| [7] | Sharma SR, Pradeep MA, Sadu N, Dube PN, Vijayan KK. First report of isolation and characterization of Photobacterium damselae subsp. damselae from cage-farmed cobia (Rachycentron canadum). Journal of Fish Diseases, 2017, 40(7): 953-958. DOI:10.1111/jfd.12557 |
| [8] | Chiu TH, Kao LY, Chen ML. Antibiotic resistance and molecular typing of Photobacterium damselae subsp. damselae, isolated from seafood. Journal of Applied Microbiology, 2013, 114(4): 1184-1192. DOI:10.1111/jam.12104 |
| [9] | Pedersen K, Skall HF, Lassennielsen AM, Bjerrum L, Olesen NJ. Photobacterium damselae subsp. damselae, an emerging pathogen in Danish rainbow trout, Oncorhynchus mykiss (Walbaum), mariculture. Journal of Fish Diseases, 2010, 32(5): 465-472. |
| [10] | Terceti MS, Vences A, Matanza XM, Dalsgaard I, Pedersen K, OsorioCR. Molecular epidemiology of Photobacterium damselae subsp. damselae outbreaks in marine rainbow trout farms reveals extensive horizontal gene transfer and high genetic diversity. Frontiers in Microbiology, 2018. |
| [11] | Terceti MS, Ogut H, Osorio CR. Photobacterium damselae subsp. damselae, an emerging fish pathogen in the Black Sea: evidence of a multiclonal origin. Applied and Environmental Microbiology, 2016, 82(13): 3736-3745. DOI:10.1128/AEM.00781-16 |
| [12] | Rivas AJ, Balado M, Lemos ML, Osorio CR. The Photobacterium damselae subsp. damselae hemolysins damselysin and HlyA are encoded within a new virulence plasmid. Infection and Immunity, 2011, 79(11): 4617-4627. DOI:10.1128/IAI.05436-11 |
| [13] | Vences A, Rivas AJ, Lemos ML, Husmann M, Osorio CR. Chromosome-encoded hemolysin, phospholipase, and collagenase in plasmidless isolates of Photobacterium damselae subsp. damselae contribute to virulence for fish. Applied and Environmental Microbiology, 2017, 83(11). |
| [14] | 许悦. 海南地区养殖石斑鱼两种细菌性疾病病原病理学研究. 海南大学硕士学位论文, 2018. |
| [15] | Wang Z, Shi C, Wang H, Wan XY, Zhang QL, Song XL, Li G, Gong M, Ye SG, Xie GS. A novel research on isolation and characterization of Photobacterium damselae subsp. damselae from Pacific white shrimp, Penaeus vannamei, displaying black gill disease cultured in China. Journal of Fish Diseases, 2020, 43(5): 551-559. DOI:10.1111/jfd.13153 |
| [16] | 王凯. 长岛地区深水网箱养殖许氏平鲉主要疾病的流行病学调查与病原病理学研究. 上海海洋大学硕士学位论文, 2018. |
| [17] | Osorio CR, Toranzo AE, Romalde JL, Barja JL. Multiplex PCR assay for ureC and 16S rRNA genes clearly discriminates between both subspecies of Photobacterium damselae. Diseases of Aquatic Organisms, 2000, 40(3): 177-183. |
| [18] | Osorio CR, Klose KE. A region of the transmembrane regulatory protein ToxR that tethers the transcriptional activation domain to the cytoplasmic membrane displays wide divergence among Vibrio species. Journal of Bacteriology, 2000, 182(2): 526-528. DOI:10.1128/JB.182.2.526-528.2000 |
| [19] | Kumar S, Stecher G, Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [20] | Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG. Clustal W and Clustal X version 2.0. Bioinformatics: Oxford, England, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [21] | Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 1987, 4(4): 406-425. |
| [22] | Tamura K, Nei M, Kumar S. Prospects for inferring very large phylogenies by using the neighbor-joining method. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(30): 11030-11035. DOI:10.1073/pnas.0404206101 |
| [23] | Felsenstein J. Confidence limits on phylogenies: an approach using the bootstrap. Evolution; International Journal of Organic Evolution, 1985, 39(4): 783-791. DOI:10.1111/j.1558-5646.1985.tb00420.x |
| [24] | Labella A, Berbel C, Manchado M, Castro D, Borrego JJ. Photobacterium damselae subsp. damselae, an emerging pathogen affecting new cultured marine fish species in Southern Spain. Recent Advances in Fish Farms. InTech, 2011. |
| [25] | Eissa IAM, Derwa HI, Ismail M, El-Lamie M, Dessouki AA, Elsheshtawy H, Bayoumy EM. Molecular and phenotypic characterization of Photobacterium damselae among some marine fishes in Lake Temsah. Microbial Pathogenesis, 2018(114): 315-322. |
| [26] | Tao Z, Shen C, Zhou S, Yang N, Wang GL, Wang YJ, Xu SL. An outbreak of Photobacterium damselae subsp. damselae infection in cultured silver pomfret Pampus argenteus in Eastern China. Aquaculture, 2018: 201-205. |
| [27] | Zhang Z, Yu Y, Wang K, Wang YG, Jiang Y, Liao MJ, Rong XJ. First report of skin ulceration caused by Photobacterium damselae subsp. damselae in net-cage cultured black rockfish (Sebastes schlegeli). Aquaculture, 2019: 1-7. |
| [28] | Shao P, Yong P, Zhou W, Sun JH, Wang YZ, Tang QP, Ren ST, Wu Z, Zhao CY, Xu YF. First isolation of Photobacterium damselae subsp. damselae from half-smooth tongue sole suffering from skin-ulceration disease. Aquaculture (Amsterdam, Netherlands), 511: 734208-734208. DOI:10.1016/j.aquaculture.2019.734208 |
| [29] | Chaban B, Hughes HV, Beeby M. The flagellum in bacterial pathogens: for motility and a whole lot more. Seminars in Cell & Developmental Biology, 2015, 46: 91-103. |
| [30] | Osorio CR, Vences A, Matanza XM, Terceti MS. Photobacterium damselae subsp. damselae, a generalist pathogen with unique virulence factors and high genetic diversity. Journal of Bacteriology, 2018, 200(15). |
| [31] | Deng YQ, Xu LW, Zhang YQ, Guo ZX, Feng J. Analysis of virulence genes and antibiotic resistance of Photobacterium damselae isolated from marine fishes in coastal South China. Acta Microbiologica Sinica, 2020, 60(11): 2606-2619. (in Chinese) 邓益琴, 徐力文, 张亚秋, 郭志勋, 冯娟. 我国华南沿海海水养殖鱼类病原菌美人鱼发光杆菌(Photobacterium damselae)分离株的毒力基因和耐药性分析. 微生物学报, 2020, 60(11): 2606-2619. |
| [32] | Hanlon RT, Forsythe JW, Cooper KM, Dinuzzo AR, Kelly M T. Fatal penetrating skin ulcers in laboratory-reared octopuses. Journal of Invertebrate Pathology, 1984, 44(1): 67-83. DOI:10.1016/0022-2011(84)90047-8 |
| [33] | Buck JD, Overstrom NA, Patton GW, Anderson HF, Gorzelany JF. Bacteria associated with stranded cetaceans from the northeast USA and southwest Florida Gulf coasts. Diseases of Aquatic Organisms, 1991, 10(2): 147-152. |
| [34] | Isenberg HD. Pathogenicity and virulence. Clinical Microbiology Reviews, 1988, 1(1): 40-53. DOI:10.1128/CMR.1.1.40 |
| [35] | Matanza XM, Osorio CR. Transcriptome changes in response to temperature in the fish pathogen Photobacterium damselae subsp. damselae: clues to understand the emergence of disease outbreaks at increased seawater temperatures. PLoS ONE, 2018, 13(12). |
