曲珊珊, 严洪珊, 林炜铁

 , 罗剑飞
, 罗剑飞

华南理工大学生物科学与工程学院, 广东 广州 510006
收稿日期:2021-03-03;修回日期:2021-04-24;网络出版日期:2021-05-07
基金项目:国家自然科学基金(91951118)
作者简介:罗剑飞,副教授,硕士生导师。研究方向为:环境微生物学、微生物生态学、环境微生物技术。研究兴趣:(1) 推动多元素耦合生物地球化学循环的微生物及其循环途径;(2) 隐藏于自然界中未知微生物类群的发现、富集和培养;(3) 修复受污染环境的微生物技术;(4) 替代化石燃料的生物能源。承担国家自然科学基金和中央高校基本科研项目等多项.
*通信作者:林炜铁, E-mail: lfwtlin@scut.edu.cn;
罗剑飞, E-mail: ljfjf2002@scut.edu.cn.
摘要:[目的] 探究化能自养硫氧化细菌Halothiobacillus sp.LS2介导的以乙炔为电子受体的厌氧硫氧化反应。[方法] 稀释涂布法测定细胞生长情况,离子色谱仪测试硫氧化动力学中SO42-和S2O32-以及基于相对荧光定量法的基因表达分析。[结果] 尽管菌株LS2在以氧气为电子受体时的最大反应速率Vmax更高,但在厌氧条件下且以乙炔为电子受体时,菌株LS2的生长量是氧气为电子受体时的2倍,且硫氧化酶基因soxB的表达量显著高于氧气作为电子受体时。[结论] 菌株LS2不仅可以以乙炔为电子受体完成厌氧硫氧化反应,且这一代谢过程的产能效率较有氧硫氧化过程更高。本研究首次发现了微生物介导的以乙炔为电子受体的厌氧硫氧化反应,对丰富硫的生物地球化学循环理论有积极意义。
关键词:厌氧硫氧化动力学乙炔soxB
Anaerobic sulfur oxidation with acetylene as electron acceptor mediated by chemoautotrophic sulfur-oxidizing bacterium Halothiobacillus sp. LS2
Qu Shanshan, Yan Hongshan, Lin Weitie

 , Luo Jianfei
, Luo Jianfei

School of Biology and Biological Engineering, South China University of Technology, Guangzhou 510006, Guangdong Province, China
Received: 3 March 2021; Revised: 24 April 2021; Published online: 7 May 2021
*Corresponding author: Weitie Lin, Weitie Lin, E-mail: lfwtlin@scut.edu.cn;
Jianfei Luo, Jianfei Luo, E-mail: ljfjf2002@scut.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China Major Research Program (91951118)
Abstract: [Objective] The study aims to investigate the acetylene as electron acceptor anaerobic sulfur oxidation mediated by the chemoautotrophic sulfur oxidizing bacterium Halothiobacillus sp. LS2. [Methods] Cell growth was measured by dilution coating method, SO42- and S2O32- in sulfur oxidation kinetics were measured by ion chromatograph, and gene expression was analyzed based on relative fluorescence quantitative method. [Results] Although strain LS2 obtained high maximum reaction rate Vmax with O2 as electron acceptor, its growth with acetylene as electron acceptor under anaerobic condition was twice that with O2 as electron acceptor, and the expression of soxB gene was significantly higher than that with O2 as electron acceptor. [Conclusion] These results indicated that strain LS2 can not only complete anaerobic sulfur oxidation with acetylene as electron acceptor, but also has higher productivity efficiency than aerobic sulfur oxidation. In this study, an anaerobic sulfur oxidation reaction using acetylene as electron acceptor was proposed for the first time, which is of positive significance to the sulfur biogeochemical cycle.
Keywords: anaerobic sulfur oxidationdynamicsacetylenesoxB
硫的生物氧化反应是一种古老的生化反应,可追溯至25亿年前的太古宙时期[1-2]。自然界中硫氧化相关酶系和微生物种类繁多,从而导致了硫氧化途径的多样性[3]。好氧条件下,硫氧化细菌(sulfur-oxidizing bacteria,SOB)以硫化物、单质硫和硫代硫酸盐等还原态硫为底物进行硫氧化反应,产生的电子通过呼吸链复合体(respiratory complex Ⅳ)传递给氧气并将质子H+泵出以驱动ATP合成;厌氧条件下,基于Complex Ⅳ的电子传递链被阻断,硫氧化反应产生的电子最终传递给NO3–/NO2–、Fe3+、Mn(Ⅳ)、N2和CO2。这些厌氧硫氧化细菌中,除了新发现的以Mn (Ⅳ)为电子受体的Sulfurimonas marisnigri[4],其他细菌的厌氧硫氧化机制已基本明确。例如:(1) 光能营养型的硫氧化细菌以CO2为主要电子受体,利用不产氧的光合作用驱动环式电子流连续泵出H+,并通过光合磷酸化作用生成ATP进行厌氧硫氧化[5-7]。(2) 嗜酸硫氧化细菌Acidithiobacillus以Fe3+为电子受体,将硫氧化作用产生的电子通过醌库(Ubiquinone/Ubiquinol,Q/QH2)传递给细胞膜上的泛醌细胞色素氧化还原酶复合体(cytochrome bc1 complex),经由细胞色素复合体(cytochrome complex I,Cyt c1)→铜蓝蛋白(rusticyanin,Rus)→Cyt c2传递给外膜上的细胞色素c,并将Fe3+还原为Fe2+;在此过程中,bc1 complex每传递1个电子即泵出1个H+并通过ATP合成酶产生能量[8-9]。(3) 与反硝化偶联的硫氧化细菌以NO3–/NO2–为电子受体,通过间质NO3–还原酶(periplasmic nitrate reductase,Nap)、cd1型NO2–还原酶(cd1 nitrite reductase,NirS)、细胞质NO3–还原酶(membrane bound nitrate reductase,Nar)、铜离子型NO2–还原酶(copper-type nitrite reductase,NirK)、NO还原酶(nitric oxide reductase,Nor)和N2O还原酶(nitrous oxide reductase,Nos)等反硝化酶的作用,每传递1个电子可泵出1–2个H+用于能量生成[10]。此外,一些具有固氮能力的SOB也可以N2为电子受体部分接收硫氧化产生的电子,但该过程不能驱动胞内质子外排,必须通过与反硝化、铁还原或锰还原作用才能促使跨膜质子梯度差的形成,进而驱动ATP的合成。除了上述物质可作为厌氧硫氧化的电子受体,自然界中还可能存在其他物质或能量代谢途径,以维持厌氧条件下硫氧化作用的进行。
我们对化能自养硫氧化细菌Halothiobacillus sp. LS2进行全基因组测序,发现其基因组中含有多种用于硫氧化的酶系基因以及固氮作用所需的基因簇,并能在好氧条件下通过固氮作用获得氮源[11],同时,我们发现该菌在好氧或厌氧条件下,也可以乙炔为电子受体进行硫氧化反应。为了阐明菌株LS2以乙炔为电子受体时的硫氧化动力学特征,本研究对其以氧气和乙炔为电子受体时的细胞生长、硫氧化动力学、硫氧化酶基因表达等进行了比较分析,以此描述了一种以乙炔为电子受体的厌氧硫氧化反应。
1 材料和方法 1.1 微生物培养
1.1.1 液体培养基(g/L): KH2PO4 0.40;MgCl2 0.12;NaCl 10;NH4Cl 0.05;A5元素液1 mL/L;pH 6.5–7.0。待灭菌完毕,加入NaHCO3 0.20;FeCl2 0.008;Na2S2O3 2.48 (试剂进行过滤除菌);厌氧培养时,培养基中加入2.0 mmol/L抗坏血酸为除氧剂。
1.1.2 固体培养基(g/L): KH2PO4 0.40;MgSO4 0.12;KNO3 0.40;NH4Cl 0.10;pH 6.5–7.0;1.5%琼脂。待灭菌完毕,加入NaHCO3 0.20;FeSO4 0.02;Na2S2O3 2.48 (试剂进行过滤除菌)。
1.1.3 菌种活化: 取保存于–80 ℃的菌株LS2,按1%接种量接入液体培养基三角瓶中,置于30 ℃振荡培养箱(ZQLY-180G)培养2 d,设置摇床转速150 r/min;梯度稀释后平板涂布至固体培养基中,置于30 ℃的生化培养箱(LRH-150)培养至菌落形成,挑单菌落进行液体培养,并作为种子液用于好氧或厌氧培养。
1.2 菌株LS2在不同气体条件下的生长曲线及硫氧化情况 在血清瓶中,分别设置顶空气体条件为5% O2、20% O2、5% O2+20% C2H2、20% C2H2,其余气体用氩气补齐;加入5.0×10–4 mol/L NH4Cl为氮源,以1.0×10–3 mol/L Na2S2O3为底物,置于30 ℃振荡培养箱(ZQLY-180G)培养2 d,设置摇床转速150 r/min,采用稀释涂布法跟踪测定每组细菌的生长量,采用离子色谱测定硫酸根的生成和底物硫代硫酸根的消耗情况(3个平行)。
1.3 以氧气和乙炔为电子受体的硫氧化动力学 通过抽滤(SHZ-D(Ⅲ)循环真空泵)收集种子液的菌体,用不含底物的新鲜培养基清洗并重悬,分装至12 mL血清瓶中(装液量为6 mL,每个底物浓度设置3个平行),分别用于好氧(20% O2)和厌氧(20% C2H2)的硫氧化动力学分析;静置1 h后,分别加入终浓度为0、0.02、0.04、0.06、0.08、0.1、0.2、0.4、0.6、0.8、1.0、1.5 mmol/L的S2O32–为底物,反应20 min后取样,用0.22 μm滤膜过滤取得的培养液,以终止反应,用离子色谱测定初始和反应终了的硫酸根浓度,稀释涂布平板法对初始菌体数量进行计数。
1.4 硫氧化酶soxB基因表达分析
1.4.1 菌株培养: 收集的菌体用不含底物的液体培养基重悬,转移至血清瓶中,顶空气体中加入20% O2、5% C2H2、20% C2H2和30% C2H2,其余用氩气补齐;加入1.0 mmol/L的S2O32–为底物,置于30 ℃恒温培养箱进行培养。
1.4.2 菌体RNA的提取: 将细菌培养液以12000 r/min的转速离心3 min (CP-80MX超高速离心机),弃上清,收集菌体至2 mL离心管中,加入1 mL RNAiso PLUS(TaKaRa),室温静置5 min后加入200 μL氯仿(大茂化学试剂)振荡混匀,12000 r/min离心2 min;收集上清液至新的离心管中,加入等体积异丙醇和1 μL核酸助沉剂(北京庄盟生物),充分混匀后于室温静置10 min;12000 r/min离心5 min,弃上清,加入1 mL 75%乙醇,充分混匀后12000 r/min离心5 min,弃上清;移除离心管中残余液体,室温干燥2 min,加入适量的RAase-free水溶解沉淀得到菌株的RNA,置于–80 ℃冰箱保存。
1.4.3 荧光定量PCR: 提取的总RNA用TransStart? One-Step gDNA Removeal and cDNA Synthesis SuperMix (北京全式金)试剂盒进行反转录,将得到的cDNA稀释10倍用于定量分析,菌株LS2的硫氧化酶soxB基因和内参基因16S rRNA引物的合成工作由广州天一辉远基因科技有限公司进行[11]。荧光定量PCR反应程序为:94 ℃ 1 min;94 ℃ 10 s,60 ℃ 34 s,重复40个循环。荧光标准曲线的扩增效率为92.3%–97.8%,标准曲线的相关系数为0.999,并采用2T–△△C相对定量方法对得到的数据进行分析。
1.5 离子色谱检测S2O32–和SO42–含量 本研究采用离子色谱法测定培养液中的S2O32–和SO42–含量。仪器:Dionex AQUION RFIC型离子色谱仪,AS16 IC分离柱,Dionex EGC Ⅲ KOH RFICTM淋洗液发生器,AS-DV自动进样器,Thermo ScientificTM DionexTM ChromeleonTM色谱数据系统(CDS)软件(Thermo公司)。实验参数:淋洗液(KOH)1.5×10–2 mol/L,流速为1 mL/min,进样量25 μL,柱温为30 ℃,背景电导0.5 μs/cm。
2 结果和分析 2.1 菌株LS2在不同气体条件下的生长情况 菌株LS2在5% O2、20% O2、5% O2+20% C2H2、20% C2H2条件下的硫氧化和生长情况如图 1所示,菌株LS2在有氧或厌氧以乙炔为电子受体时,12 h内均可将1.0 mmol/L硫代硫酸根完全氧化为硫酸根,14 h细胞数达到最大。好氧条件下,当氧气浓度为5%时,菌株LS2的最大细胞数为1.7×107 CFU/mL;当氧气浓度为20%时,菌株LS2的最大细胞数为1.4×107 CFU/mL;当气体条件为5% O2+20% C2H2时,最大细胞数为2.1× 107 CFU/mL。厌氧条件下,当乙炔浓度为20%时,菌株LS2的最大细胞数为3.3×107 CFU/mL。由此可见,乙炔为电子受体既有利于有氧条件下的菌体生长,又保证了厌氧条件下硫氧化反应的顺利发生。厌氧条件下,以乙炔为电子受体时,菌体生长量是以氧气为单一电子受体时的2倍。说明乙炔为电子受体更有利于菌体生长,它的能量代谢方式可能比氧气为电子受体时更高效。
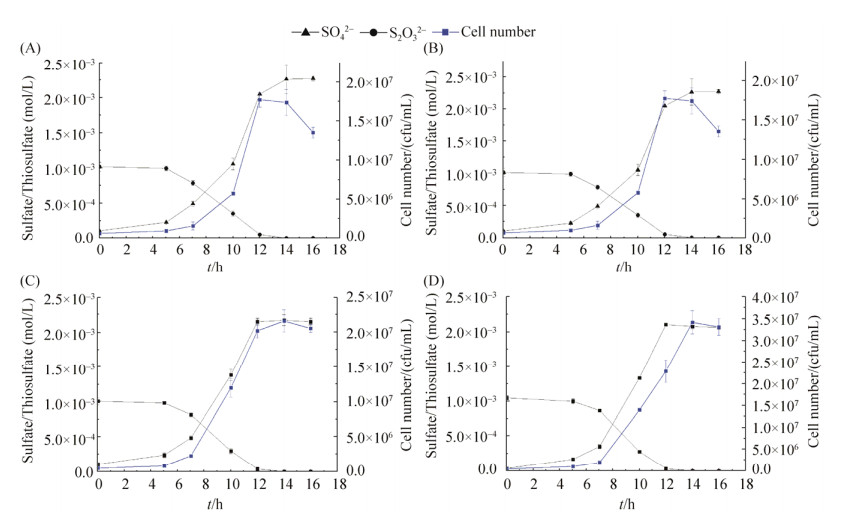 |
| 图 1 菌株LS2在5% O2 A、20% O2 B、5% O2与20% C2H2 C、20% C2H2 D条件下的硫氧化和生长情况 Figure 1 Sulfur oxidation and growth of strain LS2 under 5% O2 A, 20% O2 B, 5% O2 & 20% C2H2 C and 20% C2H2 D. Error bars represent the standard deviation of the mean (n=3). |
| 图选项 |
2.2 以氧气和乙炔为电子受体的硫氧化动力学分析 以氧气和乙炔为电子受体的硫氧化动力学分析如图 2所示,菌株LS2以氧气作为电子受体时,最大反应速率Vmax为1.9×10–5 mol/L SO42–/h (107 cells)–1,Km为3.0×10–5 mol/L S2O32–;乙炔为电子受体时,最大反应速率Vmax为1.7×10–5 mol/L SO42–/h (107 cells)–1,Km为3.0×10–5 mol/L S2O32–。可见,菌株LS2在以氧气或乙炔为电子受体时所进行的硫氧化反应具有相同的底物亲和性(Km),但厌氧硫氧化(C2H2为电子受体)速率略小于好氧硫氧化(O2为电子受体)速率。然而,相较于氧气,菌株LS2在以乙炔电子受体时有更高的生长量(图 1);可以推测,相对于有氧硫氧化反应,菌株LS2以乙炔的厌氧硫氧化过程具有更高的产能效率。
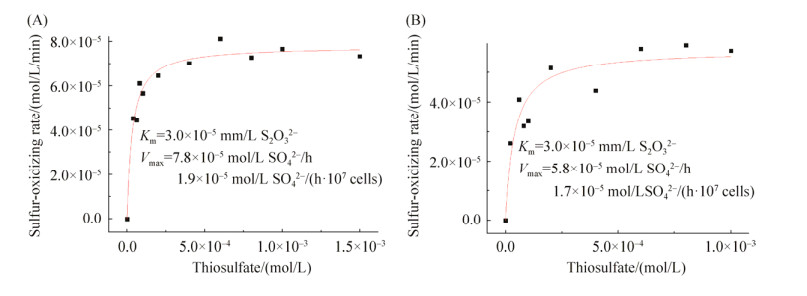 |
| 图 2 菌株LS2以氧气(A)和乙炔(B)为电子受体的硫氧化动力学 Figure 2 Sulfur oxidation kinetics of strain LS2 with oxygen (A) and acetylene (B) as electron acceptors. |
| 图选项 |
2.3 硫氧化酶soxB基因表达分析 菌株LS2硫氧化酶soxB基因在20% O2和20% C2H2,5%、20%、30% C2H2培养条件下的表达情况如图 3所示,硫氧化酶soxB基因的表达量在20% C2H2条件下比20% O2条件下高10.3%。当乙炔浓度分别为5%、20%、30%时,硫氧化酶soxB基因的表达情况随着乙炔浓度的增加而上升,在30%的实验组中表达量最高,较之5%乙炔组提高了31%,说明乙炔可能促进菌株LS2的硫氧化反应。
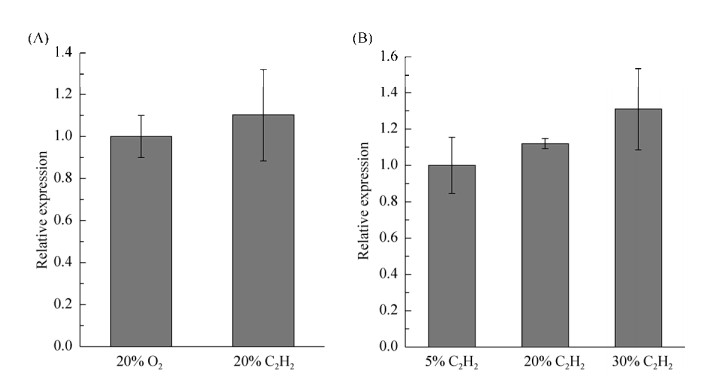 |
| 图 3 硫氧化酶soxB基因在20%氧气和20%乙炔(A),5%、20%、30%乙炔(B)的表达情况 Figure 3 The expression of sulfur oxidase soxB gene in 20% oxygen and 20% acetylene (A), 5%, 20% and 30% acetylene (B). Error bars represent the standard deviation of the mean (n=3). |
| 图选项 |
3 讨论 固氮酶作为催化乙炔还原的关键酶,在介导以乙炔为电子受体的厌氧硫氧化中发挥了核心作用。固氮酶所需电子均来源于还原态的铁氧还蛋白(ferredoxin,Fd)或黄素氧还蛋白(flavodoxin, Fld)。当前已知有6种Fd/Fld还原途径,包括Fix复合体、Rnf (Rhodobacter nitrogen fixation)复合体、丙酮酸-黄素/铁氧还蛋白氧化还原酶、FeFe氢酶、NiFe氢酶和铁氧还蛋白-NADP+氧化还原酶[12-16]。其中,Fix复合体由fixABCX基因编码,Rnf复合体由rnfABCDEG基因编码,两种复合体均可以通过NADH的氧化来还原Fd/Fld[12-14];根据文献报道,严格厌氧菌主要通过丙酮酸-黄素/铁氧还蛋白氧化还原酶、FeFe氢酶、NiFe氢酶的Fd/Fld还原途径,而好氧和兼性厌氧菌为Fd/Fld还原提供电子则需要通过NADH/NADPH的氧化。但是,Fd/Fld (Eo’=–420 mV)的电势低于NADH/ NADPH (Eo’=–320 mV),NADH/NADPH所携带的能量无法为Fd/Fld的还原提供电子而导致无法驱动固氮作用[15]。因此,好氧或兼性厌氧的化能细菌和不产氧光合细菌必须依赖于Fix或Rnf复合体介导的电子分叉(electron bifurcation)作用才能实现对Fd/Fld的还原,并以此驱动固氮作用[12, 15]。
厌氧条件下,硫氧化生成的电子一般传递给NO3–/NO2–、Fe3+、Mn(Ⅳ)等,并通过偶联的反硝化、铁还原、锰还原等作用驱动能量生成。但菌株LS2不能进行反硝化或铁还原,也无法通过这些耦合反应获得能量。基于全基因组分析,菌株LS2基因组中同时发现了fixABCX和rnfABCDEG基因,是目前唯一同时含有Fix和Rnf复合体基因的硫氧化菌[11];根据目前的文献报道,基因组中同时含有Fix复合体和Rnf复合体编码基因的细菌只有7株化能异养固氮菌以及2株互营生长的乙/丙酸氧化细菌[17-20]。菌株LS2的细胞在厌氧条件下仍可将H+泵出胞质并驱动ATP合成,其动力可能源于两种具有Electron Bifurcation功能的Fix和Rnf复合体。它们均可利用NADH(Em’= –320 mV)将铁氧还蛋白(ferredoxin,Fd)或黄素氧还蛋白(flavodoxin,Fld)还原为更低电势的Fd?/Fld? (Em’=–420 mV),从而为固氮作用提供电子。同时,Rnf复合体具有产生跨膜H+/Na+梯度的功能,电子从Fd?反向传递给NAD+时可将胞质内H+/Na+泵出从而用于驱动ATP合成,是一些化能自养产乙酸、产乙醇、产甲烷等厌氧微生物核心的能量生成方式[21-22]。此外,Rnf基因的表达与Nif基因的表达具有相关性,Rnf复合体基因对固氮酶成熟及固氮功能具有调控作用[23-25]。因此,两种electron bifurcatases不仅与能量代谢有关,还可能与固氮酶活性的激活息息相关。
可见,菌株LS2可能通过Fix和Rnf复合体架设的桥联通硫氧化反应和能量产生并将电子传递给乙炔,而乙炔作为电子受体,有氧时促进了硫氧化反应,厌氧时保障了硫氧化反应。其中,在有氧条件下,产生跨膜质子梯度的Rnf复合体是除Complex Ⅳ之外驱动ATP合成的动力,同时是厌氧条件下唯一的能量合成途径;而Fix复合体的主要作用可能是驱动Fd还原,还原态Fd将一部分电子传递给固氮酶用于乙炔还原,另一部分电子传递给Rnf复合体产生跨膜质子梯度驱动ATP的合成;根据菌株LS2以乙炔为电子受体时的生长情况和soxB基因表达情况,Fix和Rnf复合体配合介导的能量生成,不仅保证了厌氧条件下细胞生长,还有利于厌氧条件下的生长代谢。本研究首次提出了以乙炔为电子受体的厌氧硫氧化反应,说明自然界中除NO3–/NO2–、Fe3+、Mn(Ⅳ)等外,乙炔也可以作为SOB介导厌氧硫氧化的电子受体,可能还存在其他物质作为电子受体驱动厌氧硫氧化作用。因而本研究对丰富硫的生物地球化学循环理论、推动硫循环研究有积极意义。
References
| [1] | Stüeken EE, Buick R, Guy BM, Koehler MC. Isotopic evidence for biological nitrogen fixation by molybdenum-nitrogenase from 3.2 Gyr. Nature, 2015, 520(7549): 666-669. DOI:10.1038/nature14180 |
| [2] | Trail D, Watson EB, Tailby ND. The oxidation state of Hadean magmas and implications for early Earth's atmosphere. Nature, 2011, 480(7375): 79-82. DOI:10.1038/nature10655 |
| [3] | 罗剑飞. 硫氧化菌群落结构分析及其特性研究. 华南理工大学博士学位论文, 2011. |
| [4] | Henkel JV, Dellwig O, Pollehne F, Herlemann DPR, Leipe T, Schulz-Vogt HN. A bacterial isolate from the Black Sea oxidizes sulfide with manganese (Ⅳ) oxide. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(25): 12153-12155. DOI:10.1073/pnas.1906000116 |
| [5] | Eisen JA, Nelson KE, Paulsen IT, Heidelberg JF. The complete genome sequence of Chlorobium tepidum TLS, a photosynthetic, anaerobic, green-sulfur bacterium. PNAS, 2002, 99: 9509-9514. DOI:10.1073/pnas.132181499 |
| [6] | Bryant DA, Liu ZF. Green bacteria. Advances in Botanical Research. Amsterdam: Elsevier, 2013: 99-150. |
| [7] | Feniouk BA, Junge W. Proton translocation and ATP synthesis by the FoF1-ATPase of purple bacteria. The Purple Phototrophic Bacteria. Dordrecht: Springer Netherlands, 2009: 475-493. |
| [8] | Kucera J, Pakostova E, Lochman J, Janiczek O, Mandl M. Are there multiple mechanisms of anaerobic sulfur oxidation with ferric iron in Acidithiobacillus ferrooxidans?. Research Microbiology, 2016, 167(5): 357-366. DOI:10.1016/j.resmic.2016.02.004 |
| [9] | Osorio H, Mangold S, Denis Y, ?ancucheo I, Esparza M, Johnson DB, Bonnefoy V, Dopson M, Holmes DS. Anaerobic sulfur metabolism coupled to dissimilatory iron reduction in the extremophile Acidithiobacillus ferrooxidans. Applied and Environmental Microbiology, 2013, 79(7): 2172-2181. DOI:10.1128/AEM.03057-12 |
| [10] | Chen JW, Strous M. Denitrification and aerobic respiration, hybrid electron transport chains and co-evolution. Biochimica et Biophysica Acta: BBA-Bioenergetics, 2013, 1827(2): 136-144. DOI:10.1016/j.bbabio.2012.10.002 |
| [11] | 韩佳悯. 化能自养细菌Halothiobacillus sp. LS2硫氧化和固氮作用的研究. 华南理工大学硕士学位论文. |
| [12] | Poudel S, Colman DR, Fixen KR, Ledbetter RN, Zheng YN, Pence N, Seefeldt LC, Peters JW, Harwood CS, Boyd ES. Electron transfer to nitrogenase in different genomic and metabolic backgrounds. Journal Bacteriology, 2018, 200: e00757-17. |
| [13] | Buckel W, Thauer RK. Flavin-based electron bifurcation, A new mechanism of biological energy coupling. Chemical Reviews, 2018, 118(7): 3862-3886. DOI:10.1021/acs.chemrev.7b00707 |
| [14] | Boyd ES, Costas AMG, Hamilton TL, Mus F, Peters JW. Evolution of molybdenum nitrogenase during the transition from anaerobic to aerobic metabolism. Journal of Bacteriology, 2015, 197(9): 1690-1699. DOI:10.1128/JB.02611-14 |
| [15] | Müller V, Chowdhury NP, Basen M. Electron bifurcation: a long-hidden energy-coupling mechanism. Annual Review of Microbiology, 2018, 72(1): 331-353. DOI:10.1146/annurev-micro-090816-093440 |
| [16] | Greening C, Biswas A, Carere CR, Jackson CJ, Taylor MC, Stott MB, Cook GM, Morales SE. Genomic and metagenomic surveys of hydrogenase distribution indicate H2 is a widely utilised energy source for microbial growth and survival. The ISME Journal, 2016, 10(3): 761-777. DOI:10.1038/ismej.2015.153 |
| [17] | Nishizawa T, Tago K, Oshima K, Hattori M, Ishii S, Otsuka S, Senoo K. Complete genome sequence of the denitrifying and N2O-reducing bacterium Azoarcus sp. strain KH32C. Journal of Bacteriology, 2012, 194(5): 1255. DOI:10.1128/JB.06618-11 |
| [18] | Robson RL, Jones R, Robson RM, Schwartz A, Richardson TH. Azotobacter genomes: the genome of Azotobacter chroococcum NCIMB 8003(ATCC 4412). PLoS One, 2015, 10(6): e0127997. DOI:10.1371/journal.pone.0127997 |
| [19] | Nobu MK, Narihiro T, Rinke C, Kamagata Y, Tringe SG, Woyke T, Liu WT. Microbial dark matter ecogenomics reveals complex synergistic networks in a methanogenic bioreactor. The ISME Journal,, 2015, 9(8): 1710-1722. DOI:10.1038/ismej.2014.256 |
| [20] | Hidalgo-Ahumada CAP, Nobu MK, Narihiro T, Tamaki H, Liu WT, Kamagata Y, Stams AJM, Imachi H, Sousa DZ. Novel energy conservation strategies and behaviour of Pelotomaculum schinkii driving syntrophic propionate catabolism. Environmental Microbiology, 2018, 20(12): 4503-4511. DOI:10.1111/1462-2920.14388 |
| [21] | Buckel W, Thauer RK. Flavin-based electron bifurcation, a new mechanism of biological energy coupling. Chemical Reviews, 2018, 118: 3862-3886. DOI:10.1021/acs.chemrev.7b00707 |
| [22] | Schuchmann K, Müller V. Autotrophy at the thermodynamic limit of life: a model for energy conservation in acetogenic bacteria. Nature Reviews Microbiology, 2014, 12(12): 809-821. DOI:10.1038/nrmicro3365 |
| [23] | Curatti L, Brown CS, Ludden PW, Rubio LM. Genes required for rapid expression of nitrogenase activity in Azotobacter vinelandii. PNAS, 2005, 102(18): 6291-6296. DOI:10.1073/pnas.0501216102 |
| [24] | Barney BM, Plunkett MH, Natarajan V, Mus F, Knutson CM, Peters JW. Transcriptional analysis of an ammonium-excreting strain of Azotobacter vinelandii deregulated for nitrogen fixation. Applied and Environmental Microbiology, 2017, 83(20): e01534-e01517. |
| [25] | Sarkar A, K?hler J, Hurek T, Reinhold-Hurek B. A novel regulatory role of the Rnf complex of Azoarcus sp. strain BH72. Molecular Microbiology, 2012, 83(2): 408-422. DOI:10.1111/j.1365-2958.2011.07940.x |
