李袁飞1,2, 贡继尚1, 饶友生1

 , 成艳芬2
, 成艳芬2
 , 朱伟云2
, 朱伟云2 1. 南昌师范学院生物技术研究所, 江西省地方鸡种遗传改良重点实验室, 江西 南昌 330032;
2. 国家动物消化道营养国际联合研究中心, 江苏省消化道营养与动物健康重点实验室, 南京农业大学消化道 微生物研究室, 江苏 南京 210095
收稿日期:2019-06-27;修回日期:2020-08-09;网络出版日期:2020-10-10
基金项目:南昌师范学院“11531”工程建设项目;国家自然科学基金(31772627);中央高校基本科研业务费(KYDK201701);江苏省一带一路技术合作项目(BZ2018055);南昌师范学院博士科研启动基金(NSBSJJ2019002)
*通信作者:饶友生, Tel:+86-791-83812115, E-mail:rys8323571@aliyun.com;
成艳芬, Tel:+86-25-84395523, E-mail:yanfencheng@njau.edu.cn.
摘要:厌氧真菌是自然界中降解植物纤维素类物质最高效的微生物之一。近年来,大量厌氧真菌和甲烷菌共培养菌株被分离。共培养中,甲烷菌通过对厌氧真菌代谢产物的利用显著提高厌氧真菌对木质纤维素的降解;厌氧真菌通过为甲烷菌提供能量和营养物质使甲烷菌快速生成大量甲烷。全面深入地了解共培养中两者的互作关系以及共培养降解木质纤维素产甲烷的特性,将有助于研究对木质纤维素降解以及甲烷生成的调控。因此,本文主要综述了共培养的分离鉴定、多样性、互作关系以及对木质纤维素的降解。
关键词:厌氧真菌甲烷菌共培养木质纤维素降解甲烷
Advance in the co-culture of anaerobic fungi and methanogens
Yuanfei Li1,2, Jishang Gong1, Yousheng Rao1

 , Yanfen Cheng2
, Yanfen Cheng2
 , Weiyun Zhu2
, Weiyun Zhu2 1. Jiangxi Province Key Laboratory of Genetic Improvement of Indigenous Chicken Breeds, Institute of Biological Technology, Nanchang Normal University, Nanchang 330032, Jiangxi Province, China;
2. Laboratory of Gastrointestinal Microbiology, Jiangsu Key Laboratory of Gastrointestinal Nutrition and Animal Health, National Center for International Research on Animal Gut Nutrition, Nanjing Agricultural University, Nanjing 210095, Jiangsu Province, China
Received: 27 June 2019; Revised: 9 August 2020; Published online: 10 October 2020
*Corresponding author: Yousheng Rao, Tel:+86-791-83812115, E-mail:rys8323571@aliyun.com;
Yanfen Cheng, Tel:+86-25-84395523, E-mail:yanfencheng@njau.edu.cn.
Foundation item: Supported by the 11531 Engineering Construction Projects of Nanchang Normal University, by the National Natural Science Foundation of China (31772627), by the Fundamental Research Funds for the Central Universities (KYDK201701), by the Belt and Road Technical Cooperation Project in Jiangsu Province (BZ2018055) and by the Doctoral Research Startup Foundation of Nanchang Normal University (NSBSJJ2019002)
Abstract: Anaerobic fungi are among the most effective lignocelluloses-degrading microbes in nature. Recently, increasing number of the co-cultures of anaerobic fungi with methanogens have been isolated. In the co-culture, methanogens utilize metabolites produced by anaerobic fungi, enhancing the lignocelluloses-degrading ability of anaerobic fungi; anaerobic fungi provide methanogens with energy and nutrients, with which methanogens rapidly generate substantial methane. A comprehensive and in-depth understanding of the mutual interaction and the lignocelluloses-degrading methane-producing characteristics of the co-culture will be conducive to investigating the regulation of the lignocellulosic degradation and methane production. Thus, in this review, we summarized the isolation and identification, the diversity and the mutual interaction of the co-culture, and the decomposition of lignocellulosic material by the co-culture.
Keywords: anaerobic fungimethanogensco-culturedegradation of lignocellulosemethane
瘤胃是一个粗纤维降解效率极高且高度进化的天然体系,此体系中栖息着包括细菌、甲烷菌、原虫和厌氧真菌在内的大量微生物[1]。这些相互依存和制约的微生物对反刍动物瘤胃粗纤维降解、机体健康以及生产性能起着举足轻重的作用[2]。瘤胃中的厌氧真菌通过假根系统可以快速(摄食5 min之内)定殖到植物组织上,并刺穿植物细胞壁,同时通过分泌的一系列木质纤维素降解酶降解植物组织[3-4]。瘤胃内厌氧真菌对日粮中粗纤维的降解起着十分重要的作用,其粗纤维降解能力高于细菌和原虫[5-6]。虽然瘤胃内厌氧真菌的数量约为细菌的万分之一、原虫的百分之一[7],但却能降解瘤胃中8%-20%的粗饲料[8]。甲烷菌严格厌氧,它们司职有机物厌氧降解的最后一个环节。甲烷菌只能利用一些简单的化合物(如氢气、二氧化碳、甲酸等)合成甲烷,并从中获取生长所需能量。在瘤胃内,甲烷菌是主要的氢利用菌,它们通过甲烷生成作用消耗电子、维持氢分压、解除纤维降解微生物的产物抑制作用和促进植物组织的降解,对瘤胃的高效发酵起着重要作用[9]。大量研究表明,厌氧真菌和甲烷菌之间存在着十分紧密的关系:通过富集培养的方式能够得到厌氧真菌和甲烷菌的混合共培养,而通过亨氏(Hungate)厌氧滚管技术则可以分离到厌氧真菌和甲烷菌的自然共培养,并且这两种共培养能够在以木质纤维素(如稻秸、麦秸)为底物的培养基中长期稳定传代培养。为了充分了解厌氧真菌和甲烷菌两者的互作关系,本文就厌氧真菌和甲烷菌共培养的分离鉴定、共培养的多样性、共培养中两者的位置和代谢关系以及共培养对木质纤维素的降解进行综述,以期为促进厌氧真菌对粗纤维的降解、更好地理解厌氧真菌与甲烷菌之间互作关系对瘤胃粗纤维降解的作用和利用厌氧真菌和甲烷菌共培养来提高生物质资源的利用提供基础并有所启发。
1 厌氧真菌和甲烷菌共培养的分离和鉴定 1.1 共培养的分离 目前厌氧微生物主要是通过亨氏厌氧滚管技术进行分离。厌氧真菌和甲烷菌共培养的分离与厌氧真菌的分离基本一致,皆采用亨氏厌氧滚管技术进行分离。1981年,Bauchop等[10]采用亨氏厌氧滚管技术从绵羊瘤胃内分离厌氧真菌时,首次获得了一株厌氧真菌(Neocallimastix)和甲烷菌(Methanobrevibacter)的共培养。这是关于厌氧真菌和甲烷菌共培养的第一篇报道,该研究表明,甲烷菌能够与厌氧真菌紧密共存(对厌氧真菌进行多次稀释和洗脱,该培养物依然能产生甲烷);而且此共培养菌株降解纤维和产甲烷的特性很稳定(在液体培养基中经过一年的传代,其降解纤维的能力和产甲烷的能力未减退)。2002年,Joblin等[11]运用亨氏厌氧滚管技术从绵羊体内分离到了一株厌氧真菌(Neocallimastix frontalis PNK2)和甲烷菌(Methanobrevibacter smithii PS)的共培养。Cheng等[12] (2009年)通过在培养基中加入青霉素和链霉素(厌氧真菌和甲烷菌对青霉素和链霉素不敏感),利用连续传代富集的方法获得了厌氧真菌和甲烷菌的混合共培养。2011年,Jin等[13]利用青链霉素结合亨氏厌氧滚管的方法从不同草食动物食糜或粪便中分离获得了8株共培养。同样的,孙美洲等[14] (2014年)和Wei等[15] (2016年)皆利用青链霉素结合亨氏厌氧滚管的方法分别从奶牛瘤胃和牦牛瘤胃内分离得到了28株和20株共培养。目前,“青链霉素+亨氏厌氧滚管”的方法是分离两者共培养的最主要手段。
1.2 共培养的鉴定 厌氧真菌和甲烷菌共培养中同时存在着厌氧真菌和甲烷菌,因此需要采用各自相应的方法进行鉴定,以确保分离的培养物为两者的共培养。每一次挑菌之前需要对滚管中的气体进行甲烷检测,以确保甲烷菌的存在从而避免后续徒劳的工作。目前气相色谱法是甲烷检测的有效而常用的方法[10, 12, 14]。
共培养中厌氧真菌传统的鉴定方法为细胞形态学观察,该方法是通过对厌氧真菌进行染色(DAPI染色),然后利用显微镜观察菌体的生长类型、假根形态、游动孢子鞭毛数以及游动孢子的超显微结构,从而将厌氧真菌进行鉴定和分类至属[13-14, 16]。然而,目前仅仅依靠形态学特征已不能很好地对厌氧真菌进行鉴定与分类:(1)越来越多的厌氧真菌被分离和发现;(2)不同的培养基种类、培养底物和生长阶段都会对厌氧真菌的细胞形态结构产生影响[16]。因此,“细胞形态学+分子生物学”的方法已成为目前对厌氧真菌进行鉴定和分类的首选方法。ITS和28S rRNA序列分析法是目前常用的厌氧真菌分子学鉴定方法[17]。Leis等[18]和Wei等[15]利用形态学观察法结合分子生物学法(ITS序列分析法)对共培养中的厌氧真菌进行了鉴定和分类。
共培养中甲烷菌的鉴定主要采用分子生物学方法:以16S rRNA基因或mcrA基因来进行分类鉴定。Jin等[13]通过对共培养菌株中甲烷菌的16S rRNA基因进行测序,成功鉴定了8株共培养菌株中的甲烷菌。同样的,Leis等[18]和孙美洲等[14]皆采用16S rRNA基因测序的方法对共培养中的甲烷菌进行了鉴定。甲基辅酶M还原酶(MCR)是产甲烷菌生成甲烷最后一步反应的催化酶,存在所有产甲烷菌中;MCR由3个不同的亚单位(α,β,γ)组成,其中的α亚单位由mcrA基因编码[19]。Ozutsumi等[20]通过建立mcrA基因克隆文库的方法分析了瘤胃液中甲烷菌的多样性。李袁飞等[21]建立了产甲烷菌mcrA基因的T-RFLP (末端限制性片段长度多态性)分析方法,并利用该方法研究了厌氧真菌与产甲烷菌混合共培养系统中产甲烷菌的多样性。
2 厌氧真菌和甲烷菌共培养的多样性 在混合共培养中存在着多种厌氧真菌和甲烷菌,在长时间的传代培养过程中,厌氧真菌和甲烷菌的多样性变化存在一定差异。成艳芬等[22]研究表明,在每3-4 d传代1次的厌氧真菌和甲烷菌混合共培养中,从分离至第25代,甲烷菌多样性不断降低,而从第25代至第62代多样性逐渐趋于稳定。而且该混合共培养中存在着尚未分离培养的瘤胃甲烷菌。成艳芬等[23]研究发现,在每3 d传代1次的厌氧真菌和甲烷菌混合共培养中,厌氧真菌多样性在传代过程中降低,传代4次后厌氧真菌多样性趋于稳定。Cheng等[12]的结果同样表明,在厌氧真菌和甲烷菌混合共培养传代培养过程中,厌氧真菌多样性比甲烷菌多样性能更快地趋于稳定。这可能是因为培养基中的底物(供厌氧真菌利用)相对单一,而厌氧真菌则能够为甲烷菌提供相对充足的底物(如甲酸、氢气、二氧化碳)。
在目前分离得到的厌氧真菌和甲烷菌自然共培养中,一种厌氧真菌对应一种甲烷菌。Jin等[13]从不同草食动物食糜或粪便中分离到了8株共培养,共培养中的甲烷菌都为Methanobrevibacter,厌氧真菌Piromyces有6株,Neocallimastix和Anaeromyces各1株。孙美洲等[14]从荷斯坦奶牛瘤胃中分离到了28株共培养,其中的甲烷菌都为Methanobrevibacter,厌氧真菌Piromyces有15株,Neocallimastix有12株,Caeomyces有1株。Wei等[15]从牦牛瘤胃中分离到的20株共培养中,甲烷菌均为Methanobrevibacter,厌氧真菌归属于Orpinomyces (9株)、Neocallimastix (8株)和Piromyces (3株)。目前有文献报道的分离纯化的共培养菌株仅有59株,共培养中的厌氧真菌主要为Piromyces和Neocallimastix;共培养中的甲烷菌绝大部分为甲烷短杆菌属(图 1)。宿主肠道类型、地域和日粮等因素都影响着厌氧真菌和甲烷菌的多样性[24-26]。因此,大量分离不同环境下(如宿主、肠道、地域或者日粮等)厌氧真菌和甲烷菌共培养菌株可以拓展对其多样性的理解。
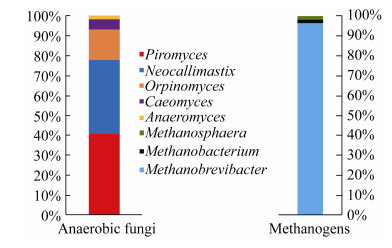 |
| 图 1 已报道的自然共培养菌株中厌氧真菌和甲烷菌属的丰度[10, 13-15, 18] Figure 1 The abundance of genus of anaerobic fungus or methanogen calculated from the reported total natural co-culture. Up to April 20, 2020, the number of the reported natural co-culture is 59. |
| 图选项 |
3 共培养中厌氧真菌和甲烷菌的关系 3.1 共培养中厌氧真菌和甲烷菌物理位置上的关系 共培养中的厌氧真菌和甲烷菌之间在物理位置上存在着十分紧密的关系。Bauchop等[10]研究表明,有大量甲烷菌紧密附着于厌氧真菌假根上。Jin等[13]和wei等[15]的结果表明,在分离得到的厌氧真菌和甲烷菌共培养中厌氧真菌和甲烷菌是“一对一”的关系,并且甲烷菌附着于厌氧真菌的假根表面(图 2)。Leis等[18]利用FISH技术研究发现,在厌氧真菌的孢子囊表面附着着大量的甲烷菌。在共培养中,甲烷菌紧密附着于厌氧真菌表面的原因可能是:甲烷菌能够更高效地利用厌氧真菌代谢产生的物质。目前还不清楚在共培养中厌氧真菌胞内是否存在着共生的甲烷菌。孙美洲等[14]在分离纯化厌氧真菌和甲烷菌共培养过程中,发现有些共培养中的甲烷菌逐渐消失,而这些厌氧真菌在甲烷菌消失后依然生长良好。这暗示厌氧真菌胞内可能没有共生的甲烷菌。另外,孙美洲等[14]也发现共培养中厌氧真菌与甲烷菌是“一对一”的共生关系。Mountfort等[27]和Nakashimada等[28]通过人为混合培养的方式将厌氧真菌与两不同属的甲烷菌进行混合培养,结果发现三者的共培养比厌氧真菌与其中任一甲烷菌的共培养对底物的降解和甲烷的生成都要高。然而,这并不能否定厌氧真菌与甲烷菌“一对一”的共生关系:首先,这些共培养是人为混合的;其次,这些共培养能否长久共存并不可知。通过将不同类型的甲烷菌接种到各厌氧真菌和甲烷菌自然共培养中并研究它们的特性将有助于进一步了解厌氧真菌与甲烷菌的共生机制。
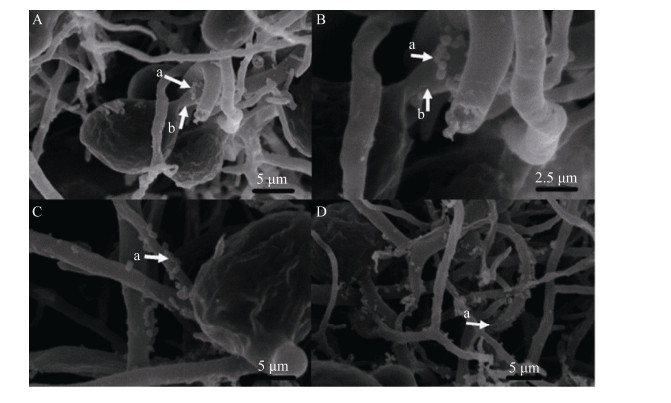 |
| 图 2 共培养(Piromyces+Methanobrevibacter)的扫描电镜图[13] Figure 2 The SEM photographs of co-culture of Piromyces and Methanobrevibacter[13]. The letter "a" indicates the indigenously associated methanogens; the letter "b" indicates the rhizoids of anaerobic fungus. |
| 图选项 |
3.2 共培养中厌氧真菌和甲烷菌在代谢上的关系 厌氧真菌发酵产生的主要代谢产物包括氢气、甲酸、乙酸、乳酸和乙醇[29-31]。甲烷菌只能利用氢气、二氧化碳、甲酸、甲醇、乙酸等简单的化合物合成甲烷,并从中获取生长所需能量[32]。在厌氧真菌和甲烷菌共培养中,厌氧真菌和甲烷菌之间通过种间氢转移可以长期稳定共存[10, 33]:厌氧真菌提供甲烷菌生长所需的能量物质;甲烷菌消除厌氧真菌代谢产物的抑制作用,促进厌氧真菌的生长代谢。共培养代谢产物中几乎没有氢气和甲酸的积累,乳酸和乙醇的含量显著低于厌氧真菌纯培养,而乙酸含量则显著高于厌氧真菌纯培养[10, 13, 33-34]。Cheng等[35]利用NMR (核磁共振光谱)技术在共培养代谢产物中检测到了柠檬酸。同样的,Li等[36]通过GC/MS技术研究发现,在共培养的厌氧真菌胞质中有柠檬酸的积累。厌氧真菌胞质中不含线粒体,而是在氢化酶体(胞内以生物膜隔离的小区室)中通过底物水平磷酸化作用生成ATP[37]。苹果酸和丙酮酸进入氢化酶体后被代谢为H2、CO2、甲酸和乙酸,同时伴随着ATP的产生;而细胞质中则主要代谢产生乳酸和乙醇[38]。在共培养中,甲烷菌改变了厌氧真菌的代谢:更多的苹果酸和丙酮酸进入氢化酶体,氢化酶体的代谢更为旺盛,产生更多的乙酸和ATP (图 3)[33-34, 39]。这种改变的原因可能为:厌氧真菌产生的氢气被甲烷菌所利用,使体系内的氢分压降低,解除了氢气对氢化酶的抑制,促进了氢化酶体内NAD(P)H生成H2的途径,因而使更多的碳水化合物进入氢化酶体,最终生成更多的乙酸和ATP;而胞质中代谢产生乳酸和乙醇皆需要NAD(P)H的参与,因此乳酸和乙醇的生成相应地被抑制[31, 33, 40]。另外,甲烷菌对甲酸的利用可以提高发酵体系的pH,使pH更有利于厌氧真菌的生长[33]。共培养所用培养基与厌氧真菌相同,培养基成分中无H2,甲酸盐的浓度很低,然而共培养中24 h就可以检测到大量甲烷[13, 15]。当共培养中的厌氧真菌被抑制时,即使在培养体系中添加了甲烷菌的能量物质(H2和CO2或甲酸盐),甲烷菌也不能在此培养体系中生长[33-34]。这表明共培养中的厌氧真菌为甲烷菌提供能量物质的同时,也提供某些生长所需的营养物质。代谢组学技术已经被用于研究共培养中厌氧真菌和甲烷菌之间的代谢关系[35-36],随着代谢组学技术的进一步发展和运用,厌氧真菌和甲烷菌之间在代谢方面的关系也将被逐步揭示。
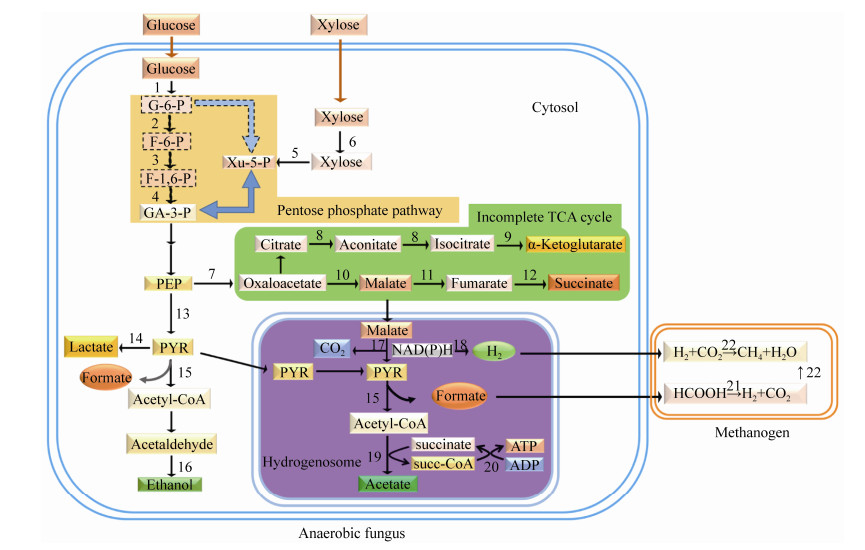 |
| 图 3 甲烷菌共存影响厌氧真菌代谢途径的模式图 Figure 3 Proposed metabolic pathway of anaerobic fungi co-cultured with methanogens. In the co-culture, hydrogen and formate produced by anaerobic fungi were the substrates available for methanogens. The numbers in the metabolic pathway indicate the following enzymes: 1: hexokinase; 2: glucose-6-phosphate isomerase; 3: phosphofructokinase; 4: fructose-bisphosphate aldolase; 5: xylulokinase; 6: xylose isomerase; 7: phosphoenolpyruvate carboxykinase; 8: aconitase; 9: isocitrate dehydrogenase; 10: malate dehydrogenase; 11: fumarase; 12: fumarate reductase; 13: pyruvate kinase; 14: lactate dehydrogenase; 15: pyruvate formate lyase; 16: aldehyde/alcohol dehydrogenase; 17: malic enzyme; 18: hydrogenase; 19: acetate:succinate CoA-transferase; 20: succinyl-CoA synthetase; 21: formate dehydrogenase; 22: methyl coenzyme-M reductase, the key enzyme of methanogenesis. |
| 图选项 |
4 厌氧真菌和甲烷菌共培养对木质纤维素的降解 厌氧真菌通过假根系统机械性穿透植物细胞壁的角质层并对木质素进行侵袭,同时分泌一系列高活性的植物细胞壁降解酶对植物细胞内腔进行酶解[16, 41]。厌氧真菌分泌的木聚糖酶的活性是目前市场上应用的木聚糖酶活性的2倍[4]。大量报道表明,相比于厌氧真菌纯培养,厌氧真菌和甲烷菌共培养对粗纤维的降解显著提高。Bauchop等[10]发现共培养发酵末期(170 h)的纤维素降解率高于纯培养发酵末期(280 h)的纤维素降解率(82% vs. 53%)。Mountfort等[27]和Nakashimada等[28]研究表明,厌氧真菌和两种甲烷菌(一种利用H2和CO2或甲酸,一种利用乙酸)的人为混合共培养降解木质纤维素的效率高于厌氧真菌与其中任一种甲烷菌的共培养。同样的,Joblin等[42]通过人为地将不同属的厌氧真菌分别和甲烷菌(Methanobrevibacter smithii PS)进行共培养,结果表明这些人为获得的共培养对燕麦秸的降解率皆高于各自的厌氧真菌纯培养。Joblin等[43]发现甲烷菌共存提高了厌氧真菌对木聚糖的降解,且共培养木聚糖酶活性比纯培养木聚糖酶活性高5倍之多。Jin等[13]和Wei等[44]研究表明,共培养对木质纤维素底物的降解率高于纯培养,其木质纤维素降解酶(如木聚糖酶、羧甲基纤维素酶、阿魏酸酯酶、乙酰酯酶等)的活性显著高于纯培养。由上述报道可知,不管是分离到的厌氧真菌和甲烷菌自然共培养还是人为混合的厌氧真菌和甲烷菌共培养,其分泌的植物细胞壁降解酶活性以及对木质纤维素的利用率都显著高于纯培养。共培养降解木质纤维素高于纯培养的主要原因为:(1)厌氧真菌代谢产生的氢气和甲酸被甲烷菌所利用,增强了厌氧真菌ATP的生成途径,因此厌氧真菌能够获取更多的能量以供其在无产物抑制条件下快速生长[34, 39];厌氧真菌快速生长,碳水化合物活性酶(CAZyme)的转录增加,进而木质纤维素降解酶的产量也显著提高[12, 45]。(2)甲烷菌共存时促进厌氧真菌糖转运体的转录[46],加速酶解产生的游离单糖的消耗[47],解除了产物增多对酶活性的反馈抑制[4, 48]。
厌氧真菌和甲烷菌共培养可以高效降解未经处理的木质纤维素,同时在短时间内可以生成大量甲烷。尽管各类厌氧真菌和甲烷菌共培养(人为混合的或者自然分离的;厌氧真菌和甲烷菌不同的组合)可以降解各种未经处理的木质纤维素材料(如稻秸、麦秸、燕麦秸、剑麻纤维等)产生甲烷,然而产甲烷的效率各异,这可能归结于:厌氧真菌对不同底物的降解存在差异;不同厌氧真菌对同一底物的降解存在差异;不同营养类型的甲烷菌对厌氧真菌代谢产物的利用效率不同;培养的时间也会影响甲烷的产量(表 1)。厌氧真菌同时代谢产生氢气、二氧化碳、甲酸和乙酸,当不同营养类型的甲烷菌(氢气营养型和乙酸营养型)同时与厌氧真菌进行共培养时可显著提高甲烷的产量[27-28]。Procházka等[49]研究表明,在半连续培养条件下添加厌氧真菌可提高沼气的产量和质量。因此,利用厌氧真菌和甲烷菌共培养降解废弃的生物质材料生产沼气具有很大的工业应用前景。通过大量分离厌氧真菌和甲烷菌共培养并研究其降解木质纤维素产甲烷的性质是筛选高效降解木质纤维素高产甲烷共培养菌株的常规方法[13, 15]。随着对厌氧真菌基因组、转录组、蛋白组以及代谢组研究的不断深入,基因工程和代谢工程将被用于高效降解木质纤维素高产甲烷共培养菌株的选育[50-51]。
表 1. 厌氧真菌和甲烷菌共培养降解木质纤维素产甲烷的量 Table 1. Methane production of co-culture of anaerobic fungus and methanogen with lignocelluloses as substrate
| Type of co-culture | Hydrogenotrophic methanogenesis | Aceticlastic methanogenesis | Substrates | Culture time | Methane production | References |
| Natural co-culture1 | + | - | Filter paper | 170 h | 2.9 mmol/g | [10] |
| Artificial co-culture2 | + | - | NH3 treated barley straw stems | 10 d | 23.3-33.3 mL/g | [42] |
| Artificial co-culture | + | - | Filter paper | 7 d | 3.0 mmol/g | [27] |
| Artificial co-culture | + | + | Filter paper | 16 d | 12.2 mmol/g | |
| Artificial co-culture | + | - | Sisal twine fiber | 7 d | 2.1 mmol/g | |
| Artificial co-culture | + | + | Sisal twine fiber | 19 d | 10.1 mmol/g | |
| Artificial co-culture | + | - | Barley straw leaf strips | 7 d | 1.7 mmol/g | |
| Artificial co-culture | + | + | Barley straw leaf strips | 19 d | 7.0 mmol/g | |
| Artificial co-culture | + | - | Non-autoclaved ryegrass stem | 6 d | 4.8-10.8 mL/g | [11] |
| Artificial co-culture | + | - | Cellulose powder | 7 d | 3.8-4.0 mmol/g | [28] |
| Artificial co-culture | - | + | Cellulose powder | 5 d | 0.5-2 mmol/g | |
| Artificial co-culture | - | + | Cellulose powder | 17 d | 0.5-3.5 mmol/g | |
| Artificial co-culture | + | + | Cellulose powder | 17 d | 6.3 mmol/g | |
| Natural co-culture | + | - | Rice straw | 4 d | 0.5-1.3 mmol/g | [13] |
| Natural co-culture | + | - | Wheat straw | 4 d | 1.0-1.2 mmol/g | |
| Natural co-culture | + | - | Maize stem | 4 d | 0.6-0.7 mmol/g | |
| Natural co-culture | + | - | Corncob | 4 d | 1.7-2.2 mmol/g | |
| Natural co-culture | + | - | Bagasse | 4 d | 1.6-1.8 mmol/g | |
| Natural co-culture | + | - | DGGS | 4 d | 1.1-1.2 mmol/g | |
| Natural co-culture | + | - | Wheat bran | 4 d | 1.1-1.2 mmol/g | |
| Natural co-culture | + | - | Wheat straw | 7 d | 0.3-1.8 mmol/g | [15] |
| 1: The co-culture obtained just by the Hungate roll-tube techniques; 2: The co-culture obtained by a mix of axenic fungal and methangenic species. +: the methanogen was present; -: the methanogen was absent. | ||||||
表选项
5 存在的问题及展望 近年来,随着越来越多的厌氧真菌和甲烷菌自然共培养被分离和研究,其相对于厌氧真菌纯培养的优势逐渐为人们所了解和重视,并有望在农业、能源等领域得到拓展和应用,然而目前仍然存在着一些亟待解决的问题。如:(1)在共培养中,厌氧真菌和甲烷菌数量之间是否存在一个最佳比例;(2)除为甲烷菌提供能源物质外,厌氧真菌还为甲烷菌提供什么营养物质;(3)甲烷菌对厌氧真菌木质纤维素酶基因的转录和表达存在什么影响。针对上述第一个问题,研究者可以人为地将厌氧真菌和甲烷菌进行不同比例的混合,测定不同比例下的生长情况以及底物的降解情况。针对上述第二个问题,研究者可以通过代谢组学来分析厌氧真菌纯培养、甲烷菌纯培养以及两者共培养中代谢产物的变化以揭示两者交互饲喂的关系。针对第三个问题,研究者可以利用转录组学和蛋白组学技术来研究厌氧真菌纯培养以及厌氧真菌和甲烷菌共培养以不同木质纤维素材料为底物时木质纤维素酶基因的转录和表达,进一步阐明甲烷菌共存对厌氧真菌产木质纤维素酶的影响。
在共培养中,厌氧真菌为甲烷菌提供能量和营养物质,维持甲烷菌的生长;甲烷菌则通过对厌氧真菌代谢产物的利用来改善厌氧真菌的生长环境,促进厌氧真菌的生长和对底物的降解。这种关系使两者能够长期稳定共存,因此,厌氧真菌和甲烷菌共培养的大量分离可为新的甲烷菌的发现或分离提供基础和新思路。当以各类木质纤维素为底物时,共培养中的厌氧真菌会分泌活性很高的木质纤维素降解酶(如木聚糖酶、纤维素水解酶和酯酶等),而且还能促使甲烷菌快速生成大量甲烷。由此可见,优良的厌氧真菌和甲烷菌共培养菌株具有很大的工业应用潜能:既可以用于木质纤维素酶也可以用于生物质能源的生产。而通过进一步研究共培养中厌氧真菌和甲烷菌之间的关系及其共生或互作的机制,将有助于调控瘤胃内粗纤维的降解以及甲烷的生成。
References
| [1] | Weimer PJ, Russell JB, Muck RE. Lessons from the cow: what the ruminant animal can teach us about consolidated bioprocessing of cellulosic biomass. Bioresource Technology, 2009, 100(21): 5323-5331. DOI:10.1016/j.biortech.2009.04.075 |
| [2] | Li FY, Guan LL. Metatranscriptomic profiling reveals linkages between the active rumen microbiome and feed efficiency in beef cattle. Applied and Environmental Microbiology, 2017, 83(9): e00061-17. |
| [3] | Edwards JE, Kingston-Smith AH, Jimenez HR, Huws SA, Sk?t KP, Griffith GW, McEwan NR, Theodorou MK. Dynamics of initial colonization of nonconserved perennial ryegrass by anaerobic fungi in the bovine rumen. FEMS Microbiology Ecology, 2008, 66(3): 537-545. DOI:10.1111/j.1574-6941.2008.00563.x |
| [4] | Solomon KV, Haitjema CH, Henske JK, Gilmore SP, Borges-Rivera D, Lipzen A, Brewer HM, Purvine SO, Wright AT, Theodorou MK, Grigoriev IV, Regev A, Thompson DA, O'Malley MA. Early-branching gut fungi possess a large, comprehensive array of biomass-degrading enzymes. Science, 2016, 351(6278): 1192-1195. DOI:10.1126/science.aad1431 |
| [5] | Akin DE, Borneman WS, Lyon CE. Degradation of leaf blades and stems by monocentric and polycentric isolates of ruminal fungi. Animal Feed Science and Technology, 1990, 31(3/4): 205-221. |
| [6] | Lee SS, Ha JK, Cheng KJ. Relative contributions of bacteria, protozoa, and fungi to in vitro degradation of orchard grass cell walls and their interactions. Applied and Environmental Microbiology, 2000, 66(9): 3807-3813. DOI:10.1128/AEM.66.9.3807-3813.2000 |
| [7] | Jenkins TC, Wallace RJ, Moate PJ, Mosley EE. Board-invited review: Recent advances in biohydrogenation of unsaturated fatty acids within the rumen microbial ecosystem. Journal of Animal Science, 2008, 86(2): 397-412. |
| [8] | Rezaeian M, Beakes GW, Parker DS. Distribution and estimation of anaerobic zoosporic fungi along the digestive tracts of sheep. Mycological Research, 2004, 108(10): 1227-1233. DOI:10.1017/S0953756204000929 |
| [9] | Ungerfeld EM. Inhibition of rumen methanogenesis and ruminant productivity: a meta-analysis. Frontiers in Veterinary Science, 2018, 5: 113. DOI:10.3389/fvets.2018.00113 |
| [10] | Bauchop T, Mountfort DO. Cellulose fermentation by a rumen anaerobic fungus in both the absence and the presence of rumen methanogens. Applied and Environmental Microbiology, 1981, 42(6): 1103-1110. DOI:10.1128/AEM.42.6.1103-1110.1981 |
| [11] | Joblin KN, Matsui H, Naylor GE, Ushida K. Degradation of fresh ryegrass by methanogenic co-cultures of ruminal fungi grown in the presence or absence of Fibrobacter succinogenes. Current Microbiology, 2002, 45(1): 46-53. DOI:10.1007/s00284-001-0078-5 |
| [12] | Cheng YF, Edwards JE, Allison GG, Zhu WY, Theodorou MK. Diversity and activity of enriched ruminal cultures of anaerobic fungi and methanogens grown together on lignocellulose in consecutive batch culture. Bioresource Technology, 2009, 100(20): 4821-4828. DOI:10.1016/j.biortech.2009.04.031 |
| [13] | Jin W, Cheng YF, Mao SY, Zhu WY. Isolation of natural cultures of anaerobic fungi and indigenously associated methanogens from herbivores and their bioconversion of lignocellulosic materials to methane. Bioresource Technology, 2011, 102(17): 7925-7931. DOI:10.1016/j.biortech.2011.06.026 |
| [14] | Sun MZ, Jin W, Li YF, Mao SY, Cheng YF, Zhu WY. Isolation and identification of cellulolytic anaerobic fungi and their associated methanogens from Holstein Cow. Acta Microbiologica Sinica, 2014, 54(5): 563-571. (in Chinese) 孙美洲, 金巍, 李袁飞, 毛胜勇, 成艳芬, 朱伟云. 瘤胃降解粗纤维产甲烷的厌氧真菌与甲烷菌共培养物的分离鉴定. 微生物学报, 2014, 54(5): 563-571. |
| [15] | Wei YQ, Yang HJ, Luan Y, Long RJ, Wu YJ, Wang ZY. Isolation, identification and fibrolytic characteristics of rumen fungi grown with indigenous methanogen from yaks (Bos grunniens) grazing on the Qinghai-Tibetan Plateau. Journal of Applied Microbiology, 2016, 120(3): 571-587. DOI:10.1111/jam.13035 |
| [16] | Haitjema CH, Solomon KV, Henske JK, Theodorou MK, O'Malley MA. Anaerobic gut fungi: advances in isolation, culture, and cellulolytic enzyme discovery for biofuel production. Biotechnology and Bioengineering, 2014, 111(8): 1471-1482. DOI:10.1002/bit.25264 |
| [17] | Edwards JE, Forster RJ, Callaghan TM, Dollhofer V, Dagar SS, Cheng YF, Chang J, Kittelmann S, Fliegerova K, Puniya AK, Henske JK, Gilmore SP, O'Malley MA, Griffith GW, Smidt H. PCR and omics based techniques to study the diversity, ecology and biology of anaerobic fungi: insights, challenges and opportunities. Frontiers in Microbiology, 2017, 8: 1657. DOI:10.3389/fmicb.2017.01657 |
| [18] | Leis S, Dresch P, Peintner U, Fliegerová K, Sandbichler AM, Insam H, Podmirseg SM. Finding a robust strain for biomethanation: anaerobic fungi (Neocallimastigomycota) from the Alpine ibex (Capra ibex) and their associated methanogens. Anaerobe, 2014, 29: 34-43. DOI:10.1016/j.anaerobe.2013.12.002 |
| [19] | Friedrich MW. Methyl-coenzyme M reductase genes: unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology, 2005, 397: 428-442. DOI:10.1016/S0076-6879(05)97026-2 |
| [20] | Ozutsumi Y, Tajima K, Takenaka A, Itabashi H. The mcrA gene and 16S rRNA gene in the phylogenetic analysis of methanogens in the rumen of faunated and unfaunated cattle. Animal Science Journal, 2012, 83(11): 727-734. DOI:10.1111/j.1740-0929.2012.01023.x |
| [21] | Li YF, Cheng YF, Zhu WY. Effects of transfer frequency on community of methanogens co-cultured with anaerobic fungi by T-RFLP. Microbiology China, 2015, 42(3): 609-619. (in Chinese) 李袁飞, 成艳芬, 朱伟云. T-RFLP分析厌氧真菌传代频率对共存产甲烷菌菌群的影响. 微生物学通报, 2015, 42(3): 609-619. |
| [22] | Cheng YF, Mao SY, Pei CX, Liu JX, Zhu WY. Detection and diversity analysis of rumen methanogens in the co-cultures with anaerobic fungi. Acta Microbiologica Sinica, 2006, 46(6): 879-883. (in Chinese) 成艳芬, 毛胜勇, 裴彩霞, 刘建新, 朱伟云. 共存于厌氧真菌分离培养液中瘤胃甲烷菌的检测及其多样性分析. 微生物学报, 2006, 46(6): 879-883. |
| [23] | Cheng YF, Zhu WY. Diversity analysis of anaerobic fungi in the co-cultures with or without methanogens by amplified ribosomal intergenic spacer analysis. Acta Microbiologica Sinica, 2009, 49(4): 504-511. (in Chinese) 成艳芬, 朱伟云. ARISA方法研究产甲烷菌共存及去除条件下瘤胃真菌多样性变化. 微生物学报, 2009, 49(4): 504-511. |
| [24] | Gruninger RJ, Puniya AK, Callaghan TM, Edwards JE, Youssef N, Dagar SS, Fliegerova K, Griffith GW, Forster R, Tsang A, McAllister T, Elshahed MS. Anaerobic fungi (phylum Neocallimastigomycota): advances in understanding their taxonomy, life cycle, ecology, role and biotechnological potential. FEMS Microbiology Ecology, 2014, 90(1): 1-17. |
| [25] | St-Pierre B, Wright ADG. Diversity of gut methanogens in herbivorous animals. Animal, 2013, 7(S1): 49-56. |
| [26] | Mi L, Yang B, Hu XL, Luo Y, Liu JX, Yu ZT, Wang JK. Comparative analysis of the microbiota between sheep rumen and rabbit cecum provides new insight into their differential methane production. Frontiers in Microbiology, 2018, 9: 575. DOI:10.3389/fmicb.2018.00575 |
| [27] | Mountfort DO, Asher RA, Bauchop T. Fermentation of cellulose to methane and carbon dioxide by a rumen anaerobic fungus in a triculture with Methanobrevibacter sp. strain RA1 and Methanosarcina barkeri. Applied and Environmental Microbiology, 1982, 44(1): 128-134. |
| [28] | Nakashimada Y, Srinivasan K, Murakami M, Nishio N. Direct conversion of cellulose to methane by anaerobic fungus Neocallimastix frontalis and defined methanogens. Biotechnology Letters, 2000, 22(3): 223-227. DOI:10.1023/A%3A1005666428494 |
| [29] | Lowe SE, Theodorou MK, Trinci AP. Growth and fermentation of an anaerobic rumen fungus on various carbon sources and effect of temperature on development. Applied and Environmental Microbiology, 1987, 53(6): 1210-1215. DOI:10.1128/AEM.53.6.1210-1215.1987 |
| [30] | Teunissen MJ, De Kort GVM, Op Den Camp HJM, Huis In T Veld JHJ. Production of cellulolytic and xylanolytic enzymes during growth of the anaerobic fungus Piromyces sp. on different substrates. Journal of General Microbiology, 1992, 138(8): 1657-1664. DOI:10.1099/00221287-138-8-1657 |
| [31] | Li YF, Sun MZ, Cheng YF, Zhu WY. Effects of associated methanogen on organic acid profile of metabolism by anaerobic fungus revealed using high performance liquid chromatography. Chinese Journal of Animal Nutrition, 2017, 29(4): 1198-1204. (in Chinese) 李袁飞, 孙美洲, 成艳芬, 朱伟云. 高效液相色谱法研究瘤胃甲烷菌共存对厌氧真菌代谢产生有机酸特性的影响. 动物营养学报, 2017, 29(4): 1198-1204. |
| [32] | Liu YC, Whitman WB. Metabolic, phylogenetic, and ecological diversity of the methanogenic archaea. Annals of the New York Academy of Sciences, 2008, 1125(1): 171-189. DOI:10.1196/annals.1419.019 |
| [33] | Li YF, Jin W, Cheng YF, Zhu WY. Effect of the associated methanogen Methanobrevibacter thaueri on the dynamic profile of end and intermediate metabolites of anaerobic fungus Piromyces sp. F1. Current Microbiology, 2016, 73(3): 434-441. DOI:10.1007/s00284-016-1078-9 |
| [34] | Li YF, Jin W, Mu CL, Cheng YF, Zhu WY. Indigenously associated methanogens intensified the metabolism in hydrogenosomes of anaerobic fungi with xylose as substrate. Journal of Basic Microbiology, 2017, 57(11): 933-940. DOI:10.1002/jobm.201700132 |
| [35] | Cheng YF, Jin W, Mao SY, Zhu WY. Production of citrate by anaerobic fungi in the presence of co-culture methanogens as revealed by 1H NMR spectrometry. Asian-Australasian Journal of Animal Sciences, 2013, 26(10): 1416-1423. |
| [36] | Li YQ, Sun MZ, Li YF, Cheng YF, Zhu WY. Co-cultured methanogen improved the metabolism in the hydrogenosome of anaerobic fungus as revealed by gas chromatography-mass spectrometry analysis. Asian-Australasian Journal of Animal Sciences, 2020, 33(12): 1948-1956. DOI:10.5713/ajas.19.0649 |
| [37] | Marvin-Sikkema FD, Gomes TMP, Grivet JP, Gottschal JC, Prins RA. Characterization of hydrogenosomes and their role in glucose metabolism of Neocallimastix sp. L2. Archives of Microbiology, 1993, 160(5): 388-396. |
| [38] | Boxma B, Voncken F, Jannink S, van Alen T, Akhmanova A, van Weelden SWH, van Hellemond JJ, Ricard G, Huynen M, Tielens AGM, Hackstein JHP. The anaerobic chytridiomycete fungus Piromyces sp. E2 produces ethanol via pyruvate: formate lyase and an alcohol dehydrogenase E. Molecular Microbiology, 2004, 51(5): 1389-1399. |
| [39] | Li YF, Cheng YF, Zhu WY. Enhancing the resistance of anaerobic fungus Piromyces sp. F1 to nitrovin by co-culture with Methanobrevibacter thaueri F1. Microbiology China, 2018, 45(1): 111-119. (in Chinese) 李袁飞, 成艳芬, 朱伟云. 共存甲烷短杆菌Methanobrevibacter thaueri F1提高梨囊鞭菌Piromyces sp. F1对硝呋烯腙的耐受性. 微生物学通报, 2018, 45(1): 111-119. |
| [40] | Bernalier A, Fonty G, Gouet P. Cellulose degradation by two rumen anaerobic fungi in monoculture or in coculture with rumen bacteria. Animal Feed Science and Technology, 1991, 32(1/3): 131-136. |
| [41] | Akin DE, Lyon CE, Windham WR, Rigsby LL. Physical degradation of lignified stem tissues by ruminal fungi. Applied and Environmental Microbiology, 1989, 55(3): 611-616. |
| [42] | Joblin KN, Campbell GP, Richardson AJ, Stewart CS. Fermentation of barley straw by anaerobic rumen bacteria and fungi in axenic culture and in co-culture with methanogens. Letters in Applied Microbiology, 1989, 9(5): 195-197. |
| [43] | Joblin KN, Naylor GE, Williams AG. Effect of Methanobrevibacter smithii on xylanolytic activity of anaerobic ruminal fungi. Applied and Environmental Microbiology, 1990, 56(8): 2287-2295. |
| [44] | Wei YQ, Long RJ, Yang H, Yang HJ, Shen XH, Shi RF, Wang ZY, Du JG, Qi XJ, Ye QH. Fiber degradation potential of natural co-cultures of Neocallimastix frontalis and Methanobrevibacter ruminantium isolated from yaks (Bos grunniens) grazing on the Qinghai Tibetan Plateau. Anaerobe, 2016, 39: 158-164. |
| [45] | Teunissen MJ, Kets EPW, Op den Camp HJM, Huis in't Veld JHJ, Vogels GD. Effect of coculture of anaerobic fungi isolated from ruminants and non-ruminants with methanogenic bacteria on cellulolytic and xylanolytic enzyme activities. Archives of Microbiology, 1992, 157(2): 176-182. DOI:10.1007/BF00245287 |
| [46] | Swift CL, Brown JL, Sepp?l? S, O'Malley MA. Co-cultivation of the anaerobic fungus Anaeromyces robustus with Methanobacterium bryantii enhances transcription of carbohydrate active enzymes. Journal of Industrial Microbiology and Biotechnology, 2019, 46(9/10): 1427-1433. DOI:10.1007/s10295-019-02188-0 |
| [47] | Joblin KN, Williams AG. Effect of cocultivation of ruminal chytrid fungi with Methanobrevibacter smithii on lucerne stem degradation and extracellular fungal enzyme activities. Letter in Applied Microbiology, 1991, 12(4): 121-124. |
| [48] | Henske JK, Gilmore SP, Haitjema CH, Solomon KV, O'Malley MA. Biomass-degrading enzymes are catabolite repressed in anaerobic gut fungi. AIChE Journal, 2018, 64(12): 4263-4270. |
| [49] | Procházka J, Mrázek J, ?trosová L, Fliegerová K, Zábranská J, Dohányos M. Enhanced biogas yield from energy crops with rumen anaerobic fungi. Engineering in Life Sciences, 2012, 12(3): 343-351. |
| [50] | Lillington SP, Leggieri PA, Heom KA, O'Malley MA. Nature's recyclers: anaerobic microbial communities drive crude biomass deconstruction. Current Opinion in Biotechnology, 2020, 62: 38-47. |
| [51] | Wilken E, Sepp?l? S, Lankiewicz TS, Saxena M, Henske JK, Salamov AA, Grigoriev IV, O'Malley MA. Genomic and proteomic biases inform metabolic engineering strategies for anaerobic fungi. Metabolic Engineering Communications, 2020, 10: e00107. |
