王小敏1,2

 , 曹东阳2, 徐祥兰2, 何孔旺2
, 曹东阳2, 徐祥兰2, 何孔旺2

1. 遵义医科大学微生物学教研室, 贵州 遵义 563000;
2. 江苏省农业科学院兽医研究所, 江苏 南京 210014
收稿日期:2019-06-26;修回日期:2019-10-31;网络出版日期:2020-03-09
基金项目:国家自然科学基金(31860706);遵义医科大学博士科研启动资金(F-872)
*通信作者:王小敏, Tel:+86-851-28642718, E-mail:WXM_ZMU@163.com;
何孔旺, E-mail:kwh2003@yeah.net.
摘要:[目的] 猪圆环病毒2型(PCV2)是严重危害世界养猪业的重要传染病,即猪圆环病毒相关疾病(PCVAD)的重要病原,其致病机制及流行规律尚未阐明。本研究对2010-2015年间采集自中国华东地区的病料进行PCV2检测,以探讨中国华东地区PCV2的分子流行病学特征,分析PCV2的遗传演化规律。[方法] 本研究利用PCR方法对384份断奶仔猪多系统衰竭综合征疑似发病猪样本进行检测,并对随机选取的42份阳性样品进行PCV2全基因组的测定和分析。[结果] 华东地区普遍存在PCV2的感染,阳性率为41.15%。序列扩增获得42株PCV2全长基因组,同源性和系统进化树分析表明,基于PCV2 ORF2和全长核苷酸序列构建的系统进化树结构是基本一致的。从病料中测得的42株PCV2与11株参考毒株序列的ORF2基因的核苷酸和氨基酸序列同源性分别为87.0%-100.0%和82.5%-100.0%。遗传进化分析显示,53株PCV2毒株可分为4个基因型,即PCV2a、PCV2b、PCV2c和PCV2d。本文获得的42株序列ORF2基因的核苷酸和氨基酸序列同源性分别为89.6%-100.0%和86.8%-100%,可分为3个基因型,即PCV2a、PCV2b和PCV2d,其中69.05%(29/42)属于PCV2d基因型,为优势基因型;PCV2b未在安徽和浙江出现。Cap蛋白的氨基酸序列分析表明,42株PCV2毒株总体多样性较低,存在4个主要的高变区,且不同基因型具有代表性突变位点。本研究获得的42株Cap序列在抗体识别的关键位点存在一定程度变异,这是否导致PCV2抗原结构、致病性以及疫苗效果的改变,还有待深入分析。[结论] 2010-2015年间,PCV2在华东地区猪群中存在较高的感染率,遗传演化相对稳定,基因型无明显地域差异,且PCV2d为优势基因型。42株PCV2 Cap序列有明显差异,但同一基因型具有代表性突变位点。本研究为探讨华东地区PCV2的遗传进化和致病机理提供了参考依据。
关键词:猪圆环病毒2型ORF2基因分子流行病学遗传变异
Molecular epidemiological analysis of porcine circovirus type 2 in East China from 2010 to 2015
Xiaomin Wang1,2

 , Dongyang Cao2, Xianglan Xu2, Kongwang He2
, Dongyang Cao2, Xianglan Xu2, Kongwang He2

1. Department of Microbiology, Zunyi Medical University, Zunyi 563000, Guizhou Province, China;
2. Institute of Veterinary Medicine, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, Jiangsu Province, China
Received: 26 June 2019; Revised: 31 October 2019; Published online: 9 March 2020
*Corresponding author: Xiaomin Wang, Tel:+86-851-28642718, E-mail:WXM_ZMU@163.com;
Kongwang He, E-mail:kwh2003@yeah.net.
Foundation item: Supported by the National Natural Science Foundation of China (31860706) and by the Doctoral Research Initiation Fund Project of Zunyi Medical University (F-872)
Abstract: [Objective] Porcine circovirus type 2 (PCV2) is one of the major etiological agents of porcine circovirus-associated diseases (PCVAD) and could cause severe economic loss to the swine industry worldwide. However, the mechanism of pathogenesis, epidemiology and molecular genetic evolution are not fully understood. Samples obtained from East China between 2010 and 2015 were detected for PCV2. In addition, 42 of 158 PCV2 positive samples were used for the whole genome amplification and sequencing to explore the genetic relationship, molecule epidemiology and genetic evolution of PCV2. [Methods] All of 384 field samples from the clinical diseased pigs between 2010 and 2015 in Jiangsu, Anhui and Zhejiang provinces of China, were screened by specific PCR to detect PCV2. The whole genome of PCV2 positive samples selected randomly was sequenced and analyzed. [Results] PCV2 is distributed widely among swine populations in East China and 158 clinical samples were positive for PCV2, indicating that the prevalence of PCV2 infection in pigs was about 41.15%. The phylogenetic tree constructed by the neighbor-joining method based on the whole genome or ORF2 gene was highly similar. The nucleotide homology and amino acid homology between the ORF2 gene of 42 isolates and 11 reference strains was between 87% to 100% and 82.5% to 100%, respectively. Phylogenetic analysis of the ORF2 genome showed that 53 strains were presented in 4 sub-genotypes (PCV2a, PCV2b, PCV2c and PCV2d). Based on comparing the nucleotides and amino acid sequences of ORF2, 42 isolates obtained in this study shared 89.6% to 100% and 86.8% to 100% identity, respectively. Genetic evolution analysis indicated that 42 isolates could be divided into three genotypes as PCV2a, PCV2b and PCV2d (69.05%), and PCV2b did not appear in Anhui and Zhejiang. Among the three genotypes, PCV2d was the predominant genotype circulating in the swine population of three provinces. Comparisons of the amino acid sequence of Cap protein revealed that 42 isolates in this study have a low variation, four major hypervariable genome regions and special substitutions among the different PCV2 genotypes. There were some aa changes in the reported antibody epitope regions of the Cap protein. However, whether those mutations could result in antigenic variations or change the pathogenicity of PCV2 and the vaccine effect should be explored further. [Conclusion] The results demonstrated that genetic evolution of PCV2 was relatively stable and PCV2b genotype was dominant in the porcine population in East China from 2010 to 2015. According to amino acid alignment data of Cap protein, different substitutions were observed in different genotypes and some representative substitutions exist in each genotype. This study provides a reference for investigating the genetic relationship, genetic evolution and pathogenesis of PCV2 in East China.
Keywords: porcine circovirus type 2ORF2 genemolecular epidemiologygenetic variation
猪圆环病毒2型(porcine circovirus type 2,PCV2)是一种流行于全球的猪圆环病毒相关疾病(PCV2-associated diseases,PCVAD)的主要致病病原,是世界养猪业正面临的日益严重的问题之一,造成了巨大的经济损失[1]。PCV2是已知感染哺乳动物最小的病毒之一,是无囊膜的单股环状负链DNA病毒,呈二十面体结构对称,长1766、1767或1768 bp。PCV2包含5个主要的开放阅读框(open reading frames,ORFs)。ORF1(Rep基因)相对保守,编码病毒复制相关蛋白(Rep和Rep’)[2];ORF2 (Cap基因)突变率较高,是与病毒致病力相关的唯一结构蛋白,即衣壳蛋白(capsid protein,Cap),参与宿主的免疫应答,是病毒主要的免疫原性蛋白[1, 3];ORF3编码病毒毒力相关蛋白,诱导PCV2感染细胞的凋亡[4];ORF4对于病毒的复制是非必需的,能够抑制caspase (cysteine- dependent aspartate-specific proteases,天冬氨酸特异性半胱氨酸蛋白酶)活性和调节感染过程中CD4+和CD8+ T淋巴细胞的数量[5];ORF5能够影响宿主细胞的转录表达、抑制I型干扰素的表达,增强PCV2的复制[6]。
研究证实,PCV2整个基因组具有较高的突变率,与许多RNA病毒突变率相似,为1.2×10–3– 6.6×10–3突变/基因组/年[7]。PCV2基因组的高突变率和重组率促进了PCV2的进化,这也是PCV2变异毒株不断出现和跨种传播的原因之一[8]。研究证实,PCV2通过点突变和基因组重组持续进化,导致新的抗原变异株不断出现[6, 9-11]。根据新的基于PCV2全基因组和Cap基因的系统进化树分型方法,PCV2可分为8个基因型,即PCV2a、PCV2b、PCV2c、PCV2d、PCV2e、PCV2f、PCV2g和PCV2h[12]。目前,PCV2a、PCV2b和PCV2d基因型流行于全球,且PCV2a有5个亚群(2A–2E),PCV2b有3个亚群(1A–1C),PCV2d有2个亚群(2d-1和2d-2)[10, 13]。PCV2c只在丹麦和巴西少数国家发现,另外也有PCV2e (PCV2a-like)和PCV2f的报道[10, 13-15]。在所有的PCV2基因型中,PCV2a在2000年前是优势基因型,随后,由于基因漂移,PCV2b基因型出现,且在过去的十年成为新的优势基因型[8-9]。研究表明,PCV2b亚型正在向PCV2d漂移[10, 16]。2010年,在中国和其他国家的PCVAD病猪中鉴定出PCV2变异株mPCV2b,该变异株目前属于PCV2d基因型[10, 17]。
尽管越来越多的猪场使用PCV2灭活疫苗或亚单位疫苗,但是PCV2感染率在中国仍然呈现上升趋势。华东地区是中国主要的养猪地域,为深入了解我国华东地区2010–2015年间猪群中PCV2的基因多样性、遗传变异和进化规律,本文对2010–2015年间采集自我国华东地区的384份断奶仔猪多系统衰竭综合征(postweaning multisystemic wasting syndrome,PMWS)疑似发病猪的临床样本进行PCV2的特异性检测;随机选取不同年份不同地区的42份PCV2阳性样本进行PCV2全基因组的扩增和测序,并与GenBank中的PCV2参考毒株序列进行遗传变异分析,以期掌握华东地区PCV2的流行病学和遗传变异,为PCVAD的防控提供理论依据。
1 材料和方法 1.1 材料 AxyPrep质粒DNA小量提取试剂盒和Agarose Gel DNA Purification试剂盒均购自Axygen公司;血液DNAout试剂盒、柱式病毒DNAout试剂盒、柱式动物DNAout试剂盒、氨苄青霉素溶液等购自北京天恩泽基因科技有限公司;2×PCR MasterMix、Ex Taq DNA聚合酶、DNA Marker DL-2000、克隆载体pMD19-T等均购自TaKaRa公司;琼脂糖购自Sigma公司;DH5α感受态细胞购自北京天根生化有限公司;引物均由上海Invitrogen公司合成。
1.2 病毒 PCV2分离株,即201105ZJ株[18]由江苏省农业科学院农业部兽用生物制品工程技术重点实验室分离、保存和馈赠。
1.3 样品采集和处理 于2010–2015年采集自我国华东地区江苏、浙江和安徽多个猪场的384份PMWS疑似发病猪的临床样品,包括血液、肺脏、腹股沟淋巴结、脾和肝脏,按照采集地和采集时间先后进行编号,–70 ℃保存备用。
将采集的组织病料和无菌PBS按照1:5比例充分研磨,制成匀浆后反复冻融3次,4 ℃ 8000 r/min离心8 min,取上清,–70 ℃保存备用。血液样品4 ℃、4000 r/min离心8 min,取血清,–70 ℃保存备用。
1.4 DNA模板的制备和PCV2的检测
1.4.1 DNA模板的制备: 按照相应的DNAout试剂盒说明书对组织、血清和细胞样本(201105ZJ株)进行病毒总DNA的提取,于–70 ℃保存备用。
1.4.2 引物设计: 参照Wang等[18]建立的方法,合成PCV2检测引物和全长基因组的扩增引物(表 1),由上海Invitrogen有限公司合成,纯度级别为PAGE。
表 1. PCV2特异性PCR引物序列 Table 1. Specific PCR primer sequences for PCV2
| Primer | Primer sequences (5'→3') | AnnealingTm /℃ | Length/bp |
| SFPCV2-1 | GGACCCCAACCACATAAAA | 56 | 1254 |
| SRPCV2-1 | CCCTAACCTATGACCCCTATGT | ||
| SFPCV2-2 | TGTTTTCGAACGCAGTGCC | 56 | 1045 |
| SRPCV2-2 | CCGTTGTCCTTGAGATCTAGGA | ||
| SFPCV2 | CGGTACCAGCTATGACGTATCCAAG | 52 | 488 |
| SRPCV2 | GCCAAGCTTTCACTTCGTAATGG | ||
| F: forward PCR primer; R: reverse PCR primer. | |||
表选项
1.4.3 特异性PCV2 PCR检测: 以上述提取的DNA为模板,以SFPCV2和SRPCV2为引物,对384份临床样品和阳性对照(PCV2分离株201105ZJ)进行PCV2的检测。分别在反应管中加入2×Taq PCR MasterMix 12.5 μL,上、下游引物各0.5 μL,DNA模板2.0 μL,最后加入ddH2O至终体系为25.0 μL。PCR反应参数:95 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 50 s,共40个循环;72 ℃ 10 min;4 ℃保存。PCR扩增产物经1.2%琼脂糖凝胶电泳,观察鉴定特异性条带,统计分析PCV2的检测结果。
1.5 PCV2全基因组的扩增、测序和分析
1.5.1 PCV2全基因组的扩增: 以PCV2检测阳性样本DNA为模板,进行PCV2全长基因组片段的PCR扩增。分别在反应管中加入Ex Taq DNA聚合酶0.25 μL、上游引物(SFPCV2-1/SFPCV2-2)、下游引物(SRPCV2-1/SRPCV2-2)各1.0 μL,10×Ex Taq Buffer (Mg2+ plus) 5.0 μL,dNTP Mixture 4.0 μL,DNA模板4.0 μL,最后加入ddH2O至终体系为50.0 μL。反应参数为:95 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 80 s,共38个循环;72 ℃ 10 min;4 ℃保存。
1.5.2 目的片段的回收、克隆和测序: PCR产物经1.0%琼脂糖凝胶电泳检测后,使用胶回收试剂盒回收目的片段,将纯化的PCR产物克隆到pMD19-T载体,转化DH5α感受态细胞,涂布于含有IPTG和X-gal的LB平板,挑取单个白色菌落接种于含有100 mg/L氨苄青霉素的LB液体培养基,置于37 ℃温箱中,220 r/min振荡培养12 h左右。按照AxyPrep质粒DNA小量提取试剂盒说明书提取质粒,经PCR鉴定后,将鉴定正确的质粒送测序。
1.5.3 数据分析: 运用DNAStar软件中MegAlign对本研究获得的42株PCV2全长序列与GenBank中收录的11株PCV2参考毒株序列进行同源性比对分析,构建系统进化树,分析本文获得序列与参考毒株间的遗传进化关系。参考毒株的GenBank登录号/名称/基因型为:AB426905 Yamagata PCV2a、EU136711 PCV2 herd1 PCV2a、AF055392 PCV2a、HQ591371 168-08-6 PCV2b、AY322002 Fh22 PCV2b、AF055394 PCV2b、EU148503 DK1980PMWSfree PCV2c、EU148505 DK1990PMWSfree PCV2c、AY181947 SD PCV2d、JX982220 KSSF1103 PCV2d和HM038030 AH PCV2d。
2 结果和分析 2.1 临床样品的检测和数据统计 对采集的384份PMWS疑似发病猪的临床样品进行PCV2的特异性PCR扩增,同时设立加入201105ZJ株DNA的阳性对照和未加DNA的空白对照。结果表明,扩增出与预期大小一致的488 bp的特异性条带。
由表 2可知,在384份临床病料中,158份为PCV2 DNA阳性,平均阳性率为41.15% (158/384)。3个省份的样品都能检测到PCV2阳性,其中浙江省的PCV2阳性率最低(38.54%),安徽省的阳性率最高(44.86%),这也表明华东地区猪群PCV2感染较为普遍,且感染率较高。
表 2. 江苏、安徽和浙江三省样品PCV2检测结果 Table 2. Detection results of PCV2 in Jiangsu, Anhui and Zhejiang Provinces
| Origin of samples | Number of samples | Positive number of PCV2 | Positive rate/% |
| Jiangsu | 181 | 73 | 40.33 |
| Zhejiang | 96 | 37 | 38.54 |
| Anhui | 107 | 48 | 44.86 |
| Total | 384 | 158 | 41.15 |
表选项
2.2 全基因组的扩增和序列测定 对来自江苏、浙江和安徽3省的42份PCV2阳性样品,进行PCV2全基因组的扩增、克隆和测序。经测序获得42株PCV2全长基因组序列,并根据样品采集时间和地点命名。
2.3 基于全长的同源性和遗传进化分析 为了解我国华东部分地区PCV2的基因多样性、遗传变异和进化规律,本文将测得的42株全基因组序列与GenBank中的11株参考毒株序列进行同源性和遗传进化分析。结果表明,53株PCV2序列的核苷酸同源性为93.6%–100.0%,201205AH株与EU148505 DK1990PMWSfree PCV2c株亲缘关系较远,只有93.6%。53株PCV2中有2株为PCV2c基因型(3.77%),10株为PCV2b基因型(18.87%),9株为PCV2a基因型(16.98%),32株为PCV2d基因型(60.38%) (图 1)。本文测得的42株华东地区PCV2流行株间核苷酸序列同源性为94.0%至100.0%,隶属于PCV2a亚型的20150423YR株与隶属于PCV2d亚型的201112ZJ的差异最大,序列同源性仅有94%。本研究获得的42株PCV2序列,6株为PCV2a基因型(14.29%),7株为PCV2b基因型(16.67%),29株为PCV2d基因型(69.05%),未发现PCV2c基因型。这些结果表明各测序毒株与参考毒株间的同源性均较高,PCV2d为华东地区的优势基因型。尽管本研究未在浙江和安徽发现PCV2b的存在,但由于测得序列数量有限,尚不能判定基因型与区域性分布之间的规律。
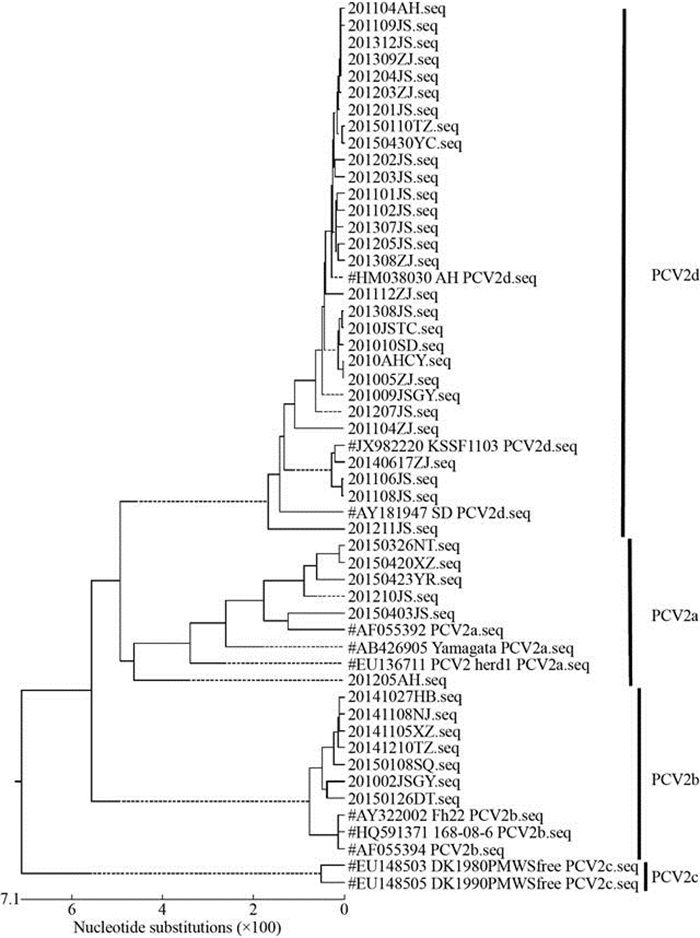 |
| 图 1 基于PCV2全长基因组序列的遗传进化树分析 Figure 1 Phylogenetic tree based on a comparison of 53 complete PCV2 genomic sequences, including the 42 sequences from this study and 11 reference sequences. The phylogenetic tree based on the nucleotide sequences of the whole genome was constructed by the neighbor-joining method using DNA Star 6.0 software. The distance estimated with the ruler represented the number of nucleotide substitutions. Genotypes were indicated with straight lines. The reference sequences were indicated with a pound sign (#). |
| 图选项 |
2.4 基于ORF2的同源性和系统进化树分析 ORF2突变率较高,是PCV2的主要免疫原基因,是研究PCV2遗传变异和分型最重要的基因,在分子流行病学调查中被广泛应用。本文将测得的42株PCV2 ORF2序列与11株属于不同亚型参考毒株的ORF2序列进行同源比对和遗传进化分析。结果表明,53株PCV2 ORF2基因全长702 bp或705 bp,核苷酸同源性为87.0%–100.0%;20150423YR株与EU148505 DK1990PMWSfree PCV2c,同源性只有87.0%,属于不同的进化分支,亲缘关系较远;本研究获得的42株PCV2 ORF2序列,核苷酸同源性为89.6%–100.0%,属于不同分支的20150423YR (PCV2a)与201211JS (PCV2d)差异最大,同源性仅为89.6%。
如图 1和图 2,基于PCV2全长基因组和基于ORF2核苷酸序列所绘制的遗传进化树相似度非常高,结构基本一致。本文测得的42株PCV2分为3个基因型,即PCV2a (6/42,14.29%)、PCV2b (7/42,16.67%)和PCV2d (29/42,69.05%),其中PCV2d在江苏、安徽和浙江广泛存在,为2010–2015年间华东地区的主要流行基因型;获得的42株PCV2与EU148505 DK1990PMWSfree PCV2c和EU148505 DK1990PMWSfree PCV2c皆不属于同一分支,亲缘关系较远。
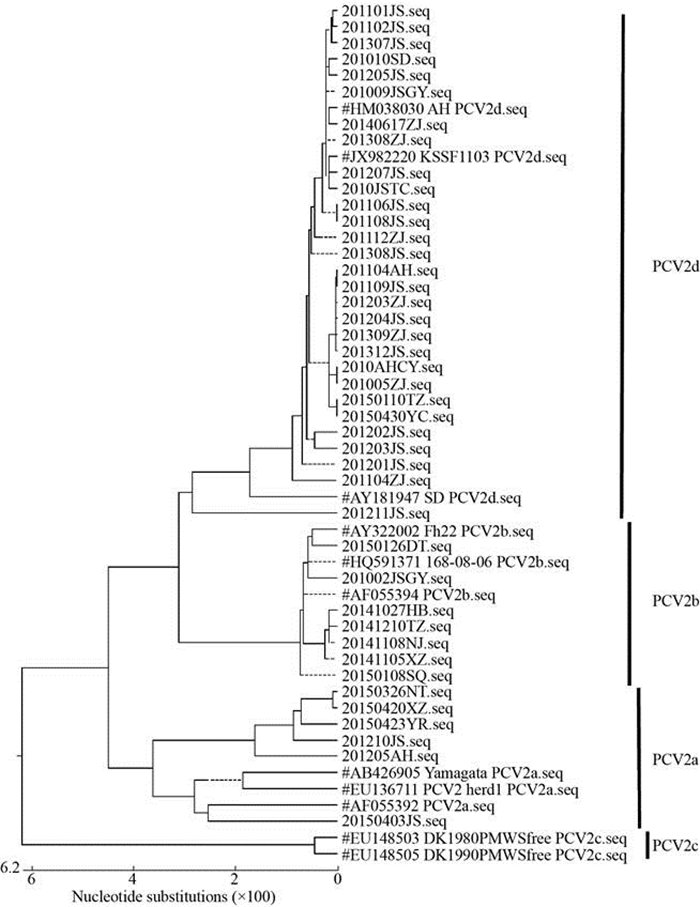 |
| 图 2 基于PCV2 ORF2基因的遗传进化树分析 Figure 2 Phylogenetic tree based on a comparison of 53 ORF2 gene, including the 42 sequences from this study and 11 reference sequences. The phylogenetic tree based on the nucleotide sequences of the nucleotides of ORF2 was constructed by the neighbor-joining method using DNA Star 6.0 software. The distance estimated with the ruler represented the number of nucleotide substitutions. Genotypes were indicated with straight lines. The reference sequences were indicated with a pound sign (#). |
| 图选项 |
2.5 Cap蛋白的氨基酸序列分析 同源性分析表明,本文测得的序列与11株国内外参考毒株的氨基酸序列同源性为82.5%–100.0%,其中201210JS与EU148505 DK1990PMWSfree PCV2c的同源性最低。本研究测得的42株Cap蛋白由233或234氨基酸组成,同源性为86.8%–100%,其中属于PCV2a分支的20150423YR与属于PCV2d的201203JS、20140617ZJ和201101JS差异最大、同源性最低。由图 3可以看出,PCV2存在4个高变区,各毒株在特异性抗原表位都存在突变,但是各毒株在同一基因型内存在一些代表性突变位点。与AB426905 Yamagata PCV2a相比,PCV2a基因型的特异性位点为T47→S/A47、R63→T/S63、M72→L72和N77→D77,PCV2b基因型的特异性位点为V57→I57、I89→R89和D210→E210,PCV2d基因型的特异性位点为F53→I53、A68→N68、T134→N/K134和S169→R/G169,PCV2c基因型因序列太少,缺乏统计意义。本文中所有的PCV2a毒株含有经典基序TNKISI (86–91),PCV2b含有经典基序SNPRSV (86–91),PCV2c和部分PCV2d (2/32)含有经典基序SNPLTV (86–91),大部分PCV2d (30/32)含有经典基序SNPLTV (86–91)和TGID (190、191、206和210)。Cap蛋白唯一的糖基化位点NYS (143–145)稳定,未发现变异。
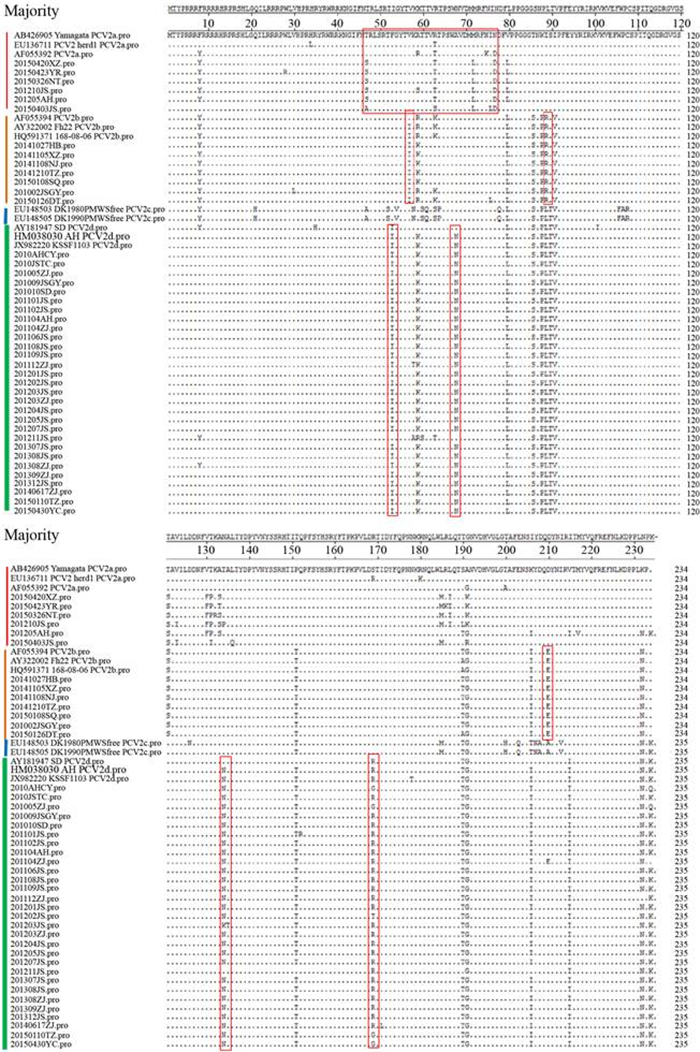 |
| 图 3 ORF2推导氨基酸多序列比对分析 Figure 3 A multiple alignment of amino acid sequences of ORF2. Sequence alignment was performed using the DNA Star 6.0 software. The regions marked with frame were representative mutation sites. |
| 图选项 |
3 讨论 PCVAD是近年危害世界养猪业的重要传染病之一,PCV2是PCVAD的主要病原。PCV2主要侵害哺乳仔猪和育肥猪,感染会导致机体内淋巴细胞的凋亡和耗竭、干扰素生成功能受损,降低机体免疫反应,引起严重的免疫抑制和多系统病变,也为其他病原体的入侵提供了有利条件。华东地区是我国养殖业发展的重要区域,因而研究华东地区PCV2的基因型分布、分子生物学特性和流行规律具有非常重要的意义。本文对我国华东地区2010至2015年PMWS疑似发病猪的PCV2流行情况进行了调查,并对其进行了分子生物学的遗传变异分析。结果显示,我国华东地区PMWS疑似发病猪样本的PCV2平均阳性率为41.15%,不同地区PCV2阳性率略有差异,安徽省的PCV2阳性率高出平均阳性率,浙江省的略低。这也表明,我国华东地区猪群中存在较为广泛的PCV2感染,危害严重,应引起重视。
PCV2的全球遗传进化分析表明,其演化轨迹为PCV2a至PCV2b,再从PCV2b至PCV2d[10]。Wang等[19]基于PCV2的ORF2和全基因组,对中国1999–2017年的74株PCV2进行基因多样性分析,发现我国除了PCV2a、PCV2b和PCV2d基因型外,还存在两种不稳定的重组基因型,即IM1和IM2基因型。王苗苗等[20]对华东地区PCV2进行遗传变异分析,结果表明,华东地区普遍存在PCV2感染,感染率达43.58%,PCV2b基因型为主,PCV2a基因型次之。在中国,除PCV2c基因型,其他基因型均共存于猪场,这也加速了PCV2的基因重组。本文测序获得42株PCV2全基因组序列,12株PCV2基因组全长为1767 bp,其余30株全长均为1768 bp。同源性分析显示,53株PCV2序列的核苷酸同源性为93.6%–100.0%,201205AH株与EU148505 DK1990PMWSfree PCV2c株同源性最低,只有93.6%,这也表明本文所得序列与国内外参考毒株之间存在较高的同源性。基于PCV2全长基因组和基于ORF2核苷酸序列所绘制的遗传进化树相似度非常高,结构基本一致。本文测得毒株与11株参考毒株可分为PCV2a (9/53)、PCV2b (10/53)、PCV2c (2/53)和PCV2d (32/53) 4个基因型,然而,201211JS在PCV2d基因型中是一个特殊的基因族。本研究获得的42株PCV2中,6株属于PCV2a、7株属于PCV2b、29株属于PCV2d,其中69.05% (29/42)属于PCV2d基因型,这也表明华东地区的优势基因型为PCV2d,该结果与近年国内大部分报道一致。尽管未发现PCV2c基因型,且未在浙江和安徽发现PCV2b的存在,但由于测得序列数量有限,尚不能判定基因型与区域性分布之间的规律。
尽管PCV2是DNA病毒,但是其基因组的突变率与单链RNA病毒的突变率相近[19-21]。ORF2编码病毒刺激机体产生中和抗体的主要结构蛋白,即Cap蛋白。ORF2变异程度远高于其他阅读框,是PCV2多个ORF中变异频率最高的一个,其较高的核苷酸替代率使得PCV2不断进化和新毒株的出现成为可能。研究表明,Cap蛋白重要的基序中存在区分PCV2基因型的关键氨基酸;Cap蛋白表面存在的多个潜在表位在免疫压力下易发生突变,从而改变PCV2的蛋白抗原性和致病力;Cap蛋白变异可能会影响PCV2与受体的结合、宿主免疫反应和病毒复制与组装,从而影响PCV2的整个生命周期。因此,Cap蛋白的少量突变就可能导致新的抗原变异或致病力增强毒株的出现[3, 10]。本研究中Cap蛋白序列分析表明,各毒株之间氨基酸序列的同源性为82.5%–100.0%;本研究获得的42株毒株之间氨基酸的同源性为86.8%–100%,与基因型PCV2c的亲缘关系较远,这也说明PCV2毒株总体多样性较低。氨基酸序列分析表明,不同亚型差异显著,其中aa 53–91、aa 121–134、aa 169–215和aa 232–234是Cap蛋白的4个高变区;同一基因型内也存在一些代表性突变位点,即PCV2a的4个特异性突变位点(47S、63T、72L和77D)、PCV2b的3个特异性突变位点(57I、89R和210E)和PCV2d的4个特异性突变位点(53I、68N、134N/K和169R/G),且大部分PCV2d在C端出现了一个赖氨酸的延伸。赖氨酸是一种限制性氨基酸,与机体免疫系统活性存在一定的相互关系,因此推测该赖氨酸的延伸可能会影响Cap蛋白的功能或病毒致病性。另外,仅有PCV2d在免疫显性诱饵表位(aa 169–180)存在突变169R/G,这可能是PCV2d成为优势基因型的原因之一。目前,接种疫苗是PCV2感染的主要防控手段,而商品化疫苗多基于PCV2a和PCV2b基因型研制而成,其交叉保护率有待进一步深入研究,这些特异性突变位点的发现为今后深入研究PCV2的进化和防控提供了参考依据。
Cap蛋白氨基酸序列分析显示,获得的42株PCV2 Cap蛋白序列在与Cap蛋白核定位有关的核定位信号区(nuclear localization signals,NLS)关键区域[21-22],即aa 12–18和aa 34–41,仅存在极少数的变异,相对比较保守。Cap蛋白唯一的糖基化位点NYS (143–145)稳定,未发现变异。Cap蛋白包含多个线性和构象特异性抗原表位[23],aa 65–87、aa 113–139、aa 132–146、aa 157–183、aa 193–207和aa 230–233,其中针对aa 117–131表位的抗体可以用来区分疫苗毒株和野毒感染[24]。研究发现Asp-70、Met-71、Asn-77、Asp-78、Glu-113、Asp-115、Asp-127、Tyr-173、Phe-174、Gln-175、Lys-179、Glu-203、Ile-206和Tyr-207位的氨基酸是重要的抗体识别位点[25-26]。在这些蛋白表位和抗体识别位点区域,本文测得的毒株都存在一定的突变,尤其是抗体识别关键点Asp-77和Lys/ILe-206的突变。这些突变是否影响PCV2毒力和产生中和抗体的能力,改变其抗原性和致病性,有待进一步的探讨和研究。本研究从分子水平对PCV2进行遗传变异分析,不仅揭示了PCV2的基因多样性,也加深了对我国华东地区PCV2亚型及流行趋势的理解。
References
| [1] | Finsterbusch T, Mankertz A. Porcine circoviruses-small but powerful. Virus Research, 2009, 143(2): 177-183. |
| [2] | Cheung AK. Transcriptional analysis of porcine circovirus type 2. Virology, 2003, 305(1): 168-180. |
| [3] | Nawagitgul P, Morozov I, Bolin SR, Harms PA, Sorden SD, Paul PS. Open reading frame 2 of porcine circovirus type 2 encodes a major capsid protein. Journal of General Virology, 2000, 81(9): 2281-2287. DOI:10.1099/0022-1317-81-9-2281 |
| [4] | Liu J, Zhu Y, Chen L, Lau J, He F, Lau A, Wang ZL, Karuppannan AK, Kwang J. The ORF3 protein of porcine circovirus type 2 interacts with porcine ubiquitin E3 ligase Pirh2 and facilitates p53 expression in viral infection. Journal of Virology, 2007, 81(17): 9560-9567. DOI:10.1128/JVI.00681-07 |
| [5] | He JL, Cao JJ, Zhou N, Jin YL, Wu JS, Zhou JY. Identification and functional analysis of the novel ORF4 protein encoded by porcine circovirus type 2. Journal of Virology, 2013, 87(3): 1420-1429. |
| [6] | Choi CY, Choi YC, Park IB, Lee CH, Kang SJ, Chun T. The ORF5 protein of porcine circovirus type 2 enhances viral replication by dampening type I interferon expression in porcine epithelial cells. Veterinary Microbiology, 2018, 226: 50-58. DOI:10.1016/j.vetmic.2018.10.005 |
| [7] | Pérez LJ, de Arce HD, Cortey M, Domínguez P, Percedo MI, Perera CL, Tarradas J, Frías MT, Segalés J, Ganges L, Nú?ez JI. Phylogenetic networks to study the origin and evolution of porcine circovirus type 2(PCV2) in Cuba. Veterinary Microbiology, 2011, 151(3/4): 245-254. |
| [8] | Zhai SL, He DS, Qi WB, Chen SN, Deng SF, Hu J, Li XP, Li L, Chen RA, Luo ML, Wei WK. Complete genome characterization and phylogenetic analysis of three distinct buffalo-origin PCV2 isolates from China. Infection, Genetics and Evolution, 2014, 28: 278-282. DOI:10.1016/j.meegid.2014.10.005 |
| [9] | Olvera A, Cortey M, Segalés J. Molecular evolution of porcine circovirus type 2 genomes:phylogeny and clonality. Virology, 2007, 357(2): 175-185. |
| [10] | Yao J, Qin YR, Zeng Y, Ouyang K, Chen Y, Huang WJ, Wei ZZ. Genetic analysis of porcine circovirus type 2(PCV2) strains between 2002 and 2016 reveals PCV2 mutant predominating in porcine population in Guangxi, China. BMC Veterinary Research, 2019, 15: 118. |
| [11] | Franzo G, Cortey M, de Castro AMMG, Piovezan U, Szabo MPJ, Drigo M, Segalés J, Richtzenhain LJ. Genetic characterisation of Porcine circovirus type 2(PCV2) strains from feral pigs in the Brazilian Pantanal:an opportunity to reconstruct the history of PCV2 evolution. Veterinary Microbiology, 2015, 178(1/2): 158-162. |
| [12] | Franzo G, Segalés J. Porcine circovirus 2(PCV-2) genotype update and proposal of a new genotyping methodology. PLoS One, 2018, 13(12): e0208585. DOI:10.1371/journal.pone.0208585 |
| [13] | Grau-Roma L, Crisci E, Sibila M, López-Soria S, Nofrarias M, Cortey M, Fraile L, Olvera A, Segales J. A proposal on porcine circovirus type 2(PCV2) genotype definition and their relation with postweaning multisystemic wasting syndrome (PMWS) occurrence. Veterinary Microbiology, 2008, 128(1/2): 23-35. |
| [14] | Cortey M, Olvera A, Grau-Roma L, Segalés J. Further comments on porcine circovirus type 2(PCV2) genotype definition and nomenclature. Veterinary Microbiology, 2011, 149(3/4): 522-523. |
| [15] | Bao F, Mi S, Luo Q, Guo H, Tu C, Zhu G, Gong W. Retrospective study of porcine circovirus type 2 infection reveals a novel genotype PCV2f. Transboundary and Emerging Diseases, 2018, 65(2): 432-440. DOI:10.1111/tbed.12721 |
| [16] | Yang S, Yin S, Shang Y, Liu B, Yuan L, Zafar Khan MU, Liu X, Cai J. Phylogenetic and genetic variation analyses of porcine circovirus type 2 isolated from China. Transboundary and Emerging Diseases, 2018, 65(2): e383-e392. DOI:10.1111/tbed.12768 |
| [17] | Jiang CG, Wang G, Tu YB, Liu YG, Wang SJ, Cai XH, An TQ. Genetic analysis of porcine circovirus type 2 in China. Archives of Virology, 2017, 162(9): 2715-2726. DOI:10.1007/s00705-017-3414-1 |
| [18] | Wang XM, He KW, Wang W, Zhou ZT, Yang GY, Mao AH, Yu ZY, Ni YX. Genetic variation of porcine circovirus type 2 isolate 201105ZJ. Agricultural Science & Technology, 2014, 15(11): 1860-1864, 1887. |
| [19] | Wang HJ, Gu JY, Xing G, Qiu XH, An ST, Wang YX, Zhang C, Liu CM, Gong WJ, Tu CC, Su S, Zhou JY. Genetic diversity of porcine circovirus type 2 in China between 1999-2017. Transboundary and Emerging Diseases, 2019, 66(1): 599-605. DOI:10.1111/tbed.13040 |
| [20] | Wang MM, Wang TT, Zhu JG. Genotyping of porcine circovirus 2 and genetic variation analysis of ORF2 in East China. Animal Husbandry & Veterinary Medicine, 2018, 50(7): 92-97. (in Chinese) 王苗苗, 王婷婷, 朱建国. 华东地区猪圆环病毒2型基因型鉴别及ORF2遗传变异分析. 畜牧与兽医, 2018, 50(7): 92-97. |
| [21] | Liu Q, Tikoo SK, Babiuk LA. Nuclear localization of the ORF2 protein encoded by porcine circovirus type 2. Virology, 2001, 285(1): 91-99. |
| [22] | Liu Q, Willson P, Attoh-Poku S, Babiuk LA. Bacterial expression of an immunologically reactive PCV2 ORF2 fusion protein. Protein Expression and Purification, 2001, 21(1): 115-120. |
| [23] | Mahé D, Blanchard P, Truong C, Arnauld C, Le Cann P, Cariolet R, Madec F, Albina E, Jestin A. Differential recognition of ORF2 protein from type 1 and type 2 porcine circoviruses and identification of immunorelevant epitopes. Journal of General Virology, 2000, 81(7): 1815-1824. DOI:10.1099/0022-1317-81-7-1815 |
| [24] | Truong C, Mahe D, Blanchard P, Le Dimna M, Madec F, Jestin A, Albina E. Identification of an immunorelevant ORF2 epitope from porcine circovirus type 2 as a serological marker for experimental and natural infection. Archives of Virology, 2001, 146(6): 1197-1211. DOI:10.1007/s007050170115 |
| [25] | Trible BR, Kerrigan M, Crossland N, Potter M, Faaberg K, Hesse R, Rowland RR. Antibody recognition of porcine circovirus type 2 capsid protein epitopes after vaccination, infection, and disease. Clinical and Vaccine Immunology, 2011, 18(5): 749-757. DOI:10.1128/CVI.00418-10 |
| [26] | Li FQ, Wang LL, Li XL. Progress on molecular biology of PCV2. Tianjin Agricultural Sciences, 2015, 21(9): 57-60. (in Chinese) 李富强, 王利丽, 李秀丽. 猪圆环病毒2型分子生物学研究进展. 天津农业科学, 2015, 21(9): 57-60. DOI:10.3969/j.issn.1006-6500.2015.09.013 |
