李兴涵1, 费小雯2, 李亚军1, 邓晓东1


1. 中国热带农业科学院热带生物技术研究所, 海南省海洋生物资源功能性成分研究与利用重点实验室, 海南 海口 571101;
2. 海南医学院基础医学与生命科学学院, 海南 海口 571199
收稿日期:2019-05-14;修回日期:2019-07-26;网络出版日期:2019-08-12
基金项目:海南省重点科技计划(ZDYF2018124);农业农村部财政专项(NFZX2018);国家自然科学基金(31870344);中国热带农业科学院基本科研业务费专项资金(19CXTD-32,1630052018001);中国热带农业科学院热带生物技术研究所基本科研业务费专项资金(1630052016009)
*通信作者:邓晓东, Tel:+86-898-66988530;E-mail:xiaodong9deng@hotmail.com.
摘要:[目的] 为研究莱茵衣藻(Chlamydomonas reinhardtii)泛素结合酶(ubiquitin-conjugating enzymes,E2)CrUBC23在莱茵衣藻油脂代谢中的作用,为高产油微藻基因工程改良和揭示藻类油脂合成及代谢调控机理奠定基础。[方法] qRT-PCR分析莱茵衣藻在低氮、低磷胁迫下泛素结合酶CrUBC23表达情况;克隆CrUBC23同源基因干涉片段和全长基因,构建RNAi干涉载体和过量表达载体,转化莱茵衣藻并检测生物量和油脂含量;构建CrUBC23-GFP融合表达载体,用农杆菌浸染洋葱表皮细胞进行亚细胞定位。[结果] 莱茵衣藻在低氮、低磷胁迫下CrUBC23基因表达量显著增加,增加幅度分别为正常培养的4.98-5.80倍和1.85-5.20倍。RNAi干扰结果显示,转基因藻细胞中性脂含量降低5.5%,总脂含量降低3.16%-17.6%。过量表达结果显示,转基因藻细胞中性脂含量增加8.8%,总脂含量增加4.51%-14.03%。[结论] CrUBC23正向调控莱茵衣藻油脂代谢,该基因定位于细胞核。
关键词:莱茵衣藻E2泛素结合酶基因沉默亚细胞定位
Ubiquitin-conjugating enzyme (E2) CrUBC23 regulates lipid accumulation in Chlamydomonas reinhardtii
Xinghan Li1, Xiaowen Fei2, Yajun Li1, Xiaodong Deng1


1. Hainan Provincial Key Laboratory for Functional Components Research and Utilization of Marine Bio-resources, Institute of Tropical Bioscience and Biotechnology, Chinese Academy of Tropical Agricultural Sciences, Haikou 571101, Hainan Province, China;
2. School of Basic Medicine and Life Science, Hainan Medical College, Haikou 571199, Hainan Province, China
Received: 14 May 2019; Revised: 26 July 2019; Published online: 12 August 2019
*Corresponding author: Xiaodong Deng, Tel: +86-898-66988530; E-mail: xiaodong9deng@hotmail.com.
Foundation item: Supported by the Key Science and Technology Program of Hainan Province (ZDYF2018124), by the Ministry of Agriculture and Rural Program-China (NFZX2018), by the National Natural Science Foundation of China (31870344), by the Chinese Academy of Tropical Agricultural Sciences (19CXTD-32, 1630052018001) and by the Institute of Tropical Biotechnology, Chinese Academy of Tropical Agricultural Sciences (1630052016009)
Abstract: [Objective] To study the lipid metabolism mechanism regulated by Ubiquitin-conjugating Enzyme (E2) CrUBC23 in Chlamydomonas reinhardtii. [Methods] The mRNA levels of ubiquitin-conjugating enzyme CrUBC23 were analyzed by qRT-PCR under low nitrogen and low phosphorus conditions in C. reinhardtii. Fragments for RNAi interference and over-expression of CrUBC23 gene were cloned, and then the RNA interference vector and over-expression vector were constructed and transformed into C. reinhardtii. The biomass and lipid content of the transgenic strain was detected. The fusion expression vector CrUBC23-GFP was constructed also, and the subcellular localization of onion epidermal cells was carried out by using Agrobacterium tumefaciens. [Results] The level of RNA in C. reinhardtii were increased significantly under low nitrogen and low phosphorus conditions, with an increase of 4.98-5.80 times and 1.85-5.20 times as much as that in normal culture, respectively. RNA interference results showed that the neutral lipid content and total lipid content of transgenic algae decreased by 5.5% and 3.16%-17.6%. While, over-expression showed that the neutral lipid content increased by 8.8% and the total lipid content increased by 4.51%-14.03%. [Conclusion] CrUBC23 is a positive regulator in lipid metabolism, and may locate in the nucleus.
Keywords: Chlamydomonas reinhardtiiubiquitin-conjugating enzymegene silencingsubcellular location
随着全球能源消耗急剧增加,石油等化石燃料的大量使用造成的环境污染问题严重影响着社会经济的可持续发展。微藻生物柴油因其具备不可比拟的优越被认可为最有可能替代石油的清洁燃料[1],然而由于高油脂含量微藻的筛选、培养、采收、破壁提油、超临界萃取等技术问题,微藻生物柴油至今还处于试验阶段[2]。目前,微藻生物柴油的技术开发重点集中在优势藻种的筛选和改良,微藻高密度培养优化与扩大化培养,微藻的采收和油脂的提取加工,微藻生物质资源的综合利用开发[3]。优势藻种的筛选和改良是微藻生物柴油生产的基础,基因工程改良技术是获得优势藻种的重要手段。现在对微藻油脂的代谢途径还了解不够,因此油脂合成及代谢相关基因的挖掘和筛选对揭示微藻总三酰甘油合成途径有很大的帮助。
研究表明,有些藻类在非生物胁迫下藻细胞油脂积累增加,这些外界条件有低氮、低磷、高盐、高pH、高温、氧化剂、激素、高光强等,其中低氮胁迫油脂积累最为明显[4-8],如胶球藻Coccomyxa sp.及三角褐指藻在缺氮条件下油脂含量从干重的20%升高至40%[7],由此可见环境因子变化对于产油微藻的生长及油脂积累具有重要的影响。本课题组对莱茵衣藻缺氮转录组分析结果显示CrUBC23基因的表达异常活跃,生物信息学分析其属于泛素结合酶(ubiquitin-conjugating enzyme,E2)蛋白家族,并对莱茵衣藻E2泛素结合酶蛋白家族进行全基因组分析,发现莱茵衣藻共有24个E2泛素结合酶[8]。CrUBC23属于泛素结合酶(ubiquitin-conjugating enzyme,E2)蛋白家族,是泛素-蛋白酶体途径(ubiquitin-proteasome pathway,UPP)的重要组成成分。UPP广泛存在于真核细胞中,是精确调控细胞质和细胞核内蛋白质有序降解的途径,并参与细胞分裂[9]、细胞凋亡[10]、细胞内信号转导[11]、肿瘤的发生[12]及MHC Ⅰ类抗原的递呈[13]等多个细胞生理过程,对维持细胞的稳态具有十分重要的作用。植物E2泛素结合酶含有一个高度保守的cysteine催化位点,E2-like具有UBCc结构域而无cysteine,因而不具有活性。植物E2泛素结合酶可以分为4个亚类:classⅠ具有UBC结构域,classⅡ包含一个C端延长的蛋白序列,class Ⅲ包含一个N端延长的蛋白序列,class Ⅳ包含C端和N端延长的蛋白序列[14]。研究表明,E2泛素结合酶参与植物泛素信号转导途径和植物对干旱、重金属、高温等非生物胁迫响应。Nurdiani等[15]发现过量表达水稻E2泛素结合酶OsSCE1水稻干旱胁迫的耐受性降低,而基因敲除后耐受性提高。Gao等[16]发现葡萄中有7个和6个VvUBC分别对冷应激和热应激反应,而在盐胁迫和缺水胁迫下没有显著的VvUBC变化。Fei等[8]发现莱茵衣藻缺氮胁迫下泛素结合酶CrUBC2表达增强,并且可以促进衣藻油脂的积累;泛素-蛋白酶体途径具有保护衣藻免受硒胁迫,但亚硒酸盐毒性引起的严重氧化应激可能阻碍UPP介导应激反应的能力[17]。
E2泛素结合酶在高等植物中的功能研究较多,但对其在低等植物中的作用知之甚少。为研究CrUBC23在莱茵衣藻油脂代谢中的作用,本研究采用PCR技术扩增了UBC23基因干涉片段和基因全长,构建了RNAi干涉载体和过量表达载体,并转化莱茵衣藻CC425,对转基因藻株生长和油脂积累进行分析;构建CrUBC23-GFP融合表达载体,对其进行了亚细胞定位,以探讨CrUBC23在莱茵衣藻油脂代谢调控中的作用。
1 材料和方法 1.1 材料 实验藻株:细胞壁缺失型藻株莱茵衣藻CC425(中国科学院水生生物研究所);培养条件:HSM固体培养基,24 ℃,光照强度110 μmol/(m2·s)继代培养保存,液体培养时接种50 mL HSM液体培养基培养,缺氮(HSM-N)和缺磷(HSM-P)培养时,用HSM液体培养基培养至对数期时,取适量藻液4000 r/min离心1 min收集藻体,ddH2O重悬藻细胞后接种到HSM-N和HSM-P液体培养基24 ℃、180 r/min振荡培养。大肠杆菌DH5α(本实验室保存)用于载体构建,采用LB培养基37 ℃、220 r/min振荡培养。农杆菌EHA105 (本实验室保存)LB培养基28 ℃振荡培养。抗生素使用:氨苄青霉素(Ampicillin) 100 μg/mL;卡那霉素(Kanamycin) 30 μg/mL;巴龙霉素(Paromomycin) 5 μg/mL,潮霉素(Hygromycin B) 10 μg/mL;5-氟吲哚(5-Fluoroindol) 5 mmol/L;L-色氨酸1.5 mmol/L;尼罗红(Nile red) 10 μg/mL;利福平(Rifampicin) 25 μg/mL;乙酰丁香酮(As) 20 mmol/L。载体:RNA干涉载体pMaa7/XIR (美国Duke大学衣藻中心);T282载体(本实验室构建);pCAMBIA1302 (实验室保存),pMD18-T (宝生物工程(大连)有限公司)。试剂:Trizol总RNA抽提试剂盒、MMLV反转录试剂盒,SYBR?Premix Ex TaqTM Ⅱ试剂盒,PCR聚合酶、限制性内切酶、DNA连接酶均购自宝生物工程(大连)有限公司;质粒抽提试剂盒、PCR产物纯化试剂盒、胶回收试剂盒等均购自生工生物工程(上海)股份有限公司。
1.2 莱茵衣藻RNA提取、cDNA合成及qRT-PCR分析 莱茵衣藻培养至对数生长期,4000 r/min离心5 min收集藻体,液氮速冻研磨后按照Trizol总RNA抽提试剂盒抽提总RNA。按照MMLV反转录试剂盒合成cDNA。按照SYBR?Premix Ex TaqTM Ⅱ试剂盒进行基因表达量检测。莱茵衣藻18S rRNA基因作为内参,采用2–△△Ct法进行相对定量计算,SPSS 22.0做统计分析。
1.3 RNA干涉载体构建 根据phytozome中C. reinhardtii数据库公布的E2泛素结合酶CrUBC23 (Cre12.g546650) CDs的序列,设计特异性引物(表 1),以cDNA为模板,PCR扩增干涉片段,插入pMD18-T,得到载体pMD-CrUBC23。根据Li等[18]莱茵衣藻RNA干涉载体构建流程构建载体。
表 1. 用于PCR扩增引物 Table 1. Primers for PCR amplification
| Primer name | Primer sequence (5′→3′) | |
| RNAi-CrUBC23/qRT-PCR | F: TCGTGGCTTGTTATCAGACG | R: ACCAGACATACCGCACAACA |
| Full-CrUBC23 | F: TTGTAGGGTAGCTGCGATGA | R: AGAGCATCTCCTGTGACTTGC |
| pC-CrUBC23 | F: CATGCCATGGTGACGGACGCGCTGGCAG | R: GGACTAGTCTAGAGCATCTCCTGTGACTTGC |
| CrUBC23-GFP | F: CATGCCATGGTGACGGACGCGCTGGCAG | R: GGACTAGTGAGCATCTCCTGTGACTTGC |
| 18S rRNA[19] pMaa7IR pCAMBIA1302 | F: GTCAGAGGTGAAATTCTTGGATTTA F: GATTTAAATGCCAGAAGGAG F: CGGTCGGCATCTACTCTATT | R: AGGGCAGGGACGTAATCAACG R: CGCGGTGAGCACCGGAACGG R: GACAGCGTCTCCGACCTGA |
表选项
1.4 全长基因克隆及过量表达、CrUBC23-GFP融合表达载体构建 莱茵衣藻CC425 cDNA为模板扩增得到全长基因,插入pMD18-T载体,PCR引入NcoⅠ和SpeⅠ酶切位点,插入pCAMBIA1302,得到过量表达载体pC-CrUBC23,GFP融合表达载体构建引入酶切位点时,引物设计取掉终止密码子,插入pCAMBIA1302 NcoⅠ和SpeⅠ位点。得到GFP融合表达载体pC-CrUBC23-GFP。
1.5 莱茵衣藻转化及油脂含量测定
1.5.1 莱茵衣藻的转化: 采用Glass Beads法[20]转化莱茵衣藻CC425。转化后藻株转移至50 mL无菌离心管中,24 ℃、120 r/min温育过夜。4000 r/min离心5 min收集藻细胞,涂布于含有L-色氨酸和巴龙霉素的TAP固体培养基培养6–8 d。挑取单克隆,接种到5-氟吲哚TAP固体培养基平板上复筛。提取基因组DNA做PCR验证,得到转基因藻株。
1.5.2 转基因藻株检测: 将转基因单克隆藻株挑取至96孔酶标板,用200 μL ddH2O混匀,用终浓度为10 μg/mL的尼罗红染色10 min,酶标仪测定575 nm吸光值。根据三酰甘油标准曲线[18]计算中性油脂含量。选取3个转基因藻株,接种到HSM培养基中,每隔1 d用酶标仪(BIO-TEKElx800)测定490 nm波长处各样品的吸光值,绘制出转化子的生长曲线,每个样品做3次重复。每隔1 d取适量藻液采用酸水解法[21](GB_T 5009.6-2003)测定总脂含量。培养第4天,用尼罗红对藻细胞染色,在荧光显微镜下观察藻细胞生长状态及油脂情况,提取RNA,检测CrUBC23 mRNA水平。
1.6 农杆菌转化洋葱表皮细胞 pC-CrUBC23-GFP融合表达载体转化农杆菌EHA105,挑取单克隆,提取质粒酶切验证。农杆菌EHA105接种于30 mL含有卡那霉素和利福平的液体LB培养基,28 ℃、200 r/min振荡培养至OD600=0.6–0.8。4000 r/min离心5 min收集细胞,1/2 MS液体培养基悬浮菌体。按照1:50的比例转接到含有As的50 mL YEB液体培养基中200 r/min、28 ℃培养过夜后, 取适量菌液,4000 r/min离心5 min,以含有10 mmol/L MgCl2和20 μmol/L As的YEB液体培养基重悬菌体。取洋葱的鳞茎,将其切成1 cm2的小块,用镊子剥取洋葱的表皮细胞, 并用无菌水浸泡、滤纸吸干表面的液体,平铺在1/2 MS固体培养上,28 ℃暗培养24 h,将预培养后的洋葱表皮在重悬后的农杆菌液中,0.8 MPa真空抽滤10–15 min。滤纸吸干表面的菌液,转入1/2 MS固体培养基中25 ℃培养24 h,光照周期为16 h光照/8 h黑暗。共聚焦显微镜488 nm紫外激发光下观察。
2 结果和分析 2.1 莱茵衣藻CC425在缺氮、缺磷培养下CrUBC23 mRNA分析 qRT-PCR结果显示,莱茵衣藻CC425在正常培养、缺氮培养(HSM-N)、缺磷培养(HSM-P)下,随着培养时间的增长CrUBC23 mRNA水平显著升高。在缺氮培养下CrUBC23 mRNA水平显著增加,第3天达到最大值,之后缓慢下降,CrUBC23 mRNA水平是对照(CC425+N)的4.98–5.80倍(图 1-A),在缺磷培养下CrUBC23 mRNA前4天均显著上升,CrUBC23 mRNA水平是对照(CC425+P)的1.85–5.20倍(图 1-B)。
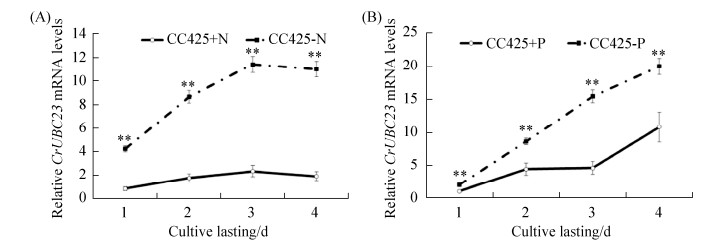 |
| 图 1 莱茵衣藻CC425缺氮(A)、缺磷(B)培养下CrUBC23 mRNA水平 Figure 1 The level of CrUBC23 mRNA of C. reinhardtii CC425 in nitrogen (A) and phosphorus (B) reduction culture statistical analysis was performed using SPSS 22.0. Significance is indicated as asterisks (**P < 0.01). |
| 图选项 |
2.2 CrUBC23生物信息学分析 CrUBC23基因全长507 bp,168 aa,编码Ubiquitin carrier protein,具有典型的UBCc功能结构域,具有半胱氨酸(cysteine)酶活性位点,属于class Ⅰ E2泛素结合酶。预测分子式为C834H1292N226O256S8,分子量18837.2 Da,pI=5.08,稳定系数45.65,脂肪系数72.40,无跨膜结构区。亚细胞定位于细胞质和细胞核(表 2)。与同源其他物种进化树分析显示,CrUBC23与数据库公布的团藻(XP_002953636)同源性较高,为93.40%,与水稻(XP_006644986)同源性较近,为77.8% (图 2)。
表 2. 蛋白同源性分析? Table 2. Homology matrix of 11 sequences?
| (%) | |||||||||||||||||||||||||||||
| Species Number | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | ||||||||||||||||||
| 1 | 100.00 | ||||||||||||||||||||||||||||
| 2 | 93.40 | 100.00 | |||||||||||||||||||||||||||
| 3 | 76.00 | 77.20 | 100.00 | ||||||||||||||||||||||||||
| 4 | 74.90 | 76.60 | 87.00 | 100.00 | |||||||||||||||||||||||||
| 5 | 74.70 | 76.50 | 84.50 | 84.50 | 100.00 | ||||||||||||||||||||||||
| 6 | 74.90 | 74.30 | 81.70 | 79.90 | 91.70 | 100.00 | |||||||||||||||||||||||
| 7 | 77.80 | 77.80 | 86.40 | 92.90 | 84.50 | 82.20 | 100.00 | ||||||||||||||||||||||
| 8 | 76.50 | 78.90 | 75.90 | 73.50 | 74.70 | 73.50 | 73.50 | 100.00 | |||||||||||||||||||||
| 9 | 77.10 | 75.90 | 84.90 | 84.30 | 88.00 | 87.30 | 87.30 | 73.50 | 100.00 | ||||||||||||||||||||
| 10 | 70.70 | 71.30 | 71.80 | 76.90 | 81.00 | 78.70 | 78.70 | 71.70 | 82.50 | 100.00 | |||||||||||||||||||
| 11 | 13.20 | 13.20 | 11.40 | 13.60 | 14.20 | 12.70 | 13.60 | 12.60 | 14.50 | 12.80 | 100.00 | ||||||||||||||||||
| 1: Chlamydomonas; 2: Volvox; 3: Nelumbo; 4: Zea; 5: Coffea; 6: Solanum; 7: Oryza; 8: Chlorella; 9: Arabidopsis; 10: Tarenaya; 11: Homo. | |||||||||||||||||||||||||||||
表选项
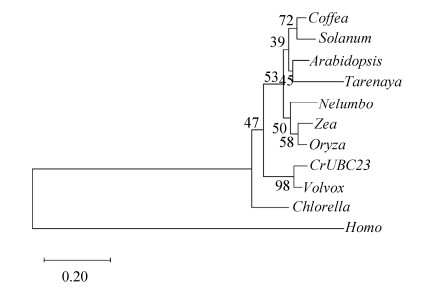 |
| 图 2 CrUBC23与其他物种同源性比较 Figure 2 Clustering analysis of CrUBC23 orthologous genes in C. reinhardtii and other species. Chlamydomonas CrUBC23: (Cre12.g546650), Volvox (EFJ45260), Nelumbo (XP_010253222), Zea (AFW84636), Coffea (CDP00072), Solanum (XP_010321377), Oryza (XP_006644986), Chlorella (EFN52212), Arabidopsis (NP_568902), Tarenaya (XP_010534487), Homo (AAH14880). |
| 图选项 |
2.3 转基因藻株的构建 通过PCR扩增获得CrUBC23干涉片段(图 3-B-a),插入pMD18-T,分别用Hind Ⅲ/BamHⅠ和XbaⅠ/SalⅠ双酶切获得正反向干涉片段(图 3-B-b),依次插入T282载体,获得中间载体,EcoRⅠ酶切中间载体获得含有CrUBC23正反向结构的片段,插入pMaa7IR/XIR (图 3-A),EcoRⅠ酶切显示,pMaa7IR/CrUBC23IR构建成功(图 3-B-d)。全长基因插入pCAMBIA1302 NcoⅠ和SpeⅠ酶切位点,得到过量表达载体pC-CrUBC23。玻璃珠法转化莱茵衣藻CC425 (图 3-C),挑取单克隆提取转基因藻株基因组DNA,进行PCR检测,结果显示pMaa7IR/CrUBC23IR (图 3-D-d)和pC-CrUBC23 (图 3-D-e)已经成功整合到莱茵衣藻CC425基因组DNA上,CrUBC23 RNAi和过量表达藻株构建成功。
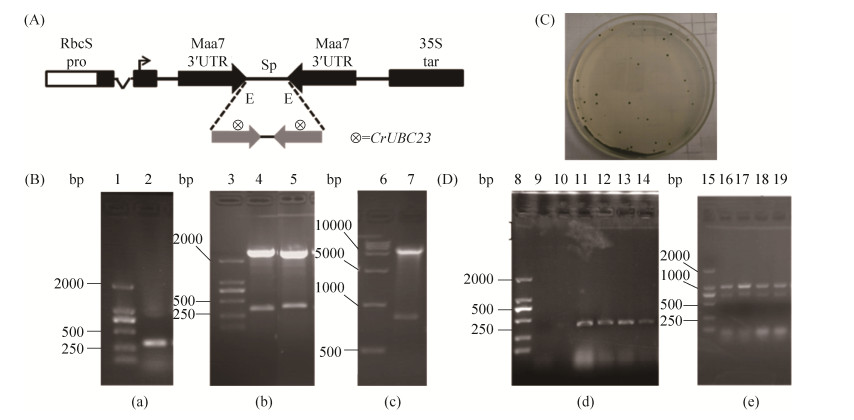 |
| 图 3 CrUBC23转基因藻株构建 Figure 3 Construction of CrUBC23 RNAi and over-expression transgenic algae. A: pMaa7IR/XIR vector. B: PCR products, a: Cloning of fragment of CrUBC23; b: Acquisition of forward and backward interference fragments; c: Identification of pMaa7IR/CrUBC23IR by EcoR I. C: Resistance screening of CrUBC23 RNAi and over-expression transgenic algae. D: PCR products, d: PCR detection of pMaa7IR/CrUBC23IR transgenic algae strains; e: PCR detection of pC-CrUBC23 transgenic algae strains. |
| 图选项 |
2.4 CrUBC23 RNAi转基因藻株生物量及油脂含量测定 尼罗红染色法测定了96株CrUBC23 RNAi转基因藻株和54株pMaa7IR/XIR转基因藻株中性脂含量,CrUBC23 RNAi转基因藻株平均NF Fluorescence/DW值为894,对照pMaa7IR/XIR平均NF Fluorescence/DW值为1390 (图 4-A和B)。选择3株转基因藻株,HSM液体培养测定生物量和油脂含量,结果显示RNAi转基因藻株生长速率减慢,生长第6天,生物量比对照pMaa7IR/XIR转基因藻株增加了4.71%–10.45% (图 4-C)。中性油脂测定显示,藻株油脂含量随着培养时间的增长而增加,第8天达到最高值,CrUBC23 RNAi转基因藻株中性脂含量比对照pMaa7IR/XIR转基因藻株平均降低5.5% (图 4-D)。培养第8天,取适量藻株用酸水解法测定油脂含量,结果显示3株CrUBC23 RNAi转基因藻株油脂含量比对照pMaa7IR/XIR转基因藻株和CC425油脂含量显著降低,降幅为3.16%–17.6% (图 4-E)。培养第4天CrUBC23 RNAi转基因藻株CrUBC23 mRNA分析显示,CrUBC23 mRNA比对照pMaa7IR/XIR转基因藻株显著降低,降低幅度为88.03%–93.2% (图 4-F),沉默效果明显。
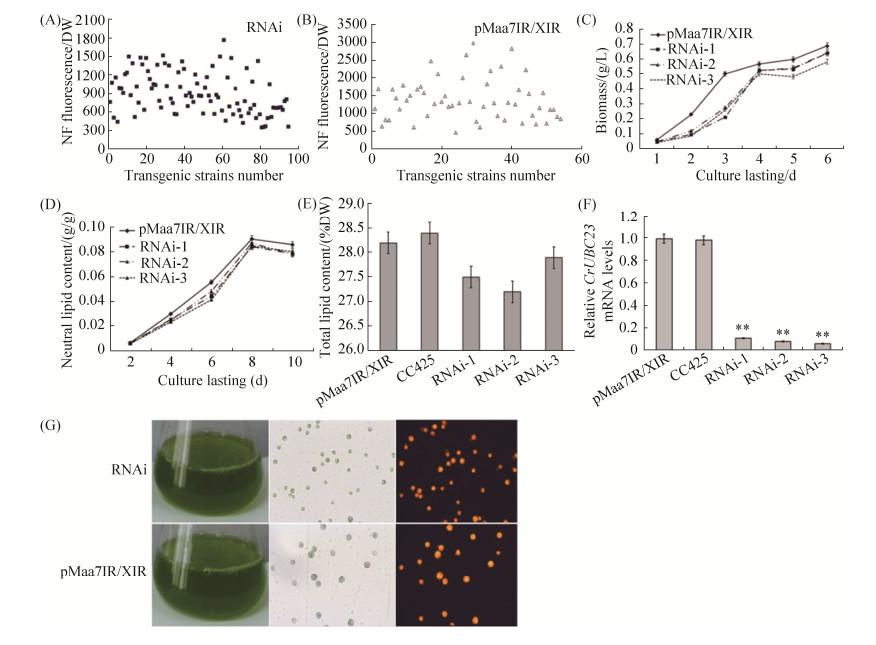 |
| 图 4 CrUBC23 RNAi转基因藻株分析 Figure 4 Analyses of the CrUBC23 RNAi transgenic algae. A, B: Neutral lipid content detected via the Nile red fluorescence method of CrUBC23 RNAi transgenic algae and the control Maa7IR/XIR transgenic line; C: Growth curves of the CrUBC23 RNAi transgenic algae; D: The neutral lipid content of the CrUBC23 RNAi transgenic algae; E: Total lipid content of the CrUBC23 RNAi transgenic algae; F: mRNA abundance of the CrUBC23 RNAi transgenic algae; G: Microscopic observations of CrUBC23 RNAi transgenic algae. After 4 days of cultivation (Nikon 80i 40×10). Statistical analysis was performed using SPSS 22.0. Significance is indicated as asterisks (**P < 0.01). |
| 图选项 |
2.5 过量表达CrUBC23转基因藻株油脂含量增加 尼罗红染色法测定了38株CrUBC23过量表达转基因藻株(pC-CrUBC23)和56株pCAMBIA1302转基因藻株(pC1302)中性脂含量,pC-CrUBC23平均NF Fluorescence/DW值为3299.67,对照pC1302平均NF Fluorescence/DW值为1665.10 (图 5-A和B)。生物量测定结果显示pC-CrUBC23与对照pC1302无显著性变化(图 5-C)。中性脂测定显示,藻株油脂含量随着培养时间的增长而增加,第6天后达到最高值,中性脂含量比对照pC1302平均增加8.8% (图 5-D)。培养第8天用酸水解法测定油脂含量,结果显示3株pC-CrUBC23油脂含量比对照pC1302增加4.51%–14.03% (图 5-E)。培养第4天CrUBC23 mRNA分析显示,pC-CrUBC23CrUBC23 mRNA比对照pC1302显著增加,表达量是对照的1.91–2.20倍(图 5-F)。
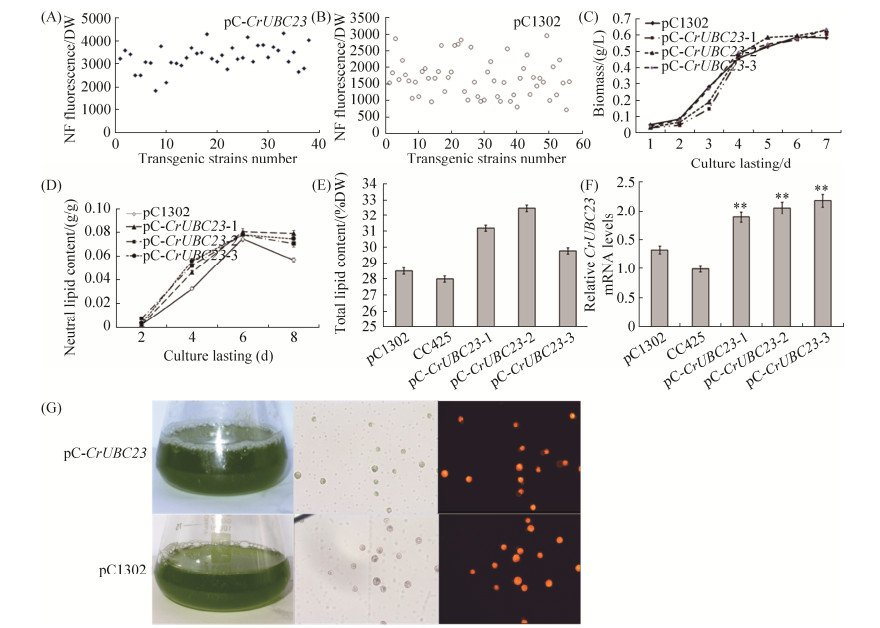 |
| 图 5 CrUBC23过量表达分析 Figure 5 Analyses of the CrUBC23 over-expression transgenic algae. A, B: Neutral lipid content detected via the Nile red fluorescence method of CrUBC23 overexpression transgenic algae and the control pCAMBIA1302 transgenic line; C: Growth curves of the CrUBC23 overexpression transgenic algae; D: The neutral lipid content of the CrUBC23 overexpression transgenic algae; E: Total lipid content of the CrUBC23 overexpression transgenic algae; F: mRNA abundance of the CrUBC23 overexpression transgenic algae; G: Microscopic observations of CrUBC23 overexpression transgenic algae. After 4 days of cultivation (Nikon 80i 40×10). Statistical analysis was performed using SPSS 22.0. Significance is indicated as asterisks (**P < 0.01). |
| 图选项 |
2.6 CrUBC23在洋葱表皮细胞中的瞬时表达 蛋白质的亚细胞定位与其功能的发挥有着密切的关系,洋葱表皮细胞体积较大,且为单层细胞,有典型的植物细胞结构,是目前研究蛋白亚细胞定位的良好材料。本研究构建了CaMV35S启动CrUBC23融合GFP的载体,利用绿色荧光蛋白的信号来确认目标蛋白的定位,农杆菌介导转化洋葱表皮细胞,以pCAMBIA1302 (pC1302)为阳性对照,ddH2O为阴性对照(CK),结果显示GFP报告基因在阳性对照细胞质和细胞核均表达,pC-CrUBC23-GFP融合蛋白表达于洋葱表皮细胞的细胞核(图 6)。细胞核是DNA复制和转录的主要场所,CrUBC23定位于细胞核,其功能可能和DNA复制和转录有关。
 |
| 图 6 CrUBC23在洋葱表皮的瞬时表达 Figure 6 Transient expression of CrUBC23 in onion epidermis. |
| 图选项 |
3 讨论 泛素-蛋白酶体途径是生物体内除溶酶体之外的另一条非常重要的蛋白质降解途径。蛋白质水解过程由泛素化和蛋白质水解两部分组成。植物E2泛素结合酶种类繁多,是连接E1泛素激活酶和E3泛素连接酶的桥梁,对泛素化修饰的特异性和精确时空性起关键作用[22]。E2主要参与DNA修复、植物的生长发育、光形态建成、缺素及抗逆胁迫响应。拟南芥中过量表达大豆GmUBC基因可以显著提高拟南芥对干旱和高盐抗性[23]。玉米泛素结合酶ZmUBC-76在盐胁迫和干旱胁迫下下调表达[24],Fei等[8]研究表明,莱茵衣藻泛素结合酶CrUBC2可以响应低氮胁迫,并可以促进莱茵衣藻油脂的积累。本研究中我们对莱茵衣藻CC425在减氮、减磷培养下CrUBC23基因表达量进行了分析,结果显示,莱茵衣藻CC425在减氮、减磷培养下CrUBC23基因均出现大幅度的增加,并随着培养时间的增长而增加。结果与转录组测序分析相一致,在减氮、减磷胁迫下,藻细胞产生各种异常或受损的蛋白,这些受损蛋白的积累,会影响细胞的各种代谢,而这个异常蛋白的降解大部分靠泛素-蛋白酶体途径来实现,E2泛素结合酶作为E1泛素激活酶和E3泛素连接酶的桥梁,具有结合并转移泛素到靶蛋白的功能,对蛋白泛素化修饰起着非常关键的作用[25]。
RNAi基因沉默技术和过量表达技术是互相验证基因功能的生物技术手段,本文针对CrUBC23基因构建了RNA干涉载体及CaMV 35S启动型过量表达载体,成功实现CrUBC23基因在莱茵衣藻CC425中的沉默和过量表达。转基因藻株结果显示,CrUBC23基因沉默后,转基因藻株总脂比对照降低了3.16%–17.6%,而过量表达后总脂含量比对照增加4.51%–14.03%,说明CrUBC23正向调控莱茵衣藻油积累。此结果趋势与Fei等[8]对E2泛素结合酶CrUBC2沉默和过量表达对莱茵衣藻油脂积累结果相吻合。目前微藻油脂代谢调控研究是微藻生物柴油研发中优势藻种改良研究的热点之一,但报道的基因主要集中在微藻三酰甘油(TAG)合成途径中的关键酶,如乙酰辅酶A羧化酶(acetyl-CoA carboxylase,ACCase)[26-27]、脂肪酸合酶(fatty acid synthase,FAS)[28]、甘油-3-磷酸酰基转移酶(glyceral 3-phosphateacyltransferase,GPAT)[29]、二脂酰甘油酰基转移酶(diacylglycerol acyltrans-ferase,DGAT)[30]、磷脂酸磷酸酶(PAP)[31],他们在三酰甘油(TAG)合成途径中起着重要的作用,然而有关调节基因的报道很少。目前有文献报道E3泛素连接酶参与脂肪酸代谢,Lü等[32]在CER9 E3泛素连接酶调控拟南芥油脂合成机理研究中发现,拟南芥CER9基因功能缺失突变体cer9-2种子含油率和Col-0野生型相比相对增加了17%,Luo等[33]在研究U-box E3泛素连接酶基因家族时发现,有效沉默CrPUB5和CrPUB14后莱茵衣藻油脂积累增加9.8%–45.0%和14.4%–61.8%。据报道大多数的E2泛素结合酶定位于细胞质,少数定位于细胞核。CrUBC23定位于细胞核,可能是由于底物性质的不同有所差异,也可能与UBCc结构域、活性位点和C末端有关[34]。细胞核是DNA复制和转录的主要场所,CrUBC23定位于细胞核,其功能可能与DNA复制和转录有关。现在报道CrUBC23同源基因CrUBC2可以与CrUBC13互作在DNA损伤修复中发挥重要的功能,并可以正向调控莱茵衣藻油脂,至于CrUBC23是否也和CrUBC2一样,具有莱茵衣藻DNA损伤修复功能,本文只验证了其可以正向调控莱茵衣藻油脂,是否具有DNA损伤修复功能以及如何调控油脂的积累,与其结合识别底物的E3泛素连接酶是什么有待进一步研究。CrUBC23 E2泛素结合酶是泛素-蛋白酶体途径的重要组成部分,在微藻各类生理生化中起着关键的作用,E2泛素结合酶同源基因是否参与微藻油脂的代谢,有待进一步研究。
综上所述,CrUBC23参与莱茵衣藻缺氮、缺磷胁迫响应,正向调控油脂积累,亚细胞定位于细胞核,其在莱茵衣藻油脂代谢调控中的作用有待进一步研究,本研究为揭示微藻油脂代谢调控网络和优势藻株基因工程改良提供了有用的理论依据。
References
| [1] | Muto M, Nojima D, Yue L, Kanehara H, Naruse K, Ujiro A, Yoshino T, Matsunaga T, Tanaka T. Potential of water surface-floating microalgae for biodiesel production: floating-biomass and lipid productivities. Journal of Bioscience and Bioengineering, 2017, 123(3): 314-318. DOI:10.1016/j.jbiosc.2016.09.015 |
| [2] | Singh J, Gu S. Commercialization potential of microalgae for biofuels production. Renewable and Sustainable Energy Reviews, 2010, 14(9): 2596-2610. DOI:10.1016/j.rser.2010.06.014 |
| [3] | Mata TM, Martins AA, Caetano NS. Microalgae for biodiesel production and other applications: a review. Renewable and Sustainable Energy Reviews, 2010, 14(1): 217-232. DOI:10.1016/j.rser.2009.07.020 |
| [4] | Longworth J, Wu DY, Huete-Ortega M, Wright PC, Vaidyanathan S. Proteome response of Phaeodactylum tricornutum, during lipid accumulation induced by nitrogen depletion. Algal Research, 2016, 18: 213-224. DOI:10.1016/j.algal.2016.06.015 |
| [5] | Hounslow E, Kapoore RV, Vaidyanathan S, Gilmour DJ, Wright PC. The search for a lipid trigger: the effect of salt stress on the lipid profile of the model Microalgal species Chlamydomonas reinhardtii for biofuels production. Current Biotechnology, 2016, 5(4): 305-313. DOI:10.2174/2211550105666160322234434 |
| [6] | Chaisutyakorn P, Praiboon J, Kaewsuralikhit C. The effect of temperature on growth and lipid and fatty acid composition on marine microalgae used for biodiesel production. Journal of Applied Phycology, 2017, 30(1): 37-45. |
| [7] | Loebl M, Cockshutt AM, Campbell DA, Finkel ZV. Physiological basis for high resistance to photoinhibition under nitrogen depletion in Emiliania huxleyi. Limnology and Oceanography, 2010, 55(5): 2150-2160. DOI:10.4319/lo.2010.55.5.2150 |
| [8] | Fei XW, Li XH, Li P, Deng XD. Involvement of Chlamydomonas DNA damage tolerence gene UBC2 in lipid accumulation. Algal Research, 2017, 22: 148-159. DOI:10.1016/j.algal.2016.12.019 |
| [9] | Yau RG, Doerner K, Castellanos ER, Haakonsen DL, Werner A, Wang N, Yang XW, Martinez-Martin N, Matsumoto ML, Dixit VM, Rape M. Assembly and function of heterotypic ubiquitin chains in cell-cycle and protein quality control. Cell, 2017, 171(4): 918-933. DOI:10.1016/j.cell.2017.09.040 |
| [10] | Li Q, Li Z, Wei S, Wang WZ, Chen Z, Zhang L, Chen L, Li BW, Sun GL, Xu JH, Li Q, Wang L, Xu ZP, Xia YW, Zhang DC, Xu H, Xu ZK. Overexpression of miR-584-5p inhibits proliferation and induces apoptosis by targeting WW domain-containing E3 ubiquitin protein ligase 1 in gastric cancer. Journal of Experimental & Clinical Cancer Research, 2017, 36: 59. |
| [11] | Shao GB, Wang RR, Sun AQ, Wei J, Peng K, Dai Q, Yang WN, Lin Q. The E3 ubiquitin ligase NEDD4 mediates cell migration signaling of EGFR in lung cancer cells. Molecular Cancer, 2018, 17(1): 24. DOI:10.1186/s12943-018-0784-2 |
| [12] | Foot N, Henshall T, Kumar S. Ubiquitination and the regulation of membrane proteins. Physiological Reviews, 2017, 97(1): 253-281. |
| [13] | Palmer AL, De Jong A, Leestemaker Y, Geurink PP, Wijdeven RH, Ovaa H, Dolan BP. Inhibition of the Deubiquitinase Usp14 diminishes direct MHC class Ⅰ antigen presentation. The Journal of Immunology, 2018, 200(3): 928-936. DOI:10.4049/jimmunol.1700273 |
| [14] | Van Wijk SJL, Timmers HTM. The family of ubiquitin-conjugating enzymes (E2s): deciding between life and death of proteins. The FASEB Journal, 2010, 24(4): 981-993. DOI:10.1096/fj.09-136259 |
| [15] | Nurdiani D, Widyajayantie D, Nugroho S. OsSCE1 encoding SUMO E2-conjugating enzyme involves in drought stress response of Oryza sativa. Rice Science, 2018, 25(2): 73-81. DOI:10.1016/j.rsci.2017.11.002 |
| [16] | Gao YY, Wang Y, Xin HP, Li SH, Liang ZC. Involvement of ubiquitin-conjugating enzyme (E2 Gene Family) in ripening process and response to cold and heat stress of Vitis vinifera. Scientific Reports, 2017, 7(1): 13290. DOI:10.1038/s41598-017-13513-x |
| [17] | Vallentine P, Hung CY, Xie JH, Hoewyk DV. The ubiquitin-proteasome pathway protects Chlamydomonas reinhardtii against selenite toxicity, but is impaired as reactive oxygen species accumulate. AoB PLANTS, 2014, 6: plu062. DOI:10.1093/aobpla/plu062 |
| [18] | Li XH, Fei XW, Deng XD. Construction of phosphofructokinase RNAi vectors and its impact on lipid accumulation in Chlamydomonas reinhardtii. Guangdong Agricultural Sciences, 2014, 41(10): 155-159. (in Chinese) 李兴涵, 费小雯, 邓晓东. 莱茵衣藻磷酸果糖激酶RNAi载体构建及其对莱茵衣藻油脂积累的影响. 广东农业科学, 2014, 41(10): 155-159. |
| [19] | Rasoul-Amini S, Ghasemi Y, Morowvat MH, Mohagheghzadeh A. PCR amplification of 18S rRNA, single cell protein production and fatty acid evaluation of some naturally isolated microalgae. Food Chemistry, 2009, 116(1): 129-136. |
| [20] | Kindle KL. High-frequency nuclear transformation of Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(3): 1228-1232. DOI:10.1073/pnas.87.3.1228 |
| [21] | Ma S, Fu LL, Wang M, Hu XW, Tan DG, Zhang JM. Comparison of extraction methods of crude fat from microalgae. China Oils and Fats, 2010, 35(5): 77-79. (in Chinese) 马帅, 付莉莉, 汪萌, 胡小文, 谭德冠, 张家明. 从微藻中提取粗脂的方法比较. 中国油脂, 2010, 35(5): 77-79. |
| [22] | Hu BL, Wan Y, Li X, Lei JG, Luo XD, Yan WG, Xie JK. Analysis on genetic diversity of phenotypic traits in rice (Oryza sativa) core collection and its comprehensive assessment. Acta Agronomica Sinica, 2012, 38(5): 829-839. (in Chinese) 胡标林, 万勇, 李霞, 雷建国, 罗向东, 严文贵, 谢建坤. 水稻核心种质表型性状遗传多样性分析及综合评价. 作物学报, 2012, 38(5): 829-839. |
| [23] | Chung E, Cho CW, So HA, Kang JS, Chung YS, Lee JH. Overexpression of VrUBC1, a mung bean E2 ubiquitin-conjugating enzyme, enhances osmotic stress tolerance in Arabidopsis. PLoS One, 2013, 8(6): e66056. DOI:10.1371/journal.pone.0066056 |
| [24] | Jue DW, Sang XL, Shu B, Liu LQ, Wang YC, Shi SY. Functional analysis of a ubiquitin-conjugating enzyme gene ZmUBC-76 in maize. Chinese Journal of Tropical Crops, 2017, 38(8): 1507-1511. (in Chinese) 决登伟, 桑雪莲, 舒波, 刘丽琴, 王一承, 石胜友. 玉米泛素结合酶基因zmubc-76的功能分析. 热带作物学报, 2017, 38(8): 1507-1511. |
| [25] | Zheng N, Shabek N. Ubiquitin ligases: structure, function, and regulation. Annual Review of Biochemistry, 2017, 86: 129-157. DOI:10.1146/annurev-biochem-060815-014922 |
| [26] | Radakovits R, Jinkerson RE, Darzins A, Posewitz MC. Genetic engineering of algae for enhanced biofuel production. Eukaryotic Cell, 2010, 9(4): 486-501. DOI:10.1128/EC.00364-09 |
| [27] | Gomma AE, Lee SK, Sun SM, Yang SH, Chung G. Improvement in oil production by increasing malonyl-CoA and glycerol-3-phosphate pools in Scenedesmus quadricauda. Indian Journal of Microbiology, 2015, 55(4): 447-455. DOI:10.1007/s12088-015-0546-4 |
| [28] | Blatti JL, Michaud J, Burkart MD. Engineering fatty acid biosynthesis in microalgae for sustainable biodiesel. Current Opinion in Chemical Biology, 2013, 17(3): 496-505. DOI:10.1016/j.cbpa.2013.04.007 |
| [29] | Niu YF, Wang X, Hu DX, Balamurugan S, Li DW, Yang WD, Liu JS, Li HY. Molecular characterization of a glycerol-3-phosphate acyltransferase reveals key features essential for triacylglycerol production in Phaeodactylum tricornutum. Biotechnology for Biofuels, 2016, 9: 60. DOI:10.1186/s13068-016-0478-1 |
| [30] | Deng XD, Gu B, Li YJ, Hu XW, Guo JC, Fei XW. The roles of acyl-CoA: diacylglycerol acyltransferase 2 genes in the biosynthesis of triacylglycerols by the green algae Chlamydomonas reinhardtii. Molecular Plant, 2012, 5(4): 945-947. DOI:10.1093/mp/sss040 |
| [31] | Deng XD, Cai JJ, Fei XW. Involvement of phosphatidate phosphatase in the biosynthesis of triacylglycerols in Chlamydomonas reinhardtii. Journal of Zhejiang University SCIENCE B, 2013, 14(12): 1121-1131. DOI:10.1631/jzus.B1300180 |
| [32] | Lü SY, Zhao HY, Des Marais DL, Parsons EP, Wen XX, Xu XJ, Bangarusamy DK, Wang GC, Rowland O, Juenger T, Bressan RA, Jenks MA. Arabidopsis ECERIFERUM9 involvement in cuticle formation and maintenance of plant water status. Plant Physiology, 2012, 159(3): 930-944. DOI:10.1104/pp.112.198697 |
| [33] | Luo QL, Li YJ, Wang WQ, Fei XW, Deng XD. Genome-wide survey and expression analysis of Chlamydomonas reinhardtii U-box E3 ubiquitin ligases (CrPUBs) reveal a functional lipid metabolism module. PLoS One, 2015, 10(3): e0122600. DOI:10.1371/journal.pone.0122600 |
| [34] | Zhang XD, Matunis MJ. Ub in charge: regulating E2 enzyme nuclear import. Nature Cell Biology, 2005, 7(1): 12-14. DOI:10.1038/ncb0105-12 |
