曾军, 吴宇澄, 林先贵


中国科学院南京土壤研究所土壤环境与污染修复重点实验室, 江苏南京 210008
收稿日期:2019-10-31;修回日期:2020-03-20;网络出版日期:2020-05-29
基金项目:国家重点基础研究发展计划(2014CB441106);江苏省自然科学基金(BK20181512);国家自然科学基金(41671266)
作者简介:曾军, 中国科学院南京土壤研究所助理研究员, 主要从事土壤有机污染微生物修复研究, 重点关注多环芳烃、全氟化合物等持久性有机污染物的微生物降解代谢过程、土壤污染物迁移转化与微生物响应过程、有机污染土壤的微生物修复技术等。近年来重点针对难降解多环芳烃的微生物代谢、酶转化、土壤PAHs转化归趋特征与微生物响应等方面展开系统研究。在本专业领域已主持和参与过国家自然科学基金、973计划、863计划、国家重点研发计划等科研项目, 相关研究成果在Soil Biology & Biochemistry, Environmental Pollution, Applied Microbiology and Biotechnology等环境微生物学国际主流杂志刊发.
*通信作者:林先贵, E-mail:xglin@issas.ac.cn.
摘要:多环芳烃是我国土壤环境质量标准中要求严格管控的一类持久性有机污染物,利用微生物技术修复有机污染土壤具有绿色、经济等突出特点,应用前景广泛。目前多学科的协同发展和新技术的研究应用,为多环芳烃土壤微生物转化机制与污染生态过程等方面带来了新的认识,同时对修复技术的实际应用与调控提供了新的思考方向。本文以多环芳烃污染土壤微生物修复为主体,从污染土壤微生物修复应用技术、多环芳烃微生物降解特征、土壤体系污染物归趋规律与微生物作用及土壤污染微生物群落响应与研究技术等方面进行综合评述,并针对现存应用技术瓶颈和理论空白作进一步思考和展望。
关键词:多环芳烃土壤微生物修复群落结构与功能
Advances in microbial remediation of soils polluted by polycyclic aromatic hydrocarbons
Jun Zeng, Yucheng Wu, Xiangui Lin


Key Laboratory of Soil Environment and Pollution Remediation, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, Jiangsu Province, China
Received: 31 October 2019; Revised: 20 March 2020; Published online: 29 May 2020
*Corresponding author: Xiangui Lin, E-mail:xglin@issas.ac.cn.
Foundation item: Supported by the National Key Basic Research Program of China (2014CB441106), by the Natural Science Foundation of Jiangsu Province (BK20181512) and by the National Natural Science Foundation of China (41671266)
Abstract: Polycyclic aromatic hydrocarbons are a group of persistent organic pollutants that require strict control according to the national standard for soil environmental quality of China. Remediation using microbial process is recognized as economical and eco-friendly technology with great potential. Recent advances in microbial remediation with regards to biotransformation mechanisms and microbial response to soil pollution open up new perspectives on the field-scale bioremediation and management for polycyclic aromatic hydrocarbon-contaminated soil. The review provides an overview of microbial remediation of soil polluted by polycyclic aromatic hydrocarbons, surveying comprehensively from remediation technology, biodegradation characteristics, to microbial response to soil pollution. At last, some theoretical bottlenecks in development of microbial remediation techniques are discussed.
Keywords: polycyclic aromatic hydrocarbonssoilmicrobial remediationcommunity composition and function
多环芳烃(polycyclic aromatic hydrocarbons,PAHs)具有致癌、致畸、致突变等“三致”效应,是美国环保署优先控制的一类持久性有机污染物,据《全国土壤污染状况调查公报》,我国土壤PAHs点位超标率高达1.4%,土壤环境质量总体不容乐观。自然环境中有机污染物的去除主要通过微生物的降解或转化作用而实现,微生物修复技术即是利用这种生物过程进行土壤污染修复的方法,它与物理、化学方法相比更为环境友好、绿色经济。在农田污染土壤应用微生物技术期望满足修复兼顾农业生产的现实需求,以实现边生产边修复;在场地污染环境将微生物技术耦合更为高效的物理、化学技术则是为了实现修复工程的节能减耗。
微生物修复针对石油烃、有机农药、多氯联苯等污染土壤具有较好的效果,而对于疏水性更高、生物降解更难的PAHs,其修复难度更大。发展更为完善的PAHs污染土壤微生物修复技术,除了明晰污染物自身的生物降解代谢特征,还应结合实际环境中的土壤结构和性质、污染物理化特性、微生物群落组成和功能等进行综合考虑。本文重点就难降解PAHs的微生物代谢机制和土壤体系下PAHs微生物转化过程等方面作系统介绍,并就PAHs污染土壤微生物修复技术现存问题与发展趋势进行探讨。
1 PAHs污染土壤的微生物修复应用技术 有机污染土壤修复技术包括物理修复、化学修复、生物修复等。物理、化学修复技术利用温度、电动力、化学氧化等方式将污染物从土壤去除,起效快、周期短,但工程耗能较高且对土壤性质破坏较大;生物修复技术通过植物或微生物对污染物的吸收、降解或转化作用实现污染土壤修复,工程周期相对较长,但具有成本低且环境友好等独特优势,在农田污染土壤修复应用更为广泛。现行微生物修复技术对石油烃、多氯联苯、杀虫剂等多种有机污染土壤具有较好效果[1-3],但针对强疏水性PAHs的修复难度较大。微生物修复可分为原位和异位技术应用[4],原位技术不需将污染土壤搬离现场进行修复,通常采用土壤翻耕、营养物质添加、接种外源菌剂等措施在原位土壤进行修复作业,其修复成本低适用于大面积污染土壤修复;异位技术需要将污染土壤挖出,采用生物堆制法、制床法、泥浆生物器反应法等进行处理,土壤和修复剂在异位均匀混合能提供更充分的通氧因此降解效果更佳,但因修复成本高更适用于点源污染土壤。
土壤微生物修复从技术原理可划分为生物刺激和生物强化两大类型[5]。生物刺激是通过添加营养物质等刺激土著微生物降解活性来修复土壤的方法。有机污染土壤的有机质水平往往较高,其他营养物质的快速消耗可能限制土著微生物发挥降解作用,因此通过营养元素调整土壤碳、氮、磷等元素配比形成利于PAHs微生物降解的环境条件[6];另一方面,刺激方法也基于污染物的微生物转化代谢特征而直接强化特定过程,如研究发现芳烃结构化学物质可通过共代谢作用增强土壤难降解PAHs的生物降解[7-8]。实际应用中,生物刺激剂多选择植物秸秆、畜禽粪便、活性污泥等廉价工农业废弃物[9],通过原位添加或异位堆肥等方式进行土壤修复,它们在修复土壤的同时还能提升农田土壤肥力实现废弃资源再利用。其中发酵牛粪和植物秸秆等在调节土壤营养比、刺激土著降解真菌上发挥作用[10],菇渣废弃物自身包含大量白腐真菌残体及木质素降解酶,其修复机制则是木质素降解酶对PAHs的转化作用[11-12]。此外,鼠李糖脂等生物表面活性剂也是常用的土壤修复刺激剂[13],它在促进土壤PAHs溶出增强生物降解等方面发挥积极作用。
生物刺激法在实际土壤修复中更为常用,在自然环境中有机污染物降解微生物数量较低的时候,就需要考虑采用生物强化方法引入外源高效降解菌。强化采用的菌剂可来自实验室预培养的微生物纯培养物,也有经污染物预富集处理而饱含降解微生物的土壤[14]。通常外来接种降解微生物会受到土著生物的生态位竞争而难以成功定殖于新环境,而外源降解菌能否建立生态位是影响生物强化效率的一个重要因素。生物强化对于生物利用相对更好的石油烃、硝基苯、有机农药等往往能有效加速其降解进程[1, 3, 15],但是对于疏水性更强、生物利用性低的PAHs,因微生物难以建立合适的生态位而无法产生有效的修复效果。相比而言,采用酶制剂的生物强化修复更为直接有效,研究表明漆酶能够高效转化土壤中苯并[a]芘等高环PAHs[16],它通过提升污染物矿化、增强污染物与土壤有机质结合的方式实现污染物的环境脱毒[17],酶制剂实际应用最大的问题在于其容易失活、应用成本高;也有研究提出往土壤中引入含降解基因的质粒,通过质粒转化作用提高土著微生物的污染物降解活性。
联合其他修复方法的微生物修复技术往往能更大发挥降解性能,针对PAHs污染土壤目前发展出电动-微生物耦合修复、植物-微生物联合修复等有效技术。电动-微生物耦合修复技术利用电场作用促进土壤污染物和营养物质的传质过程,同时增强微生物迁移提高其与污染物接触进而促进污染物降解,该技术对低渗透性、传质低的粘土类土壤具有独特应用优势[18]。有研究表明,PAHs降解菌在电场作用下从土壤表层迁移至底层[19],电动耦合技术显著提高了土壤中的PAHs的微生物降解[20],同时通过添加表面活性剂可进一步强化该耦合技术修复效果。植物-微生物联合修复是农田污染土壤常用的原位修复技术,植物根系为微生物在土壤中的定殖提供了微环境,根系分泌物等也强化了根际区域PAHs的微生物降解,同时接种根瘤菌、菌根真菌等对植物促生微生物,可以改善植物在污染环境中的生长而提高修复效率[21];近年来,热脱附-微生物联合技术在场地污染土壤修复中引起关注,在场地热修复处理过程中耦合微生物强化降解作用有望降低热能输出从而控制工程应用成本[22-23]。实际土壤污染复杂、场景多变,在修复工程中需结合现场状况制定相匹配的微生物修复组合措施。
2 难降解PAHs的微生物代谢特征 2.1 细菌和真菌对PAHs的降解 解析难降解PAHs的微生物代谢特征为发展有效的微生物修复技术提供聚焦靶标,目前研究主要集中于细菌和真菌对PAHs污染物的代谢过程[24]。
细菌能以唯一碳源形式将PAHs直接矿化为CO2和H2O,降解速率通常与PAHs环数成反比。据报道,细菌对4环及以下的PAHs矿化效率较高,而对5环及以上PAHs的转化效果不明显,但在共代谢条件下可以促5环以上PAHs的降解[25]。细菌好氧降解PAHs通常通过羟基化双加氧、脱氢以及开环双加氧的步骤进行(图 1)[26]。其中,羟基化双加氧酶催化步骤是细菌降解PAHs的限速步骤,它为PAHs引入分子氧,产生顺式(cis-)二氢二醇,顺式二氢二醇通过脱氢、开环步骤继续被代谢降解。此外,某些细菌还存在细胞色素P450单加氧酶途径[27],它形成反式(trans-)二氢二醇,脱氢后形成PAHs二醇。该物质可进入矿化途径,也可通过形成复合物的方式进入解毒途径。
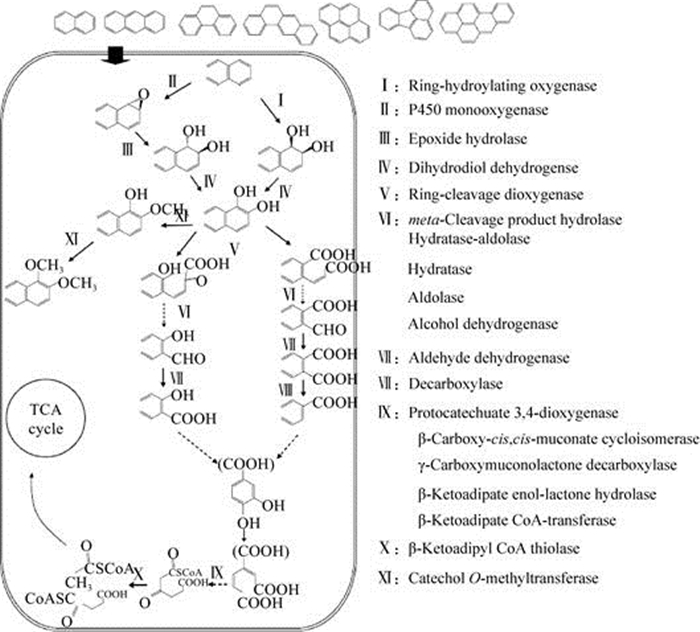 |
| 图 1 细菌对PAHs的主要降解途径[26] Figure 1 Degradative pathway of polycyclic aromatic hydrocarbons by aerobic bacteria[26]. |
| 图选项 |
真菌对PAHs的降解与细菌不同,一方面,它对PAHs的降解通常以共代谢形式进行,另一方面真菌对高环PAHs具有偏好性降解。据报道,许多真菌尤其白腐真菌都具有PAHs转化能力,但是真菌一般是将PAHs转化成水溶性更高的代谢产物而非直接矿化[28],Kotterman等[29]研究表明,烟管菌BOS55虽然可以降解高达70%的苯并[a]芘,但其矿化率仅为10%左右。真菌转化PAHs主要有细胞色素P450单加氧酶和木质素降解酶两条途径(图 2)[24]。研究表明,非木腐真菌采用P450单加氧酶转化PAHs,而木腐真菌同时拥有P450单加氧酶和木质素降解酶两套体系,在酶系协同作用下可矿化PAHs[30]。细胞色素P450单加氧酶为PAHs引入单分子氧形成环氧化物,通过水解形成反式二氢二醇,该物质再进一步单加氧形成PAHs终极致毒产物二氢二醇环氧化物;同时二氢二醇也可被脱氢酶还原成二醇,通过结合形成O-甲基、葡糖苷、葡糖苷酸和硫酸盐等复合物实现污染物脱毒[31]。木质素降解酶包括漆酶、木质素过氧化物酶和锰过氧化物酶,它们通过单电子攻击形成自由激发态羟基,再氧化PAHs形成醌,有的PAHs-醌可直接开环而被矿化[32]。
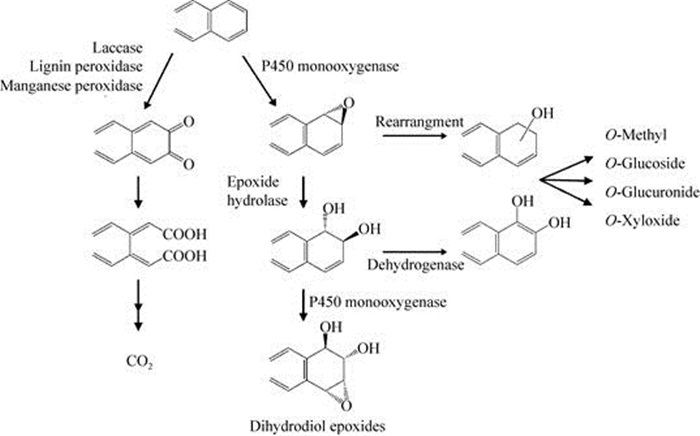 |
| 图 2 真菌对PAHs的主要降解途径[24] Figure 2 Catabolic pathway of polycyclic aromatic hydrocarbons by fungi[24]. |
| 图选项 |
2.2 PAHs降解过程中真菌和细菌的协同作用 真菌和细菌对PAHs的降解特性虽然不同却可以互补,即真菌可以转化高环PAHs,而细菌对中、低环PAHs具有更好的矿化作用,有研究发现二者结合可显著提高高环PAHs的矿化效率。Kotterman等[29]研究表明,单独接种烟管菌BOS55时苯并[a]芘矿化率为10%左右,加入活性污泥后矿化率显著提高至25%。而Boonchan等[33]直接采用了真菌-细菌共培养体降解高环PAHs,发现共培养体能有效去除屈、苯并[a]蒽、二苯并[a, h]蒽和苯并[a]芘等,其中苯并[a]芘矿化率高达53%。研究认为,共培养体系中真菌对高环PAHs发起初始攻击,然后代谢产物由细菌进行后续矿化。
而真菌-细菌协同作用在多介质、多界面、非均一性的土壤环境中更能发挥重要作用[34]。在非饱和多相土壤环境下,PAHs降解细菌自身迁移受到限制,真菌菌丝表面的液膜可以牵引细菌并帮助其在土壤环境中迁移[35],同时真菌菌丝也可吸收PAHs,通过胞质流动转运土壤PAHs给降解细菌[36]。这种协同作用增强了污染物的生物可利用性从而促进其生物降解[37]。相比而言,真菌-细菌对高环PAHs代谢的协同研究却鲜有报道。通过代谢途径可发现它们降解PAHs可能存在某些协同代谢的节点:真菌P450单加氧酶途径产生反式二氢二醇在脱氢酶作用下形成二醇类物质,真菌木质素降解酶途经产生的PAHs-醌类也可由PAHs降解细菌中的o-醌类氧化还原酶转化成PAHs二醇[38],二醇类物质可直接进入细菌代谢途经被矿化。解毒方面,醌的还原减弱了醌类-DNA加合物的形成,而PAHs降解细菌中儿茶酚-O-甲基转移酶,可催化二醇形成甲基化复合物[39],缓解活性氧造成的DNA损伤。同时,已报道的细菌的PAHs-醌开环途径[40],也为真菌-细菌协同代谢过程提供了可能线索。
2.3 PAHs微生物降解过程中的其他协同作用 除了真菌-细菌联合作用,PAHs的微生物代谢普遍存在协同现象,比如微生物菌群降解效果优于单一种类微生物[8]、低环PAHs作为共代谢底物可刺激高环PAHs的微生物降解[41],这些现象都暗示了微生物代谢PAHs所进化出的协同代谢策略。普遍认为,微生物降解PAHs是一个由高环到低环逐级开环并降解的过程,所以较为“经济”的做法,是微生物可能针对不同种类但结构类似的PAHs共享一套代谢途径。研究表明,高环PAHs降解细菌往往能降解其他种类的PAHs[42],而酶学研究也揭示该降解特性与PAHs羟基化双加氧酶的广谱催化特质相关联,PAHs羟基化双加氧酶容纳大分子底物的催化口袋可实现不同种类PAHs的转化作用[43-44]。
目前,PAHs微生物降解的研究也多注重于单个微生物的转化过程,严重缺乏对微生物群体协同代谢方面的认识,而解析协同代谢机制却是对传统认识的重要补充。通过对协同代谢的认识,我们在传统芘降解菌的筛选过程中引入更容易降解的菲,成功获取了多种类的芘降解菌[41],而Wang等[45]采用四氯乙烯代替多氯联苯获取了长期难以研究的多氯联苯厌氧脱氯菌。据调研,真菌-细菌协同代谢和底物共代谢是目前针对苯并[a]芘等难降解PAHs的较为高效的代谢形式,然而其中的协同代谢机制却远未阐明,解析协同过程有望发展和完善难降解PAHs的微生物修复新技术、新理论。
3 土壤中PAHs环境归趋与微生物群落响应 有机污染物进入土壤体系除了微生物降解,还将涉及吸附、结合、再释放等更复杂的物质迁移和转化过程,这些过程综合影响了污染物在土壤中的消减,直接决定污染物环境生态风险。此外,土壤体系的复杂性还体现于土壤微生物降解不同于传统纯培养研究的单一过程,其更多反映为多种群微生物作用下的土壤污染物环境行为。在土壤PAHs转化过程中,微生物直接参与了污染物的降解和转化,影响了污染物在土壤中的吸附结合[46],同时还作为环境敏感因子反馈污染物在土壤中的环境化学行为变化[47]。
3.1 微生物参与土壤PAHs的环境归趋 在一个封闭的土壤微域体系中,一部分PAHs被生物矿化成CO2直接离开土壤,或转变成无毒的生物质组分残留于土壤[48];一部分与土壤腐殖质结合,形成生物可利用性极低的不可提取结合态残留(non-extractable residues,NER)。上述两过程均可实现污染物脱毒[46],而残余的可提取态污染物,因生物有效性较高对土壤生态环境造成毒害产生风险。
微生物在土壤有机污染物的环境归趋过程中发挥重要作用,它不仅主控了污染物的降解矿化,还参与绝大部分NER的形成过程[46]。研究证实,活性土壤(即非灭菌)中的NER形成量远高于灭菌土壤。土壤腐殖质与PAHs结构的π键形成π-π电子供体-受体作用力而增强结合固定,活性土壤中微生物通过对母体化合物基团进行生物修饰,增强了这种共价(酯键、醚键)或非共价(氢键)的结合过程[49]。此外,微生物还促成土壤有机粘土聚合物的形成,通过物理包埋作用增强土壤对有机污染物的固定结合[50],土壤PAHs的环境归趋行为与土壤微生物群落响应存在密切关联[51]。因此,PAHs在土壤中归趋行为不仅受到污染物自身理化性质影响,也与它们的生物有效性直接相关。如菲在土壤中主要通过矿化形成CO2离开土壤,而苯并[a]芘则大部分与土壤有机质结合固定而实现脱毒,所以针对不同污染物的环境归趋行为应采用适宜的微生物强化修复措施,如易降解PAHs可重点发展降解矿化技术,难降解PAHs更需关注固定稳定化方法[17, 52]。
3.2 土壤微生物对PAHs污染的响应 PAHs污染物的消减过程势必引发土壤生态系统反馈,微生物作为“活跃分子”可敏感捕捉到这种细微变化[47]。微生物一方面作为分解者反馈污染物降解过程,导致大量土著降解菌被富集[53],另一方面污染物因生态毒性改变土壤微生物群落结构与功能[54]。区别于传统的化学提取模拟方法,微生物群落对环境污染过程的响应更为真实,特别在生物有效性极低的中、低浓度自然PAHs污染土壤,化学提取法根本无法获取其生物有效态信息,而微生物群落却能够敏感指示污染状况。
据报道,传统生态预测模型中引入微生物参数可实现更精准的土壤生态功能预测[55]。目前,针对自然污染土壤环境的微生物研究不多,相对而言地下水的研究更为深入。研究者初步阐释了地下水污染物(如厌氧脱氯、铀还原等)自然转化过程[56]和修复应用下[57]污染物转化迁移的内在生物学机制。He等[58]研究表明,微生物功能多样性信息可以更好地预测地下水污染状况。土壤环境因高度异质及更低的生物有效性,其研究难度比水体更大。同时,自然农田多属于中、低浓度污染,土壤微生物群落组成分异是在长期污染和消减过程中逐渐进化形成,低剂量、长期污染输入的生态反馈过程揭示更具现实生态意义[59]。
3.3 土壤污染微生物群落与功能解析 组学技术日趋成熟,在土壤复杂体系研究PAHs的微生物降解过程发挥重要作用。功能基因扩增子测序是认识环境PAHs降解关键基因多样性的重要手段[60],宏基因组技术通过大规模DNA鸟枪法测序,结合基因组装配与基因功能识别,针对土壤中所有微生物重建污染物降解途径[61]。宏转录组分析土壤中的RNA组分更具有功能意义,目前已被用于土壤微生物对菲的响应研究[62]。宏蛋白质组将提取的土壤蛋白质经双向电泳分离后进行质谱分析,通过功能注释研究PAHs降解中的酶系[63]。
组学技术往往针对土壤中所有微生物,而土壤降解微生物实际仅占极小部分,稳定同位素探针(stable isotope probing,SIP)方法可帮助更好地聚焦PAHs降解微生物[64]。采用13C-PAHs为底物,土壤中降解微生物利用该底物合成13C标记的DNA、RNA、蛋白质等生物大分子,通过适当的分析手段即可聚焦至目标降解菌。已有研究利用DNA-SIP技术研究了不同土壤中PAHs降解菌的多样性[65],探索了环境条件、修复处理等对降解菌组成的影响[66],并揭示了降解微生物对PAHs的选择性[67]。近来,SIP与组学技术的联用为揭示土壤中PAHs降解菌的功能特征提供了有力保障,如通过SIP-宏基因组/宏转录组对基因功能进行识别[68],利用SIP-宏蛋白质组分析降解转化酶[69]、采用SIP-代谢组鉴定土壤中污染物代谢产物[70],通过这些技术重建出土壤PAHs代谢途径,从而为污染土壤修复提供理论指导。
4 研究展望 微生物技术因独特的技术优势在有机污染土壤修复中应用前景广泛,PAHs疏水性强、生物降解难,其实际修复难度大。PAHs污染土壤微生物修复技术的应用与调控,不仅需要明晰高效的微生物转化代谢机制,更应立足真实环境加深土壤中PAHs微生物转化过程和污染生态认识,这其中涉及环境学、土壤学、生物学、生态学等多学科交叉,其中两个问题值得重点思考与关注。
4.1 阐明难降解PAHs的微生物协同代谢过程与机制 难降解PAHs的微生物协同作用广泛存在,但目前相关认识严重不足,阐明高效PAHs代谢过程是建立微生物修复技术的基础。代谢协同作用可体现在微生物群落协同代谢、真菌-细菌协同代谢、不同污染物降解相互促进代谢等,其中又重点涉及不同代谢途径间的关键节点的研究,包括关键协同微生物类群、节点代谢产物类型与限速酶等,节点信息将为促进微生物协同降解技术的发展、应用与调控奠定理论基础。
4.2 发展土壤体系污染物微生物代谢过程的基础理论 土壤环境中污染物的微生物转化过程更为复杂,无法照搬纯培养体系理论认知,实际修复应用不仅要考虑微生物降解过程,还需考量微生物代谢对污染物吸附结合等的影响,利用同位素示踪技术解析污染物环境归趋行为,对不同污染物发展相匹配的微生物修复技术具有指引作用。
PAHs生物有效性低,其强化技术受限于降解菌的成功定殖从而难以发挥应有作用,破解此难题需加深土壤污染生态学方面的认识。揭示土壤污染物环境化学行为相耦合的微生物作用与响应规律,有助于解析土壤污染降解的关键微生物类群和途径,结合室内短期培育和野外长期效应则能从微观机理和宏观现象等多角度揭示土壤微生物对PAHs污染的响应规律,而蛋白组、代谢组和SIP等现代分子生物学技术的发展为揭示污染物微生物“黑箱”过程与机制提供重要研究手段。
References
| [1] | Chaudhary DK, Kim J. New insights into bioremediation strategies for oil-contaminated soil in cold environments. International Biodeterioration & Biodegradation, 2019, 142: 58-72. |
| [2] | Sharma JK, Gautam RK, Nanekar SV, Weber R, Singh BK, Singh SK, Juwarkar AA. Advances and perspective in bioremediation of polychlorinated biphenyl-contaminated soils. Environmental Science and Pollution Research, 2018, 25(17): 16355-16375. DOI:10.1007/s11356-017-8995-4 |
| [3] | Cycoń M, Mrozik A, Piotrowska-Seget Z. Bioaugmentation as a strategy for the remediation of pesticide-polluted soil:a review. Chemosphere, 2017, 172: 52-71. DOI:10.1016/j.chemosphere.2016.12.129 |
| [4] | Teng Y, Luo YM, Li ZG. Principles and techniques of microbial remediation of polluted soils. Soils, 2007, 39(4): 497-502. (in Chinese) 滕应, 骆永明, 李振高. 污染土壤的微生物修复原理与技术进展. 土壤, 2007, 39(4): 497-502. |
| [5] | Megharaj M, Ramakrishnan B, Venkateswarlu K, Sethunathan N, Naidu R. Bioremediation approaches for organic pollutants:a critical perspective. Environment International, 2011, 37(8): 1362-1375. DOI:10.1016/j.envint.2011.06.003 |
| [6] | Bamforth SM, Singleton I. Bioremediation of polycyclic aromatic hydrocarbons:current knowledge and future directions. Journal of Chemical Technology and Biotechnology, 2005, 80(7): 723-736. DOI:10.1002/jctb.1276 |
| [7] | Kanaly R, Bartha R, Fogel S, Findlay M. Biodegradation of[14C]benzo[a]pyrene added in crude oil to uncontaminated soil. Applied and Environmental Microbiology, 1997, 63(11): 4511-4515. DOI:10.1128/AEM.63.11.4511-4515.1997 |
| [8] | Kanaly RA, Bartha R, Watanabe K, Harayama S. Rapid mineralization of benzo[a]pyrene by a microbial consortium growing on diesel fuel. Applied and Environmental Microbiology, 2000, 66(10): 4205-4211. DOI:10.1128/AEM.66.10.4205-4211.2000 |
| [9] | Han XM, Hu HW, Shi XZ, Zhang LM, He JZ. Effects of different agricultural wastes on the dissipation of PAHs and the PAH-degrading genes in a PAH-contaminated soil. Chemosphere, 2017, 172: 286-293. DOI:10.1016/j.chemosphere.2017.01.012 |
| [10] | Cébron A, Beguiristain T, Bongoua-Devisme J, Denonfoux J, Faure P, Lorgeoux C, Ouvrard S, Parisot N, Peyret P, Leyval C. Impact of clay mineral, wood sawdust or root organic matter on the bacterial and fungal community structures in two aged PAH-contaminated soils. Environmental Science and Pollution Research, 2015, 22(18): 13724-13738. DOI:10.1007/s11356-015-4117-3 |
| [11] | Li XZ, Wu YC, Lin XG, Zhang J, Zeng J. Dissipation of polycyclic aromatic hydrocarbons (PAHs) in soil microcosms amended with mushroom cultivation substrate. Soil Biology and Biochemistry, 2012, 47: 191-197. DOI:10.1016/j.soilbio.2012.01.001 |
| [12] | García-Delgado C, D'Annibale A, Pesciaroli L, Yunta F, Crognale S, Petruccioli M, Eymar E. Implications of polluted soil biostimulation and bioaugmentation with spent mushroom substrate (Agaricus bisporus) on the microbial community and polycyclic aromatic hydrocarbons biodegradation. Science of the Total Environment, 2015, 508: 20-28. DOI:10.1016/j.scitotenv.2014.11.046 |
| [13] | Zhang Y, Miller RM. Effect of rhamnolipid (biosurfactant) structure on solubilization and biodegradation of n-alkanes. Applied and Environmental Microbiology, 1995, 61(6): 2247-2251. DOI:10.1128/AEM.61.6.2247-2251.1995 |
| [14] | Lamberts RF, Johnsen AR, Andersen O, Christensen JH. Univariate and multivariate characterization of heavy fuel oil weathering and biodegradation in soil. Environmental Pollution, 2008, 156(2): 297-305. DOI:10.1016/j.envpol.2008.02.008 |
| [15] | Labana S, Pandey G, Paul D, Sharma NK, Basu A, Jain RK. Pot and field studies on bioremediation of p-nitrophenol contaminated soil using Arthrobacter protophormiae RKJ100. Environmental Science & Technology, 2005, 39(9): 3330-3337. |
| [16] | Wu YC, Teng Y, Li ZG, Liao XW, Luo YM. Potential role of polycyclic aromatic hydrocarbons (PAHs) oxidation by fungal laccase in the remediation of an aged contaminated soil. Soil Biology and Biochemistry, 2008, 40(3): 789-796. DOI:10.1016/j.soilbio.2007.10.013 |
| [17] | Zeng J, Zhu QH, Wu YC, Shan J, Ji R, Lin XG. Oxidation of benzo[a]pyrene by laccase in soil enhances bound residue formation and reduces disturbance to soil bacterial community composition. Environmental Pollution, 2018, 242: 462-469. DOI:10.1016/j.envpol.2018.06.075 |
| [18] | Pazos M, Rosales E, Alcántara T, Gómez J, Sanromán M. Decontamination of soils containing PAHs by electroremediation:a review. Journal of Hazardous Materials, 2010, 177(1/3): 1-11. |
| [19] | Wick LY, Mattle PA, Wattiau P, Harms H. Electrokinetic transport of PAH-degrading bacteria in model aquifers and soil. Environmental Science & Technology, 2004, 38(17): 4596-602. |
| [20] | Niqui-Arroyo JL, Ortega-Calvo JJ. Integrating biodegradation and electroosmosis for the enhanced removal of polycyclic aromatic hydrocarbons from creosote-polluted soils. Journal of Environmental Quality, 2007, 36(5): 1444-1451. DOI:10.2134/jeq2006.0516 |
| [21] | Wang XM, Teng Y, Tu C, Luo YM, Greening C, Zhang N, Dai SX, Ren WJ, Zhao L, Li ZG. Coupling between nitrogen fixation and tetrachlorobiphenyl dechlorination in a rhizobium-legume symbiosis. Environmental Science & Technology, 2018, 52(4): 2217-2224. |
| [22] | Marcet TF, Cápiro NL, Morris LA, Hassan SM, Yang Y, L?ffler FE, Pennell KD. Release of electron donors during thermal treatment of soils. Environmental Science & Technology, 2018, 52(6): 3642-3651. |
| [23] | Marcet TF, Cápiro NL, Yang Y, L?ffler FE, Pennell KD. Impacts of low-temperature thermal treatment on microbial detoxification of tetrachloroethene under continuous flow conditions. Water Research, 2018, 145: 21-29. DOI:10.1016/j.watres.2018.07.076 |
| [24] | Peng RH, Xiong AS, Xue Y, Fu XY, Gao F, Zhao W, Tian YS, Yao QH. Microbial biodegradation of polyaromatic hydrocarbons. FEMS Microbiology Reviews, 2008, 32(6): 927-955. DOI:10.1111/j.1574-6976.2008.00127.x |
| [25] | Zeng J, Lin XG, Zhang J, Li XZ. Isolation of polycyclic aromatic hydrocarbons (PAHs)-degrading Mycobacterium spp. and the degradation in soil. Journal of Hazardous Materials, 2010, 183(1/3): 718-723. |
| [26] | Kim SJ, Kweon O, Jones RC, Edmondson RD, Cerniglia CE. Genomic analysis of polycyclic aromatic hydrocarbon degradation in Mycobacterium vanbaalenii PYR-1. Biodegradation, 2008, 19(6): 859-881. DOI:10.1007/s10532-008-9189-z |
| [27] | Brezna B, Kweon O, Stingley RL, Freeman JP, Khan AA, Polek B, Jones RC, Cerniglia CE. Molecular characterization of cytochrome P450 genes in the polycyclic aromatic hydrocarbon degrading Mycobacterium vanbaalenii PYR-1. Applied Microbiology and Biotechnology, 2006, 71(4): 522-532. |
| [28] | Aranda E. Promising approaches towards biotransformation of polycyclic aromatic hydrocarbons with Ascomycota fungi. Current Opinion in Biotechnology, 2016, 38: 1-8. DOI:10.1016/j.copbio.2015.12.002 |
| [29] | Kotterman MJJ, Vis EH, Field JA. Successive mineralization and detoxification of benzo[a]pyrene by the white rot fungus Bjerkandera sp. strain BOS55 and indigenous microflora. Applied and Environmental Microbiology, 1998, 64(8): 2853-2858. DOI:10.1128/AEM.64.8.2853-2858.1998 |
| [30] | Bezalel L, Hadar Y, Cerniglia CE. Enzymatic mechanisms involved in phenanthrene degradation by the white rot fungus Pleurotus ostreatus. Applied and Environmental Microbiology, 1997, 63(7): 2495-2501. DOI:10.1128/AEM.63.7.2495-2501.1997 |
| [31] | Penning TM, Burczynski ME, Hung CF, McCoull KD, Palackal NT, Tsuruda LS. Dihydrodiol dehydrogenases and polycyclic aromatic hydrocarbon activation:generation of reactive and redox active o-quinones. Chemical Research in Toxicology, 1999, 12(1): 1-18. |
| [32] | Hammel KE, Gai WZ, Green B, Moen MA. Oxidative degradation of phenanthrene by the ligninolytic fungus Phanerochaete chrysosporium. Applied and Environmental Microbiology, 1992, 58(6): 1832-1838. DOI:10.1128/AEM.58.6.1832-1838.1992 |
| [33] | Boonchan S, Britz ML, Stanley GA. Degradation and mineralization of high-molecular-weight polycyclic aromatic hydrocarbons by defined fungal-bacterial cocultures. Applied and Environmental Microbiology, 2000, 66(3): 1007-1019. DOI:10.1128/AEM.66.3.1007-1019.2000 |
| [34] | Worrich A, Stryhanyuk H, Musat N, K?nig S, Banitz T, Centler F, Frank K, Thullner M, Harms, H, Richnow HH, Miltner A, Kastner M, Wick LY. Mycelium-mediated transfer of water and nutrients stimulates bacterial activity in dry and oligotrophic environments. Nature Communications, 2017, 8: 15472. DOI:10.1038/ncomms15472 |
| [35] | Furuno S, P?zolt K, Rabe C, Neu TR, Harms H, Wick LY. Fungal mycelia allow chemotactic dispersal of polycyclic aromatic hydrocarbon-degrading bacteria in water-unsaturated systems. Environmental Microbiology, 2010, 12(6): 1391-1398. |
| [36] | Schamfu? S, Neu TR, Van Der Meer JR, Tecon R, Harms H, Wick LY. Impact of mycelia on the accessibility of fluorene to PAH-degrading bacteria. Environmental Science & Technology, 2013, 47(13): 6908-6915. |
| [37] | Otto S, Banitz T, Thullner M, Harms H, Wick LY. Effects of facilitated bacterial dispersal on the degradation and emission of a desorbing contaminant. Environmental Science & Technology, 2016, 50(12): 6320-6326. |
| [38] | Kim YH, Engesser KH, Cerniglia CE. Two polycyclic aromatic hydrocarbon o-quinone reductases from a pyrene-degrading Mycobacterium. Archives of Biochemistry and Biophysics, 2003, 416(2): 209-217. DOI:10.1016/S0003-9861(03)00297-2 |
| [39] | Zeng J, Lin XG, Zhang J, Zhu H, Chen H, Wong MH. Successive transformation of benzo[a]pyrene by laccase of Trametes versicolor and pyrene-degrading Mycobacterium strains. Applied Microbiology and Biotechnology, 2013, 97(7): 3183-3194. DOI:10.1007/s00253-012-4120-2 |
| [40] | Schuler L, Chadhain SMN, Jouanneau Y, Meyer C, Zylstra GJ, Hols P, Agathos SN. Characterization of a novel angular dioxygenase from fluorene-degrading Sphingomonas sp. strain LB126. Applied and Environmental Microbiology, 2008, 74(4): 1050-1057. DOI:10.1128/AEM.01627-07 |
| [41] | Zeng J, Zhu QH, Li YJ, Dai YL, Wu YC, Sun YH, Miu LY, Chen H, Lin XG. Isolation of diverse pyrene-degrading bacteria via introducing readily utilized phenanthrene. Chemosphere, 2019, 222: 534-540. DOI:10.1016/j.chemosphere.2019.01.189 |
| [42] | Kweon O, Kim SJ, Holland RD, Chen HY, Kim DW, Gao Y, Yu LR, Baek S, Baek DH, Ahn H, Cerniglia CE. Polycyclic aromatic hydrocarbon metabolic network in Mycobacterium vanbaalenii PYR-1. Journal of Bacteriology, 2011, 193(17): 4326-4337. DOI:10.1128/JB.00215-11 |
| [43] | Zeng J, Zhu QH, Wu YC, Chen H, Lin XG. Characterization of a polycyclic aromatic ring-hydroxylation dioxygenase from Mycobacterium sp. NJS-P. Chemosphere, 2017, 185: 67-74. DOI:10.1016/j.chemosphere.2017.07.001 |
| [44] | Kweon O, Kim SJ, Kim DW, Kim JM, Kim HL, Ahn Y, Sutherland JB, Cerniglia CE. Pleiotropic and epistatic behavior of a ring-hydroxylating oxygenase system in the polycyclic aromatic hydrocarbon metabolic network from Mycobacterium vanbaalenii PYR-1. Journal of Bacteriology, 2014, 196(19): 3503-3515. DOI:10.1128/JB.01945-14 |
| [45] | Wang SQ, Chng KR, Wilm A, Zhao SY, Yang KL, Nagarajan N, He JZ. Genomic characterization of three unique Dehalococcoides that respire on persistent polychlorinated biphenyls. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(33): 12103-12108. DOI:10.1073/pnas.1404845111 |
| [46] | K?stner M, Nowak KM, Miltner A, Sch?ffer A. (Multiple) isotope probing approaches to trace the fate of environmental chemicals and the formation of non-extractable 'bound' residues. Current Opinion in Biotechnology, 2016, 41: 73-82. DOI:10.1016/j.copbio.2016.05.002 |
| [47] | Smith MB, Rocha AM, Smillie CS, Olesen SW, Paradis C, Wu LY, Campbell JH, Fortney JL, Mehlhorn TL, Lowe KA, Earles JE, Phillips J, Techtmann SM, Joyner DC, Elias DA, Bailey KL, Hurt RA, Preheim SP, Sanders MC, Yang J, Mueller MA, Brooks S, Watson DB, Zhang P, He ZL, Dubinsky EA, Adams PD, Arkin AP, Fields MW, Zhou JZ, Alm EJ, Hazen TC. Natural bacterial communities serve as quantitative geochemical biosensors. mBio, 2015, 6(3): e00326-15 |
| [48] | Trapp S, Brock AL, Nowak K, K?stner M. Prediction of the formation of biogenic nonextractable residues during degradation of environmental chemicals from biomass yields. Environmental Science & Technology, 2018, 52(2): 663-672. |
| [49] | Mitchell PJ, Simpson MJ. High affinity sorption domains in soil are blocked by polar soil organic matter components. Environmental Science & Technology, 2013, 47(1): 412-419. |
| [50] | Gevao B, Semple KT, Jones KC. Bound pesticide residues in soils:a review. Environmental Pollution, 2000, 108(1): 3-14. |
| [51] | Zhu QH, Wu YC, Zeng J, Wang XX, Zhang TL, Lin XG. Influence of bacterial community composition and soil factors on the fate of phenanthrene and benzo[a]pyrene in three contrasting farmland soils. Environmental Pollution, 2019, 247: 229-237. DOI:10.1016/j.envpol.2018.12.079 |
| [52] | Wu YC, Ding QM, Zhu QH, Zeng J, Ji R, Dumont MG, Lin XG. Contributions of ryegrass, lignin and rhamnolipid to polycyclic aromatic hydrocarbon dissipation in an arable soil. Soil Biology and Biochemistry, 2018, 118: 27-34. DOI:10.1016/j.soilbio.2017.11.022 |
| [53] | Hazen TC, Dubinsky EA, DeSantis TZ, Andersen GL, Piceno YM, Singh N, Jansson JK, Probst A, Borglin SE, Fortney JL, Stringfellow WT, Bill M, Conrad ME, Tom LM, Chavarria KL, Alusi TR, Lamendella R, Joyner DC, Spier C, Baelum J, Auer M, Zemla ML, Chakraborty R, Sonnenthal EL, D'Haeseleer P, Holman HYN, Osman S, Lu ZM, Van Nostrand JD, Deng Y, Zhou JZ, Mason OU. Deep-sea oil plume enriches indigenous oil-degrading bacteria. Science, 2010, 330(6001): 204-208. DOI:10.1126/science.1195979 |
| [54] | Mason OU, Scott NM, Gonzalez A, Robbins-Pianka A, Baelum J, Kimbrel J, Bouskill NJ, Prestat E, Borglin S, Joyner DC, Fortney JL, Jurelevicius D, Stringfellow WT, Alvarez-Cohen L, Hazen TC, Knight R, Gilbert JA, Jansson JK. Metagenomics reveals sediment microbial community response to deepwater horizon oil spill. The ISME Journal, 2014, 8(7): 1464-1475. DOI:10.1038/ismej.2013.254 |
| [55] | Miki T, Ushio M, Fukui S, Kondoh M. Functional diversity of microbial decomposers facilitates plant coexistence in a plant-microbe-soil feedback model. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(32): 14251-14256. DOI:10.1073/pnas.0914281107 |
| [56] | Flynn TM, Sanford R, Ryu H, Bethke CM, Levine AD, Ashbolt NJ, Domingo JWS. Functional microbial diversity explains groundwater chemistry in a pristine aquifer. BMC Microbiology, 2013, 13: 146. DOI:10.1186/1471-2180-13-146 |
| [57] | Van Nostrand JD, Wu LY, Wu WM, Huang ZJ, Gentry TJ, Deng Y, Carley J, Carroll S, He ZL, Gu BH, Luo J, Criddle CS, Watson DB, Jardine PM, Marsh TL, Tiedje JM, Hazen TC, Zhou JZ. Dynamics of microbial community composition and function during in situ bioremediation of a uranium-contaminated aquifer. Applied and Environmental Microbiology, 2011, 77(11): 3860-3869. DOI:10.1128/AEM.01981-10 |
| [58] | He ZL, Zhang P, Wu LW, Rocha AM, Tu QC, Shi Z, Wu B, Qin YJ, Wang JJ, Yan QY, Curtis D, Ning DL, van Nostrand JD, Wu LY, Yang YF, Elias DA, Watson DB, Adams MWW, Fields MW, Alm EJ, Hazen TC, Adams PD, Arkin AP, Zhou JZ. Microbial functional gene diversity predicts groundwater contamination and ecosystem functioning. mBio, 2018, 9(1): e02435-17. DOI:10.1128/mBio.02435-17 |
| [59] | Clark CM, Cleland EE, Collins SL, Fargione JE, Gough L, Gross KL, Pennings SC, Suding KN, Grace JB. Environmental and plant community determinants of species loss following nitrogen enrichment. Ecology Letters, 2007, 10(7): 596-607. DOI:10.1111/j.1461-0248.2007.01053.x |
| [60] | Liang CY, Huang Y, Wang Y, Ye QH, Zhang ZT, Wang H. Distribution of bacterial polycyclic aromatic hydrocarbon (PAH) ring-hydroxylating dioxygenases genes in oilfield soils and mangrove sediments explored by gene-targeted metagenomics. Applied Microbiology and Biotechnology, 2019, 103(5): 2427-2440. DOI:10.1007/s00253-018-09613-x |
| [61] | Zhang SF, Hu Z, Wang H. Metagenomic analysis exhibited the co-metabolism of polycyclic aromatic hydrocarbons by bacterial community from estuarine sediment. Environment International, 2019, 129: 308-319. DOI:10.1016/j.envint.2019.05.028 |
| [62] | de Menezes A, Clipson N, Doyle E. Comparative metatranscriptomics reveals widespread community responses during phenanthrene degradation in soil. Environmental Microbiology, 2012, 14(9): 2577-2588. DOI:10.1111/j.1462-2920.2012.02781.x |
| [63] | Bastida F, Jehmlich N, Lima K, Morris BEL, Richnow HH, Hernández T, Von Bergen M, García C. The ecological and physiological responses of the microbial community from a semiarid soil to hydrocarbon contamination and its bioremediation using compost amendment. Journal of Proteomics, 2016, 135: 162-169. DOI:10.1016/j.jprot.2015.07.023 |
| [64] | Dumont MG, Murrell JC. Stable isotope probing-linking microbial identity to function. Nature Reviews Microbiology, 2005, 3(6): 499-504. DOI:10.1038/nrmicro1162 |
| [65] | Jones MD, Crandell DW, Singleton DR, Aitken MD. Stable-isotope probing of the polycyclic aromatic hydrocarbon-degrading bacterial guild in a contaminated soil. Environmental Microbiology, 2011, 13(10): 2623-2632. DOI:10.1111/j.1462-2920.2011.02501.x |
| [66] | Jones MD, Singleton DR, Carstensen DP, Powell SN, Swanson JS, Pfaender FK, Aitken MD. Effect of incubation conditions on the enrichment of pyrene-degrading bacteria identified by stable-isotope probing in an aged, PAH-contaminated soil. Microbial Ecology, 2008, 56(2): 341-349. DOI:10.1007/s00248-007-9352-9 |
| [67] | Rodgers-Vieira EA, Zhang ZF, Adrion AC, Gold A, Aitken MD. Identification of anthraquinone-degrading bacteria in soil contaminated with polycyclic aromatic hydrocarbons. Applied and Environmental Microbiology, 2015, 81(11): 3775-3781. DOI:10.1128/AEM.00033-15 |
| [68] | Thomas F, Corre E, Cébron A. Stable isotope probing and metagenomics highlight the effect of plants on uncultured phenanthrene-degrading bacterial consortium in polluted soil. The ISME Journal, 2019, 13(7): 1814-1830. DOI:10.1038/s41396-019-0394-z |
| [69] | Herbst FA, Bahr A, Duarte M, Pieper DH, Richnow HH, Von Bergen M, Seifert J, Bombach P. Elucidation of in situ polycyclic aromatic hydrocarbon degradation by functional metaproteomics (protein-SIP). Proteomics, 2013, 13(18/19): 2910-2920. |
| [70] | Tian ZY, Vila J, Yu M, Bodnar W, Aitken MD. Tracing the biotransformation of polycyclic aromatic hydrocarbons in contaminated soil using stable isotope-assisted metabolomics. Environmental Science & Technology Letters, 2018, 5(2): 103-109. |
