刘艳如

 , 李鑫, 董盼盼, 黄建忠, 舒正玉
, 李鑫, 董盼盼, 黄建忠, 舒正玉

福建师范大学生命科学学院, 工业微生物发酵技术国家地方联合工程研究中心, 教育部工业微生物工程中心, 福建 福州 350108
收稿日期:2018-10-08;修回日期:2019-01-25;网络出版日期:2019-02-27
基金项目:国家自然科学基金项目(31370802,31870787);福建省科技厅重点项目(2013H0021);福建省自然科学基金项目(2017J01441)
*通信作者:刘艳如, Tel/Fax:+86-591-22868212, E-mail:yrliu@fjnu.edu.cn;
舒正玉, Tel/Fax:+86-591-22868212, E-mail:shuzhengyu@fjnu.edu.cn.
摘要:[目的] 中温伯克霍尔德菌胞外脂肪酶LipA在工业领域具有重要的应用价值。利用蛋白质工程技术来提高其热稳定性,对开发脂肪酶LipA酶制剂及提高其应用范围及应用效果,具有重要的意义。[方法] 利用生物信息学软件Castp、Voronoia和Cave分析LipA分子中存在的空腔及其组成氨基酸残基;利用FoldX软件构建上述氨基酸残基的突变体电子文库,并基于空腔效应(体积变小)、自由能变化值(降低)和空间结构特点等对前述突变体电子文库进行筛选。从突变体电子文库中选择具有代表性的突变体,通过基因工程技术,引入突变。经诱导表达后,实验验证并筛选出热稳定性的突变体。[结果] 构建了一个由58个突变体组成的电子文库;并对其中17个代表性的突变体进行了实验验证;筛选到2个热稳定性有明显提高的突变体LipA-His15Pro和LipA-Ala210Val;其叠加突变体LipA-His15Pro/Ala210Val的T5012较野生型LipA提高了8℃,在55℃下的半衰期较野生型脂肪酶LipA提高了23.1倍。[结论] 基于空腔填充技术构建热稳定性伯克霍尔德菌胞外脂肪酶LipA突变体,是一种行之有效的策略。
关键词:伯克霍尔德菌脂肪酶LipA空腔填充效应T5012半衰期
Effect of cavity-filling mutations on thermostability of lipase LipA from Burkholderia sp.
Yanru Liu

 , Xin Li, Panpan Dong, Jianzhong Huang, Zhengyu Shu
, Xin Li, Panpan Dong, Jianzhong Huang, Zhengyu Shu

National & Local United Engineering Research Center of Industrial Microbiology and Fermentation Technology, Engineering Research Center of Industrial Microbiology, Ministry of Education; College of Life Sciences, Fujian Normal University, Fuzhou 350108, Fujian Province, China
Received: 8 October 2018; Revised: 25 January 2019; Published online: 27 February 2019
*Corresponding author: Liu Yanru, Tel/Fax:+86-591-22868212, E-mail:yrliu@fjnu.edu.cn;
Shu Zhengyu, Tel/Fax:+86-591-22868212, E-mail:shuzhengyu@fjnu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31370802 and 31870787), by the Key Project from Science and Technology Bureau of Fujian Province (2013H0021) and by the Natural Science Funds of Fujian Province (2017J01441)
Abstract: [Objective] To improve the thermostability of lipase LipA from Burkholderia sp., a series of cavity-filled lipase LipA mutants were constructed and evaluated. [Methods] Amino acid residuals consisted of cavities in 3D structural model of lipase LipA were predicted using Castp, Voronoia and Cave, and then computational libraries of every amino acid residuals were constructed using software packages FoldX. The following types of mutants were excluded from the computational libraries:(1) volume of the cavity was increased; (2) value of free energy, ΔΔG, was over -0.5 kcal/mol. Total 58 mutants were screened from the computational libraries, and 17 mutants were selected to verify the mutation effect. [Results] Thermostability of lipase mutants (LipA-His15Pro and LipA-Ala210Val) were improved and T5012 value of lipase LipA-His15Pro and lipase LipA-Ala210Val increased by 4 ℃ and 2 ℃, respectively. Thermostability of the superimposing mutant lipase LipA-His15Pro/Ala210Val was further improved and the half-life (t1/2) at 55 ℃ increased by 23.1 times. [Conclusion] Cavity-filling mutation was a feasible technique to improve the thermostability of lipase LipA from Burkholderia sp.
Keywords: Burkholderia sp.lipase LipAcavity-filling mutationT5012half-life
脂肪酶(lipase,EC3.1.1.3)又称为三酰甘油酯水解酶,具有独特的油水界面激活的催化特性。脂肪酶除了可以催化酯的水解反应外,还可以催化酯的合成、转酯化、酯交换、醇解和氨解等反应类型[1]。微生物脂肪酶催化的各类反应,具有高效的专一性,严谨的对映体选择性、位点选择性或链长选择性等优点,已被广泛应用于食品加工、手性化合物合成、洗涤剂、造纸、废水处理及生物柴油酶法合成等领域[2]。天然微生物脂肪酶欠佳的热稳定性,一定程度上限制了其在工业领域的规模化应用。为了克服这一障碍,化学修饰技术、固定化技术和蛋白质工程改造技术等均被应用于酶蛋白热稳定性的改良[3]。近年来,随着生物信息学、计算化学和结构生物学的快速发展,蛋白质工程改造技术正被越来越多的研究人员应用于酶蛋白的分子改良,并取得显著效果。
利用蛋白质工程技术改良酶蛋白的热稳定性主要有两种策略:理性设计(或半理性设计)和定向进化。过去20年中,利用定向进化策略已成功改良各种酶蛋白,获得了系列优良的热稳定性酶蛋白突变体[4]。但定向进化技术依赖对庞大突变文库进行高通量筛选,方可获得理想突变体,或需要耗费大量的人力物力资源,或依赖酶标仪等高端设备及高通量筛选技术。近年来,随着越来越多的蛋白质3D结构被解析,结合越来越成熟的生物信息学的手段,基于理性设计(或半理性设计)策略,构建小型smart突变文库,快速获得理想的热稳定性突变体蛋白质分子,获得了突破性的进展。如Floor等以Sphingomonas paucimobilis卤代烷脱盐酶(haloalkane dehalogenase) LinB为研究对象,从150个突变体的小型文库中筛选到半衰期延长了200多倍的阳性突变体[5];Wijma等以Rhodococcus erythropolis柠檬烯-1, 2-环氧化物水解酶(limonene-1, 2-epoxide hydrolase)为研究对象,从包含64个突变体的小型文库中,筛选到21个热稳定提高的突变体。其中最优热稳定性突变体,表观熔点温度(apparent melting temperature)从50 ℃提高到85 ℃,半衰期提高了250多倍[6]。
蛋白质分子的空腔(cavity)是由于组成蛋白质的氨基酸在天然状态下包装(packing)不够致密而在蛋白分子表面或内部形成的空洞[7-8]。不同位置的空腔对蛋白质的性质和功能的影响不同,如:蛋白质结构域与结构域之间的空腔,对于结构域之间的相对运动和构象变化具有重要的功能;而蛋白质内部的空腔,可能影响到蛋白质的稳定性和催化活性[9-10]。对蛋白质分子的空腔进行改造,可显著提高其温度稳定性。如对Bacillus circulans木聚糖酶(xylanase)分子表面的空腔进行理性设计,获得的突变体热稳定性(t1/2)提高了15倍[11];而对Bacillus stearothermophilus普鲁兰酶(neopullulanase)分子内的空腔进行理性设计,获得的突变体热稳定性(t1/2)提高了13倍[12]。
本课题组制备的中温Burkholderia sp. ZYB002脂肪酶LipA在造纸等领域具有潜在的应用价值[13-14]。为进一步提高其应用效果,本课题组基于B-factor筛选突变位点[15]和理性设计盐桥等策略[16],显著提高了Burkholderia sp.脂肪酶LipA的热稳定性,突变体LipA-Phe221Asp/Asn125Glu的T5012较野生型LipA提高了11.1 ℃,t1/2延长了49.6倍。本论文报道了基于空腔填充效应,通过理性设计策略,成功改良脂肪酶LipA分子的热稳定性实验结果。
1 材料和方法 1.1 材料
1.1.1 菌株与载体: Burkholderia sp. ZYB002菌株胞外脂肪酶LipA及其伴侣蛋白LipB的编码基因均由本实验室克隆并提交NCBI核酸数据库(登录号为:EU768869)。表达载体pACYC-lipA/lipB及重组菌株E. coli BL21(DE3)-pACYC-lipA/lipB均由本实验室构建并保存。
1.1.2 工具酶、引物及试剂: PrimeSTAR HS DNA Polymerase、Quick Dpn I内切酶及标准分子量的DNA Marker均购于宝生物工程(大连)有限公司;Wizard SV Gel和PCR Clean-Up System等试剂盒购于普洛麦格(北京)生物技术有限公司;4-硝基苯月桂酸酯(p-nitripheny laurate,pNPL)购于Sigma (美国)公司;氯霉素购于鼎国生物技术有限公司(北京);其他试剂均为分析纯。本实验使用的寡核苷酸引物对由生工生物工程(上海)股份有限公司合成,具体序列如表 1。
表 1. 本实验所用的PCR引物对 Table 1. Pairs of the primers used in this study
| Primers | Mutation sites | Oligonucleotide sequence (5′→3′) | Tm/℃ | Annealing temperature/℃ |
| BF1F | LipA-H15P | ATCATCCTCGTTCCGGGGCTCTCG | 65.8 | 62 |
| BF1R | GGTACCCGAGAGCCCCGGAACGAG | 69.0 | ||
| BF2F | LipA-D21M | GGCTCTCGGGTACCATGAAGTACG | 61.9 | 56 |
| BF2R | GCCCGCGTACTTCATGGTACC | 61.5 | ||
| BF3F | LipA-D21V | GGGCTCTCGGGTACCGTTAAGTAC | 61.7 | 55 |
| BF3R | CGCGTACTTAACGGTACCCGAG | 60.4 | ||
| BF4F | LipA-D21R | GGGCTCTCGGGTACCCGTAAGTAC | 63.8 | 58 |
| BF4R | CCGCGTACTTACGGGTACCCG | 63.4 | ||
| BF5F | LipA-D36I | ACGGCATCCAGGAGATTCTGCAAC | 62.1 | 56 |
| BF5R | CCGTTCTGTTGCAGAATCTCCTGG | 60.5 | ||
| BF6F | LipA-E63Y | CGCGGCTATCAGTTGCTCGCTTAC | 63.2 | 58 |
| BF6R | AACTGATAGCCGCGCCCGTTC | 63.6 | ||
| BF7F | LipA-N82L | GGCGACCAAGGTGCTGCTCGTC | 66.8 | 62 |
| BF7R | GTGGCCGACGAGCAGCACCTTG | 67.0 | ||
| BF8F | LipA-V84I | GACCAAGGTGAATCTCATTGGCCAC | 62.2 | 57 |
| BF8R | CCTGGCTGTGGCCAATGAGATTC | 63.0 | ||
| BF9F | LipA-A210V | CCTGCTGTATTCGTGGGTTGGCAC | 63.4 | 58 |
| BF9R | ATCGCCGTGCCAACCCACGAATAC | 64.9 | ||
| BF10F | LipA-A210I | CCTGCTGTATTCGTGGATTGGCAC | 61.0 | 56 |
| BF10R | ATCGCCGTGCCAATCCACGAATAC | 62.4 | ||
| BE11F | LipA-A210L | CCTGCTGTATTCGTGGCTGGGCAC | 65.8 | 60 |
| BF11R | ATCGCCGTGCCCAGCCACGAATAC | 67.2 | ||
| BF12F | LipA-A210C | CCTGCTGTATTCGTGGTGTGGCAC | 63.4 | 58 |
| BF12R | ATCGCCGTGCCACACCACGAATAC | 64.8 | ||
| BF13F | LipA-A210M | CCTGCTGTATTCGTGGATGGGCAC | 63.1 | 58 |
| BF13R | ATCGCCGTGCCCATCCACGAATAC | 64.5 | ||
| BF14F | LipA-T280L | AAGGTGCTGAGCCTGAGCTACAAG | 61.6 | 56 |
| BF14R | TTCCACTTGTAGCTCAGGCTCAGC | 61.6 | ||
| BF15F | LipA-N285V | ACGAGCTACAAGTGGGTTCACCTC | 61.3 | 55 |
| BF15R | TCGTCGAGGTGAACCCACTTGTAG | 60.9 | ||
| BF16F | LipA-E289V | ACCACCTCGACGTTATCAACCAGC | 61.7 | 55 |
| BF16R | GAGCAGCTGGTTGATAACGTCGAG | 60.3 | ||
| BF17F | LipA-V296L | CAACCAGCTGCTCGGCCTGCGCGG | 73.6 | 63 |
| BF17R | ATACGCGCCGCGCAGGCCGAGCAG | 74.6 |
表选项
1.2 热稳定性脂肪酶LipA突变体电子文库的构建及筛选 利用生物信息学构建热稳定性脂肪酶LipA突变体电子文库的基本步骤如下:(1)使用Castp[17]、Voronoia[18]和Cave[19]软件预测出LipA 3D分子结构中存在的空腔数量及其对应的氨基酸残基组成;(2)分析并筛选出上述3个软件预测结果中,脂肪酶LipA的空腔及其氨基酸残基组成的交集;并利用FoldX[20]构建上述氨基酸残基的突变体电子文库;(3)筛选出电子文库中自由能变化小于-0.5 kcal/mol的突变体;并进一步利用Castp筛选出突变后空腔体积变小的突变体,最终获得的结构合理的突变体构成突变电子文库[5, 21];(4)从上述预测的各个空腔中,选择有代表性的突变体,利用基因工程技术引入突变;经诱导表达后,测定每个突变体的突变效应,进而评估空腔效应;(5)进一步构建并筛选阳性突变体所在空腔的其他突变体。
1.3 脂肪酶lipA突变文库的构建 利用表 1的引物对,以质粒pACYC-lipA/lipB为模板,通过滚环PCR扩增引入突变。PCR扩增程序为:95 ℃ 5 min;95 ℃,30 s;不同引物对的退火温度参考表 1,退火时间30 s;72 ℃延伸6 min;25个循环;72 ℃延伸7 min。利用Dpn I内切酶37 ℃下酶切PCR扩增产物6 h。酶切产物经纯化回收后热击转化E. coli BL21(DE3)。突变基因经测序验证。
1.4 脂肪酶lipA及其突变体基因的诱导表达及粗酶液的制备 脂肪酶LipA及突变体LipA粗酶液的制备参照刘艳如等(2017)[16]。测序正确的转化子利用LB液体培养基培养至OD600达到0.6-0.9后,加入终浓度为1 mmol/L的IPTG,30 ℃诱导培养16 h。离心收集菌体,经超声裂解后,再次离心收集裂解上清液作为粗酶液。
1.5 蛋白浓度测定 采用Bradfored法测定野生型LipA与突变体LipA粗酶液的蛋白浓度[22]。
1.6 脂肪酶酶活的测定 脂肪酶酶活的测定采用比色法[23]。酶学反应体系如下:20 mmol/L pH 7.4的Na2HPO4-NaH2PO4缓冲体系,终浓度为0.33 mmol/L的4-硝基苯月桂酸酯,加入合适稀释度的脂肪酶酶液30 μL,40 ℃反应5 min。记录410 nm下反应体系吸光度的变化值(在上述反应条件下,ε410nm为0.0131 L/(μmol·cm))。在上述的条件下,每分钟释放1 μmol对硝基苯酚所需要的酶量定义为一个酶活单位(U)。
1.7 热稳定性提高的脂肪酶突变体的初筛 脂肪酶热稳定性的测定参照刘艳如[16]等。调整野生型脂肪酶LipA和突变体脂肪酶LipA粗酶液到相同的蛋白质浓度;55 ℃水浴12 min后迅速置于冰上冷却。室温下静置30 min后,分别测定野生型脂肪酶LipA及突变体脂肪酶LipA的残余酶活。筛选出残余酶活较野生型脂肪酶LipA有提高的突变体脂肪酶LipA。
1.8 脂肪酶及其突变体T5012和t1/2的测定 T5012和t1/2测定参考Zhao和Arnold[24]。
T5012的测定:将脂肪酶LipA(或突变体)置于45-70 ℃热处理12 min,迅速水浴降至室温。依次测定脂肪酶最适反应条件下的酶活,计算出残余酶活为未处理时酶活的50%时所对应的温度。
t1/2的测定:在pH 7.4的条件下,将脂肪酶于55 ℃水浴处理,不同处理时间分别测定脂肪酶残余酶活。当脂肪酶残余酶活降至未处理脂肪酶酶活50%时对应的处理时间即为脂肪酶的半衰期。
1.9 分子动力学模拟 利用Gromacs 5.1.4软件对脂肪酶LipA及突变体LipA-His15Pro/Ala210Val在55 ℃下进行分子动力学模拟,分析其RMSD值的变化趋势。LipA及突变体LipA-His15Pro/Ala210Val催化三联体的His均质子化处理。在CHARMM27力场下,向含平衡TIP3P水分子的六面体盒子内,添加Na+平衡体系的电荷,以达到电中性。体系与盒子边的最小距离为0.9 nm。采用最陡下降法(steepest descents)对系统进行能量最小化,用NVT和NPT对系统进行平衡,使其系统温度为328 K,压力为1.05 bar。在此条件下进行时长为40 ns的分子动力学模拟。
2 结果和分析 2.1 脂肪酶LipA 3D结构中存在的空腔及组成空腔的氨基酸残基分析 利用Castp、Voronoia和Cave软件预测脂肪酶LipA的三维结构中存在的空腔数目分别为35个、20个和24个。3个软件均预测出的空腔(共12个)及其氨基酸残基组成如表 2。从表 2中可以看出,不同软件预测出的每个空腔的氨基酸残基的组成存在一定的差异。
表 2. 脂肪酶LipA 3D分子结构中,经Castp、Voronoia和Cave均预测出的空腔及其氨基酸残基组成 Table 2. Predicted cavities in the 3D structure of LipA using Castp, Voronoia and Cave, respectively
| Cavity number | Cavity-lining residues | Castp | Voronoia | Cave | Cavity number | Cavity-lining residues | Castp | Voronoia | Cave | |
| 1 | Pro113 | + | + | + | 7 | Gln292 | + | + | + | |
| Ser117 | + | + | + | Gly295 | + | + | + | |||
| Leu167 | + | + | + | Val296 | + | + | + | |||
| Leu266 | + | + | + | Arg297 | + | + | + | |||
| 2 | Asp55 | + | + | + | Pro304 | + | + | + | ||
| Asp56 | + | + | + | 8 | Ile110 | + | + | + | ||
| Gly57 | + | + | + | Trp209 | + | + | + | |||
| Gly60 | - | - | + | Trp284 | + | + | + | |||
| Glu63 | + | + | + | Glu289 | + | + | + | |||
| Tyr95 | - | + | + | Pro304 | + | + | + | |||
| 3 | Ala213 | - | - | + | Val307 | + | + | + | ||
| Ile214 | + | + | + | Ile308 | + | + | + | |||
| Asp228 | + | + | + | 9 | Thr112 | - | - | + | ||
| Ser230 | - | - | + | Ser208 | + | + | + | |||
| Thr245 | + | + | + | Trp209 | + | + | + | |||
| Leu248 | + | + | + | Ala210 | + | + | + | |||
| Phe249 | - | - | + | Ala272 | + | + | + | |||
| Asn285 | + | + | + | Leu273 | + | + | + | |||
| 4 | Asn82 | + | + | + | Val277 | + | + | + | ||
| Leu83 | + | + | + | Thr280 | + | + | + | |||
| Val84 | + | + | + | 10 | Ile33 | + | + | + | ||
| Ser106 | + | + | + | Asp36 | + | + | + | |||
| Thr108 | + | + | + | Leu37 | + | + | + | |||
| Leu315 | + | - | + | Val305 | + | + | + | |||
| 5 | Leu13 | - | + | + | Ile308 | + | + | + | ||
| His15 | + | + | + | Arg309 | + | + | + | |||
| Thr20 | + | + | + | 11 | Ser230 | + | + | + | ||
| Asp21 | + | + | + | Thr231 | - | - | + | |||
| Tyr29 | + | + | + | Asp242 | + | + | + | |||
| Trp30 | + | + | + | Ser244 | + | + | + | |||
| Val46 | + | - | + | Thr245 | + | + | + | |||
| Ala47 | - | - | + | Asn285 | + | + | + | |||
| 6 | Gly111 | + | + | + | Leu287 | + | + | + | ||
| Ala210 | + | + | + | 12 | Ile11 | + | + | + | ||
| Gly211 | + | + | + | Leu13 | + | + | + | |||
| Asn263 | + | + | + | Leu37 | + | + | + | |||
| Asp264 | + | + | + | Val84 | + | + | + | |||
| Val267 | + | + | + | Ile110 | + | + | + | |||
| Ser268 | + | + | + | Trp209 | + | + | + | |||
| Lys269 | + | + | + | Ile308 | + | + | + | |||
| Ala272 | + | + | + | His311 | + | + | + | |||
| +: Amino acids found in the predicted cavity; -: No appearance of amino acids in the predicted cavity. | ||||||||||
表选项
2.2 脂肪酶LipA突变体电子文库的构建 对上述3个软件均预测为组成空洞的氨基酸残基,利用FoldX软件,构建并筛选上述氨基酸残基的突变体电子文库(筛选标准:ΔΔG<-0.5 kcal/mol作为热稳定性提高的阳性突变体),共获得68个阳性突变体;对前述68个突变体的空腔效应利用Castp评估后,筛除10个空腔体积增大的突变体,最终获得58个符合预期目标的突变体,组成脂肪酶LipA的突变体电子文库(表 3)。
表 3. 计算机辅助设计的脂肪酶LipA的突变体电子文库 Table 3. Computationally designed libraries for rapid lipase LipA stabilization
| Mutation site | ΔΔG/(kcal/mol) | Cavity volume changes/?3 | Mutation site | ΔΔG/(kcal/mol) | Cavity volume changes/?3 |
| LipA-His15Pro | -2.027 | -3.2 | LipA-Val277Leu | -0.804 | -40.3 |
| LipA-Asp21Val | -0.573 | -18.2 | LipA-Thr280Leu | -2.324 | -25.5 |
| LipA-Asp21Met | -0.823 | -18.2 | LipA-Thr280Val | -1.871 | -24.4 |
| LipA-Asp21Arg | -1.061 | -18.2 | LipA-Thr280Phe | -0.628 | -19.9 |
| LipA-Asp36Met | -0.656 | -40.3 | LipA-Thr280Met | -1.230 | -25.8 |
| LipA-Asp36Ile | -0.661 | -40.3 | LipA-Asn285Val | -0.537 | -21.3 |
| LipA-Asp55Val | -1.113 | -12.6 | LipA-Asn285Ile | -1.102 | -21.3 |
| LipA-Asp55Ile | -1.548 | -12.6 | LipA-Glu289Val | -2.209 | -7 |
| LipA-Asp56Met | -1.419 | -12.2 | LipA-Glu289Pro | -1.328 | -14.2 |
| LipA-Glu63Pro | -0.927 | -12.2 | LipA-Glu289Lys | -2.169 | -18.9 |
| LipA-Glu63Tyr | -0.581 | -12.2 | LipA-Glu289Ala | -1.269 | -0.6 |
| LipA-Asn82Leu | -0.962 | -22.5 | LipA-Glu289Gln | -2.370 | -13.9 |
| LipA-Val84Ile | -0.617 | -28.7 | LipA-Glu289Thr | -0.843 | -21.2 |
| LipA-Ser106Ala | -1.050 | -5.3 | LipA-Gln292Met | -1.265 | -10.7 |
| LipA-Ser106Ile | -0.943 | -22.5 | LipA-Val296Tyr | -0.618 | -12.4 |
| LipA-Ser106Cys | -1.050 | -22.5 | LipA-Gln292Leu | -1.577 | -12.8 |
| LipA-Ser106Asn | -0.825 | -5.3 | LipA-Val296Trp | -0.688 | -43 |
| LipA-Thr108Leu | -1.566 | -22.5 | LipA-Val296Phe | -0.766 | -17.7 |
| LipA-Ala210Val | -1.043 | -16.1 | LipA-Val296Met | -1.008 | -15.7 |
| LipA-Ala210Ile | -1.620 | -54.1 | LipA-Val296Lys | -0.567 | -10.1 |
| LipA-Ala210Leu | -1.156 | -54.1 | LipA-Val296Leu | -0.848 | -17 |
| LipA-Ala210Met | -0.659 | -54.1 | LipA-Val296Ile | -0.655 | -56.4 |
| LipA-Ala210Cys | -0.577 | -54.1 | LipA-Arg309Leu | -1.5157 | -40.3 |
| LipA-Asp228Phe | -1.641 | -21.3 | LipA-Arg309Ile | -0.597 | -20.8 |
| LipA-Asp228Leu | -1.746 | -21.3 | LipA-Val305Leu | -0.524 | -14.8 |
| LipA-Asp228Met | -1.838 | -8.1 | LipA-His311Phe | -1.506 | -9.2 |
| LipA-Thr245Val | -1.530 | -52.6 | LipA-His311Met | -2.993 | -41.2 |
| LipA-Thr245Met | -0.511 | -45.9 | LipA-His311Leu | -2.587 | -46.7 |
| LipA-Leu266Phe | -1.029 | -11.7 | LipA-Ser106Thr | -0.500 | -22.5 |
表选项
2.3 脂肪酶lipA基因微型突变文库的构建 上述58个突变体分属11个空腔(表 2的1号空腔中未能筛选到符合要求的电子突变体)。从每个空腔中选择一个具有代表性的突变体(选择的突变位点及对应的突变氨基酸残基见表 4),利用定点突变技术,构建突变基因,引入突变氨基酸残基。由于3号空腔中选择的突变体LipA-Asp228Leu未能构建成功,因此实际只构建10个lipA突变基因,并考察对应的10个空腔的突变效应。经诱导表达、脂肪酶LipA突变体的热稳定性分析后,仅LipA-His15Pro和LipA-Ala210Val表现为阳性效应(分别属于第5个空腔和第6个空腔,如表 2)。在此基础上,进一步构建表 3中属于第5个空腔(LipA-Asp21Val、LipA-Asp21Met和LipA-Asp21Arg)和第6个空腔的突变体(LipA-Ala210Ile、LipA-Ala210Leu、LipA-Ala210Cys和LipA-Ala210Met)。各个突变体的突变效应如表 4。最后将筛选获得的最优单点突变体LipA-His15Pro和LipA-Ala210Val进行叠加突变。55 ℃下热处理12 min后,突变体LipA-His15Pro、LipA-Ala210Val和LipA-His15Pro/Ala210Val的残余酶活较野生型分别提高了29.9%、33.7%和60.0%。
表 4. 55 ℃处理12 min后不同突变体的残余酶活 Table 4. The residual activity of different mutants heated 12 min at 55 ℃
| Mutation site | Increase rate of residual activity/% |
| LipA-His15Pro | 29.9±2.9 |
| LipA-Asp21Met | -1.4±5.8 |
| LipA-Asp21Val | -10.0±4.8 |
| LipA-Asp21Arg | -11.9±0.5 |
| LipA-Asp36Ile | n.d. |
| LipA-Glu63Tyr | n.d. |
| LipA-Asn82Leu | n.d. |
| LipA-Val84Ile | 11.7±1.5 |
| LipA-Ala210Val | 33.7±2.0 |
| LipA-Ala210Leu | -35.8±0.9 |
| LipA-Ala210Cys | -18.0±7.0 |
| LipA-Ala210Met | n.d. |
| LipA-Thr280Leu | n.d. |
| LipA-Asn285Val | n.d. |
| LipA-Glu289Val | n.d. |
| LipA-Val296Leu | -19.9±1.9 |
| LipA-Ala210Ile | 7.8±0.7 |
| LipA-His15Pro/Ala210Val | 60.0±5.7 |
| n.d.: Not determined. | |
表选项
2.4 野生型脂肪酶及其脂肪酶突变体T5012的测定 野生型脂肪酶与热稳定提高的脂肪酶突变体的T5012的测定结果如图 1。野生型脂肪酶的T5012为53 ℃,脂肪酶突变体LipA-His15Pro、LipA-Ala210Val和LipA-His15Pro/Ala210Val的T5012较野生型脂肪酶均有一定程度的提高,分别为57 ℃、55 ℃和61 ℃ (图 1)。
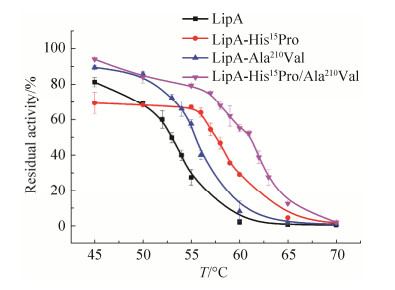 |
| 图 1 野生型脂肪酶及其脂肪酶突变体T5012值的测定 Figure 1 The T5012 of wild type lipase LipA and LipA mutants. Error bars represent standard errors. |
| 图选项 |
2.5 野生型脂肪酶及脂肪酶突变体t1/2的测定 野生型脂肪酶与热稳定脂肪酶突变体的t1/2的测定结果如图 2。野生型脂肪酶LipA的t1/2为5.2 min,而热稳定脂肪酶突变体LipA-His15Pro、LipA-Ala210Val和LipA-His15Pro/Ala210Val的t1/2分别为50 min、15 min和120 min。
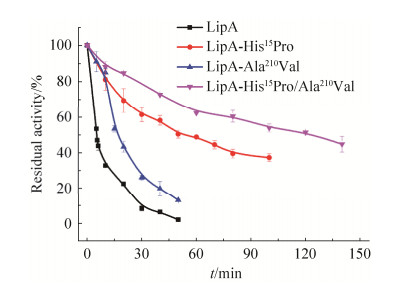 |
| 图 2 野生型脂肪酶LipA及其热稳定突变体t1/2的测定 Figure 2 The t1/2 value of wild type lipase LipA and LipA mutants. Error bars represent standard errors. |
| 图选项 |
2.6 热稳定脂肪酶突变体LipA提高热稳定性的分子机制 LipA分子第15位的组氨酸突变为脯氨酸后,其所处的空腔体积减小了3.2 ?3,而第210位的丙氨酸突变为缬氨酸后,其所处的空腔体积减小了16.1 ?3 (图 3)。通过对蛋白质内部的疏水核心(hydrophobic core)进行理性设计改造,缩小空腔体积,提高蛋白质分子的疏水核心的包装度(packing),有助于提高蛋白质分子的热稳定性[25-26]。分子动力学模拟结果进一步表明:热稳定性脂肪酶突变体LipA-His15Pro/Ala210Val的RMSD曲线较野生型脂肪酶LipA的RMSD曲线明显降低(图 4)。
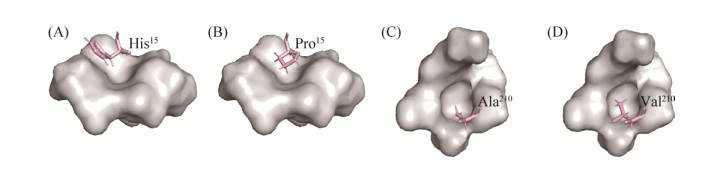 |
| 图 3 两个阳性突变体突变前后的空腔结构 Figure 3 Cavity model of lipase A and lipase A mutants. A/B: Comparison of cavity volume between lipase LipA and lipase LipA-His15Pro; C/D: Comparison of cavity volume between lipase LipA and lipase LipA-Ala210Val. |
| 图选项 |
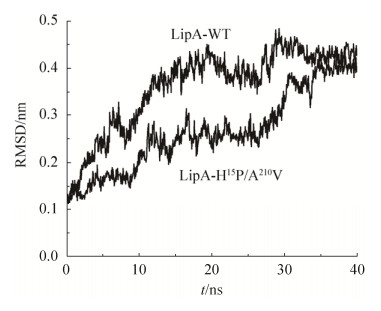 |
| 图 4 野生型脂肪酶LipA及其突变体的RMSD值 Figure 4 The RMSD values of wild type lipase LipA and lipase LipA mutant. |
| 图选项 |
3 讨论 本文利用生物信息学手段分析出伯克霍尔德菌脂肪酶LipA分子3D结构中共存在12个空腔,并对其中的11个空腔进行了“空腔填充”突变(另外一个空腔由于突变体构建失败而未能考察)。实验结果表明:脂肪酶突变体LipA-His15Pro和LipA-Ala210Val的空腔体积缩小,热稳定性有所提高。其中突变体LipA-His15Pro所在的空腔较突变前仅缩小了3.2 ?3,而突变体LipA-Ala210Val所在的空腔较突变前缩小了16.1 ?3 (表 3),但突变体LipA-His15Pro的突变效应优于LipA-Ala210Val (图 2)。除了本文讨论的空腔效应外,突变体LipA-His15Pro应该还存在其他影响脂肪酶突变体热稳定性的分子机制,如解折叠自由能差(ΔΔG) (从表 3中可以看出,本位点突变导致ΔΔG降低了2.027 kcal/mol),或脯氨酸效应(His15所在位置为柔性区域)等。上述两个突变位点的叠加突变进一步提高了“空腔填充”效果,叠加突变体LipA-His15Pro/Ala210Val较野生型脂肪酶LipA在55 ℃下的半衰期延长了23.1倍。使用本研究中的表达体系,lipA基因及其对应的伴侣蛋白基因lipB未能实现高效表达,因此本研究中未能纯化出重组脂肪酶LipA及其突变体,并测定相应的比活力(specificity)、米氏常数(Km)及催化效率(Kcat/Km)等。后续研究中,我们将进一步优化表达体系,提高lipA及lipB的表达量,纯化出重组脂肪酶LipA及其对应的突变体,并比较比活力、米氏常数及催化效率的差异,以更好地理解突变效应及突变机制。
本文中我们并未对组成11个空腔的所有氨基酸残基进行突变。在第一轮筛选过程中,基于氨基酸残基的R基团的大小、疏水性、空腔填充效应及突变体自由能变化值ΔΔG,仅从每个空腔中选择一个有代表性的突变体进行验证是否存在“空腔填充”效应(表 3和表 4)。经验证存在“空腔填充”效应的突变体所在的空腔(如His15Pro所在的第5个空腔和Ala210Val所在的第6个空腔),才进一步构建并验证组成该空腔的其他氨基酸残基突变体的突变效应。实验结果表明,改造蛋白质分子内部的空腔,利用具有较大R基团的疏水氨基酸残基替换,可以取得较好的突变效应[27]。但对位于蛋白质分子表面的空腔进行理性设计改造,仅仅考虑“空腔填充”效应是不够的;除此之外,组成空腔的氨基酸残基的柔性(flexibility)、氨基酸残基与周围临近氨基酸残基的互作效应等,对蛋白质分子的稳定性也存在显著的影响[11]。
我们以具有重要工业应用价值的伯克霍尔德菌胞外脂肪酶LipA作为热稳定蛋白质理性设计(或半理性设计)改造的模型分子,先后利用“B-factor”值进行定点饱和突变筛选热稳定脂肪酶LipA突变体[15];理性设计盐桥构建热稳定脂肪酶LipA突变体[16]及本文基于“空腔填充”效应构建热稳定脂肪酶LipA突变体。此外,我们还对胆固醇酯酶的β-转角(β-turn)进行了优化设计,并基于“脯氨酸”效应理性设计胆固醇酯酶突变体(实验室尚未发表数据)。我们在实验中发现,各类理性设计热稳定蛋白质分子的方法中,基于盐桥效应构建的突变体突变效果相对较为理想;基于单一突变效应理性设计构建的热稳定突变体,很难突破10-15 ℃的瓶颈(ΔTm)[28];多效应联合的多点突变虽然可以显著改善酶蛋白分子的热稳定性[29],但在实验中我们也发现,即使突变位点远离活性中心(实验中,我们对脂肪酶LipA或胆固醇酯酶突变位点均限定为:距离活性中心催化三联体Ser侧链—OH氧原子12 ?的范围外),但多点突变仍然经常导致酶蛋白的催化效率(Kcat/Km)显著降低。如何平衡和取舍酶蛋白的热稳定性和催化效率,依然存在巨大的挑战。
致谢
感谢中国科学院武汉物理与数学研究所段漠杰博士和福建师范大学生命科学学院研究生江虹同学在分子动力学模拟中给予的指导和帮助。
References
| [1] | Pandey A, Benjamin S, Soccol CR, Nigam P, Krieger N, Soccol VT. The realm of microbial lipases in biotechnology. Biotechnology and Applied Biochemistry, 1999, 29: 119-131. |
| [2] | Hasan F, Shah AA, Hameed A. Industrial applications of microbial lipases. Enzyme and Microbial Technology, 2006, 39(2): 235-251. DOI:10.1016/j.enzmictec.2005.10.016 |
| [3] | Pucci F, Rooman M. Physical and molecular bases of protein thermal stability and cold adaptation. Current Opinion in Structural Biology, 2017, 42: 117-128. DOI:10.1016/j.sbi.2016.12.007 |
| [4] | Eijsink VGH, G?seidnes S, Borchert TV, van den Burg B. Directed evolution of enzyme stability. Biomolecular Engineering, 2005, 22(1-3): 21-30. DOI:10.1016/j.bioeng.2004.12.003 |
| [5] | Floor RJ, Wijma HJ, Colpa DI, Ramos-Silva A, Jekel PA, Szymański W, Feringa BL, Marrink SJ, Janssen DB. Computational library design for increasing Haloalkane dehalogenase stability. Chem Bio Chem, 2014, 15(11): 1660-1672. DOI:10.1002/cbic.201402128 |
| [6] | Wijma HJ, Floor RJ, Jekel PA, Baker D, Marrink SJ, Janssen DB. Computationally designed libraries for rapid enzyme stabilization. Protein Engineering, Design and Selection, 2014, 27(2): 49-58. DOI:10.1093/protein/gzt061 |
| [7] | Hubbard SJ, Gross KH, Argos P. Intramolecular cavities in globular proteins. Protein Engineering, Design and Selection, 1994, 7(5): 613-626. DOI:10.1093/protein/7.5.613 |
| [8] | Hubbard SJ, Argos P. Cavities and packing at protein interfaces. Protein Science, 1994, 3(12): 2194-2206. DOI:10.1002/pro.5560031205 |
| [9] | Hubbard SJ, Argos P. A functional role for protein cavities in domain: domain motions. Journal of Molecular Biology, 1996, 261(2): 289-300. DOI:10.1006/jmbi.1996.0460 |
| [10] | Merski M, Shoichet BK. Engineering a model protein cavity to catalyze the Kemp elimination. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(40): 16179-16183. DOI:10.1073/pnas.1208076109 |
| [11] | Joo JC, Pohkrel S, Pack SP, Yoo YJ. Thermostabilization of Bacillus circulans xylanase via computational design of a flexible surface cavity. Journal of Biotechnology, 2010, 146(1/2): 31-39. |
| [12] | Ece S, Evran S, Janda JO, Merkl R, Sterner R. Improving thermal and detergent stability of Bacillus stearothermophilus neopullulanase by rational enzyme design. Protein Engineering, Design and Selection, 2015, 28(6): 147-151. DOI:10.1093/protein/gzv001 |
| [13] | Liu YR, Qiu FJ, Shu ZY, Wang ZZ, Qiu LQ, Li X, Jiang XZ, Huang JZ. Purification and enzymatic characterization of the extracellular lipase from Burkholderia sp. ZYB002. Journal of Fujian Normal University (Natural Science Edition), 2014, 30(3): 100-105. (in Chinese) 刘艳如, 邱芳锦, 舒正玉, 王作镇, 邱黎清, 李欣, 江贤章, 黄建忠. 伯克霍尔德菌ZYB002胞外脂肪酶的分离纯化及其酶学性质分析. 福建师范大学学报(自然科学版), 2014, 30(3): 100-105. |
| [14] | Shu ZY, Wu JG, Chen D, Cheng LX, Zheng Y, Chen JP, Huang JZ. Optimization of Burkholderia sp. ZYB002 lipase production for pitch control in thermomechanical pulping (TMP) processes. Holzforschung, 2012, 66(3): 341-348. |
| [15] | Liu YR, Qiu LQ, Huang JZ, Zhao BC, Wang ZZ, Zhu XL, Gao YY, Shu ZY. Screening for mutants with thermostabe lipase A from Burkholderia sp. ZYB002. Acta Microbiologica Sinica, 2015, 55(6): 748-754. (in Chinese) 刘艳如, 邱黎清, 黄建忠, 赵丙春, 王作镇, 朱晓兰, 高媛媛, 舒正玉. 热稳定性伯克霍尔德菌脂肪酶A突变体的筛选. 微生物学报, 2015, 55(6): 748-754. |
| [16] | Liu YR, Zhao BC, Dong PP, Qiu LQ, Huang JZ, Zhu XL, Wang ZZ, Shu ZY. Computer-aid screening of thermostable lipase LipA from Burkholderia sp. ZYB002. Acta Microbiologica Sinica, 2017, 57(7): 1014-1025. (in Chinese) 刘艳如, 赵丙春, 董盼盼, 邱黎清, 黄建忠, 朱晓兰, 王作镇, 舒正玉. 理性设计盐桥构建伯克霍尔德菌脂肪酶热稳定突变体. 微生物学报, 2017, 57(7): 1014-1025. |
| [17] | Dundas J, Ouyang Z, Tseng J, Binkowski A, Turpaz Y, Liang J. Castp: computed atlas of surface topography of proteins with structural and topographical mapping of functionally annotated residues. Nucleic Acids Research, 2006, 34: W116-W118. DOI:10.1093/nar/gkl282 |
| [18] | Rother K, Hildebrand PW, Goede A, Gruening B, Preissner R. Voronoia: Analyzing packing in protein structures. Nucleic Acids Research, 2009, 37(Database issue): D393-D395. |
| [19] | Bu?a J, Hayryan S, Hu CK, Skrivanek J, Wu MC. Enveloping triangulation method for detecting internal cavities in proteins and algorithm for computing their surface areas and volumes. Journal of Computational Chemistry, 2009, 30(3): 346-357. DOI:10.1002/jcc.21060 |
| [20] | Maupetit J, Derreumaux P, Tuffery P. Pep-fold: an online resource for de novo peptide structure prediction. Nucleic Acids Research, 2009, 37(Web Server issue): W498-W503. |
| [21] | Krieger E, Koraimann G, Vriend G. Increasing the precision of comparative models with YASARA NOVA—a self-parameterizing force field. Proteins, Structure, Function, and Bioinformatics, 2002, 47(3): 393-402. DOI:10.1002/prot.10104 |
| [22] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [23] | Hiol A, Jonzo MD, Druet D, Comeau L. Production, purification and characterization of an extracellular lipase from Mucor hiemalis f. hiemalis. Enzyme and Microbial Technology, 1999, 25(1/2): 80-87. |
| [24] | Zhao HM, Arnold FH. Directed evolution converts subtilisin E into a functional equivalent of thermitase. Protein Engineering, Design and Selection, 1999, 12(1): 47-53. |
| [25] | Abraham T, Abraham T, Pack SP, Yoo YJ. Stabilization of Bacillus subtilis lipase a by increasing the residual packing. Biocatalysis, 2005, 23(3/4): 217-224. |
| [26] | Lazar GA, Handel TM. Hydrophobic core packing and protein design. Current Opinion in Chemical Biology, 1998, 2(6): 675-679. DOI:10.1016/S1367-5931(98)80102-6 |
| [27] | Vriend G, Berendsen HJC, van der Zee JR, van der Burg B, Venema G, Eijsink VGH. Stabilization of the neutral protease of Bacillus stearothermophilus by removal of a buried water molecule. Protein Engineering, Design and Selection, 1991, 4(8): 941-945. DOI:10.1093/protein/4.8.941 |
| [28] | Wijma HJ, Floor RJ, Janssen DB. Structure- and sequence-analysis inspired engineering of proteins for enhanced thermostability. Current Opinion in Structural Biology, 2013, 23(4): 588-594. DOI:10.1016/j.sbi.2013.04.008 |
| [29] | Reetz MT, Carballeira JD, Vogel A. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability. Angewandte Chemie, 2006, 118(46): 7909-7915. DOI:10.1002/ange.200602795 |
