谷天燕, 刘晓楠, 李玲聪, 刘妍池, 胡少锋, 吕晨茵, 刘华, 赵国刚


河北农业大学生命科学学院, 河北 保定 071000
收稿日期:2018-09-25;修回日期:2018-10-15;网络出版日期:2018-12-06
基金项目:国家重点研发计划(2017YFD0201100);河北省留学人员科技活动项目(优秀类)(C2015005015);农业部饲料生物技术重点实验室开放基金
*通信作者:赵国刚, Tel:+86-312-7528250, Fax:+86-312-7528272, E-mail:zhgg@hebau.edu.cn.
摘要:[目的] 通过两端融合表达几丁质结合结构域来提高几丁质酶的活性和生物防治植物病原真菌能力。[方法] 以苜蓿链霉菌(Streptomyces alfalae)ACCC40021中唯一的GH19家族几丁质酶为模板,构建两端融合表达几丁质结合结构域的几丁质酶,并进行原核表达;利用3,5-二硝基水杨酸法(DNS)测定几丁质酶活。[结果] 成功构建了CatDChiB(催化结构域)、rChiB(含N-端几丁质结合结构域)、DChBDChiB(含两端几丁质结合结构域)三种形式的酶,并在大肠杆菌中得到了高效表达;与CatDChiB和rChiB相比,DChBDChiB显著地提高了对α-几丁质、胶体几丁质和黑曲霉几丁质的结合能力和活性;同时增强了其对病原真菌长枝木霉的抑制作用。[结论] 两端融合表达几丁质结合结构域是简单有效的提高几丁质酶活性及抗真菌活性的策略。
关键词:碳水化合物结合模块(CBMs)几丁质结合结构域(ChBD)苜蓿链霉菌(Streptomyces alfalae)几丁质酶植物真菌病害
Enhancing antifungal activity of chitinase by fusion of chitin binding domain at both termini
Tianyan Gu, Xiaonan Liu, Lingcong Li, Yanchi Liu, Shaofeng Hu, Chenyin Lv, Hua Liu, Guogang Zhao


College of Life Sciences, Hebei Agricultural University, Baoding 071000, Hebei Province, China
*Corresponding author: Guogang Zhao, Tel:+86-312-7528250, Fax:+86-312-7528272, E-mail:zhgg@hebau.edu.cn.
Foundation item: Supported by the National Key Research and Development Plan of China (2017YFD0201100), by the Science Project Grant for Oversea Personnel in Hebei Province (Excellent Class) (C2015005015) and by the Open Project Program of Key Laboratory for Feed Biotechnology of the Ministry of Agriculture, China
Abstract: [Objective] To develop a simple method to improve the enzymatic and antifungal activity of chitinase by fusion of chitin binding domain at both termini. [Methods] The chimeric and truncated chitinases were constructed using the unique glycoside hydrolase family 19 chitinase in Streptomyces alfalae ACCC40021 as template, and expressed in Escherichia coli. The 3, 5-dinitrosalicylic acid (DNS) method was used to determine the enzyme activity with colloid chitin as the substrate. [Results] CatDChiB (catalytic domain), rChiB (N-terminal chitin-binding domain) and DChBDChiB (double chitin-binding domain) were successfully constructed and expressed in E. coli BL21(DE3). Compared to CatDChiB and rChiB, DChBDChiB improved the binding ability and activity towards colloidal chitin, α-chitin and chitin from Aspergillus niger. Furthermore, antifungal activity was enhanced against plant pathogenic fungus Trichoderma longibranchiatum. [Conclusion] A simple, feasible and efficient method was developed to improve the enzymatic and antifungal activity of chitinase by fusion of chitin binding domain on both C-and N-terminus of protein.
Keywords: carbohydrate binding moduleschitin-binding domainStreptomyces alfalaechitinasephytopathogenic fungi
碳水化合物结合模块(carbohydrate binding modules,CBMs)是糖苷水解酶结构中没有催化多糖水解功能的底物识别模块[1]。CBMs在底物识别和催化活性两方面对酶起着至关重要的作用[2-3]。CBMs能够增强糖苷水解酶对不溶性多糖底物(例如几丁质、纤维素)的结合能力,进一步提高其催化活性[4-5]。根据氨基酸序列的相似性,CBMs分为84个家族,其中识别几丁质的几丁质结合结构域(chitin-binding domain,ChBD)属于CBM5、CBM12和CBM18家族[1]。
几丁质酶是一种作用于几丁质产生N-乙酰葡萄糖胺和几丁寡糖的糖苷水解酶[6-7]。根据序列同源性,可分为GH18家族的几丁质酶和GH19家族的几丁质酶[8-9],GH18家族的几丁质酶存在于细菌中,GH19家族的几丁质酶主要存在于高等植物中,仅在放线菌和海洋细菌中发现了GH19家族的几丁质酶[10]。放线菌中的GH19家族的几丁质酶来源于植物同源基因的水平转移,使其能够分解真菌细胞壁的几丁质,在抗真菌的过程中处于优势地位[11]。大多数几丁质酶的结构主要由信号肽、几丁质催化域和几丁质结合结构域ChBD三部分组成[12]。植物来源的几丁质酶中的ChBD主要在N端,属于CBM18家族;而细菌和真菌来源的几丁质酶ChBD在N端或C端,属于CBM5或12家族[13]。研究表明,几丁质结合结构域对于几丁质酶酶活和抗真菌活力至关重要[1, 14-15]。
植物真菌病害是造成世界范围内粮食作物和蔬菜水果减产或死亡的主要原因。目前,对植物真菌病害的防治主要依赖化学杀菌剂。化学杀菌剂的长期使用容易引起植病真菌产生抗性,从而降低其效果;另一方面,化学杀菌剂的生产和使用造成了生态系统的破坏以及对人类健康的潜在威胁。而生物防治植物真菌病害具有高效、无污染的特点,是科学研究和社会可持续发展的重要方向。几丁质酶能够分解真菌的细胞壁以抑制真菌生长,因而被作为最有潜力的生防试剂。迄今为止,已有很多不同来源的几丁质酶被发现和性质表征。但是,天然几丁质酶的低活力和高生产成本限制了其广泛使用。因此,利用基因工程技术,开发新型高效几丁质酶是必要的。
苜蓿链霉菌(Streptomyces alfalae) ACCC40021属于放线菌,是从陕西泾阳分离出的有益根际细菌,已经广泛地用作商品化的土壤微生物肥料和抑制植物病原真菌的生防制剂[16-17]。通过对苜蓿链霉菌ACCC40021的基因组序列(GenBank accession No. CP015588)进行CAZy(Carbohydrate active enzyme database)分析,发现唯一的GH19家族的几丁质酶SaChiB基因。结构分析表明在SaChiB N-端具有1个几丁质结合结构域。本研究通过构建并表达CatDChiB (催化结构域)、rChiB (含N-端几丁质结合结构域)、DChBDChiB (含两端几丁质结合结构域)三种形式的几丁质酶蛋白,进行抗真菌功能分析,证明简单的两端融合表达ChBD能够显著地增强几丁质酶对不同几丁质的活性和抗真菌活性。
1 材料和方法 1.1 菌株 苜蓿链霉菌ACCC40021(Streptomyces alfalae ACCC40021)、病原真菌长枝木霉(Trichoderma longibranchiaum) ACCC 31615由中国农业微生物菌种保藏中心提供。大肠杆菌(Escherichia coli) Trans1-T1和BL21 (DE3)购自全式金生物(北京)有限公司。
1.2 主要试剂 PfuDNA聚合酶、限制性内切酶和T4连接酶购自全式金生物(北京)有限公司。硫酸卡那霉素、异丙基β-D-半乳糖苷(IPTG)、黑曲霉、几丁质和其他化学试剂均购自生工生物工程(上海)股份有限公司。α-几丁质购自Sigma(美国)。
黑曲霉几丁质的制备:0.2 mL的碱性蛋白酶2.4 L FG加到5%的黑曲霉水溶液中,在60 ℃下温育12 h后,12000 r/min离心15 min。沉淀在100 ℃下煮沸15 min后,水洗3次,在65 ℃下干燥12 h,得到的黑曲霉粉重新称重,备用。
胶体几丁质制备:10 g几丁质加200 mL浓盐酸,放转子搅拌至沉淀溶解,于4 ℃放置24 h后,加500 mL 50%乙醇,搅拌5 min,离心去上清,加蒸馏水调pH至7左右,最后用蒸馏水定容至500 mL,得到2%胶体几丁质,4 ℃保存备用。
1.3 pET-30a(+)-CatDChiB、pET-30a(+)-rChiB和pET-30a(+)-DChBDChiB的构建 从NCBI中获取蛋白SaChiB的核苷酸序列(GenBank accession No. MH828721)以及氨基酸序列(Refseq: WP0766829881),用signal IP预测其信号肽,得到其成熟蛋白的序列。通过SMART预测出其ChBD序列,且分别设计专一性引物(表 1)。
表 1. 实验所用引物 Table 1. Primers used in the study
| Primer | Sequence (5′→3′) | Restriction site |
| rChiB F | CGCGAATTCACGGCGTCCGCGGCC | EcoRⅠ |
| rChiB R | AGCAAGCTTGCAGCTCAGGTTGGAGC | Hind Ⅲ |
| CatDChiB F | ATATATGAATTCGGCGCCTGCGGCGGCGGCAC | EcoRⅠ |
| CatDChiB R | AGCAAGCTTGCAGCTCAGGTTGGAGC | Hind Ⅲ |
| ChBDChiB F | CGCAAGCTTACGGCGTCCGCGGCC | Hind Ⅲ |
| ChBDChiB R | ATATATCTCGAGCGCGTCCGCCCAGACGTCGG | XhoⅠ |
| Italics sections are protective bases, and underlined sequences are restriction enzyme sites. | ||
表选项
以苜蓿链霉菌ACCC40021基因组为模板,以rChiB F和rChiB R为上下游引物扩增编码成熟几丁质酶SaChiB (去除信号肽)的目的片段rChiB,片段长度为810 bp;以CatDChiB F和CatDChiB R为上下游引物扩增CatDChiB,片段长度为660 bp;以ChBDChiB F和ChBDChiB R为上下游引物扩增ChBDChiB,片段大小为150 bp。PCR反应体系为:1 μL 10 μmol上游引物F和下游引物R,10 μL 5 × TransStart FastPfu Fly Buffer,4 μL 2.5 mmol/L dNTPs,1μL TransStart FastPfu Fly DNA Polymerase,1 μL链霉菌ACCC40021基因组,ddH2O补足至50 μL。PCR程序为98 ℃ 3 min,98 ℃ 30 s,58 ℃ 30 s,72 ℃ 1 min,共40个循环,72 ℃ 10 min。核酸电泳检测其大小,回收目的片段,目的片段rChiB、CatDChiB和载体pET-30a(+)用限制性内切酶EcoR Ⅰ和Hind Ⅲ酶切,T4连接酶连接,转化到大肠杆菌Trans1-T1感受态中,挑选阳性克隆,送华大基因测序得到重组质粒pET-30a(+)-rChiB和pET-30a(+)-CatDChiB;将目的片段ChBDChiB和测序正确的重组质粒pET-30a(+)-rChiB用Hind Ⅲ和XhoⅠ酶切,T4连接酶连接,转化到大肠杆菌Trans1-T1中,挑选阳性克隆送华大基因测序得到重组质粒pET-30a(+)-DChBDChiB,DChBDChiB大小为966 bp,与预期一致。
1.4 重组几丁质酶的原核表达及纯化 将pET-30a(+)-CatDChiB、pET-30a(+)-rChiB和pET-30a(+)-DChBDChiB转化到大肠杆菌表达菌株BL21(DE3)中,涂布于带有50 mg/mL硫酸卡那霉素的LB固体培养基上,37 ℃过夜培养。挑取单克隆接种于2 mL (含50 mg/mL硫酸卡那霉素) LB液体培养基中,37 ℃、220 r/min过夜培养。以1/100的接种量接种于200 mL含50 mg/mL硫酸卡那霉素LB液体培养基中,37 ℃、220 r/min培养2–3 h,当OD600=0.6–0.8时,加入诱导剂终浓度为0.1 mmol/L的IPTG,20 ℃、200 r/min、20 h,于12000 r/min、10 min、4 ℃下离心,收集沉淀,将沉淀溶于20 mL lysis buffer (50 mmol/L NaH2PO4,300 mmol/L NaCl和10 mmol/L咪唑,pH 8.0)中,在冰上用超声波细胞破碎仪进行破碎,以40%的振幅超声破碎后,细胞裂解液于12000 r/min、10 min、4 ℃离心收集,上清即为粗酶液,粗酶液用Ni-NTA纯化,纯化步骤为:将粗酶液加载到Ni柱填料中,用washing buffer (50 mmol/L NaH2PO4,300 mmol/L NaCl和50 mmol/L咪唑,pH 8.0)去除杂蛋白,再用250 mmol/L elution buffer (50 mmol/L NaH2PO4,300 mmoL/L NaCl和250 mmol/L咪唑,pH 8.0)和350 mmol/L elution buffer (50 mmol/L NaH2PO4,300 mmol/L NaCl和350 mmoL/L咪唑,pH 8.0)洗脱并收集带有活性的蛋白,全程4 ℃操作,并用聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamidegel electrophoresis,SDS-PAGE)检测其纯化程度。得到的纯蛋白用30 kDa的超滤管(Sigma,美国)去除咪唑,最后溶在50 mmol/L磷酸盐缓冲液(pH=8)中,放在–20 ℃待用。
1.5 几丁质酶的活性测定 用3, 5-二硝基水杨酸法(3, 5-dinitrosalicylic acid,DNS)测定还原糖的方法检测其活性[6],标准体系为:0.1 mL几丁质酶,0. 2 mL 2%胶体几丁质,0.3 mL 50 mmol/L磷酸盐缓冲液(pH=8),45 ℃反应1 h后用DNS终止反应,100 ℃加热10 min,在λ=540 nm下测定其吸光度。一个酶活单位定义为:在最适条件下,每分钟分解胶体几丁质释放1 μmol GlcNAc所需的酶量。
1.6 重组几丁质酶对不同几丁质的活性 在最适条件(45 ℃,50 mmol/L磷酸盐缓冲液pH=8)下,分别对CatDChiB、rChiB和DChBDChiB进行活性测定,底物为胶体几丁质、α-几丁质和黑曲霉几丁质。
1.7 重组几丁质酶对不溶性底物结合能力分析 取2 μmol/L CatDChiB、rChiB和DChBDChiB分别与1 mg/mL胶体几丁质、α-几丁质和黑曲霉于50 mmol/L磷酸盐缓冲液(pH=8)中充分混匀,于4 ℃下,在旋转混合器上反应1 h后,10000 r/min、5 min收集上清,上清即为未结合的蛋白,通过BCA法测蛋白浓度,蛋白结合率按照公式(1)计算[18]。
 | 公式(1) |
1.9 数据分析 所有数据至少设置3个独立重复实验,均使用GraphPad Prism 5计算其平均值及标准偏差(Standard Deviation,SD)(平均值±SD),方差分析(One-way ANOVA),并且对于小于或等于0.05的P值被认为是统计学显著。
2 结果和分析 2.1 SaChiB蛋白序列分析 通过NCBI CD-search (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析其结构,如图 1所示,SaChiB属于GH19家族的几丁质酶,其结构由信号肽、几丁质结合结构域和催化结构域(GH19几丁质酶)三部分构成,其中几丁质结合结构域属于CBM5家族。
 |
| 图 1 SaChiB结构示意图 Figure 1 Schematic structures of SaChiB. |
| 图选项 |
2.2 重组几丁质酶的构建及原核表达 成功构建了去除ChBD、N端带有ChBD和两端带有ChBD的三种重组质粒pET-30a(+)-CatDChiB、pET-30a(+)-rChiB和pET-30a(+)-DChBDChiB,其结构示意图如图 2-A所示;将成功构建并测序正确的重组质粒转化到大肠杆菌表达菌株BL21 (DE3)进行原核表达,经Ni-NTA纯化后,进行SDS-PAGE分析,如图 2-B所示,三种蛋白经Ni-NTA纯化后得到了单一的目的条带,且蛋白大小CatDChiB为31.0 kDa,rChiB为36.3 kDa,DChBDChiB为41.5 kDa,与预计大小一致。
 |
| 图 2 CatDChiB、rChiB和DChBDChiB的结构示意图和SDS-PAGE分析 Figure 2 Schematic structures and SDS-PAGE analysis of CatDChiB, rChiB and DChBDChiB. A: Schematic structures of CatDChiB, rChiB and DChBDChiB. B: SDS-PAGE analysis of CatDChiB, rChiB and DChBDChiB. Lane M:Standard protein marker; lane 1: purified protein CatDChiB after Ni-NTA; lane 2: purified protein rChiB after Ni-NTA; lane 3: purified protein DChBDChiB after Ni-NTA. |
| 图选项 |
2.3 重组几丁质酶对不溶性底物结合能力分析 CatDChiB、rChiB和DChBDChiB对不溶性底物α-几丁质、胶体几丁质、黑曲霉的结合能力如图 3所示,CatDChiB、rChiB和DChBDChiB对α-几丁质的结合能力分别为34.24%、58.29%、60.52%;对胶体几丁质的结合能力分别为22.29%、50.52%、52.10%;对黑曲霉的结合能力分别为47.39%、54.94%、78.42%。结果证明,融合表达ChBD能够增强几丁质酶对α-几丁质、胶体几丁质、黑曲霉的结合能力。
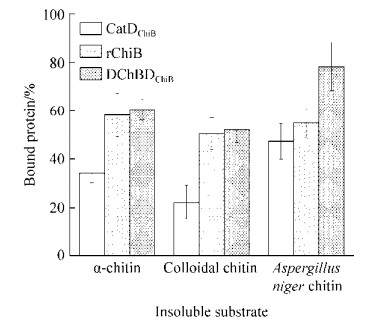 |
| 图 3 CatDChiB、rChiB和DChBDChiB对不溶性底物的结合能力 Figure 3 Insoluble substrate binding ability of CatDChiB, rChiB and DChBDChiB. Data are presented as mean±SD and are representatives of three independent experiments. |
| 图选项 |
2.4 重组几丁质酶对不同几丁质的活性 CatDChiB、rChiB和DChBDChiB分别在45 ℃、50 mmol/L磷酸盐(pH=8)缓冲液下测定其对胶体几丁质、α-几丁质和黑曲霉的活性,如表 2所示,DChBDChiB对胶体几丁质、α-几丁质和黑曲霉的活性最高,rChiB次之,CatDChiB最低,DChBDChiB与rChiB、CatDChiB相比差异显著。
表 2. 重组几丁质酶对不同几丁质的活性 Table 2. Recombinant chitinases activity towards different chitin
| Substrate | CatDChiB /(U/mg) | rChiB /(U/mg) | DChBDChiB /(U/mg) |
| Colloidal chitin | 23.55±0.03 | 28.37±0.50 | 33.19±0.46*** |
| α-chitin | 0.93±0.13 | 2.40±0.06 | 2.81±0.02* |
| Aspergillus niger chitin | 0.47±0.05 | 0.50±0.02 | 0.93±0.12*** |
| Data are presented as mean±SD and asterisk indicates to be statistically signifcant compared with rChiB (* represents P < 0.05; *** represents P < 0.01). | |||
表选项
2.5 抗真菌活性 CatDChiB、rChiB和DChBDChiB对长枝木霉的抗真菌活性如图 4所示,对照对长枝木霉没有抑制作用,DChBDChiB相较于CatDChiB和rChiB有较明显抑制区域,且CatDChiB、rChiB和DChBDChiB的抑制区域为6.76 mm、11.91 mm、17.93 mm。这说明两端加入ChBD能够明显增强几丁质酶活性进而增强其对植物病原真菌长枝木霉的抑制作用。
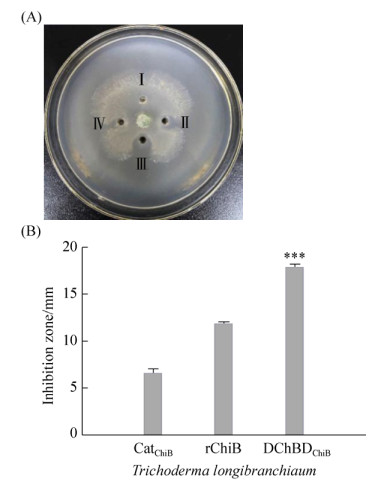 |
| 图 4 CatDChiB、rChiB和DChBDChiB抗真菌活性 Figure 4 Antifungal activity of CatDChiB, rChiB and DChBDChiB. (A): Trichoderma longibranchiaum. (Ⅰ) control (10 mmol/L citrate buffer pH 8 without enzyme); (Ⅱ) 0.67 pmol of purified rChiB (in 10 mmol/L citrate buffer pH 8); (Ⅲ) 0.67 pmol of purified DChBDChiB (in 10 mmol/L citrate buffer pH 8); (Ⅳ) 0.67 pmol of purified CatDChiB (in 10 mmol/L citrate buffer pH 8). (B):inhibition zone diameter of pathogenic fungal growth. Data are presented as mean±SD and *** indicates to be statistically signifcant compared with rChiB (P < 0.01). |
| 图选项 |
3 讨论 本研究通过构建三种重组蛋白CatDChiB、rChiB和DChBDChiB,并在大肠杆菌BL21(DE3)中得到了高效表达,首次成功构建了两端带有ChBD的几丁质酶,并研究了其对不同几丁质的活性和抗真菌活性。
ChBD能够增强对不同几丁质的结合能力和活性。来源于Bacillus circulans WL-12中的ChBD对粉末几丁质和胶体几丁质有明显提高[19]。来自Trichoderma atroviride PTCC5220的C端融合表达ChBD的几丁质酶Chit42与原酶相比,对不溶性几丁质的活性为(1.4±0.3) U/mL、(2.4±0.1) U/mL,提高了1.71倍,对胶体几丁质的活性为(7.76±0.6) U/mL、(10.0±0.7) U/mL,提高了1.29倍[14];在Aeromonassp. No. 10S-24 GH19家族的几丁质酶中,去除ChBD的几丁质酶比原酶对胶体几丁质、晶体几丁质的活性分别下降了30%、65%[15]。本研究通过重组几丁质酶对不溶性底物结合实验表明(图 3):DChBDChiB、rChiB较CatDChiB对α-几丁质、胶体几丁质和黑曲霉的结合能力分别提高了1.76倍、1.70倍;2.34倍、2.27倍;1.65倍、1.16倍;重组几丁质酶对不同几丁质活性实验表明(表 2),DChBDChiB、rChiB较CatDChiB对α-几丁质、胶体几丁质和黑曲霉分别提高了3.02倍、2.58倍;1.4倍、1.2倍;1.98倍、1.06倍。两端融合表达ChBD (DChBDChiB)对底物结合能力和活性最强,且不影响酶的催化结构域的结构和活性中心。本研究融合表达的ChBD根据氨基酸序列同源性分类,属于CBM5。CBM5是Type A类“表面结合”碳水化合物结合模块(surface-binding CBMs),具有独立的空间结构,可以结合不溶性、高度结晶的几丁质或纤维素,进一步提高糖苷水解酶的催化活性[1]。本研究结果证明,两端融合表达ChBD不影响几丁质酶的催化结构域和活性中心的结构,显著地提高了酶对不溶性底物的结合能力和酶活。
ChBD对于几丁质酶抗真菌的作用有显著影响。来自Streptomyces griseus HUT6037的ChiC是第一个被发现的来自细菌的GH19家族的几丁质酶[20],抑制T. reesei实验表明,带有ChBDChiC的天然的ChiC与CatDChiC相比,抗真菌活性提高了10倍左右[21];来自Trichoderma atroviride PTCC5220的Chit42的C端加入一个ChBD之后,明显地提高了抗真菌活性[14]。我们通过抗真菌实验(图 4)发现,DChBDChiB、rChiB较CatDChiB的抗真菌作用分别提高了2.65倍、1.76倍,这说明两端带有ChBDChiB(DChBDChiB)能明显增强对长枝木霉的抑制作用。本研究是首次报道两端融合表达ChBD来提高几丁质酶对长枝木霉的抗真菌能力,该重组几丁质酶可作为一种潜在的生防制剂应用于农业领域。
本研究成功构建了pET-30a(+)-CatDChiB、pET-30a(+)-rChiB和pET-30a(+)-DChBDChiB并在大肠杆菌中得到了高效表达,DChBDChiB相较于CatDChiB和rChiB而言,其在底物结合、活性以及抗真菌活性上都有显著提高。我们的研究结果表明两端融合表达ChBD是简单高效地提高几丁质酶的活性和抗真菌能力的策略;同时也提供了一种潜在的、新型高效的防治植物真菌病害的生防制剂。
References
| [1] | Boraston AB, Bolam DN, Gilbert HJ, Davies GJ. Carbohydrate-binding modules: fine-tuning polysaccharide recognition. Biochemical Journal, 2004, 382(3): 769-781. DOI:10.1042/BJ20040892 |
| [2] | Igarashi K, Koivula A, Wada M, Kimura S, Penttil? M, Samejima M. High speed atomic force microscopy visualizes processive movement of Trichoderma reesei cellobiohydrolase Ⅰ on crystalline cellulose. Journal of Biological Chemistry, 2009, 284(52): 36186-36190. DOI:10.1074/jbc.M109.034611 |
| [3] | Igarashi K, Uchihashi T, Koivula A, Wada M, Kimura S, Okamoto T, Penttil? M, Ando T, Samejima M. Traffic jams reduce hydrolytic efficiency of cellulase on cellulose surface. Science, 2011, 333(6047): 1279-1282. DOI:10.1126/science.1208386 |
| [4] | Arola S, Linder MB. Binding of cellulose binding modules reveal differences between cellulose substrates. Scientific Reports, 2016, 6: 35358. DOI:10.1038/srep35358 |
| [5] | Chhabra SR, Kelly RM. Biochemical characterization of Thermotoga maritima endoglucanase Cel74 with and without a carbohydrate binding module (CBM). FEBS Letters, 2002, 531(2): 375-380. DOI:10.1016/S0014-5793(02)03493-2 |
| [6] | Yang SQ, Fu X, Yan QJ, Jiang ZQ, Wang J. Biochemical characterization of a novel acidic exochitinase from Rhizomucor miehei with antifungal activity. Journal of Agricultural and Food Chemistry, 2016, 64(2): 461-469. DOI:10.1021/acs.jafc.5b05127 |
| [7] | Fu X, Yan QJ, Wang J, Yang SQ, Jiang ZQ. Purification and biochemical characterization of novel acidic chitinase from Paenicibacillus barengoltzii. International Journal of Biological Macromolecules, 2016, 91: 973-979. DOI:10.1016/j.ijbiomac.2016.06.050 |
| [8] | Mallakuntla MK, Vaikuntapu PR, Bhuvanachandra B, Das SN, Podile AR. Transglycosylation by a chitinase from Enterobacter cloacae subsp. cloacae generates longer chitin oligosaccharides. Scientific Reports, 2017, 7: 5113. DOI:10.1038/s41598-017-05140-3 |
| [9] | Vaikuntapu PR, Rambabu S, Madhuprakash J, Podile AR. A new chitinase-D from a plant growth promoting Serratia marcescens GPS5 for enzymatic conversion of chitin. Bioresource Technology, 2016, 220: 200-207. DOI:10.1016/j.biortech.2016.08.055 |
| [10] | Prakash NAU, Jayanthi M, Sabarinathan R, Kangueane P, Mathew L, Sekar K. Evolution, homology conservation, and identification of unique sequence signatures in GH19 family chitinases. Journal of Molecular Evolution, 2010, 70(5): 466-478. DOI:10.1007/s00239-010-9345-z |
| [11] | Kawase T, Yokokawa S, Saito A, Fujii T, Nikaidou N, Miyashita K, Watanabe T. Comparison of enzymatic and antifungal properties between family 18 and 19 chitinases from S. coelicolor A3(2). Bioscience, Biotechnology, and Biochemistry, 2006, 70(4): 988-998. DOI:10.1271/bbb.70.988 |
| [12] | Kezuka Y, Ohishi M, Itoh Y, Watanabe J, Mitsutomi M, Watanabe T, Nonaka T. Structural studies of a two-domain chitinase from Streptomyces griseus HUT6037. Journal of Molecular Biology, 2006, 358(2): 472-484. DOI:10.1016/j.jmb.2006.02.013 |
| [13] | Limón MC, Margolles-Clark E, Benítez T, Penttil? M. Addition of substrate-binding domains increases substrate-binding capacity and specific activity of a chitinase from Trichoderma harzianum. FEMS Microbiology Letters, 2001, 198(1): 57-63. DOI:10.1111/fml.2001.198.issue-1 |
| [14] | Matroodi S, Motallebi M, Zamani M, Moradyar M. Designing a new chitinase with more chitin binding and antifungal activity. World Journal of Microbiology and Biotechnology, 2013, 29(8): 1517-1523. DOI:10.1007/s11274-013-1318-0 |
| [15] | Kojima M, Yoshikawa T, Ueda M, Nonomura T, Matsuda Y, Toyoda H, Miyatake K, Arai M, Fukamizo T. Family 19 chitinase from Aeromonas sp. No. 10S-24: role of chitin-binding domain in the enzymatic activity. The Journal of Biochemistry, 2005, 137(2): 235-242. DOI:10.1093/jb/mvi022 |
| [16] | Tao TS, Yue YY, Liang SF, Sang JL, Yin SY. The antibiotic strain 5406-Streptomyces jingyangensis N. SP. Acta Microbiologica Sinica, 1979, 19(3): 249-254. (in Chinese) 陶天申, 岳莹玉, 梁绍芬, 桑金隆, 尹莘耘. 5406抗生菌--泾阳链霉菌新种. 微生物学报, 1979, 19(3): 249-254. |
| [17] | She WQ, Sun ZF, Yi L, Zhao SM, Liang YX. Streptomyces alfalfae sp. nov. and comparisons with its closest taxa Streptomyces silaceus, Streptomyces flavofungini and Streptomyces intermedius. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(1): 44-49. DOI:10.1099/ijsem.0.000671 |
| [18] | Suma K, Podile AR. Chitinase A from Stenotrophomonas maltophilia shows transglycosylation and antifungal activities. Bioresource Technology, 2013, 133: 213-220. DOI:10.1016/j.biortech.2013.01.103 |
| [19] | Hashimoto M, Ikegami T, Seino S, Ohuchi N, Fukada H, Sugiyama J, Shirakawa M, Watanabe T. Expression and characterization of the chitin-binding domain of chitinase A1 from Bacillus circulans WL-12. Journal of Bacteriology, 2000, 182(11): 3045-3054. DOI:10.1128/JB.182.11.3045-3054.2000 |
| [20] | Ohno T, Armand S, Hata T, Nikaidou N, Henrissat B, Mitsutomi M, Watanabe T. A modular family 19 chitinase found in the prokaryotic organism Streptomyces griseus HUT 6037. Journal of Bacteriology, 1996, 178(17): 5065-5070. DOI:10.1128/jb.178.17.5065-5070.1996 |
| [21] | Itoh Y, Kawase T, Nikaidou N, Fukada H, Mitsutomi M, Watanabe T, Itoh Y. Functional analysis of the chitin-binding domain of a family 19 chitinase from Streptomyces griseus HUT6037: substrate-binding affinity and cis-dominant increase of antifungal function. Bioscience, Biotechnology, and Biochemistry, 2002, 66(5): 1084-1092. DOI:10.1271/bbb.66.1084 |
