朱菲莹1, 张屹1, 肖姬玲1, 魏林2, 梁志怀1


1. 湖南省农业生物技术研究所, 湖南 长沙 410125;
2. 湖南省植物保护研究所, 湖南 长沙 410125
收稿日期:2019-01-24;修回日期:2019-03-13;网络出版日期:2019-03-29
基金项目:公益性行业(农业)科研专项(201503110-03);国家重点研发计划(2017YFD0200606);湖南省农业科技创新资金项目(2018QN37)
*通信作者:梁志怀, E-mail:liangzhihuainky@163.com.
摘要:[目的] 研究施用生物有机肥处理土壤后对西瓜枯萎病发生的影响,明确可能与西瓜枯萎病发病密切相关的土壤微生物群落结构。[方法] 设置2组不同年份材料对比,依次为施用生物有机肥后2016年发病期(CK2016、T12016)和2017年发病期(CK2017、T12017)。基于16S rRNA序列测定,利用Illumina Miseq高通量测序平台对施用生物有机肥后不同年份的土壤微生物群落组成和差异进行测序分析。[结果] 试验结果发现,不同年份的土壤微生物群落中alpha多样性指数并无明显的差异,但是施用生物有机肥后的土壤相比对照土壤中细菌群落多样性略有增高。不同年份土壤细菌群落结构在门水平上差异不显著,其中变形菌门和硬壁菌门是构成这两个年份土壤细菌的重要组成部分,同时也是比较稳定的微生物类群。在属水平上分析发现主要动态变化菌属为芽孢杆菌属、肠球菌属、乳球菌属及水恒杆菌属。通过Spearman分析发现它们与西瓜枯萎病发生率的关系均为负相关。[结论] 施用生物有机肥可帮助西瓜连作土壤的生态修复,对枯萎病防治起到一定的作用。通过对施用生物有机肥的不同年份土壤微生物种群结构动态变化及对枯萎病发生率呈正负相关的微生物菌属的分析研究,为如何利用调节土壤微生物群落结构来防治西瓜枯萎病提供了新的思路。
关键词:西瓜土壤微生物群落生物有机肥高通量测序枯萎病
Regulation of soil microbial community structures and watermelon Fusarium wilt by using bio-organic fertilizer
Feiying Zhu1, Yi Zhang1, Jiling Xiao1, Lin Wei2, Zhihuai Liang1


1. Hunan Agricultural Biotechnology Research Institute, Changsha 410125, Hunan Province, China;
2. Institute of Plant Protection, Hunan Province, Changsha 410125, Hunan Province, China
Received: 24 January 2019; Revised: 13 March 2019; Published online: 29 March 2019
*Corresponding author: Zhihuai Liang, E-mail:liangzhihuainky@163.com.
Foundation item: Supported by the Special Fund for Agro-scientific Research in the Public Interest (201503110-03), by the National Key Research and Development Program of China (2017YFD0200606) and by Funds for Independent Innovation of Agricultural Science and Technology in Hunan Province (2018QN37)
Abstract: [Objective] The objective of our study was to compare the soil bacterial communities by using bio-organic fertilizer in disease occurring period, and to excavate the closely related bacterial genera to Watermelon Fusarium wilt. [Methods] The V3-V4 region of the 16S rRNA gene in bacterial DNA was amplified using the polymerase chain reaction technique and sequenced by the Illumina Miseq platform to diversity analysis of the soil microbial community composition after application of bio-organic fertilizer in different years (2016 onset stage and 2017 onset stage). [Results] There was no significant difference in alpha diversity indices among soil microbial community diversity at different years, but the diversity richness increased slightly after applying bio-organic fertilizer by comparison with the control. At phylum level, the Proteobacteria and Firmicutes were the relatively stable bacterial communities in these two periods. However, at genus level, the major dynamic bacteria were Bacillus, Enterococcus, Lactococcus and Mizugakiibacter. Furthermore, the Spearman analysis showed that they were negatively correlated with the watermelon Fusarium wilt disease morbidity. [Conclusion] The application of bio-organic fertilizer plays a certain role in the control of watermelon Fusarium wilt by helping ecological restoration of microbial communities in continuously cropped watermelon-growing soil. Thus, the approach to identify the dynamic changes of soil microbes and find out their relationships between the incidence of watermelon Fusarium wilt provided a new idea of helping control watermelon Fusarium wilt by adjusting soil microbial community structure.
Keywords: watermelonsoil microbesbio-organic fertilizerIllumina MiseqFusarium wilt
土壤微生物在土壤生态系统中的有机物质转化和能量循环中具有举足轻重的作用。如土壤生态系统中养分循环速度的高低,对于稳定土壤生态系统、抵御不良环境、预防和调控土传病害等具有重要作用。生物有机肥施用,促进了土壤中多种微生物的大量繁殖,增加了土壤中微生物的群体数量,改善了土壤结构,提高了土壤中微生物的活性,从而调节土壤微生物生态平衡,有利于抑制了农作物病害的发生[1-2]。近年来,许多研究表明添加生物有机肥在改良设施蔬菜等土壤方面发挥着重要作用[3-5]。西瓜枯萎病(Fusarium oxysporum)是西瓜栽培生产中的一种最重要的病害,严重影响西瓜产量和品质[3]。研究生物有机肥施用来调节土壤微生物群落结构,以达到防治西瓜枯萎病的目的,对提高西瓜产量和品质及瓜农收入、实现西瓜栽培可持续发展等具有重要生产指导意义[6]。长期施用生物有机肥可以提高土壤肥力,改善土壤理化性质,调控土壤微生物群落结构,使退化的土壤生态系统得以快速恢复[7, 8]。施用生物有机肥可增加土壤中有机碳,既有助于促进土壤中生物多样性的恢复,也可对土壤中病原菌种群数量的增加起到一定的抑制作用。已有较多研究表明,施加不同外源有机物对调控土壤微生物群落结构存在明显差异。生物有机肥的施用不仅可以增进土壤肥力,而且有助于改善作物根系微生态环境的理化性状,进而提高有益微生物的活性和农作物的抗病性[3, 5, 8, 12]。Chen研究发现,在大田施用微生物菌剂不仅可显著提高土壤微生物多样性,而且可改变土壤微生物的功能多样性[9]。Zhou等研究发现在温室盆栽条件下连作黄瓜土壤中真菌群落多样性呈动态变化,发现土壤真菌种群变化与黄瓜生长状况紧密相关[10]。Li等施用拮抗菌和生物有机肥,促进了黄瓜生长,而且起到了防控黄瓜土传立枯病,降低根际土壤病原菌数量及其改善根际土壤微生物区系的作用,结果发现以生物有机肥的处理效果最好[11]。
本课题组在大田试验中研究了不同肥料组合对西瓜枯萎病发生的影响,发现施用棉隆+生物有机肥可以重建土壤生态环境,土壤细菌的多样性得到了提高,对西瓜枯萎病具有显著的防治效果[12]。本研究以西瓜早佳8424为材料,选用南京农业大学研制的馕播王牌复合微生物肥进行土壤处理,通过高通量测序平台检测,对施用生物有机肥后不同时期土壤微生物多样性的动态变化以及对西瓜枯萎病发生的影响进行差异比较分析。以期获得土壤微生物群落结构在门和属水平上的动态变化规律,找到与西瓜枯萎病发生呈正负相关的微生物菌群,为利用生物有机肥调节土壤微生物群落结构以达到防治西瓜枯萎病的目的提供有效途径。
1 材料和方法 1.1 试验区概况 本试验在2016–2017年于湖南省农业科学院高桥科研基地(长沙市长沙县高桥镇)进行,为连作3年的西瓜大棚种植区。土壤质地为沙壤土,pH 4.77,有机质23.6 g/kg,总氮1.64 g/kg,碱解氮107.5 mg/kg,总磷1.09 g/kg,有效磷252.97 mg/kg,总钾23.7 g/kg,速效钾262.5 mg/kg[9]。
1.2 试验设计 试验在连作3年的西瓜大棚(30 m×6 m)内进行。设置试验组不同时期对比:T12016 (2016年发病期,生物有机肥处理),CK2016 (2016年发病期,对照),T12017 (2017年发病期,生物有机肥处理),CK2017 (2017年发病期,对照)。供试作物为西瓜早佳8424(由新疆农人公司提供)。供试生物有机肥为馕播王牌复合微生物肥,由南京农业大学提供,总养分N+P2O5+K2O=8%,有机质>20%,有效活菌数>0.2亿/g,主要菌种为芽孢杆菌。每年3月份施用生物有机肥280 kg/棚,撒施后立即旋耕(深度30 cm)。并同时将西瓜苗置于营养钵内培育,30–35 d后进行移栽,常规施肥管理,畦宽3 m,株距50 cm,双蔓整枝对爬,密度360株/667 m2。
1.3 田间病害调查 调查统计西瓜生育期内枯萎病的发生情况,并计算发病率。发病率(%)=(发病株数/调查总株数)×100。
由表 1可见,生物有机肥施用后,西瓜枯萎病发病率显著降低。2017年较2016年施用生物有机肥发病率有所降低,分别为54%和48%,说明连续施用生物有机肥可能抑制西瓜枯萎病的发生。
表 1. 西瓜发病情况 Table 1. Disease incidence of watermelon plants
| Treatment | Total plant number | Diseased plant number | Disease rate/% |
| T12016 | 89.33±6.11a | 48.00±14.00a | 54±0.15a |
| T12017 | 76.67±1.15b | 36.67±1.53b | 48±0.02b |
| CK2016 | 89.33±1.15a | 82.67±1.53c | 93±0.27c |
| CK2017 | 89.33±1.15a | 61.00±16.52d | 80±0.23d |
| Data were expressed as mean±SD (n=3). Different lower-case letters showed significant difference at 0.05 level according to Duncan’s multiple range test at 0.05 level. | |||
表选项
1.4 田间取样 分别在2016–2017年西瓜枯萎病发病期不同处理的试验区内采集土样。在大棚内按“S”形随机选择植株,用土壤取样器采取10–20 cm深的土壤,在每个大棚中选取5点土壤样品混为1个重复,设置3个重复,4个时期共取12份土样装密封袋带回实验室。去除土壤中植物根系和石块,过60目标准筛后分成2份。其中1份样品风干后用于土壤理化性质的检测,另外1份样品装入10 mL无菌离心管中置于–80 ℃冰箱保存作测序分析用。
1.5 基于16S rRNA的土壤微生物高通量测序分析 选用OMEGA Soil DNA kit (200)试剂盒提取土壤样本中总DNA。对细菌16S rRNA基因V3–V4高变区进行PCR扩增(Bio-Rad MyCycler普通PCR仪),引物序列为:343F-798R。PCR扩增采用HiFi Hot Start Ready Mix试剂盒(反应体系为:2×HiFi Hot Start Ready Mix 15 μL,P1 1 μL,P2 1 μL,DNA 50 ng,ddH2O 13 μL,T:30 μL)。扩增反应程序为:94 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,30个循环,72 ℃ 7 min。将产物送交上海美吉生物有限公司进行Illumina Miseq高通量测序。
1.6 数据处理与分析 Illumina Miseq测序所得数据称为raw data,获得raw data后,对原始双端测序数据进行去杂及拼接等处理,去除低质量碱基序列。然后,通过对去杂后的数据进行去嵌合体序列处理,从而得到较优质的序列,进而作下游分析。利用Fast QC软件对测序原始数据进行质量控制,然后拼接,过滤掉低质量的序列。基于这些高质量序列进行OTUs (Operational taxonomic units)的聚类及物种分类,将物种注释与OTUs结合,从而得到每个样品的OTUs及分类谱系分析的基本结果。首先挑选OTUs,即对所有的优质序列采用CD-HIT方法对OTU进行分类,将97%相似度即对应物种分类到属水平作为分类标准。随后以在OTU分类中丰度最大序列为代表序列。与Greengenes数据库进行比对后最终得到系统发育树和OTU分类表格,最后执行α多样性以及β多样性的分析。利用MOTHUR软件进行微生物群落的α多样性分析,包括SHANNON指数和SIMPSON指数。基于tax_summary_a文件夹中的数据表,利用R语言工具作图(物种Venn图,群落柱形图等);利用冗余分析技术(Redundancy analysis,RDA)及Spearman相关系数,对土壤样品、微生物群落结构和西瓜植株的病害进行关联性分析。部分表格数据整理与分析采用EXCEL 2010及SPSS Statistics 17.0软件完成。
2 结果和分析 2.1 土壤样品测序有效性 通过对供试12份土壤样品的16S rRNA高通量测序分析,抽平后共获得69036条有效序列,各组文库的覆盖率均达97%以上,以表明所建的文库可真实体现样本中的细菌实际数量。
2.2 不同年份土壤微生物群落OTU特异性分析 通过对不同年份采集的土壤样品中细菌群落Venn图(图 1)的比较分析,可以清晰地展现出它们的微生物群落OTU数目组成、特异性、重叠或相似性。不同时期生物有机肥处理与对照之间存在的共有细菌OTUs数量为460个,其中CK2016特有222个OTUs,T12016特有75个OTUs;CK2017特有84个OTUs,T12017特有85个OTUs。CK2016与CK2017共有800个OTUs,T12016与T12017共有677个OTUs。表明生物有机肥处理后不同时期的土壤细菌群落整体结构变化不大,但也存在个别差异。
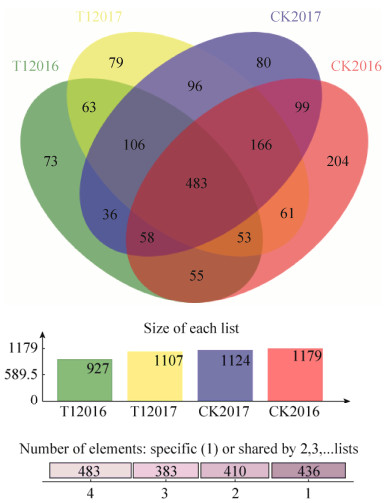 |
| 图 1 不同年份土壤样品中微生物群落OTU维恩图 Figure 1 Venn diagram of soil bacterial community OTUs in soil samples from different years. Different colors represent different groups. The overlapping part represents the species common to multiple groupings. The non-overlapping part represents the species specific to the group, and the number represents the corresponding number of species. |
| 图选项 |
2.3 土壤微生物群落α多样性 单样本的多样性(α多样性)可以反映微生物群落的丰富度和多样性。其中Simpson指数表示随机选取的2条序列属于同一OTU的概率,其指数数值越大说明样本的多样性就越低。Shannon指数常用于反映群落多样性,其指数数值越大说明样本的多样性就越高。Student’s t test用于比较2个平均数的差异是否显著。由图 2可以看出,通过对4组土壤样品微生物α多样性的分析,施用生物有机肥后土壤细菌群落多样性增高,但是差异不显著。
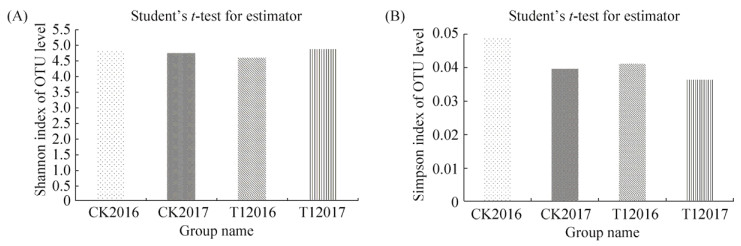 |
| 图 2 不同年份土壤细菌多样性指数 Figure 2 Diversity indices of soil bacterial communities in different samples from different years. The abscissa is the group name and the ordinate is the exponential average of each group. This chart shows the significant differences between samples. A: Shannon index; B: Simpson index. |
| 图选项 |
2.4 土壤中微生物群落结构动态变化及分析
2.4.1 在门水平上土壤细菌群落结构动态变化: 在门水平上,不同时期检测出生物有机肥处理后土壤样品中共有优势细菌从高到低分别为变形菌门(Proteobacteria)、硬壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、拟杆菌门(Bacteroidetes)、糖杆形菌门(Saccharibacteria)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)。其中T12016样品中的主要细菌占比为变形菌门33.66%、硬壁菌门32.08%、酸杆菌门7.24%、芽单胞菌门5.85%;T12017样品中的主要细菌占比为硬壁菌门27.53%、变形菌门24.24%、酸杆菌门15.16%、拟杆菌门6.47%;CK2016样品中的主要细菌占比为硬壁菌门31.06%、变形菌门25.69%、酸杆菌门15.90%、绿弯菌门5.70%;CK2017样品中的主要细菌占有比为变形菌门26.2%、硬壁菌门19.62%、酸杆菌门16.03%、拟杆菌门10.05%、绿弯菌门8.83% (图 3)。
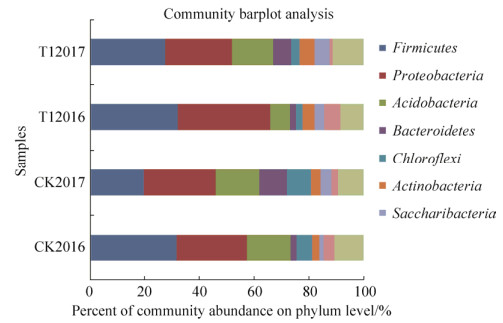 |
| 图 3 门水平上不同年份土壤样品中的主要细菌群落相对丰度 Figure 3 Relative community abundance of main soil bacterial communities during different years on phylum level. The ordinate is the sample name, the abscissa is the proportion of the species in the modified sample. The columns of different colors represent different species, and the length of the columns represents the proportion of the species, others < 0.05. |
| 图选项 |
2.4.2 在属水平土壤细菌群落结构的动态变化: 由图 4可见,在共有细菌中占优势的菌属分别为芽孢杆菌属(Bacillus)、肠球菌属(Enterococcus)、乳球菌属(Lactococcus)、水恒杆菌属(Mizugakiibacter)、罗丹菌属(Rhodanobacter)、根霉菌属(Rhizomicrobium)、芽单胞菌属(Gemmatimonas)、硫杆菌属(Thiobacillus)、多变芽孢杆菌属(Effusibacillus)及Chryseolinea。在T12016样品中的主要细菌占比为肠球菌属11.50%、芽孢杆菌属9.34%、乳球菌属7.06%、水恒杆菌属4.69%、罗丹菌属3.55%、芽单胞菌属2.79%、根霉菌属2.78%;在T12017样品中的主要细菌占比为芽孢杆菌属18.01%、乳球菌属5.23%、罗丹菌属3.73%、水恒杆菌属2.72%、根霉菌属1.95%。在CK2016样品中的主要细菌占比为芽孢杆菌属13.20%、肠球菌属3.34%、多变芽孢杆菌属2.80%、芽单胞菌属1.04%;在CK2017样品中的主要细菌占有比为芽孢杆菌属13.02%、乳球菌属3.93%、硫杆菌属3.09%、罗丹菌属2.53%、Chryseolinea 2.11%。
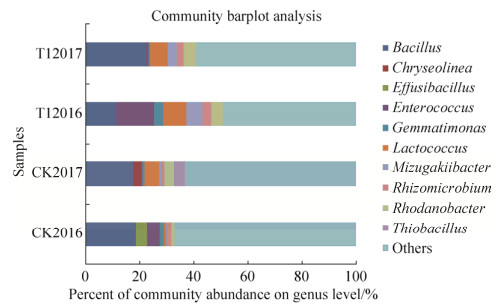 |
| 图 4 属水平上不同年份土壤样品主要细菌群落的相对丰度 Figure 4 Relative community abundance of main soil bacterial communities during different years on genus level. The ordinate is the sample name, the abscissa is the proportion of the species in the modified sample. The columns of different colors represent different species, and the length of the columns represents the proportion of the species, others < 0.02. |
| 图选项 |
2.5 土壤微生物与西瓜枯萎病发病率正负相关菌群分析 如表 2所示,从属水平上看,与枯萎病发病率呈正相关的细菌菌群按相关性系数大小排列为硝化螺旋菌属(Nitrospira)、Chryseolinea、Acidibacter;与西瓜枯萎病发病率呈负相关的菌群按相关性系数大小排列为水恒杆菌属、酸杆菌属、乳球菌属、鞘氨醇单胞菌属(Sphingomonas)、罗丹菌属、根霉菌属、克洛诺菌属(Cronobacter)、芽孢杆菌属细菌。
表 2. 土壤中细菌与枯萎病发病率之间的Spearman相关性 Table 2. Spearman correlation coefficients between the soil bacterial diversity and the disease incidence
| Genus | Correlation coefficient |
| Mizugakiibacter | –0.687 |
| Acidobacterium | –0.673 |
| Lactococcus | –0.535 |
| Sphingomonas | –0.322 |
| Rhodanobacter | –0.305 |
| Rhizomicrobium | –0.189 |
| Cronobacter | –0.156 |
| Bacillus | –0.147 |
| Enterococcus | –0.095 |
| Thiobacillus | –0.086 |
| Gemmatinonas | –0.077 |
| Acidibacter | 0.06 |
| Chryseolinea | 0.429 |
| Nitrospira | 0.531 |
表选项
3 讨论 3.1 调控前后不同时期土壤微生物群落结构变化与功能恢复 土壤微生物的菌群数量及其种类分布不仅作为影响植物生长发育的重要因子,而且与植物病害发生存在着密切联系[13-14]。Philippot等研究指出,作为土壤健康状态的重要指标,其中土壤微生物群落结构多样性成为保护土壤生态系统的重要媒介[15]。土壤微生物群落的多样性又受到土壤各种特性的影响,且不同施肥处理对土壤微生物影响也不同。越来越多的研究发现,植物根际微生态的失衡可能是引起作物连作障碍的主要原因[7, 16]。连作条件使“植物-土壤-微生物”三者的稳定关系在农田生态系统受到干扰。因此,植物通过根际分泌物影响根际微生物群落结构和组成,反之,微生物对植株的物质和能量循环产生相应的响应机制,对植物的信息传递等产生重要影响,进而影响了植物的生长发育和健康[17-18]。
本文通过施用生物有机肥调查研究了其在不同时期对西瓜枯萎病发生的影响。试验结果发现,不同年份的土壤细菌群落中Simpson指数和Shannon指数并无明显的差异,说明施用生物有机肥后的土壤相比对照土壤中的细菌群落多样性增高,但是差异不显著。在门水平上的分析表明,生物有机肥施用后在2个调查时期(2016年发病期,2017年发病期)的土壤样品中的主要细菌群落动态变化表现为:变形菌门33.66%–24.24%,疵壁菌门32.08%–27.53%,酸杆菌门7.24%–15.16%。其中变形菌门、疵壁菌门在这2个时期的土壤微生物中占比最高,说明它们不仅是构成这2个时期土壤微生物的重要组成部分,而且比较稳定。而在属水平上分析发现主要动态变化菌属为:芽孢杆菌属9.34%–18.01%,肠球菌属11.5%–0.8%,乳球菌属7.06%–5.23%,水恒杆菌属4.69%–2.72%,根霉菌属2.78%–1.95%,其中芽孢杆菌属细菌占比最多。
3.2 土壤微生物群落结构中与西瓜枯萎病发病正负相关菌群分析 本课题组于2013年开始在西瓜连作障碍治理示范区开展了植物-土壤-微生态基础理论研究,长期监测不同措施对连作西瓜病害的防控效果,发现施用棉隆+生物有机肥可通过重建土壤生态环境提高土壤细菌多样性,从而对西瓜枯萎病有显著的防治作用[12]。
本试验通过对生物多样性相对丰度排名前50的土壤细菌菌属与西瓜枯萎病发病率之间相关性的分析,发现生物有机肥处理后土壤中主要菌群大都与西瓜枯萎病发病率呈负相关。其中芽孢杆菌属、肠球菌属、乳球菌属、水恒杆菌属等在两个调查时期为主要发生动态变化的菌属,与西瓜枯萎病呈负相关。芽孢杆菌属在2个调查时期均是土壤中主要细菌类群,说明其在西瓜的生长中起着重要作用。有研究表明,芽孢杆菌属对诸多枯萎病具有生防作用,它具有繁殖快、适应性广、易培养等特点而被越来越多的研究者认可并应用。Rhizomicrobium是Ueki等在2010年从水稻根中分离到的1株兼性厌氧发酵芽孢杆菌[19]。Prakash等从受污染的富含硝酸盐的含水层中分离得到Rhodanobacter,属于反硝化细菌属[20]。肠球菌属是一类存在于人类和动物肠道的菌群之一[21-23],近几年研究发现其广泛存在于土壤[24]、水[25]及植物[26]中。研究发现Enterococcus faecium LKE12能通过产生赤霉素和生长素等植物激素促进植物生长[26]。乳球菌属由Schleifer等(1985)提议设立,该菌引起人类的各种感染与肠球菌相似[27-28],乳酸乳球菌广泛用于食品生产[29]和医疗应用。Golomb等通过对拟南芥叶组织溶解物中乳酸乳球菌的生长过程进行转录组分析,发现菌株KF147中与碳水化合物代谢相关基因被诱导表达[30]。同时,Altin的研究认为从土壤中分离出的一种乳球菌菌株[31]可作为一种与金属转运酶相关的新型生物材料。水恒杆菌属最先被日本科学家从淡水湖沉积物中分离发现[32-33]。课题组在前期调查过程中,通过西瓜根际土壤细菌群落多样性的研究发现,水恒杆菌属多样性丰度在非连作西瓜根际土壤中较连作土壤中明显升高[2]。因此,挖掘此类菌株有望筛选出对西瓜枯萎病具有生防作用的微生物菌剂。
本文研究发现,土壤中存在与西瓜枯萎病发病率呈正相关的一些细菌菌群,如硝化螺旋菌属、Chryseolinea、Acidibacter。硝化螺旋菌属在土壤[34]中作为一种好氧化能自养的亚硝酸盐氧化菌,在植物硝化过程中起着关键作用[35]。有研究报道指出硝化螺旋菌属是氮循环微生物群落的关键组成部分[36]。Chryseolinea是由德国科学家于2013年发现的一种新细菌,隶属于拟杆菌门[37],我国科学家也从土壤样本中分离得到类似菌株[38]。Acidibacter最早是由Falagany等[39]从西班牙西南部废弃金属矿的1个坑湖中分离鉴定出的嗜酸铁还原γ-变形杆菌。但是,对于Chryseolinea和Acidibacter 2个属的功能还鲜有报道。这些与西瓜枯萎病发病率呈正相关的细菌菌群可能通过分泌物促使植株发病,也可降解土壤中有机物质,产生某些有利于西瓜枯萎病菌侵染植株的物质,从而导致西瓜枯萎病发生;这些微生物菌群丰度的变化可能影响植株抗病性发生改变。
4 结论 本试验研究结果表明,施用生物有机肥后在不同时期土壤细菌alpha多样性并无明显差异,说明2016和2017年西瓜枯萎病发生时期的土壤细菌群落结构动态变化不大。该实验表明,大田施用生物有机肥后的短时间内,土壤细菌种群的变化不大,说明生物有机肥对土壤细菌群落重建、恢复和维持等是一个缓慢的过程。根际微生物群落与植物健康密切相关,调控根际土壤微生物可增强植物抵御病虫害的能力,因此,合理的农业管理和土壤改良措施可以调控土壤微生物的结构和功能,从而诱导植物产生获得性免疫以抵御病虫害发生。本研究利用高通量测序技术发现的与西瓜枯萎病发病率呈正负相关的微生物菌群,为如何利用调节土壤微生物群落结构来防治西瓜枯萎病提供了新思路。随着分子生物技术的高速发展,运用现代农业生物技术的手段探索建立生物资源多样性与栽培措施协调性的技术体系,为在西瓜连作条件下采取有效措施调控植物根际土壤微生物菌群结构及改变根际土壤理化性状,以便稳定和提高西瓜产量和品质,提供了新的理论依据。
References
| [1] | Huang LF, Song LX, Xia XJ, Mao WH, Shi K, Zhou YH, Yu JQ. Plant-soil feedbacks and soil sickness: from mechanisms to application in agriculture. Journal of Chemical Ecology, 2013, 39(2): 232-242. DOI:10.1007/s10886-013-0244-9 |
| [2] | Berendsen RL, Pieterse CMJ, Bakker PAHM. The rhizosphere microbiome and plant health. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001 |
| [3] | Ling N, Deng KY, Song Y, Wu YC, Zhao J, Raza W, Huang QW, Shen QR. Variation of rhizosphere bacterial community in watermelon continuous mono-cropping soil by long-term application of a novel bioorganic fertilizer. Microbiological Research, 2014, 169(7/8): 570-578. |
| [4] | Shen ZZ, Ruan YZ, Chao X, Zhang J, Li R, Shen QR. Rhizosphere microbial community manipulated by 2 years of consecutive biofertilizer application associated with banana Fusarium wilt disease suppression. Biology and Fertility of Soils, 2015, 51(5): 553-562. DOI:10.1007/s00374-015-1002-7 |
| [5] | Yuan SF, Li MY, Fang ZY, Liu Y, Shi W, Pan B, Wu K, Shi JX, Shen B, Shen QR. Biological control of tobacco bacterial wilt using Trichoderma harzianum amended bioorganic fertilizer and the arbuscular mycorrhizal fungi Glomus mosseae. Biological Control, 2016, 92: 164-171. DOI:10.1016/j.biocontrol.2015.10.013 |
| [6] | Zhu FY, Li JG, Zhang Y, Xiao JL, Liang ZH. Watermelon rhizosphere soil bacterial diversity affects the occurrence of fusarium wilt. Chinese Agricultural Science Bulletin, 2018, 34(17): 69-76. (in Chinese) 朱菲莹, 李基光, 张屹, 肖姬玲, 梁志怀. 西瓜根际土壤细菌群落多样性对枯萎病发生的影响. 中国农学通报, 2018, 34(17): 69-76. |
| [7] | Marcote I, Hernández T, Garc??a C, Polo A. Influence of one or two successive annual applications of organic fertilisers on the enzyme activity of a soil under barley cultivation. Bioresource Technology, 2001, 79(2): 147-154. DOI:10.1016/S0960-8524(01)00048-7 |
| [8] | Zhao QY, Dong CX, Yang XM, Mei XL, Ran W, Shen QR, Xu YC. Biocontrol of Fusarium wilt disease for Cucumis melo melon using bio-organic fertilizer. Applied Soil Ecology, 2011, 47(1): 67-75. DOI:10.1016/j.apsoil.2010.09.010 |
| [9] | Chen L, Xun WB, Sun L, Zhang N, Shen QR, Zhang RF. Effect of different long-term fertilization regimes on the viral community in an agricultural soil of southern china. European Journal of Soil Biology, 2014, 62: 121-126. DOI:10.1016/j.ejsobi.2014.03.006 |
| [10] | Zhou JZ, Wu LY, Deng Y, Zhi XY, Jiang YH, Tu QC, Xie JP, Van Nostrand JD, He ZL, Yang YF. Reproducibility and quantitation of amplicon sequencing-based detection. The ISME Journal, 2011, 5(8): 1303-1313. DOI:10.1038/ismej.2011.11 |
| [11] | Li R, Shen ZZ, Sun L, Zhang RF, Fu L, Deng XH, Shen QR. Novel soil fumigation method for suppressing cucumber Fusarium wilt disease associated with soil microflora alterations. Applied Soil Ecology, 2016, 101: 28-36. DOI:10.1016/j.apsoil.2016.01.004 |
| [12] | Zhu FY, Tian C, Zhang Y, Xiao JL, Wei L, Liang ZH. Effects of different fertilization treatments on soil microbial community structure and the occurrence of watermelon wilt. Chinese Journal of Biological Control, 2018, 34(4): 589-597. (in Chinese) 朱菲莹, 田程, 张屹, 肖姬玲, 魏林, 梁志怀. 不同施肥处理对土壤微生物群落结构及西瓜枯萎病发生的影响. 中国生物防治学报, 2018, 34(4): 589-597. |
| [13] | Li XG, Ding CF, Zhang TL, Wang XX. Fungal pathogen accumulation at the expense of plant-beneficial fungi as a consequence of consecutive peanut monoculturing. Soil Biology and Biochemistry, 2014, 72: 11-18. DOI:10.1016/j.soilbio.2014.01.019 |
| [14] | Faure D, Vereecke D, Leveau JHJ. Molecular communication in the rhizosphere. Plant and Soil, 2009, 321(1/2): 279-303. |
| [15] | Philipp O, Hamann A, Osiewacz HD, Koch I. The autophagy interaction network of the aging model Podospora anserina. BMC Bioinformatics, 2017, 18(1): 196. |
| [16] | Chemidlin Prevost-Boure N, Maron PA, Ranjard L, Nowak V, Dufrene E, Damesin C, Soudani K, Lata JC. Seasonal dynamics of the bacterial community in forest soils under different quantities of leaf litter. Applied Soil Ecology, 2011, 47(1): 14-23. DOI:10.1016/j.apsoil.2010.11.006 |
| [17] | Levy A, Salas Gonzalez I, Mittelviefhaus M, Clingenpeel S, Herrera Paredes S, Miao JM, Wang KR, Devescovi G, Stillman K, Monteiro F, Rangel Alvarez B, Lundberg DS, Lu TY, Lebeis S, Jin Z, McDonald M, Klein AP, Feltcher ME, Rio TG, Grant SR, Doty SL, Ley RE, Zhao BY, Venturi V, Pelletier DA, Vorholt JA, Tringe SG, Woyke T, Dangl JL. Genomic features of bacterial adaptation to plants. Nature Genetics, 2018, 50(1): 138-150. DOI:10.1038/s41588-017-0012-9 |
| [18] | Bulgarelli D, Rott M, Schlaeppi K, Ver Loren Van Themaat E, Ahmadinejad N, Assenza F, Rauf P, Huettel B, Reinhardt R, Schmelzer E, Peplies J, Gloeckner FO, Amann R, Eickhorst T, Schulze-Lefert P. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota. Nature, 2012, 488(7409): 91-95. DOI:10.1038/nature11336 |
| [19] | Ueki A, Kodama Y, Kaku N, Shiromura T, Satoh A, Watanabe K, Ueki K. Rhizomicrobium palustre gen. nov., sp. nov., a facultatively anaerobic, fermentative stalked bacterium in the class Alphaproteobacteria isolated from rice plant roots. The Journal of General and Applied Microbiology, 2010, 56(3): 193-203. DOI:10.2323/jgam.56.193 |
| [20] | Prakash O, Green SJ, Jasrotia P, Overholt WA, Canion A, Watson DB, Brooks SC, Kostka JE. Rhodanobacter denitrificans sp. nov., isolated from nitrate-rich zones of a contaminated aquifer. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(10): 2457-2462. |
| [21] | Mete E, Kaleli ?, Cevahir N, Demir M, Akkaya Y, Kiri? Sat?lm?? ?. Evaluation of virulence factors in enterococcus species. Mikrobiyoloji Bulteni, 2017, 51(2): 101-114. DOI:10.5578/mb.53992 |
| [22] | Beukers AG, Zaheer R, Goji N, Amoako KK, Chaves AV, Ward MP, McAllister TA. Comparative genomics of Enterococcus spp. isolated from bovine feces. BMC Microbiology, 2017, 17: 52. DOI:10.1186/s12866-017-0962-1 |
| [23] | Neumann B, Prior K, Bender JK, Harmsen D, Klare I, Fuchs S, Bethe A, Zühlke D, G?hler A, Schwarz S, Schaffer K, Riedel K, Wieler LH, Werner G. A core genome multilocus sequence typing scheme for Enterococcus faecalis. Journal of Clinical Microbiology, 2019, 57(3): e01686-18. |
| [24] | Ali SA, Bin-Asif H, Hasan KA, Rehman M, Abbasi A. Molecular assessment of virulence determinants, hospital associated marker (IS16 gene) and prevalence of antibiotic resistance in soil borne Enterococcus species. Microbial Pathogenesis, 2017, 105: 298-306. DOI:10.1016/j.micpath.2017.02.041 |
| [25] | Martzy R, Kolm C, Brunner K, Mach RL, Krska R, ?inkovec H, Sommer R, Farnleitner AH, Reischer GH. A loop-mediated isothermal amplification (LAMP) assay for the rapid detection of Enterococcus spp. in water. Water Research, 2017, 122: 62-69. DOI:10.1016/j.watres.2017.05.023 |
| [26] | Lee KE, Radhakrishnan R, Kang SM, You YH, Joo GJ, Lee IJ, Ko JH, Kim JH. Enterococcus faecium LKE12 cell-free extract accelerates host plant growth via gibberellin and indole-3-acetic acid secretion. Journal of Microbiology and Biotechnology, 2015, 25(9): 1467-1475. DOI:10.4014/jmb.1502.02011 |
| [27] | Kimoto-Nira H, Nagakura Y, Kodama C, Shimizu T, Okuta M, Sasaki K, Koikawa N, Sakuraba K, Suzuki C, Suzuki Y. Effects of ingesting milk fermented by Lactococcus lactis H61 on skin health in young women: a randomized double-blind study. Journal of Dairy Science, 2014, 97(9): 5898-5903. DOI:10.3168/jds.2014-7980 |
| [28] | Komano Y, Shimada K, Naito H, Fukao K, Ishihara Y, Fujii T, Kokubo T, Daida H. Efficacy of heat-killed Lactococcus lactis JCM 5805 on immunity and fatigue during consecutive high intensity exercise in male athletes: a randomized, placebo-controlled, double-blinded trial. Journal of the International Society of Sports Nutrition, 2018, 15: 39. DOI:10.1186/s12970-018-0244-9 |
| [29] | Goodman LB, Lawton MR, Franklin-Guild RJ, Anderson RR, Schaan L, Thachil AJ, Wiedmann M, Miller CB, Alcaine SD, Kovac J. Lactococcus petauri sp. nov., isolated from an abscess of a sugar glider. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(11): 4397-4404. DOI:10.1099/ijsem.0.002303 |
| [30] | Golomb BL, Marco ML. Lactococcus lactis metabolism and gene expression during growth on plant tissues. Journal of Bacteriology, 2015, 197(2): 371-381. |
| [31] | Alt?n G, Nikerel E, ?ahin F. Draft genome sequence of magnesium-dissolving Lactococcus garvieae A1, isolated from soil. Genome Announcements, 2017, 5(21): e00386-17. |
| [32] | Kojima H, Tokizawa R, Fukui M. Mizugakiibacter sediminis gen. nov., sp. nov., isolated from a freshwater lake. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(12): 3983-3987. |
| [33] | Watanabe T, Kojima H, Fukui M. Draft genome sequence of Mizugakiibacter sediminis skMP5T. Genome Announcements, 2015, 3(5): e01185-15. |
| [34] | Han S, Luo XS, Liao H, Nie HL, Chen WL, Huang QY. Nitrospira are more sensitive than Nitrobacter to land management in acid, fertilized soils of a rapeseed-rice rotation field trial. The Science of the Total Environment, 2017, 599-600: 135-144. DOI:10.1016/j.scitotenv.2017.04.086 |
| [35] | Daims H, Wagner M. Nitrospira. Trends in Microbiology, 2018, 26(5): 462-463. DOI:10.1016/j.tim.2018.02.001 |
| [36] | Daims H, Lebedeva EV, Pjevac P, Han P, Herbold C, Albertsen M, Jehmlich N, Palatinszky M, Vierheilig J, Bulaev A, Kirkegaard RH, Von Bergen M, Rattei T, Bendinger B, Nielsen PH, Wagner M. Complete nitrification by Nitrospira bacteria. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461 |
| [37] | Kim JJ, Alkawally M, Brady AL, Rijpstra WIC, Sinninghe Damsté JS, Dunfield PF. Chryseolinea serpens gen. nov., sp. nov., a member of the phylum Bacteroidetes isolated from soil. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(2): 654-660. |
| [38] | Wang JJ, Chen Q, Li YZ. Chryseolinea flava sp. nov., a new species of Chryseolinea isolated from soil. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(11): 3518-3522. DOI:10.1099/ijsem.0.003022 |
| [39] | Falagán C, Johnson DB. Acidibacter ferrireducens gen. nov., sp. nov.: an acidophilic ferric iron-reducing gammaproteobacterium. Extremophiles, 2014, 18(6): 1067-1073. DOI:10.1007/s00792-014-0684-3 |
