卓娜1, 伊丽1,2, 浩斯娜1, 吉日木图1,2


1. 内蒙古农业大学 乳制品生物技术与工程教育部重点实验室, 内蒙古 呼和浩特 010018;
2. 内蒙古骆驼研究院, 内蒙古 巴丹吉林 737300
收稿日期:2018-10-31;修回日期:2019-01-17;网络出版日期:2019-03-13
基金项目:双一流学科创新团队建设(NDSC2018-14);内蒙古自治区科技创新引导项目(KCMS2018048);内蒙古自治区科技重大项目;内蒙古自然科学基金(2018BS03017);高层次人才引进科研启动项目(NDY2017-28)
*通信作者:吉日木图, Tel/Fax:+86-471-3968532; E-mail:yeluotuo1999@vip.163.com.
摘要:[目的] 传统发酵乳制品是一类未经任何处理自然发酵而成的,其微生态环境未遭破坏,从而乳酸菌的生物学特性和基因多样性得到了很好的保留,具有开发和利用价值。自然发酵酸驼乳常用来治疗多种疾病且效果良好,与其中丰富的乳酸菌资源有着密不可分的联系。然而,目前有关自然发酵酸驼乳微生物菌群及多样性相关研究甚少。因此进一步挖掘内蒙古地区双峰驼自然发酵酸驼乳微生物群落结构和多样性是至关重要的。[方法] 本研究采用Illumina Miseq测序技术,测定了苏尼特和阿拉善双峰驼的自然发酵酸驼乳中微生物16S rRNA Ⅴ3-Ⅴ4区序列,并对群落结构和多样性进行了比较分析。[结果] 多样性分析表明,苏尼特双峰驼酸驼乳中微生物群落丰富度和种群差异性比阿拉善双峰驼酸驼乳大,细菌多样性也高。在门水平上,苏尼特和阿拉善双峰驼酸驼乳中的菌群均以厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为主。在属水平上,苏尼特双峰驼酸驼乳主要以乳杆菌属(Lactobacillus)和乳球菌属(Lactococcus)为优势菌群,阿拉善双峰驼酸驼乳以乳杆菌属(Lactobacillus)和醋酸杆菌属(Acetobacter)为优势菌属。此外,肠杆菌属(Enterobacter)、拉乌尔菌属(Raoultella)和明串珠菌属(Leuconostoc)等的含有食源性致病菌和环境污染菌的菌属被检出。综上所述,不同地区不同品种酸驼乳的乳酸菌种类及优势菌群有较大差异,存在显著的地理差异。[结论] 通过本研究,不仅对苏尼特和阿拉善双峰驼自然发酵酸驼乳乳酸菌的组成和种类有了明确的认知,为评估发酵酸驼乳微生物群落对消费者身体健康的影响提供了数据基础的同时为今后筛选优势菌群和挖掘新型益生菌奠定基础。
关键词:骆驼乳微生物多样性16S rRNA基因序列分析法
Application of 16S rRNA high-throughput sequencing for comparative study of the microbial diversity of traditional fermented bactrian camel milk from Alxa bactrian camel and Sonid bactrian camel
Na Zhuo1, Li Yi1,2, Sina Hao1, Tu Jirimu1,2


1. Key Laboratory of Dairy Biotechnology and Bioengineering, Ministry of Education, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia Autonomous Region, China;
2. Inner Mongolia Institute of Camel Research, Badanjiran 737300, Inner Mongolia Autonomous Region, China
Received: 31 October 2018; Revised: 17 January 2019; Published online: 13 March 2019
*Corresponding author: Tu Jirimu, Tel/Fax: +86-471-3968532; E-mail: yeluotuo1999@vip.163.com.
Foundation item: Supported by the Double-class Discipline Innovation Team Building Project (NDSC2018-14), by the Inner Mongolia Science and Technology Innovation Guide Project (KCMS2018048), by the Inner Mongolia Science and Technology Major Project, by the Natural Science Foundation of Inner Mongolia (2018BS03017) and by High-level Scientific Research Foundation for the Introduction of Talent (NDY2017-28)
Abstract: [Objective] Traditional fermented camel milk is a valuable source of food for people living in the arid areas, and it is often used to treat a variety of disease with good effects. Its nutritional and medical functions are closely related to its abundant lactic acid bacteria. However, the studies of bacterial diversity of fermented camel milk are scattered and the amount of available data are still limited. Therefore, it is necessary to explore the microbiota composition and diversity of the traditional fermented camel milk. [Methods] We sequenced the Ⅴ3-Ⅴ4 region of 16S rRNA genes in fermented camel milk of Alxa and Sonid Bactrian camel by Illumina Miseq platform, and the microbiota composition and diversity were compared and analyzed. [Results] The results of Alpha diversity showed that the fermented camel milk from Sonid has higher bacterial diversity and microbial community richness than those of Alxa. At the phylum level, Firmicutes and Proteobacteria were dominant in both of them. At the genus level, Lactobacillus and Lactococcus were dominant in Sonid samples, Lactobacillus and Acetobacter were dominant in Alxa samples. In addition, we also found some foodborne pathogens and environmentally contaminating bacteria, such as Enterobacter, Raoultella and Leuconostoc. There were significant differences in the types and dominant bacteria in fermented camel milk in different regions. [Conclusion] These results provide insights into the microbiota community and diversity of traditional fermented camel milk, and also lay the foundation for screening dominant bacteria and excavating new probiotics in the further studies.
Keywords: camel milkmicrobial diversity16S rRNA high-throughput sequencing
驼乳是荒漠地区游牧人民的主要乳源之一。驼乳与人乳相似,其乳糖和胆固醇含量较低,矿物质和维生素C含量高,且含有丰富的溶菌酶、乳铁蛋白、免疫球蛋白等保护性蛋白[1]。驼乳同人乳一样含有高浓度的α-乳白蛋白、乳铁蛋白和β-酪蛋白,不含致敏因子β-乳球蛋白[2];驼乳脂肪球颗粒小易于消化吸收。驼乳具有自然酸度高的特点,平均为22°T (牛奶为16–18°T,马奶为6.5°T)[3],酸化后可长期保存不腐败,因此当地牧民也常将驼乳加工为发酵乳饮用。发酵酸驼乳风味独特,营养丰富,具有调节胃肠功能、提高人体免疫力、降血压、降血脂等保健功效,同时对肺结核、风湿、神经性疾病、血液病等疾病具有良好的治疗作用,是食疗保健的最佳饮品[4]。发酵食品在发酵过程中有多种微生物参与,将原料中的蛋白质分解为氨基酸和多肽,脂肪分解为短链的挥发脂肪酸和酯类物质,微生物的代谢产物与原料分解产物共同形成了发酵食品特有的风味和质构特征品质[5]。环境的差异和宿主动物的来源不同直接影响着原奶的微生物群落结构组成;而原奶加工乳制品中微生物组不仅受到环境条件、来源动物的影响,也受到乳品加工过程的影响[6-7]。在气候独特条件下生存的骆驼,其分泌的乳汁,乃至其发酵制品,都蕴藏着丰富的微生物资源(自然环境中的乳酸菌资源),极有可能成为优良益生菌的来源。
随着分子生物学技术的不断发展,新的技术——高通量测序技术以其高读长、精度高、通量高、无偏性的自身优势被广泛应用研究微生物的群落多样性[8]。近年来逐步建立起来的16S rRNA基因序列分析法,通过提取样品中微生物的总DNA,直接从核酸水平对样品进行分析研究,避免了样品中微生物的丢失,而样品序列的多样性和不同序列的丰度在一定程度上反映了原始样品中微生物种群的多样性和丰度[9]。到目前为止,以16S rRNA对发酵酸驼乳进行全面的微生物多样性的研究较少,因此开展骆驼乳微生物组的研究将非常必要。
本研究的目的是通过采集内蒙古不同地区自然发酵酸驼乳,采用分子生物学技术16S rRNA基因序列分析法对酸驼乳的微生物群落多样性和构成进行比较分析,更加全面地展现内蒙古地区骆驼乳制品的微生物多样性的同时为今后筛选优势菌群和挖掘新型益生菌奠定基础。
1 材料和方法 1.1 材料与试剂 E.Z.N.A.?soil试剂盒(Omega Bio-tek,Norcross,GA,U.S.),AxyPrep DNA凝胶回收试剂盒(AXYGEN公司),Tris-HCl缓冲液,50 mL冻存管,液氮。
1.2 仪器与设备 PCR仪:ABI GeneAmp? 9700型,Eppendorf AG 22331 Hamburg高速冷冻离心机:德国艾本德,QuantiFluorTM-ST蓝色荧光定量系统:Promega公司。
1.3 样品采集 采用人工采集法,从内蒙古锡林郭勒盟苏尼特右旗(Sonid,S),阿拉善盟阿拉善左旗(Alxa,A)自然放牧地区收集以成年双峰驼驼乳自然发酵而成的酸驼乳,每一个样品采集50 mL于无菌无酶冻存管中,所有样品立即放入液氮速冻,之后转移至–80 ℃冰箱待用。共计16份样品,均采自牧民家,详细情况见表 1。
表 1. 样品及其采集地 Table 1. Samples and collection place
| Sample type | Sample number | Collection place |
| Fermented camel milk | A1 | Alxa Left Banner Aolunbulage |
| Fermented camel milk | A2 | Alxa Left Banner Aolunbulage |
| Fermented camel milk | A3 | Alxa Left Banner Bayanhaote |
| Fermented camel milk | A4 | Alxa Left Banner Aolunbulage |
| Fermented camel milk | A5 | Alxa Left Banner Barunbieli |
| Fermented camel milk | A6 | Alxa Left Banner Bayanhaote |
| Fermented camel milk | A7 | Alxa Left Banner Bayanhaote |
| Fermented camel milk | A8 | Alxa Left Banner Jilantai |
| Fermented camel milk | A10 | Alxa Left Banner Bayanhaote |
| Fermented camel milk | S1 | Sonid Right Banner Erennaoer |
| Fermented camel milk | S2 | Sonid Right Banner Saihantala |
| Fermented camel milk | S5 | Sonid Right Banner Aqituwula |
| Fermented camel milk | S6 | Sonid Right Banner Saihantala |
| Fermented camel milk | S7 | Sonid Right Banner Aqituwula |
| Fermented camel milk | S8 | Sonid Right Banner Aqituwula |
| Fermented camel milk | S9 | Sonid Right Banner Erennaoer |
表选项
1.4 总DNA提取 根据E.Z.N.A.?soil试剂盒(Omega Bio-tek,Norcross,GA,U.S.)说明书进行总DNA抽提,DNA浓度和纯度利用NanoDrop2000进行检测,利用1%琼脂糖凝胶电泳检测DNA提取质量。
1.5 16S rRNA Ⅴ3-Ⅴ4区的PCR扩增及测序 用提取的总DNA作为模板,以338F (5′-ACTCCTACGGGAGGCAGGAG-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′)为引物,对细菌的16S rRNA Ⅴ3-Ⅴ4可变区进行扩增。扩增体系(20 μL):4 μL 5×缓冲液,2 μL的2.5 mmol/L dNTPs,引物(5 μmol/L)均为0.8 μL,0.4 μL的FastPfu聚合酶,0.2 μL BSA,10 ng模板DNA,补充ddH2O至20 μL。PCR反应参数:a. 1×(3 min 95 ℃);b.循环数×(30 s 95 ℃;30 s退火温度℃;45 s 72 ℃);c. 10 min 72 ℃,10 ℃保存user。利用Illumina公司的Miseq PE300平台进行测序。PCR扩增及测序工作由上海美吉生物医药科技有限公司完成。
1.6 数据分析 原始测序序列使用Trimmomatic软件质控,使用FLASH软件进行拼接。然后用Usearch (vsesion 7.0)软件得到干净的序列,根据97%的相似度对序列进行OTU聚类;使用UCHIME软件剔除嵌合体。利用RDP classifier (http://rdp.cme.msu.edu/)对每条序列进行物种分类注释,比对Silva数据库(Release128)。比较分析样品中微生物群落结构的结构及组成。
2 结果和分析 2.1 样品序列数的特点 通过细菌的16S rRNA Ⅴ3-Ⅴ4区的测序发现,16份自然发酵酸驼乳样本共得到高质量质控序列846974个序列标签,平均长度为447 bp。所有序列按97%的相似度进行OTU分类,总共聚成418个OTU,详细情况见表 2。通过统计OTU得到了域1、界1、门18、纲36、目78、科132、属236、种330的结果。
表 2. 酸驼乳样品的信息和序列丰度 Table 2. Information and sequence abundance of fermented camel milk samples
| Group | Raw reads | OTU | Coverage/% |
| A1 | 51409 | 31 | 99.97 |
| A2 | 47401 | 25 | 99.97 |
| A3 | 44061 | 60 | 99.95 |
| A4 | 51368 | 45 | 99.98 |
| A5 | 44739 | 92 | 99.98 |
| A6 | 43784 | 122 | 99.94 |
| A7 | 49621 | 65 | 99.96 |
| A8 | 66950 | 47 | 99.96 |
| A10 | 43978 | 39 | 99.98 |
| S1 | 52123 | 53 | 99.97 |
| S2 | 56663 | 37 | 99.97 |
| S5 | 51682 | 146 | 99.95 |
| S6 | 70823 | 58 | 99.95 |
| S7 | 48508 | 78 | 99.96 |
| S8 | 57790 | 58 | 99.97 |
| S9 | 66074 | 290 | 99.98 |
表选项
丰度等级曲线可以反映样品中所含物种的丰富程度和均匀程度。根据图 1可知,A组A2样本曲线跨度较小,曲线下降陡峭,说明OTU之间的丰度差异较大,均匀度越低,细菌的组成较单一而且其优势菌种明显。而S组S9的曲线跨度较大,曲线平坦,下降缓慢,说明S9物种的组成较丰富且物种组成的均匀度较高,相比其他样品其优势的菌种不明显。A组的9个样本分布较集中,S组7个样本中的5个样本聚集在一起。
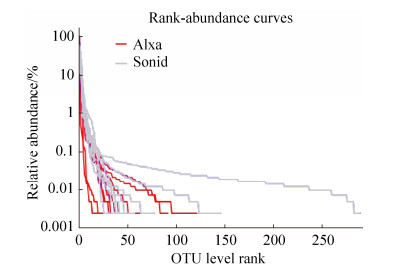 |
| 图 1 酸驼乳微生物的丰度等级曲线 Figure 1 Rank-Abundance curve of fermented camel milk samples. |
| 图选项 |
2.2 酸驼乳中细菌群落的多样性分析
2.2.1 Alpha多样性分析: Shannon、Simpson、Ace和Chao1指数是用于反映样本微生物群落的丰富度和多样性的参数。通过图 2可知,S组的Shannon指数显著高于A组(P≤0.001),而A组的Simpson指数显著高于S组(P≤0.001)。S组的ACE和Chao1指数尽管高于A组,但是不显著(P > 0.05)。这说明两组酸驼乳微生物群落丰富度和种群有差异,其中S组微生物群落丰富度和种群差异性较大,细菌多样性高。
 |
| 图 2 酸驼乳微生物多样性指数 Figure 2 Alpha diversity index of fermented camel milks amples. *: 0.01 < P≤0.05; **: 0.001 < P≤0.01; ***: P≤0.001. |
| 图选项 |
2.2.2 Beta多样性分析: Beta多样性用于比较不同样本微生物群落构成的差异。距离矩阵表示群落中每2个样本间的差异。由图 3可知,16个样本共聚为两大分支。S组7个样本之间的细菌群落结构相似性较高,共同聚集在上方。A组9个样本之间的细菌群落结构相似性较高,聚集在下方,说明2个品种的双峰驼酸驼乳微生物群落结构存在地理分布差异。
 |
| 图 3 酸驼乳细菌群落聚类分析图 Figure 3 Clustering analysis of fermented camel milk bacterial community. |
| 图选项 |
对样品间的距离矩阵进行主成分分析,作出Beta多样性的三维主坐标分析(PCoA)图,如图 4所示。主坐标分析发现其第一主成分、第二主成分和第三成分的贡献率分别为61.43%、12.85%和8.74%,其中可以明显看出A组的9个样本聚集在一起,距离较近,说明阿拉善双峰驼酸驼乳组的样本之间的微生物组成结构较为相似。而S组的7个样本聚集的距离较为分散,分为2个分布区,每个区内的样本聚集较近,说明S组的样本之间的微生物组成结构也有所不同。该结果进一步说明,2个品种的双峰驼酸驼乳微生物存在较大的差异。
 |
| 图 4 酸驼乳细菌群落结构的三维主坐标分析 Figure 4 3D principal co-ordinates analysis of bacterial community structure of fermented camel milk samples. |
| 图选项 |
2.3 样品群落组成分析
2.3.1 物种Venn图分析: Venn图分析可以比较直观地表现环境样本在不同的分类水平上的组成相似性及重叠情况。如图 5所示,重叠部分表示2个品种的双峰驼酸驼乳组中共有的物种,有169个物种。没有重叠的部分表示2个品种的双峰驼酸驼乳组所特有的物种,S组有221个物种,A组有28个物种。因此,S组总的物种有390个,A组总的物种有197个。
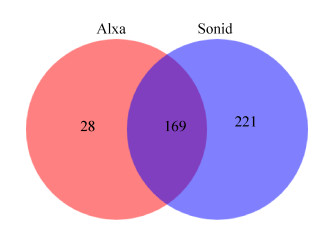 |
| 图 5 酸驼乳物种Venn图 Figure 5 Venn diagrams of bacterial OTUs in two groups of fermented camel milk samples. |
| 图选项 |
2.3.2 细菌门分类水平的比较: 采用QIIME软件分析各样品在门分类水平上菌群的组成和结构(图 6),共鉴定出5个细菌门,分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)和Others。16个样品均有分布且相对丰度较高的菌门为厚壁菌门和变形菌门,其中A组的2个菌门分别占细菌总序列的88.50%和11.32%,S组的分别为80.17%和18.30%。由此可知,2个不同品种的发酵酸驼乳的优势菌群门均为厚壁菌门和变形菌门。
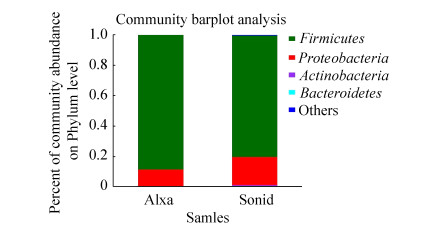 |
| 图 6 酸驼乳样本门分类水平菌群分布图 Figure 6 Bacteria community structure of different samples in phylum. |
| 图选项 |
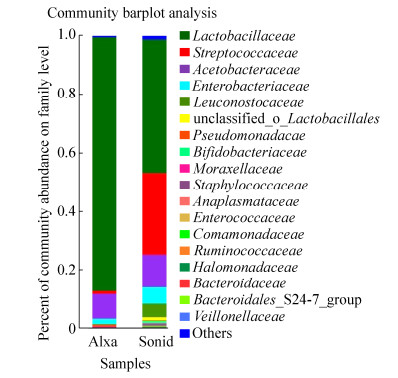 |
| 图 7 酸驼乳样本科分类水平菌群分布图 Figure 7 Bacteria community structure of different samples in family. |
| 图选项 |
2.3.3 细菌科分类水平的比较: 科水平上共得到了19个细菌科,其中丰度最高的前10个细菌科是乳杆菌科(Lactobacillaceae)、链球菌科(Streptococcaceae)、乙酰杆菌科(Acetobacteraceae)、肠杆菌科(Enterobacteriaceae)、明串珠菌科(Leuconostocaceae)、unclassified-o-Lactobacillales、假单胞菌科(Pseudomonadaceae)、双歧菌科(Bifidobacteriaceae)、鼠尾草科(Moraxellaceae)和葡萄球菌科(Staphylococcaceae)。其中A组的优势菌科是乳酸菌科和乙酰杆菌科,分别占细菌总序列的87.02%和8.48%。而S组的优势菌科为乳酸菌科和链球菌科,分别占细菌总序列的45.44%和28.29%。
2.3.4 细菌属分类水平的比较: 属水平上共得到了23个,其中丰度最高的前15个细菌属分别是乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、醋酸杆菌属(Acetobacter)、肠杆菌属(Enterobacter)、明串珠菌属(Leuconostoc)、unclassified-o- Lactobacillales、假单胞菌属(Pseudomonas)、双歧杆菌属(Bifidobacterium)、拉乌尔菌属(Raoultella)、不动杆菌属(Acinetobacter)、巨型球菌属(Macrococcus)、沃尔巴克氏体属(Wolbachia)、沙雷氏菌属(Serratia)、葡糖杆菌属(Gluconobacter)和链球菌属(Streptococcus)。16个样品均有分布且相对丰度较高的细菌属分别是乳杆菌属、乳球菌属、醋酸杆菌属和肠杆菌属。其中S组的优势菌属是乳杆菌属和乳球菌属,分别占45.33%和28.02%。而A组的优势菌属为乳杆菌属和醋酸杆菌属,分别占87.02%和8.26%。
2.3.5 聚类热图(Heatmap图)分析: Heatmap图是以颜色梯度来表征二维矩阵或表格中的数据大小,呈现群落物种组成信息,反映不同分组或样本在各分类水平上群落组成的相似性和差异性。从Heatmap聚类分析的结果(图 9)可以看出,在属水平上,A组和S组的Lactobacillus、Lactococcus、Acetobacter、Enterobacter、Leuconostoc、Pseudomonas、Bifidobacterium等属所占比例较大,即相对丰度高。该结果印证了Beta多样性分析中属水平的结果(图 8)。
 |
| 图 8 酸驼乳样本属分类水平菌群分布图 Figure 8 Bacteria community structure of different samples in genus. |
| 图选项 |
 |
| 图 9 含系统发育进化树的热图(属水平) Figure 9 Heatmap graph with phylogenetic tree (genus level). |
| 图选项 |
 |
| 图 10 酸驼乳微生物科分类水平上的Wilcoxon秩和检验 Figure 10 Wilcoxon rank-sum test or Mann-Whitney U test on the family level of fermented camel milk samples. |
| 图选项 |
 |
| 图 11 酸驼乳微生物属分类水平上的Wilcoxon秩和检验 Figure 11 Wilcoxon rank-sum test or Mann-Whitney U test on the genus level of fermented camel milk samples. |
| 图选项 |
2.4 组间差异分析 组间差异显著性检验根据得到的群落丰度数据,运用严格的统计学方法可以检测不同组或样本微生物群落中表现出的丰富度差异的物种,进行假设性检验,评估观察到的差异的显著性。Wilcoxon秩和检验结果显示,在科分类水平上2个品种的双峰驼酸驼乳样本在乳杆菌科(Lactobacillaceae)、链球菌科(Streptococcaceae)、间孢囊菌科(Intrasporangiaceae)、皮杆菌科(Dermabacteraceae)和草酸杆菌科(Oxalobacteraceae)、Family_XI_o_Clostridiales上有显著性差异(0.01 < P≤0.05)。其中A组各科所占的相对丰度依次为87.0200%、1.0240%、0%、0%、0%和0%,而S组各科所占的相对丰度依次为45.3300%、28.2900%、0.0080%、0.0067%、0.0060%和0.0027%。
在属分类水平上的检验说明两个品种的双峰驼酸驼乳样本在乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、Ruminococcaceae_UCG-005、Prevotellaceae-NK3B31-group、诺尔士菌属(Knoellia)、短状杆菌属(Brachybacterium)、Pseudarthrobacter、盐水球菌属(Salinicoccus)、马赛菌属(Massilia)和Parasutterella有显著性差异(0.01 < P≤0.05,0.001 < P≤0.01)。其中A组各属所占的相对丰度依次为87.0200%、0.9666 %、0%、0%、0%、0%、0%、0%、0%和0%。,而S组各属所占的相对丰度依次为45.3300%、28.0200%、0.0211%、0.0080%、0.0074%、0.0067%、0.0064%、0.0057%、0.0050%和0.0047%。
3 讨论 内蒙古传统发酵乳制品是牧民以牛、马、骆驼、山羊、绵羊乳为原料,不添加任何食品发酵剂,借助自然环境发酵而成的乳制品,其蕴含了大量的微生物资源,而由于地理条件、气候环境及制作工艺等因素影响,不同地区的传统发酵酸驼乳中微生物组成具有较大差异。阿拉善牧民制作酸驼乳的方法是将制作奶皮取掉的熟奶或生奶盛入打酥油的木桶里,放2–3 d(冬天放热处),也可加少许黄米(起发酵作用),驼乳发酵变酸,用特制的木柞反复捣2 h左右把酥油分离出来,木桶中留下的稀一些的酸奶就叫“艾日格”,属于搅拌发酵[10]。而苏尼特发酵酸驼乳的制作方法属于静置发酵,将新鲜驼乳不经过消毒直接放入专用的木桶放置室内发酵(温度一般为20 ℃),酸驼乳被饮用后再补充适当体积的新鲜驼乳。为了揭示不同地区、不同环境和不同方法制作的传统发酵酸驼乳微生物差异,本研究采用高通量测序对阿拉善和苏尼特双峰驼自然发酵酸驼乳中的微生物组成和多样性进行了测定。
通过研究发现,苏尼特双峰驼和阿拉善双峰驼自然发酵酸驼乳的细菌多样性、微生物群落组成各不相同。S组展示了较高的物种丰度,其种群差异性较大,细菌多样性高,与A组微生物群落结构存在显著的地理分布差异。在门水平上,2个不同品种的双峰驼酸驼乳的优势菌门均为厚壁菌门和变形菌门,但2个细菌门的丰度不同,该结果与前人的研究结果相一致[11-13],其中张敏等[12]的研究结果表明酸驼乳中的厚壁菌门的丰度为89.96%–98.00%,变形菌门为17.20%,Fugl等[11]的结果中分别为85%和15%。该结果也与酸牛乳、酸马乳、发酵牦牛乳的研究结果相一致[14-17]。在属水平上,S组的优势菌属是乳杆菌属和乳球菌属,而A组的优势菌属为乳杆菌属和醋酸杆菌属,其中乳杆菌属为两组最优势的菌属。大多数发酵乳中的微生物均以乳杆菌属、乳球菌属、肠球菌属等菌属为主[18-20],其中乳杆菌属作为优势菌属可能是因为其具有较强的耐酸性造成的,乳杆菌属的耐酸性比乳球菌属和链球菌属都高[21]。张敏等[12]、张哲等[22]的研究表明,酸驼乳中的乳杆菌属的丰度分别为82.05%、60.13%,这与本研究结果相一致。醋酸杆菌属适于在偏高的温度下生长,且醋酸杆菌属含有乙醇脱氢酶具有很强的将乙醇氧化成乙酸的能力。与苏尼特地区相比,阿拉善地区属温带荒漠地区,干旱少雨,日常温度较高,由此可推断阿拉善自然发酵酸驼乳中醋酸杆菌属丰度较高与其环境有着一定的关系。另外,通过测定两个地区自然发酵酸驼乳pH值发现,阿拉善地区酸驼乳的pH值为3.60,苏尼特酸驼乳的pH值为4.03,可进一步推断是由阿拉善酸驼乳中大量的醋酸杆菌属将乙醇氧化为乙酸导致酸度增加。
此外,本次研究中也有肠杆菌属、明串珠菌属、假单胞菌属、拉乌尔菌属、不动杆菌属、链球菌属等被鉴定出。上述菌属里有食源性致病菌和环境污染菌,这些属在Delbes等[23]和张敏等[12]的研究报道中也出现过。A组主要以肠杆菌属(Enterobacter)和拉乌尔菌属(Raoultella)为主,S组以肠杆菌属(Enterobacter)和明串珠菌属(Leuconostoc)为主。发酵食品中微生物种类多,同时细菌源性的污染危害着食品的安全,对人体健康也带来了潜在的安全隐患。这些污染可能与原乳中的细菌污染有关。原乳中的微生物一般是来自不同动物的乳房、不同的农场和挤奶环境、与奶直接接触的挤奶工人和容器[24]。另一种可能是采样时的手法操作不规范或运输样本等出现的现象。
4 结论 本研究对内蒙古苏尼特和阿拉善双峰驼自然发酵酸驼乳共16份样本以16S rRNA基因序列分析法进行群落微生物多样性研究发现,2个不同品种的双峰驼酸驼乳微生物优势菌门均为厚壁菌门和变形菌门。苏尼特双峰驼酸驼乳以乳杆菌属和乳球菌属为优势菌属,而阿拉善双峰驼酸驼乳以乳杆菌属和醋酸杆菌属为优势菌属。这说明两个不同品种的酸驼乳微生物群落结构存在显著的地理分布差异。此外,也检测到了食源性致病菌和环境污染菌的存在。该研究对苏尼特和阿拉善双峰驼自然发酵酸驼乳的微生物多样性和乳酸菌的组成种类有了明确的认知,为评估发酵酸驼乳微生物群落对消费者身体健康的影响提供了数据基础的同时为今后筛选优势菌群和挖掘新型益生菌奠定基础。
References
| [1] | Yadav AK, Kumar R, Priyadarshini L, Singh J. Composition and medicinal properties of camel milk: a review. Asian Journal of Dairy and Food Research, 2015, 34(2): 83-91. DOI:10.5958/0976-0563.2015.00018.4 |
| [2] | Ehlayel M, Bener A, Abu Hazeima K, Al-Mesaifri F. Camel milk is a safer choice than goat milk for feeding children with cow milk allergy. ISRN Allergy, 2011, 2011: 391641. |
| [3] | Nie XM. Research on the key technology of acid camel milk producing. Master Dissertation of Xinjiang Agricultural University, 2008. (in Chinese) 聂小苗.酸驼乳加工关键技术研究.新疆农业大学硕士学位论文, 2008. http://cdmd.cnki.com.cn/Article/CDMD-10758-2008066432.htm |
| [4] | He JX, Hasisurong, Narenbatu, Jirimutu. Research progress in health care function of camel milk. Food Science, 2009, 30(23): 504-507. (in Chinese) 何俊霞, 哈斯苏荣, 那仁巴图, 吉日木图. 驼乳医疗保健作用的研究进展. 食品科学, 2009, 30(23): 504-507. DOI:10.3321/j.issn:1002-6630.2009.23.113 |
| [5] | Shu C, Wu CS, Zhong CP, Suo HY, Li L. Progress of research methods for microbial diversity in fermented foods. Food Science, 2013, 34(15): 397-402. (in Chinese) 舒畅, 吴春生, 钟慈平, 索化夷, 李林. 发酵食品微生物多样性研究方法进展. 食品科学, 2013, 34(15): 397-402. DOI:10.7506/spkx1002-6630-201315076 |
| [6] | Devirgiliis C, Zinno P, Perozzi G. Update on antibiotic resistance in foodborne Lactobacillus and Lactococcus species. Frontiers in Microbiology, 2013, 4: 301. |
| [7] | Cavanagh D, Fitzgerald GF, McAuliffe O. From field to fermentation: the origins of Lactococcus lactis and its domestication to the dairy environment. Food Microbiology, 2015, 47: 45-61. DOI:10.1016/j.fm.2014.11.001 |
| [8] | Mi QL, Li XM, Guan Y, Gao Q, Gui YF, Zhu ZH, Yao JH. Application of high-throughput sequencing in food microbial ecology: a review. Food Science, 2016, 37(23): 302-308. (in Chinese) 米其利, 李雪梅, 管莹, 高茜, 桂永发, 朱洲海, 夭建华. 高通量测序在食品微生物生态学研究中的应用. 食品科学, 2016, 37(23): 302-308. DOI:10.7506/spkx1002-6630-201623049 |
| [9] | Li DP, Guo MZ, Xu WT. Advances and applications on methodology of 16S rRNA sequencing in gut microbiota analysis. Biotechnology Bulletin, 2015, 31(2): 71-77. (in Chinese) 李东萍, 郭明璋, 许文涛. 16S rRNA测序技术在肠道微生物中的应用研究进展. 生物技术通报, 2015, 31(2): 71-77. |
| [10] | Lang XH, Chen ZM. Alxa bactrian camel milk products traditional production process. Animal Husbandry and Feed Science, 2010, 31(8): 81-82, 85. (in Chinese) 郎小红, 陈泽明. 阿拉善双峰驼奶制品传统制作工艺. 畜牧与饲料科学, 2010, 31(8): 81-82, 85. DOI:10.3969/j.issn.1672-5190.2010.08.039 |
| [11] | Fugl A, Berhe T, Kiran A, Hussain S, Laursen MF, Bahl MI, Hailu Y, S rensen KI, Guya ME, Ipsen R, Hansen EB. Characterisation of lactic acid bacteria in spontaneously fermented camel milk and selection of strains for fermentation of camel milk. International Dairy Journal, 2017, 73: 19-24. DOI:10.1016/j.idairyj.2017.04.007 |
| [12] | Zhang M, Zhang Y, Huang LL, Liu YD, Zhou H, Ni YQ. Application of 16S rDNA high-throughput sequencing for comparative study of the microbial diversity of dairy products from western and northern Xinjiang, China. Food Science, 2017, 38(20): 27-33. (in Chinese) 张敏, 张艳, 黄丽丽, 刘忆冬, 周红, 倪永清. 基于16S rDNA高通量测序方法比较新疆西北部地区乳品中微生物的多样性. 食品科学, 2017, 38(20): 27-33. DOI:10.7506/spkx1002-6630-201720005 |
| [13] | Shangpliang HNJ, Rai R, Keisam S, Jeyaram K, Tamang JP. Bacterial community in naturally fermented milk products of Arunachal Pradesh and Sikkim of India analysed by high-throughput amplicon sequencing. Scientific Reports, 2018, 8(1): 1532. DOI:10.1038/s41598-018-19524-6 |
| [14] | Chen HC, Wang SY, Chen MJ. Microbiological study of lactic acid bacteria in kefir grains by culture-dependent and culture-independent methods. Food Microbiology, 2008, 25(3): 492-501. DOI:10.1016/j.fm.2008.01.003 |
| [15] | Quigley L, O'Sullivan O, Beresford TP, Ross RP, Fitzgerald GF, Cotter PD. High-throughput sequencing for detection of subpopulations of bacteria not previously associated with artisanal cheeses. Applied and Environmental Microbiology, 2012, 78(16): 5717-5723. DOI:10.1128/AEM.00918-12 |
| [16] | Xu HY, Liu WJ, Gesudu Q, Sun ZH, Zhang JC, Guo Z, Zheng Y, Hou QC, Yu J, Qing YT, Kwok LY, Menhe B, Zhang HP. Assessment of the bacterial and fungal diversity in home-made yoghurts of Xinjiang, China by pyrosequencing. Journal of the Science of Food and Agriculture, 2015, 95(10): 2007-2015. DOI:10.1002/jsfa.6912 |
| [17] | Liu WJ, Xi XX, Sudu Q, Kwok L, Guo Z, Hou QC, Menhe B, Sun TS, Zhang HP. High-throughput sequencing reveals microbial community diversity of Tibetan naturally fermented yak milk. Annals of Microbiology, 2015, 65(3): 1741-1751. DOI:10.1007/s13213-014-1013-x |
| [18] | Federici S, Ciarrocchi F, Campana R, Ciandrini E, Blasi G, Baffone W. Identification and functional traits of lactic acid bacteria isolated from Ciauscolo salami produced in Central Italy. Meat Science, 2014, 98(4): 575-584. DOI:10.1016/j.meatsci.2014.05.019 |
| [19] | Henríquez-Aedo K, Durán D, Garcia A, Hengst MB, Aranda M. Identification of biogenic amines-producing lactic acid bacteria isolated from spontaneous malolactic fermentation of chilean red wines. LWT-Food Science and Technology, 2016, 68: 183-189. DOI:10.1016/j.lwt.2015.12.003 |
| [20] | Nero LA, de Mattos MR, Barros Mde A, Ortolani MB, Beloti V, Franco BD. Listeria monocytogenes and Salmonella spp. in raw milk produced in Brazil: occurrence and interference of indigenous microbiota in their isolation and development. Zoonoses Public Health, 2008, 55(6): 299-305. DOI:10.1111/j.1863-2378.2008.01130.x |
| [21] | Bao QH, Liu WJ, Yu J, Wang WH, Qing MJ, Chen X, Wang F, Zhang JC, Zhang WY, Qiao JM, Sun TS, Zhang HP. Isolation and identification of cultivable lactic acid bacteria in traditional yak milk products of Gansu Province in China. The Journal of General and Applied Microbiology, 2012, 58(2): 95-105. DOI:10.2323/jgam.58.95 |
| [22] | Zhang Z, Liu XM, Chen W. Isolation and identification of lactic acid bacteria and yeast and biodiversity analysis of traditional fermented camel milk in Inner Mongolia. Journal of Chinese Institute of Food Science and Technology, 2018, 18(7): 230-238. (in Chinese) 张哲, 刘小鸣, 陈卫. 内蒙古传统发酵驼乳中乳酸菌和酵母菌的分离鉴定及其生物多样性分析. 中国食品学报, 2018, 18(7): 230-238. |
| [23] | Delbès C, Ali-Mandjee L, Montel MC. Monitoring bacterial communities in raw milk and cheese by culture-dependent and -independent 16S rRNA gene-based analyses. Applied and Environmental Microbiology, 2007, 73(6): 1882-1891. DOI:10.1128/AEM.01716-06 |
| [24] | Oliver SP, Jayarao BM, Almeida RA. Foodborne pathogens in milk and the dairy farm environment: food safety and public health implications. Foodborne Pathogens and Disease, 2005, 2(2): 115-129. DOI:10.1089/fpd.2005.2.115 |
