许春敏1, 林霄3, 李蕾2, 王淑萍2, 杨帆2, 徐石海3, 邓海2, 周永军2, 林厚文1,2


1.江西中医药大学, 江西 南昌 330004;
2.上海交通大学医学院附属仁济医院药学部, 上海 200127;
3.暨南大学药学院, 广东 广州 510632
收稿日期:2018-01-23;修回日期:2018-04-01;网络出版日期:2018-04-20
基金项目:国家自然科学基金(31670096, 31628001, 31600014)
*通信作者:林厚文。Tel/Fax:+86-21-68383339;E-mail:franklin67@126.com
摘要:[目的] 多肽化合物Surugamides(sgm)生物合成基因簇包含4个非核糖体多肽合酶(NRPS)基因surA-D, 负责2个NRPS生物合成途径。已有报道确认surA基因与Surugamide A产物相关, 而surB基因与sgm F产物相关, 但对surC和surD基因功能的归属尚没有实验证据。本工作拟在之前研究的基础上进一步确认surA和surD负责Surugamide A产物生物合成, 为基因工程改造Surugamides生物合成途径以及研究其NRPS蛋白之间的识别机制提供理论依据。[方法] 从海绵中分离放线菌并通过16S rRNA基因序列比对分析其分类单元。通过在线数据库antiSMASH分析基因组序列, 发现天然产物生物合成基因簇。通过UPLC-Q-TOF-MS和13C NMR鉴定化合物结构。把构建完成的同源重组双交换质粒导入链霉菌宿主后筛选基因缺失或替换突变株。[结果] 从胄甲海绵来源链霉菌S.albidoflavus LHW3101基因组中发现了Surugamides生物合成基因簇, 确认了该菌株发酵产物中的化合物sgm A和sgm F。构建了surB和surC基因同时缺失的突变株RJ9, 发现RJ9不再产sgm F而仍然产Surugamide A。在缺失突变surB和surC基因的同时在surD基因前引入了组成型强启动子ermEp*, 结果发现RJ9产Surugamide A水平是野生型菌株的约2倍。[结论] 确认了surB和surC基因与sgm A产物无关。在surD基因前引入强启动子后显著提高了Surugamide A的产量, 提示surD基因与sgm A产物相关, 结合已报到surA基因与Surugamide A产物相关的证据, 进一步确认了surA和surD基因负责Surugamide A生物合成的推论。
关键词:微生物天然产物非核糖体多肽合酶生物合成基因簇
Elucidation of an intercalation structure of the gene cluster for nonribosomal peptide Surugamides biosynthesis
Xu Chunmin1, Lin Xiao3, Li Lei2, Wang Shuping2, Yang Fan2, Xu Shihai3, Deng Hai2, Zhou Yongjun2, Lin Houwen1,2


1.Jiangxi University of Traditional Chinese Medicine, Nanchang 330004, Jiangxi Province, China;
2.Department of Pharmacy, Ren Ji Hospital, School of Medicine, Shanghai Jiao Tong University, Shanghai 200127, China;
3.College of Pharmacy, Jinan University, Guangzhou 510632, Guangdong Province, China
Received 23 January 2018; Revised 1 April 2018; Published online 20 April 2018
*Corresponding author: Houwen Lin, Tel/Fax:+86-21-68383339; E-mail:franklin67@126.com
Supported by the National Natural Science Foundation of China (31670096, 31628001, 31600014)
Abstract: [Objective] The Surugamides (sgm) biosynthetic gene cluster has been reported to contain four NRPS genes surA-D which give two unrelated NRPS pathways.It was experimentally confirmed that surA and surB were essential for Surugamide A and Surugamide F production respectively.However, the functions of surC and surD genes are not verified with experimental evidence.This work is designed to confirm if surA and surD genes are responsible for Surugamide A biosynthesis so as to pave the way to either genetically engineering of Surugamides biosynthetic pathways or study the recognition mechanism of their NRPS proteins. [Methods] The Actinomycetes isolated from marine sponge were identified by analyzing their 16S rRNA gene sequences.The natural product biosynthetic gene clusters were analyzed by submitting genomic sequences to the online database antiSMASH.The chemical structures of isolated compounds were elucidated by UPLC-Q-TOF-MS and 13C NMR.To generate the mutation of gene deletion and replacement, a plasmid was constructed with two fragments used for target homologous recombination.The plasmid was then transformed into the target strain for screening of double-crossover mutants. [Results] We discovered Surugamides gene cluster from the genome of Streptomyces albidoflavus LHW3101 isolated from marine sponge Dactylospongia elegans.The compound Surugamide A and Surugamide F were then identified from the fermentation extract of the strain.The surB and surC gene in Surugamides gene cluster were replaced with a constitutive and strong promoter ermEp*, which was located just before the transcription frame of surD gene.The resulted mutant RJ9 lost the production of Surugamide F but kept on producing Surugamide A with around two-fold yield of the wide type strain. [Conclusion] Gene surB and surC are verified to be not related to sgm A production.It is reported that surA is essential for Surugamide A production.And a remarkable yield improvement for Surugamide A was achieved after introducing a strong promoter before the open reading frame of surD in this work.Therefore we concluded that surA and surD take charge of the biosynthesis of Surugamide A.
Keywords: microbial natural productsnonribosomal peptide synthasebiosynthetic gene cluster
微生物来源非核糖体多肽(nonribosomal peptide,NRP)类化合物是重要的药物发现源头,例如目前临床使用的免疫抑制剂环孢霉素(cyclosporin)、抗生素万古霉素(vancomycin)、达托霉素(daptomycin)和青霉素(penecillin)以及2015年发现的抑制所有金黄色葡萄球菌和结核分支杆菌变种的新型抗生素teixobactin均属于微生物来源的NRP类化合物[1-2]。微生物来源NRP类化合物是通过非核糖体肽合成酶[3](nonribosomal peptide synthetase,NRPS)介导生物合成的[3]。在NRPS生物合成过程中酶催化功能域的模块化和装配线排列的特点使得对其工程化改造和目标产物预测成为可能[4]。NRPS含有的多个延伸模块(module)呈装配线模式依次传递延伸中的肽链,每个模块含有基本的功能域负责肽链合成,包括合成肽键的缩合功能域(condensation)、活化并加载氨基酸的腺苷酰化功能域(adenylation)以及肽酰基载体蛋白(peptide carrier protein/thiolation),在NRPS装配线末端通常含有负责肽链释放的硫酯酶[5]。此外在延伸模块中还可能存在一些非必需的修饰功能域,如负责氨基酸构型转化的差向异构酶(epimerase)功能域和在氨基上引入甲基的氮-甲基转移酶(N-methyltransferase)功能域。相比于化学合成多肽化合物需要昂贵的基团保护试剂和有毒溶剂,微生物生物合成多肽仅需要廉价的发酵培养基,可规模化生产并降低对环境的污染,因此发现并研究微生物来源NRPS生物合成途径对开发药用活性多肽类化合物具有重要价值。
我们从胄甲海绵(Dactylospongia elegans)中分离获得了链霉菌S. albidoflavus LHW3101,从其发酵产物中发现了环八肽化合物Surugamide A (sgm A)和链状十肽化合物Surugamide F (sgm F),基因组分析发现了该化合物对应的NRPS类型生物合成基因簇。该基因簇包含4个NRPS基因A–D,前期研究基于缺失突变A和B基因的结果,推测A和D基因负责sgm A的合成,而位于A和D基因之间的B和C基因负责sgm F的合成[6-7]。本研究通过构建B和C基因的缺失突变株以及为D基因引入强启动子,进一步明确了sgm A生物合成的必需基因,为基因工程改造sgm生物合成途径以及研究其NRPS蛋白之间的识别机制提供了研究基础。
1 材料和方法 1.1 材料
1.1.1 主要试剂和仪器: 超保真DNA聚合酶为High-Fidelity PCR Master Mix(NEB Phusion?),普通DNA聚合酶为2×fast Taq PCR Master Mix (上海柏根生物),质粒提取试剂盒购自上海捷瑞生物工程有限公司,PCR片段回收试剂盒为Zymoclean Gel DNA Recovery Kit,限制性内切酶均为NEB生产,U-Clone Master Mix for DNA Cloning试剂盒购自百诺威科技(武汉)有限公司,抗生素均购自生工生物工程(上海)股份有限公司,色谱级乙腈和甲醇为德国默克公司产品,分析纯试剂乙酸乙酯和甲醇购自上海化学试剂有限公司。液质联用(LC-MS)为Waters HPLC-ACQUITY QDa,高分辨质谱为Waters UPLC-QTOF-MS/MS,中压色谱系统购自三泰科技(常州)有限公司。振荡摇床(ZQZY-70BS)为上海知楚仪器有限公司生产。
1.1.2 培养基: SP2(g/L):酵母提取物5,麦芽提取物2,葡萄糖2,海盐18。M1(g/L):淀粉10,酵母提取物4,蛋白胨2,琼脂粉18,磷酸二氢钾2,海盐18,25 μg/mL萘啶酮酸,50 μg/mL放线菌酮。LB (g/L):胰蛋白胨10,酵母提取物5,氯化钠5,葡萄糖2。SFM (g/L):低温黄豆饼粉20,甘露醇20,琼脂粉20,pH 7.2,MgCl2 10 mmol/L。TSBY (g/L):大豆胰蛋白胨30,酵母提取物5,蔗糖103,海盐18,pH 7.2。
1.1.3 菌株和质粒: Streptomyces albidoflavus LHW3101由本实验室分离获得,Streptomyces albus J1074为通用模式菌株,RJ9为S. albidoflavus LHW3101来源的基因缺失突变株。E. coli DH10B用于质粒构建,E. coli ET12567/pUZ8002用于双亲结合转移。质粒pRJ26为本工作构建,用于获得基因缺失突变株RJ9。质粒pYH7携带apramycin(apr)抗性(AprR)基因,是大肠杆菌-放线菌穿梭质粒,用于构建基因缺失突变株[8]。质粒pIB139用于PCR模板,扩增组成型强启动子ermEp*[9]。
1.1.4 PCR引物和模板: 用于PCR扩增同源重组交换臂的引物为:L-surBC-S (5'-GACTGATCAAG GCGAATACTTCAGAGGCTGGCGTACGTCCTGTCC-3'),L-surBC-A (5'-ACACTCGCATGCATACT AGAGAATCCCCCTGGGGCTGGTACCTG-3'),R- surBC-S (5'-TGCCGGTTGGTAGGATCCACATAT GACCACGCCCTCGTCGCGGGA-3'),ermP-sur-S (5'-AGGGGGATTCTCTAGTATGCATGCGAGTGTC-3')和ermP-sur-A (5'-TGGTCATATGTGGATCCT ACCAACCGGCAC-3')。验证突变株RJ9的引物为surBC-2S (5'-ATGTGGTGCATCGACAGCAGCAG-3')和surBC-2A (5'-TCAACTCCAACGGCAAGGTG AC-3')以及ermP-sur-S与ermP-sur-A。用于菌种鉴定16S rRNA基因扩增的引物为27F (5'-AGAGTT TGATCCTGGCTCAG-3'),1492R (5'-GGTTACCTT GTTACGACTT-3')。用于筛选突变株的基因组DNA快速提取方法为:用牙签从单菌落上挑取菌丝体于PCR管,加入20 μL 10% Chelex? 100 Resin (Bio-Rad),100 ℃加热20 min,取上清1 μL作为PCR模板。
1.2 海绵共附生放线菌分离和鉴定 取胄甲海绵1 cm×1 cm方块,用1.2%无菌海盐水洗涤3次后充分研磨后加入10 mL无菌海盐水得到样品液,依次做10–1、10–2和10–3梯度稀释,各取200 μL分别涂布于M1培养基平板上,28 ℃培养14 d,选取放线菌生长表型的菌落在ISP2平板上划线获得单菌落,从单菌落上取菌丝体提取基因组DNA作为模板,用引物27F和1492R扩增16S rRNA基因,通过琼脂糖胶电泳分离和回收1.6 kb的PCR片段并进行测序,序列输入在线数据库Ezbiocloud分析菌株与已发现菌株的相似度。
1.3 克隆构建 以LHW3101菌株基因组DNA为模板PCR扩增同源交换臂,用引物L-surA-S和L-surA-A扩增获得1657 bp左臂,用引物R-surA-S和R-surA-A扩增获得1638 bp右臂。以质粒pIB139为模板,PCR扩增包含组成型强启动子ermEp*的212 bp片段,引物为ermP-sur-S和ermP-sur-A。用EcoR I和Hind Ⅲ双酶切质粒pYH7后,通过琼脂糖凝胶电泳回收7086 bp载体片段。通过Gibson DNA assembly的方法[10]连接以上4个片段。反应条件为:5 μL U-Clone Master Mix、5 μL DNA片段(包括约50 μg载体片段,每个插入片段100–200 μg)、50 ℃反应60 min。反应结束后取5 μL反应液通过钙转化转入E. coli DH10B,通过菌落PCR用引物surBC-2S和surBC-2A筛选目标质粒pRJ26的克隆。
1.4 基因缺失突变株筛选 先将质粒pRJ26通过钙转化导入E. coli ET12567/pUZ8002获得用于双亲接合转移的大肠杆菌。将大肠杆菌过夜培养液按1/100 (V/V)转接LB培养基,培养3–4 h后取1.5 mL菌液离心(6000 r/min,30 s)收集菌体,通过重悬浮和离心的方法用LB培养基洗涤菌体3次,然后把菌体悬浮于150 μL LB培养基待用。从平板挑取链霉菌LHW3101的单菌落接种TSBY培养基,30 ℃、220 r/min培养36 h后取1 mL菌液离心(10000 r/min,1 min)收集菌体,用LB培养基洗涤菌体2次后把菌体悬浮于150 μL LB培养基待用。把大肠杆菌和链霉菌的菌液混和后涂布在SFM平板(10 mmol/L Mg2+),30 ℃培养12 h后用1 mL抗生素水溶液覆盖平板,保证水吹干后平板抗生素浓度为10 μg/mL apramycin和25 μg/mL萘啶酮酸。覆盖后的平板于30 ℃培养4 d后可见AprR单菌落。挑取AprR单菌接种SFM平板(10 μg/mL apramycin)进行抗性复证,选取抗性正确的单菌落在SFM平板上转接3次,收集孢子并梯度稀释孢子悬浊液后涂布在SFM平板,30 ℃培养3 d后挑取单菌落筛选AprS菌株,提取基因组DNA作为PCR模板,用引物surBC-2S和surBC-2A筛选双交换突变株以及引物对ermP-sur-S和ermP-sur-A筛选双交换突变株,通过对PCR产物进行测序确认突变株。
1.5 链霉菌发酵及化合物提取 小量液体发酵及化合物提取:从SFM平板挑取单菌落接种到10 mL TSBY培养基,用100 mL弹簧三角瓶,30 ℃、220 r/min培养36 h,取1 mL菌液作为种子同样条件发酵2 d。取0.6 mL发酵液于1.5 mL离心管,加入0.6 mL乙酸乙酯,加热40 ℃并1000 r/min振荡30 min,萃取液14000 r/min离心2 min,收集有机相于1.5 mL离心管,待乙酸乙酯挥发完,加入400 μL甲醇溶解样品,经14000 r/min离心5 min后做LC-MS分析,上样量为20 μL。
大量液体发酵及化合物提取:将小量液体发酵种子液按10% (V/V)接种到150 mL TSBY培养基,用500 mL弹簧三角瓶在相同条件下培养28 h后得到二级种子,再按同样比例接种到2 L弹簧三角瓶,每瓶装500 mL TSBY培养基,同样条件发酵5 d。发酵液用等体积的乙酸乙酯萃取3次,减压旋蒸萃取液后收集浸膏。
1.6 化合物Surugamides色谱分离和纯化 将16 g浸膏经过凝胶色谱柱Sephadex LH-20进行初步分离,洗脱溶剂为甲醇。LC-MS分析跟踪目标馏分,对目标馏分再进行中压液相色谱分离,ODS色谱柱(15 μm, 100 ?,20 g),甲醇梯度洗脱条件:0–30 min (60%→80%),30–60 min (80%),60–90 min (80%→100%),流速10 mL/min。LC-MS分析跟踪目标馏分,对目标馏分再通过制备型HPLC做最终纯化,洗脱溶剂为乙腈:水(0.1%甲酸)=43:57,色谱柱为Phenomenex Venus Prep C18 (5 μm,250 mm×10 mm),流速3 mL/min。
1.7 LC-MS及NMR分析条件 常规HPLC分析条件为:A相为水(0.1%甲酸),B相为乙腈(MeCN);MeCN洗脱梯度为0– 17 min (30%→69.7%),17.0–17.1 min (69.7%→ 100%),17.1–21.0 min (100%),21.0–21.1 min (100%→ 30%),21.1–26.0 min (30%);流速0.7 mL/min;检测波长190 nm–400 nm;柱温25 ℃,色谱柱为Waters Xbridge C18 (5 μm,250 mm×4.6 mm)。低分辨质谱为Waters ACQUITY QDa,正离子模式。UPLC流动相为MeCN和0.1%甲酸水溶液,洗脱梯度为0–0.2 min (5% MeCN),0.2–5.0 min (100% MeCN),5–7 min (100% MeCN),7.0–7.1 min (5% MeCN),7.1–9.0 min (5% MeCN),色谱柱为Acquity UPLC BEH C18 (1.7 μm,2.1 mm×50 mm)。ESI-Q-TOF高分辨质谱分析采用正离子模式。核磁数据在Bruker Avance Ⅲ 600 MHz核磁共振仪上采集,核磁样品溶剂DMSO-d6。
1.8 基因组测序及生物信息学分析 通过细菌基因组DNA抽提试剂盒(天根)提取和纯化样本DNA,利用超声波将样本DNA随机打断,通过末端修复、连接接头,选择200-350 bp的DNA文库片段,使用Aglient 2100系统对文库进行质检,然后上机测序。测序平台为Illumina Hiseq Xten,测序方法为双端测序,最终产生大于4G的原始FASTQ数据。通过FASTQC进行数据质控,Trimmomatic去除接头和质量差的碱基,保留长度大于40 bp的序列,使用SPAdes 3.6软件进行基因组de novo拼接,获得scaffold片段长度大于200 kb。用软件Prokka进行基因预测和注释。通过在线服务器Ezbiocloud (https://www.ezbiocloud.net/dashboard)分析16S rRNA序列,通过次级代谢产物生物合成基因簇预测网站antiSMASH (http://antismash.secondarymetabolites.org/)预测基因组次生代谢产物生物合成基因簇,通过NRPS-PKS在线分析网站(http://www.nii.res.in/nrps-pks.html)分析NRPS大蛋白的催化功能域构成。Surugamides生物合成基因簇DNA序列已提交GenBank数据库,序列编号MH070261。
2 结果和分析 2.1 从海绵中分离获得S. albidoflavus LHW3101 将采自中国南海永兴岛的胄甲海绵(Dactylospongia elegans)粉末悬浊液梯度稀释后涂平板培养10–30 d,共获得放线菌生长表型的单菌落32个。通过在线服务器Ezbiocloud对16S rRNA基因序列进行比对分析,发现其中一株菌与S. albidoflavus DSM 40455T相似度达到100%[11],命名该菌株为S. albidoflavus LHW3101。LC-MS分析LHW3101菌株发酵产物后发现其次生代谢产物较为丰富,所以对其进行了基因组扫描测序。基因组序列经生物信息学分析和基因注释后发现了5个NRPS类型基因簇。
2.2 非核糖体多肽Surugamides生物合成基因簇分析 将LHW3101基因组序列递交AntiSMASH数据库进行比对分析,发现一个包含18个肽链延伸功能模块的大型NRPS基因簇,该基因簇与来源于S. albus J1074的Surugamides(sgm)生物合成基因簇几乎完全相同,2个基因簇结构和基因大小完全相同,仅有2个NRPS基因存在微小差别(99%的相同度)(图 1,表 1)。前期研究发现sgm基因簇应包括2个NRPS生物合成途径,推测负责环八肽化合物sgm A生物合成的基因是surA和surD,而嵌入在这2个基因之间的surB和surC基因可能负责链状十肽化合物sgm F的生物合成[6-7]。两个NRPS生物合成体系均没有后修饰步骤和硫酯酶功能域。
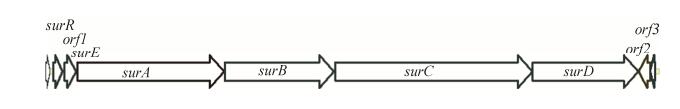 |
| 图 1 链霉菌LHW3101和J1074来源的Surugamides生物合成基因簇结构相同 Figure 1 The same gene organizations are found for the Surugamides gene clusters derived respectively from S. albidoflavus LHW3101 and S. albus J1074. |
| 图选项 |
表 1. 链霉菌LHW3101和J1074来源Surugamides基因簇蛋白序列比较分析 Table 1. The protein similarity analysis of Surugamides biosynthetic gene clusters derived respectively from S. albidoflavus LHW3101 and S. albus J1074
| ORF name | aa (LHW3101 or J1074) | Identity/% | Predicted function |
| SurR | 138 | 100 | Gent family DNA-binging transcriptional regulator |
| ORF1 | 368 | 100 | Membrane protein |
| SurE | 451 | 100 | Beta-lactmase |
| SurA | 5726 | 99 | NRPS (A-T, C-A-T-E, C-A-T-E, C-A-T) |
| SurB | 4256 | 100 | NRPS (A-T, C-A-T-E, C-A-T, C-A-T) |
| SurC | 7691 | 100 | NRPS (C-A-T, C-A-T-E, C-A-T, C-A-T-E, C-A-T, C-A-T-E) |
| SurD | 4114 | 99 | NRPS (C-A-T, C-A-T-E, C-A-T-E) |
| ORF2 | 452 | 99 | Drug resistance transporter |
| ORF3 | 203 | 100 | TetR family transporter regulator |
表选项
可能负责sgm F生物合成的NRPS蛋白SurB和SurC共加载10个氨基酸(图 2-A),包括1个色氨酸(起始模块)、2个亮氨酸(第一和五延伸模块)、3个缬氨酸(第二、六和八延伸模块)、1个苏氨酸(第三延伸模块)、2个丙氨酸(第七和九延伸模块)以及加载于第4个延伸模块的β-氨基酸3-amino-2(R)-methylpropionic acid(AMPA)。该NRPS装配线含有的4个差向异构酶(epimerase,E)功能域分别位于第一、五、七和九延伸模块,负责转化L-型氨基酸为D-型氨基酸。尚未在sgm基因簇边界处找到可能负责AMPA生物合成的基因。
 |
| 图 2 Surugamide F(A)和Surugamide A(B)生物合成基因簇及其生物合成途径推导 Figure 2 The gene clusters and proposed biosynthetic pathways for surugamide F (A) and surugamide A (B). |
| 图选项 |
可能负责sgm A生物合成的NRPS蛋白SurA和SurD共加载8个氨基酸(图 2-B),SurA包括一个起始模块和4个延伸模块,各模块A功能域加载的氨基酸依次为异亮氨酸、丙氨酸、异亮氨酸、异亮氨酸、赖氨酸;SurD含3个延伸模块,其A功能域分别加载异亮氨酸、苯丙氨酸和亮氨酸。第一、三、六和七延伸模块中包含的差向异构酶功能域应该负责sgm A环肽分子对应氨基酸的D型构相的生成。
基于以上对S. albidoflavus LHW3101来源的非核糖体多肽Surugamides生物合成基因簇的生物信息学分析,提示该菌具有生产sgm A和sgm F的潜力。
2.3 化合物Surugamides的分离纯化和结构鉴定 对S. albidoflavus LHW3101和S. albus J1074的发酵产物乙酸乙酯萃取物进行了高分辨质谱UPLC-ESI-QTOF分析对比,发现两者均有化合物sgm A和sgm F对应的分子离子峰:sgm A,[M+H]+=912.6277(计算值912.6281,误差值–0.4 mg/L);sgm F,[M+H]+=1056.6486(计算值1056.6452,误差值3.2 mg/L)(图 3,图 4)。为进一步确证LHW3101发酵产物中的sgm A化合物的结构,对其进行了36 L液体发酵。用乙酸乙酯萃取发酵液后获得16 g浸膏,经凝胶色谱柱Sephadex HP-20分离得到A–E 5个馏分。把含有目标化合物的B馏分上样中压液相ODS制备柱,通过LC-MS检测获得目标馏分,最终通过制备型HPLC分离获得sgm A化合物9.7 mg。对化合物sgm A进行了13C NMR分析,通过对比文献报道碳谱化学位移确证了其结构的正确性[12](表 2)。由于sgm F产量很低而未获得足够的化合物进行NMR测试,但其高分辨质谱数据和预期完全符合。
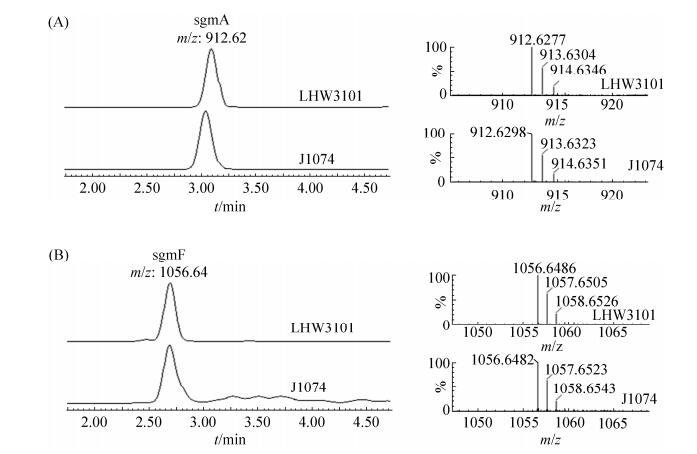 |
| 图 3 超高压液相-四级杆-飞行时间质谱(UPLC-Q-TOF-MS)分析确认链霉菌LHW3101和J1074发酵提取物中的化合物sgm A(A)和sgm F(B) Figure 3 UPLC-Q-TOF-MS analysis of the sgm A (A) and sgm F (B) in the fermentation extract of strain LHW3101 and J1074. The left part is the extracted molecular ion peak and the right one is the mass signal of target molecular ion. |
| 图选项 |
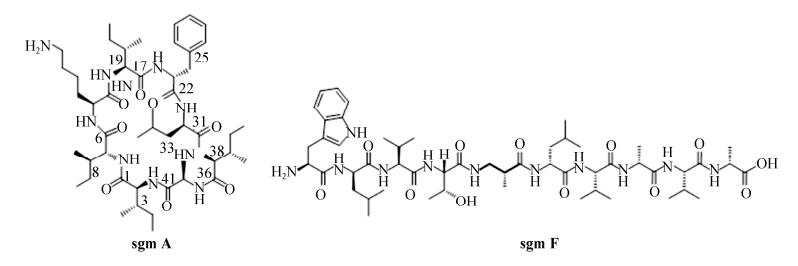 |
| 图 4 sgm A和sgm F的化学结构图 Figure 4 The chemical structure of sgm A and sgm F. |
| 图选项 |
表 2. 与文献比对确认sgm A的NMR碳谱归属(在DMSO-d6溶剂中检测) Table 2. NMR assignments for sgm A analyzed in DMSO-d6 by comparing with the published data
| Residue | Position | Experiment11] | |
| δC | δC | ||
| Ile1 | 1 | ND | 172.1 |
| 2 | 57.8 | 57.8 | |
| 3 | 34.8 | 35.3 | |
| 3’ | 14.7 | 14.8 | |
| 4 | 24.5 | 24.7 | |
| 5 | 11.5 | 11.9 | |
| Ile2 | 6 | 171.0 | ND |
| 7 | 57.0 | 57.0 | |
| 8 | 35.9 | 36.3 | |
| 8’ | 14.5 | 14.8 | |
| 9 | 25.7 | 26.0 | |
| 10 | 11.4 | 11.9 | |
| Lys | 11 | ||
| 12 | 51.7 | 52.1 | |
| 13 | 31.4 | 31.7 | |
| 14 | 22.1 | 22.3 | |
| 15 | 29.1 | 28.2 | |
| 16 | 38.8 | 39.7 | |
| Ile3 | 17 | 171.1 | ND |
| 18 | 58.2 | 58.2 | |
| 19 | 35.4 | 35.9 | |
| 19’ | 15.0 | 15.0 | |
| 20 | 25.0 | 25.1 | |
| 21 | 11.1 | 11.4 | |
| Phe | 22 | 171.3 | 171.5 |
| 23 | 54.8 | 55.0 | |
| 24 | 36.4 | 36.8 | |
| 25 | 138.1 | 138.0 | |
| 26,30 | 129.0 | 129.3 | |
| 27,29 | 128.1 | 128.4 | |
| 28 | 126.3 | 126.6 | |
| Leu | 31 | 172.9 | 172.9 |
| 32 | 52.7 | 52.7 | |
| 33 | ND | 40.4 | |
| 34 | 24.4 | 24.6 | |
| 35 | 21.3 | 21.7 | |
| 35’ | 23.2 | 23.5 | |
| Ile4 | 36 | 169.8 | 169.8 |
| 37 | 57.6 | 57.7 | |
| 38 | 35.8 | 36.1 | |
| 38’ | 15.2 | 15.4 | |
| 39 | 23.9 | 24.2 | |
| 40 | 10.6 | ND | |
| Ala | 41 | 172.6 | 172.8 |
| 42 | 48.1 | 48.2 | |
| 43 | 18.9 | 19.2 | |
表选项
2.4 确认sgm A和sgm F的生物合成相关基因 为了确认镶嵌于sgm基因簇内部的surB和surC基因仅负责sgm F的生物合成而与sgm A无关,构建了用于缺失基因surB和surC的同源重组双交换载体pRJ26,同时在该同源交换臂中间装载了组成型强启动子ermEp*,使得缺失surB和surC的同时在surD基因阅读框前面引入强启动子ermEp*(图 5-A)。通过双亲接合转移把pRJ26导入宿主菌LHW3101后筛选同源重组双交换突变株。用引物surBC-2S和surBC-2A对候选突变株进行PCR筛选,双交换突变株PCR目标产物为1122 bp (图 5-B左),野生型的PCR目标产物因理论长度为39 kb所以无法扩增出产物,同时用引物ermp-sur-A和ermp-sur-S进一步验证ermEp*已引入surD基因前,目标产物为212 bp (图 5-B右),经过测序确认两对引物验证突变株的PCR产物,最终获得突变株RJ9。LC-MS分析RJ9液体发酵乙酸乙酯萃取物发现化合物sgm F不再产生,而sgm A的产量提高到野生型产量约2倍水平(图 5-C)。
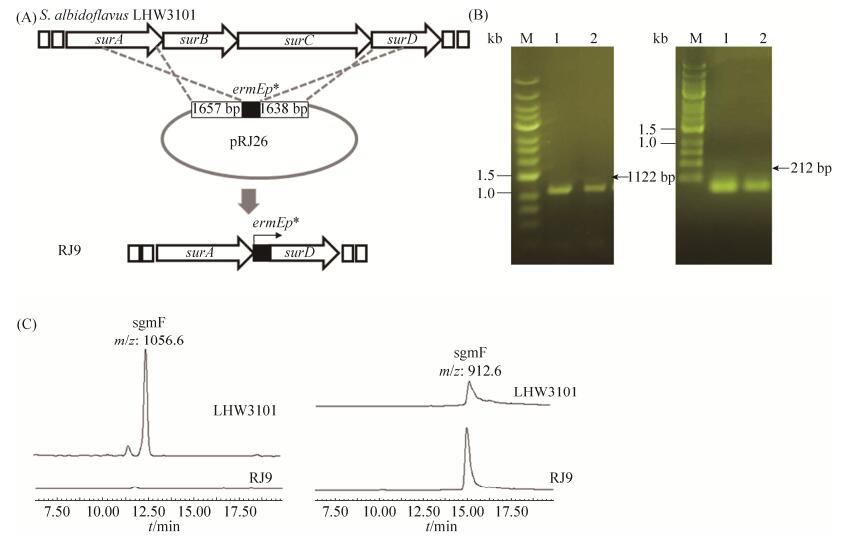 |
| 图 5 surB-C被替换突变株RJ9的构建(A,B)以及发酵产物LC-MS检测(C) Figure 5 Construction and LC-MS analysis of the surB-C replacement mutant RJ9. A: Replacement of surB-surC genes fragment with a constitutive strong promter ermEp* just before the open reading frame of surD by homologous recombination; B: PCR confirmation of mutant RJ9. The PCR product of either positive control (PC) or RJ9 is 1122 bp for primer surBC-2S and surBC-2A (left gel) and 212 bp for primer ermP-sur-S and ermP-sur-A (right gel); C: LC-MS analysis of the fermentation extract of RJ9 and LHW3101. The ion peaks corresponding to sgmF and sgm A are labeled. |
| 图选项 |
3 讨论 Surugamides(sgm)生物合成基因簇最先发现于海洋链霉菌Streptomyces sp. JAMM992中[7],但其DNA序列一直未公布。2017年许飞等再次从S. albus J1074中发现了该基因簇,并对surA和surB基因分别进行了基因中断突变,发现surA是化合物Surugamide A (sgm A)生物合成必需,而surB为化合物Surugamide F (sgm F)生物合成必需,结合对NRPS大蛋白功能域构成的分析,推测NRPS基因surA和surD负责sgmA生物合成,而surB和surC则专一负责sgmF的生物合成[6]。我们从中国西沙胄甲海绵(Dactylospongia elegans)中分离到了链霉菌S. albidoflavus LHW3101,在对其基因组天然产物生物合成基因簇分析中发现了sgm基因簇,确认了LHW3101代谢产物中的化合物sgm A和sgm F。前期报道对基因surC和surD功能归属尚无实验证据,我们通过基因缺失突变确认了surB和surC基因与sgm A产物无关,在surD基因编码框前引入强启动子后显著提高了sgm A的产量,从而进一步确证了surA和surD基因负责sgm A生物合成的推论。此外,surB和surC基因编码的NRPS功能域构成与化合物sgm F链状十肽的结构对应,同时缺失这两个基因后sgm F不再产生,提示surB和surC可能负责sgm F的生物合成。
Sgm生物合成途径还存在一些未解之谜值得深入探索。两个NRPS生物合成途径均缺乏负责链释放或环化功能的硫酯酶功能域,推测位于sgm基因簇左边界的内酰胺酶(lactamase)基因或基因簇外部的硫酯酶基因负责完成这一功能,类似的情况如南昌霉素(nanchangmycin)聚酮生物合成末端释放由单独存在硫酯酶通过反式作用机制负责完成[13]。此外sgm F肽链构成中含有的β-氨基酸AMPA需要相关的基因负责合成,该前体也发现于链霉菌来源化合物Vicenistatin的聚酮生物合成途径中,是由谷氨酸经谷氨酸变位酶和脱羧酶负责合成的[14],用这2个蛋白的序列分别对S. albidoflavus LHW3101基因组进行BLAST比对搜索却并未找到同源基因。
References
| [1] | Kries H. Biosynthetic engineering of nonribosomal peptide synthetases. Journal of Peptide Science, 2016, 22(9): 564-570. DOI:10.1002/psc.v22.9 |
| [2] | Ling LL, Schneider T, Peoples AJ, Spoering AL, Engels I, Conlon BP, Mueller A, Sch berle TF, Hughes DE, Epstein S, Jones M, Lazarides L, Steadman VA, Cohen DR, Felix CR, Fetterman KA, Millett WP, Nitti AG, Zullo AM, Chen C, Lewis K. A new antibiotic kills pathogens without detectable resistance. Nature, 2015, 17(7535): 455-459. |
| [3] | Walsh CT. Insights into the chemical logic and enzymatic machinery of NRPS assembly lines. Natural Product Reports, 2016, 33(2): 127-135. DOI:10.1039/C5NP00035A |
| [4] | Wang SY. Advances in the study of the mechanism and application of nonribosomal peptide synthetases. Acta Microbiologica Sinica, 2007, 47(4): 734-737. (in Chinese) 王世媛. 非核糖体肽合成酶(NRPSs)作用机理与应用的研究进展. 微生物学报, 2007, 47(4): 734-737. DOI:10.3321/j.issn:0001-6209.2007.04.034 |
| [5] | Horsman ME, Hari TPA, Boddy CN. Polyketide synthase and non-ribosomal peptide synthetase thioesterase selectivity:logic gate or a victim of fate?. Natural Product Reports, 2016, 33(2): 183-202. DOI:10.1039/C4NP00148F |
| [6] | Xu F, Nazari B, Moon K, Bushin LB, Seyedsayamdost MR. Discovery of a cryptic antifungal compound from Streptomyces albus J1074 using high-throughput elicitor screens. Journal of the American Chemical Society, 2017, 139(27): 9203-9212. DOI:10.1021/jacs.7b02716 |
| [7] | Ninomiya A, Katsuyama Y, Kuranaga T, Miyazaki M, Nogi Y, Okada S, Wakimoto T, Ohnishi Y, Matsunaga S, Takada K. Biosynthetic gene cluster for surugamide A encompasses an unrelated decapeptide, surugamide F. Chembiochem, 2016, 17(18): 1709-1712. DOI:10.1002/cbic.v17.18 |
| [8] | Sun YH, He XY, Liang JD, Zhou XF, Deng ZX. Analysis of functions in plasmid pHZ1358 influencing its genetic and structural stability in Streptomyces lividans 1326. Applied Microbiology and Biotechnology, 2009, 82(2): 303-310. DOI:10.1007/s00253-008-1793-7 |
| [9] | Wilkinson CJ, Hughes-Thomas ZA, Martin CJ, B hm I, Mironenko T, Deacon M, Wheatcroft M, Wirtz G, Staunton J, Leadlay PF. Increasing the efficiency of heterologous promoters in actinomycetes. Journal of Molecular Microbiology and Biotechnology, 2002, 4(4): 417-426. |
| [10] | Gibson DG, Young L, Chuang RY, Venter JC, Hutchison Ⅲ CA, Smith HO. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods, 2009, 6(5): 343-345. DOI:10.1038/nmeth.1318 |
| [11] | Hain T, Ward-Rainey N, Kroppenstedt RM, Stackebrandt E, Rainey FA. Discrimination of Streptomyces albidoflavus strains based on the size and number of 16S-23S ribosomal DNA intergenic spacers. International Journal of Systematic Bacteriology, 1997, 47(1): 202-206. DOI:10.1099/00207713-47-1-202 |
| [12] | Takada K, Ninomiya A, Naruse M, Sun Y, Miyazaki M, Nogi Y, Okada S, Matsunaga S. Surugamides A-E, cyclic octapeptides with four D-amino acid residues, from a marine Streptomyces sp.:LC-MS-aided inspection of partial hydrolysates for the distinction of D-and L-amino acid residues in the sequence. Journal of Organic Chemistry, 2013, 78(13): 6746-6750. DOI:10.1021/jo400708u |
| [13] | Liu TG, You DL, Valenzano C, Sun YH, Li JL, Yu Q, Zhou XF, Cane DE, Deng ZX. Identification of NanE as the thioesterase for polyether chain release in nanchangmycin biosynthesis. Chemistry & Biology, 2006, 13(9): 945-955. |
| [14] | Miyanaga A, Iwasawa S, Shinohara Y, Kudo F, Eguchi T. Structure-based analysis of the molecular interactions between acyltransferase and acyl carrier protein in vicenistatin biosynthesis. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(7): 1802-1807. DOI:10.1073/pnas.1520042113 |
