李想1,2, 孙岩1,2, 刘俊杰1, 姚钦1, 王光华1


1.中国科学院东北地理与农业生态研究所, 中国科学院黑土区农业生态重点实验室, 黑龙江 哈尔滨 150081;
2.中国科学院大学, 北京 100049
收稿日期:2018-04-18;修回日期:2018-07-10;网络出版日期:2018-07-19
基金项目:国家自然科学基金(41571246)
*通信作者:王光华, Tel:+86-451-86602745;Fax:+86-451-86603736;E-mail:wanggh@iga.ac.cn
摘要:[目的] 揭示我国东北典型湿地沉积物中T4型噬菌体g23基因的多样性,明确湿地环境T4型噬菌体群落分布特征,为噬菌体生态学研究提供数据支撑。[方法] 采用简并性引物MZIA1bis和MZIA6对采自东北6个地点不同类型湿地沉积物土壤DNA进行PCR扩增,采用克隆测序方法,解析沉积物中T4型噬菌体g23基因组成,通过UniFrac分析T4型噬菌体群落结构在湿地沉积物中与其他环境中的差异。[结果] 在东北湿地沉积物中共得到262条不同的g23基因序列,构建的系统进化树分析表明,我国东北湿地沉积物T4型噬菌体g23基因分布与海洋、湖泊及稻田生态系统中g23基因亲缘关系较近,而与东北旱地黑土g23基因分布较远;以g23基因群集表征的T4型噬菌体群落在不同地点湿地中分异明显。[结论] 东北湿地生态系统T4型噬菌体群落结构复杂多样,存在着一些未知的噬菌体类群。
关键词:T4型噬菌体g23基因湿地沉积物系统进化树Unifrac分析
T4-type bacteriophage genetic diversity in wetland sediments in northeast China
Xiang Li1,2, Yan Sun1,2, Junjie Liu1, Qin Yao1, Guanghua Wang1


1.Key Laboratory of Mollisols Agroecology, Northeast Institute of Geography and Agroecology, Chinese Academy of Sciences, Harbin 150081, Heilongjiang Province, China;
2.University of Chinese Academy of Sciences, Beijing 100049, China
Received 18 April 2018; Revised 10 July 2018; Published online 19 July 2018
*Corresponding author: Guanghua Wang, Tel: +86-451-86602745; Fax: +86-451-86603736; E-mail: wanggh@iga.ac.cn
Supported by the National Natural Science Foundation of China (41571246)
Abstract: [Objective] To provide scientific data for studying the ecology of bacteriophage in natural environments, the genetic diversity and distribution patterns of g23 of T4-type bacteriophage in wetland sediments were revealed. [Methods] Degenerate primers MZIA1bis and MZIA6 were used to PCR amplification of g23 gene of T4-type bacteriophages, and the PCR products were cloned and sequenced to reveal the diversity of g23 sequences in 6 different wetland sediment samples obtained from northeast China. In addition, the Unifrac analysis was used to compare the T4-type bacteriophage community structures in wetland sediments with those in other environments. [Results] In total, 262 different g23 clones of T4-type bacteriophage were obtained. Neighbor-joining phylogenetic tree showed that the distribution of g23 genes in wetland sediments was similar to those in aquatic ecosystems such as oceans and lakes and paddy ecosystems, but distinctly different from those in upland black soils. In addition, the T4-type bacteriophage community as evaluated by g23 gene assembles was remarkably different among different wetlands. [Conclusion] The composition of T4-type bacteriophage community was complex and diverse in wetland ecosystem, and the novel g23 groups of T4-type bacteriophage existed in wetland sediments in northeast China.
Keywords: T4-type bacteriophageg23 genewetland sedimentphylogenetic treeUnifrac analysis
噬菌体,即细菌病毒,是自然环境中病毒群落的主要组成部分[1]。噬菌体最早于1915年和1917年被Twort和D’Herelle发现,距今已有100多年的历史,对它的研究促进了现代分子生物学技术的发展[2]。噬菌体对微生物群落结构形成有着巨大的影响,在营养物质流动和能量循环中起着至关重要的作用[3]。噬菌体被认为是引起基因水平移动的主要载体,是驱动生命进化的主要动力[4]。自然环境中噬菌体数量庞大,仅海洋中就超过1030[5]。但目前我们对环境中噬菌体多样性及其群落分布特征的了解还很少。
g23基因是编码T4型噬菌体主要壳蛋白(major capsid protein)的基因[6],其基因片段存在高度保守的序列,Filée等[7]发现多株T4型噬菌体基于g23基因与基于几个保守基因的系统发育分析高度一致,并首次设计出简并性引物MZIABis/MZIA6,利用该对引物从不同海洋病毒浓缩液DNA中直接PCR扩增出g23基因,发现除少数序列与已报道的T-evens等类群的同源关系较近外,大部分序列属于新建立的5个海洋T4型噬菌体新类群(Marine Group Ⅰ-Ⅴ),开拓了利用免培养的分子生物学技术研究环境中T4型噬菌体基因多样性的先例。近年来,针对T4型噬菌体g23基因多样性的研究呈现多元化趋势,我们前文已对自然环境中g23基因多样性研究进展进行了综述[8],最近,又有研究者对东北旱地黑土、北极冰川、东北稻田、三江湿地、珠江口沉积物及九龙江口等环境下T4型噬菌体多样性进行了报道[9-16],这些研究表明g23基因分布与其来源环境关系很大,T4型噬菌体基因多样性异常丰富。g23基因被认为是一个研究不同环境中T4型噬菌体多样性的重要分子标记基因[17]。
湿地是地球陆地表层生态系统的重要组成部分,它是由水陆相互作用而形成的具有特殊功能的自然综合体,介于水生生态系统和陆生生态系统之间的过渡类型,兼具两生态系统的特点。湿地常被比喻为“地球之肾”,在陆地生态系统元素循环中具有重要作用,同时具有净化水质、调节气候、维持生物多样性等功能[18]。湿地因植被种类多样性、季节性水分波动、微环境中氧化还原反应交替发生等特点,决定其为天然的物种基因库,是研究微生物生态学的热点领域之一。尽管国际上近10多年来对海洋和淡水湖泊环境下噬菌体遗传多样性等报道很多,但对自然湿地生态系统的研究还鲜有报道,而对我国湿地环境下T4型噬菌体g23基因多样性的工作仅限于郑春雨等在2013年针对我国三江湿地的研究[14]。东北是我国湿地的主要集中分布区之一,具有多种类型的湿地资源。鉴于以往的研究已阐述了T4型噬菌体在海洋、湖泊、稻田和旱地黑土环境中多样性的特点,UniFrac分析结果也证实来自上述四类环境中的T4型噬菌体g23基因序列群集分布差异较大,表明不同的生态系统存在独特的T4型噬菌体群落的分布[12]。我们推测,在我国东北湿地可能会存在未知的T4型噬菌体g23基因序列及集群。因此,本研究通过对我国东北典型湿地沉积物样品的采集,采用分子生物学技术解析T4型噬菌体g23基因多样性及其群落分布特征,比较不同湿地沉积物样点之间,以及湿地和其他环境之间的T4型噬菌体g23基因组成异同,揭示T4型噬菌体在湿地环境中的分布特征。
1 材料和方法 1.1 湿地沉积物的采集 于2016年7月和8月采集6个地点的我国东北典型湿地沉积物(图 1),包括黑龙江北部大兴安岭南瓮河沼泽湿地(NWH: 51°07′44.82″N,125°07′54.78″E)、东部建三江洪河沼泽湿地(HH: 47°34′26.10″N,133°14′21.24″E)、西部齐齐哈尔扎龙沼泽湿地(ZL: 47°11′53.58″N,124°14′07.80″E)、南部兴凯湖湖泊湿地(XKH: 45°21′54.28″N,132°19′02.47″E)、辽宁丹东鸭绿江口滨海湿地(YLJK: 39°49′22.50″N,124°03′32.64″E)和辽宁辽河口滨海湿地(LHK: 40°56′52.26″N,121°47′34.98″E)。湿地沉积物土样为湿地表层土壤样品(0-10 cm),每个样点采样时表层覆盖1 m以上的水层,每个采样点多点采集混合后放入冰盒中运回实验室。
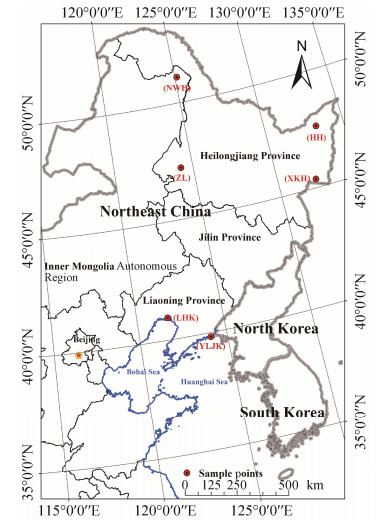 |
| 图 1 本研究东北湿地采样地点分布图 Figure 1 Sampling locations of different wetlands in northeast China for this study. YLJK, NWH, HH, LHK, XKH and ZL indicate wetlands of Yalujiangkou, Nanwenghe, Honghe, Liaohekou, Xingkaihu and Zhalong, respectively. |
| 图选项 |
1.2 湿地沉积物总DNA提取及PCR扩增 取0.5 g湿地沉积物样品,采用土壤DNA提取试剂盒Fast DNA? Spin Kit for Soil(MP Biomedicals,USA),并按说明书操作方法提取湿地沉积物土壤DNA。提取的DNA经1%的琼脂糖凝胶电泳检测后,存放于-20℃冰箱中保存待用。
采用简并性引物MZIA1bis(5′-GAT ATT TGI GGI GTT CAG CCI ATG A-3′)和MZIA6(5′-CGC GGT TGA TTT CCA GCA TGA TTT C-3′)对T4型噬菌体g23基因进行PCR扩增[7],该引物组合扩增出的T4型噬菌体g23基因片段长度相差很大。PCR产物的长度在380-700 bp范围内可能是g23基因,而不在这个范围内的PCR产物均不是g23基因[7]。PCR扩增体系为50 μL,其中含有引物浓度为50 pmol的上游和下游引物各0.5 μL,1-2 μL的DNA模板,10 μL的EasyTaq buffer (TransGen Biotech,北京),5 μL的dNTPs (2.5 mmol/L each;TransGen Biotech,北京),1 μL Easy Taq DNA聚合酶(TransGen Biotech,北京),添加灭菌超纯水至要求的体积。阴性对照以无菌ddH2O为模板。PCR反应程序为:94 ℃ 5 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1 min,35个循环;72 ℃ 5 min。
1.3 克隆与测序 从1.5%琼脂糖凝胶上切取目标范围内的PCR产物,用QIAEX Ⅱ胶回收试剂盒(QIAEX Ⅱ Gel Extraction Kit,德国)进行DNA胶纯化,纯化的DNA片段连接至pMD18-T质粒载体上(TaKaRa,大连,中国),转化大肠杆菌Escherichia coli DH5α感受态细胞。随机从每个转化子中挑选50个阳性克隆,再用MZIA1bis/MZIA6引物进行菌体PCR扩增,循环数降至26。挑选的阳性克隆过夜培养菌液,递交测序公司(华大基因,深圳)进行Sanger测序。
1.4 系统进化分析 克隆的核苷酸序列采用欧洲生物信息研究所网站(http://www.ebi.ac.uk/) EMBOSS Transeq程序翻译成氨基酸序列。使用NCBI网站(http://www.ncbi.nlm.nih.gov/)上的BLAST (Basic Local Alignment Search Tool)搜索程序在氨基酸水平上对获得g23阳性克隆进行最高相似性比对。通过MEGA 6.0软件[19],采用NJ法构建系统发育树。应用QIIME (1.9.0 version)软件将序列同源性≥97%的序列归为同一操作单元(OTU),将获得的OTU统计结果在QIIME上进行多样性和UniFrac分析[20]。从QIIME软件上获得主坐标点数据,并利用R (version 3.4.3)软件的vegan (v.2.4-5)绘制主坐标分布图(PCoA)。
1.5 提交序列 本研究获得的T4型噬菌体g23基因序列已提交至NCBI网站,其序列号为MH160835-MH161096。
2 结果和分析 2.1 湿地沉积物DNA提取、PCR扩增、胶回收及克隆筛选 湿地沉积物提取DNA (图 2-A)后,PCR扩增g23基因在400-500 bp有条带(图 2-B),经胶回收获取目的基因(图 2-C),各样品筛选阳性克隆的位置在400-500 bp的范围(图 2-D)。随机挑取410个阳性克隆递交测序公司进行测序,经NCBI数据库比对,获得了333条g23基因序列。各个样品组内在核苷酸水平上去除重复序列后,剩余262条不同的g23基因序列,其中44、24、73、33、57和31个克隆分别来自鸭绿江口、南瓮河、洪河、辽河口、兴凯湖和扎龙湿地。扩增出的g23基因序列(含引物)长度为391-502 bp,翻译成长度为130-167个氨基酸残基。
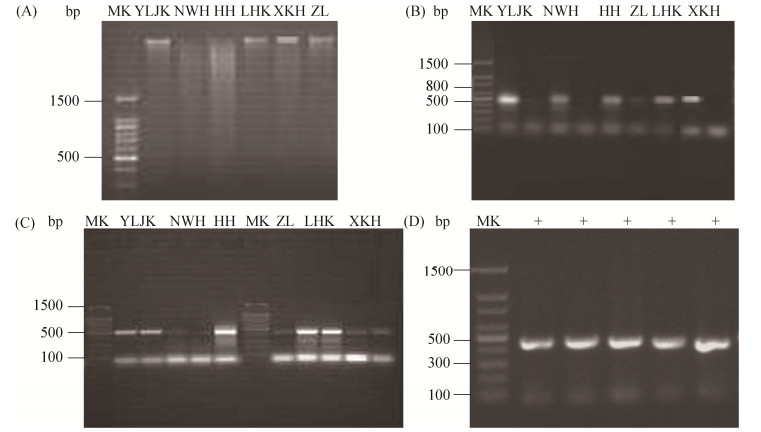 |
| 图 2 东北湿地沉积物中DNA提取(A)、g23基因的PCR扩增(B)、胶纯化(C)及阳性克隆检测(D) Figure 2 Profiles of DNA extraction (A), PCR amplification (B), gel purification (C) and positive clone (D) of g23 gene of T4-type bacteriophages obtained from wetland sediments in Northeast China. MK and + represent DNA marker and positive clone, respectively. YLJK, NWH, HH, LHK, XKH and ZL indicate wetland sediments of Yalujiangkou, Nanwenghe, Honghe, Liaohekou, Xingkaihu and Zhalong, respectively. |
| 图选项 |
2.2 g23基因的最近亲缘序列分析 在NCBI网站上,基于氨基酸水平上经BLASTp比对结果显示,本研究获得的g23基因序列与其他环境来源(稻田、旱地黑土、海水和淡水湖泊等)的g23基因序列具有62%-100%相似度。具体来说,南瓮河、洪河、扎龙、兴凯湖、鸭绿江口和辽河口湿地沉积物样品与已报道序列的相似度分别为73%-95%、63%-99%、68%-99%、62%-99%、68%-99%和69%-100%。图 3为6个湿地沉积物样品中获得的g23基因序列与已报道的g23基因序列最高相似性的分布范围。由图可知,各湿地沉积物中g23基因序列的最高相似度绝大部分大于70%,其中来自南瓮河湿地的g23基因序列的最高相似度均大于74%。另外,有13.64% (6/44)来自鸭绿江口滨海湿地的g23基因序列的最高相似度小于70%,有13.70% (10/73)来自洪河湿地的g23基因序列的最高相似度小于70%。
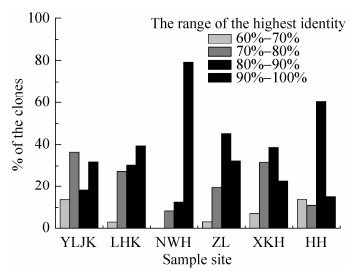 |
| 图 3 不同地点湿地沉积物g23克隆与GenBank中g23氨基酸序列最高同源性所占比例 Figure 3 The proportion of clones obtained from different wetland sediments having the highest range of BLAST search identity with known g23 amino acid sequences in GenBank. The different gray bars indicate the range of the highest identity. YLJK, LHK, NWH, ZL, XKH and HH indicate wetland sediments of Yalujiangkou, Liaohekou, Nanwenghe, Zhalong, Xingkaihu and Honghe, respectively. |
| 图选项 |
2.3 g23基因系统进化分析 将本研究获得的262条g23基因序列在氨基酸水平上构建系统进化树(图 4)。依据系统进化树结构可将序列划分成13个进化簇(Cluster Ⅰ-XIII),每个进化簇与数据库中已知序列最高相似度在73.5%±15.8%至96.8%±1.5%之间。
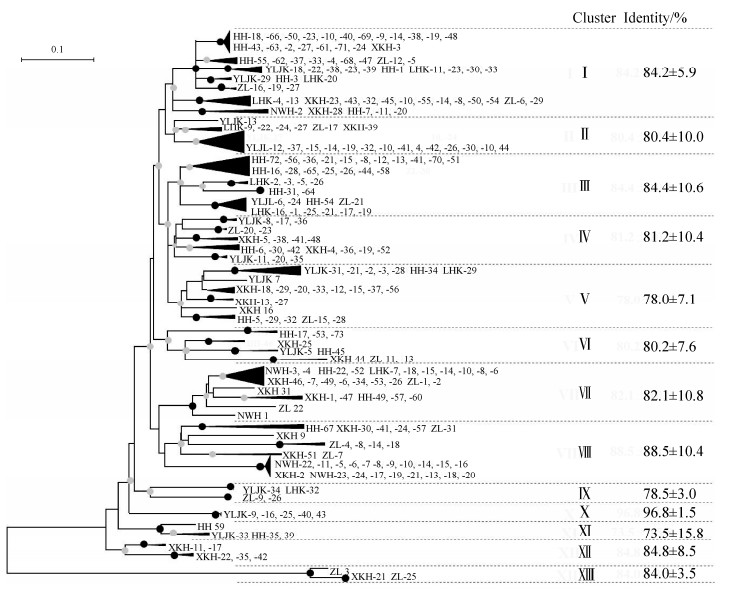 |
| 图 4 东北湿地沉积物中T4型噬菌体g23序列在氨基酸水平构建的系统进化树 Figure 4 Neighbor-joining phylogenetic tree of g23 sequences obtained from wetland sediments of this study. The black and gray circles at the internal nodes indicate the bootstrap support value with at least 90% and 50% respectively. The scale bar represents a 10% amino acid sequence difference. |
| 图选项 |
将本研究获得的g23序列与已发表的稻田生态系统[11-12, 21-24]中的T4型噬菌体g23序列进行比较(图 5-A)。由图可知,湿地沉积物中大部分g23序列不能形成明显的独立分支(cluster)。因此我们认为,湿地沉积物中的T4型噬菌体g23基因多样性与稻田生态系统中的亲缘关系较近。
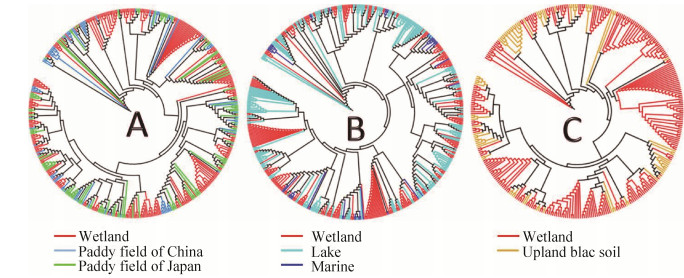 |
| 图 5 东北湿地沉积物与其他生态系统中T4型噬菌体g23基因系统进化分析 Figure 5 Phylogenetic analysis of g23 amino acid sequences obtained from wetland sediments in this study with those obtained from paddy field soils of northeast China and Japan (A), with those from marine waters and lake waters (B), and with those from upland black soils (C). |
| 图选项 |
将本研究获得的g23序列与已发表的海洋生态系统[7]、湖泊生态系统[25-29]中的g23序列进行比较(图 5-B)。由图可知,本研究获得的序列除小部分能独立于海洋、湖泊环境而形成一些小的分支外,多数序列划分到与海洋、湖泊共享分支中。因此我们认为,湿地沉积物中的T4型噬菌体g23基因多样性与海洋、湖泊生态系统中g23基因也具有较近的亲缘关系。
将本研究获得g23序列与已报道的东北旱地黑土[9-10]中g23基因序列进行比较(图 5-C)。由图可以明显看出,湿地沉积物T4型噬菌体与东北旱地黑土T4型噬菌体不同。大部分湿地沉积物中的g23序列能独立于旱地黑土,单独形成一些分支,说明湿地沉积物中的T4型噬菌体不同于旱地黑土环境。
2.4 T4型噬菌体g23基因序列的UniFrac分析 利用UniFrac分析法,将本研究获得g23基因序列与海洋水体、湖泊水体、北极冰川水体、中国东北稻田水体和土壤、日本稻田水体和土壤、中国东北旱地黑土以及珠江河口湿地沉积物中的代表性g23基因序列进行比较分析(图 6)。主坐标分析(PCoA)图显示,不同环境的g23序列分布在4个象限,大多集中分布在轴心附近。来自中国东北稻田土壤、中国东北旱地黑土和中国东北三江湿地土壤的样品列位于第二象限,海洋、湖泊、稻田水及三江湿地的水体样品位于第三、四象限较近的位置,星号标记是本研究不同的湿地沉积物样点,6个地点的g23基因群落分布较分散,分别在第一、三、四象限。具体分布情况为:南瓮河湿地、鸭绿江口湿地和扎龙湿地分布在第一象限,且南瓮河湿地群落与其他湿地相距较远。洪河湿地分布在第三象限,与中国东北稻田水体环境来源的g23基因群集的PCoA距离较近,兴凯湖湖泊湿地和辽河口湿地沉积物群落分布在第四象限,与水体环境群落位置较近,且兴凯湖湖泊湿地与淡水湖泊、湿地水体以及北极冰川水体的样品位置靠近,表明这些样点的T4型噬菌体群集相似性较高。
 |
| 图 6 湿地沉积物中T4噬菌体与其他生态系统中T4型噬菌体的UniFrac分析 Figure 6 UniFrac analysis of T4-type bacteriophage community among wetland sediments, wetland water, marine water, lake freshwater, Pearl river estuary sediment, paddy soil of Japanese, paddy soil of northeast China, paddy floodwater of Japan, paddy floodwater and upland black soil of northeast China. |
| 图选项 |
3 讨论和结论 本研究获得的262条T4型噬菌体g23基因序列,基于氨基酸序列水平上与NCBI数据库中的g23基因序列进行比对,我们发现来自湿地沉积物样品中的g23基因序列有大约24%(63/262)的序列与数据库中序列的相似度低于75%,表明湿地沉积物样品中存在着新的g23基因序列集群。从图 4系统发育树每组进化簇内序列与已报道的序列平均相似度分析发现,cluster Ⅴ、cluster Ⅸ和cluster Ⅺ与已知序列相似度低,分别为78.0%±7.1%、78.5%±3.0%和73.5%±15.8%。因此这3个进化簇中的g23序列可能是新发现的基因,这3个进化簇也很可能是新的g23基因序列集群。
自2005年Filée[7]首次发现海洋生态系统中T4型噬菌体与最初划分出的T-evens、Pseudo T-evens、Schizo T-evens和ExoT-evens不同后,陆续有研究者报道出海洋[7]、湖泊[25-29]、日本稻田[21-23]、中国东北稻田[11-12, 24]、中国东北旱地黑土[9-10]、珠江口沉积物[15]以及九龙江口[16]中发现的g23基因分布与其生存环境关系很大,T4型噬菌体基因多样性异常丰富,它的群落结构在湖泊、海洋和稻田环境中具有较大的差异性。根据以前的研究,来自海洋、淡水湖泊、稻田和旱地土壤环境的T4型噬菌体通常形成针对其环境特定的不同群体。作为介于水生环境和陆地环境之间的过渡区域,湿地可能会汇集来自不同环境的不同病毒群落,本研究从湿地沉积物样本中获得的g23基因群落与之前描述的海洋组、湖泊组、稻田组和旱地黑土组有关(图 5)。另外,本研究获得的T4型噬菌体g23基因序列大部分独立于旱地黑土而存在,但与海洋、湖泊等水生生态系统和稻田生态系统中的T4型噬菌体g23基因序列相近,很可能是由于湿地和稻田都是生长水生植被的浅水生态系统,并且稻田属于人工湿地生态系统的一种。
T4型噬菌体g23基因序列的集合或群集(assemblage)可以表征T4型噬菌体群落结构[7]。通过对g23基因群集的UniFrac分析,可以评估不同环境下T4型噬菌体群落结构组成的异同[14]。本研究基于PC1/PC2的主坐标分析(PCoA)图显示,中国东北地区土壤g23群集的样点位置相对集中,包括中国东北稻田土壤、旱地黑土、三江湿地土壤等,而距离海洋、湖泊、日本稻田土壤和水体较远,这一发现进一步支持了地理分隔是决定T4型噬菌体群落分布的主要因素之一[12]。有趣的是,虽然是从全球范围内采集到的3个海水样本和5个湖泊淡水样本,但是根据其采样环境,这些样本位置相对较集中,而即便是与中国东北土壤样品同步采集的稻田水样、三江湿地水样,g23群集在水体中的位置则明显位于与土壤不同的区域(图 6)。由此表明,生态过程是决定T4型噬菌体群落分布的另一个重要因素[12]。本实验获得的东北湿地沉积物g23群集不同于土壤中的T4型噬菌体,而与水生生态系统较靠近,这可能是由于湿地是生长水生植物的浅水生态系统,其中,兴凯湖湖泊湿地的g23群集与其他湖泊中的g23群集很好地聚在一起,表明湖泊环境中的T4型噬菌体群落相似度高于其他生态系统[28]。值得一提的是,虽然本实验中选取的6个湿地样点在地理位置上相对较近(相对于其他采样点),但基于PCoA图显示未聚在一起,说明不同的湿地中T4型噬菌体群落结构差异明显。
综上所述,我们采用PCR-克隆测序技术,以T4型噬菌体g23基因作为分子标记基因对东北典型湿地沉积物中T4型噬菌体进行调查,发现东北典型湿地沉积物中存在着丰富的T4型噬菌体基因序列,既包括来自不同生态系统的T4型噬菌体类群,又包括新的湿地环境g23基因类群;湿地沉积物中g23基因分布与海洋、湖泊及稻田生态系统中g23基因亲缘关系较近,与东北旱地黑土g23基因亲缘关系较远;PCoA分析表明T4型噬菌体群落在不同地点湿地中分异明显。
References
| [1] | Wommack KE, Colwell RR. Virioplankton:viruses in aquatic ecosystems. Microbiology and Molecular Biology Reviews, 2000, 64(1): 69-114. |
| [2] | Mann NH. The third age of phage. PLoS Biology, 2005, 3(5): e182. DOI:10.1371/journal.pbio.0030182 |
| [3] | Weinbauer MG. Ecology of prokaryotic viruses. FEMS Microbiology Reviews, 2004, 28(2): 127-181. DOI:10.1016/j.femsre.2003.08.001 |
| [4] | Paul JH. Microbial gene transfer:an ecological perspective. Journal of Molecular Microbiology and Biotechnology, 1999, 1(1): 45-50. |
| [5] | Brüssow H, Hendrix RW. Phage genomics:small is beautiful. Cell, 2002, 108(1): 13-16. DOI:10.1016/S0092-8674(01)00637-7 |
| [6] | Hambly E, Tétart F, Desplats C, Wilson WH, Krisch HM, Mann NH. A conserved genetic module that encodes the major virion components in both the coliphage T4 and the marine cyanophage S-PM2. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(20): 11411-11416. DOI:10.1073/pnas.191174498 |
| [7] | Filée J, Tétart F, Suttle CA, Krisch HM. Marine T4-type bacteriophages, a ubiquitous component of the dark matter of the biosphere. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(35): 12471-12476. DOI:10.1073/pnas.0503404102 |
| [8] | Wang GH, Liu JJ, Kimura M. Genetic diversity of major capsid genes (g23) of T4-type bacteriophages in natural environments-a review. Acta Microbiologica Sinica, 2011, 51(6): 732-739. (in Chinese) 王光华, 刘俊杰, KimuraM. 自然环境中T4型噬菌体g23基因多样性研究进展. 微生物学报, 2011, 51(6): 732-739. |
| [9] | Wang GH, Yu ZH, Liu JJ, Jin J, Liu XB, Kimura M. Molecular analysis of the major capsid genes (g23) of T4-type bacteriophages in an upland black soil in Northeast China. Biology and Fertility of Soils, 2011, 47(3): 273-282. DOI:10.1007/s00374-010-0533-1 |
| [10] | Liu JJ, Wang GH, Zheng CY, Yuan XH, Jin J, Liu XB. Specific assemblages of major capsid genes (g23) of T4-type bacteriophages isolated from upland black soils in Northeast China. Soil Biology and Biochemistry, 2011, 43(9): 1980-1984. DOI:10.1016/j.soilbio.2011.05.005 |
| [11] | Liu JJ, Wang GH, Wang Q, Liu JD, Jin J, Liu XB. Phylogenetic diversity and assemblage of major capsid genes (g23) of T4-type bacteriophages in paddy field soils during rice growth season in Northeast China. Soil Science and Plant Nutrition, 2012, 58(4): 435-444. DOI:10.1080/00380768.2012.703610 |
| [12] | Liu JJ, Yu ZH, Wang XZ, Jin J, Liu XB, Wang GH. The distribution characteristics of the major capsid gene (g23) of T4-type phages in paddy floodwater in Northeast China. Soil Science and Plant Nutrition, 2016, 62(2): 133-139. DOI:10.1080/00380768.2016.1163507 |
| [13] | Bellas CM, Anesio AM. High diversity and potential origins of T4-type bacteriophages on the surface of Arctic glaciers. Extremophiles, 2013, 17(5): 861-870. DOI:10.1007/s00792-013-0569-x |
| [14] | Zheng CY, Wang GH, Liu JJ, Song CC, Gao HX, Liu XB. Characterization of the major capsid genes (g23) of T4-type bacteriophages in the wetlands of northeast China. Microbial Ecology, 2013, 65(3): 616-625. DOI:10.1007/s00248-012-0158-z |
| [15] | He MQ, Cai LL, Zhang CL, Jiao NZ, Zhang R. Phylogenetic diversity of T4-type phages in sediments from the subtropical pearl river estuary. Frontiers in Microbiology, 2017, 8: 897. DOI:10.3389/fmicb.2017.00897 |
| [16] | Liu L, Cai LL, Zhang R. Co-existence of freshwater and marine T4-like myoviruses in a typical subtropical estuary. FEMS Microbiology Ecology, 2017, 93(11): fix119. |
| [17] | Adriaenssens EM, Cowan DA. Using signature genes as tools to assess environmental viral ecology and diversity. Applied and Environmental Microbiology, 2014, 80(15): 4470-4480. DOI:10.1128/AEM.00878-14 |
| [18] | McJannet D, Wallace J, Keen R, Hawdon A, Kemei J. The filtering capacity of a tropical riverine wetland:Ⅱ. Sediment and nutrient balances. Hydrological Processes, 2012, 26(1): 53-72. DOI:10.1002/hyp.v26.1 |
| [19] | Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [20] | Lozupone C, Knight R. UniFrac:a new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology, 2005, 71(12): 8228-8235. DOI:10.1128/AEM.71.12.8228-8235.2005 |
| [21] | Fujii T, Nakayama N, Nishida M, Sekiya H, Kato N, Asakawa S, Kimura M. Novel capsid genes (g23) of T4-type bacteriophages in a Japanese paddy field. Soil Biology and Biochemistry, 2008, 40(5): 1049-1058. DOI:10.1016/j.soilbio.2007.11.025 |
| [22] | Nakayama N, Asakawa S, Kimura M. Comparison of g23 gene sequence diversity between Novosphingobium and Sphingomonas phages and phage communities in the floodwater of a Japanese paddy field. Soil Biology and Biochemistry, 2009, 41(2): 179-185. DOI:10.1016/j.soilbio.2008.06.008 |
| [23] | Wang GH, Murase J, Taki K, Ohashi Y, Yoshikawa N, Asakawa S, Kimura M. Changes in major capsid genes (g23) of T4-type bacteriophages with soil depth in two Japanese rice fields. Biology and Fertility of Soils, 2009a, 45(5): 521-529. DOI:10.1007/s00374-009-0362-2 |
| [24] | Wang GH, Jin J, Asakawa S, Kimura M. Survey of major capsid genes (g23) of T4-type bacteriophages in rice fields in Northeast China. Soil Biology and Biochemistry, 2009b, 41(2): 423-427. DOI:10.1016/j.soilbio.2008.11.012 |
| [25] | López-Bueno A, Tamames J, Velázquez D, Moya A, Quesada A, Alcamí A. High diversity of the viral community from an Antarctic lake. Science, 2009, 326(5954): 858-861. DOI:10.1126/science.1179287 |
| [26] | Butina TV, Belykh OI, Maksimenko SY, Belikov SI. Phylogenetic diversity of T4-like bacteriophages in Lake Baikal, East Siberia. FEMS Microbiology Letters, 2010, 309(2): 122-129. |
| [27] | Huang HZ, Cheng K, Xu M, Zhao YJ. Genetic diversity of T4 virioplankton, inferred from g23 gene, in Wuhan Donghu Lake. China Environmental Science, 2011, 31(3): 443-447. (in Chinese) 黄慧珍, 程凯, 许敏, 赵以军. 基于g23基因的武汉东湖T4类浮游病毒遗传多样性. 中国环境科学, 2011, 31(3): 443-447. |
| [28] | Butina TV, Belykh OI, Potapov SA, Sorokovikova EG. Diversity of the major capsid genes (g23) of T4-like bacteriophages in the eutrophic Lake Kotokel in East Siberia, Russia. Archives of Microbiology, 2013, 195(7): 513-520. DOI:10.1007/s00203-013-0884-8 |
| [29] | Zhong X, Jacquet S. Differing assemblage composition and dynamics in T4-like myophages of two neighbouring sub-alpine lakes. Freshwater Biology, 2014, 59(8): 1577-1595. DOI:10.1111/fwb.2014.59.issue-8 |
