李秋霞1#, 曹理想1#, 谭红铭1, 孙健2, 崔勇3, 徐润林1


1.中山大学生命科学学院, 广东 广州 510275;
2.国家文物局水下文化遗产保护中心, 北京 100192;
3.广东省文物保护与考古研究所, 广东 广州 510075
收稿日期:2017-10-09;修回日期:2017-12-16;网络出版日期:2018-01-19
基金项目:国家文物局专项(413174400001)
*通信作者:徐润林, Tel:+86-20-84110786;E-mail:xurunlin_1960@yahoo.com
#共同第一作者
摘要:[目的]通过对"南海Ⅰ号"沉船出水木质文物标本中细菌类群的分析,了解饱水木质文物中的细菌类群并推测细菌对木质文物损害的机制。[方法]应用Illumina MiSeq测序平台对采自该沉船的10份木质文物标本中细菌V3-V4序列进行测序与分析,比较各标本中细菌群落的组成差异。[结果]根据97%序列相似性得到3 780不同的细菌OTUs,分属34目、35科的187个属;多数细菌OTU属于变形菌门(Proteobacteria)细菌,占全部细菌OTU的52.9%,在细菌纲水平上γ-变形菌纲(γ-Proteobacteria)(占17.9%)是丰度最高的细菌纲。德沃氏菌属(Devosia)(3.5%)是"南海一号"沉船样品丰度最高的属,其他分别属于甲基娇养杆菌属(Methylotenera)(2.4%)、鼠尾菌属(Muricauda)(1.2%)。其中氢噬胞菌属(Hydrogenophaga)、中国农大湖积物杆菌(Lacibacter cauensis)、德氏食酸菌(Acidovorax delafieldii)、德沃斯氏菌属(Devosia)、沉积物杆状菌属(Sediminibacterium)、缺陷短孢单胞菌(Brevundimonas diminuta)和门多萨假单胞菌(Pseudomonas mendocina)在所有样品中均可检测到。[结论]"南海Ⅰ号"沉船出水木质文物存在着种类丰富的好氧与厌氧细菌种类,多种细菌类群具有较好的纤维素降解能力与铁硫元素转化能力,控制细菌群落中参与分解纤维素的细菌与铁硫循环菌活性对于保护木质文物有重要作用。
关键词: 饱水木质文物 细菌群落 高通量测序 “南海Ⅰ号”沉船
Bacterial communities in the waterlogged wooden cultural relics from the "Nanhai No. 1" shipwreck
Qiuxia Li1#, Lixiang Cao1#, Hongming Tan1, Jian Sun2, Yong Cui3, Runlin Xu1


1.School of Life Sciences, Sun Yat-sen University, Guangzhou 510275, Guangdong Province, China;
2.Underwater Cultural Heritage Protection Center, State Administration of Cultural Heritage, Beijing 100192, China;
3.The Cultural Relics Protection and Archaeology Institute of Guangdong, Guangzhou 510075, Guangdong Province, China
Received 9 October 2017; Revised 16 December 2017; Published online 19 January 2018
*Corresponding author: Xu Runlin, Tel:+86-20-84110786;E-mail:xurunlin_1960@yahoo.com
Supported by the Special Research Project of Underwater Heritage Protection from the State Administration of Cultural Heritage of China (413174400001)
Those authors contributed equally work
Abstract: [Objective]To understand the distribution of microorganisms and their effects on the waterlogged wooden relics collected from the "Nanhai No. 1" shipwreck.[Methods]Prokaryotic microorganisms in 10 wooden relics samples collected from the "Nanhai No. 1" shipwreck were identified by using high throughput sequencing technique. The composition and diversity of the bacterial communities in each sample were analyzed.[Results]Total 121 394 313 bacterial sequences with an average reading length of 458 bp were obtained. There were 3 780 different OTUs based on the similarity of the 97% sequences. According to the sequence in the GenBank database, 34 orders, 35 families and 187 genera of bacteria were involved in the wooded relics. Among these bacteria, most species were from Proteobacteria, and the species from Bacteroidetes ranked in the second. In the class level, the species from γ-Proteobacteria and α-Proteobacteria contributed more on community diversity. Hydrogenophaga, Lacibacter cauensis, Acidovorax delafieldii, Devosia, Sediminibacterium, Brevundimonas diminuta and Pseudomonas mendocina were found in all samples, and Muricauda, Thiobacillus, Rhodoplanes, Emticicia, Methylotenera mobilis and Flavobacterium were appeared with higher frequency of bacterial taxa. Principal component analysis showed that there were some differences in the composition of the bacterial community in different cultural relics.[Conclusion]There were rich biodiversity in prokaryotic microorganisms, both aerobic and anaerobic, in the archaeological wood from "Nanhai No. 1" shipwreck. The bacteria species, which participate in the decomposition of cellulose in the bacterial communities, resulted in a great challenge to the protection of cultural relics. At the same time, impact from iron-sulfur cycle bacteria should be taken into consideration due to they were existing in these relics.
Key words: waterlogged wooden relics bacterial community high-throughput sequencing technology "Nanhai No.1" shipwreck
木质文物是集建筑、雕刻、彩绘、装饰等于一体的综合艺术形式,具有很高的科学、艺术、历史和欣赏价值。木质文物保护是文物保护工作者的重要工作内容。在木质文物的长期保存过程中,容易发生干缩、变形、开裂、起翘、粉化、脱落、霉斑、变色等多种损害类型,木质文物的基本结构单位是高等植物细胞,所以木质文物损害以生物损害为主,其次为物理、化学损害[1]。因此从本质上讲,对木质文物的保护就是要防止构成文物的植物细胞的分解。一般认为:在经历了长年的地下埋藏、水浸及自然降解,细胞壁内的半纤维素、纤维素和木质素等主要成分由于细菌的分解作用出现了大量的流失,细胞壁强度降低,胞壁变薄,整个细胞变得极为脆弱,加上胞内的含水率一般都超过200%以上,细胞形态的维持从依靠细胞壁为主转为由进入到细胞内的水来支撑[2]。与自然界中其他有机物分解过程相似,虽然木质材料在海洋环境的分解过程相对缓慢,其分解和降解过程也必然是细菌参与的结果[3]。因此了解海底沉船木质文物中细菌的类群对于认识沉船木质文物生物损害机制具有重要意义。
发掘出水的海底木质文物面临着从低氧(或厌氧)环境转为有氧环境的变化,这一变化过程必然导致木质文物中细菌类群的变化以及细菌对木质文物分解或降解活动的加剧。长期以来,由于微生物学检测技术的局限,人们对水下木质文物降解过程中的细菌类群研究较少,影响了对水下木质文物的合理保护。国外采用细菌16S rRNA基因文库构建与DGGE技术对采自英格兰、德国、意大利、挪威、瑞典和荷兰的108份不同年代的饱水木质文物样本中的细菌群落进行分析,鉴定出了囊泡短波单胞菌(Brevundimonas vesicularis)、假单胞菌属、伯杰氏菌属(Bergeyella sp.)、金黄杆菌属(Chryseobacterium sp.)等多种细菌[4]。随着细菌测序技术的发展,国外****利用新型测序技术对饱水木质文物的细菌性病害开展系统研究,基本阐明了细菌在饱水木材内环境中的类群变化与生命活动特点[5-7]。而在我国,到目前为止,针对文物细菌性病害的研究或调查主要集中在石质、壁画或岩画等无机质文物[8-11],而对各类出土(水)饱水木质文物的细菌性病害虽已引起重视[2, 12-13],但具体研究并不多见[14]。随着我国沿海越来越多木质沉船被发现,由此面临的水下木质文物的生物损伤状况评估、后期的文物清理和保护等涉及到细菌学问题也会越来越多。
随着国内对文物保护的重视,多学科的研究人员参与到木质文物保护中。为更准确地了解饱水木质文物中细菌类群的组成和分布,进一步探讨水下木质文物分解与细菌间的联系[4, 15],本研究以我国2007年发掘出水的宋代古沉船“南海Ⅰ号”为例,利用高通量测序技术分析该沉船部分木质文物内部的细菌群落组成,希望所得结果能为水下木质文物状况的评估和保护提供参考。
1 材料和方法 1.1 研究对象简介 “南海Ⅰ号”是1987年发现于广东省阳江海域的一艘宋代早期(距今约800多年)的木质远洋商船。沉没地点距海岸边33 km。沉船长30.4 m、宽9.8 m、高4.2 m。该沉船沉没的原始位置处于海面下24 m的淤泥底质环境中,沉船表面覆盖有1.0–1.5 m厚的淤泥。经过多次水下勘查,确认该沉船船体保存较好,上下甲板以下部分结构基本完整,船舱内满载大量陶瓷、金属、漆器和石质文物。经过多方面学科的论证和实地勘查,国家及广东省有关部门联合,该沉船于2007年12月被整体打捞出水并移入阳江海陵岛的广东海上丝绸之路博物馆水下保存,供进一步的考古发掘和文物保护[16]。
1.2 样品采集 实验所用样品于2013年直接采集自沉船木料。使用灭菌解剖刀自水下木材刮取5 g样品,放入50 mL圆锥试管,置于冰上,运输到实验室;另取1 g样品立刻放入液氮罐,用于DNA提取。先后共取得10个木材样品,各样品所属文物信息见表 1。
表 1. 实验中各木质样品的来源 Table 1. Samples collected from the wooden relics
| Sample ID of the wooden relics | Sample number used in the experiment |
| T0301②: Wood-13 | T3 |
| 2014NHIT0302② | T4 |
| T0301③: Wood-12 (Nanhai No.1) | T17 |
| 2014NHIT0502④-24 | T18 |
| T0301②: Wood-9 (Nanhai No.1) | T13 |
| 2014NHIT0501③: Wood-4 | T16 |
| T0301②: Wood-4 | T1 |
| 2014NHIT0102②: Wood-12 | T6 |
| 2014NHIT0502④: Wood-9 | T2 |
| 2014NHIT0501③: Wood-33 | T5 |
表选项
在本次调查中,选取了“南海Ⅰ号”沉船的10个木质文物样品,样品编号见表 1。
1.3 总DNA提取 称取0.5 g木材样品,经液氮研磨后,使用PowerSoil? DNA Isolation Kit (Mo Bio)土壤DNA提取试剂盒,按照试剂盒说明书的提取步骤,提取总DNA。1%凝胶电泳检测总DNA浓度和纯度后,使用微量紫外分光仪定量分析DNA浓度和纯度。
1.4 PCR扩增和IlluminaMiSeq测序 Illumina MiSeq测序按照下列步骤进行:细菌通用引物(27F和1492R引物)扩增饱水古木的细菌16S rRNA基因全长(1.2 kb)。PCR反应混合液(25 μL)包括1×PCR buffer (TaKaRa),200 μmol/L dNTP,每个引物0.2 μmol/L,3 mmol/L MgCl2,2.5 UTaqDNA聚合酶(TaKaRa)。PCR程序:94 ℃ 5 min;94 ℃ 1 min;54 ℃ 1 min;72 ℃ 1 min,35个循环,最后一步72 ℃延伸10 min。PCR产物进一步用S-D-Bact-0341-b-S- 17 (5′-CCTACG GGNGGCWGCAG-3′)和S-D-Bact-0785-a-A-21 (5′- GACTACHVGGGTATCTAATCC-3′)扩增细菌16S rRNA基因V3-V4高变区[17]。正反引物加上接头,连接序列。不同的样品的反向引物加上不同的标签序列以便区分混合在一起测序的样品。所有的PCR反应在30 μL容量的PCR管进行,PCR混合液包括有15 μL Phusion?高保真PCR MasterMix (New England Biolabs)和0.5 U AccuPrimerTMTaq DNA聚合酶(LifeTechnologies,USA);0.2 μmol/L正反引物,10 ng模板DNA。PCR程序:98 ℃ 1 min;98 ℃ 10 s;50 ℃ 30 s;72 ℃ 60 s;30个循环;72 ℃ 5 min。PCR扩增完成后,取2 μL PCR产物2%凝胶电泳检测扩增效果。样品的3次PCR产物混合,用GeneJET凝胶提取试剂盒(ThermoScientific)纯化,Agilent2100Bioanalyzer分析混合PCR产物;高灵敏度DNA芯片(Agilent Technologies,德国)产物均一化。根据NEBNext? Ultra?DNA文库制备试剂盒的说明书制备文库并索引标记。Qubit@2.0 Fluorometer (ThermoScientific)和Agilent Bioanalyzer 2100 system (Agilent Technologies,Germany)检测文库质量。整个测序过程在Illumina MiSeq (北京贝瑞和康公司)测序平台进行。
1.5 数据拼接和预处理 应用FLASH软件(V1.2.7)[18],依据overlapping双末端序列去除正反引物序列,根据不同的标签序列,所有同一个标签的序列归类到一个样品,即获得原始tags。参照Mago? & Salzberg[18]的方法对原始tags进一步过滤低质量tags。用QIIME软件(V1.7.0)[19]检测获得的clean tags的质量,并去除低质量tags[20]。用UCHIME算法[21-22]去除嵌合体。通过UPARSE软件包(Uparsev7.0.1001)[23],使用UPARSE-OTU和UPARSE-OUTref算法[19],将得出的有效序列按照97%序列相似性分成不同的操作分类单元,最后根据RDPclassfier序列生成微生物分类信息[24]。提交序列信息到NCBI(the National Center for Biotechnology Information),序列读数获得的序列号为SRP062743。
1.6 数据分析 应用QIIME软件包,通过主要成分分析(PCA)进行群类分析,QIIME计算加权和非加权的unifrac距离,用于β多样性的系统发育分析[24],不同微生物类群的系统发育关系利用KRONA[25]进一步列出。
2 结果和分析 2.1 木质文物样品中的细菌科属类群 经质量过滤序列,获得平均读长为458 bp的共121394313个细菌16S rRNA基因V3–V4序列,根据97%序列相似性分属3780不同的细菌OTUs。根据对细菌OTU的对比分析,发现多数细菌OTU属于变形菌门(Proteobacteria)细菌,占全部细菌OTU的52.9%,尤其在T16样品占的比例最高(占72.5%);拟杆菌门(Bacteroidetes)属于丰度仅次于变形菌门的细菌,占22.1%,在T3样品占的比例最高(达47.7%);其他丰度较高的细菌OTU分别属于绿弯菌门(Chloroflexi) (12.6%)、放线菌门(Actinobacteria) (2.4%)、厚壁菌门(Firmicutes) (2.1%)、酸杆菌门(Acidobacteria) (1.6%)、浮霉菌门(Planctomycetes) (1.2%)和疣微菌门(Verrucomicrobia) (0.2%) (图 1)。
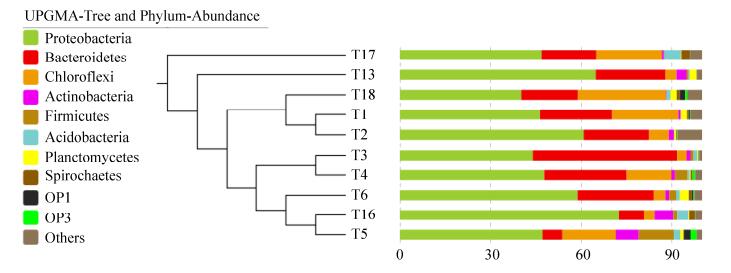 |
| 图 1 Weighted unifrac分析显示的“南海Ⅰ号”部分木质文物样品中细菌类群 Figure 1 Weighted unifrac analysis on bacterial groups in the wooden samples from "Nanhai No. 1" shipwreck. |
| 图选项 |
在细菌纲水平上γ-变形菌纲(γ-Proteobacteria) (占17.9%)是丰度最高的细菌纲,在T2样品中所占比例最高(占有29.7%);α-变形菌纲(α-Proteobacteria) (17.6%)是丰度第二的纲,其中在T13样品中占36.9%;其他丰度较高的纲有β-变形菌纲(β-Proteobacteria) (16.0%)、脱卤拟球菌纲(Dehalococcoidetes) (10.1%)、纤维粘网菌(Cytophagia) (9.3%)、黄杆菌纲(Flavobacteriia) (6.5%)、Saprospirae纲(3.2%)、厌氧绳菌纲(Anaerolineae) (2.2%)和梭菌纲(Clostridia) (1.6%)。
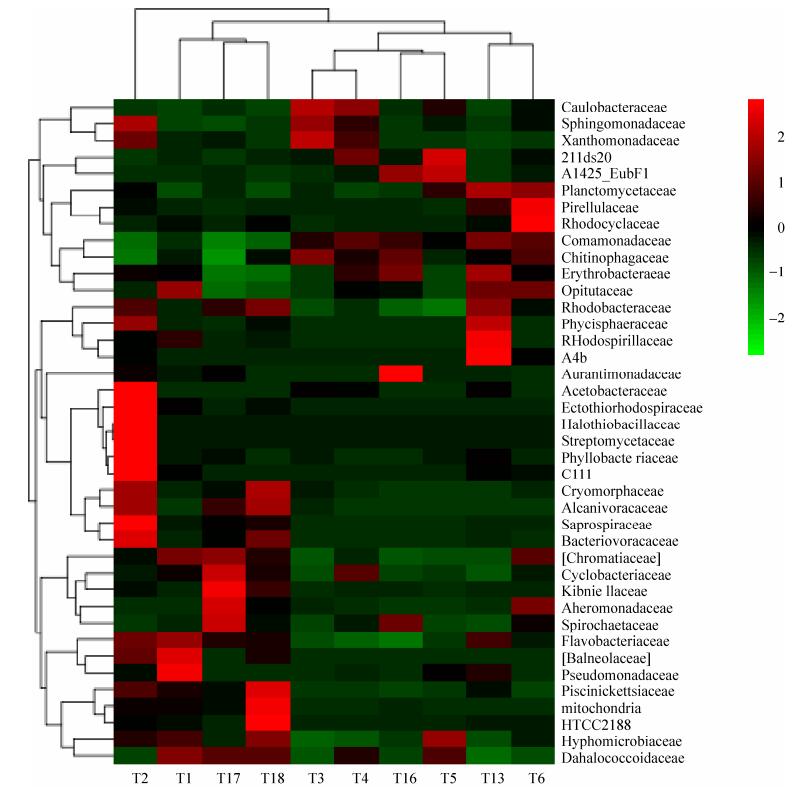
在目的分类界元上,共检测到有34个目的细菌(图 2),主要类群包括有脱卤拟球菌目(Dehalococcoidales) (9.6%)、噬纤维菌目(Cytophagales) (9.3%)、伯克氏菌目(Burkholderiales) (8.8%)、交替单胞菌目(Alteromonadales) (7.6%)、根瘤菌目(Rhizobiales) (6.9%)和黄杆菌目(Flavobacteriales) (6.5%)。
 |
| 图 2 “南海Ⅰ号”部分木质文物样品中检测到的34目细菌 Figure 2 34 bacteria Orders detected in wooden relics samples from "Nanhai No.1" shipwreck. |
| 图选项 |
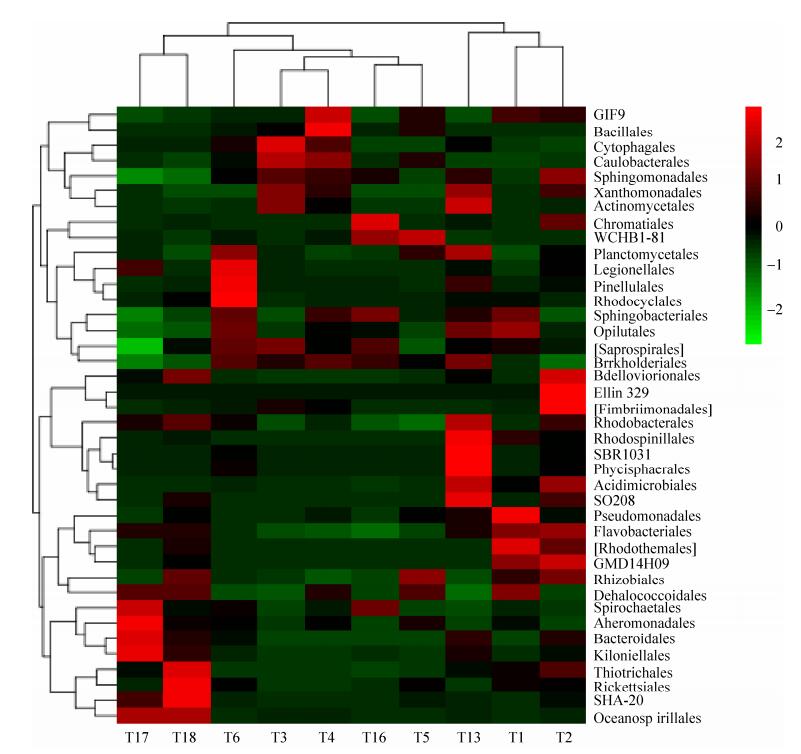
在科水平上,共检出丰度较高的35个科的细菌,其中脱卤拟球菌科(Dehalococcoidaceae)最高,占9.6%;噬纤维菌科(Cytophagaceae) (7.7%)、生丝微菌科(Hyphomicrobiaceae) (5.1%)、黄杆菌科(Flavobacteriaceae) (4.9%)和红螺菌科(Rhodospirillaceae) (4.0%)呈现出来较高的丰度(图 3)。
 |
| 图 3 “南海Ⅰ号”部分木质文物样品检测到的35科细菌 Figure 3 35 bacteria Families detected in wooden relics samples from "Nanhai No.1" shipwreck. |
| 图选项 |
在细菌属水平上,共检测出细菌属187个,其中丰度较高的细菌属37个(图 4)。德沃氏菌属(Devosia) (3.5%)是“南海一号”沉船木质样品发现的丰度最高的属,其他所占比例较高的类群分别属于甲基娇养杆菌属(Methylotenera) (2.4%)、鼠尾菌属(Muricauda) (1.2%)、嗜酸菌属(Acidovorax) (1.2%)、黄杆菌属(Flavobacterium) (1.7%)、海杆菌属(Marinobacter) (1.0%)、红游动菌属(Rhodoplanes) (0.9%)、湖积物杆菌属(Lacibacter) (0.4%)、泥杆菌属(Lutibacterium) (0.6%)、中生根瘤菌属(Mesorhizobium) (0.4%)、鞘脂菌属(Sphingobium) (0.9%)、硫杆菌属(Thiobacillus) (0.9%)和盐硫杆菌属(Halothiobacillus) (0.8%)。
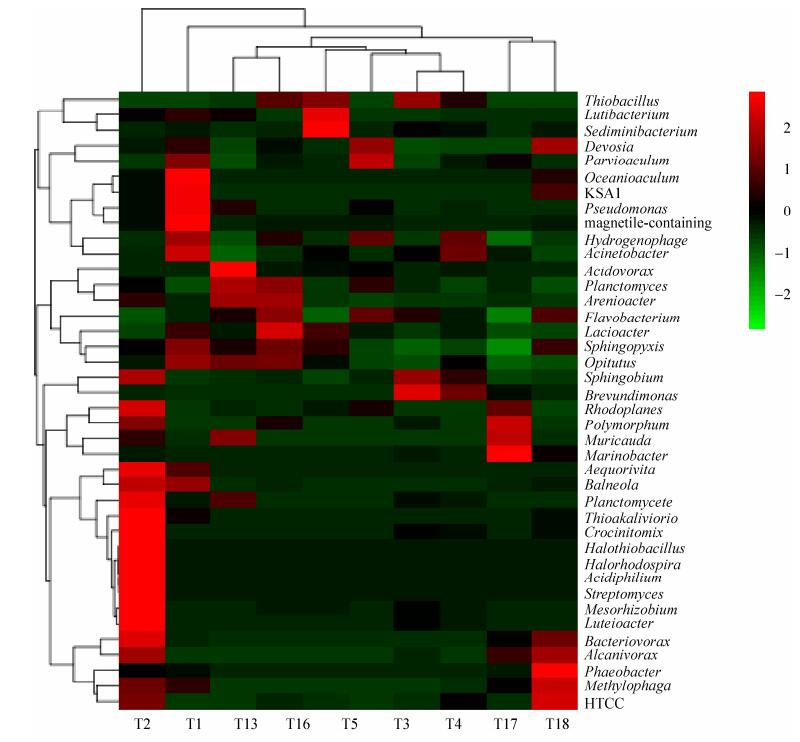 |
| 图 4 “南海Ⅰ号”部分木质文物样品中丰度较高的37属细菌 Figure 4 37 bacteria Genera with high abundance in wooden relics samples from "Nanhai No.1" shipwreck. |
| 图选项 |
2.2 各木质文物样品中优势菌群 表 2列出了“南海Ⅰ号”沉船的10个木质样品中细菌的分布信息。
表 2. “南海Ⅰ号”木质样品中细菌属水平上的分布 Table 2. The distribution of bacteria genera in the wooden relics samples from "Nanhai No. 1" shipwreck
| Sample number | OTUs | Amount of bacterial genus | Representative genus or species |
| T3 | 413 | 113 | Methylotenera mobilis, Sphingobium, Brevundimonas diminuta, Thiobacillus |
| T4 | 428 | 110 | Thiobacillus, Methylotenera mobilis, Brevundimonas diminuta, Bacillus |
| T17 | 260 | 60 | Muricauda, Marinobacter, Pseudoalteromonas, Rhodoplanes, Idiomarina |
| T18 | 409 | 82 | Devosia, Methylophaga, Idiomarina |
| T13 | 395 | 76 | Acidovorax delafieldii, Emticicia, Muricauda, Saccharospirillum |
| T16 | 257 | 66 | Devosia, Thiobacillus, Lutibacterium, Sediminibacterium |
| T1 | 420 | 73 | Devosia, Oceanibaculum indicum, Hydrogenophaga, Pseudomonas mendocina |
| T6 | 480 | 107 | Thiobacillus, Lacibacter cauensis, Flavobacterium |
| T2 | 395 | 80 | Rhodoplanes, Halothiobacillus, Sphingobium, Mesorhizobium |
| T5 | 323 | 77 | Methylotenera mobilis, Devosia, Flavobacterium, Hydrogenophaga, Caldicoprobacter |
表选项
在所有检测到的细菌中,氢噬胞菌属(Hydrogenophaga)、中国农大湖积物杆菌(Lacibacter cauensis)、德氏食酸菌(Acidovorax delafieldii)、德沃斯氏菌属(Devosia)、沉积物杆状菌属(Sediminibacterium)、缺陷短孢单胞菌(Brevundimonas diminuta)和门多萨假单胞菌(Pseudomonas mendocina)出现在所有受检木质文物中;鼠尾杆菌属(Muricauda)、硫杆菌属(Thiobacillus)、红游动菌属(Rhodoplanes)、菌保所杆菌属(Emticicia)、活动甲基娇养杆菌(Methylotenera mobilis)和黄杆菌属(Flavobacterium)出现在9个受试样品中。
2.3 木质文物样品中细菌类群的Alpha多样性分析 通过计算Chao1、PD_whole_tree、OS、Shannon指数和Simpson指数等参数,得出各受检样品中微生物的alpha多样性(表 3)。
表 3. “南海Ⅰ号”沉船木质样品中细菌群落的α-多样性分析 Table 3. The α- diversity indices of bacterial communities in sunken wood from "Nanhai No.1"
| Samples | Structural parameters of bacterial community* | |||||
| Chao1 | ACE | Shannon index | Simpson index | Coverage | OS | |
| T3 | 484.25 | 499.88 | 4.58 | 0.83 | 0.997 | 413 |
| T4 | 497.49 | 517.66 | 5.51 | 0.94 | 0.996 | 428 |
| T17 | 282.68 | 305.96 | 4.72 | 0.92 | 0.998 | 260 |
| T18 | 423.81 | 443.09 | 5.91 | 0.96 | 0.998 | 409 |
| T13 | 417.00 | 431.84 | 5.77 | 0.93 | 0.997 | 395 |
| T16 | 310.05 | 328.50 | 4.82 | 0.91 | 0.997 | 257 |
| T1 | 455.57 | 494.42 | 5.98 | 0.96 | 0.996 | 420 |
| T6 | 525.50 | 547.42 | 6.07 | 0.95 | 0.995 | 480 |
| T2 | 463.70 | 479.99 | 5.99 | 0.96 | 0.996 | 395 |
| T5 | 390.23 | 401.80 | 5.48 | 0.95 | 0.996 | 323 |
| *Chao1: the index of species richness, the larger the value, the more the total number of species; ACE: an index used to estimate the number of OUTs in the community; Coverage: the sequencing depth index; OS: the number of OTUs observed. | ||||||
表选项
2.4 不同类群细菌在木质文物样品中的分布 虽然所检测木质文物样品中均存在多种细菌,但不同样品中细菌群落组成差别较大(图 5),如在样品T5中,丰度最高的细菌属是甲基娇养杆菌属(Methylotenera);在样品T2、T6、T17、T16、T1和T3中主要的细菌属分别属于红游动菌属(Rhodoplanes)、黄草杆菌(Flavobacterium)、鼠尾菌属(Muricauda)、泥杆菌属(Lutibacterium)、鞘脂菌属(Sphingobium)和食酸菌(Acidovorax)。
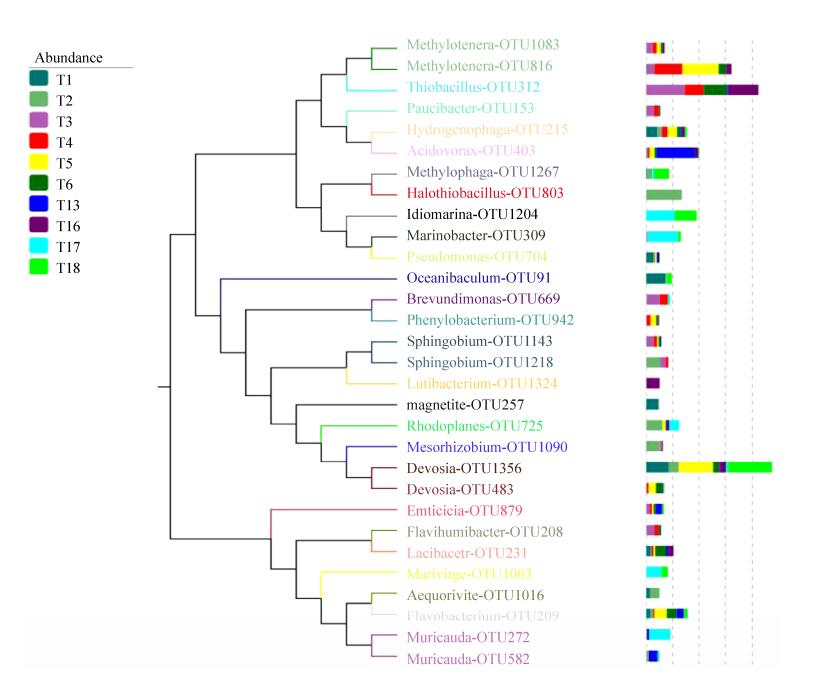 |
| 图 5 “南海Ⅰ号”沉船木质文物样品中的细菌类群系统发育分析 Figure 5 Phylogenetic analysis of bacterial groups in the wooden relics samples from "Nanhai No. 1" shipwreck. |
| 图选项 |
将不同样品细菌高通量测序数据进行主成分分析,根据非加权UniFrac距离生成PCA图(图 6)。沉船木质样品中OTUs组成的比较表明,样品T3与T5的矩阵距离相距较远,显示T3样品和T5样品之间的微生物群落结构组成相差较大。样品T6与T4的矩阵距离较近,显示二者样品质检的细菌类群信息较为接近。相同的道理,样品T1和T2的细菌类群结构也较为接近。
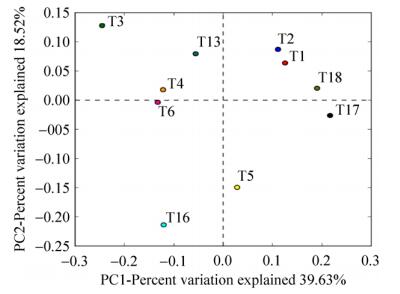 |
| 图 6 “南海Ⅰ号”木质文物样品中细菌群落组成的PCA分析 Figure 6 PCA analysis of bacterial community compositions in the wooden relics samples from "Nanhai No. 1" shipwreck. |
| 图选项 |
3 讨论 通过分析文物中的细菌类群了解危害文物的细菌种类是近年来文物保护中的常见的措施。在欧洲国家,已分离到多种与木质文物腐蚀有关的好氧菌或厌氧菌,最常见的好氧菌有噬胞菌(Cytophaga sp.)、纤维弧菌(Cellvibrio sp.);厌氧菌有梭状芽孢杆菌(Clostridium sp.)、芽孢杆菌(Bacillus sp.)、假单胞菌(Pseudomonas sp.)、黄杆菌(Arthrobacter sp.)、黄草杆菌(Flavobacterium sp.)和螺旋菌(Spirillum sp.)[26-27]。随着分子生物学技术在微生物多样性研究中的广泛应用,越来越多的细菌类群被识别和鉴定[28]。采用分子分析技术对采自英格兰、德国、意大利、挪威、瑞典和荷兰的108份不同年代的饱水木质文物样本中的细菌群落进行分析,鉴定出包括囊泡短波单胞菌(Brevundimonas vesicularis)、假单胞菌、纤维弧菌、伯杰菌(Bergeyella sp.)、金黄杆菌(Chryseobacterium sp.)、黄草杆菌、螺杆菌(Helicobacter sp.)、不动杆菌(Acinetobacter sp.)、土壤杆菌(Agrobacterium sp.)、草酸杆菌(Oxalobacter sp.)、红球菌(Rhodococcus sp.)、芽孢杆菌、食酸菌(Acidovorax sp.)、红杆菌(Rhodobacter sp.)、梭菌(Clostridium sp.)以及草酸杆菌科的Herbaspirillum sp.和Massilia sp.在内的多个菌种;其中以黄草杆菌和噬胞菌最为常见[7]。
本研究从“南海Ⅰ号”10份木质文物标本中鉴定出涵盖了多个门、纲、34目、35科的187属细菌,反映出了该沉船木质文物中的细菌有非常高的生物多样性。细菌群落的α-多样性分析(表 3)也验证了这一点。这一结果明显高于陆上文物的细菌菌群[9-10],但低于沉没于东海浙江沿岸的“小白礁Ⅰ号”(另文发表)。
对于木质文物,其物质基础是纤维素。保证纤维素不被降解是木质文物保护的基础,因此必须考虑文物表面和内部是否有可降解纤维素的细菌存在。本研究检出的全部细菌中,与纤维素分解有关的种类有不少。拟杆菌(Bacteroidetes)是所见细菌类群丰度较高的一类,噬胞菌-黄杆菌(Cytophagaceae-Flavobacterium,CF)群组又在其中占主要比例。噬细胞菌可能利用多糖复合物比如琼脂糖、纤维素和羧甲基纤维素或者几丁质,产乙酸盐、丙酸盐、琥珀酸盐等终产物。这群细菌均为严格好氧菌,许多也是兼性厌氧菌,形态多样,短杆,滑行。海水环境中CF细菌表现出较高丰度和多样性,可能与分解有机物质如纤维素相关[4, 29],梭菌纲(Clostridia)多为厌氧菌,且大部分有纤维素降解能力[30]。交替单胞菌(Alteromonadales)专性嗜压,兼性厌氧利用纤维二糖[31]。另外,疣微菌门(Verrucomicrobia)成员广泛存在于温带至深海低温水域,大多为厌氧菌,具有纤维素降解和硝酸盐还原能力[32]。这些微生物可能与腐蚀木头和产生还原性化合物作为化能自养方式的能量来源有关[33]。正是由于好氧或厌氧降解纤维素的细菌共同存在,对饱水木质文物而言,无论有氧还是无氧,只要文物处于湿润环境,细菌对纤维素的降解都可能发生。
除了直接利用纤维素作为碳源的细菌外,本研究还发现了种类众多的参与铁硫循环菌,如丰度较大的脱卤拟球菌、硫杆菌、盐硫杆菌和海杆菌等。脱卤杆菌通常被认为是参与硫酸盐还原、铁还原、产甲烷或者产乙酸的微生物,其代谢产物又为硫酸盐还原菌提供反应底物(H2S)[34]。硫杆菌一般为化能无机自养,具有氧化Fe和S的能力,参与铁硫生物代谢循环[35]。盐硫杆菌属硫氧化菌,能将还原性H2S氧化成硫酸等[36]。海杆菌为铁氧化菌,能促进Fe离子生成并催化还原性硫化合物生成硫酸[37]。有研究表明,埋于海底沉积物的饱水木质文物通常容易积累还原性铁硫复合物[38-39]。一旦文物被打捞出水进入有氧环境,其积累的还原性铁硫复合物接触氧后被氧化生成硫酸。在酸性条件下,木质文物中的纤维素和半纤维素将出现酸解而使得木质软化;植物细胞壁出现降解,导致木质文物整体结构受到影响[40-43]。
在陆上的湿润环境中,木质材料的纤维素的分解主要是由真菌类微生物完成的;在海洋水环境中,真菌类微生物一般分布较少,尤其在水下低氧环境中,真菌类的存在几乎是不可能的,因此海洋水下木质文物纤维素的分解就更侧重于细菌。总而言之,发掘出水的木质文物必然面临着从低氧(或厌氧)环境转为有氧环境变化,这一变化势必导致微生物对木质文物分解或降解活动的加剧。本研究从“南海Ⅰ号”沉船部分木质文物内部鉴定出了众多类群细菌。由于它们的生理特性差异很大,并有多个类群在厌氧或好氧条件下具有直接分解纤维素的能力。在后期的文物保护上就必须考虑控制细菌的继续繁衍。文物中的水分是细菌(包括厌氧和好氧菌)生长的必要条件之一,也是到目前为止人们可较好控制的条件。因此尽快使得文物脱水干燥可能是文物保护最好的办法,同时在文物处理过程中还要考虑铁硫等元素的清除。
References
| [1] | Zheng LP, Xi ZK, Wu XZ, Wang YF. Investigation on diseases of wood relics in library and their preservation in Chongqing area. Journal of Chongqing Normal University (Edition of Social Sciences), 2008(6): 80-86. (in Chinese) 郑利平, 席周宽, 武仙竹, 王运辅. 重庆地区馆藏木质文物病害调查与保护. 重庆师范大学学报(哲学社会科学版), 2008(6): 80-86. |
| [2] | Xu RL. Research on bacteria causing damage in water-saturated archaeological wood and their identification. Sciences of Conservation and Archaeology, 2013, 25(3): 104-110. (in Chinese) 徐润林. 饱水木质文物的细菌病害及其诊断技术的进展. 文物保护与考古科学, 2013, 25(3): 104-110. |
| [3] | Bj?rdal CG, Nilsson T, Daniel G. Microbial decay of waterlogged wood found in Sweden applicable to archaeology and conservation. International Biodeterioration & Biodegradation, 1999, 43(1/2): 63-73. |
| [4] | Landy ET, Mitchell JI, Hotchkiss S, Eaton RA. Bacterial diversity associated with archaeological waterlogged wood:ribosomal RNA clone libraries and denaturing gradient gel electrophoresis (DGGE). International Biodeterioration & Biodegradation, 2008, 61(1): 106-116. |
| [5] | Holt DM. Bacterial degradation of lignified wood cell walls in aerobic aquatic habitats:Decay patterns and mechanisms proposed to account for their formation. Journal of the Institute of Wood Science, 1983, 9(5): 212-223. |
| [6] | Holt DM, Jones EBG. Bacterial degradation of lignified wood cell walls in anaerobic aquatic habitats. Applied and Environmental Microbiology, 1983, 46(3): 722-727. |
| [7] | Holt DM, Jones EBG, Furtado SEJ. Bacterial decay of wood in aquatic habitats//Record of the 1979 Annual Convention of the British Wood Preserving Association. Cambridge, 1979. |
| [8] | Yu M, Zhu XD, Pan J. Advance in detection methods of microbes on historic stones-a review. Acta Microbiologica Sinica, 2011, 51(11): 1447-1453. (in Chinese) 于淼, 朱旭东, 潘皎. 石质文物微生物检测技术的研究进展. 微生物学报, 2011, 51(11): 1447-1453. |
| [9] | Ge YQ, Li ZM, Sun YZ, Pan J, Wen HA, Guo H. Application of molecular biological techniques in identifying pathogenic microbes on mural paintings. Sciences of Conservation and Archaeology, 2012, 24(2): 14-21. (in Chinese) 葛琴雅, 李哲敏, 孙延忠, 潘皎, 文华安, 郭宏. 壁画菌害主要种群之分子生物学技术检测. 文物保护与考古科学, 2012, 24(2): 14-21. |
| [10] | Yan F, Ge QY, Li Q, Yu M, Zhu XD, Pan J. Analysis of microbial community on the surface of the historic stone and nearby rock samples in Yungang Grottoes. Acta Microbiologica Sinica, 2012, 52(5): 629-636. (in Chinese) 颜菲, 葛琴雅, 李强, 于淼, 朱旭东, 潘皎. 云冈石窟石质文物表面及周边岩石样品中微生物群落分析. 微生物学报, 2012, 52(5): 629-636. |
| [11] | Ma X, Mao L, Ma YT, Wu FS, Wang WF, Ma XJ, An LZ, Feng HY. A review on microbial community ecology in prehistoric Lascaux Cave paintings. Dunhuang Research, 2010(6): 115-120. (in Chinese) 马旭, 毛琳, 马燕天, 武发思, 汪万福, 马晓军, 安黎哲, 冯虎元. 拉斯科洞穴史前壁画微生物生态学研究进展. 敦煌研究, 2010(6): 115-120. |
| [12] | Liu L. Microbial corrosion and anti-corrosion measures for waterlogged wood objects. China Cultural Heritage Scientific Research, 2014(4): 83-85. (in Chinese) 刘亮. 出土饱水木质文物的微生物腐蚀及防腐措施. 中国文物科学研究, 2014(4): 83-85. |
| [13] | Chen JC, Huang X, Chen XL, Chen ZH. Corrosion type and conservation of archaeological waterlogged wood. Materials Review, 2015, 29(6): 96-101. (in Chinese) 陈家昌, 黄霞, 陈晓琳, 陈中行. 出土饱水木质文物的腐蚀病害类型与保护研究进展. 材料导报, 2015, 29(6): 96-101. |
| [14] | Fan XD, Lei Q, Qiu ZM, Zhang J, Wang J, Ma LA. Survey of the species and quantity of microbes from waterlogged wooden lacquer in "Taoist Temple No 1" Chu tomb in Jiangling. Journal of Yangtze University (Natural Science Edition), 2015, 12(15): 51-53. (in Chinese) 范晓丹, 雷琼, 邱祖明, 章俊, 汪俭, 马立安. 江陵天星观一号楚墓出土饱水木漆器微生物种类及数量调查. 长江大学学报(自然科学版), 2015, 12(15): 51-53. |
| [15] | Helms AC, Martiny AC, Hofman-Bang J, Ahring BK, Kilstrup M. Identification of bacterial cultures from archaeological wood using molecular biological techniques. International Biodeterioration & Biodegradation, 2004, 53(2): 79-88. |
| [16] | 刘志远. 2011年"南海Ⅰ号"的考古试掘. 北京: 科学出版社, 2011. |
| [17] | Degnan PH, Ochman H. Illumina-based analysis of microbial community diversity. The ISME Journal, 2012, 6(1): 183-194. DOI:10.1038/ismej.2011.74 |
| [18] | Mago? T, Salzberg SL. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [19] | Edgar RC. UPARSE:highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [20] | Bokulich NA, Subramanian S, Faith JJ, Gevers D, Gordon JI, Knight R, Mills DA, Caporaso JG. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing. Nature Methods, 2013, 10(1): 57-59. DOI:10.1038/nmeth.2276 |
| [21] | Wang Q, Garrity GM, Tiedje JM, Cole JR. Na?ve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [22] | Haas BJ, Gevers D, Earl AM, Feldgarden M, Ward DV, Giannoukos G, Ciulla D, Tabbaa D, Highlander SK, Sodergren E, Methé B, DeSantis TZ, The Human Microbiome Consortium, Petrosino JF, Knight R, Birren BW. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [23] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Pe?a AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [24] | K?ljalg U, Nilsson RH, Abarenkov K, Tedersoo L, Taylor AFS, Bahram M, Bates ST, Bruns TD, Bengtsson-Palme J, Callaghan TM, Douglas B, Drenkhan T, Eberhardt U, Due?as M, Grebenc T, Griffith GW, Hartmann M, Kirk PM, Kohout P, Larsson E, Lindahl BD, Lücking R, Martín MP, Matheny PB, Nguyen NH, Niskanen T, Oja J, Peay KG, Peintner U, Peterson M, P?ldmaa K, Saag L, Saar I, Schüβler A, Scott JA, Senés C, Smith ME, Suija A, Taylor DL, Telleria MT, Weiss M, Larsson KH. Towards a unified paradigm for sequence-based identification of fungi. Molecular Ecology, 2013, 22(21): 5271-5277. DOI:10.1111/mec.12481 |
| [25] | Ondov BD, Bergman NH, Phillippy AM. Interactive metagenomic Visualization in a Web browser. BMC Bioinformatics, 2011, 12: 385. DOI:10.1186/1471-2105-12-385 |
| [26] | Daniel GF, Nilsson T, Singh AP. Degradation of lignocellulosics by unique tunnel-forming bacteria. Canadian Journal of Microbiology, 1987, 33(10): 943-948. DOI:10.1139/m87-166 |
| [27] | Tiano P. Biodegradation of cultural heritage: decay mechanisms and control methods//Ninth ARIADNE Workshop "Historic Material and their Diagnostic". ARCCHIP, Prague, 2002. |
| [28] | Ye JY, Luo GY. Progress in the biodiversity of nonculturable microorganisms and microbial molecular ecology. Microbiology, 2004, 31(5): 111-115. (in Chinese) 叶姜瑜, 罗固源. 未培养微生物的研究与微生物分子生态学的发展. 微生物学通报, 2004, 31(5): 111-115. |
| [29] | Fandino LB, Riemann L, Steward GF, Azam F. Population dynamics of Cytophaga-Flavobacteria during marine phytoplankton blooms analyzed by real-time quantitative PCR. Aquatic Microbial Ecology, 2005, 40: 251-257. DOI:10.3354/ame040251 |
| [30] | Kato S, Haruta S, Cui ZJ, Ishii M, Igarashi Y. Effective cellulose degradation by a mixed-culture system composed of a cellulolytic Clostridium and aerobic non-cellulolytic bacteria. FEMS Microbiology Ecology, 2004, 51(1): 133-142. DOI:10.1016/j.femsec.2004.07.015 |
| [31] | Nogi Y, Hosoya S, Kato C, Horikoshi K. Psychromonas hadalis sp. nov., a novel piezophilic bacterium isolated from the bottom of the Japan Trench. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(6): 1360-1364. DOI:10.1099/ijs.0.64933-0 |
| [32] | Schlesner H, Jenkins C, Staley JT. The phylum Verrucomicrobia: a phylogenetically heterogeneous bacterial group//Dworkin M, Falkow S, Rosenberg E, Schleifer KH, Stackebrandt E. The Prokaryotes. New York: Springer, 2006: 881-896. |
| [33] | Bessette S, Fagervold SK, Romano C, Martin D, Le Bris N, Galand PE. Diversity of bacterial communities on sunken woods in the Mediterranean Sea. Journal of Marine Science and Technology, 2014, 22(1): 60-66. |
| [34] | Finster K. Microbiological disproportionation of inorganic sulfur compounds. Journal of Sulfur Chemistry, 2008, 29(3/4): 281-292. |
| [35] | Preston J, Watts JEM, Jones M. Novel bacterial community associated with 500-year-old unpreserved archaeological wood from King Henry VⅢ's Tudor warship the Mary Rose. Applied and Environmental Microbiology, 2012, 78(24): 8822-8828. DOI:10.1128/AEM.02387-12 |
| [36] | Kelly DP, Wood AP. Reclassification of some species of Thiobacillus to the newly designated genera Acidithiobacillus gen. nov., Halothiobacillus gen. nov. and Thermithiobacillus gen. nov. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(2): 511-516. DOI:10.1099/00207713-50-2-511 |
| [37] | Amin SA, Green DH, Al Waheeb D, G?rdes A, Carrano CJ. Iron transport in the genus Marinobacter. BioMetals, 2012, 25(1): 135-147. DOI:10.1007/s10534-011-9491-9 |
| [38] | Wetherall KM, Moss RM, Jones AM, Smith AD, Skinner T, Pickup DM, Goatham SW, Chadwick AV, Newport RJ. Sulfur and iron speciation in recently recovered timbers of the Mary Rose revealed via X-ray absorption spectroscopy. Journal of Archaeological Science, 2008, 35(5): 1317-1328. DOI:10.1016/j.jas.2007.09.007 |
| [39] | Sandstrom M, Jalilehvand F, Damian E, Fors Y, Gelius U, Jones M, Salome M. Sulfur accumulation in the timbers of King Henry VⅢ's warship Mary Rose:a pathway in the sulfur cycle of conservation concern. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(40): 14165-14170. DOI:10.1073/pnas.0504490102 |
| [40] | Kim YS, Singh AP, Nilsson T. Bacteria as important degraders in waterlogged archaeological woods. Holzforschung, 1996, 50(5): 389-392. DOI:10.1515/hfsg.1996.50.5.389 |
| [41] | Sandstr?m M, Jalilehvand F, Persson I, Gelius U, Frank P, Hall-Roth I. Deterioration of the seventeenth-century warship Vasa by internal formation of sulphuric acid. Nature, 2002, 415(6874): 893-897. DOI:10.1038/415893a |
| [42] | Fors Y. Sulfur-related conservation concerns in marine archaeological wood: the origin, speciation and distribution of accumulated sulfur with some remedies for the Vasa. Doctor Dissertation of Stockholm University, 2008. |
| [43] | Almkvist G, Persson I. Analysis of acids and degradation products related to iron and sulfur in the Swedish warship Vasa. Holzforschung, 2008, 62(6): 694-703. |
