郭睿1#, 王海朋1#, 陈华枝1, 熊翠玲1, 郑燕珍1, 付中民1, 赵红霞2, 陈大福1


1.福建农林大学蜂学学院, 福建 福州 350002;
2.广东省生物资源应用研究所, 广东 广州 510260
收稿日期:2017-11-02;修回日期:2018-01-18;网络出版日期:2018-01-31
基金项目:国家自然科学基金(31702190);现代农业产业技术体系建设专项资金(CARS-44-KXJ7);福建省教育厅中青年教师教育科研项目(JAT170158);福建农林大学科技创新专项基金(CXZX2017343);福建省大学生创新创业训练计划(201610389053)
作者简介:陈大福, 博士, 副教授, 硕士生导师。现任福建农林大学蜂学学院副院长; 农业部现代农业产业技术体系(蜂)岗位科学家; 中国养蜂学会蜜蜂保护专业委员会副主任; 中国蜂产品协会蜂产品生产专业委员会副主任。主要从事蜜蜂病害防治方面的教学与科研, 所在课题组长期致力于蜜蜂病原微生物及其与蜜蜂的互作机制研究, 主持或参与国家自然科学基金项目等国家级和省级项目多项, 发表学术论文50余篇, 参加全国高等农林院校"十一五"规划教材3部教材及4部著作的编写工作
*通信作者:陈大福, Tel/Fax:+86-591-83726835, E-mail:dfchen826@fafu.edu.cn
#并列第一作者
摘要:[目的]本研究利用small RNA-seq技术对球囊菌的纯培养进行测序,对球囊菌的microRNAs miRNAs)进行预测、鉴定和分析,进而构建miRNAs-mRNAs的调控网络。[方法]利用Illumina Hiseq Xten平台对球囊菌菌丝与孢子进行测序,通过相关生物信息学软件对球囊菌的miRNAs进行预测和分析,通过茎环(Stem-loop)PCR对部分miRNAs进行鉴定,利用Cytoskype软件构建miRNAs-mRNAs的调控网络。[结果]本研究共获得48268696条clean reads,预测出118个球囊菌的miRNAs,它们的长度分布介于18–25 nt之间,不同长度的miRNA的首位碱基偏好性差异明显。Stem-loop PCR验证结果显示共有10个miRNAs能够扩增出符合预期的目的片段,说明多数miRNAs可能真实存在。共预测出6529个球囊菌miRNAs的靶基因,其中5725个能够注释到Nr、Swissprot、KOG、GO和KEGG数据库。进一步分析结果显示有24个靶基因注释在MAPK信号通路。Cytoskype软件分析结果显示球囊菌的miRNAs与mRNAs之间存在复杂的调控网络,绝大多数的miRNAs处于调控网络的内部且同时结合多个mRNAs。[结论]本研究率先对球囊菌的miRNAs及miRNAs-mRNAs调控网络进行全面分析,研究结果丰富了对球囊菌miRNAs的认识,为其基础生物学信息提供了有益补充,也为阐明球囊菌致病的分子机理打下了一定基础。
关键词: 蜜蜂 球囊菌 microRNA 靶基因 调控网络
Identification of Ascosphaera apis microRNAs and investigation of their regulation networks
Rui Guo1#, Haipeng Wang1#, Huazhi Chen1, Cuiling Xiong1, Yanzhen Zheng1, Zhongmin Fu1, Hongxia Zhao2, Dafu Chen1


1.College of Bee Science, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
2.Guangdong Institute of Applied Biological Resources, Guangzhou 510260, Guangdong Province, China
Received 2 November 2017; Revised 18 January 2018; Published online 31 January 2018
*Corresponding author: Dafu Chen, Tel/Fax:+86-591-83726835, E-mail:dfchen826@fafu.edu.cn
Supported by the National Natural Science Foundation of China (31702190), by the Earmarked Fund for China Agriculture Research System (CARS-44-KXJ7), by the Teaching and Scientific Research Fund of Education Department of Fujian Province (JAT170158), by the Scientific and Technical Innovation Fund of Fujian Agriculture and Forestry University (CXZX2017343) and by the Undergraduate Innovation and Entrepreneurship Training Program of Fujian Province (201610389053)
#These authors contributed equally to this work
Abstract: [Objective]In this study, pure culture of A. apis was sequenced using sRNA-seq technology, followed by prediction, identification and analysis of A. apis microRNAs. The microRNAs-mRNAs regulation network was further constructed.[Methods]Illumina Hiseq Xten platform was used to sequence mycelium and spores of A. apis, and corresponding softwares were used to predict and analyze A. apis microRNAs, some of which were identified via Stem-loop PCR. Cytoskype software was used to construct A. apis microRNAs-mRNAs regulation network.[Results]A total of 48268696 clean reads were obtained, and 118 miRNAs of A. apis were predicted, whose length was distributed between 18 nt and 25 nt. The preference of the first base of miRNAs with different length was obviously various. Stem-loop PCR result showed target fragments with expected sizes were amplified from 10 microRNAs, implying most of the predicted microRNAs' true existence. In total, 6529 target genes of A. apis microRNAs were predicted, and among them 5725 could be annotated in Nr, Swissprot, KOG, GO and KEGG databases. Further investigation demonstrated 24 target genes were annotated in MAPK signaling pathway. Cytoskype software analysis suggested complicated regulation networks exist between microRNAs and mRNAs in A. apis, and majority of miRNAs inside the networks bind to several mRNAs.[Conclusion]Our findings enrich the understanding of A. apis microRNAs, provide beneficial supplement for basic biology information of A. apis, and lay some foundation for illustrating the molecular mechanism regulating the pathogenesis of this widespread fungal pathogen.
Key words: honeybee Ascosphaera apis microRNA target gene regulation network
蜜蜂球囊菌(Ascosphaera apis,简称球囊菌)特异性侵染蜜蜂幼虫而导致白垩病。该病作为典型的蜜蜂真菌性病害,给世界各国养蜂业造成较大损失[1]。近20年来,国内外****在球囊菌的分类鉴定[2]、形态学[3]、病理学[4]、流行病学[5]、侵染过程[6]、免疫防御[7]及疾病防治[8]等方面开展了一系列研究,但其生长发育机制和致病机理仍缺乏分子水平的深入研究[9],相关组学研究[10]也十分有限。2006年,球囊菌基因组的公布[11]为其分子生物学及组学研究奠定了基础。本课题组前期组装并注释了球囊菌的参考转录组[12],利用转录组数据大规模挖掘球囊菌的SSR位点[13],并对胁迫不同抗性蜜蜂肠道球囊菌的基因表达谱和转录组变化进行了全面解析[14-15]。
真核生物基因组编码大量的具有功能的非编码RNA (non-coding RNAs,ncRNAs)。根据长度,ncRNAs可分为长度小于200 nt的小RNA,如microRNA (miRNA),以及长度大于200 nt的长链非编码RNA (long non-coding RNAs,lncRNAs)[16]。miRNA通过序列互补配对与靶mRNA的特定区域结合,从而下调其表达水平或使其降解[17]。近年来,随着高通量测序技术与生物信息学方法的快速发展和融合,大量的动植物miRNAs被预测和鉴定出来[18-19]。2010年,Lee等通过深入分析QDE-2蛋白相关的小RNA,鉴定出粗糙脉孢菌(Neurospora crassa)中的miRNA及5种小RNA的生物合成途径[20],这是有关真菌miRNA研究的首例报道。此后,在核盘菌(Sclerotinia sclerotiorum)[21]、绿僵菌(Metarhizium anisopliae)[22]、里氏木霉(Trichoderma reesei)[23]和新月弯孢菌(Curvularia lunata)[24]等真菌中也陆续预测出miRNAs。Liu等利用Illumina HiSeq技术对C. lunata的分生孢子和菌丝进行测序,预测出1012个已注释miRNAs和48个未注释miRNAs,并基于生物信息学分析结果推测这些miRNAs的靶基因在分子孢子的萌发和菌丝生长过程发挥重要作用[24]。Lin等利用small RNA-seq (sRNA-seq)技术对立枯丝核菌(Rhizoctonia solani)感染后6个时间点的水稻叶片上的菌丝进行测序,预测出177个R. solani的miRNAs,并通过miRNA的靶基因分析得到15个候选的致病miRNAs[25]。然而对于球囊菌,至今仍无miRNA及其孢子萌发、菌丝生长和致病性等方面作用的相关研究报道。为最大限度地预测和鉴定球囊菌的miRNAs,本研究在前期研究基础上利用sRNA-seq技术对实验室条件下培养的球囊菌菌丝和孢子混合样品进行测序,结合生物信息学和分子生物学方法对球囊菌的miRNA进行预测、分析及鉴定,进而构建球囊菌的miRNAs-mRNAs调控网络。研究结果可为球囊菌的基础生物学信息提供有益的补充,也为在分子水平阐明其致病的分子机理打下一定基础。
1 材料和方法 1.1 供试球囊菌 本研究使用的球囊菌菌株由福建农林大学蜂学学院蜜蜂保护学实验室保存并活化。
1.2 主要试剂及仪器 Small RNA文库构建试剂盒NEB Next Ultra small RNA Sample Library Prep Kit for Illumina RNA为美国NEB公司产品。抽提试剂盒和Superscript Ⅱ Reverse Transcriptase为日本TaKaRa公司产品。DNA marker为广东东盛生物科技有限公司产品。其他试剂均为国产分析纯。
超净工作台购自苏州安泰空气技术有限公司,超低温冰箱购自日本Panasonic电器公司,PCR仪购自美国Bio Rad公司,核酸电泳槽为北京六一生物科技有限公司产品,凝胶成像仪为上海培清科技有限公司产品,超微量核酸浓度测定仪Nanodrop 2000为美国Thermo Fisher公司产品。
1.3 测序样品准备、small RNA文库构建及深度测序 参照陈大福等[26]的方法对球囊菌进行活化。将实验室保存的球囊菌菌株接种至马铃薯葡萄糖琼脂(PDA)培养基,置于37 ℃生化箱恒温培养10 d,待培养皿中黑色孢子囊和白色菌丝较多时,在超净台中将孢子囊和菌丝刮至无菌EP管,随后立即将EP管放入液氮速冻,再转移至超低温冰箱保存备用。为最大限度地预测和鉴定球囊菌的miRNAs,本研究选取球囊菌菌丝和孢子的混合样品作为测序对象。上述测序样品设3个生物学重复(Aa-1、Aa-2、Aa-3)。
委托北京百迈克生物科技有限公司对上述3个样品进行单端测序。测序平台为Illumina Hiseq Xten。本研究测得的转录组数据已上传美国国家生物技术信息中心(NCBI)数据库,BioProject号:PRJNA395108。
1.4 生物信息学分析
1.4.1 测序数据质控与评估: 使用Qubit 2.0对文库的浓度进行检测,将文库浓度稀释至1 ng/μL,使用Agilent 2100 bioanalyzer对insert size进行检测,使用qPCR方法对文库的有效浓度进行准确定量,以保证文库质量。测序得到的原始序列含有接头序列或低质量序列,为了保证信息分析的准确性,按如下标准对原始数据进行质量控制:(1)对于每个样本,将碱基质量值小于20的和碱基数量超过50%的reads序列去掉;(2)去除未知碱基N含量大于等于10%的reads;(3)去除没有3′接头序列的reads;(4)剪切掉3′接头序列,去除短于18或长于30个核苷酸的序列,从而得到高质量序列(clean reads)。
1.4.2 sRNA分类注释及参考基因组比对: 利用Bowtie软件[27],将clean reads分别与Silva、GtRNAdb、Rfam及Repbase数据库进行序列比对,过滤核糖体RNA (rRNA)、转运RNA (tRNA)、核内小RNA (snRNA)、核仁小RNA (snoRNA)等ncRNA以及重复序列,获得包含miRNA的unannotated reads。利用Bowtie软件将unannotated reads与参考基因组(https://www.ncbi.nlm.nih.gov/genome/?term=ascosphaera%20apis)进行序列比对,获取在参考基因组上的位置信息,即为mapped reads。
1.4.3 miRNA预测与分析: 利用miRDeep2软件[28]将比对到参考基因组上的reads与miRBase数据库中的已知miRNA前体序列进行比对,来鉴定已知miRNA的表达。同时,通过reads比对到基因组上的位置信息得到可能的前体序列,基于reads在前体序列上的分布信息及前体结构能量信息,采用贝叶斯模型经打分最终实现新miRNA的鉴定。对各样本中miRNA进行表达量的统计,并用TPM算法[29]对表达量进行归一化处理。
1.4.4 miRNA靶基因预测、分析及调控网络构建: 根据已知miRNA和新预测的miRNA (novel miRNA)与对应物种的基因序列信息,利用TargetFinder软件[30]进行靶基因预测。利用BLAST软件将预测靶基因序列与Nr、Swiss-Prot、KOG、GO、KEGG和Pfam数据库比对,获得靶基因的注释信息。利用Cytoskype软件[31]构建miRNA- mRNA调控网络。
1.4.5 Novel miRNA的茎环(Stem-loop)PCR验证: 利用RNA抽提试剂盒(TaKaRa,日本)提取球囊菌的总RNA。参照Chen等[32]的方法,利用DNAMAN软件(Lynnon Biosoft公司,美国)设计novel miRNA的Stem-loop引物、上游引物和下游引物,设计好的引物委托生工生物工程(上海)股份有限公司合成。利用Stem-loop引物,按照cDNA第一链合成试剂盒(TaKaRa,日本)说明书进行miRNA的反转录,反转录得到的cDNA作为模板进行常规PCR。PCR体系(20 μL)包括:cDNA模板1 μL,上下游引物各1 μL,PCR mix 10 μL,无菌水7 μL。PCR程序如下:95 ℃ 5 min;95 ℃ 50 s,49 ℃ 50 s,72 ℃ 50 s,35个循环;72 ℃ 10 min。PCR产物经2%的琼脂糖凝胶电泳检测。
2 结果和分析 2.1 sRNA-seq数据的质控与评估 实验室条件下活化的球囊菌如图 1所示。球囊菌菌丝与孢子样品的sRNA-seq共产生70612299条raw reads,过滤低质量、含N、长度小于18 nt和大于30 nt的reads后,得到48268696条clean reads,平均Q30达到98.75% (表 1)。上述结果说明本研究得到的高通量测序数据质量良好,可用于下一步分析。将各样品的clean reads比对球囊菌的参考基因组,平均比对上的reads数约为2732116条,平均比对率约为52.66% (表 2)。
 |
| 图 1 实验室条件下的球囊菌纯培养 Figure 1 Ascosphaera apis pure culture under laboratory condition. |
| 图选项 |
表 1. sRNA-seq数据统计 Table 1. Overview of sRNA-seq datasets
| Samples | Raw reads | Low quality | Containing ‘N’ reads | Length < 18 | Length > 30 | Clean reads | Q30/% |
| Aa-1 | 28107459 | 0 | 0 | 6826867 | 1254478 | 20026114 | 98.71 |
| Aa-2 | 18958888 | 0 | 0 | 7620276 | 844015 | 10494597 | 98.84 |
| Aa-3 | 23545952 | 0 | 0 | 3464066 | 2333901 | 17747985 | 98.69 |
表选项
表 2. sRNA-seq数据比对参考基因组信息统计 Table 2. Mapping of sRNA-seq data to the reference genome of A. apis
| Samples | Total reads | Mapped reads | Mapping ratio/% |
| Aa-1 | 12372063 | 5451506 | 44.06 |
| Aa-2 | 1872934 | 1071438 | 57.21 |
| Aa-3 | 2951258 | 1673403 | 56.70 |
表选项
2.2 球囊菌miRNA的预测与分析 利用miRDeep2软件,通过reads比对到参考基因组上的位置和二级结构预测,共预测出118个球囊菌的miRNAs。这些miRNAs长度分布介于18–25 nt之间,其中,分布在20、22和23 nt长度的miRNA数最多(图 2-A)。miRNA的碱基偏好性分析结果显示,不同长度的miRNA的首位碱基偏好性差异明显,如长度为18 nt、19 nt和23 nt的miRNAs首位碱基多为C (45.00%–66.67%),长度为20 nt和22 nt的miRNAs首位碱基多为A和G (57.89%–63.16%),长度为24 nt的miRNAs首位碱基多为A和U (62.50%) (图 2-B)。随机挑选12个miRNAs进行stem-loop PCR验证,电泳结果显示共有10个miRNAs (conservative_AZGZ01000001.1_ 1600, conservative_AZGZ01000001.1_ 170, conservative_ AZGZ01000002.1_13849, conservative_AZGZ01000004.1_33180, conservative_AZGZ01000005.1_36837, conservative_AZGZ01000006.1_39913, conservative_ AZGZ01000006.1_ 40023, conservative_AZGZ01000006.1_ 40522, conservative_ AZGZ01000007.1_42036, conservative_ AZGZ01000007.1_ 42616)能够扩增出符合预期的目的片段(图 3),说明本研究预测出的球囊菌miRNA中有相当比例可能真实存在。本研究使用的引物序列详见表 3。
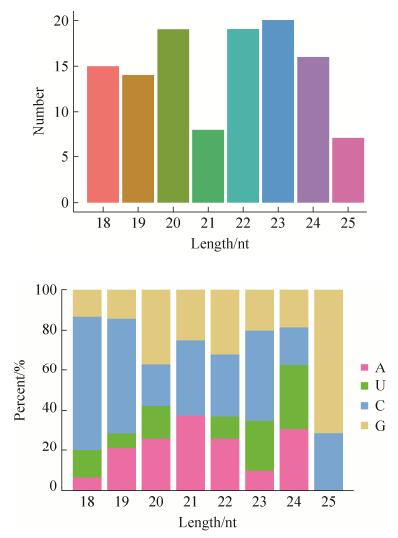 |
| 图 2 球囊菌miRNAs的特征分析 Figure 2 Analysis of characteristics of A. apis miRNAs. A: Length distribution of A. apis miRNA; B: First nucleotide bias of A. apis miRNA. |
| 图选项 |
 |
| 图 3 球囊菌novel miRNAs的stem-loop PCR鉴定 Figure 3 Identification of novel miRNAs of A. apis via stem-loop PCR. M: DNA marker. |
| 图选项 |
表 3. 本研究使用的引物 Table 3. Primers used in this study
| Primer name | Primer sequence |
| Loop-1 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCATTTTCG |
| Loop-2 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGCCTCAAT |
| Loop-4 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCCACTTCA |
| Loop-5 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGGGGAAGA |
| Loop-6 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAAGCACAC |
| Loop-8 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCTCAAAGA |
| Loop-9 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCAACTGAG |
| Loop-10 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGTGGTTGC |
| Loop-11 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCTTTTCTT |
| Loop-12 | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGAGCCCAC |
| Aa-1F | ATGGATTTGGAGATCC |
| Aa-2F | CACTTGGTGTCCGCGTC |
| Aa-4F | GAAGAAGGTCTGTTGTT |
| Aa-5F | TCTGCATCAGTGAATC |
| Aa-6F | GAGGATGTCGCTGT |
| Aa-8F | GCGGCTCTGGGAGGATA |
| Aa-9F | TTGCCGTCGGAGAGAC |
| Aa-10F | GTTGGTATCGTC |
| Aa-11F | AGAACGTTGAGGCTG |
| Aa-12F | CGGGTGGGTG |
| Aa-R | CTCAACTGGTGTCGTGGA |
表选项
2.3 球囊菌miRNA的靶基因的预测及功能注释 利用TargetFinder软件对预测出的118个球囊菌miRNAs的靶基因进行预测,共预测出6529个靶基因。BLAST比对结果显示,共有5725个靶基因获得注释信息,能够注释到Nr数据库、Swissprot数据库、KOG数据库、GO数据库和KEGG数据库的靶基因数量分别为5720、4191、3719、3202和2716个。
KOG数据库注释结果显示,上述靶基因注释到RNA加工与修饰和信号转导机制等26个直系同源蛋白家族(图 4),其中,注释到一般性功能预测的靶基因最多(551 genes),其次为翻译、核糖体结构及生物合成(252 genes),氨基酸转运及新陈代谢(180 genes),翻译(150 genes),碳水化合物转运及新陈代谢(143 genes),复制、重组及修复(142 genes),说明球囊菌的miRNAs参与糖类和蛋白质等物质代谢过程、生物信息传递过程的调控。GO数据库注释结果显示,上述靶基因涉及42个GO条目,注释基因数最多的是代谢进程(2258 genes)、氧化磷酸化(1492 genes)、细胞进程(1846 genes)、结合(1513 genes)、单一组织进程(1408 genes)、细胞组件(1246 genes)、细胞(1243 genes)、细胞器(849 genes)、细胞膜(582 genes)、大分子复合物(537 genes),说明球囊菌的miRNAs在细胞组分、分子功能和生物学进程等方面发挥广泛的调控作用(图 5)。KEGG数据库注释结果显示,上述靶基因注释到99个pathways上,注释基因数最多的是氨基酸的生物合成(103 genes)、核糖体(89 genes)、剪接体(74 genes)、蛋白质的内质网加工(63 genes)、RNA转运(61 genes)、减数分裂(49 genes)、RNA降解(48 genes)、氧化磷酸化(45 genes)、过氧化物酶体(43 genes)和泛素介导的蛋白水解(42 genes)等(图 6-A),说明球囊菌的miRNAs在调控物质和能量代谢等方面发挥重要功能。进一步分析发现,有24个球囊菌miRNAs的靶基因富集在MAPK信号通路(图 6-B),说明球囊菌的miRNAs对该通路具有调控作用。
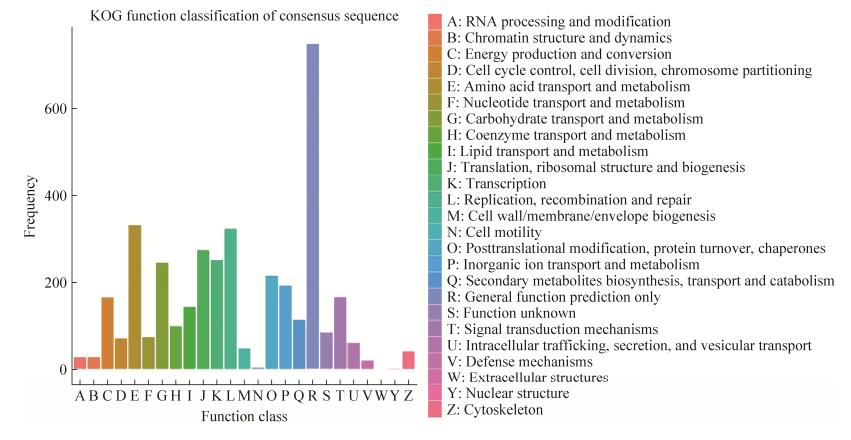 |
| 图 4 球囊菌miRNAs的靶基因的KOG数据库注释 Figure 4 KOG database annotation of target genes of A. apis miRNAs. |
| 图选项 |
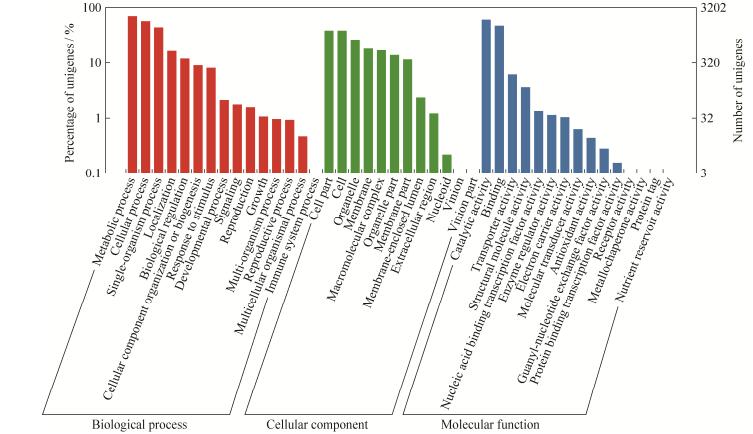 |
| 图 5 球囊菌miRNAs的靶基因的GO数据库注释 Figure 5 GO database annotation of target genes of A. apis miRNAs. |
| 图选项 |
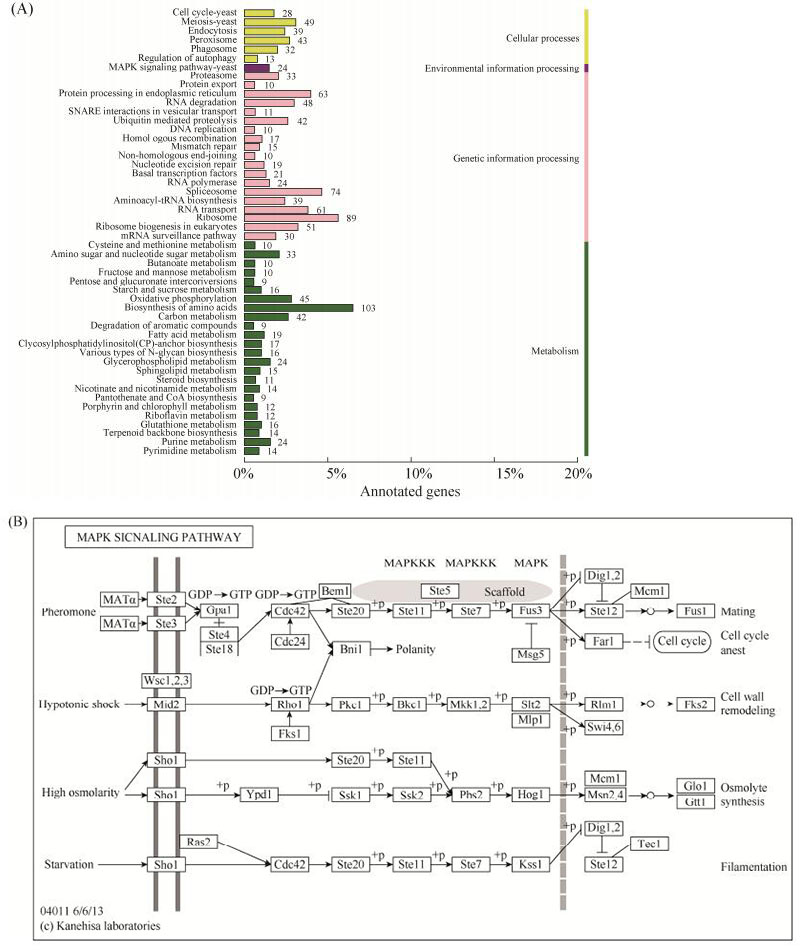 |
| 图 6 球囊菌miRNAs的靶基因的KEGG数据库注释 Figure 6 KEGG database annotation of target genes of A. apis miRNAs. A: Pathway annotation; B: General map of MAPK signaling pathway. |
| 图选项 |
进一步利用靶向关系构建球囊菌的miRNAs- mRNAs调控网络,结果显示仅conservative_ AZGZ01000009.1_44970结合1个靶基因(gene794),其余117个miRNAs均能结合3个及以上的靶基因。利用Cytoscape软件对上述调控网络进行可视化,结果显示绝大多数miRNAs处于调控网络的内部且与多个靶基因靶向结合,球囊菌的miRNAs与mRNAs之间存在复杂的调控网络(图 7)。
 |
| 图 7 球囊菌的miRNAs-mRNAs调控网络 Figure 7 miRNAs-mRNAs regulation networks in A. apis. Red ovals indicate A. apis mRNAs, and green rhombuses indicate A. apis miRNAs. |
| 图选项 |
3 讨论 目前,miRNA已被证明广泛存在于多种真菌[20, 22-24, 33-36]。Huang等利用RNA-seq技术对东方蜜蜂微孢子虫(Nosema ceranae)感染的西方蜜蜂中肠进行测序,结合real-time PCR技术鉴定出6个N. ceranae的miRNAs[36]。本研究利用sRNA-seq技术对实验室纯培养的球囊菌菌丝和孢子进行测序,基于高质量的测序数据预测出118个miRNAs,RT-PCR验证结果显示随机选取的12个miRNAs中有10个成功扩增出目的片段,成功扩增率达到83.33%,初步表明预测出的多数miRNAs真实存在。本研究预测出的miRNAs丰富了球囊菌的miRNA信息,可为深入研究球囊菌miRNAs在孢子萌发、菌丝生长、侵染和增殖过程中的功能提供重要的参考信息。
本研究中,球囊菌miRNAs的靶基因注释到多达2433个GO条目,涉及细胞组分(2364 genes)、分子功能(5114 genes)和生物学进程(4675 genes)的诸多方面。其中,有43个靶基因注释到包括菌丝生长(21 genes)在内的12个生长相关的GO条目;有27个靶基因注释到包括分生孢子发育(3 genes)在内的9个发育相关的GO条目;有8个靶基因注释到包括有性生殖(1 gene)在内的4个增殖相关GO条目;有157个基因注释到包括胁迫应答(13 genes)在内的49个应激反应相关GO条目;有348个基因注释到包括翻译起始调控(15 genes)在内的156个生物学调控相关GO条目。上述结果表明miRNAs广泛参与球囊菌的生长、发育、增殖和应激等生命活动的调控。KEGG数据库注释结果显示,有54个靶基因参与球囊菌的11条氨基酸代谢通路,包括酪氨酸代谢(4 genes)、色氨酸代谢(6 genes)和组氨酸代谢(1 gene)等;有105个靶基因参与球囊菌的13条碳水化合物代谢通路,包括淀粉和蔗糖代谢(16 genes)、肌醇磷酸盐代谢(3 genes)和三羧酸循环(2 genes)等;有66个靶基因参与球囊菌的7条脂代谢通路,包括甘油磷脂代谢(24 genes)、类固醇的生物合成(11 genes)和花生四烯酸代谢(2 genes)等;有38个靶基因参与球囊菌的2条核苷酸代谢通路,包括嘌呤代谢(24 genes)和嘧啶代谢(14 genes)。上述结果表明相应的miRNAs在球囊菌的物质代谢方面具有重要的调控功能。此外,还发现57个靶基因参与球囊菌的3条能量代谢通路,分别为氧化磷酸化(45 genes)、硫代谢(7 genes)和氮代谢(5 genes),表明相应的miRNAs参与球囊菌的能量代谢调控。本研究中,分别有10、119和270个靶基因富集在DNA复制、RNA转录及蛋白质翻译,表明相应的miRNAs在球囊菌的生物信息的传递过程具有重要的调控作用。
昆虫的消化道上皮围食膜和体壁的角质层一般由几丁质和蛋白质组成,对于昆虫保护体内器官和抵御病原入侵至关重要[37]。昆虫病原真菌侵染宿主主要依靠酶的降解和机械压力的联合作用[38]。郑志阳等结合SDS-PAGE技术和酶的活性染色方法对健康和白垩病患病蜜蜂幼虫血淋巴进行检测,发现后者的血淋巴中含有多种蛋白酶和脂酶活性[39]。本研究发现,球囊菌miRNAs的靶基因中含有多达24种蛋白酶编码基因,包括ATP依赖的蛋白酶编码基因(gene5852)、半胱氨酸蛋白酶编码基因(gene2062)和自体吞噬的丝氨酸蛋白酶编码基因(gene1853)等;含有18种脂酶编码基因,例如磷脂酶B编码基因(gene1981)、分泌性脂酶编码基因(gene908)和胞外脂酶编码基因(gene4284)等。几丁质酶是昆虫病原真菌的重要毒力因子[40]。本研究中,球囊菌miRNAs的靶基因包含4个几丁质酶编码基因,分别为内切几丁质酶1编码基因(gene1223)、类型几丁质酶编码基因(gene2593)及几丁质酶3编码基因(gene2594,gene5143);推测球囊菌的miRNAs可通过调控蛋白酶和脂酶基因的表达水平影响蛋白酶和脂酶的分泌,从而促进其在宿主体内的增殖过程。
MAPK信号通路在真菌致病性及响应环境胁迫方面作用关键[41]。前期研究发现,对于胁迫意蜂幼虫的球囊菌,有48个差异表达基因(DEGs)富集在MAPK信号通路上且都呈现出上调趋势[15],说明该通路在球囊菌胁迫意蜂幼虫的过程中被激活而表现活跃;但对于胁迫中蜂幼虫的球囊菌,有11个DEGs富集在MAPK信号通路且都呈现出下调趋势[14],说明该通路在球囊菌胁迫中蜂幼虫的过程中受到抑制;基于前期研究结果,我们推测MAPK信号通路在意蜂幼虫、中蜂幼虫的球囊菌抗性差异方面扮演重要角色,不同抗性蜜蜂幼虫可能通过与球囊菌的互作调控其MAPK信号通路。本研究发现球囊菌miRNAs的靶基因中有2个(gene3094,gene3824)富集在MAPK信号通路,其中MAPK激酶激酶编码基因(gene3094)受到多达12个miRNAs的靶向调控,包括conservative_AZGZ01000009.1_44870、conservative_ AZGZ01000016.1_10151和conservative_ AZGZ01000017.1_11003等;MAPK激酶激酶3编码基因(gene3824)受到3个miRNAs的靶向调控,分别为conservative_AZGZ01000046.1_35555、unconservative_AZGZ01000004.1_33206和conservative_AZGZ01000007.1_42616;上述结果表明球囊菌的多个miRNAs参与MAPK信号通路的调控,这些miRNAs在球囊菌的侵染和增殖过程、球囊菌与蜜蜂幼虫互作过程中的功能有待于进一步研究。本研究仅对球囊菌的菌丝和孢子进行了测序,研究结果可揭示miRNAs与球囊菌的功能或代谢通路的潜在关联性,若要在组学水平深入解析球囊菌miRNAs的表达谱和变化规律,则需要对球囊菌胁迫的不同时间点的蜜蜂幼虫肠道进行测序,测序获得的混合数据经过滤后与本研究获得的miRNA数据进行比较分析,进而对差异表达miRNAs进行靶基因预测及分析,这是下一步的工作重点。
一个miRNA可同时靶向调控多个靶基因,一个靶基因也能同时被多个miRNAs靶向调控[18]。本研究中,Cytoscape软件分析结果显示绝大多数的球囊菌miRNAs处于调控网络的内部(图 7)且同时与多个mRNAs存在靶向结合关系,球囊菌的miRNAs-mRNAs间存在复杂的调控关系,推测miRNAs作为球囊菌的关键调控因子,在其生命活动中扮演重要角色。综而述之,本研究利用sRNA-seq技术对球囊菌的菌丝和孢子进行深度测序,对球囊菌的miRNAs进行了预测、分析、鉴定以及miRNAs-mRNAs的调控网络构建。研究结果丰富了对球囊菌miRNAs的认识,为其基础生物学信息提供了有益补充,也为阐明球囊菌致病的分子机理打下了一定基础。
References
| [1] | Aronstein KA, Murray KD. Chalkbrood disease in honey bees. Journal of Invertebrate Pathology, 2010, 103: S20-S29. DOI:10.1016/j.jip.2009.06.018 |
| [2] | Lumbsch HT, Huhndorf SM. Outline of Ascomycota. Myconet, 2007, 13: 1-58. |
| [3] | Chorbiński P. Enzymatic activity of strains of Ascosphaera apis. Medycyna Weterynaryjna, 2003, 59(11): 1019-1022. |
| [4] | Winston ML. The biology of the honeybee. Cambridge: Harvard University Press, 1991. |
| [5] | Flores JM, Spivak M, Gutiérrez I. Spores of Ascosphaera apis contained in wax foundation can infect honeybee brood. Veterinary Microbiology, 2005, 108(1/2): 141-144. |
| [6] | Theantana T, Chantawannakul P. Protease and β-N-acetylglucosaminidase of honey bee chalkbrood pathogen Ascosphaera apis. Journal of Apicultural Research, 2008, 47(1): 68-76. DOI:10.1080/00218839.2008.11101426 |
| [7] | Evans JD, Spivak M. Socialized medicine:individual and communal disease barriers in honey bees. Journal of Invertebrate Pathology, 2010, 103: S62-S72. DOI:10.1016/j.jip.2009.06.019 |
| [8] | Hornitzky M. Literature review of chalkbrood. A report for the rural industries research and development corporation. Publication No. 01/150, Kingston: ACT, 2001. |
| [9] | Aronstein KA, Murray KD, de León JH, Qin X, Weinstock GM. High mobility group (HMG-box) genes in the honeybee fungal pathogen Ascosphaera apis. Mycologia, 2007, 99(4): 553-561. DOI:10.1080/15572536.2007.11832549 |
| [10] | Cornman RS, Bennett AK, Murray KD, Evans JD, Elsik CG, Aronstein K. Transcriptome analysis of the honey bee fungal pathogen, Ascosphaera apis:implications for host pathogenesis. BMC Genomics, 2012, 13(1): 285-298. DOI:10.1186/1471-2164-13-285 |
| [11] | Qin X, Evans JD, Aronstein KA, Murray KD, Weinstock GM. Genome sequences of the honey bee pathogens Paenibacillus larvae and Ascosphaera apis. Insect Molecular Biology, 2006, 15(5): 715-718. DOI:10.1111/imb.2006.15.issue-5 |
| [12] | Zhang ZN, Xiong CL, Xu XJ, Huang ZJ, Zheng YZ, Luo Q, Liu M, Li WD, Tong XY, Zhang Q, Liang Q, Guo R, Chen DF. De novo assembly of a reference transcriptome and development of SSR markers for Ascosphaera apis. Acta Entomologica Sinica, 2017, 60(1): 34-44. (in Chinese) 张曌楠, 熊翠玲, 徐细建, 黄枳腱, 郑燕珍, 骆群, 刘敏, 李汶东, 童新宇, 张琦, 梁勤, 郭睿, 陈大福. 蜜蜂球囊菌的参考转录组de novo组装及SSR分子标记开发. 昆虫学报, 2017, 60(1): 34-44. |
| [13] | Li WD, Xiong CL, Wang HQ, Hou ZX, Tong XY, Zhang L, Fu ZM, Zheng YZ, Chen DF, Guo R. Large scale development of SSR molecular markers of Ascosphaera apis based on RNA-seq data. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2017, 46(4): 434-438. (in Chinese) 李汶东, 熊翠玲, 王鸿权, 侯志贤, 童新宇, 张璐, 付中民, 郑燕珍, 陈大福, 郭睿. 基于RNA-seq数据大规模挖掘蜜蜂球囊菌的SSR分子标记. 福建农林大学学报(自然科学版), 2017, 46(4): 434-438. |
| [14] | Guo R, Chen DF, Huang ZJ, Liang Q, Xiong CL, Xu XJ, Zheng YZ, Zhang ZN, Xie YN, Tong XY, Hou ZX, Jiang LL, Dao C. Transcriptome analysis of Ascosphaera apis stressing larval gut of Apis cerana cerana. Acta Microbiologica Sinica, 2017, 57(12): 1865-1878. (in Chinese) 郭睿, 陈大福, 黄枳腱, 梁勤, 熊翠玲, 徐细建, 郑燕珍, 张曌楠, 解彦玲, 童新宇, 候志贤, 江亮亮, 刀晨. 球囊菌胁迫中华蜜蜂幼虫肠道过程中病原的转录组学研究. 微生物学报, 2017, 57(12): 1865-1878. |
| [15] | Chen DF, Guo R, Xiong CL, Liang Q, Zheng YZ, Xu XJ, Huang ZJ, Zhang ZN, Zhang L, Li WD, Tong XY, Xi WJ. Transcriptomic analysis of Ascosphaera apis stressing larval gut of Apis mellifera ligustica (Hyemenoptera:Apidae). Acta Entomologica Sinica, 2017, 60(4): 401-411. (in Chinese) 陈大福, 郭睿, 熊翠玲, 梁勤, 郑燕珍, 徐细建, 黄枳腱, 张曌楠, 张璐, 李汶东, 童新宇, 席伟军. 胁迫意大利蜜蜂幼虫肠道的球囊菌的转录组分析. 昆虫学报, 2017, 60(4): 401-411. |
| [16] | St Laurent G, Wahlestedt C, Kapranov P. The landscape of long non-coding RNA classification. Trends in Genetics, 2015, 31(5): 239-251. DOI:10.1016/j.tig.2015.03.007 |
| [17] | Lucas KJ, Zhao B, Liu SP, Raikhel AS. Regulation of physiological processes by microRNAs in insects. Current Opinion in Insect Science, 2015, 11: 1-7. DOI:10.1016/j.cois.2015.06.004 |
| [18] | Bartel DP. MicroRNAs:Target recognition and regulatory functions. Cell, 2009, 136(2): 215-233. DOI:10.1016/j.cell.2009.01.002 |
| [19] | Griffiths-Jones S, Saini HK, van Dongen S, Enright AJ. miRBase:tools for microRNA genomics. Nucleic Acids Research, 2008, 36(Database issue): D154-D158. |
| [20] | Lee HC, Li LD, Gu WF, Xue ZH, Crosthwaite SK, Pertsemlidis A, Lewis ZA, Freitag M, Selker EU, Mello CC, Liu Y. Diverse pathways generate microRNA-like RNAs and Dicer-independent small interfering RNAs in fungi. Molecular Cell, 2010, 38(6): 803-814. DOI:10.1016/j.molcel.2010.04.005 |
| [21] | Zhou JH, Fu YP, Xie JT, Li B, Jiang DH, Li GQ, Cheng JS. Identification of microRNA-like RNAs in a plant pathogenic fungus Sclerotinia sclerotiorum by high-throughput sequencing. Molecular Genetics & Genomics, 2012, 287(4): 275-282. |
| [22] | Zhou Q, Wang ZX, Zhang J, Meng HM, Huang B. Genome-wide identification and profiling of microRNA-like RNAs from Metarhizium anisopliae during development. Fungal Biology, 2012, 116(11): 1156-1162. DOI:10.1016/j.funbio.2012.09.001 |
| [23] | Kang K, Zhong JS, Jiang L, Liu G, Gou CY, Wu Q, Wang Y, Luo J, Gou DM. Identification of microRNA-like RNAs in the filamentous fungus Trichoderma reesei by solexa sequencing. PLoS One, 2013, 8(10): e76288. DOI:10.1371/journal.pone.0076288 |
| [24] | Liu T, Hu J, Zuo YH, Jin YZ, Hou JM. Identification of microRNA-like RNAs from Curvularia lunata associated with maize leaf spot by bioinformation analysis and deep sequencing. Molecular Genetics & Genomics, 2016, 291(2): 587-596. |
| [25] | Lin RM, He LY, He JY, Qin PG, Wang YR, Deng QM, Yang XT, Li SC, Wang SQ, Wang WM, Liu HN, Li P, Zheng AP. Comprehensive analysis of microRNA-Seq and target mRNAs of rice sheath blight pathogen provides new insights into pathogenic regulatory mechanisms. DNA Research, 2016, 23(5): 415-425. DOI:10.1093/dnares/dsw024 |
| [26] | Chen DF, Guo R, Xiong CL, Liang Q, Zheng YZ, Xu XJ, Zhang ZN, Huang ZJ, Zhang L, Wang HQ, Xie YN, Tong XY. Transcriptome of Apis cerana cerana larval gut under the stress of Ascosphaera apis. Scientia Agricultura Sinica, 2017, 50(13): 2614-2623. (in Chinese) 陈大福, 郭睿, 熊翠玲, 梁勤, 郑燕珍, 徐细建, 张曌楠, 黄枳腱, 张璐, 王鸿权, 解彦玲, 童新宇. 中华蜜蜂幼虫肠道响应球囊菌早期胁迫的转录组学. 中国农业科学, 2017, 50(13): 2614-2623. DOI:10.3864/j.issn.0578-1752.2017.13.019 |
| [27] | Langmead B, Trapnell C, Pop M, Salzberg SL. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology, 2009, 10: R25. DOI:10.1186/gb-2009-10-3-r25 |
| [28] | Friedl?nder MR, Mackowiak SD, Li N, Chen W, Rajewsky N. miRDeep2 accurately identifies known and hundreds of novel microRNA genes in seven animal clades. Nucleic Acids Research, 2012, 40(1): 37-52. DOI:10.1093/nar/gkr688 |
| [29] | Fahlgren N, Howell MD, Kasschau KD, Chapman EJ, Sullivan CM, Cumbie JS, Givan SA, Law TF, Grant SR, Dangl JL, Carrington JC. High-throughput sequencing of Arabidopsis microRNAs:evidence for frequent birth and death of miRNA genes. PLoS One, 2007, 2(2): e219. DOI:10.1371/journal.pone.0000219 |
| [30] | Allen E, Xie ZX, Gustafson AM, Carrington JC. microRNA-directed phasing during trans-acting siRNA biogenesis in plants. Cell, 2005, 121(2): 207-221. DOI:10.1016/j.cell.2005.04.004 |
| [31] | Smoot ME, Ono K, Ruscheinski J, Wang PL, Ideker T. Cytoscape 2.8:new features for data integration and network visualization. Bioinformatics, 2011, 27(3): 431-432. DOI:10.1093/bioinformatics/btq675 |
| [32] | Chen CF, Ridzon DA, Broomer AJ, Zhou ZH, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research, 2005, 33(20): e179. DOI:10.1093/nar/gni178 |
| [33] | Raman V, Simon SA, Romag A, Demirci F, Mathioni SM, Zhai JX, Meyers BC, Donofrio NM. Physiological stressors and invasive plant infections alter the small RNA transcriptome of the rice blast fungus, Magnaporthe oryzae. BMC Genomics, 2013, 14(1): 326-344. DOI:10.1186/1471-2164-14-326 |
| [34] | Jiang N, Yang YP, Janbon G, Pan J, Zhu XD. Identification and functional demonstration of miRNAs in the fungus Cryptococcus neoformans. PLoS One, 2012, 7(12): e52734. DOI:10.1371/journal.pone.0052734 |
| [35] | Chen Y, Gao QX, Huang MM, Liu Y, Liu ZY, Liu X, Ma ZH. Characterization of RNA silencing components in the plant pathogenic fungus Fusarium graminearum. Scientific Reports, 2015, 5: 12500. DOI:10.1038/srep12500 |
| [36] | Huang Q, Evans JD. Identification of microRNA-like small RNAs from fungal parasite Nosema ceranae. Journal of Invertebrate Pathology, 2016, 133: 107-109. DOI:10.1016/j.jip.2015.12.005 |
| [37] | Guo R, Wang SM, Xue RY, Cao GL, Hu XL, Huang ML, Zhang YQ, Lu YH, Zhu LY, Chen F, Liang Z, Kuang SL, Gong CL. The gene expression profile of resistant and susceptible Bombyx mori strains reveals cypovirus-associated variations in host gene transcript levels. Applied Microbiology and Biotechnology, 2015, 99(12): 5175-5187. DOI:10.1007/s00253-015-6634-x |
| [38] | Du Y, Xiong CL, Shi XL, Zheng YZ, Fu ZM, Xu XJ, Chen DF, Guo R. Transcriptome analysis of differentially expressed genes in Ascosphaera apis stressing the 6-day-old larval gut of Apis mellifera ligustica. Acta Agriculturae Zhejiangensis, 2017, 29(7): 1119-1128. (in Chinese) 杜宇, 熊翠玲, 史秀丽, 郑燕珍, 付中民, 徐细建, 陈大福, 郭睿. 意大利蜜蜂6日龄幼虫肠道内球囊菌的差异表达基因分析. 浙江农业学报, 2017, 29(7): 1119-1128. |
| [39] | Zheng ZY, Li JH, Liang Q, Chen DF. Ascosphaera apis secretes multiple extracellular enzymes to infect honeybee larvae. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2011, 40(3): 280-284. (in Chinese) 郑志阳, 李江红, 梁勤, 陈大福. 蜜蜂球囊菌分泌多种胞外酶侵染蜜蜂幼虫. 福建农林大学学报(自然科学版), 2011, 40(3): 280-284. |
| [40] | Liu PH, Zhou G, Chen DF, Liang Q. Effects of metal ions on the activity of the chitinase from Ascosphaera apis. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2008, 37(1): 96-98. (in Chinese) 刘鹏虎, 周刚, 陈大福, 梁勤. 金属离子对蜜蜂球囊菌几丁质酶活力的影响. 福建农林大学学报(自然科学版), 2008, 37(1): 96-98. |
| [41] | Rodríguez-Pe?a JM, García R, Nombela C, Arroyo J. The high-osmolarity glycerol (HOG) and cell wall integrity (CWI) signalling pathways interplay:a yeast dialogue between MAPK routes. Yeast, 2010, 27(8): 495-502. DOI:10.1002/yea.1792 |
