陈国锋1, 孔令新1, 王辂2, 褚以文2, 由德林1


1.上海交通大学生命科学技术学院, 微生物代谢国家重点实验室, 上海 200240;
2.成都大学四川抗菌素工业研究所, 四川 成都 610052
收稿日期:2017-04-14;修回日期:2017-06-14;网络出版日期:2017-07-18
基金项目:国家自然科学基金(31630002,31170085,31470183)
*通信作者:由德林, Tel:+86-21-62932943;E-mail:dlyou@sjtu.edu.cn
摘要:[目的]研究黄脂菌素产生菌灰黄链霉菌中编码ArsR家族转录调控蛋白(Arsenical resistance regulator)的xanR3基因的功能。[方法]利用大肠杆菌和链霉菌双亲本接合转移的方法,构建xanR3基因缺失突变株及回补突变株。利用cDNA在相邻同方向的基因间隔区进行PCR确定黄脂菌素生物合成基因簇中的转录单元。利用荧光定量RT-PCR方法进行突变株中黄脂菌素生物合成基因簇转录水平的检测。[结果]对得到的xanR3基因缺失突变株及回补突变株进行发酵,发现xanR3基因缺失突变株产黄脂菌素能力下降,回补菌株中黄脂菌素产量相比缺失突变株有一定程度的恢复,但仍未达到野生型水平。经鉴定,黄脂菌素生物合成基因簇中共有18个共转录单元,其中4个共转录单元在?xanR3突变株中转录水平明显下降。[结论]ArsR家族转录调控基因xanR3是黄脂菌素生物合成的正调控基因。
关键词: 黄脂菌素 AsrR家族转录调控蛋白 RT-PCR 生物合成
Function of AsrR family transcriptional regulatory gene xanR3 in the biosynthetic cluster of xantholipin
Guofeng Chen1, Lingxin Kong1, Lu Wang2, Yiwen Chu2, Delin You1


1.State Key Laboratory of Microbial Metabolism, School of Life Sciences & Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China;
2.Sichuan Industrial Institute of Antibiotics, Chengdu University, Chengdu 610052, Sichuan Province, China
Received 14 April 2017; Revised 14 June 2017; Published online 18 July 2017
*Corresponding author: Delin You, Tel:+86-21-62932943;E-mail:dlyou@sjtu.edu.cn
Supported by the National Natural Science Foundation of China (31630002, 31170085, 31470183)
Abstract: [Objective]We revealed the function of the ArsR (Arsenical resistance regulator) family regulator XanR3 in xantholipin biosynthetic gene cluster.[Methods]Genetically, the xanR3 disrupted mutant CGF03 and complementary strain CGF06 were constructed by conjugation between S. flavogriseus SIIA-A02191 and Escherichia coli ET12567/pUZ8002. The operons within xantholipin biosynthesis cluster were grouped by RT-PCR and the transcriptional level was analyzed in the mutant CGF-3 by quantitative real-time RT-PCR.[Results]Inactivation of the xanR3 gene dramatically reduced xantholipin biosynthesis, and its complementation partially restored xantholipin production. Totally, genes in xantholipin biosynthetic gene cluster were assigned into 18 co-transcription units, and the disruption of xanR3 directly down-regulated the transcription of four operons as xanO1-xanS1, xanB1-xanB3, xanT-xanO9 and xanO10-xanC3.[Conclusion]XanR3, a ArsR family transcriptional regulator, is an activator of xantholipin biosynthesis.
Key words: xantholipin AsrR family transcriptional regulator RT-PCR biosynthesis
黄脂菌素(Xantholipin,图 1)是2003年Terui等[1]在筛选热休克蛋白HSP47基因表达抑制剂的过程中发现的。它由灰黄链霉菌(Streptomycesflavogriesus)发酵产生,属于多环呫吨酮(Xanthone)类化合物。多环呫吨酮类化合物(图 1)含有高度氧化且呈角状折叠排列的六环骨架和典型的呫吨酮核心环(图 1所示A–C环)[2]。多环呫吨酮类化合物独特的分子结构赋予其多样性的生物活性,包括抗细菌(主要抗革兰氏阳性菌,如金黄色葡萄球菌)、抗真菌(如抗酵母)、抗寄生虫(如抗鸡球虫)以及抗肿瘤细胞等活性[3]。自1959年俄国科学家从Actinomycealbusvar fugatas的发酵液中首次分离得到albofungin,目前共分离得到约40多种多环呫吨酮类化合物[4-7]。
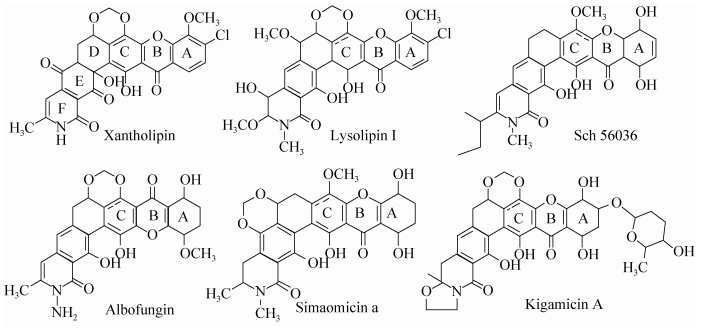 |
| 图 1 微生物来源的多环呫吨酮类代表性化合物[8] Figure 1 Examples of polycyclic xanthone antibiotics. Ring A, B and C were the characteristic xanthone ring nucleus[8]. |
| 图选项 |
黄脂菌素除了可以有效地抑制热休克蛋白HSP47基因的表达(半抑制浓度IC50为2×10–7 mol/L),同时还具有极强的抗真菌、抗革兰氏阳性菌活性。此外,黄脂菌素还可以有效抑制人类成纤维细胞胶原分子的合成(半抑制浓度IC50为2.7×10–8 mol/L),暗示黄脂菌素可以作为治疗纤维化疾病的药物[1]。针对黄脂菌素抗肿瘤细胞活性的研究中还发现其对人类鼻咽腔癌细胞KB细胞株和骨髓性白血病细胞株HL60都具有极高的活性(半抑制浓度分别为IC50 < 2×10–9 mol/L,IC50 < 3×10–7 mol/L),具有作为抗肿瘤药物开发的潜力[2]。然而,黄脂菌素产生菌较低的产素水平(约10 mg/L)是限制其未来开发应用的主要因素。前期研究已克隆鉴定了黄脂菌素生物合成基因簇,研究了黄脂菌素生物合成基因簇中关键结构基因的功能以及黄脂菌素中核心呫吨酮环形成机制,确定了黄脂菌素生物合成途径[8],本研究在此基础上,继续深入研究黄脂菌素生物合成中调控蛋白的具体功能,揭示调节基因对黄脂菌素产量的影响机制。预期对黄脂菌素生物合成调控机制的研究,将深化对黄脂菌素生物合成的理解,为通过基因工程进行定向遗传改良构建黄脂菌素高产菌株提供理论依据。
通过对黄脂菌素生物合成基因簇的生物信息学分析,发现3个可能的转录调控基因(xanR1、xanR2和xanR3)。其中,XanR3蛋白序列中包含典型的螺旋-转角-螺旋(HTH,helix-turm-helix) DNA结合结构域和锌离子的结合位点,属于ArsR家族调节蛋白。尽管随着基因组测序的展开,在许多链霉菌中发现了ArsR同源蛋白,但它们的功能并不清楚。本研究利用PCR-targeting的方法,以阿泊拉霉素抗性基因置换xanR3基因的部分片段构建了?xanR3突变株,利用整合型回补质粒pJTU824载体构建了?xanR3::xanR3回补菌株,通过对基因缺失突变株、回补菌株和野生型菌株SIIA-A02191发酵产量的对比分析,考察了xanR3对黄脂菌素产量的影响,为黄脂菌素生物合成调控机制研究奠定了基础,并为后续提高黄脂菌素产量提供了参考。
1 材料和方法 1.1 实验材料
1.1.1 菌株与质粒: 黄脂菌素产生菌S. flavogriseus SIIA-A02191、大肠杆菌DH10B、ET12567/pUZ8002以及质粒pJTU824、质粒pIJ773、fosmid 22E4均由上海交通大学微生物代谢国家重点实验室保藏。调控基因xanR3缺失突变株CGF-3与回补突变株CGF-6为本研究构建。
1.1.2 主要试剂仪器: PCR反应使用TaKaRa Taq试剂盒,1 kb Plus DNA ladder为Invitrogen公司产品,RNA提取试剂盒为北京赛百盛基因技术有限公司Total RNA Isolation Kit,RNA反转录试剂盒为TaKaRa公司PrimeScript? RT Reagent Kit with gDNA Eraser (Perfect Real Time),荧光实时定量(q-Real time) PCR反应试剂盒为上海翊圣生物科技有限公司HieffTM qPCR SYBR? Green Master Mix (low Rox Plus)。限制性内切酶EcoR Ⅰ和Nde Ⅰ购自Fermentas公司。ISP3 (燕麦)培养基:燕麦5 g,青岛琼脂5 g,ddH2O 250 mL,加入250 μL微量元素母液(FeSO4?7H2O 0.1 g,MnCl2?4H2O 0.1 g,ZnSO4?7H2O 0.1 g,ddH2O 100 mL;加1 mol/L HCl 50 μL)用4 mol/L NaOH调节pH到7.0–7.5,灭菌,用于灰黄链霉菌及其衍生菌株产孢。孢子预萌发培养液:酵母粉1 g,水解酪蛋白氨基酸1 g,定容至100 mL,灭菌。接合转移培养基:黄豆饼粉20 g,自来水1000 mL,灭菌过滤定容至1000 mL,分装补加甘露醇5 g/250 mL,琼脂5 g/250 mL,燕麦5 g/250 mL,加入250 μL微量元素母液,用4 mol/L NaOH调节pH到7.2,灭菌。种子培养基:葡萄糖10 g,可溶性淀粉25 g,酵母提取物2 g,鱼粉5 g,棉籽粉3 g,酪蛋白水解物(Casein) 3 g,ddH2O 1 L,调节pH至7.2,补加碳酸钙2 g,灭菌。发酵培养基:葡萄糖10 g,糊精25 g,燕麦粉20 g,棉子粉10 g,鱼粉5 g,酵母提取物2 g,ddH2O 1 L,调节pH至7.2,碳酸钙3 g,灭菌。高效液相色谱(HPLC)使用Agilent 1200 Series,液质联用[LC-MS(n)]分析使用Agilent 1100 series LC/MSD Trap System,色谱柱均采用Agilent TC-C18反相柱(5.0 μm,250 mm×4.6 mm)。
1.2 xanR3基因敲除突变株CGF-3的构建 参照PCR-targeting的操作方法,碱裂解法提取质粒pIJ773,并用限制性内切酶EcoR Ⅰ和Hind Ⅲ切下包含阿泊拉霉素抗性基因aac(3)IV、oriT以及FRT序列大小约为1.4 kb的片段。然后,以此片段为模板设计用于xanR敲除的一对引物targetR3-F/R (表 1),将PCR扩增后得到的片段,利用大肠杆菌Red重组系统与包含黄脂菌素生物合成基因簇的fosmid 22E4进行重组,获得可用于敲除的质粒pCGF03。最后,用含此质粒的大肠杆菌ET12567/pUZ8002与野生型灰黄链霉菌进行两亲本接合转移,30 ℃、28 h后用阿泊拉霉素和萘啶酮酸进行覆盖,7 d后即可挑取双交换接合转移子。将接合转移子接种至液体种子培养基中培养3 d,提取基因组DNA,并在xanR3基因的两侧设计一对验证引物YZR3-F/R (表 1)对其进行PCR验证。本研究中,经过PCR扩增,双交换突变株产物片段大小为4.1 kb,野生型对照菌株得到产物大小为3.4 kb (图 2)。
表 1. 构建xanR3基因敲除突变株和回补菌株所用PCR引物 Table 1. Primers used for construction and verification of the xanR3 disruption mutant strain and the complementation strain
| Primer | Sequence (5′→3′) |
| targetR3-F | CAGGTACGGCCGCGGGTG GCGGCCGCCGGGCTCGACCGGATTCCGGGGATCCGTCGACC |
| targetR3-R | GCGCAGGGCGGTCAGGTTCTGGGACACGCCCGCCGGGGATGTAGGCTGGAGCTGCTTC |
| YZR3 | ACGCCCTGACGGTGATGCT |
| YZR3 | CCAGTAGTCAAGGGACCGCTG |
| huibuR3-F | AAACATATGCGCGAATCCTCGGGGGGG |
| huibuR3-R | AAAGAATTCTCACCCTGCGAGGAGGGA |
表选项
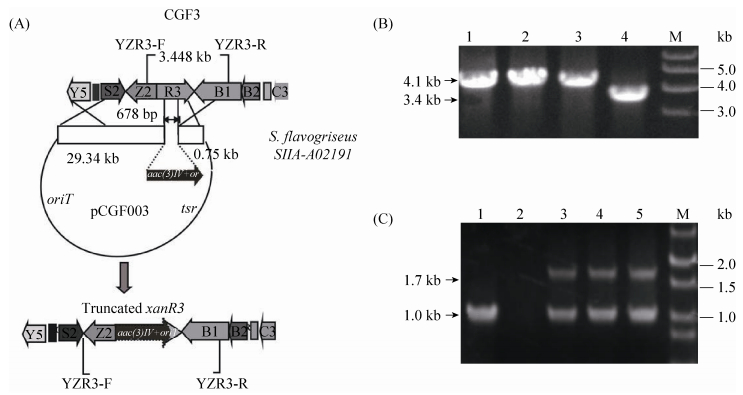 |
| 图 2 xanR3基因敲除突变株的构建及验证(A,B)和回补突变株CGF-6的验证(C) Figure 2 Construction and verification of the xanR3 disruption mutant CGF-3 and the complementation strain CGF-6. A: the construction of the xanR3 disruption mutant CGF-3. The 0.68 kb region in xanR3 was replaced by 1.4 kb aac(3)IV gene. B: PCR verfication of the xanR3 disruption mutant CGF-3. M: 1 kb DNA ladder; lane 1–3: double crossover recombinant; lane 4: wild-type strain. C: PCR verification of the complementary strain CGF-6. M: 1 kb DNA ladder; lane 1: wild-type strain; lane 2: pJTU824 control; lane 3–5: complementary strains CGF-6. |
| 图选项 |
1.3 xanR3基因回补菌株CGF-6的构建 利用目的基因xanR3首末端序列,设计一对PCR引物huibuR3-F/R,以野生型灰黄链霉菌基因组DNA为模板,扩增得到xanR3,经限制性内切酶EcoR Ⅰ和Nde Ⅰ酶切后,连接到整合型载体pJTU824上,获得回补质粒pCGF06。将质粒pCGF06利用前述接合转移的方法整合到?xanR3突变株CGF-3中。在回补菌株CGF-6的基因组中,同时含有被阿泊拉霉素抗性片段部分替换的xanR3基因和整合型质粒pJTU824所携带的完整的xanR3基因,因此经过PCR扩增可同时得到两条大小分别为1.0 kb和1.7 kb片段的接合转移子即为?xanR3回补菌株CGF-6。
1.4 灰黄链霉菌发酵及发酵产物的分离与检测 将野生型菌株SIIA-A02191、突变株CGF-3和回补菌株CGF-6的孢子按0.1%的接种量接种于种子摇瓶中,30 ℃、220 r/min培养3 d,将种子液按1/20的量转接于80 mL发酵培养基的摇瓶中继续培养6 d。所得发酵液利用乙酸乙酯萃取粗提,用1 mL甲醇溶解后,用于HPLC或者LC/MS检测分析。HPLC检测流动相A相为含0.2%甲酸的水溶液,B相为乙腈,流速为0.6 mL/min,检测波长为274 nm。检测程序为:0–5 min,30%等度洗脱;5–35 min,30%–70%梯度洗脱;35–40 min,70%–80%梯度洗脱;40–41 min,80–100%梯度洗脱;41–45 min,100%等度洗脱;45–46 min,100%–30%梯度洗脱;46–60 min,30%等度洗脱(此处的百分比表示的是有机相乙腈在水溶液中所占的体积百分数)。
LC/MS分析条件为:正离子模式,干燥气流10 mL/min,喷雾器压力为30 psi,干燥气体温度350 ℃,分子量扫描范围为200至1000,多级质谱断裂分析轰击电压为1.0–1.5 V,检测程序设置与高效液相色谱(HPLC)检测相同。
1.5 灰黄链霉菌总RNA的提取与反转录PCR 将野生型菌株SIIA-A02191、突变株CGF-3和突变回补菌株CGF-6转入发酵培养基中培养48 h后,分别取1 mL菌液,按照Total RNA Isolation Kit操作说明提取RNA。Nanodrop测定浓度及OD260/OD280,并配合琼脂糖凝胶电泳确定RNA的质量与含量。加入2 μL 5×gDNA Erase Buffer,1 μL gDNA Eraser,1 μg RNA,加RNaseFree H2O至10 μL,42 ℃、2 min对基因组DNA进行消化,利用1对反向基因间隔区两侧设计的引物进行PCR验证无基因组残留,引物序列为xanWR2-F/R (表 2)。确认无基因组DNA残留后,取上步反应液按照反转录试剂盒操作说明配制成20 μL体系,将所提取RNA反转录为cDNA。
表 2. 确定黄脂菌素的生物合成基因转录本所用的PCR引物 Table 2. Primers used for the identification of the transcripts in xantholipin biosynthetic gene cluster
| Primer | Gene tested | Forward (5'→3') | Reverse (5′→3′) |
| xanWR2 | xanW-xanR2 | ATCTGGATTGTGCAGGGTTGA | GTGACACTGCCCGTGGTGA |
| xanP-ben | xanP-xanG | TTCAGCAGCGACTTCCACCAC | TCGGCACCAGCGCATACAG |
| xanA-ben | xanA-xanR1 | GAGCGGCGCAACGTGGAGAT | CCTGGAAGAACGAGCAGTGGG |
| xanR1-ben | xanR1-xanO2 | ACGCCCACTGCTCGTTCTTC | TCGGCAGGGTCACTTCCAT |
| xanO2-ben | xanO2-xank | ACGCCATCGACGACATCCC | CACGCGGTACACGCACAGAC |
| xanU-ben | xanU-xanO3 | ATCGGCAAGGGCATGTGG | CGCTTCCTGTGCGTTCGT |
| xanO3-ben | xanO3-xanL | TCACTCCAAGACCCGTTACACC | CATCCAGATTTCCACGCACA |
| xanL-ben | xanL-xanH | ACCCTCGCTCCTGCCCTCTT | AACCACGCATGTATTTCTCCAA |
| xanM1-ben | xanM1-xanZ1 | GAGCCACCGAAGGCAAGACC | GAACGAGTAGTACGCCCGCAGA |
| xanN-ben | xanN-xanW | CTACGGAGCACAGTGAGACGC | ACGGCACCACCGCACAGAT |
| xanM2-ben | xanM2-xanO4 | AGGAAGTGGTCGGTGCCC | CCTTCGTCTCCTGGCGGTC |
| xanM3-ben | xanM3-xanO5 | CAGAACCCGCACATCCTCAA | CGACCAGCGAACGTATCCC |
| xanO5-ben | xanO5-xanS2 | TGAACTGGCTGCTGCTGTGG | GGCGATGCCGATGGTGAA |
| xanB2-ben | xanB2-xanB3 | CCCACTCGACCTCGATGCTG | CCAACGATCCGGGAACACG |
| xanB3-ben | xanB3-xanO6 | GCTTCTGGCGGATGGTCTGTT | ACGAGGTTGAGGCGGCCTACTG |
| xanZ3-ben | xanZ3-xanO8 | ACGGGATGTGGCTGATGGC | CGCACTGCTGCTCTGACTGAC |
| xanO8-ben | xanO8-xanC1 | AGCGAGATGGTGACGGTGGC | GATCAAGCAGAAGGTCGAGGC |
| xanC1-ben | xanC1-xanE | GCGTCGATCACGATGCTGTT | CCGCACCCAAGACGCTGAC |
| xanT-ben | xanT-xanO9 | ACACCGTCGAGGGCTTCATC | ATCGCCACATCGGCTTCC |
| xanO10-ben | xanO10-xanZ4 | GGCATCGTCGGGTCCCAGTT | CCATCGTCAACATCGGTTCGTC |
表选项
1.6 黄脂菌素生物合成基因共转录单元的确认 将同方向有交叉重叠序列的2个基因视为同一个转录单元,转录方向相反的2个相邻基因视为2个不同的转录单元。对其余同方向且有间隔区序列的2个基因设计引物,进行PCR验证,引物设计原则为正向引物和反向引物分别在间隔区的两侧,PCR扩增的片段应包含间隔区序列。黄脂菌素的生物合成基因簇共54 kb,含有49个基因,为确定其共转录单元本研究设计19对引物,以野生型菌株SIIA-A02191总RNA反转录获得的cDNA为模板进行PCR验证(表 2)。
1.7 黄脂菌素生物合成基因转录水平检测 采用荧光相对定量Real-time PCR法进行检测,比较样品中各转录单元相对于内参基因hrdB的数量变化。具体的是利用公式??Ct=(Ct目标基因–Ct内参基因)样品组–(Ct目标基因–Ct内参基因)参照组,通过比较Ct值(每个反应管内的荧光信号强度达到设定阈值所需循环数),计算出样品中目标基因的相对变化量。对18个目标基因和内参基因hrdB分别设计引物(表 3),Real-time PCR反应采用HieffTM qPCR SYBR? Green Master Mix (low Rox Plus)试剂盒20 μL体系。使用ABI 7500 Fast,循环条件为95 ℃ 2 min;95 ℃ 10 s,60 ℃ 20 s,72 ℃ 20 s,40个循环。溶解曲线:95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s,1个循环。
表 3. Real-time PCR实验所用引物 Table 3. Primers used for quantitative real-time PCR
| Primer | Forward (5′→3′) | Reverse (5′→3′) |
| hrdB-RT | GTTCCTGTACGGGACCAGTC | TGCGGTCGAGAAGTTCGACTA |
| xanO1-RT | GCTGCGGGAGCCGAACCACT | ACCTCGTCGCCCTCGCCATC |
| xanR1-RT | GGCTACGGCTACGCGGTGAA | GGATGCTGACGGCGAAGTGG |
| xanL-RT | AGCACCACGATCTCGGACTG | GGCTGGGACCGGACTACTACA |
| xanH-RT | TTCCACCCAGATCCTCGTCA | TGTGCCTCCACCTTGTCCC |
| xanZ1-RT | ACCGTGCCGGACATCGAC | TGAGGGGCTCGTAACTGTCGTA |
| xanW-RT | CGGAGCGCAGGTCCATCT | GCCTACTACACGAACGTCAAGC |
| xanR2-RT | GTCAAGCGATAACCGGAAGAAT | GCCCACACCGAGGCGTTCGGC |
| xanM2-RT | GTCGTGCCCGAGTGAGGTGT | TGATCCGCAGCCTGAACC |
| xanO4-RT | GTCGCCCTTGCCGATGGTGT | CCTGTTCTGGGACTCGTTCGTG |
| xanM3-RT | ACCCGCACATCCTCAAGCA | GGTCGAAGACGTGACCCTCC |
| xanZ2-RT | GGCAAGCACCTCGTCGCT | CAGAACCAGTACGGCATCGG |
| xanR3-RT | GCTGCTGCTGACCGAACTGG | TCACCCTGCGAGGAGGGATT |
| xanB1-RT | CGAGGAGGAGATCCGCAAGG | GAGGAAGCCACCGACCCA |
| xanO6-RT | TCGCTGACGATGAAGTAGACGC | TCCGCATGGAGATCAACCC |
| xanC1-RT | CTTCGGCCTCGACCTTCTG | TGCGGCTGTTCTGGGAGTA |
| xanE-RT | CAGGCGATGGACTGGTAGGC | GTACGAACTGGCCGTGGTGAC |
| xanT-RT | AGCGCCCGCATCCTGTCCA | CCATCGCACGAGCTTCTCC |
| xanO10-RT | ACATTGCACAGATGCGAGACCG | CGCAACTGGGACCCGACGAT |
表选项
2 结果和分析 2.1 xanR3基因缺失突变株与回补菌株的构建及发酵产物的分析 为了确认xanR3基因的功能,利用大肠杆菌和链霉菌属间双亲本接合转移,对其进行体内缺失及回补实验,构建了xanR3基因缺失突变株CGF-3和缺失突变株的体内回补菌株CGF-6 (图 2)。
将野生型菌株SIIA-A02191和CGF-3、CGF-6进行液体摇瓶发酵。30 ℃发酵6 d后,丙酮和乙酸乙酯对发酵液进行粗提,所得发酵产物利用HPLC分析。从LC-MS检测结果(图 3)中可以看出,xanR3基因敲除后突变株CGF-3仍然有黄脂菌素产生,但产量只有野生型菌株SIIA-A02191的10%左右,暗示xanR3基因在黄脂菌素生物合成过程中起正调控作用。xanR3基因回补后,回补菌株产量较缺失突变株有一定提高,产量恢复到野生型的40%左右,再次证明xanR3的正调控功能。
 |
| 图 3 敲除突变株CGF-3与回补突变株CGF-6发酵产物的LC-MS检测 Figure 3 LC-MS analysis of the xanR3 disruption mutant CGF-3 and complementation strain CGF-6. A: HPLC analysis of the fermentation extracts of CGF-6 (ⅰ), CGF-3 (ⅱ), wild type (ⅲ), and xantholipin standard (ⅳ). B: quantitative analysis of xantholipin produced by mutant strains. The yield of xantholipin in wild type strain is represented by 1, whereas it is represented by the fold to that of WT in mutants CGF-3 and CGF-6. C: validation of xantholipin from the fermentation extracts by mass spectrum analysis, CGF-6 (ⅰ), CGF-3 (ⅱ), wild type (ⅲ), and xantholipin standard (ⅳ). |
| 图选项 |
2.2 黄脂菌素生物合成基因共转录本的确定 为了确认xanR3基因在黄脂菌素生物合成过程中的调控作用,对其生物合成基因的共转录单元进行了确认。同方向有交叉重叠序列的2个基因视为同一转录单元(xanO1-xanS1,xanJ-xanP,xanG-xanA,xanK-xanU,xanH-xanM1,xanB1-xanB2,xanO6-xanZ3,xanV-xanC3),其余同方向有间隔区的2个基因设计引物进行PCR验证,验证其是否在同一转录本上。前期研究发现野生型菌株SIIA-A02191转入发酵培养基中48 h后即可检测到黄脂菌素的合成,因此本研究取48 h发酵液中的菌体提取RNA,将其逆转录成cDNA后作为模板进行PCR,并根据电泳结果判断相邻2个基因的共转录关系(图 4),黄脂菌素生物合成基因共有18个转录单元:xanO1-xanS1、xanJ-xanO3、xanL、xanH-xanM1、xanZ1、xanN-xanW、xanR2、xanM2、xanO4、xanM3-xanS2、xanZ2、xanR3、xanB1-xanB3、xanO6-xanO8、xanC1、xanE-xanC2、xanT-xanO9和xanO10-xanC3。
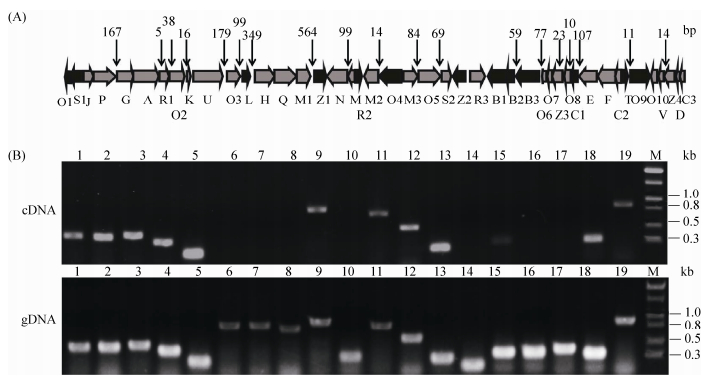 |
| 图 4 黄脂菌素的生物合成基因簇和相邻基因共转录分析 Figure 4 Organization of the gene cluster for xantholipin biosynthesis and verification of the operons in xantholipin biosynthesis gene cluster. A: organization of the gene cluster for xantholipin biosynthesis in which the vertical solid arrows show the position of primers used for quantitative real-time PCR, the gap is the intergenic spacer between adjacent genes. The numbers above the gene cluster represent the lengths (bp) of the intergenic regions. Genes are assigned in two colors: black and gray, the same direction genes which belong to the same mRNA transcription units were marked in the same color. B: PCR confirmation of the amplicons fragments on an ethidium bromide-dyed agarose gel. The amplicons were designed to covering of the adjacent genes, genomic DNA (gDNA) was used as the positive control. lane 1: xanP-xanG 465 bp; lane 2:xanA-xanR1 453 bp; lane 3: xanR1-xanO2 485 bp; lane 4: xanO2-xank 485 bp; lane 5: xanU-xanO3 266 bp; lane 6: xanO3-xanL 833 bp; lane 7: xanL-xanH 832 bp; lane 8: xanM1-xanZ1 762 bp; lane 9: xanN-xanW 888 bp; lane 10: xanM2-xanO4 309 bp; lane 11: xanM3-xanO5 799 bp; lane 12: xanO5-xanS2 553 bp; lane 13: xanB2-xanB3 278 bp; lane 14: xanB3-xanO6 182 bp; lane 15: xanZ3-xanO8 345 bp; lane 16: xanO8-xanC1 349 bp; lane 17: xanC1-xanE 390 bp; lane 18: xanT-xanO9 324 bp; lane 19: xanO10-xanZ4 882 bp; M: 1 kb DNA ladder. |
| 图选项 |
2.3 xanR3基因缺失突变株CGF-3和野生型菌株SIIA-A02191中黄脂菌素的生物合成基因转录水平的检测 确定基因簇上的共转录单元后,在各转录本内部设计引物,一般选取该转录本的第一个基因按Real-time PCR引物设计标准设计,目的片段大小为200–300 bp,共设计Real-time PCR引物18对及一对内参引物hrdB。先将引物以野生型基因组为模板PCR扩增,确认是否有非特异扩增,电泳无杂带,则初步断定所设计引物可用于Real-time PCR反应。根据??Ct计算公式,在突变株CGF-3中目标基因按照系统算法测得的数值大于2.0或者低于0.5,即判断该基因的转录被显著增强或者抑制。荧光定量结果显示(图 5),突变株内不同基因转录受到的影响不同,其中xanO1-xanS1、xanB1-xanB3、xanT-xanO9、xanO10-xanC3转录水平下降明显,为野生型的10%–30%之间,转录水平下降的基因涉及黄脂菌素生物合成中的延伸单元的供应、核心骨架形成以及生物合成后修饰等方面。其中,xanB1-xanB3编码乙酰辅酶A羧化酶,参与丙二酰辅酶A的合成;xanO10-xanC3中的xanD编码Ⅱ型聚酮合酶的亚基,影响聚酮碳链的延伸,xanC3编码环化酶,影响合成前体的形成,xanO10、xanZ4和xanO1均编码氧化还原相关蛋白,影响分子骨架的氧化还原修饰。这些可能受XanR3调控的基因转录水平的降低,与突变株黄脂菌素产量降低相一致,也再次表明xanR3是黄脂菌素生物合成过程中的正调控基因。
 |
| 图 5 黄脂菌素的生物合成基因转录水平检测 Figure 5 Transcriptional analysis of the xantholipin biosynthetic gene cluster in wild type strain and mutants CGF-3 by quantitative realtime RT-PCR. The relative expression value of xanO1, xanO10, xanT and xanB1 were lower than 0.5 indicating less mRNA produced in mutant strains than that in the wild-type strain. Error bars were calculated from three independent experiments each of which was performed in duplicate. |
| 图选项 |
3 讨论 本研究构建了xanR3基因缺失突变株CGF-3,发现突变株CGF-3中黄脂菌素产量下降为野生型的10%左右,体内回补后回补菌株CGF-6的产量上升为野生型的40%左右,进一步通过荧光定量RT-PCR,确定了xanR3作为正调控基因可以调控黄脂菌素生物合成相关基因的转录。生物信息学分析发现XanR3属于ArsR转录调控家族蛋白,该家族蛋白可作为金属离子感受器发挥转录调节作用[9],XanR3蛋白的C末端含有ArsR家族结合DNA的HTH结构域,暗示XanR3可能同样作为转录调控因子发挥调控作用。研究也发现链霉菌中的ArsR蛋白可以调控相关抗生素分子的生物合成,例如在S. coelicolor中发现SCO0608基因可编码AsrR家族同源蛋白,敲除该基因可加速孢子的形成及十二烷基灵菌红素和放线菌素的产生[10]。虽然该家族蛋白广泛存在于链霉菌中,但这类蛋白参与调控抗生素生物合成的机制尚待进一步研究。本研究中,xanR3基因的缺失使得参与黄脂菌素生物合成前体供应、骨架合成以及后修饰作用的基因转录量明显下降,同时该突变株中黄脂菌素产量明显降低,证明XanR3是黄脂菌素生物合成途径的正调控因子。本研究为探究黄脂菌素生物合成过程的调控机制及黄脂菌素高产菌株的构建提供了理论基础。
References
| [1] | Terui Y, Chu YW, Li JY, Ando T, Yamamoto H, Kawamura Y, Tomishima Y, Uchida S, Okazaki T, Munetomo E, Seki T, Yamamoto K, Murakami S, Kawashima A. Xantholipin, a novel inhibitor of HSP47 gene expression produced by Streptomyces sp.. Tetrahedron Letters, 2003, 44(29): 5427-5430. DOI:10.1016/S0040-4039(03)01318-2 |
| [2] | Zhang WK, Wang L, Kong LX, Wang T, Chu YW, Deng ZX, You DL. Unveiling the post-PKS redox tailoring steps in biosynthesis of the type Ⅱ polyketide antitumor antibiotic xantholipin. Chemistry & Biology, 2012, 19(3): 422-432. |
| [3] | Winter DK, Sloman DL, Porco Jr JA. Polycyclic xanthone natural products:structure, biological activity and chemical synthesis. Natural Product Reports, 2013, 30(3): 382-391. DOI:10.1039/c3np20122h |
| [4] | Kobayashi K, Nishino C, Ohya J, Sato S, Mikawa T, Shiobara Y, Kodama M. Actinoplanones A and B, new cytotoxic polycyclic xanthones from Actinoplanes sp.. The Journal of Antibiotics, 1988, 41(4): 502-511. DOI:10.7164/antibiotics.41.502 |
| [5] | Malkina ND, Dudnik YV, Lysenkova LN, Lazhko EI, Galatenko OA, Katrukha GS. 167-A, a new antibiotic produced by a mutant of an inactive wild strain of Amycolata autotrophica. The Journal of Antibiotics, 1994, 47(3): 342-348. DOI:10.7164/antibiotics.47.342 |
| [6] | Solov'Eva NK, Rudaia SM. Characteristics of the organism producing a new antifungal antibiotic albofungin. Antibiotiki, 1958, 4: 5-10. |
| [7] | Nakagawa A, Omura S, Kushida K, Shimizu H, Lukacs G. Structure of cervinomycin, a novel xantone antibiotic active against anaerobe and mycoplasma. The Journal of Antibiotics, 1987, 40(3): 301-308. DOI:10.7164/antibiotics.40.301 |
| [8] | Kong LX, Zhang WK, Chooi YH, Wang L, Cao B, Deng ZX, Chu YW, You DL. A multifunctional monooxygenase XanO4 catalyzes xanthone formation in xantholipin biosynthesis via a cryptic demethoxylation. Cell Chemical Biology, 2016, 23(4): 508-516. DOI:10.1016/j.chembiol.2016.03.013 |
| [9] | Shi WP, Wu JH, Rosen BP. Identification of a putative metal binding site in a new family of metalloregulatory proteins. The Journal of Biological Chemistry, 1994, 269(31): 19826-19829. |
| [10] | Yang YH, Kim JN, Song E, Kim E, Oh MK, Kim BG. Finding new pathway-specific regulators by clustering method using threshold standard deviation based on DNA chip data of Streptomyces coelicolor. Applied Microbiology and Biotechnology, 2008, 80(4): 709-717. DOI:10.1007/s00253-008-1574-3 |
