曲道峰, 沈杨, 张聪聪, 韩剑众


浙江工商大学食品与生物工程学院, 浙江 杭州 310018
收稿日期:2017-01-14;修回日期:2017-06-30;网络出版日期:2017-07-10
基金项目:浙江省科技计划项目(2017C32036);食品科学与工程浙江省重中之重一级学科(2017SICR118)
*通信作者:韩剑众, Tel/Fax:+86-571-28008964;E-mail:hanjz99@zjgsu.edu.cn
摘要:成簇规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR),是存在于多数细菌和古菌中的遗传结构,能够有效防御外源DNA的入侵(质粒、噬菌体等),进而防御外源基因的水平转移。[目的]本研究以沙门氏菌属中常见的鸡伤寒沙门氏菌(Salmonella gallinarum)、鼠伤寒沙门氏菌(Salmonella typhimurium)、猪霍乱沙门氏菌(Salmonella choleraesuis)以及肠炎沙门氏菌(salmonella enteritidis)等30个菌株为研究对象。探索CRISPR位点在不同沙门氏菌种中的结构差异。[方法]通过生物信息学的方法比较间隔序列与插入序列的同源性以及CRISPR位点与质粒数量关系。[结果]30株沙门氏菌中均存在CRISPR结构,包括CRISPR位点61个以及可疑位点12个。重复序列和cas1基因均不能作为这4类细菌的分类依据。[结论]虽然我们发现CRISPR位点数量与间隔区数量和质粒数量之间均不存在统计学关系,但间隔序列整合子、耐药基因等移动遗传原件具有一定的同源性,说明沙门氏菌在进化过程中不断受外源基因的侵袭。
关键词: 沙门氏菌 CRISPR 间隔区 质粒
Genomics comparison between CRISPR loci and plasmid in Salmonella enteric
Daofeng Qu, Yang Shen, Congcong Zhang, Jianzhong Han


College of Food and Biotechnology, Zhejiang Gongshang University, Hangzhou 310018, Zhejiang Province, China
Received 14 January 2017; Revised 30 June 2017; Published online 10 July 2017
*Corresponding author: Jianzhong Han, Tel/Fax:+86-571-28008964;E-mail:hanjz99@zjgsu.edu.cn
Supported by the Zhejiang Science and Technology Plan Project (2017C32036) and by the Food Science and Engineering-the Most Important Discipline of Zhejiang Province (2017SICR118)
Abstract: Clustered regularly interspaced short palindromic repeats (CRISPR) is the genetic structure found in most of bacteria and archaea. The function of CRISPR is defending against the invasion of exogenous DNA (plasmid, phage, etc.), preventing heterologous gene transfer.[Objective]This study is to study the structural differences of CRISPR loci in several Salmonella sp.[Methods]The molecular structure of CRISPR and the relationship between CRISPR and plasmid was analyzed in 30 strains, including Salmonella gallinarum, S. typhimurium, S. choleraesuis and S. enteritidis by bioinformatics.[Results]CRISPR structure existed in the 30 strains of Salmonella, totally 61 conformed CRISPR loci and 12 possible ones. Neither repeat sequence nor cas1 gene could be used for the classification of these strains.[Conclusion]Although the number of CRISPR loci and spacer sequence have no statistical relationship with the number of plasmid, the spacer sequence has genetic homology with plasmid, transposons, integron and resistant genes. This suggests that Salmonella sp. are attacked by exogenous genes constantly during evolution, and CRISPR loci of Salmonella sp. play a role in resisting exogenous genes.
Key words: Salmonella CRISPR spacer plasmid
短回文重复序列首先在大肠杆菌中被发现,后来在其他的原核生物以及古细菌中陆续发现了类似的正向重复序列,2002年Jansen等将该类序列命名为CRISPR[1-2]。CRISPR系统由重复间隔序列、前导序列和cas基因组成,其最明显的结构特征是由一段不连续的重复序列(Direct repeat, DR)和插入其中的间隔序列(Spacer sequence)构成的重复间隔序列[3-4]。重复序列是具有高度保守性且具有回文结构的短小序列,其大小一般不超过40 bp,间隔序列很多源于基因移动原件,如噬菌体和质粒[5]。前导序列(Leader sequence)是位于CRISPR位点5′端、与重复序列相连、长约300-500 bp、富含AT碱基的序列,在同一物种内相对保守,新的间隔序列一般出现在前导序列和第一个重复序列之间[1]。目前研究认为前导序列含有CRISPR转录过程中需要的启动子[6]。此外,在CRISPR座附近存在cas基因(CRISPR-associated),该基因具有多种类型,目前已经发现cas1-cas10等多种不同的类型[7]。cas基因目前也只有在CRISPR结构中才被发现,cas1-cas6基因被认为是CRISPR结构的核心基因,在多种CRISPR结构中出现,其中几乎每一个CRISPR结构中都存在cas1和cas2基因,因此这两个基因被用作鉴别CRISPR系统的分子标记[8]。有研究表明cas基因编码的cas蛋白具有核酸酶、聚合酶、解旋酶和整合酶等多种功能[9]。CRISPR/cas9基因编辑技术已成为目前生物信息界的研究热点。近年来,研究者们认为CRISPR结构能够很好地抵御外源DNA的侵袭如噬菌体、质粒等。Mojica等将含有CRISPR的菌株和不含CRISPR的同类菌株进行对比,发现含有CRISPR结构的菌株能够更好地抵御外源DNA的入侵[10]。目前对于CRISPR的研究大多集中在CRISPR抵御噬菌体的侵入方面,对于CRISPR干预质粒水平转移的方面研究较少。
沙门氏菌(Salmonella)是一类常见的食源性致病菌,主要存在于人和动物的肠道内,属于致病能力极强的革兰氏阴性短杆菌。感染沙门氏菌的人或带菌者的粪便污染食品,可引起人的食物中毒,据统计在各国细菌食物中毒的案例中,由沙门氏菌引起的食物中毒占首位。目前国内对CRISPR的研究主要集中在志贺氏菌和乳酸菌,而对沙门氏菌的研究鲜有报道。本文对基因组测序的30株沙门氏菌的CRISPR结构进行了比较分析,探索CRISPR位点在不同沙门氏菌种中的结构差异,并比较间隔序列与插入序列的同源性以及CRISPR位点与质粒数量关系。
1 材料和方法 1.1 材料 30株沙门氏菌的基因组全序列已收录到NCBI数据库,其中包括12株鼠伤寒沙门氏菌、4株猪霍乱沙门氏菌、3株鸡伤寒沙门氏菌以及11株肠炎沙门氏菌的基因组序列,GenBank编号见表 1。
表 1. 30株沙门氏菌基因组序列信息汇总 Table 1. Summary of information on the genome sequence of 30 strains of Salmonella
| Strains | Assembly |
| Typhimurium str. LT2 | GCA_000006945.1 |
| Typhi str. CT18 | GCA_000195995.1 |
| Typhi str. Ty2 | GCA_000007545.1 |
| Typhimurium str. 14028S | GCA_000022165.1 |
| Typhimurium str. D23580 | GCA_000027025.1 |
| Typhimurium str. ST4/74 | GCA_000188735.1 |
| Typhimurium str. SL1344 | GCA_000210855.2 |
| Typhimurium str. UK-1 | GCA_000213635.1 |
| Typhi str. P-stx-12 | GCA_000245535.1 |
| Typhimurium str. 798 | GCA_000252875.1 |
| Typhimurium str. T000240 | GCA_000283735.1 |
| Typhimurium str. U288 | GCA_000380325.1 |
| Choleraesuis str. SC-B67 | GCA_000008105.1 |
| Choleraesuis str. ATCC 10708 | GCA_000487295.2 |
| Choleraesuis | GCA_000742815.1 |
| Choleraesuis str. SCSA50 | GCA_000187565.1 |
| Gallinarum str. 287/91 | GCA_000009525.1 |
| Gallinarum/pullorum str. RKS5078 | GCA_000235545.1 |
| Gallinarum/pullorum str. CDC1983-67 | GCA_000462995.1 |
| Enteritidis str. P125109 | GCA_000009505.1 |
| Enteritidis str. 77-1427 | GCA_000280315.2 |
| Enteritidis str. CDC_2010K_0968 | GCA_000335875.2 |
| Enteritidis str. 18569 | GCA_000335875.2 |
| Enteritidis str. EC20121175 | GCA_000623055.2 |
| Enteritidis str. EC20111175 | GCA_000626335.1 |
| Enteritidis str. EC20120200 | GCA_000626095.2 |
| Enteritidis str. SA20082034 | GCA_000625855.2 |
| Enteritidis str. EC20120002 | GCA_000624395.2 |
| Enteritidis str. EC20121177 | GCA_000624175.2 |
| Enteritidis str. EC20120916 | GCA_000624155.1 |
表选项
1.2 沙门氏菌中CRISPR位点的查找 沙门氏菌的CRISPR结构分析通过CRISPR Database (http://CRISPR.i2bc.paris-saclay.fr/CRISPR/)数据库的查找以及CRISPR Finder (http://CRISPR.i2bc.paris-saclay.fr/Server/)和CRT软件进行查找预测。本文根据菌名前3位字母和CRISPR结构种类对菌株的CRISPR结构进行命名。
1.3 CRISPR结构的生物信息学分析 采用Clustal W软件和Clcgenomicswb 8软件对CRISPR Finder查找的CRISPR结构进行多重序列比对,Draft基因组中的cas基因通过Domain search (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)以及本地BLAST完成。前导序列的多序列对比分析通过Clustal W软件以及本地BLAST完成。采用MEGA 7.0软件对沙门氏菌的同向重复序列进行遗传进化聚类分析,判断CRISPR能否成为菌种分类的标示基因。采用同样的方法对cas基因也做类似的聚类分析。参数均采用软件默认参数:Start Angle:0 Clock;Radius:300 Pixels;Center Hole:20%。
1.4 间隔序列的同源序列查找 将各菌种的spacer区序列提交到IS Finder网站和INTERGRALL网站进行BLAST,通过在线比对(https://www-is.biotoul.fr/blast.php)分析spacer区序列的同源序列是否来源于外源DNA (采用默认参数)。
1.5 CRISPR与质粒数量的相关性分析 在NCBI下载查找对应菌株的质粒并下载完整序列,采用非参数检验Wilcoxon rank sum test比较分析菌株CRISPR数量与菌株质粒数量之间的关系。所有统计分析由Origin 8.5完成。
2 结果和分析 2.1 沙门氏菌CRISPR结构位点的基因组分布情况 30株沙门氏菌CRISPR位点分析结果表明,共发现CRISPR位点61个,每株沙门氏菌均含有至少一个CRISPR位点,最高不超过3个CRISPR位点,且发现的CRISPR位点均位于染色体上,在质粒中没有发现CRISPR位点。30株沙门氏菌的平均CRISPR个数为2个,与肠炎沙门氏菌的CRISPR平均个数一致,低于鼠伤寒沙门氏菌的2.4个,高于猪霍乱沙门氏菌的1个和鸡伤寒沙门氏菌的1.7个。鼠伤寒沙门氏菌的CRISPR个数差异最大,其中Entericaserovar Typhi str. Ty2、Entericaserovar Typhi str. CT18、Salmonella Entericaserovar Typhi str. P-stx-12 CRISPR位点个数最少均只有1个。11株肠炎沙门的CRISPR位点个数与平均数最为接近均为2个,且这两类菌株的CRISPR结构相对保守。
对30株沙门氏菌的CRISPR-cas系统进行分类,发现沙门氏菌中的CRISPR-cas系统均属于Ⅰ-E和Ⅰ-F类型(包含cas3和cas7基因),对Ⅱ类和Ⅲ类Cas系统均没有检出。对本研究发现的59个CRISPR结构进行结构分析,根据其结构的相似性,将检出的CRISPR结构分为4个类别(CRISPR1-CRISPR4),但在CRISPR1-4结构中除了重复序列数量差异明显之外,其Cas结构具有较强的保守性(图 1)。CRISPR 2和CRISPR 3结构在4种沙门氏菌中都普遍存在,而CRISPR 1结构仅在Salmonella Enterica serovar Typhi str. CT18和Typhimurium str. LT2菌株的基因组中发现,CRISPR 2结构最为常见,3株鸡伤寒沙门氏菌均为CRISPR 4。其中肠炎沙门氏菌的CRISPR结构最为稳定,均为CRISPR 4。CRISPR 3结构最为特殊,在cas区前端具有两段重复区,该结构仅在鼠伤寒沙门氏菌中检出。具有相同CRISPR-Cas结构的菌株其各自的重复区个数存在着显著的差异,重复区个数少的具有3-4个,多的可达30个以上。
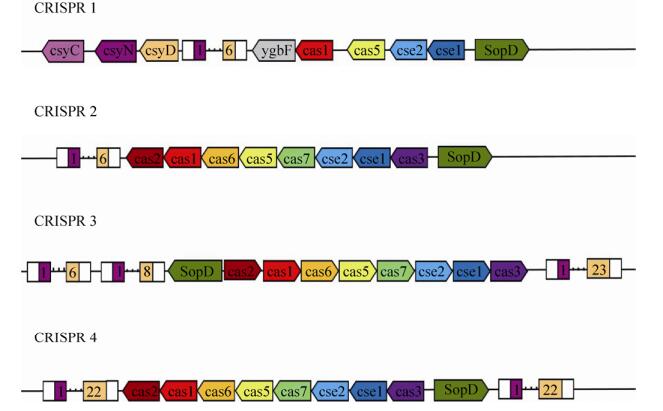 |
| 图 1 30株沙门氏菌中4类CRISPR位点结构示意图 Figure 1 Structure diagram of 4 types of CRISPR sites in 30 strains of Salmonella. Different color arrows represent different genes and directions; The blank box represents the repeated area, and the box with the number represents the interval and the interval number. According to the number of duplicates and the location of the whole CRISPR structure, the structure of CRISPR in 30 strains of Salmonella was divided into four categories, including Typhimurium str. lt2 and Typhi str. ct18. There are 6 strains of CRISPR 2 including Typhi STR. Ty2, Typhi STR. P-stx-12. CRISPR 3 includes 7 strains of Typhimurium str. 14028S, Typhimurium str. D23580. CRISPR 4 includes 15 strains like Choleraesuis str. Sc-b67, Choleraesuis str and ATCC 10708. |
| 图选项 |
2.2 重复序列与Cas蛋白的进化分析 对30株沙门氏菌的重复序列进行对比分析,我们发现重复序列在四类沙门氏菌种具有很强的保守性,但也不完全一致。在同种沙门氏菌中发现了三类差异较为明显的重复序列,其余重复序列的差异仅仅体现在部分碱基的突变导致个别碱基出现差异(表 2,图 2)。以重复序列构建的系统进化树表明本研究的四类沙门氏菌相互混杂,不能彼此区分开,因此不能根据重复序列对属于同种的菌株进行分类(图 3)。已有研究表明,可以根据CRISPR结构的差异对细菌进行分类[11],但本文发现除了肠炎沙门氏菌只具有CRISPR 4结构外,其余3类细菌都至少含有两类CRISPR结构,因此也不能根据CRISPR结构对同种的细菌菌株进行分类。在同一类细菌中也存在完全不同的重复序列,如Gallinarum str. 287/91和Gallinarum/pullorum str. RKS5078 (表 2)。
表 2. CRISPR重复序列的分类分析 Table 2. Classification analysis of CRISPR repeating sequence
| CRISPR | Repeat sequence (5′→3′) | Size of repeat sequence/bp | Size of spacer sequence/bp |
| Typ 1 | CGGTTTATCCCCGCTGGCGCGGGGAACAC | 29 | 32 |
| Typ 2 | CGGTTTATCCCCGCTGGCGCGGGGAACAC | 29 | 32 |
| Typ 3 | CGGTTTATCCCCGCTGGCGCGGGGAACAC | 29 | 32 |
| Typ 4 | CGGTTTATCCCCGCTGGCGCGGGGAACAC | 29 | 32 |
| Cho 4 | CGGTTTATCCCCGCTGGCGCGGGGAACAC | 29 | 32 |
| GTGTTCCCCGCGCCAGCGGGGATAAACCG | 29 | 32 | |
| Cho 2 | GTTTATCCCCGCTAGCGCGGGGAACAC | 27 | 34 |
| Gal 2 | GTGTTCCCCGCGCCAGCGGGGATAAACCG | 29 | 32 |
| GTTTATCCCCGCTAGCGCGGGGAACAC | 27 | 34 | |
| Ent 4 | CGGTTTATCCCCGCTGGCGCGGGGAACAC | 29 | 32 |
| GTGTTCCCCGCGCCAGCGGGGATAAACCG | 29 | 32 | |
| The table lists the sequence of repeat of eight representative strains and the size of repeat sequences and spacer sequences. | |||
表选项
 |
| 图 2 8株沙门氏菌重复区序列 Figure 2 The repeat sequence of 8 strains of salmonella. |
| 图选项 |
 |
| 图 3 利用鸡伤寒沙门氏菌、鼠伤寒沙门氏菌、猪霍乱沙门氏菌,肠炎沙门氏菌30个菌株的重复序列构建系统发生树拓扑结构图 Figure 3 Use of repeat sequence Salmonella typhi, Salmonella Typhimurium, Salmonella Cholera and Salmonella Enteritidis 30 strains to build evolutionary tree. |
| 图选项 |
CRISPR结构中重复区的重复数在同种细菌中的不同类菌株之间存在明显的差异,而且同类细菌中重复数也存在较大差异,不存在规律性。鼠伤寒沙门氏菌中的38个CRISPR位点中共含有重复个数466个(包括可疑位点的确定位点),可疑位点的重复序列长度均为28 bp,确定位点的重复序列长度均为29 bp,其中确定位点中重复数最高的可达33个。猪霍乱沙门氏菌中含有7个CRISPR位点,包含重复区个数86个(均为确定位点),其中重复数最高可达35个,最低的仅含有5个重复数。鸡伤寒沙门氏菌中含有CRISPR位点6个,含有重复区个数34个,肠炎沙门氏菌中的20个CRISPR位点中包含重复序列个数为232个,该菌种的CRISPR结构最为稳定,重复序列重复个数也最为稳定均处于9到13之间,重复区序列长度也均为29 bp。
本研究中,我们在发现的绝大多数CRISPR位点附近发现了Cas蛋白基因,且绝大多数Cas蛋白基因均位于两段重复序列之间。同时,我们发现处于两段重复序列之间的Cas蛋白基因的排列顺序具有高度的一致性,依次为cas2-cas1-cas6-cas5-cas7-cse7-cse2-cse1-cas3。Salmonella Entericaserovar Typhi str. CT18和Salmonella Typhimurium str. LT2 CRISPR结构与其他结构具有明显的不同,其重复序列于Cas蛋白基因之间,且不含cas3基因。经分析本次研究发现的CRISPR结构中均含有cas1基因,因此我们对cas1基因进行聚类分析(图 4),发现cas1基因与重复序列类似,具有较强的保守性,也不能对同种细菌的不同菌株进行区分。
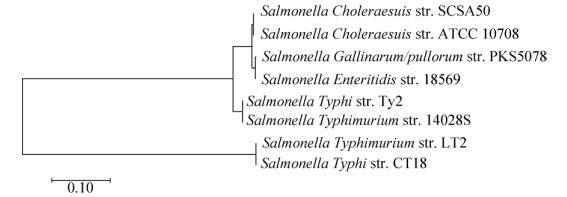 |
| 图 4 利用8株具有代表性菌株的cas1基因序列构建系统发生树拓扑图 Figure 4 Use of cas1 genes of 8 typically strains to build evolutionary tree. |
| 图选项 |
2.3 间隔序列和前导序列的特征分析 在30株沙门氏菌的CRISPR位点中共含有间隔序列747条,它们的长度具有高度的统一性,我们发现重复序列长度为29 bp的CRISPR位点其间隔序列平均长度均为32 bp,重复序列长度为27 bp的CRISPR位点其间隔序列长度均为34 bp。我们把一个重复序列和与其相邻的一个间隔序列称为一个重复单元,我们发现30株沙门氏菌CRISPR位点的一个重复单元序列长度几乎全都为61 bp,我们推测在CRISPR位点附近有着能够严格调控重复单元序列长度的功能基因存在。我们将这些间隔序列递交给INTEGRALL网站进行blast的比对,发现不能搜索到与间隔序列完全匹配到的移动原件基因,但是大部分间隔序列中的12-14 bp长度的序列能够检索到与之完全匹配的来源于其他菌种的质粒、整合子、转座子、耐药基因等移动基因序列,如Typhi str. Ty2菌株CRISPR结构中第一个间隔序列有12 bp序列与质粒pB10-2中的TnpA基因具有同源性(TnpA是一类转座酶基因),Choleraesuis str. ATCC 10708菌株CRISPR结构中的第二个间隔序列有14 bp序列与blaIMP-25 (一类碳青霉烯酶耐药基因)具有同源性(图 5),也有一部分间隔序列不能搜索到与之同源的基因,其原因可能是目前INTEGRALL数据库中的移动原件基因数据不够完整导致。以上结果表明了间隔序列中的部分碱基序列与一些可移动基因具有同源性,但对于32 bp长度的间隔序列仅有12 bp序列能够完全匹配移动原件基因的原因有待进一步探索和研究。同类细菌的不同株细菌间隔区不具有同源性,这表明同类细菌的CRISPR结构可能与细菌所处的环境有关。
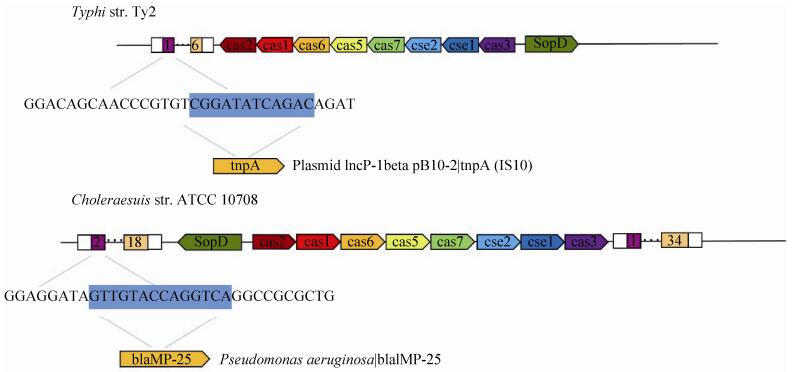 |
| 图 5 菌株Typhi str. Ty2和Choleraesuis str. ATCC 10708的CRISPR位点中间隔序列同源序列分析 Figure 5 The spacer homologous sequence analysis of Salmonella Typhi str. Ty2 and Salmonella Choleraesuis str. ATCC 10708. The TnpA gene in plasmid pb10-2 is a kind of transpoase, and blaIMP-25 is a type of carbapene-resistant gene. |
| 图选项 |
有研究表明,CRISPR位点的上游端可能存在一段前导序列,其长度大约在300-500 bp之间[2]。我们对CRISPR1-6结构的5′侧翼区的1000 bp序列进行多重序列对比,发现该区段存在较多的AAAAA和TTTT结构且存在较多的碱基突变(图 6),说明该区域为基因移动的频繁区域,与基因的启动调控有关。
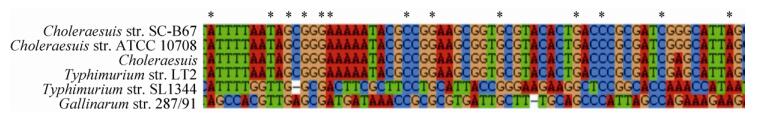 |
| 图 6 5株沙门氏菌前导序列多重序列对比图 Figure 6 Sequence comparison of sequence of 5 Salmonella leader sequences. |
| 图选项 |
2.4 间隔序列数量与质粒数量的相关性分析 对30株沙门氏菌的质粒数和可移动质粒数进行统计分析。沙门氏菌的基因组中质粒均在0-3个之间,其中鼠伤寒沙门氏菌的平均质粒个数为1.5个,具有结合转移区的平均质粒数为1个;猪霍乱沙门氏菌的平均质粒数为1个且均具有结合转移区;鸡伤寒沙门氏菌的基因组没有质粒基因;肠炎沙门氏菌基因组中的平均质粒数为0.3个且均具有结合转移区。在分析沙门氏菌CRISPR位点数量与质粒数量之间的相关性时我们并未发现两者之间具有相关性,CRISPR位点少的基因组质粒数不一定多。从间隔序列的同源性来说,沙门氏菌中的CRISPR的确有抵御外源基因入侵的能力,但与质粒在比较分析间隔序列数量与质粒数量之间关系时并没有发现间隔序列数量越多质粒就越少的现象,我们推测出现该情况的原因可能与质粒的类型有关,可能某些质粒存在抵抗CRISPR结构的功能,这需要对质粒结构进一步深入研究。
3 讨论 沙门氏菌是最为常见的食源性致病菌,能够引起人和动物肠道疾病。美国疾病控制与预防中心曾统计报道:每年将近1/6的人口因食用污染食品而引起不同症状的食物中毒现象,其中沙门氏菌是引起食物中毒和疾病暴发的主要病原菌, 而最为主要的就是鼠伤寒沙门氏菌、肠炎沙门氏菌、鸡伤寒沙门氏菌和猪霍乱沙门氏菌。目前认为人类沙门氏菌感染和食物中毒主要源于被沙门氏菌感染的禽类、蛋类及其他禽类产品[12]。
CRISPR系统是能够抵御质粒、噬菌体、转座子等移动基因原件入侵的防御体系,其中CRISPR/Cas9基因编辑技术已经发展成为一种最有效的基因编辑技术[13]。CRISPR系统能够抑制携带耐药基因的质粒、转座子、整合子等外源基因的入侵,从而防止耐药基因在细菌间的传播,尤其是致病细菌之间的传播,这对临床治疗和畜牧业都具有重要的意义[14-15]。本文对沙门氏菌的CRISPR位点进行生物信息学分析,有助于了解CRISPR位点在沙门氏菌中的分布情况和结构等信息,分析推测外源质粒与CRISPR之间的关系,其中我们发现间隔序列与许多耐药基因和转座子、整合子、质粒具有不同程度的同源性,这也证实了Bikard等的研究[5],对设计干预致病菌获得耐药性具有基因层面的指导意义。
30株细菌中均至少存在一个CRISPR位点,通过CRISPR位点分析可将其分为4类。在对沙门氏菌的CRISPR-Cas系统进行分类时我们发现沙门氏菌基因组中的CRISPR-Cas系统基本均属于Ⅰ类中的Ⅰ-E亚类型,这与王鹏飞等[16]研究志贺氏菌、大肠杆菌和沙门氏菌CRISPR结构特征生物信息学分析的结果一致。从CRISPR位点在4类细菌基因组的分布来看,在数量上并没有显著差异,但本次研究发现的CRISPR结构均位于染色体基因上,在质粒上没有检出,并且发现重复序列具有一定的保守性,主要发现三类差异较大的重复序列,在同一类细菌中也存在不同重复序列的情况,在对重复序列进行生物进化分析后表明重复序列不能对同种细菌下的不同类细菌进行分类,说明CRISPR基因很可能是通过基因水平转移得到的,并非源自自身进化。本次研究主要发现了4类结构不同的CRISPR类型(CRISPR1-4),同种细菌的不同类细菌具有不同的结构类型但间隔序列的长度具有高度统一性,这表明不同类型的CRISPR结构的作用机制可能是一致的。
通过对cas基因的研究,我们发现30株沙门氏菌的cas基因排列顺序具有高度统一性,与重复序列的保守性一样,cas1基因也具有类似的保守性,与有关文献报道的结果相一致[17]。这说明沙门氏菌在进化过程中可能面临着类似的选择压力。前导序列在同种细菌中不具有很强的保守性,存在较多碱基突变和缺失的情况,这表明前导序列是一段较为活跃的基因,可能具有启动CRISPR基因表达的作用,这与Yoshinao等[18]关于细菌基因组分析结果一致。通过对间隔区序列的分析,我们发现虽然间隔序列不能完全在INTEGRALL数据库中发现匹配序列,但间隔序列的部分序列可以找到与之完全匹配的序列,而这些序列绝大部分来源于质粒、噬菌体、转座子、整合子和耐药基因。这表明沙门氏菌中的CRISPR位点确实具有抵御外源基因侵入的功能,但在研究沙门氏菌CRISPR位点数量与质粒数量以及间隔序列数量与质粒数量之间的相关性时,并没有发现两者之间具有统计学的相关性。我们推测出现该情况的原因可能与质粒的类型有关,可能某些质粒存在抵抗CRISPR结构的功能,这需要对质粒结构进一步深入研究。
References
| [1] | Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(3): 653-663. DOI:10.1099/mic.0.27437-0 |
| [2] | Jansen R, van Embden JDA, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [3] | Reeks J, Naismith JH, White MF. CRISPR interference:a structural perspective. Biochemical Journal, 2013, 453(2): 155-166. DOI:10.1042/BJ20130316 |
| [4] | Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [5] | Bikard D, Hatoum-Aslan A, Mucida D, Marraffini LA. Crispr interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection. Cell Host & Microbe, 2012, 12(2): 177-186. |
| [6] | Barrangou P, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [7] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [8] | Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions. Microbiology, 2010, 64: 475-493. DOI:10.1146/annurev.micro.112408.134123 |
| [9] | Marraffini LA, Sontheimer EJ. CRISPR interference:RNA-directed adaptive immunity in bacteria and archaea. Nature Reviews Genetics, 2010, 11(3): 181-190. DOI:10.1038/nrg2749 |
| [10] | Mojica FJM, Díez-Villase?or C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Molecular Microbiology, 2000, 36(1): 244-246. DOI:10.1046/j.1365-2958.2000.01838.x |
| [11] | Baird-Parker AC. Food borne salmonellosis. The Lancet, 1990, 336(8725): 1231-1235. DOI:10.1016/0140-6736(90)92844-8 |
| [12] | Luber P. Cross-contamination versus undercooking of poultry meat or eggs-which risks need to be managed first?. International Journal of Food Microbiology, 2009, 134(1-2): 21-28. DOI:10.1016/j.ijfoodmicro.2009.02.012 |
| [13] | Mussolino C, Cathomen T. RNA guides genome engineering. Nature Biotechnology, 2013, 31(3): 208-209. DOI:10.1038/nbt.2527 |
| [14] | Bachmann NL, Petty NK, Zakour NLB, Szubert JM, Savill J, Beatson SA. Genome analysis and CRISPR typing of Salmonella enterica serovar Virchow. BMC Genomics, 2014, 15: 389. DOI:10.1186/1471-2164-15-389 |
| [15] | Shariat N, Timme RE, Pettengill JB, Barrangou R, Dudley EG. Characterization and evolution of Salmonella CRISPR-Cas systems. Microbiology, 2015, 161(2): 374-386. DOI:10.1099/mic.0.000005 |
| [16] | Wang PF. Bioinformatics analysis of CRISPR structures and feature in Shigella, Escherichia coli and Salmonella. Doctor Dissertation of Zhengzhou University, 2015. 王鹏飞. 志贺菌、大肠杆菌和沙门菌CRISPR结构特征生物信息学分析. 郑州大学博士学位论文, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10459-1015302504.htm |
| [17] | Horvath P, Co?té-Monvoisin AC, Romero DA, Boyaval P, Fremaux C, Barrangou R. Comparative analysis of CRISPR loci in lactic acid bacteria genomes. International Journal of Food Microbiology, 2009, 131(1): 62-70. DOI:10.1016/j.ijfoodmicro.2008.05.030 |
| [18] | Azuma Y, Hosoyama A, Matsutani M, Furuya N, Horikawa H, Harada T, Hirakawa H, Kuhara S, Matsushita K, Fujita N, Shirai M. Whole-genome analyses reveal genetic instability of Acetobacter pasteurianus. Nucleic Acids Research, 2009, 37(17): 5768-5783. DOI:10.1093/nar/gkp612 |
