王梦, 危双, 王婷, JosefVoglmeir, 刘丽


南京农业大学食品科学技术学院, 江苏 南京 210095
收稿日期:2017-03-11;修回日期:2017-05-27;网络出版日期:2017-07-03
基金项目:国家自然科学基金(31371739,31671854);中央高校基本科研业务费(KYRC201209)
*通信作者:刘丽, Tel:+86-25-84399553;Fax:+86-25-84399513;E-mail:lichen.liu@njau.edu.cn
摘要:[目的]对细菌Solitalea canadensis中编码β-N-乙酰氨基己糖苷酶的基因进行克隆,通过原核表达获得重组β-N-乙酰氨基己糖苷酶,并研究其酶学性质。[方法]以Solitalea canadensis基因组DNA为模板,使用加尾PCR的方法克隆编码β-N-乙酰氨基己糖苷酶的基因,构建含有组氨酸标签的重组表达载体,并将重组质粒导入大肠杆菌BL21(DE3)中进行原核表达。重组蛋白经Ni-NTA纯化,以对硝基苯酚-β-乙酰氨基葡萄糖(pNP-β-GlcNAc)为底物研究其酶学性质,包括最适温度、最适pH以及金属离子和抑制剂的影响。[结果]从菌株Solitalea canadensis克隆得到了β-N-乙酰氨基己糖苷酶基因片段(GeneBank:WP_014682183.1),全长2586 bp,重组表达所得蛋白表观分子量约为97 kDa,最适pH 6.0,最适温度42℃,但不稳定,半衰期小于5 min。该酶对十二烷基磺酸钠(SDS)敏感,活性受Triton X-100和尿素的抑制。此外二糖分子也能不同程度地抑制该重组酶的活性,特异性抑制剂PugNAc(O-(2-Acetamido-2-deoxy-D-glucopyranosylideneamino)N-phenylcarbamate)对该酶的IC50为2 μmol/L。该重组酶蛋白除能水解对硝基苯酚-β-乙酰氨基葡萄糖苷和对硝基苯酚-β-乙酰氨基半乳糖(pNP-β-GalNAc)外,还能对O-链聚糖核心结构Core Ⅱ末端的乙酰氨基葡萄糖进行水解。[结论]本文首次从Solitalea canadensis中克隆得到能水解末端β 1-6连接的乙酰氨基葡萄糖而不能水解β 1-4连接键的β-N-乙酰氨基己糖苷酶,并对其进行了酶学性质研究和底物特异性分析,为开发高效特异性强的糖链分析工具酶提供理论基础。
关键词: Solitalea canadensis β-N-乙酰氨基己糖苷酶 酶学特性
Gene cloning, expression, purification and characterization of a beta-N-acetylhexosaminidase from Solitalea canadensis
Meng Wang, Shuang Wei, Ting Wang, Josef Voglmeir, Li Liu


College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095, Jiangsu Province, China
Received 11 March 2017; Revised 27 May 2017; Published online 3 July 2017
*Corresponding author: Li Liu, Tel:+86-25-84399553;Fax:+86-25-84399513;E-mail:lichen.liu@njau.edu.cn
Supported by the National Natural Science Foundation of China (31371739, 31671854) and by the Fundamental Research Funds for the Central Universities (KYRC201209)
Abstract: [Objective]We intended to discover and characterize a novel beta-N-acetylhexosaminidase from Solitalea canadensis.[Methods]Genomic DNA extracted from Solitalea canadensis was used as the template for gene cloning of the beta-N-acetylhexosaminidase using PCR reaction. The PCR product was digested with restriction endonucleases Nde Ⅰ and Xho Ⅰ, then ligated to pET-30a vector. After plasmid was transformed into E.coli BL21 (DE3) cells, the recombinant enzyme was expressed by IPTG induction and purified with nickel affinity chromatography. Characterization of recombinant SoCaHexNAc including optimal pH and temperature, metal ions dependency and inhibitor was done using pNP-β-GlcNAc as the substrate. Effect of different chemical compounds and disaccharides on the enzyme activity was also measured.[Results]A beta-N-acetylhexosaminidase gene with an open reading fragment of 2586 bp was successfully obtained, which encodes 856 amino acids with a putative molecular size of 97 kDa. The results of SDS-PAGE revealed that the recombinant SoCaHexNAc (GeneBank accession number:WP_014682183.1) was expressed and purified successfully. Characterization of the enzyme showed that the optimum pH of SoCaHexNAc is 6.0, and the optimum temperature is 42℃ with a half-life being less than 5 minutes. The recombinant SoCaHexNAc was sensitive to SDS and could be partly inhibited by Trition X-100 and urea. Different concentrations of lactose, maltose and cellobiose could also inhibit the activity of SoCaHexNAc to different extends. The IC50 of a specific β-N-acetylhexosaminidase inhibitor, PugNAc, was 2 μmol/L. The substrate specificity result showed that the recombinant SoCaHexNAc was active to pNP-GlcNAc and pNP-GalNAc. When being used for the hydrolysis of GlcNAc from natural glycans, the recombinant SoCaHexNAc exhibited linkage specificity evidenced by the fact that only β 1, 6-linked GlcNAc in Core Ⅱ structure, but not the β 1, 4-linked GlcNAc in NGA2 structure, was removed, although the terminal GlcNAc was the exceptional terminal sugar in both substrates.[Conclusion]A beta-N-actylhexosamindase with activity specifically towards β 1, 6-linked but not β 1, 4-linked GlcNAc was discovered and characterized from Solitalea canadensis for the first time. The results of characterization and substrate specificity showed it might be a potential novel tool enzyme which could be used in structure analysis of glycans.
Key words: Solitalea canadensis beta-N-acetylhexosaminidase enzymatic characterization
糖链的结构分析是糖组学研究的重要内容之一,其主要任务是研究组成糖链的各种单糖的种类、数量、连接位点及键型等。糖链不像蛋白质一样是基因编码的产物,有特定的排列规律。组成糖链的单糖种类和数量多变,单糖之间的连接方式(包括连接位点、连接键型)也异常复杂,存在大量同分异构体,导致糖链结构分析工作任务艰巨。长期以来,科研工作者一直致力于糖链结构分析技术和方法的开发和改良,目前主要使用的技术方法包括:高效液相色谱(HPLC)、质谱(MS)、核磁共振(NMR)、毛细管电泳和糖芯片技术等。其中外切糖苷酶技术结合高效液相色谱是最常用的对糖链进行初步结构解析的方法之一。外切糖苷酶能够特异性地将某种特定键型的末端单糖从糖链上解离下来,反应条件温和,催化效率高并具有很强的底物特异性。使用外切糖苷酶技术结合高效液相色谱,能够明确糖链的单糖组成、连接位点和键型等详细信息,被认为是一种绿色高效的糖链结构分析手段。乙酰氨基葡萄糖(GlcNAc)和乙酰氨基半乳糖(GalNAc)广泛存在于各种糖结构中,使用β-N-乙酰氨基己糖苷酶特异性水解末端不同键型连接的乙酰氨基葡萄糖和乙酰氨基半乳糖将为复杂的糖链分析工作带来巨大便利。
β-N-乙酰氨基己糖苷酶(β-N-acetylhexosaminidase,EC 3.2.1.52) 是一类能特异性水解糖链非还原末端乙酰氨基己糖的糖苷酶,通常同时具有β-N-乙酰氨基葡萄糖苷酶和β-N-乙酰氨基半乳糖苷酶活性,而且对β-N-乙酰氨基葡萄糖的水解活性大于对β-N-乙酰氨基半乳糖[1]。β-N-乙酰氨基己糖苷酶根据催化原理的不同分属于GH20、GH3或GH84三个蛋白家族[2],尤以GH20家族中的β-N-乙酰氨基己糖苷酶数目最多。该酶广泛存在于人类、哺乳动物、昆虫、植物以及微生物中,参与糖蛋白、糖脂、氨基聚糖等的代谢过程,具有重要生物学功能[3-8]。目前用于糖结构分析的β-N-乙酰氨基己糖酶主要有两种,分别来自刀豆和肺炎双球菌。这两种酶都是广谱性酶,能够水解包括1-2、1-3、1-4和1-6在内的所有β连接乙酰氨基葡萄糖,因此只能区分α和β连接键型,不能区别连接位点,且两种酶都不能水解N-糖链中平分型的末端乙酰氨基葡萄糖。此外,来自肺炎双球菌的酶只对GlcNAc有活性,不能水解末端GalNAc。由此可见,现有的β-N-乙酰氨基己糖酶在糖链结构研究的应用中还存在不足之处,尤其缺乏对不同键型的选择性,因此开发一种特异性水解某单一键型、具有高度底物选择性的β-N-乙酰氨基己糖苷酶十分必要。
Solitalea canadensis (F. canadensis)是一种从土壤中分离得到的革兰氏阴性菌[9],其全基因组信息已经公布,但关于Solitalea canadensis源的β-N-乙酰氨基己糖苷酶至今未见描述。本文针对Solitalea canadensis基因组中编码β-N-乙酰氨基己糖苷酶的基因,利用分子生物学手段进行了克隆,构建了表达载体,成功在大肠杆菌中进行了异源表达,并对其酶学特性进行了研究,测定了底物特异性,以期开发用于糖链结构分析的新工具酶。
1 材料和方法 1.1 菌种和载体 菌株Solitalea canadensis DSM3403,购自德国菌种保藏中心;敲除β-半乳糖苷酶基因的大肠杆菌菌株BL21(DE3) 和Top 10购自天根(北京)生化科技有限公司,其中Top 10经抗噬菌体改造命名为BMMach1 T1,保存于本实验室;pTG-19载体购自上海捷瑞生物工程有限公司;pET-30a载体购自Novagen公司。
1.2 主要试剂和生物信息学软件 DNA回收试剂盒、质粒提取试剂盒均购自Axygen公司(北京);改良型Bradford蛋白浓度测定试剂盒购自生物工程(上海)股份有限公司;rTaq DNA聚合酶购自TaKaRa公司(大连);T4连接酶、限制性内切酶Nde Ⅰ、Xho Ⅰ购自Thermo Scientific公司;细菌培养所用抗生素分别为氨苄青霉素(100 μg/mL)、卡那霉素(50 μg/mL);液相色谱仪流动相均为色谱纯,购自默克公司;引物合成和基因测序由金斯瑞(南京)生物科技有限公司完成。序列比对使用BLAST工具(https://blast.ncbi.nlm.nih.gov/Blast.cgi),多重序列比对使用Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/),进化树使用软件MEGA 6.0.6绘制,二级结构预测使用在线软件ESPript 3.0 (http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)完成。
1.3 Solitalea canadensis β-N-乙酰氨基己糖苷酶基因扩增 根据NCBI数据库中Solitalea canadensis编码β-N-乙酰氨基己糖苷酶的DNA序列,使用软件Primer Premier 5进行引物设计,并在前引物和后引物的5′端分别添加Nde Ⅰ和Xho Ⅰ限制性内切酶酶切位点。前引物F:5′-GGAATTCCATATGATGA AAAATTTCACACTTAGTTTG-3′ (Nde Ⅰ),后引物R:5′-CCGCTCGAGGTTCTTGATCTCCTCAATTC TG-3′ (Xho Ⅰ)。Solitalea canadensis基因组DNA参照Mahuku[10]等报道的方法进行提取。以基因组DNA为模板进行PCR扩增,PCR程序为:95 ℃ 5 min,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min,循环35次,72 ℃ 10 min。扩增产物使用1%琼脂糖凝胶进行电泳分离,并按Axygen DNA回收试剂盒说明进行切胶回收。
1.4 表达载体的构建 回收得到的基因片段在T4连接酶作用下与pTG-19载体连接得到重组质粒,并热激转化至BMMach1 T1感受态细胞,使用蓝白斑筛选重组子,并挑取白色菌落以M13通用引物进行PCR鉴定。按Axygen质粒提取试剂盒说明对阳性克隆菌落抽提质粒,所得质粒用限制性内切酶Nde Ⅰ和Xho Ⅰ进行双酶切,酶切后的目的基因片段与经相同酶切处理的pET-30a载体连接,并将连接产物转化至BMMach1 T1感受态细胞中,使用菌落PCR进行鉴定,将阳性克隆送样至测序公司测序,得到构建成功的重组质粒pET30a-SoCaHexNAc。
1.5 β-N-乙酰氨基己糖苷酶的外源表达和纯化 提取重组质粒pET30a-SoCaHexNAc,转化入E. coli BL21(DE3) 感受态细胞。挑取单菌落于5 mL含卡那霉素(50 μg/mL)的LB培养基中过夜培养,然后接种至含有400 mL LB培养基的摇瓶中扩大培养,直至菌液浓度达到OD600约为0.5,加入IPTG至终浓度1 mmol/L,18 ℃诱导表达24 h。
4 ℃、4000 r/min离心收集菌体,将菌体沉淀重悬于10 mL裂解液(100 mmol/L NaCl,50 mmol/L Tris-HCl pH 8.0,1% Triton X-100,1 mmol/L PMSF),超声破碎细胞,12000 r/min、4 ℃离心20 min,收集上清,使用Ni-NTA亲和层析进行纯化,镍柱使用5倍柱体积的平衡缓冲液(50 mmol/L Tris/HCl pH 8.0,50 mmol/L NaCl)进行平衡后上样,并使用10倍柱体积的结合缓冲液(50 mmol/L Tris/HCl pH 8.0,50 mmol/L NaCl,20 mmol/L咪唑)冲洗去除非特异结合的蛋白,再用洗脱缓冲液(50 mmol/L Tris/HCl pH 8.0,50 mmol/L NaCl,500 mmol/L咪唑)洗脱重组蛋白,280 nm波长检测柱下液,收集蛋白峰即纯化后的重组酶。
对诱导前后、上清和纯化蛋白进行SDS-PAGE,分析重组蛋白的表达水平、纯化情况和分子量,分离胶电泳电压100 V,浓缩胶电泳电压80 V。蛋白含量的测定使用基于Bradford法的蛋白定量试剂盒,以牛血清蛋白(BSA)为标准绘制曲线,具体操作参照试剂盒说明进行。
1.6 酶活力的测定 以对硝基苯酚-β-乙酰氨基葡萄糖苷(pNP-β-GlcNAc)为底物,通过对硝基苯酚(pNP,p-nitrophenol)的生成量计算重组β-N-乙酰氨基己糖苷酶的活力。一个酶活力单位(1 U)定义为37 ℃条件下,每分钟水解产生1 μmol对硝基苯酚所需的酶量。配置一系列不同浓度的pNP标准溶液:0.01、0.02、0.05、0.10、0.20、0.50、1.00、2.00、5.00 mmol/L。取25 μL pNP标准溶液与50 μL 1 mol/L Na2CO3溶液混匀,取30 μL于酶标仪405 nm波长测定吸光值,绘制不同浓度pNP与吸光度标准曲线。25 μL酶活力测定的反应体系含1 mmol/L pNP-β-GlcNAc,50 mmol/L缓冲液。向反应体系中加入酶溶液10 μL,37 ℃孵育10 min后立即加入50 μL 1 mol/L Na2CO3溶液终止反应,取30 μL于384孔板中,使用酶标仪检测405 nm波长下吸光度。每个反应设置3个重复,以不加酶的反应体系为空白对照。根据读取的吸光度和标准曲线换算出产物生成量,计算酶活力。
1.7 重组N-乙酰氨基己糖苷酶的酶学特性研究
1.7.1 最适pH的测定: 以pNP-β-GlcNAc为底物,分别检测不同pH值缓冲体系对酶活力的影响。所用缓冲体系分别为磷酸氢二钠-柠檬酸溶液(pH 3.0-8.0)、Tris-HCl (pH 8.0-9.0)、Na2CO3-NaHCO3 (pH 9.0-10.0) 为缓冲液,缓冲液pH梯度0.5。反应体系同上述酶活力测定方法。
1.7.2 最适温度和不同温度下稳定性的测定: 在最适pH下测定酶反应的最适温度,测定温度分别为4、16、25、37、42、45、50、55、60、65和70 ℃。反应体系和操作方法同最适pH的测定。温度稳定性试验分别将酶溶液(含0.5% BSA)在37 ℃和42 ℃下孵育1、2、5、10、15、30 min和1 h (以不加酶和未经温度孵育的酶为对照)分别测定酶活性。
1.7.3 金属离子和不同化学物质对酶活性影响的测定: 在最适条件下,分别向上述反应体系中加入Mg2+、Mn2+、Zn2+、Ni2+、Cu2+、Fe3+、Ca2+ (金属离子终浓度为2 mmol/L),按上述步骤测定金属离子对酶活性的影响,同时设置不含金属离子和含2 mmol/L EDTA的反应体系作为对照。以同样的方法分别测定不同化学物质对酶活性的影响,包括尿素(终浓度0.1、0.5、1.0 mol/L),β-巯基乙醇(2-ME,终浓度1、10、50 mmol/L),Triton X-100 (终浓度0.1%、0.5%、1.0%,V/V),SDS (终浓度0.1%、0.5%、1.0%,M/V)。
1.7.4 二糖分子对酶活性影响的测定: 在最适条件下,向反应体系分别加入终浓度5、25和50 mmol/L不同二糖,包括蔗糖、乳糖、麦芽糖、松二糖、密二糖和纤维二糖,测定不同二糖分子对酶活性的影响,操作步骤如上所述。
1.7.5 特异性抑制剂(PugNAc)对酶活性影响的测定: 以pNP-β-GlcNAc为底物,使用酶标仪测定PugNAc [O-(2-Acetamido-2-deoxy-D-glucopyranosylideneamino)N-phenylcarbamate]对酶活性的抑制作用。PugNAc的终浓度设置如下:1、2、5、10、20、50 pmol/L,0.1、0.2、0.5、1.0、2.0、5.0、10.0、20.0、50.0 nmol/L,1.0、2.0、5.0、10.0、20.0、50.0 μmol/L和0.1 mmol/L。操作步骤如上所述。1.8 重组N-乙酰氨基己糖苷酶底物特异性鉴定 重组酶对于不同pNP-单糖活性的测定:使用酶标仪分别以对硝基苯酚-β-葡萄糖苷(pNP-β-glucose)、对硝基苯酚-β-半乳糖苷(pNP-β-galactose)、对硝基苯酚-β-乙酰葡萄糖苷(pNP-β-GlcNAc)、对硝基苯酚-β-乙酰半乳糖苷(pNP-β-GalNAc)、对硝基苯酚-α-葡萄糖苷(pNP-α-glucose)、对硝基苯酚-α-半乳糖苷(pNP-α-galactose)、对硝基苯酚-α-乙酰氨基葡萄糖苷(pNP-α-GlcNAc)和对硝基苯酚-α-岩藻糖苷(pNP-α-Fucose)为底物实时测定OD405。反应体系中底物终浓度2 mmol/L,缓冲体系为50 mmol/L pH 6.0的磷酸盐-柠檬酸缓冲液,反应温度37 ℃,反应时间1 h。
重组酶对于pNP-Core Ⅱ活性的测定:以pNP-Core Ⅱ为底物(终浓度0.1 mmol/L),缓冲体系为50 mmol/L pH 6.0的磷酸盐-柠檬酸缓冲液,37 ℃分别反应1、2、5 h,95 ℃加热5 min终止反应。反应产物使用高效液相色谱(HPLC)方法进行测定。色谱条件:流动相A为含0.1%三氟乙酸(TFA)的水,流动相B为含0.1% TFA的乙腈;流速0.8 mL/min,紫外检测器检测波长330 nm,色谱柱选用HyperCloneTM 5 μm ODS (C18) 120? 250 mm×4.6 mm,进样体积20 μL。洗脱程序:0-6.0 min流动相B比例线性提高至90%,6.0-8.0 min流动相B比例保持不变,8.0-9.0 min流动相B比例降至20%。
重组酶对于2AB-NGA2活性的测定:以2AB标记的NGA2为底物(终浓度1 μmol/L),缓冲体系为20 mmol/L pH 6.0 MES缓冲液,37 ℃反应12 h,加入乙腈至终浓度80%终止反应,离心取上清,使用超高压液相色谱(UPLC)检测酶反应产物。色谱条件:流动相A为50 mmol/L NH4COOH (pH 4.5),流动相B为乙腈;流速0.5 mL/min;柱温60 ℃,荧光检测器激发波长330 nm和发射波长420 nm,色谱柱为Waters ACQUITY UPLC? BEH Glycan Column 1.7 μm×2.1 mm×150 mm,进样体积45 μL。洗脱程序:流动相B比例0-6 min由95%降至78%,6.0-20 min由78%降至70%,20-21 min升至100%,23-25 min降至95%。
2 结果和分析 2.1 基因克隆、载体构建与序列分析 以Solitalea canadensis基因组DNA为模板使用所设计引物进行基因扩增,琼脂糖凝胶电泳显示成功得到一全长约2500 bp的DNA片段,命名为SoCaHexNAc (图 1-A)。扩增所得DNA片段经酶切和连接,构建得到重组质粒pET30a-SoCaHexNAc。测序结果表明克隆得到的重组β-N-乙酰氨基己糖苷酶基因与Solitalea canadensis基因序列匹配,完整开放阅读框全长2586 bp,共编码861个氨基酸残基,带有C-端组氨酸标签。BLAST数据库信息显示该蛋白属GH20家族。系统发育进化树显示该基因所编码蛋白序列与目前已知的来自玉米螟Ostrinia furnacalis (ABI81756.1)、人类乙酰氨基己糖苷酶的β亚基(NP_000512.1)、Paenivacillus sp. Ts12 (3GH4)、Treptomyces plicatus (1HP4)、Streptococcus pneumonia (5A69 & WP_000679939.1)、Talaromyces flavus (AEQ33603.1)、Serratia marcescens(ETX49365.1)、Aggregatibacter actinomycetemcomitans(1YHT)、Serratia marcescens (WP_016928753.1) 的GH20家族β-N-乙酰氨基己糖苷酶有不同程度的相似性(图 1-B)。氨基酸序列多重比对结果显示,尽管在分子进化树上进化关系远近不同,但来自真核和原核生物的11个不同β-N-乙酰氨基己糖苷酶在氨基酸序列Val313-Lys373、Tyr408-Lys451、His518-Glu523、Lys602-Trp605、Ile701-Trp735之间都具有保守性。二级结构预测也显示这几段序列内所含α螺旋和β折叠,数量和位置极为类似,可能与形成(β/α)8桶状催化结构域密切相关(图 2)。第522位的天冬氨酸(Asp,D)和第523位的谷氨酸(Glu,E)的2个酸性氨基酸在所有酶中都高度保守,可能与催化活性中心有关。
 |
| 图 1 PCR扩增产物电泳结果及与不同来源GH20家族β-N-乙酰氨基己糖苷酶进化树分析 Figure 1 Agarose gel electrophoresis and phylogenetic analysis of β-N-acetylhexosaminidase from Solitaleacanadensis. A: M: DNA marker; SoCaHexNAc: PCR product of SoCaHexNAc. B: phylogenetic tree constructed using Maximum Likelihood method by the software Mega 6 based on the amino acid sequences of β-N-acetylhexosaminidase from different sources, accession numbers were given in brackets and the enzyme identified in this study was with gray shade. Bootstrap values (n=500 replicates) were reported above branches as percentages, the length of branches were shown under branches. |
| 图选项 |
 |
| 图 2 SoCaHexNAc与其他来源的GH20家族β-N-乙酰氨基己糖苷酶的序列多重对比图 Figure 2 Multiple sequence alignment of β-N-acetylhexosaminidases from Solitalea canadensis and other sources. The multiple sequence alignment was performed by Clustal Omega, and secondary structure indicated along the top of alignment (α-helix; β-sheet) was constructed using ESPript 3.0 based on the structure of 1YHT. Identical residues are in red, highly conserved residues are shaded in red. |
| 图选项 |
2.2 重组蛋白的表达和纯化 重组大肠杆菌IPTG诱导前后菌体、裂解所得上清液和镍亲和层析纯化组分的SDS-PAGE分析结果如图 3所示。通过比较诱导前后菌体的蛋白组成,可以发现IPTG诱导后菌体在分子量100 kDa左右具有一明显增强条带,同时上清液和纯化组分都在相同位置具有信号,应为所表达重组蛋白SoCaHexNAc,其表观分子量与理论分子量(97.5 kDa)相吻合,表明经Ni-NTA纯化得到重组蛋白。Bradford法测定的重组蛋白浓度为1.4 mg/mL,比活力为33.8 U/mg。
 |
| 图 3 重组蛋白SDS-PAGE Figure 3 SDS-PAGE of recombinant protein from Solitalea canadensis. M: marker; 1: cell pallet before induction; 2: cell pallet after induction with IPTG; 3: supernatant after cell lysis; 4: protein after purification on Ni-NTA agarose column. |
| 图选项 |
2.3 酶学性质研究
2.3.1 最适pH: 不同pH缓冲体系对重组SoCaHexNAc的活性影响如图 4-A所示。重组SoCaHexNAc最适pH为6.0,在pH 5.0-7.5范围内酶活力可保持60%以上,当pH低于5.0时酶的活性迅速下降。
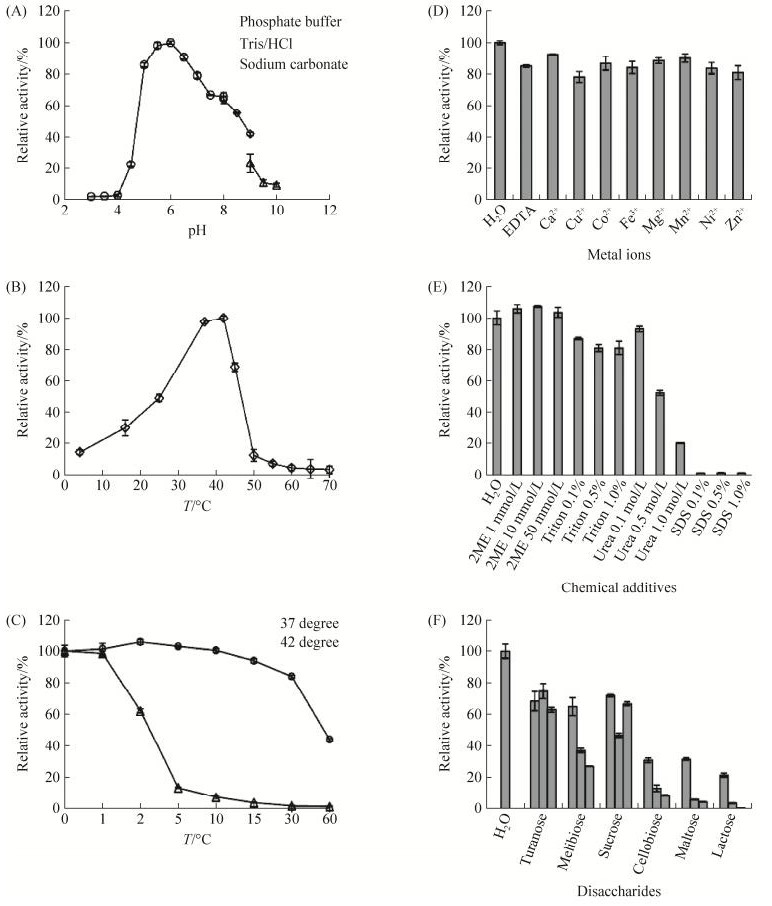 |
| 图 4 SoCaHexNAc酶学性质研究 Figure 4 Characterization of the recombinant β-N-acetylhexosaminidase, SoCaHexNAc. A: pH optima assayed at 37 ℃ in buffers ranging from pH 3.0–10.0. B: temperature optima assayed at the optimum pH from 4 ℃ to 70 ℃. C: thermostability of SoCaHexNAc done with enzymes incubated at 37 ℃ and 42 ℃ for different time intervals. D: effect of metal ions measured by adding different metal ions with a final concentration of 2 mmol/L. E: effect of different chemical additives. F: effect of different disaccharides with concentrations of 5, 25 and 50 mmol/L. |
| 图选项 |
2.3.2 最适温度及温度稳定性: 重组SoCaHexNAc的表观最适温度为42 ℃ (图 4-B)。当反应温度高于42 ℃时,酶活性迅速降低,50 ℃时酶活性仅为12%。温度稳定性试验显示,42 ℃环境下该酶不稳定,但在温度37 ℃孵育30 min仍可以保持原有酶活力的80%以上(图 4-C)。尽管重组SoCaHexNAc表观最适温度为42 ℃,但该温度下酶并不稳定,由于37 ℃下该酶的稳定性相对较好,因此在后续的实验中,我们选用了37 ℃为反应温度而不是最适温度42 ℃。
2.3.3 金属离子和化学添加剂对酶活性的影响: 金属离子影响的测定结果显示不同金属离子对酶活性均无显著影响(图 4-D)。不同浓度的2-ME和Triton X-100对酶的活性无显著影响。尿素对重组SoCaHexNAc的抑制作用具有剂量效应,1 mol/L尿素能够抑制约80%的酶活性。SDS对酶活性的抑制作用明显,0.1% SDS即可抑制90%以上的酶活性(图 4-E)。
2.3.4 二糖对酶活性的影响: 密二糖、纤维二糖、麦芽糖和乳糖对重组SoCaHexNAc酶活性具有抑制作用,其中乳糖的抑制作用最为明显,50 mmol/L的乳糖可以完全抑制酶活性。松二糖和蔗糖也具有一定抑制作用,但抑制剂浓度与抑制作用没有明显的线性关系(图 4-F)。
2.3.5 特异性酶抑制剂PugNAc对酶活性的抑制作用: 0-2 nmol/L PugNAc不影响酶的活性,PugNAc浓度大于2 nmol/L时,PugNAc对重组酶SoCaHexNAc产生抑制作用并具有量效关系,其IC50为2 μmol/L。2.4 底物特异性
2.4.1 重组SoCaHexNAc对pNP-单糖的底物特异性研究: 在384孔板中依次加入8 μL酶反应液(含50 mmol/L pH 6.0的磷酸盐-柠檬酸缓冲液和1 mmol/L不同的pNP-单糖),向各个孔中同时加入2 μL酶液,立即测定405 nm下实时吸光度,设置不加酶的对照组。结果显示SoCaHexNAc可以特异性水解pNP-β-GlcNAc和pNP-β-GalNAc,其中pNP-β-GlcNAc的水解速率高于pNP-β-GalNAc,对其他底物无作用(图 5-A)。
 |
| 图 5 SoCaHexNAc底物特异性 Figure 5 Activity detection of recombinant SoCaHexNAc. A: substrate specificity assays of SoCaHexNAc to pNP-monosaccharides base on micro-plate reader; B: determination of hydrolyze activity to pNP-Core Ⅱ based on HPLC; C: activity of SoCaHexNAc to 2AB-NGA2. |
| 图选项 |
2.4.2 重组SoCaHexNAc对O-链寡糖及N-链寡糖标准品末端乙酰氨基葡萄糖水解活性的研究: 在37 ℃以及最适pH条件下,分别以O-链寡糖pNP-Core Ⅱ和N-链寡糖2AB-NGA2为底物,使用HPLC分别测定底物的生成量。重组SoCaHexNAc能够水解pNP-Core Ⅱ末端β 1-6连接的乙酰氨基葡萄糖,随反应时间增加,底物峰(保留时间4.2 min)不断降低,而保留时间为5.4 min的产物峰逐渐升高,当反应5 h时,pNP-Core Ⅱ基本完全反应(图 5-B)。而重组SoCaHexNAc对2AB-NGA2中β 1-4连接的乙酰氨基葡萄糖无作用,反应12 h后仍无产物峰生成(图 5-C)。3 讨论 本研究首次从细菌Solitalea canadensis克隆得到一种新的β-N-乙酰氨基己糖苷酶基因,并通过原核表达体系对其进行了重组表达,得到有活性的重组β-N-乙酰氨基己糖苷酶SoCaHexNAc。系统发育进化树显示,来源于真核和原核的11个GH20家族β-N-乙酰氨基己糖苷酶可以分为2个亚类,SoCaHexNAc与Serratia marcescens来源的β-N-乙酰氨基己糖苷酶同属一个亚类,进化关系最近,而与另一亚类的昆虫和人体来源的β-N-乙酰氨基己糖苷酶距离较远。尽管如此,SoCaHexNAc与同一亚类和不同亚类之间的β-N-乙酰氨基己糖苷酶之间的氨基酸序列多重比对仍显示存在某些氨基酸残基高度保守,如SoCaHexNAc第327位和522位的天冬氨酸、第330位精氨酸、第429位的脯氨酸、第438位的组氨酸、第520位甘氨酸、第523位的谷氨酸、第605位和709位的色氨酸,尤其是第523位的精氨酸和第524位的谷氨酸在所有11个样本中完全一致。此外,SoCaHexNAc氨基酸序列中还存在一些相对保守的区段,而根据这些区段推测出的α-螺旋和β-折叠等二级结构类型与已知晶体结构的1YHT相比,在数目和位置上都高度一致。根据已报道的GH20家族中β-N-乙酰氨基己糖苷酶晶体结构的研究[11-13]和二级结构的分析,我们推断SoCaHexNAc氨基酸序列中这些保守片段所形成的α螺旋和β折叠可能位于酶催化口袋区域,参与形成(β/α)8桶状结构,并将Asp-Glu这2个与催化相关的氨基酸残基包围在内部,构成催化活性中心。根据系统发育进化树、氨基酸序列多重比对分析以及二级结构分析结果综合判断SoCaHexNAc酶具有明显的GH20 β-N-乙酰氨基己糖苷酶家族特征,应分属于GH20家族。
重组SoCaHexNAc酶学性质研究表明该酶最适pH值为6.0,与之前报道的β-N-乙酰氨基己糖苷酶类似,都处于弱酸性至中性范围内,如细菌Aeromonas hydrophila来源的β-N-乙酰氨基己糖苷酶rNAG-B最适pH值为6.0[14],Bacillusstearothermophilus来源的β-HexNAcase最适pH处于6.5左右[15],Vibrio furnissii来源的pXE18和pXE28最适pH值7.0[16],灰翅夜蛾来源的Sfhex最适pH值5.5[17],玉米螟的OfHex1最适pH值为7.0[18]。本文所述重组酶来源于土壤细菌Solitaleacanadensis,表观最适温度为42 ℃,而该细菌培养温度为18-30 ℃[9],显然,该酶的最适温度与细菌Solitalea canadensis生长的环境温度并不一致。来源于细菌Solitalea canadensis的其他酶也有类似发现,如有研究指出来源于该菌种的甘露糖苷酶最适反应温度为37 ℃也并非其最适生长温度[19]。类似现象在其他土壤细菌中也有发现,如关于内切葡聚糖酶的报道[20]指出自淡水沼泽森林土壤中分离的芽孢杆菌Paenibacillus sp. M33中内切葡聚糖酶的最适温度为50 ℃,高于其菌体存在的环境温度。分离自我国无锡某地羊圈土壤中的细菌Brevibacillus parabrevis CGMCC 10798最适培养温度为37 ℃,该菌来源的丝氨酸蛋白酶最适温度却是60 ℃[21],由此发现重组酶的最适温度与来源菌株的生长温度并没有严格的必然关系。此外,在酶的稳定性试验中,我们发现虽然SoCaHexNAc表观最适温度为42 ℃,但其在该温度下并不稳定,37 ℃下酶的稳定性明显增强。酶的热稳定性也是制约其应用价值的重要因素,在酶的实际应用中不仅需要酶具有较高催化速率迅速催化化学反应,也希望其具有良好的稳定性可以长期保存,因此在后续的研究中我们将尝试对酶进行改造,以提高其温度的稳定性。
研究中还发现该酶会受二糖分子不同程度的影响,尤以乳糖对其的影响突出,而类似的特性在其他关于该酶的研究中未见报道。生物信息学表明GH20家族β-N-乙酰氨基己糖苷酶催化活性中心含有高度保守序列His/Asn-Xaa-Gly-Ala/Cys/ Gly/Met-Asp-Glu-Ala/Ile/Leu/Val[22],其中谷氨酸和天冬酰胺作为2个重要的催化位点,参与催化的过程。有文献指出该酶的催化机制为底物辅助催化[23],即底物2位碳原子上的乙酰氨基基团中的羰基氧作为分子内亲核试剂使之形成一个环状离子中间体,谷氨酸和天冬酰胺上的羧基作为一般酸/碱提供质子,促使糖苷键的断裂和糖基的水解。由于乳糖等二糖分子中不存在乙酰氨基,无法形成离子中间体,导致酶催化反应不能进行,但二糖分子的结构和酶的天然底物结构相似,能与底物竞争酶的催化中心,导致表观酶活性被抑制,这也与二糖分子对酶活性抑制作用的研究结果相吻合。
此外底物特异性初步研究还发现,重组SoCaHexNAc可以水解O-链寡糖核心结构Core Ⅱ末端乙酰氨基葡萄糖,而不能水解N-链糖核心结构NGA2的末端乙酰氨基葡萄糖,这可能是由于重组酶对底物有一定的选择性和专一性,CoreII末端乙酰氨基葡萄糖是以β-1, 6键连接而NGA2末端乙酰氨基葡萄糖是以β-1, 4键与其他单糖连接的,该重组酶对β-1, 6键表现活性,而不能水解β-1, 4键连接的末端乙酰氨基葡萄糖。未来我们还将进一步探索重组SoCaHexNAc的底物特异性,检测更多不同的底物,比较重组SoCaHexNAc对不同连结位点的糖链以及对O-糖链和N-糖链的活性差异。目前为止,虽有多个不同来源的β-N-乙酰氨基己糖苷酶被克隆表达,但表现出的酶学性质和底物特异性各不相同,如有的酶能水解线性壳聚寡糖却不能水解N-链寡糖,有的只能特异性水解1种或2种连接位点,这导致不同酶的应用价值和应用范围不尽相同。因此,以重组SoCaHexNAc为模板,通过重组突变,对活性中心进行改造,以期发掘出针对特定结构的寡糖外切糖苷工具酶,对糖链结构分析技术的开发和改良具有重要意义。
References
| [1] | Cabezas JA. Some comments on the type references of the official nomenclature (IUB) for β-N-acetylglucosaminidase, β-N-acetylhexosaminidase and β-N-acetylgalactosaminidase. The Biochemical Journal, 1989, 261(3): 1059-1060. |
| [2] | Slámová K, Bojarová P, Petrásková L, K?en V. β-N-Acetylhexosaminidase:what's in a name…?. Biotechnology Advances, 2010, 28(6): 682-693. DOI:10.1016/j.biotechadv.2010.04.004 |
| [3] | Landels EC, Green PM, Ellis IH, Fensom AH, Bobrow M. Beta-hexosaminidase splice site mutation has a high frequency among non-Jewish Tay-Sachs disease carriers from the British Isles. Journal of Medical Genetics, 1992, 29(8): 563-567. DOI:10.1136/jmg.29.8.563 |
| [4] | Katta S, Ankati S, Podile AR. Chitooligosaccharides are converted to N-acetylglucosamine by N-acetyl-β-hexosaminidase from Stenotrophomonas maltophilia. FEMS Microbiology Letters, 2013, 348(1): 19-25. DOI:10.1111/fml.2013.348.issue-1 |
| [5] | Yang Q, Liu T, Liu FY, Qu MB, Qian XH. A novel β-N-acetyl-D-hexosaminidase from the insect Ostrinia furnacalis (Guenée). The FEBS Journal, 2008, 275(22): 5690-5702. DOI:10.1111/j.1742-4658.2008.06695.x |
| [6] | Jin YL, Jo YY, Kim KY, Shim JH, Kim YW, Park RD. Purification and characterization of β-N-acetylhexosaminidase from rice seeds. Journal of Biochemistry and Molecular Biology, 2002, 35(3): 313-319. |
| [7] | Oikawa A, Itoh E, Ishihara A, Iwamura H. Purification and characterization of β-N-acetylhexosaminidase from maize seedlings. Journal of Plant Physiology, 2003, 160(9): 991-999. DOI:10.1078/0176-1617-01089 |
| [8] | Slámová K, Kulik N, Fiala M, Krejzová-Hofmeisterová J, Ettrich R, K?en V. Expression, characterization and homology modeling of a novel eukaryotic GH84β-N-acetylglucosaminidase from Penicillium chrysogenum. Protein Expression and Purification, 2014, 95: 204-210. DOI:10.1016/j.pep.2014.01.002 |
| [9] | Christensen P. Flexibacter canadensis sp. nov. International Journal of Systematic Bacteriology, 1980, 30(2): 429-432. DOI:10.1099/00207713-30-2-429 |
| [10] | Mahuku GS. A simple extraction method suitable for PCR-based analysis of plant, fungal, and bacterial DNA. Plant Molecular Biology Reporter, 2004, 22(1): 71-81. DOI:10.1007/BF02773351 |
| [11] | Ramasubbu N, Thomas LM, Ragunath C, Kaplan JB. Structural analysis of Dispersin B, a biofilm-releasing glycoside hydrolase from the Periodontopathogen Actinobacillus actinomycetemcomitans. Journal of Molecular Biology, 2005, 349(3): 475-486. DOI:10.1016/j.jmb.2005.03.082 |
| [12] | Mark BL, Vocadlo DJ, Knapp S, Triggs-Raine BL, Withers SG, James MNG. Crystallographic evidence for substrate-assisted catalysis in a bacterial β-hexosaminidase. The Journal of Biological Chemistry, 2001, 276(13): 10330-10337. DOI:10.1074/jbc.M011067200 |
| [13] | Langley DB, Harty DWS, Jacques NA, Hunter N, Guss JM, Collyer CA. Structure of N-acetyl-β-D-glucosaminidase (GcnA) from the endocarditis pathogen Streptococcus gordonii and its complex with the mechanism-based inhibitor NAG-thiazoline. Journal of Molecular Biology, 2008, 377(1): 104-116. DOI:10.1016/j.jmb.2007.09.028 |
| [14] | Lan XQ, Zhang X, Kodaira R, Zhou ZY, Shimosaka M. Gene cloning, expression, and characterization of a second β-N-Acetylglucosaminidase from the chitinolytic bacterium Aeromonas hydrophila Strain SUWA-9. Bioscience, Biotechnology, and Biochemistry, 2008, 72(2): 492-498. DOI:10.1271/bbb.70573 |
| [15] | Sakai K, Narihara M, Kasama Y, Wakayama M, Moriguchi M. Purification and characterization of thermostable β-N-acetylhexosaminidase of Bacillus stearothermophilus CH-4 isolated from Chitin-containing compost. Applied and Environmental Microbiology, 1994, 60(8): 2911-2915. |
| [16] | Chitlaru E, Roseman S. Molecular cloning and characterization of a novel β-N-acetyl-D-glucosaminidase from Vibrio furnissii. The Journal of Biological Chemistry, 1996, 271(52): 33433-33439. DOI:10.1074/jbc.271.52.33433 |
| [17] | Tomiya N, Narang S, Park J, Abdul-Tahman B, Choi O, Singh S, Hiratake J, Sakata K, Betenbaugh MJ, Palter KB, Lee YC. Purification, characterization, and cloning of a Spodoptera frugiperda Sf9β-N-acetylhexosaminidase that hydrolyzes terminal N-acetylglucosamine on the N-glycan core. The Journal of Biological Chemistry, 2006, 281(28): 19545-19560. DOI:10.1074/jbc.M603312200 |
| [18] | Liu T, Liu FY, Yang Q, Yang J. Expression, purification and characterization of the chitinolytic β-N-acetyl-D-hexosaminidase from the insect Ostrinia furnacalis. Protein Expression and Purification, 2009, 68(1): 99-103. DOI:10.1016/j.pep.2009.06.004 |
| [19] | Liu FF, Kulinich A, Du YM, Liu L, Voglmeir J. Sequential processing of mannose-containing glycans by two α-mannosidases from Solitalea canadensis. Glycoconjugate Journal, 2016, 33(2): 159-168. DOI:10.1007/s10719-016-9651-9 |
| [20] | Kanchanadumkerng P, Sakka M, Sakka K, Wiwat C. Characterization of endoglucanase from Paenibacillus sp. M33, a novel isolate from a freshwater swamp forest. Journal of Basic Microbiology, 2017, 57(2): 121-131. DOI:10.1002/jobm.201600225 |
| [21] | Zhang RX, Gong JS, Su C, Zhang DD, Tian H, Dou WF, Li H, Shi JS, Xu ZH. Biochemical characterization of a novel surfactant-stable serine keratinase with no collagenase activity from Brevibacillus parabrevis CGMCC 10798. International Journal of Biological Macromolecules, 2016, 93: 843-851. DOI:10.1016/j.ijbiomac.2016.09.063 |
| [22] | Gutternigg M, Kretschmer-Lubich D, Paschinger K, Rendi? D, Hader J, Geier P, Ranftl R, Jantsch V, Lochnit G, Wilson IBH. Biosynthesis of Truncated N-linked oligosaccharides results from non-orthologous hexosaminidase-mediated mechanisms in nematodes, plants, and insects. The Journal of Biological Chemistry, 2007, 282(38): 27825-27840. DOI:10.1074/jbc.M704235200 |
| [23] | Mark BL, James MNG. Anchimeric assistance in hexosaminidases. Canadian Journal of Chemistry, 2002, 80(8): 1064-1074. DOI:10.1139/v02-130 |
