陈亚迎1, 刘壮1, 吕朋安1, 申军士1,2

 , 朱伟云1
, 朱伟云1 1.江苏省消化道营养与动物健康重点实验室, 南京农业大学消化道微生物研究室, 江苏 南京 210095;
2.俄亥俄州立大学动物科学系, 美国俄亥俄州 哥伦布 43210
收稿日期:2016-12-29;修回日期:2017-02-22;网络出版日期:2017-02-28
基金项目:国家自然科学基金青年基金(31402101);江苏省自然科学基金青年基金(BK20140696);中央高校基本科研业务费专项资金(Y020150023)
*通信作者:Tel:+86-25-84395523;E-mail:shenjunshi@njau.edu.cn
摘要: [目的]通过体外静态模拟瘤胃发酵法研究溶菌酶对瘤胃发酵、甲烷生成及微生物菌群结构的影响。[方法]采用单因素多水平试验设计,溶菌酶添加水平分别为0(L-0,对照组)、0.1 mg/100 mL(L-0.1)、1 mg/100 mL(L-1)、10 mg/100 mL(L-10)和100 mg/100 mL(L-100),定时测定产气量和甲烷产量,培养24 h后,发酵液用于发酵参数和微生物菌群数量的qPCR测定,其中L-0、L-1和L-100三个组发酵液同时进行16S rRNA基因Illumina高通量测序。[结果]与对照组相比,低剂量溶菌酶添加(L-0.1组)不影响甲烷产量、氨氮浓度、干物质消失率、有机物消失率和总挥发性脂肪酸等瘤胃发酵参数(P>0.05);随着剂量提高,L-1处理组甲烷产量、氨氮浓度显著降低(P<0.05),丙酸浓度显著增加(P<0.05),并且干物质消失率、有机物消失率和总挥发性脂肪酸不受影响(P>0.05);而较高剂量组(L-10和L-100组)虽然甲烷产量显著降低,丙酸浓度显著增加(P<0.05),但干物质消失率和有机物消失率也显著降低(P<0.05)。qPCR结果显示高剂量组(L-100组)总菌、原虫、甲烷菌数量与对照组相比显著降低(P<0.05),而L-0.1、L-1和L-10组总菌、真菌和原虫数量与对照组相比均无显著变化(P>0.05)。高通量测序主成分分析(PCA)显示对照组与溶菌酶添加组间瘤胃细菌组成的明显区分,说明添加溶菌酶显著改变了瘤胃细菌菌群结构。溶菌酶通过增加月形单胞菌和琥珀酸弧菌等丙酸生成菌的相对丰度,使更多的氢被用于生成丙酸,导致甲烷产量降低;溶菌酶可抑制普雷沃氏菌和拟杆菌属等蛋白降解菌的生长,进而减少蛋白质过度降解,降低氨氮浓度。[结论]添加适宜浓度(1 mg/100 mL)的溶菌酶可通过调控瘤胃微生态改变瘤胃发酵模式,降低瘤胃甲烷和氨的生成,短期内并不影响饲料消化。
关键词: 溶菌酶 体外发酵 甲烷 发酵参数 菌群结构
Effects of lysozyme on in vitro fermentation, methanogenesis and microbial community structure of the rumen
Yaying Chen1, Zhuang Liu1, Peng'an Lv1, Junshi Shen1,2

 , Weiyun Zhu1
, Weiyun Zhu1 1.Laboratory of Gastrointestinal Microbiology, Jiangsu Key Laboratory of Gastrointestinal Nutrition and Animal Health, College of Animal Science and Technology, Nanjing Agricultural University, Nanjing 210095, Jiangsu Province, China;
2.Department of Animal Sciences, The Ohio State University, Columbus, OH 43210, USA
Received 29 December 2016; Revised 22 February 2017; Published online 28 February 2017
*Corresponding author: Tel: +86-25-84395523; E-mail: shenjunshi@njau.edu.cn
Supported by the National Natural Science Foundation of China for the Youth (31402101), by the Jiangsu Provincial Natural Science Foundation for the Youth (BK20140696) and by the Fundamental Research Funds for the Central Universities (Y020150023)
Abstract: [Objective]We studied the effects of lysozyme on in vitro rumen fermentation, methanogenesis and microbial community structure.[Methods]Lysozyme was added to in vitro ruminal cultures at 5 doses: with 0 (L-0, Control), 0.1 mg/100 mL (L-0.1), 1 mg/100 mL (L-1), 10 mg/100 mL (L-10), and 100 mg/100 mL (L-100). Total gas and methane production were measured at different time of incubation. Culture samples were collected at 24 h for analysis of fermentation parameters and functional microbial populations. In addition, samples of L-0, L-1, and L-100 collected at 24 h were also used subjected to metagenomics analysis of bacterial community using Illumina sequencing of 16S rRNA gene amplicons.[Results]Compared with control, methane production, ammonia concentration, dry matter digestibility, organic matter digestibility and total volatile fatty acid concentration were not influenced by L-0.1 (P>0.05). Methane production and NH3-N concentration were reduced, and propionate concentration was increased by L-1 (P < 0.05), whereas dry matter digestibility, organic matter digestibility or total volatile fatty acid were not affected (P>0.05). Methane production was reduced, and propionate concentration was increased by L-10 and L-100 (P < 0.05), but dry matter digestibility and organic matter digestibility were also decreased significantly (P < 0.05). The qPCR results showed that total bacteria, fungi and methanogens were significantly reduced by L-100 (P < 0.05), but were not influenced (P>0.05) by L-0.1, L-1, or L-10. Principal Component Analysis of the sequencing data showed clear differences in the composition of the ruminal bacterial community between the control and the lysozyme treatments, demonstrating evident impact of the lysozyme addition. The abundance of propionate-producing bacteria (e.g., Selenomonas and Succinivibrio) was increased by lysozyme, resulting in more hydrogen being directed to production of propionate instead of methane. Moreover, the reduced ammonia concentration in L-1 was probably due to the lower abundance of proteolytic bacteria (e.g., Prevotella and Bacteroides) inhibited by lysozyme.[Conclusion]Appropriate lysozyme addition (1 mg/100 mL) can be used to modulate ruminal microbial ecology and reduce methanogenesis and ammoniagenesis by rumen microbiome without adversely affecting feed digestion or fermentation in short-term.s
Key words: lysozyme in vitro fermentation methane fermentation parameter microbial community structure
溶菌酶 (Lysozyme) 是一种稳定的碱性蛋白酶,可通过破坏细菌细胞壁肽聚糖中N-乙酰胞壁酸和N-乙酰氨基葡萄糖之间的β-1, 4糖苷键,引起细菌裂解[1]。溶菌酶在人、动物、植物及各种微生物中广泛存在,是生物体内重要的非特异性免疫因子。研究发现在饲料中添加溶菌酶具有替代抗生素的效果,能提高猪和禽类的生产性能[2-3],用含人乳溶菌酶基因的转基因奶山羊羊乳饲喂仔猪和羔羊,可增加羔羊日增重,调节仔猪小肠微生物菌群[4];断奶仔猪补充饲喂溶菌酶可改善肠道形态并提高肠道免疫水平[5]。然而,国内外对溶菌酶应用的研究工作主要集中于单胃动物中,对反刍动物瘤胃功能的影响尚不清楚。在瘤胃中,革兰氏阴性菌主要产生丙酸和琥珀酸等,而大部分革兰氏阳性菌会产生氢气、CO2和甲酸等甲烷前体物,有部分革兰氏阳性菌还有高效脱氨基产氨功能,过快地产氨而不能被瘤胃及时利用致使氮源的浪费[6]。早期研究发现溶菌酶对革兰氏阳性菌有显著抑制效果,而对其他菌几乎无影响[1]。因此,我们推测溶菌酶可以在不影响瘤胃发酵的情况下抑制甲烷和过多氨氮的产生,从而提高瘤胃对能量和氮元素的利用。本试验旨在通过体外静态模拟瘤胃发酵法研究溶菌酶对瘤胃发酵、甲烷生成及微生物菌群结构的影响,初步探讨溶菌酶对瘤胃发酵调控的微生物机制。
1 材料和方法 1.1 试验材料 试验所用溶菌酶提取自鸡蛋清,购自合肥博美生物科技有限责任公司,由Amresco进口分装;活性:≥20000 U/mg;CAS:12650-88-3。发酵底物为0.5 g混合精料 (玉米68%、豆粕32%) 和0.5 g粉碎羊草。
1.2 试验设计 试验采用单因素多水平试验设计,分为对照组和4个试验组。对照组不添加任何添加剂;溶菌酶试验组添加水平分别为0.1 mg/100 mL (L-0.1)、1 mg/100 mL (L-1)、10 mg/100 mL (L-10) 和100 mg/100 mL (L-100)。每个处理4个重复且均测定相应指标。采用波杂山羊为瘤胃液供体,进行体外发酵。
1.3 瘤胃液接种及体外培养 瘤胃液取自南京农业大学动物房3头成年波杂公山羊[体重 (25±2) kg],日粮精粗比为50:50[精料补充料500 g (玉米65%、豆粕26%、麦麸5%、NaCl 1%、NaHCO3 1%、CaHPO4 1%、预混料1%),羊草与苜蓿干草各250 g],每日8:00和17:00等量饲喂,自由饮水。按上述日粮饲喂10 d后口腔采集瘤胃液,采用申军士等[7]方法将采集的3头成年波杂公山羊瘤胃液混匀后,过滤备用。参照Theodorou等[8]方法配制缓冲液,pH调至7.00。厌氧条件下按体积比10:90的比例将过滤的瘤胃液与缓冲液充分混合,并按每瓶100 mL混合液厌氧分装至180 mL的发酵瓶,置于39 ℃条件下培养24 h。
1.4 指标测定 发酵过程中动态测定0、3、6、9、12、24 h产气量与甲烷产量。参照Theodorou等[8]方法测定产气量;参考Wang等[9]和Yang等[10]描述的条件使用气相色谱仪 (GC-2014,Shimadzu,Japan) 测定甲烷浓度,并结合产气量计算出甲烷产量;发酵24 h后立即用pH计 (Ecoscan pH 5,Singapore) 测pH。随后冰浴发酵瓶终止发酵,采集发酵液样品,冻存于-20 ℃冰箱。待测样品参照Weatherburn[11]方法测定氨态氮 (NH3-N);参考申军士等[7]方法测定挥发性脂肪酸 (VFA);参照马艳艳等[12]方法测定干物质 (DMD) 及有机物质消失率 (OMD)。
1.5 微生物菌群数量及细菌多样性分析 1.5.1?发酵液DNA提取:1mL发酵液样品解冻后在9000×g离心力、4 ℃离心5 min,弃上清加入1 mL CTAB (hexadecyltrimethylammonium bromide),十六烷基三甲基溴化铵) 转移至锆珠管中,利用Bead Beat提取瘤胃微生物总DNA[13-14]。提取的发酵液微生物DNA使用Nanodrop 2000分光光度计 (Thermo Fisher Scientific, Inc., Madison, Wisconsin, USA) 测定DNA浓度 (ng/μL),样品260 nm与280 nm OD比值在1.8-2.0,-20 ℃保存备用。
1.5.2?Real-time PCR定量菌群:参考申军士等[7]方法构建20 μL反应体系,使用ABI7500 Real-time PCR仪对瘤胃发酵液中总菌、真菌、原虫、甲烷菌、硫还原菌 (Sulfate-reducing bacteria,SRB) 进行定量分析。分别以总菌的16S rRNA基因,以真菌和原虫的18S rRNA基因,以甲烷菌的甲基辅酶M还原酶 (mcrA) 基因和硫还原菌的亚硫酸还原酶 (dsrA) 基因作为模板制作各目标菌定量的标准曲线。Real-time PCR引物序列见表 1。
表 1. 瘤胃目标微生物实时定量PCR引物序列 Table 1. Primers used for real-time PCR quantification of rumen target organisms
| Target organisms | Primers | Sequences (5′→3′) | Annealing temp./℃ | References |
| Total bacteria | 1114F | CGGCAACGAGCGCAACCC | 60 | [15] |
| 1275R | CCATTGTAGCACGTGTGTAGCC | |||
| Fungi | Fungi-F | GAGGAAGTAAAAGTCGTAACAAGGTTTC | 60 | [15] |
| Fungi-R | CAAATTCACAAAGGGTAGGATGATT | |||
| Protozoa | Prot316F | GCTTTCCGWTGGTAGTGTATT | 54 | [16] |
| Prot539R | CTTGCCCTCYAATCGTWCT | |||
| Methanogens | McrA-F | TTCGGTGGATCDCARAGRGC | 60 | [17] |
| McrA-R | GBARGTCGWAWCCGTAGAATCC | |||
| Sulfate-reducing | DsrA-F | CCAACATGCACGGYTCCA | 60 | [18] |
| bacteria | DsrA-R | CGTCGAACTTGAACTTGAACTTGTAGG |
表选项
1.5.3?瘤胃细菌MiSeq高通量测序与分析:以样品总DNA为模板,用细菌通用引物16S rDNA的V3-V4区域作为目标DNA序列,用引物338F (5′-ACTCCTRCGGGAGGCAGCAG-3′) 和806R (5′-GGACTACCVGGGTATCTAAT-3′) 进行PCR扩增;PCR过程在ABI Gene Amp? 9700上进行,采用20 μL反应体系,包括0.4 μL FastPfu Polymerase (北京全式金生物技术有限公司),4 μL 5 × FastPfu buffer,正反向引物 (5 μmol/L) 各0.4 μL,2 μL dNTPs (2.5 mmol/L),总DNA模板10 ng,补ddH2O至20 μL。PCR反应扩增条件为95 ℃预变性2 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,25个循环;72 ℃延伸20 min。扩增完成后PCR产物用含溴化乙锭的2%琼脂糖凝胶电泳鉴定并用PCR纯化试剂盒 (Axygen,美国) 纯化和回收,回收的PCR产物用QuantiFlourTM-ST fluorometer[普洛麦格 (北京) 生物技术有限公司)]定量,并按测序要求进行混合。然后送至上海美吉生物医药科技有限公司,使用Illumina MiSeq PE300测序平台完成高通量测序。使用软件FLASH将MiSeq测序得到的paired-end数据切除原始序列两端的测序接头后进行片段拼接[19],然后根据引物barcode信息将序列分成不同的文库对序列质量进行质控和过滤。在Usearch软件中 (version 7.1) 对序列进行归类操作,选出与OTU代表序列相似性在97 %以上的序列,生成OTU表格;通过QIIME软件平台在16S rRNA Silva细菌比对数据库中 (Silva version 119) [20]对OTU进行物种分类注释。对各个样品进行α多样性分析,计算样品间丰度指数 (Ace、Chao1)、多样性指数 (Shannon、Simpson) 及各样本文库覆盖率 (Coverage)。使用Canoco软件 (version 4.5) 进行主成分分析 (PCA) 和作图。
1.6 数据分析 微生物定量数据经StepOneTM Software v2.2.2初步计算,与其余各试验数据经Excel整理后,利用SAS 9.2广义线性模型 (GLM) 进行方差分析和显著性分析,采用Duncan法进行多重比较。显著性水平为P < 0.05。
2 结果和分析 2.1 不同溶菌酶水平下产气量和甲烷产量的变化 2.1.1?产气量变化:由表 2可得,与对照组相比,溶菌酶试验组随着溶菌酶剂量增加总产气量逐渐降低,且L-10和L-100总产气量与L-0相比均显著降低 (P < 0.05)。
2.1.2?甲烷产量变化:由表 2可知,体外培养过程中,与对照组相比,添加不同剂量溶菌酶均可抑制甲烷产生,其中L-1、L-10和L-100可显著降低甲烷产量 (P < 0.05),并呈显著的剂量效应 (P < 0.05)。与对照组相比,L-0.1、L-1、L-10和L-100组甲烷产量分别减少了3.43%、8.58%、16.74%和48.50%。
2.2 不同溶菌酶水平下瘤胃发酵参数的变化 2.2.1?pH值变化:由表 2可知,体外培养24 h后,所有组pH值均在正常范围内,与对照组相比,L-0.1和L-100组的pH值无显著影响 (P > 0.05),但L-1和L-10组pH值显著降低 (P < 0.05)。
表 2. 溶菌酶对体外发酵24 h总产气量、甲烷产量、pH、氨氮浓度、干物质和有机物消化率的影响 Table 2. Effects of lysozyme on total gas and methane production, pH, ammonia concentration, DM and OM digestibility in the in vitro rumen mixed cultures at 24 h
| Items | Different supplement level/(mg/100 mL) | SEM | P-value | ||||||
| 0 | 0.1 | 1 | 10 | 100 | Trt | Linear | Quadratic | ||
| Total gas/mL | 182.1a | 182.0a | 178.1a | 170.9b | 133.7c | 1.58 | < 0.01 | < 0.01 | 0.01 |
| Methane/mL | 23.3a | 22.5a | 21.3b | 19.4c | 12.0d | 0.33 | < 0.01 | < 0.01 | < 0.01 |
| pH | 6.68a | 6.69a | 6.64b | 6.65b | 6.69a | 0.01 | < 0.01 | < 0.01 | < 0.01 |
| NH3-N/(mg/dL) | 14.5bc | 14.2c | 13.7d | 15.0ab | 15.1a | 0.18 | < 0.01 | < 0.01 | < 0.01 |
| DM digestibility/% | 54.1a | 54.6a | 54.2a | 52.2b | 41.9c | 0.27 | < 0.01 | < 0.01 | 0.01 |
| OM digestibility/% | 56.0a | 56.0a | 55.5a | 53.3b | 42.8c | 0.309 | < 0.01 | < 0.01 | < 0.01 |
| L-0: no additives; L-0.1: lysozyme 0.1 mg/100 mL; L-1: lysozyme 1 mg/100 mL; L-10: lysozyme 10 mg/100 mL; L-100: lysozyme 100 mg/100 mL; SEM: standard error means; a-d Means within a row with different superscripts differ (P < 0.05). The same as follows. | |||||||||
表选项
2.2.2?干物质 (DMD) 及有机物消失率 (OMD) 变化:由表 2可知,与对照组相比,L-0.1和L-1组DMD及OMD均无显著变化 (P > 0.05),但L-10和L-100组DMD及OMD显著降低 (P < 0.05)。
2.2.3?氨态氮浓度 (NH3-N) 变化:由表 2可知,L-1组氨态氮浓度显著低于对照组 (P < 0.05),L-100组氨态氮浓度显著高于对照组(P < 0.05),其他溶菌酶组与对照组无显著差异。
2.2.4?挥发性脂肪酸 (VFA) 浓度变化:由表 3可知,体外发酵24 h后,与对照组相比,L-100组总VFA浓度显著降低 (P < 0.05),而L-0.1、L-1和L-10组总VFA浓度均无显著差异 (P > 0.05);各溶菌酶试验组乙酸浓度随溶菌酶添加水平的提高而先升高后降低,且L-1组显著高于对照组 (P < 0.05),L-100组显著低于对照组 (P < 0.05);各试验组丙酸浓度均随溶菌酶添加水平的提高而显著增加 (P < 0.05),乙丙比显著降低 (P < 0.05),且均呈显著的剂量效应 (P < 0.05);各试验组的丁酸和戊酸的浓度均显著低于对照组 (P < 0.05),并且也呈现显著的剂量效应 (P < 0.05)。此外,L-100组显著降低了总支链挥发性脂肪酸 (BCVFA) 和异戊酸浓度 (P < 0.05)。
表 3. 溶菌酶对体外发酵24 h挥发性脂肪酸的影响 Table 3. Effect of lysozyme on volatile fatty acid concentration in the in vitro rumen mixed cultures at 24 h
| Items | Different supplement level/(mg/100 mL) | SEM | P-value | ||||||
| 0 | 0.1 | 1 | 10 | 100 | Trt | Linear | Quadratic | ||
| Total VFA/(mmol/L) | 64.8a | 65.4a | 67.0a | 64.1a | 56.3b | 1.020 | < 0.01 | < 0.01 | 0.60 |
| Acetate/(mmol/L) | 38.4b | 39.3ab | 40.5a | 37.9b | 30.9c | 0.590 | < 0.01 | < 0.01 | 0.49 |
| Propionate/(mmol/L) | 15.9c | 16.6c | 18.8b | 20.2a | 21.0a | 0.330 | < 0.01 | < 0.01 | < 0.01 |
| Butyrate/(mmol/L) | 8.32a | 7.49b | 5.48c | 4.32d | 3.42e | 0.170 | < 0.01 | < 0.01 | < 0.01 |
| Isobutyrate/(mmol/L) | 0.70a | 0.62a | 0.75a | 0.73a | 0.40b | 0.065 | 0.01 | < 0.01 | 0.35 |
| Valerate/(mmol/L) | 0.49a | 0.38b | 0.32bc | 0.28c | 0.14d | 0.026 | < 0.01 | < 0.01 | < 0.01 |
| Isovalerate/(mmol/L) | 1.16a | 1.17a | 1.13a | 0.62b | 0.40c | 0.016 | < 0.01 | < 0.01 | < 0.01 |
| Acetate/Propionate | 2.42a | 2.36b | 2.15c | 1.88d | 1.47e | 0.013 | < 0.01 | < 0.01 | < 0.01 |
| Total BCVFA/(mmol/L) | 1.86a | 1.78a | 1.87a | 1.34b | 0.80c | 0.073 | < 0.01 | < 0.01 | < 0.01 |
表选项
2.3 发酵液微生物变化 2.3.1?不同溶菌酶水平下功能菌群数量的变化:由表 4可知,与对照组相比,L-100组总菌数量显著降低 (P < 0.05),其余试验组中总菌数量均无显著变化 (P > 0.05);真菌不受溶菌酶添加剂量影响 (P > 0.05);L-100组中原虫、甲烷菌和硫还原菌数量显著低于对照组 (P < 0.05),而L-0.1、L-1和L-10组原虫、甲烷菌和硫还原菌 (SRB) 数量与对照组相比均无显著变化 (P > 0.05)。
表 4. 溶菌酶对体外发酵功能菌群数量的影响 Table 4. Effects of lysozyme on microbial populations (log copy numbers/mL) in the in vitro rumen mixed cultures
| Items | Different supplement level/(mg/100 mL) | SEM | P-value | ||||||
| 0 | 0.1 | 1 | 10 | 100 | Trt | Linear | Quadratic | ||
| Total bacteria | 12.30a | 12.30a | 12.31a | 12.33a | 11.77b | 0.031 | < 0.01 | < 0.01 | 0.05 |
| Fungi | 7.73 | 7.82 | 7.89 | 7.91 | 7.84 | 0.083 | 0.62 | 0.85 | 0.32 |
| Protozoa | 10.33 | 10.36 | 10.37 | 10.40 | 10.21 | 0.043 | 0.06 | 0.01 | 0.22 |
| Methanogens | 8.81a | 8.77a | 8.82a | 8.80a | 8.65b | 0.023 | < 0.01 | < 0.01 | 0.63 |
| SRB | 8.27 | 8.29 | 8.30 | 8.35 | 8.23 | 0.026 | 0.056 | 0.02 | 0.04 |
| SRB: sulfate-reducing bacteria | |||||||||
表选项
2.3.2?发酵液微生物区系变化:高通量测序结果表明各组细菌平均有效序列数超过25171条,有效序列平均长度为437 bp,各组物种覆盖率均在99.7%以上,说明本次测序结果能代表样本中菌群的真实情况。表 5显示L-1组发酵液中的微生物
表 5. 溶菌酶对体外发酵液细菌菌群丰度和多样性的影响 Table 5. Effects of lysozyme on in vitro fermentation bacterial richness and diversity
| Items | Value | SEM | P-value | ||||
| L-0 | L-1 | L-100 | Trt | Linear | Quadratic | ||
| OTU | 625.8a | 606.8b | 275.0c | 4.930 | < 0.01 | < 0.01 | 0.053 |
| Ace | 654.3a | 646.5a | 333.3b | 5.180 | < 0.01 | < 0.01 | 0.55 |
| Chao1 | 658.8a | 651.5a | 336.8b | 6.420 | < 0.01 | < 0.01 | 0.67 |
| Shannon index | 4.83a | 4.48b | 2.64c | 0.062 | < 0.01 | < 0.01 | < 0.01 |
| Simpson | 0.024c | 0.044b | 0.219a | 0.005 | < 0.01 | < 0.01 | 0.02 |
| Coverage | 0.998 | 0.998 | 0.997 | 0.000 | 0.32 | 0.20 | 0.44 |
| 丰度指数Ace指数和Chao1指数与对照组相比无显著变化 (P > 0.05),而高剂量L-100组比对照组显著降低 (P < 0.01);表示微生物多样性指数的Shannon指数随溶菌酶剂量的增加显著降低,Simpson指数则显著升高 (P < 0.01)。 | |||||||
表选项
由图 1可看出,Illumina高通量测序细菌16S rRNA基因主成分分析 (PCA) 结果显示不同处理组间瘤胃细菌组成呈组别聚集,且被明显地区分,说明溶菌酶添加剂量显著影响细菌菌群结构组成。
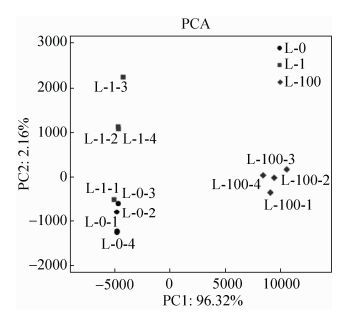 |
| 图 1. 体外发酵液细菌菌群主成分分析 Figure 1. Principal component analysis (PCA) of in vitro fermentation bacterial communities. |
| 图选项 |
高通量测序共检测到18个门,87个属。由表 6可知,其中变形菌门 (Proteobacteria)、拟杆菌门 (Bacteroidetes) 和厚壁菌门 (Firmicutes) 是主要的优势门类,占总菌数量的88.4%以上。变形菌门相对丰度随溶菌酶添加水平的提高而显著增加 (P < 0.05),拟杆菌门相对丰度则随溶菌酶添加水平的提高而显著降低 (P < 0.05),且两者均呈现显著剂量效应 (P < 0.05);而厚壁菌门相对丰度只在L-100组中显著降低 (P < 0.05)。除此之外,含量较少的纤维杆菌门 (Fibrobacteres) 随溶菌酶添加而显著降低 (P < 0.05),梭杆菌门 (Fusobacteria)、软壁菌门 (Tenericutes) 在L-100组中显著增加 (P < 0.05)。溶菌酶添加剂量对互养菌门 (Synergistetes)、螺旋体门 (Spirochaetae) 和黏胶球形菌门 (Lentisphaerae) 等相对丰度较低的门类无显著影响 (P > 0.05)。
表 6. 溶菌酶对体外发酵液细菌菌群相对丰度的影响 Table 6. Effects of lysozyme on relative abundance (%) of in vitro fermentation bacterial communities at phylum and genus level (accounted for≥0.5% of total sequences each in at least one treatment)
| Phylum | Genus/other | Relative abundance/% | SEM | P-value | ||||
| L-0 | L-1 | L-100 | Trt | Linear | Quadratic | |||
| Proteobacteria | Total | 24.20c | 36.90b | 53.70a | 1.610 | < 0.01 | < 0.01 | < 0.01 |
| Ruminobacter | 12.56b | 21.59a | 3.03c | 1.036 | < 0.01 | < 0.01 | < 0.01 | |
| Succinivibrio | 10.15b | 11.72b | 50.17a | 0.654 | < 0.01 | < 0.01 | 0.23 | |
| Unclassified Succinivibrionaceae | 0.90a | 3.05a | 0.05b | 0.097 | < 0.01 | < 0.01 | < 0.01 | |
| Bacteroidetes | Total | 41.60a | 32.70b | 22.50c | 2.130 | < 0.01 | < 0.01 | 0.02 |
| Prevotella | 14.30a | 9.67ab | 4.54b | 1.824 | 0.01 | < 0.01 | 0.11 | |
| Bacteroides | 1.62a | 0.88b | 0.05c | 0.173 | < 0.01 | < 0.01 | 0.02 | |
| Unclassified Rikenellaceae | 9.72b | 8.67b | 15.69a | 1.428 | 0.02 | < 0.01 | 0.60 | |
| Unclassified Bacteroidales | 8.45a | 7.57a | 1.90b | 0.756 | < 0.01 | < 0.01 | 0.46 | |
| Unclassified Peptococcaceae | 7.32a | 5.71a | 0.29b | 0.718 | < 0.01 | < 0.01 | 0.16 | |
| Firmicutes | Total | 27.50a | 25.80a | 12.20b | 1.830 | < 0.01 | < 0.01 | 0.55 |
| Pseudobutyrivibrio | 3.34a | 2.60b | 0.26c | 0.136 | < 0.01 | < 0.01 | < 0.01 | |
| Ruminococcus | 3.26a | 4.00a | 0.56b | 0.476 | < 0.01 | < 0.01 | 0.28 | |
| Butyrivibrio | 2.16a | 2.43a | 0.01b | 0.248 | < 0.01 | < 0.01 | 0.43 | |
| Succiniclasticum | 0.96b | 0.88b | 3.37a | 0.337 | < 0.01 | < 0.01 | 0.83 | |
| Saccharofermentans | 1.08a | 1.04a | 0.00b | 0.060 | < 0.01 | < 0.01 | 0.76 | |
| Selenomonas | 0.18b | 0.23b | 3.66a | 0.224 | < 0.01 | < 0.01 | 0.96 | |
| Anaerovibrio | 0.13b | 0.13b | 0.58a | 0.019 | < 0.01 | < 0.01 | 0.80 | |
| Unclassified Christensenellaceae | 5.15a | 4.02b | 0.26c | 0.334 | < 0.01 | < 0.01 | 0.05 | |
| Unclassified Lachnospiraceae | 4.46a | 4.25a | 0.06b | 0.314 | < 0.01 | < 0.01 | 0.71 | |
| Unclassified Ruminococcaceae | 3.51a | 3.23a | 0.10b | 0.231 | < 0.01 | < 0.01 | 0.47 | |
| Unclassified Clostridiales | 0.80a | 0.72a | 0.30b | 0.049 | < 0.01 | < 0.01 | 0.30 | |
| Unclassified Veillonellaceae | 0.43b | 0.32b | 2.47a | 0.190 | < 0.01 | < 0.01 | 0.64 | |
| Fibrobacteres | Total | 2.92a | 1.83b | 0.00c | 0.319 | < 0.01 | < 0.01 | 0.04 |
| Fibrobacter | 2.92a | 1.83b | 0.00c | 0.319 | < 0.01 | < 0.01 | 0.04 | |
| Synergistetes | Total | 1.44 | 1.17 | 2.10 | 0.305 | 0.14 | 0.06 | 0.54 |
| Unclassified Synergistaceae | 0.95 | 0.80 | 1.25 | 0.206 | 0.33 | 0.17 | 0.59 | |
| Pyramidobacter | 0.32b | 0.24b | 0.75a | 0.077 | < 0.01 | < 0.01 | 0.45 | |
| Lentisphaerae | Total | 0.88 | 0.46 | 0.45 | 0.118 | 0.05 | 0.15 | 0.03 |
| Unclassified Lentisphaerae | 0.75a | 0.33b | 0.35b | 0.104 | 0.03 | 0.16 | 0.02 | |
| Tenericutes | Total | 0.32b | 0.35b | 1.38a | 0.142 | < 0.01 | < 0.01 | 0.93 |
| Anaeroplasma | 0.20b | 0.26b | 1.30a | 0.133 | < 0.01 | < 0.01 | 0.81 | |
| Fusobacteria | Total | 0.01b | 0.01b | 6.28a | 0.412 | < 0.01 | < 0.01 | 0.92 |
| Fusobacterium | 0.01b | 0.01b | 6.28a | 0.412 | < 0.01 | < 0.01 | 0.92 | |
表选项
由表 6得,相对丰度大于0.5%的26种属类中,普雷沃氏菌属 (Prevotella) 相对丰度随溶菌酶剂量提高而降低,L-100组相对于对照组L-0显著降低 (P < 0.05);与对照组相比,L-1组瘤胃杆菌 (Ruminobacter) 菌属相对丰度显著增加,而L-100组显著降低 (P < 0.05);琥珀酸弧菌属 (Succinivibrio) 相对丰度随溶菌酶添加水平的提高而显著增加 (P < 0.05);随溶菌酶剂量增加,丝状杆菌属 (Fibrobacter)、拟杆菌属 (Bacteroides)、假丁酸弧菌属 (Pseudobutyrivibrio) 和Unclassified Christensenellaceae等4个菌属的相对丰度均显著降低,并呈现显著的剂量效应 (P < 0.05)。瘤胃球菌属 (Ruminococcus)、丁酸弧菌属 (Butyrivibrios)、Saccharofermentans、未分类消化球菌科 (Unclassified Peptococcaceae)、未分类拟杆菌目 (Unclassified Bacteroidales)、未分类毛螺菌科 (Unclassified Lachnospiraceae)、未分类瘤胃菌科 (Unclassified Ruminococcaceae) 和Unclassified Clostridiale等8个菌属的相对丰度在L-100组中显著降低 (P < 0.05);未分类理研菌科 (Unclassified Rikenellaceae)、Succiniclasticum、未分类韦荣氏球菌科 (Unclassified Veillonellaceae)、Pyramidobacter、月形单胞菌属 (Selenomonas)、厌氧支原体属 (Anaeroplasma)、厌氧弧菌属 (Anaerovibrio) 和梭形杆菌属 (Fusobacterium) 等8个菌属相对丰度在L-100组中显著升高 (P < 0.05)。
3 讨论 饲料在瘤胃微生物作用下,被降解产生甲烷、CO2和H2等气体。研究表明,体外培养时产气量与饲料OMD呈高度正相关性,产气量随着饲料发酵程度的提高而增加[21]。本研究发现,L-10和L-100组虽然能显著降低甲烷产量,但总产气量、DMD和OMD也会受到抑制,这种变化与瘤胃菌群丰富度和多样性的变化密切相关。瘤胃中主要的纤维降解菌多属于革兰氏阳性菌,如瘤胃球菌 (Ruminococcus)、丁酸弧菌 (Butyvibrio)、假丁酸弧菌 (Pseudobutyrivibrio)。此外,革兰氏阴性菌丝状杆菌 (Fibrobacter) 和普雷沃氏菌 (Prevotella) 等对纤维的降解也起着重要作用[22-23]。本试验高通量测序结果表明,L-100组抑制了上述5类最主要的纤维分解菌,而L-1组无此抑制效应,说明高剂量溶菌酶可能是通过裂解纤维分解菌,使纤维分解菌数量下降进而导致总产气量、DMD和OMD降低。
反刍动物排放的甲烷主要由甲烷菌产生,甲烷产量与甲烷菌数量密切相关。本研究qPCR结果显示,低剂量溶菌酶并不抑制甲烷菌,说明甲烷产量的减少是由于溶菌酶改变了甲烷前体物的代谢途径。研究发现,饲料降解生成的H2与CO2易被甲烷菌利用合成甲烷,此途径产生的甲烷可占瘤胃甲烷总产量的80%[24];生成甲烷是瘤胃主要的排H2途径,其能缓解瘤胃过高的氢分压,维持动态平衡[25],因此,对H2的有效利用可在抑制甲烷方面起到重要作用。前人研究发现,有一部分瘤胃微生物,如硫还原菌 (SRB)、硝酸盐还原菌、产乙酸菌 (Acetogens) 和延胡索酸还原菌等,可与甲烷菌竞争利用氢,能有效缓解氢分压并减少甲烷产生[25]。SRB利用硫酸盐电子受体沉积氢生成H2S,但其竞争氢的能力通常弱于甲烷菌[25]。本研究qPCR结果显示SRB数量不受溶菌酶影响,说明溶菌酶并未加强这类代谢途径。产乙酸菌可将CO2经乙酰辅酶A代谢途径完成终端电子转移,还原成乙酸或丁酸等非甲烷物质;研究发现瘤胃中主要的产乙酸菌有醋酸菌属 (Acetobacterium)、真杆菌属 (Eubacterium) 和梭菌属 (Clostridium) 等[26],但本研究高通量结果显示此3类菌属未成为优势菌群,表明溶菌酶未增强产乙酸菌途径。琥珀酸丝状杆菌 (Fibrobacter succinogenes) 和反刍兽新月形单胞菌 (Selenomonas ruminantium) 等延胡索酸还原菌能将延胡索酸还原成琥珀酸,可进一步生成丙酸[25]。本研究试验组丙酸浓度均显著增加,月形单胞菌相对丰度增加,丝状杆菌相对丰度降低,我们推测甲烷产量受抑制的原因是由于H2更多地被延胡索酸还原菌 (反刍兽新月形单胞菌) 所利用进而使丙酸浓度升高。
瘤胃发酵产生的VFA是反刍动物的重要能量来源,与瘤胃微生物有密切联系。研究发现,琥珀酸丝状杆菌 (Fibrobacter succinogenes)、白色瘤胃球菌 (Ruminococcus albus) 和黄色瘤胃球菌 (Ruminococcus flavefaciens) 是主要生成乙酸的细菌[27];此外,原虫也可胞吞有机物发酵产生乙酸和丁酸[28]。本试验结果表明,溶菌酶对原虫数量无显著影响,因此推测L-1组瘤胃球菌相对丰度的增加导致了乙酸浓度的增加,而L-100组琥珀酸丝状杆菌和瘤胃球菌相对丰度的减少致使乙酸浓度降低。丙酸生成主要通过琥珀酸 (succinate pathway) 或丙烯酸盐 (acrylate pathway) 两种途径,而绝大部分丙酸经琥珀酸途径合成[27]。其中,琥珀酸弧菌 (Succinivibrio) 是主要的琥珀酸产生菌[29],反刍兽新月形单胞菌 (Selenomonas ruminantium) 是利用琥珀酸生成丙酸的主要菌属[27]。本研究高通量结果显示溶菌酶处理组琥珀酸弧菌丰度升高,月形单胞菌相对丰度也升高,说明溶菌酶通过琥珀酸途径增加了丙酸的生成。前人研究发现丁酸弧菌属是丁酸的来源之一[27];Fraga等[30]研究发现假丁酸弧菌属和未分类的毛螺杆菌科 (Unclassified Lachnospiraceae) 的数量也与丁酸产量呈显著正相关。本研究发现溶菌酶处理组原虫数量和丁酸弧菌相对丰度不受影响,因此推测溶菌酶组丁酸浓度降低可能与假丁酸弧菌属相对丰度显著降低有关。丙酸增加,乙丙比降低,说明溶菌酶使瘤胃发酵类型趋向于丙酸型,有利于能量利用。此外,PCA显示不同处理组瘤胃细菌组成被明显区分开来,说明溶菌酶添加显著改变了瘤胃细菌菌群结构,结合本次瘤胃发酵结果,表明溶菌酶可使氢更多地被甲烷菌以外的瘤胃微生物利用,进而减少甲烷产生并改善瘤胃发酵。
瘤胃中NH3-N浓度反映瘤胃内氨氮产生速率和氨氮被利用速率的关系,是评价N利用效率的一项重要指标。对N利用相关的瘤胃微生物研究发现,瘤胃原虫对蛋白降解有重要作用,去原虫可使瘤胃氨氮浓度降低[31]。研究表明,普雷沃氏菌属 (Prevotella) 在瘤胃众多微生物中含量最为丰富,其中栖瘤胃普雷沃氏菌 (Prevotella ruminicola) 和Prevotella bryantii对蛋白质消化有积极作用[32],栖瘤胃普雷沃氏菌还是瘤胃中唯一产生二肽基肽酶 (dipeptidyl peptidase) 的微生物[33],抑制其生长可减少蛋白降解。此外,栖瘤胃拟杆菌 (Bacteroides ruminicola) 和溶纤维丁酸弧菌 (Butyrivibrio fibrisolvens) 也是重要的蛋白降解菌[32, 34],对蛋白降解及氨氮生成有显著影响。本试验中,溶菌酶对原虫数量和丁酸弧菌相对丰度无显著影响,而普雷沃氏菌属和拟杆菌属的相对丰度在添加溶菌酶后显著降低,因此,L-1组氨氮浓度显著降低说明其可能与发酵体系中普雷沃氏菌属和拟杆菌属被抑制有关。而L-100组氨氮浓度增加可能是由于总菌数量显著降低,导致细菌利用氨合成微生物蛋白效率降低,最终造成氨氮累积所致。另外,支链挥发性脂肪酸 (BCVFA) 和戊酸是氨基酸脱氨基的产物[32],本研究发现试验组BCVFA和戊酸均显著降低,表明溶菌酶抑制了氨基酸利用微生物的脱氨基作用。
4 结论 添加适宜浓度 (1 mg/100 mL) 的溶菌酶可降低瘤胃甲烷和氨的生成,增加丙酸比例以及降低乙丙比,短期内并不影响饲料消化。微生物组学分析结果表明这种发酵模式的改变与瘤胃微生物菌群结构及多样性的变化密切相关。
参考文献
| [1] | Callewaert L, Michiels CW. Lysozymes in the animal kingdom.Journal of Biosciences, 2010, 35(1): 127–160DOI:10.1007/s12038-010-0015-5. |
| [2] | Humphrey BD, Huang N, Klasing KC. Rice expressing lactoferrin and lysozyme has antibiotic-like properties when fed to chicks.The Journal of Nutrition, 2002, 132(6): 1214–1218. |
| [3] | Oliver WT, Wells JE. Lysozyme as an alternative to antibiotics improves growth performance and small intestinal morphology in nursery pigs.Journal of Animal Science, 2013, 91(7): 3129–3136DOI:10.2527/jas.2012-5782. |
| [4] | Maga EA, Walker RL, Anderson GB, Murray JD. Consumption of milk from transgenic goats expressing human lysozyme in the mammary gland results in the modulation of intestinal microflora.Transgenic Research, 2006, 15(4): 515–519DOI:10.1007/s11248-006-0014-3. |
| [5] | Nyachoti CM, Kiarie E, Bhandari SK, Zhang G, Krause DO. Weaned pig responses to Escherichia coli K88 oral challenge when receiving a lysozyme supplement.Journal of Animal Science, 2012, 90(1): 252–260DOI:10.2527/jas.2010-3596. |
| [6] | Green BL, McBride BW, Sandals D, Leslie KE, Bagg R, Dick P. The impact of a monensin controlled-release capsule on subclinical ketosis in the transition dairy cow.Journal of Dairy Science, 1999, 82(2): 333–342DOI:10.3168/jds.S0022-0302(99)75240-9. |
| [7] | Shen JS, Liu Z, Chen YY, Lv PA, Zhu WY. Effects of nisin on in vitro fermentation, methanogenesis and functional microbial populations of the rumen.Acta Microbiologica Sinica, 2016, 56(8): 1348–1357(in Chinese). 申军士, 刘壮, 陈亚迎, 吕朋安, 朱伟云. 乳酸链球菌素对瘤胃体外发酵、甲烷生成及功能菌群数量的影响.微生物学报, 2016, 56(8): 1348–1357. |
| [8] | Theodorou MK, Williams BA, Dhanoa MS, McAllan AB, France J. A simple gas production method using a pressure transducer to determine the fermentation kinetics of ruminant feeds.Animal Feed Science and Technology, 1994, 48(3/4): 185–197. |
| [9] | Wang XF, Mao SY, Liu JH, Zhang LL, Cheng YF, Jin W, Zhu WY. Effect of the gynosaponin on methane production and microbe numbers in a fungus-methanogen co-culture.Journal of Animal and Feed Sciences, 2011, 20(2): 272–284DOI:10.22358/jafs/66184/2011. |
| [10] | Yang CJ, Mao SY, Long LM, Zhu WY. Effect of disodium fumarate on microbial abundance, ruminal fermentation and methane emission in goats under different forage:concentrate ratios.Animal, 2012, 6(11): 1788–1794DOI:10.1017/S1751731112000857. |
| [11] | Weatherburn MW. Phenol-hypochlorite reaction for determination of ammonia.Analytical Chemistry, 1967, 39(8): 971–974DOI:10.1021/ac60252a045. |
| [12] | Ma YY, Cheng YF, Zhu WY. Different origins and stubbles of alfalfa:evaluation of nutritional value by an in vitro fermentation method.Chinese Journal of Animal Nutrition, 2014, 26(8): 2421–2432(in Chinese). 马艳艳, 成艳芬, 朱伟云. 体外发酵法评价不同产地、不同茬次苜蓿的营养价值.动物营养学报, 2014, 26(8): 2421–2432. |
| [13] | Bürgmann H, Pesaro M, Widmer F, Zeyer J. A strategy for optimizing quality and quantity of DNA extracted from soil.Journal of Microbiological Methods, 2001, 45(1): 7–20DOI:10.1016/S0167-7012(01)00213-5. |
| [14] | Zoetendal EG, Akkermans ADL, De Vos WM. Temperature gradient gel electrophoresis analysis of 16S rRNA from human fecal samples reveals stable and host-specific communities of active bacteria.Applied and Environmental Microbiology, 1998, 64(10): 3854–3859. |
| [15] | Denman SE, McSweeney CS. Development of a real-time PCR assay for monitoring anaerobic fungal and cellulolytic bacterial populations within the rumen.FEMS Microbiology Ecology, 2006, 58(3): 572–582DOI:10.1111/fem.2006.58.issue-3. |
| [16] | Sylvester JT, Karnati SKR, Yu ZT, Morrison M, Firkins JL. Development of an assay to quantify rumen ciliate protozoal biomass in cows using real-time PCR.The Journal of Nutrition, 2004, 134(12): 3378–3384. |
| [17] | Denman S, Tomkins N, McSweeney C. Quantitation and diversity analysis of ruminal methanogenic populations in response to the antimethanogenic compound bromochloromethane.FEMS Microbiology Ecology, 2007, 62(3): 313–322DOI:10.1111/fem.2007.62.issue-3. |
| [18] | Devkota S, Wang Y, Musch MW, Leone V, Fehlnerpeach H, Nadimpalli A, Antonopoulos DA, Jabri B, Chang EB. Dietary-fat-induced taurocholic acid promotes pathobiont expansion and colitis in Il10-/- mice.Nature, 2012, 487(7405): 104–108. |
| [19] | Edgar RC. Search and clustering orders of magnitude faster than BLAST.Bioinformatics, 2010, 26(19): 2460–2461DOI:10.1093/bioinformatics/btq461. |
| [20] | Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Gl?ckner FO. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools.Nucleic Acids Research, 2013, 41(D1): D590–D596DOI:10.1093/nar/gks1219. |
| [21] | Menke KH, Steingass H. Estimation of the energetic feed value obtained from chemical analysis and in vitro gas production using rumen fluid.Animal Research and Development, 1988, 28: 7–55. |
| [22] | Koike S, Kobayashi Y. Fibrolytic rumen bacteria:their ecology and functions.Asian-Australasian Journal of Animal Sciences, 2009, 22(1): 131–138DOI:10.5713/ajas.2009.r.01. |
| [23] | Thoetkiattikul H, Mhuantong W, Laothanachareon T, Tangphatsornruang S, Pattarajinda V, Eurwilaichitr L, Champreda V. Comparative analysis of microbial profiles in cow rumen fed with different dietary fiber by tagged 16S rRNA gene pyrosequencing.Current Microbiology, 2013, 67(2): 130–137DOI:10.1007/s00284-013-0336-3. |
| [24] | Guo YQ, Hu WL, Liu JX. Methanogens and manipulation of methane production in the rumen.Acta Microbiologica Sinica, 2005, 45(1): 145–148(in Chinese). 郭嫣秋, 胡伟莲, 刘建新. 瘤胃甲烷菌及甲烷生成的调控.微生物学报, 2005, 45(1): 145–148. |
| [25] | Morgavi DP, Forano E, Martin C, Newbold CJ. Microbial ecosystem and methanogenesis in ruminants.Animal, 2010, 4(7): 1024–1036DOI:10.1017/S1751731110000546. |
| [26] | Drake HL, Küsel K, Matthies C. Acetogenic prokaryotes//Rosenberg E, DeLong EF, Lory S, Stackebrandt E, Thompson F. The Prokaryotes:Prokaryotic Physiology and Biochemistry. Berlin Heidelberg:Springer, 2013:3-60. |
| [27] | Mitsumori M, Sun WB. Control of rumen microbial fermentation for mitigating methane emissions from the rumen.Asian-Australasian Journal of Animal Sciences, 2008, 21(1): 144–154DOI:10.5713/ajas.2008.r01. |
| [28] | Hillman K, Williams AG, Lloyd D. Postprandial variations in endogenous metabolic activities of ovine rumen ciliate protozoa.Animal Feed Science and Technology, 1995, 52(3/4): 237–247. |
| [29] | Pope PB, Smith W, Denman SE, Tringe SG, Barry K, Hugenholtz P, McSweeney CS, McHardy AC, Morrison M. Isolation of Succinivibrionaceae implicated in low methane emissions from Tammar wallabies.Science, 2011, 333(6042): 646–648DOI:10.1126/science.1205760. |
| [30] | Fraga M, Perelmuter K, Valencia MJ, Martínez M, Abin-Carriquiry A, Cajarville C, Zunino P. Evaluation of native potential probiotic bacteria using an in vitro ruminal fermentation system.Annals of Microbiology, 2014, 64(3): 1149–1156DOI:10.1007/s13213-013-0753-3. |
| [31] | Firkins JL, Yu Z, Morrison M. Ruminal nitrogen metabolism:perspectives for integration of microbiology and nutrition for dairy.Journal of Dairy Science, 2007, 90(S): E1–E16. |
| [32] | Patra AK, Yu ZT. Effects of vanillin, quillaja saponin, and essential oils on in vitro fermentation and protein-degrading microorganisms of the rumen.Applied Microbiology and Biotechnology, 2014, 98(2): 897–905DOI:10.1007/s00253-013-4930-x. |
| [33] | Wallace RJ. Ruminal microbial metabolism of peptides and amino acids.The Journal of Nutrition, 1996, 126(4S): 1326S–1334S. |
| [34] | Chen G, Russell JB. More monensin-sensitive, ammonia-producing bacteria from the rumen.Applied and Environmental Microbiology, 1989, 55(5): 1052–1057. |
