邓洁, 吴巧芬, 高华, 徐悦, 欧倩, 武波, 蒋承建


亚热带农业生物资源保护与利用国家重点实验室, 广西大学生命科学与技术学院, 广西 南宁 530004
收稿日期:2017-03-25;修回日期:2017-05-28;网络出版日期:2017-06-26
基金项目:国家自然科学基金(21262003);2017年度广西高等教育创优计划教学相关项目-优势特色专业项目(优质本科专业)
*通信作者:蒋承建, Tel:+86-771-3239403;Fax:+86-771-3237873;E-mail:jiangcj@gxu.edu.cn
摘要:[目的]完成一个来源于碱性污染土壤宏基因组文库的新的L-半胱亚磺酸脱羧酶基因undec1A的鉴定,研究其酶学性质并利用非理性设计技术对其进行分子改造。[方法]以pETBlue-2为表达载体构建包含undec1A基因的重组表达质粒,转化至宿主细胞E.coli Tuner(DE3)pLacI中构建重组表达克隆,采用镍亲和层析完成了酶蛋白的分离纯化,完成其生化特征研究,利用连续易错PCR技术对Undec1A蛋白进行分子改造。[结果]生物信息学分析结果揭示Undec1A蛋白与已知的L-半胱亚磺酸脱羧酶存在类似的辅酶结合位点和底物识别催化基序等。分子对接结果显示氨基酸残基Val237、Asp239、Asp266、Ile267、Ala268和Lys298等决定了与底物分子L-半胱亚磺酸的识别和结合催化。以L-半胱亚磺酸作为底物,重组Undec1A蛋白的最适作用pH为7.0,最适作用温度为35℃;分子动力学常数Km为(1.557±0.015)mmol/L,Vmax为(49.07±3.19)μmol/(L·min),kcat为(45.80 ±1.32)/min。利用连续易错PCR技术完成了亲本酶的分子改造,分离筛选到了一个酶活力更高的突变酶Undec1A-1180。在优化条件下,Undec1A-1180的比活力较亲本酶提高了约5.62倍。[结论]本研究为构建牛磺酸的生物合成工艺提供了理论参考,因而具有重要的实践意义。
关键词: 牛磺酸 L-半胱亚磺酸脱羧酶 宏基因组文库 连续易错PCR技术 突变酶
Protein engineering by random mutagenesis and analysis of a metagenome-derived cysteine sulfinate decarboxylase
Jie Deng, Qiaofen Wu, Hua Gao, Yue Xu, Qian Ou, Bo Wu, Chengjian Jiang


State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources, College of Life Science and Technology, Guangxi University, Nanning 530004, Guangxi Province, China
Received 25 March 2017; Revised 28 May 2017; Published online 26 June 2017
*Corresponding author: Chengjian Jiang, Tel:+86-771-3239403;Fax:+86-771-3237873;E-mail:jiangcj@gxu.edu.cn
Supported by the National Natural Science Foundation of China (21262003), and by the 2017 Excellent Teaching Program of Guangxi High Education-Program of Advantage and Characteristic Specialty (High Quality of Undergraduate Program)
Abstract: [Objective]The aim of this study was to identify a novel L-cysteine sulfinate decarboxylase from alkaline polluted soil metagenome and to use non-rational design method to improve the enzyme.[Methods]L-cysteine sulfinate decarboxylase gene undec1A was cloned into pETBlue-2 vector and expressed in Escherichia coli Tuner (DE3) pLacI. The recombinant Undec1A protein was purified to homogeneity. The original Undec1A protein was characterized and a mutagenesis library was constructed with sequential error-prone PCR method, then an interesting variant was identified.[Results]Multiple sequence alignment analysis showed that Undec1A protein shared the similar pyridoxal 5'-phosphate binding sites and the substrate recognition motif with the other known L-cysteine sulfinate decarboxylases. Molecular docking results indicated that amino acid residues Val237, Asp239, Asp266, Ile267, Ala268 and Lys298 contributed to the decarboxylation of L-cysteine sulfinate acid. Recombinant Undec1A protein had an apparent Km of (1.557±0.015) mmol/L, a Vmax of (49.07±3.19) μmol/(L·min), and a kcat of (45.80 ±1.32)/min at the optimal reaction conditions of 35℃ and pH 7.0 when using L-cysteine sulfinate acid as the substrate. Furthermore, the protein engineering approach of random mutagenesis via sequential error-prone PCR was applied on the original Undec1A protein. Compared with the original Undec1A protein, the best variant of Undec1A-1180 in the random mutagenesis library, exhibited 5.62-folds at the optimal reaction conditions of 35℃ and pH 7.0.[Conclusion]These results are the first step towards a better understanding of the properties of Undec1A protein. Protein engineering with error-prone PCR paves the way toward the metagenome-derived genes for biotechnological applications.
Key words: taurine L-cysteine sulfinate decarboxylase metagenomic library sequential error-prone PCR mutation
牛磺酸(Taurine)又名牛胆酸,化学名为2-氨基乙磺酸,分子式为C2H7NO3S,结构式为H2N-CH2-CH2-SO3H,是动物体内含量最丰富的游离氨基酸,也是人体必需的氨基酸之一[1]。牛磺酸具有多种独特的生理功能,在消炎、镇痛、解热、降血糖、维持正常视觉机能、调节神经传导和脂类的消化与吸收、参与内分泌活动、增加心脏收缩能力、提高免疫能力等方面发挥重要作用,因此作为一种添加剂被广泛应用于食品、饲料和医药等工业领城[2]。
目前市场上流行的牛磺酸生产方法主要有化学合成法和生物提取法等[3]。在生物组织中,牛磺酸的生物合成主要是由蛋氨酸和半胱氨酸代谢的中间产物——半胱亚磺酸(Cysteine sulfinic acid,CSA)经L-半胱亚磺酸脱羧酶(Cysteine sulfinate decarboxylase,CSD,EC 4.1.1.29) 脱羧成亚牛磺酸,再经氧化生成,即半胱亚磺酸代谢途径[4]。体外生物合成牛磺酸反应条件温和,安全,产品质量高,而且没有环境污染,具有重要的经济效益和环保意义。但是目前已发现的L-半胱亚磺酸脱羧酶活性都非常低[5],CSD的活性直接反映了生物合成牛磺酸的能力。因此,从自然环境体系中分离新的L-半胱亚磺酸脱羧酶基因构建高效基因工程菌研发生物合成牛磺酸的新工艺显得非常重要。
L-半胱亚磺酸脱羧酶较早分离于肝脏组织,被认为是牛磺酸合成途径中的限速酶,L-半胱亚磺酸脱羧酶活性的高低,直接制约牛磺酸的合成量[6]。已经证实来自于脑组织的CSD和分离于肝脏组织的CSD酶都具有脱羧半胱亚磺酸盐和磺基丙氨酸的功能,然而不同的是,相对于肝脏组织的CSD,脑组织中的CSD并不完全依赖于磷酸吡哆醛[7]。不同来源诸如水牛、狗和小鼠脑组织的L-半胱亚磺酸脱羧酶可以分成两大类,即CSD Ⅰ型和CSD Ⅱ型,其中CSD Ⅱ型具有谷氨酸脱羧酶的功能;而CSD Ⅰ型主要分离于肝脏组织,与牛磺酸的体内和体外合成具有直接联系[8]。Reymond等完成了来自老鼠肝脏组织的L-半胱亚磺酸脱羧酶的基因克隆和序列分析等工作。生物信息学分析表明相关基因编码的蛋白属于CSD Ⅱ型,和谷氨酸脱羧酶具有明显的序列一致性,是一个包含493个氨基酸的多肽,分子量大小为55.2 kDa[9]。
目前有关L-半胱亚磺酸脱羧酶的研究主要来源于真核生物细胞[10]。众所周知,微生物是一个巨大的基因资源库。分子生态学技术研究表明:自然界中除了少部分可用传统纯培养方法获得的微生物以外,还存在着大量的“未培养微生物”。未培养微生物在其物种类群、新陈代谢途径、生理生化反应等方面都存在更为广泛的多样性,是更为丰富的可供人类开发利用的资源[11]。Handelsman等经过系统总结后提出的“宏基因组文库技术”概念把环境微生物的研究提升到了一个新层次。宏基因组文库技术绕开了“纯培养技术”的瓶颈,使得人们在基因组水平上直接开发利用自然界中丰富的未培养微生物资源变成现实[12]。从自然环境体系宏基因组中分离新的L-半胱亚磺酸脱羧酶进行作用机制研究具有重要的理论意义。但是目前国际上鲜见有关来自于环境宏基因组文库中新的L-半胱亚磺酸脱羧酶的鉴定报道。
前期研究中我们构建了碱性污染土壤宏基因组文库,基于酶的功能筛选和序列特异性筛选策略,从文库中分离到了一个新的氨基酸脱羧酶基因undec1A[13]。本研究中我们以L-半胱亚磺酸作为底物,完成了Undec1A蛋白具有L-半胱亚磺酸脱羧酶的功能分析,利用连续易错PCR技术,构建了突变酶库,完成了一个具有较高酶活力的突变酶Undec1A-1180的鉴定研究。本研究拓宽了人们对未培养微生物来源的L-半胱亚磺酸脱羧酶的认识,也为构建新的牛磺酸微生物工艺提供了技术参考。
1 材料和方法 1.1 主要仪器和试剂 分光光度计UV mini-124为Shimadzu产品;离心机为Eppendorf公司产品;PCR仪为EDC-810东胜创新生物科技有限公司产品。T4 DNA连接酶购自TaKaRa公司;Pfu DNA聚合酶购自天根生物有限公司;质粒提取试剂盒购于上海生物工程有限公司;胶回收及PCR产物纯化试剂盒购于Bio Flux公司;其他各种酶制剂购自Promega公司;各种化学试剂均为国产分析纯。
1.2 DNA处理和蛋白质分析 所有DNA处理方法包括基因克隆、亚克隆、E. coli感受态细胞的制备以及PCR都依据标准操作技术[14]或者产品说明,特别标明除外。蛋白质的准备和分析包括E. coli中蛋白的提取、定量以及SDS-PAGE也是遵照标准操作技术[15]。
1.3 文库来源和基因来源 本研究所用文库为课题组前期已构建的碱性污染土壤宏基因组文库。该文库以pGEM-3Zf(+)为克隆载体,包含大约30000个克隆,外源DNA插入片段平均大小为3.5 kb左右。基因undec1A为课题组前期利用基于序列特异性直接测序分离得到,基因大小为1077 bp,可以编码一个由359个氨基酸残基组成的多肽[13]。
1.4 DNA序列分析和基因结构鉴定 DNA序列分析主要是用ABI Prism 3700 DNA测序仪中的bigdye终结者V3.1的循环测序(Applied Biosystems,USA)。利用在线蛋白质翻译工具Expasy (http://www.expasy.org/tools/dna.html)进行蛋白质翻译。在NCBI (the National Center for Biotechnology Information) Entrez页面的蛋白质和核酸数据库在线相关软件进行序列分析比较(http://www.ncbi.nlm.nih.gov//)。利用BLAST 2.0软件进行序列相似性查找。用Vector NTI (Informax,North Bethesda,MD,USA)中的Align X组件进行同源蛋白序列分析,评分矩阵为blosum62mt2。通过推断出的氨基酸序列比较,完成了一个编码新的L-半胱亚磺酸脱羧酶的基因undec1A的鉴定。并以Neighbor-Joining计算模型为基础的Molecular Evolutionary Genetics Analysis 6.0 (MEGA,Version 6.0)[16]软件构建了进化树。采用引导值评估进化树的可靠性(1000次重复)。采用Swiss-Model服务器(https://swissmodel.expasy.org/)进行酶蛋白的同源建模;模型评估在服务器UCLA-SAVES (http://services.mbi.ucla.edu/SAVES/)上进行;使用软件Autodock 4.2完成底物分子L-半胱亚磺酸和Undec1A蛋白的分子对接;使用PyMOL软件完成建模分子的图形观察。
1.5 L-半胱亚磺酸脱羧酶基因的突变体库构建和筛选 本研究中PCR引物委托于上海生物工程有限公司合成。根据生物信息学分析的序列特征,设计以下引物扩增目标ORF (open reading frame):上游引物:5′-ATAGTCGACATGATCACCCCTCTT ACGCTGGCAAC-3′;下游引物:5′-CGACGCGTG TGAACCAGGGTAAGTATCTTCCG-3′。在上、下游引物中分别引入Sal Ⅰ和Mlu Ⅰ酶切位点(下划线部分)。配制10×易错Buffer:1 mol/L KCl,1 mol/L Tris-HCl;10×dNTP为2 mmol/L dGTP、2 mmol/L dATP、10 mmol/L dCTP和10 mmol/L dTTP。100 μL易错PCR反应体系包含:10 μL10×易错Buffer,10 pmol/L引物,10 U Taq DNA聚合酶,20 ng质粒模板DNA,4 mmol/L MgCl2和0.5 mmol/L MnCl2等。PCR反应体系设置为:96 ℃ 2 min;94 ℃ 40 s,65 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min,扩增得到1077 bp左右的DNA片段。用质量分数为1.0%的琼脂糖凝胶电泳鉴定易错PCR产物,利用胶回收试剂盒切胶回收PCR产物,于–20 ℃保存。第一轮PCR产物经过分离纯化后用作第二轮易错PCR模板。将最终易错PCR产物经胶回收分离纯化后,利用限制性酶Sal Ⅰ和Mlu Ⅰ酶切消化后与经过同样处理的pGEM-3Zf(+)连接,转化感受态细胞E. coli DH5α,构建突变体库。大量抽提突变文库中重组阳性质粒DNA,与限制性内切酶Sal Ⅰ和Mlu Ⅰ双酶切后的pETBlue-2载体进行连接,转化感受态细胞E. coli Tuner (DE3) pLac?,利用氨基酸自动分析仪(Beckman 6300,USA)进行突变体库的分离筛选[17]。酶基因在大肠杆菌系统中的表达和酶蛋白的分离纯化参照文献[13]执行。
1.6 重组蛋白活性测定和最适温度以及最适pH 重组Undec1A蛋白活性测定采用氨基酸自动分析方法[17],酶活力的计算方式为pH 7.0、35 ℃条件下1 s内转化1 μmol/L L-半胱亚磺酸所需要的酶量为1个酶活力单位。最适pH和最适温度的测定都是采用标准测定方法[17]。所有实验均需重复3次,取平均值。测定pH对Undec1A活性影响时,pH梯度为pH 4.5–6.0 (20 mmol/L醋酸缓冲液)、pH 6.0–7.5 (20 mmol/L磷酸缓冲液)和pH 7.0–9.0 (Tris-HCl缓冲液),缓冲体系中添加5 mmol/L DTT。测定不同温度条件对酶活性的影响时,在20 mmol/L磷酸缓冲液(pH 7.0) 中测定不同温度(20–50 ℃)下的酶活性。
1.7 酶动力学参数测定 重组Undec1A蛋白的动力学参数主要是通过在35 ℃、20 mmol/L磷酸缓冲液(pH 7.0) 中,测定不同浓度下(0.25–15.00 mmol/L) L-半胱亚磺酸减少的速度获得。利用酶动力学程序进行双倒数作图获得酶学参数Km和Vmax等[18]。
2 结果和分析 2.1 新的L-半胱亚磺酸脱羧酶基因的分离筛选和分析 基于序列特异性直接测序分析策略,课题组完成了undec1A基因(GenBank数据库登录号:EF015465.1) 的分离筛选。利用LC-MS技术完成了Undec1A蛋白(GenBank数据库登录号:ABJ80896.1) 能够催化L-半胱氨酸脱羧生成β-巯基乙胺的鉴定,结合相应的脱羧酶生物信息学分析结果,证明Undec1A蛋白具备L-半胱氨酸脱羧酶的功能[13]。生物信息学分析比较结果暗示在DNA水平上,undec1A基因和已知的L-半胱亚磺酸脱羧酶没有任何一致性。在氨基酸水平上,Undec1A蛋白与来自Danio rerio的L-半胱亚磺酸脱羧酶(NP_001007349.1) 的相似性最高,为26.1%;与来自Ceratosolen solmsi marchali的L-半胱亚磺酸脱羧酶(XP_011497415) 的相似度较高,为21.8%。功能区域和已知的L-半胱亚磺酸脱羧酶也具有类似的保守基序(图 1),诸如底物识别基序和PLP结合基序等,但是保守性一般。
 |
| 图 1 Undec1A蛋白和其他L-半胱亚磺酸脱羧酶的多重序列分析比较结果 Figure 1 Multiple sequence alignment of the Undec1A protein with other CSDs. From top to bottom, except the first of Undec1A protein. Other proteins were including: the sequences are from the Ceratosolen solmsi marchali (GenBank accession number: XP_011497415.1), the Danio rerio (GenBank accession number: NP_001007349.1), the Rattus norvegicus (GenBank accession number: 2113203A), and the Bos taurus (GenBank accession number: DAA29988). |
| 图选项 |
以PDB数据库中Bacillus anthracis来源的磷酸式泛酰巯基乙胺半胱氨酸脱羧酶(PDB ID: 5int)为模板,该模板与Undec1A的一致性为45.75%,利用Swiss-Model服务器[19],完成了Undec1A蛋白的同源建模(图 2-A)。利用分子对接技术[20],完成了Undec1A分子和底物分子L-半胱亚磺酸之间的分子对接(图 2-B)。对接模型揭示了Undec1A蛋白与L-半胱亚磺酸可能的结合位点为Val237、Asp239、Asp266、Ile267、Ala268和Lys298残基等,这些残基均与L-半胱亚磺酸之间形成氢键。这些结果也揭示了Undec1A蛋白与已知的L-半胱亚磺酸脱羧酶具有相似的底物结合识别催化模式。
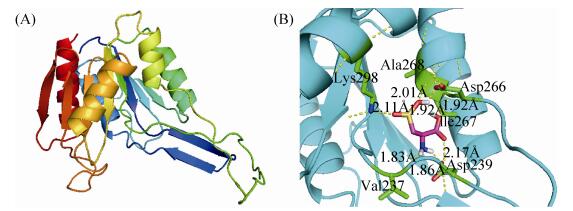 |
| 图 2 Undec1A的同源建模模型(A)及Undec1A与L-半胱亚磺酸的分子对接结果(B) Figure 2 Homology modeling structure (A) and the docking result (B) of Undec1A with L-cysteine sulfinate acid, respectively. |
| 图选项 |
系统进化分析结果揭示了Undec1A蛋白与Danio rerio和Papilio Xuthus等来源的L-半胱亚磺酸脱羧酶具有亲缘关系,但是进化关系较为疏远(图 3)。根据保守的酶的活性位点,相类似的L-半胱亚磺酸残基的催化功能等分子结构特征分析,初步将Undec1A蛋白鉴定为一个新的L-半胱亚磺酸脱羧酶。
 |
| 图 3 Undec1A蛋白和其他L-半胱亚磺酸脱羧酶的进化关系分析 Figure 3 Phylogenetic relationship of Undec1A protein with related CSDs. The parentheses indicated the different sources of CSDs. The phylogenetic tree was constructed by the neighbor-joining method using MEGA 6.0 and 1000 bootstrap replicates are indicated at branching points. The Undec1A was shown with red triangle. The GenBank accession number and original genus of other CSD were also shown on the tree. The scale bar represents the number of changes per amino acid position. |
| 图选项 |
2.2 L-半胱亚磺酸脱羧酶最适pH和最适作用温度 利用pETBlue-2表达载体,构建了包含undec1A基因的重组表达质粒;将重组表达质粒导入E. coli Tuner (DE3) pLac?感受态细胞,构建了包含目的基因的重组表达克隆。完成了酶基因在大肠杆菌系统中的高效表达,采用镍柱亲和层析技术,完成了酶蛋白的分离纯化。
以L-半胱亚磺酸为底物,发现在pH 6.0–8.0的范围内,Undec1A蛋白对L-半胱亚磺酸保持相对比较高的活力(> 80%)。在pH 7.0左右的情况下,酶活力相对最高。因此,Undec1A蛋白的最适作用pH值为7.0。在pH 5.5–8.0的范围内,Undec1A蛋白对pH值的变化表现出了较高的耐受性,酶的活力保持相对较高,变化不明显。发现在30–40 ℃温度范围内,Undec1A蛋白对L-半胱亚磺酸保持相对比较高的活力(> 70%)。在35 ℃左右的情况下,酶活力相对最高。因此,Undec1A蛋白的最适作用温度为35 ℃。
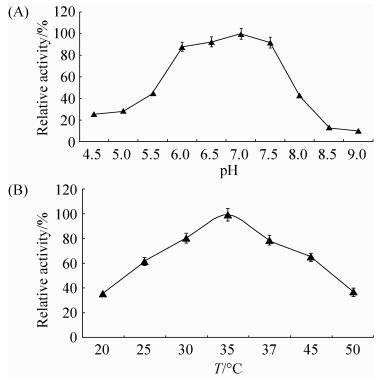 |
| 图 4 Undec1A蛋白的最适pH (A)和最适作用温度(B) Figure 4 The optimal pH (A) and temperature (B) of recombinant Undec1A protein. |
| 图选项 |
2.3 底物谱特征 在pH 7.0、作用温度为35 ℃的条件下,检测重组Undec1A蛋白对不同底物的脱羧情况(表 1)。结果表明Undec1A蛋白对L-Cysteine、L-Asparaginate、L-Glutamate和L-Cysteine sulfinate acid等均具备较好的脱羧效果。
表 1. Undec1A蛋白对不同底物的作用效果检测 Table 1. Relative activities of Undec1A protein toward different substrates
| Substrates | Relative activity/% |
| L-Cysteine | 99.67±2.52 |
| L-Alanine | 16.57±1.82 |
| L-Proline | 41.73±2.45 |
| L-Asparaginate | 88.97±2.37 |
| L-Glutamate | 84.33±1.68 |
| L-Cysteine sulfinate acid | 75.10±2.75 |
表选项
2.4 分子动力学参数 在pH 7.0、作用温度为35 ℃的条件下,调整底物L-半胱亚磺酸的浓度为:0.25、1.00、3.00、5.00、8.00、15.00 mmol/L。测定不同底物浓度下Undec1A蛋白的酶活力数据。根据Lineweaver-Burk作图,Undec1A蛋白对L-半胱亚磺酸的Km为(1.557±0.015) mmol/L,Vmax为(49.07±3.19) μmol/(L·min),kcat为(45.8±1.32)/min。
2.5 突变酶的分离筛选 利用连续易错PCR技术,我们构建了一个大约包含10000个阳性克隆左右的突变体库。基于氨基酸脱羧酶的功能筛选策略和序列特异性的直接测序分离筛选相结合的策略,分离得到了一个让人感兴趣的突变蛋白Undec1A-1180。与原始氨基酸序列比较,发现Undec1A-1180有4处氨基酸发生改变,分别为Val81Leu、Phe240Ser、Ile250Ser和Asp266Leu。
以L-半胱亚磺酸盐酸盐为底物,突变蛋白Undec1A-1180在pH 7.0和35 ℃下活性最高。表观米氏常数Km为(1.105±0.022) mmol/L,最大反应速率Vmax为(108.67±3.55) μmol/(L·min),催化常数kcat为(88.57± 2.12) ? /min。与亲本酶比较,突变酶Undec1A-1180的作用最适条件没有太大的变化,但是与底物的亲和力增强,催化底物转化为产物的能力增强。在优化条件下,比活力为(24.1±1.6) U/mg protein,较亲本酶增加了约5.62倍。
3 讨论 当前有关水牛、狗、鱼类和小鼠等脑、肝脏组织等不同真核细胞体系来源的L-半胱亚磺酸脱羧酶研究较为深入[21]。相关研究也指出不同来源的L-半胱亚磺酸脱羧酶在结构和功能上均存在相当大的差异性(表 2)。来自于脑组织的CSD和分离于肝脏组织的CSD酶都具有脱羧半胱亚磺酸盐和磺基丙氨酸的功能[9],然而不同的是,相对于肝脏组织的CSD,脑组织中的CSD并不完全依赖于磷酸吡哆醛[4, 6, 22]。分离于脑组织中的CSD已经被证实在生物合成γ-氨基丁酸(GABA)过程中具有谷氨酸脱羧酶(GAD)的功能[23]。后来有关水牛、狗和小鼠脑组织中的CSD研究结果进一步说明了L-半胱亚磺酸脱羧酶可以分成两大类,即CSD Ⅰ型和CSD Ⅱ型,其中CSD Ⅱ型具有谷氨酸脱羧酶的功能;而CSD Ⅰ型主要分离于肝脏组织,与牛磺酸的体内和体外合成具有直接联系[22-24]。在体外实验中,以半胱亚磺酸为底物,CSD Ⅰ型的Km值为0.05–0.20 mmol/L[7, 25],这个浓度和脑组织中半胱亚磺酸的浓度0.1–0.2 mmol/L是非常接近的;CSD Ⅱ型的Km值一般是5–6 mmol/L。相对来说,CSD Ⅱ型对半胱亚磺酸的亲和力比CSD Ⅰ型低25–100倍[25]。更加值得重视的是,CSD Ι型作用的底物谱很窄,它只对半胱亚磺酸和磺基丙氨酸起作用;但是CSD Ⅱ型可以表达GAD活性,可以被谷氨酸抑制[8]。根据Undec1A蛋白可以表达GAD活性的功能特征,推测Undec1A蛋白是CSD Ⅱ型L-半胱亚磺酸脱羧酶。
表 2. 不同来源的CSDs性质比较 Table 2. Properties of CSDs from various sources
| Source | pH | T/℃ | Km/(mmol/L) | kcat | Reference |
| UndeclA | 7.0 | 35 | 1.557±0.015 | 45.80±1.32/min | This study |
| UndeclA-1180 | 7.0 | 35 | 1.105±0.022 | 88.57士2.12/min | This study |
| Rainbow trout CSD | 6.8 | 37 | 2.300±0.240 | 4.72±0.36/s | Wang et al.[21] |
| Japanese flounder CSD | 6.8 | 37 | 3.450±0.380 | 1.52士0.13/s | Wang et al.[21] |
| Rat liver CSD | 7.4-7.8 | - | 0.045-0.050 | - | Oertel et al.[22] |
| Rat brain CSD Ⅰ | 7.4-7.8 | - | 0.050-0.100 | - | Oertel et al.[22] |
| Rat brain CSD Ⅱ | 7.4-7.5 | - | 6 | - | Oertel et al.[22] |
表选项
有研究通过原核生物细胞体系来源的系列CSD的结构研究,发现了新的底物识别基序和活性位点。功能研究结果指出在某些细菌中存在相同或类似的CSD,也发现细菌中含有半胱氨酸双加氧酶可以氧化L-半胱氨酸生成CSA,这些相关证据支持在原核生物中存在一个可能的细菌牛磺酸生物合成途径的构想[17]。在此前研究中,我们指出Undec1A蛋白具有能够使L-半胱氨酸脱羧生成β-巯基乙胺的功能[13, 26]。当前研究也论证了在相关作用条件下,Undec1A蛋白具备L-半胱亚磺酸脱羧酶的活性。我们推测未培养微生物来源的酶基因由于结构上的差异性,很可能具备一些意料之外的功能特征。本研究拓宽了人们对未培养微生物来源的新酶基因的功能认识,也为阐述生物组织在合成牛磺酸过程中的L-半胱亚磺酸脱羧酶酶活力低的瓶颈问题提供新的参考依据。
总的来说,本研究利用宏基因组学技术,完成了一个碱性污染土壤微生物来源的具有L-半胱亚磺酸脱羧酶活性的新基因undec1A的鉴定研究。多重序列比对和系统进化关系分析以及同源建模结果揭示了Undec1A蛋白和已知的L-半胱亚磺酸脱羧酶具有一定的序列相似度。在Undec1A蛋白序列中发现了相应的保守基序和关键氨基酸残基位点。完成了重组Undec1A蛋白的系列生化性质诸如酶活力、最适pH和最适作用温度等的研究,证明了Undec1A具备多底物谱特征。利用连续易错PCR技术,完成了突变体库的构建,并从文库中分离得到了一个感兴趣的突变酶Undec1A-1180。本研究对挖掘新的L-半胱亚磺酸脱羧酶基因资源和构建新的牛磺酸生物合成工艺具有参考意义。
References
| [1] | Lambert IH, Kristensen DM, Holm JB, Mortensen OH. Physiological role of taurine-from organism to organelle. Acta Physiologica, 2015, 213(1): 191-212. DOI:10.1111/apha.12365 |
| [2] | Huxtable RJ. Physiological actions of taurine. Physiological Reviews, 1992, 72(1): 101-163. |
| [3] | Gu YL, Shi F, Yang HZ, Deng YQ. Leaching separation of taurine and sodium sulfate solid mixture using ionic liquids. Separation and Purification Technology, 2004, 35(2): 153-159. DOI:10.1016/j.seppur.2003.08.004 |
| [4] | Tappaz M, Almarghini K, Legay F, Remy A. Taurine biosynthesis enzyme cysteine sulfinate decarboxylase (CSD) from brain:the long and tricky trail to identification. Neurochemical Research, 1992, 17(9): 849-859. DOI:10.1007/BF00993260 |
| [5] | Haga Y, Kondo H, Kumagai A, Satoh N, Hirono I, Satoh S. Isolation, molecular characterization of cysteine sulfinic acid decarboxylase (CSD) of red sea bream Pagrus major and yellowtail Seriola quinqueradiata and expression analysis of CSD from several marine fish species. Aquaculture, 2015, 449: 8-17. DOI:10.1016/j.aquaculture.2015.04.004 |
| [6] | Kim HW, Yoon SH, Park T, Kim BK, Park KK, Lee DH. Gene expressions of taurine transporter and taurine biosynthetic enzyme during mouse and chicken embryonic development. Advances in Experimental Medicine and Biology, 2006, 583: 69-77. DOI:10.1007/978-0-387-33504-9 |
| [7] | Li JH, Ling YQ, Fan JJ, Zhang XP, Cui S. Expression of cysteine sulfinate decarboxylase (CSD) in male reproductive organs of mice. Histochemistry and Cell Biology, 2006, 125(6): 607-613. DOI:10.1007/s00418-005-0095-8 |
| [8] | Ueki I, Stipanuk MH. 3T3-L1 adipocytes and rat adipose tissue have a high capacity for taurine synthesis by the cysteine dioxygenase/cysteinesulfinate decarboxylase and cysteamine dioxygenase pathways. Journal of Nutrition, 2009, 139(2): 207-214. |
| [9] | Reymond I, Sergeant A, Tappaz M. Molecular cloning and sequence analysis of the cDNA encoding rat liver cysteine sulfinate decarboxylase (CSD). Biochimica et Biophysica Acta (BBA)-Gene Structure and Expression, 1996, 1307(2): 152-156. DOI:10.1016/0167-4781(96)00068-1 |
| [10] | Junyent F, Utrera J, Camins A, Pallàs M, Romero R, Auladell C. Synthesis, uptake and release of taurine in astrocytes treated with 8-Br-cAMP. Neuroscience Letters, 2009, 467(3): 199-202. DOI:10.1016/j.neulet.2009.10.032 |
| [11] | Monier JM, Demanèche S, Delmont TO, Mathieu A, Vogel TM, Simonet P. Metagenomic exploration of antibiotic resistance in soil. Current Opinion in Microbiology, 2011, 14(3): 229-235. DOI:10.1016/j.mib.2011.04.010 |
| [12] | Schloss PD, Handelsman J. Metagenomics for studying unculturable microorganisms:cutting the Gordian knot. Genome Biology, 2005, 6(8): 229. DOI:10.1186/gb-2005-6-8-229 |
| [13] | Jiang CJ, Wu B. Molecular cloning and functional characterization of a novel decarboxylase from uncultured microorganisms. Biochemical and Biophysical Research Communications, 2007, 357(2): 421-426. DOI:10.1016/j.bbrc.2007.03.159 |
| [14] | Sambrook J, Russell DW. Molecular Cloning:A Laboratory Manual. 3rd edition. New York: Cold Spring Harbor Laboratory Press, 2001. |
| [15] | Reisinger V, Eichacker LA. Isolation of membrane protein complexes by blue native electrophoresis. Methods in Molecular Biology, 2008, 424: 423-431. DOI:10.1007/978-1-60327-064-9 |
| [16] | Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:molecular evolutionary genetics analysis version 6:molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [17] | Agnello G, Chang LL, Lamb CM, Georgiou G, Stone EM. Discovery of a substrate selectivity motif in amino acid decarboxylases unveils a taurine biosynthesis pathway in prokaryotes. ACS Chemical Biology, 2013, 8(10): 2264-2271. DOI:10.1021/cb400335k |
| [18] | Stanislawski J. Enzyme Kinetics, version 1.5. Fort Pierce, Fla: Trinity Software, 1991. |
| [19] | Biasini M, Bienert S, Waterhouse A, Arnold K, Studer G, Schmidt T, Kiefer F, Cassarino TG, Bertoni M, Bordoli L, Schwede T. SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information. Nucleic Acids Research, 2014, 42(W1): W252-W258. DOI:10.1093/nar/gku340 |
| [20] | Gaboyer F, Burgaud G, Alain K. Physiological and evolutionary potential of microorganisms from the Canterbury Basin subseafloor, a metagenomic approach. FEMS Microbiology Ecology, 2015, 91(5): pii:fiv029. |
| [21] | Wang X, He G, Mai KS, Xu W, Zhou HH. Differential regulation of taurine biosynthesis in rainbow trout and Japanese flounder. Scientific Reports, 2016, 6: 21231. DOI:10.1038/srep21231 |
| [22] | Oertel WH, Schmechel DE, Weise VK, Ransom DH, Tappaz ML, Krutzsch HC, Kopin IJ. Comparison of cysteine sulphinic acid decarboxylase isoenzymes and glutamic acid decarboxylase in rat liver and brain. Neuroscience, 1981, 6(12): 2701-2714. DOI:10.1016/0306-4522(81)90114-7 |
| [23] | Tappaz M, Bitoun M, Reymond I, Sergeant A. Characterization of the cDNA coding for rat brain cysteine sulfinate decarboxylase:brain and liver enzymes are identical proteins encoded by two distinct mRNAs. Journal of Neurochemistry, 1999, 73(3): 903-912. |
| [24] | Tappaz ML. Taurine biosynthetic enzymes and taurine transporter:molecular identification and regulations. Neurochemical Research, 2004, 29(1): 83-96. DOI:10.1023/B:NERE.0000010436.44223.f8 |
| [25] | Almarghini K, Barbagli B, Tappaz M. Production and characterization of a new specific antiserum against the taurine putative biosynthetic enzyme cysteine sulfinate decarboxylase. Journal of Neurochemistry, 1994, 62(4): 1604-1611. |
| [26] | Jiang CJ, Shen PH, Yan B, Wu B. Biochemical characterization of a metagenome-derived decarboxylase. Enzyme and Microbial Technology, 2009, 45(1): 58-63. DOI:10.1016/j.enzmictec.2009.03.003 |
