王静, 翟璐, 张销寒, 李风玲, 肖亚中, 彭惠


安徽大学生命科学学院, 安徽省微生物与生物催化工程技术研究中心, 安徽 合肥 230039
收稿日期:2017-03-31;修回日期:2017-06-02;网络出版日期:2017-06-09
基金项目:国家自然科学基金(31470207);安徽省****科学基金(170808J04)
*通信作者:彭惠, Tel:+86-510-63861928;E-mail:pph0259@126.com
摘要:[目的]获得能高效降解大米生淀粉的α-淀粉酶。[方法]将来源Clostridium butyricum T-7的α-淀粉酶的淀粉结合结构域与能快速偏好性降解大米生淀粉的α-淀粉酶AmyP的催化结构域融合重组表达。[结果]融合蛋白AmyP-Clo保留了野生型AmyP催化优势的基础上,对大米生淀粉的比活为(373.9±8.4)U/mg,4 h内对5%大米生淀粉的最终降解率为(42.7±1.1)%,对大米生淀粉的最高结合率为(71.1±1.6)%,这些数据相比AmyP分别提高了3.1、2.8和1.3倍。[结论]融合蛋白AmyP-Clo能高效降解生大米淀粉,是一个具有优良应用价值的酶。
关键词: α-淀粉酶 大米生淀粉 融合表达 淀粉结合结构域
Hydrolysis of raw rice starch by a chimeric α-amylase engineered with α-amylase from Clostridium butyricum T-7
Jing Wang, Lu Zhai, Xiaohan Zhang, Fengling Li, Yazhong Xiao, Hui Peng


Engineering Technology Research Center of Microorganisms and Biocatalysis, School of Life Sciences, Anhui University, Hefei 230039, Anhui Province, China
Received 31 March 2017; Revised 2 June 2017; Published online 9 June 2017
*Corresponding author: Hui Peng, Tel:+86-510-63861928;E-mail:pph0259@126.com
Supported by the National Natural Science Foundation of China (31470207) and by the Science Fund for Distinguished Young Scholars of Anhui Province, China (170808J04)
Abstract: [Objective]To obtain efficient raw rice starch-digesting enzymes.[Methods]α-Amylase AmyP can hydrolyze raw rice starch efficiently. We constructed a chimeric α-amylase (AmyP-Clo) by fusion of the catalytic domain of AmyP and a starch binding domain of α-amylase from Clostridium butyricum T-7.[Results]AmyP-Clo retained the advantages of AmyP, and increased catalytic efficiency towards raw rice starch. The specific activity was (373.9±8.4) U/mg. The final hydrolysis degree was (42.7±1.1)% for the hydrolysis of 5% raw rice starch suspension after 4 h. The maximal fraction of bound protein was (71.1±1.6)%.[Conclusion]AmyP-Clo could efficiently hydrolyze raw rice starch.
Key words: α-amylase raw rice starch chimeric protein starch binding domain
大米是世界约50%人口的主粮,其重要性不言而喻。但是大米淀粉的深加工应用却远没有小麦、玉米等淀粉广泛[1]。一个重要原因是大米生淀粉的成分和结构远比小麦、玉米等淀粉复杂[2]。近几年,随着无需糊化(高温破坏生淀粉结构的耗能过程)就能直接降解生淀粉的酶的深入研究,生淀粉降解酶在方便食品、医药慢消化淀粉、多孔淀粉缓释剂、新型脂肪替代品等方面显示了广阔的应用前景[3-4]。但是大部分生淀粉降解酶无法降解大米生淀粉,或是降解能力很差。α-淀粉酶(EC 3.2.1.1) 是三大类生淀粉降解酶之一。目前报道的α-淀粉酶中只有十余个酶具有降解大米生淀粉的能力[5-7],而这十余个酶中仅有2个酶降解大米生淀粉的能力比降解小麦淀粉强,它们分别是来自蚯蚓(Eisenia foetida)的Amy Ⅱ [5]和来自Laceyellasacchari的淀粉酶[7]。其余的α-淀粉酶降解小麦、玉米等生淀粉的能力都远比大米强。此外,由于生淀粉具有致密的结晶结构,绝大部分生淀粉降解酶的降解速度都很慢, 常常需要24 h以上。已知的α-淀粉酶中仅有2个能快速降解生淀粉。1个来自Anoxybacillus flavothermus,降解5%的玉米生淀粉仅需2-3 h[8];另1个来自Bacillus licheniformis,降解1%的玉米生淀粉只需3 h[9]。缓慢的降解速度极大影响了酶的应用。
α-淀粉酶AmyP是前期工作中从海洋宏基因组文库中获得的一个新型淀粉酶[10]。它不仅对大米生淀粉具有绝对的偏好降解特性(比活是其他淀粉的4倍以上),而且对各种生淀粉的降解速度都很快,尤其是大米生淀粉,整个降解时间不超过4 h,起始30 min内效率极高。这种“快速偏好降解大米生淀粉”的特性从未在其他已知的生淀粉降解酶中发现过[6]。随后,我们发现AmyP虽然拥有一个淀粉结合结构域(SBD),但是该SBD对AmyP降解生淀粉的贡献较小[11]。本文报道通过替换SBD,将来源Clostridium butyricum的SBDClo[12]与AmyP△SBD融合,得到了一个具有更高的降解大米生淀粉能力的融合酶AmyP-Clo。
1 材料和方法 1.1 克隆、表达和纯化融合蛋白AmyP-Clo 来源C. butyricum的SBDClo基因由上海生工合成,在N和C端分别引入酶切位点EcoR Ⅰ和Xho Ⅰ。含有缺失SBD的AmyPΔSBD重组表达载体pET28a-AmyPΔSBD由本实验构建[11]。使用EcoR Ⅰ和Xho Ⅰ酶切SBDClo和pET28a-AmyPΔSBD,然后采用标准的分子克隆技术将片段连接,在表达载体pET28a上形成融合基因AmyP-Clo。质粒转化大肠杆菌BL21 (DE3) 后,16 ℃诱导表达,AmyP-Clo蛋白通过Ni2+亲和层析一步纯化。野生型AmyP的重组表达载体pET28a-AmyP由本实验室构建[10],同样的方法得到纯化的AmyP。
1.2 生淀粉酶活测定和蛋白浓度 采用3, 5-二硝基水杨酸(DNS)法测定生淀粉降解后释放出的还原糖的量[13],以葡萄糖为标准。一个标准反应体系包含50 μL适当稀释的纯酶和550 μL 10% (W/V)大米或玉米生淀粉。酶和淀粉均配置在50 mmol/L磷酸钠钾缓冲液(pH 7.5) 中,在40 ℃反应10 min,立刻放置于冰冷的0.3 mol/L Na2CO3溶液中,骤冷终止反应[14],随后4 ℃ 8000×g离心5 min,取上清测定还原糖的量。生淀粉使用前用MilliQ水洗涤5次,除去可能的还原糖。猪胰淀粉酶(pig pancreas, PPA,购自Sigma)的最适反应条件是pH 7.0 (50 mmol/L磷酸钠钾缓冲液)和37 ℃。一个标准酶活单位(U)定义为每分钟释放出1 μmol还原糖所需的酶量。蛋白浓度采用Bradford法测定,牛血清白蛋白作为标准。
1.3 温度和pH对活性的影响 在40 ℃测定不同pH值对大米生淀粉降解活性的影响。缓冲液为50 mmol/L乙酸-乙酸钠缓冲液(pH 4.0到6.0)、磷酸钠钾缓冲液(pH 6.0到8.5) 和甘氨酸-NaOH缓冲液(pH 8.5到10.0),间隔区间为0.5个pH。在最适pH 7.5下测定温度对降解活性的影响,温度范围为10 ℃到65 ℃,间隔区间为5 ℃。
1.4 大米和玉米生淀粉的酶解动力学 采用5%的大米或玉米生淀粉为底物,分别加入60 U的AmyP-Clo、AmyP和PPA,在这3个酶各自的最适pH和温度条件下进行酶解。加入0.2%的甲苯到反应混合液中防止可能的微生物污染[15],终反应体系为6 mL。反应混合液置于37 ℃或40 ℃的水浴锅中温和摇晃(100 r/min)反应4 h,期间间隔不同的时间取样,每次取样0.6 mL。取出的样品4 ℃ 8000×g离心5 min,取上清测定还原糖的量。
1.5 生淀粉吸附能力测定 往1 mL的50 mmol/L磷酸盐缓冲液(pH 7.5) 中分别加入0-90 mg的大米生淀粉和玉米生淀粉,振荡后形成悬浊液,然后分别加入1.8 μmol/L的AmyP-Clo和AmyP。为了避免酶解反应对吸附的干扰,将试管放置在冰水混合的水浴锅中温和摇晃(100 r/min)孵育1 h。然后4 ℃ 12000×g离心10 min,取100 μL上清用Bradford法测定蛋白含量,然后绘制吸附曲线。空白对照组用等体积缓冲液替代SBD蛋白。吸附率(%)=(总蛋白-上清中残留的蛋白)/总蛋白×100%。
2 结果和分析 2.1 融合蛋白AmyP-Clo构建、表达与纯化 委托上海生工直接合成SBDClo的基因861个碱基,然后运用常规的分子克隆技术,与AmyP△SBD的基因连接,成功构建了融合基因的重组表达质粒pET28a-AmyP-Clo。质粒转化大肠杆菌后,低温16 ℃诱导后实现可溶性表达,无包涵体。重组表达的AmyP-Clo蛋白已经融合了载体上的His-tag标签,直接采用Ni2+亲和层析获得纯品(图 1)。实验室前期构建的pET28a-AmyP质粒也同样表达纯化,获得野生型AmyP的纯酶。AmyP-Clo蛋白有779个氨基酸,理论分子量为85.7 kDa,从图 1中可见重组表达的AmyP-Clo分子量与理论分子量吻合。野生型AmyP的分子量为70.3 kDa。
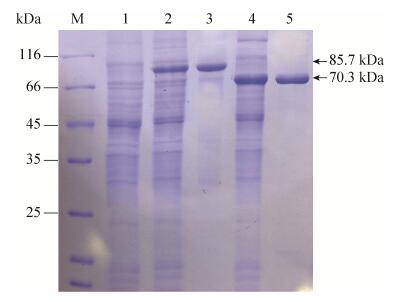 |
| 图 1 SDS-PAGE分析融合蛋白AmyP-Clo和野生型AmyP的表达与纯化 Figure 1 SDS-PAGE analysis of the chimeric AmyP-Clo and wild-type AmyP expression and purification. M: size marker proteins; lane 1: the cell lysates of E. coli harboring pET28a; lane 2: the cell lysates of E. coli harboring pET28a-AmyP-Clo; lane 3: the purified AmyP-Clo protein; lane 4: the cell lysates of E. coli harboring pET28a-AmyP; lane 5: the purified AmyP protein. |
| 图选项 |
2.2 pH和温度对融合蛋白AmyP-Clo降解活性的影响 以大米生淀粉为底物,AmyP-Clo的最适pH为7.0 (图 2-A),与野生型AmyP相比,降低了0.5。虽然最适pH发生了变化,但是酶活随pH变化的曲线整体上和野生型AmyP的曲线类似,差别较小。AmyP-Clo的最适温度没有改变,依然与野生型AmyP的一样,都为40 ℃ (图 2-B)。AmyP-Clo的酶活随温度变化的曲线整体上和野生型AmyP的曲线也类似,但是各温度下的酶活都比野生型AmyP更高一点。例如,30 ℃和50 ℃时AmyP-Clo的酶活为最高酶活的80%和75%;而同样温度时AmyP的酶活仅为最高酶活的53%和37%。这表明AmyP-Clo的温度适应性更好了,可以在更为宽泛的温度下降解生淀粉。
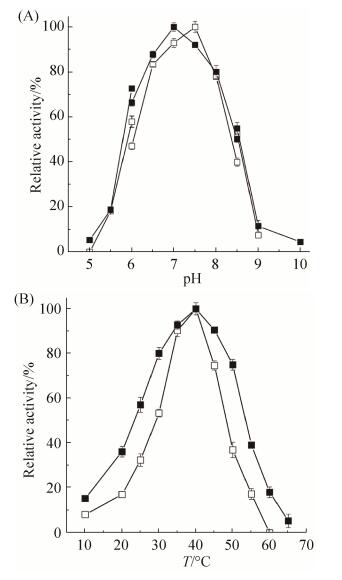 |
| 图 2 pH (A)和温度(B)对AmyP-Clo(■)和AmyP(□)生淀粉降解酶活的影响 Figure 2 Effects of pH (A) and temperature (B) on the activity of AmyP-Clo (■) and AmyP (□) towards raw starch. Values are the means±SD of three experiments (n=3). |
| 图选项 |
2.3 AmyP-Clo降解大米生淀粉和玉米生淀粉 在最适条件下,AmyP-Clo降解大米生淀粉和玉米生淀粉的比活分别为(373.9±8.4) U/mg和(150.2±4.4) U/mg。野生型AmyP在最适条件下对大米生淀粉和玉米生淀粉的比活分别为(118.5± 0.6) U/mg[6]和(39.6±1.4) U/mg[16]。因此,融合蛋白AmyP-Clo的比活力分别提高了3.1和3.8倍。
为了进一步衡量比较AmyP-Clo的降解能力,将AmyP-Clo、AmyP和PPA在酶量相同的条件下加到5%的大米生淀粉的悬浊液中,测定其在4 h内的酶解动力学如图 3-A所示。AmyP-Clo的酶解动力学曲线和AmyP类似,依然是典型的两阶段式,即先为快速降解阶段,然后是缓慢降解阶段。作为对照的典型生淀粉酶PPA则是持续增长模式。与AmyP相比,AmyP-Clo对大米生淀粉的降解能力提高2.8倍,4 h的最终降解率达到(42.7±1.1)%,而AmyP的降解率为(15.5±0.5)%。相同时间内的PPA的降解率仅为(11.1±0.3)%。
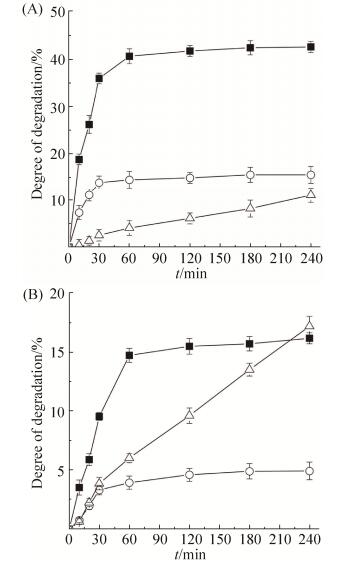 |
| 图 3 融合蛋白AmyP-Clo(■)、AmyP(○)和PPA(?)对大米(A)和玉米生淀粉(B)的降解 Figure 3 Ability of the chimeric AmyP-Clo (■), AmyP (○) and PPA (?) to digest raw rice (A) and corn (B) starches. Values are the means±SD of three experiments (n=3). |
| 图选项 |
这3个酶对5%的玉米生淀粉的酶解动力学如图 3-B所示。与AmyP相比,AmyP-Clo对玉米生淀粉的降解能力提高3.3倍,4 h的最终降解率达到(16.2±0.4)%,而AmyP的降解率为(4.9±0.2)%。在多种生淀粉中,PPA对玉米生淀粉的降解能力最强[17]。4 h时PPA的降解率为(17.2±0.6)%,略高于AmyP-Clo。
2.4 AmyP-Clo对大米生淀粉和玉米生淀粉的吸附能力 SBD的主要功能是吸附生淀粉颗粒,进而帮助酶在淀粉颗粒表面聚集,提高酶降解生淀粉颗粒的能力。AmyP-Clo对大米和玉米生淀粉的降解能力都比野生型AmyP显著提高,那么SBDClo对生淀粉的结合能力极可能比SBDAmyP强。为了验证融合蛋白AmyP-Clo降解生淀粉能力的提高确实是由于SBD结合能力的提高所致,在蛋白的摩尔质量相同的条件下,我们测定了AmyP-Clo和AmyP对大米(图 4-A)和玉米生淀粉(图 4-B)的结合能力。结果表明,融合蛋白AmyP-Clo对这两种生淀粉的结合能力确实都明显高于野生型AmyP的结合能力。对大米生淀粉而言,AmyP-Clo的最高结合率为(71.1±1.6)%,AmyP的最高结合率为(54±1.4)%;对玉米生淀粉而言,AmyP-Clo的最高结合率为(86.6±2.5)%,AmyP的最高结合率为(32.5±1.1)%。这说明,融合蛋白AmyP-Clo有了对生淀粉结合能力更强的SBDClo的帮助,能更有效地结合生淀粉,进而展示出比野生型AmyP更强的生淀粉降解能力。
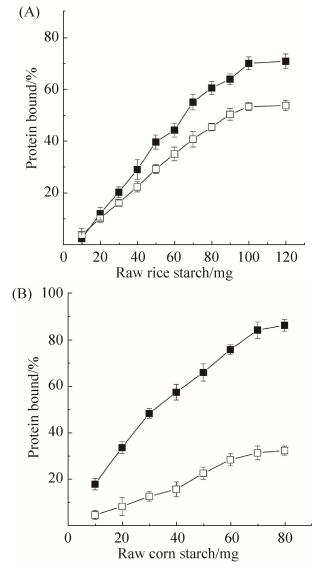 |
| 图 4 融合蛋白AmyP-Clo(■)和AmyP(□)对大米(A)和玉米生淀粉(B)的结合能力 Figure 4 Ability of the chimeric AmyP-Clo (■) and AmyP (□) to bind raw rice (A) and corn (B) starches. Values are the means±SD of three experiments (n=3). |
| 图选项 |
3 讨论 不同植物来源的生淀粉具有不同的颗粒大小、表面微结构和内部结晶模式,因此酶对不同来源的生淀粉的降解能力相差很大。α-淀粉酶AmyP在降解大米生淀粉方面具有几个显著优势:比活高、反应速度快、最适反应温度(40 ℃)远低于淀粉的糊化温度、不需要添加Ca2+。因此AmyP是一个具有应用价值的生淀粉降解酶。我们注意到将AmyP自身的SBD去除后,虽然AmyP△SBD对大米生淀粉的降解能力有所下降,但是下降幅度不算很大[11]。因此,理论上可以采用替换SBD的办法进一步提高其降解能力。能降解大米生淀粉的α-淀粉酶不多,综合考虑酶的最适作用温度和pH等因素之后,选择来源C. butyricum T-7的淀粉酶的SBDClo作为对象[12]。这个酶降解生淀粉的能力依次是玉米>小麦>大米>土豆。
与预计结果类似,融合蛋白AmyP-Clo对大米和玉米生淀粉的降解能力都得到了显著提高,分别为野生型AmyP的3.1和3.8倍。单就提高倍数而言,对玉米生淀粉的比活增加得更多,这应该是由于来源 C. butyricum T-7的淀粉酶的最适作用生淀粉是玉米。SBD在生淀粉降解过程中的功能是“捕获”底物。因此,催化结构域对最终的降解率、酶的作用方式的贡献更大。从图 3中可见,融合蛋白AmyP-Clo对大米和玉米生淀粉的酶解动力学过程与野生型AmyP的高度相似,保持了典型的两阶段式。只是由于AmyP-Clo的比活提高,整个降解率均提高。对优势底物大米生淀粉(5%),AmyP-Clo在4 h的最终降解率达到(42.7±1.1)%,是AmyP的2.8倍,是商品化的PPA的3.8倍。这表明AmyP-Clo是一个具有优良应用价值的酶。另外,AmyP-Clo对玉米生淀粉的降解率虽然也显著提高为AmyP的3.3倍,但是AmyP本身对玉米生淀粉的降解能力不强,AmyP-Clo在4 h的最终降解率略低于PPA。通过融合SBD提高酶的活力在多个酶中获得过成功。例如:大麦α-淀粉酶融合来源Aspergillus niger的葡糖糖化酶的SBD[18],Bacillus acidicola的α-淀粉酶融合来源Geobacillus thermoleovorans的α-淀粉酶的SBD[19],它们的酶活都显著增加。
为了验证底物结合能力与降解能力之间的关系,我们测定了融合蛋白AmyP-Clo和野生型AmyP对生大米和玉米的结合能力。AmyP-Clo确实对这两种生淀粉的结合能力都明显强于AmyP,对生大米的最高结合率是AmyP的1.3倍,对生玉米的最高结合率是AmyP的2.7倍。比较出人意外的是AmyP-Clo对生大米的最高结合率提高的倍数只有1.3倍,但是比活增加了3.1倍,最终降解率增加了2.8倍。这也暗示了如果能寻找到与生大米淀粉结合能力更佳的SBD,就有进一步提高酶活的可能性。
References
| [1] | Bemiller JN. Starch modification:challenges and prospects. Starch/St?rke, 1997, 49(4): 127-131. DOI:10.1002/(ISSN)1521-379X |
| [2] | Buléon A, Colonna P, Planchot V, Ball S. Starch granules:structure and biosynthesis. International Journal of Biological Macromolecules, 1998, 23(2): 85-112. DOI:10.1016/S0141-8130(98)00040-3 |
| [3] | Sun HY, Zhao PJ, Ge XY, Xia YJ, Hao ZK, Liu JW, Peng M. Recent advances in microbial raw starch degrading enzymes. Applied Biochemistry and Biotechnology, 2010, 160(4): 988-1003. DOI:10.1007/s12010-009-8579-y |
| [4] | Robertson GH, Wong DWS, Lee CC, Wagschal K, Smith MR, Orts WJ. Native or raw starch digestion:a key step in energy efficient biorefining of grain. Journal of Agricultural and Food Chemistry, 2006, 54(2): 353-365. DOI:10.1021/jf051883m |
| [5] | Ueda M, Asano T, Nakazawa M, Miyatake K, Inouye K. Purification and characterization of novel raw-starch-digesting and cold-adapted α-amylases from Eisenia foetida. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 2008, 150(1): 125-130. DOI:10.1016/j.cbpb.2008.02.003 |
| [6] | Lei Y, Peng H, Wang Y, Liu YT, Han F, Xiao YZ, Gao Y. Preferential and rapid degradation of raw rice starch by an α-amylase of glycoside hydrolase subfamily GH13_37. Applied Microbiology and Biotechnology, 2012, 94(6): 1577-1584. DOI:10.1007/s00253-012-4114-0 |
| [7] | Lomthong T, Chotineeranat S, Kitpreechavanich V. Production and characterization of raw starch degrading enzyme from a newly isolated thermophilic filamentous bacterium, Laceyella sacchari LP175. Starch/St?rke, 2015, 67(3/4): 255-266. |
| [8] | Tawil G, Viks?-Nielsen A, Rolland-Sabaté A, Colonna P, Buléon A. Hydrolysis of concentrated raw starch:a new very efficient α-amylase from Anoxybacillus flavothermus. Carbohydrate Polymers, 2012, 87(1): 46-52. DOI:10.1016/j.carbpol.2011.07.005 |
| [9] | Bo?i? N, Ruiz J, López-Santín J, Vuj?i? Z. Production and properties of the highly efficient raw starch digesting α-amylase from a Bacillus licheniformis ATCC 9945a. Biochemical Engineering Journal, 2011, 53(2): 203-209. DOI:10.1016/j.bej.2010.10.014 |
| [10] | Liu Y, Lei Y, Zhang XC, Gao Y, Xiao YZ, Peng H. Identification and phylogenetic characterization of a new subfamily of α-amylase enzymes from marine microorganisms. Marine Biotechnology, 2012, 14(3): 253-260. DOI:10.1007/s10126-011-9414-3 |
| [11] | Peng H, Zheng YY, Chen MJ, Wang Y, Xiao YZ, Gao Y. A starch-binding domain identified in α-amylase (AmyP) represents a new family of carbohydrate-binding modules that contribute to enzymatic hydrolysis of soluble starch. FEBS Letters, 2014, 588(7): 1161-1167. DOI:10.1016/j.febslet.2014.02.050 |
| [12] | Tanaka T, Ishimoto E, Shimomura Y, Taniguchi M, Oi S. Purification and some properties of raw starch-binding amylase of Clostridium butyricum T-7 isolated from mesophilic methane sludge. Agricultural and Biological Chemistry, 1987, 51(2): 399-405. |
| [13] | Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar. Analytical Chemistry, 1959, 31(3): 426-428. DOI:10.1021/ac60147a030 |
| [14] | Tahir R, Ellis PR, Butterworth PJ. The relation of physical properties of native starch granules to the kinetics of amylolysis catalysed by porcine pancreatic α-amylase. Carbohydrate Polymers, 2010, 81(1): 57-62. DOI:10.1016/j.carbpol.2010.01.055 |
| [15] | Hamilton LM, Kelly CT, Fogarty WM. Raw starch degradation by the non-raw starch-adsorbing bacterial alpha amylase of Bacillus sp. IMD 434. Carbohydrate Research, 1998, 314(3/4): 251-257. |
| [16] | Peng H, Lei Y, Liu YT, Wang Y. Degradation of raw corn starch by an α-amylase (AmyP) from marine environment. China Biotechnology, 2012, 32(7): 79-83. (in Chinese) 彭惠, 雷寅, 刘源涛, 汪颖. 海洋环境来源的淀粉酶AmyP对生玉米淀粉的降解特性. 中国生物工程杂志, 2012, 32(7): 79-83. |
| [17] | Iefuji H, Chino M, Kato M, Iimura Y. Raw-starch-digesting and thermostable α-amylase from the yeast Cryptococcus sp. S-2:purification, characterization, cloning and sequencing. Biochemical Journal, 1996, 318(3): 989-996. DOI:10.1042/bj3180989 |
| [18] | Juge N, N?hr J, Le Gal-Co?ffet MF, Kramh?ft B, Furniss CSM, Planchot V, Archer DB, Williamson G, Svensson B. The activity of barley α-amylase on starch granules is enhanced by fusion of a starch binding domain from Aspergillus niger glucoamylase. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2006, 1764(2): 275-284. DOI:10.1016/j.bbapap.2005.11.008 |
| [19] | Parashar D, Satyanarayana T. A chimeric α-amylase engineered from Bacillus acidicola and Geobacillus thermoleovorans with improved thermostability and catalytic efficiency. Journal of Industrial Microbiology & Biotechnology, 2016, 43(4): 473-484. |
