黄胜1, 虞沂2


1. 湖北民族学院医学院, 湖北 恩施 445000;
2. 武汉大学药学院, 组合生物合成与新药发现教育部重点实验室, 湖北 武汉 430072
收稿日期: 2015-09-21;修回日期:2015-12-10; 网络出版日期:2015-12-16
基金项目:国家自然科学基金(31570033)
通讯作者:虞沂,Tel/Fax:+86-27-68752491;E-mail:yu_yi@whu.edu.cn
摘要: 二硫吡咯酮类抗生素是一类具有独特的吡咯酮二硫杂环戊二烯(4H-[1,2] 二硫 [4,3-b] 吡咯-5-酮)骨架的化合物的总称。基于N-7位酰基侧链的不同以及N-4位是否含有甲基,可分为N-methyl-Nacylpyrrothine、N-acylpyrrothine和thiomarinols等类别。迄今为止,已有27种该类化合物被报道,重要代表包括全霉素(holomycin)、硫藤黄菌素(thiolutin)、金霉素(aureothricin)以及最近发现的thiomarinols。就生物活性而言,二硫吡咯酮类抗生素具有广谱的抗细菌活性,对多种微生物,包括革兰氏阴性菌、革兰氏阳性菌以及寄生虫都有较好的杀灭活性。甚至一些二硫吡咯酮衍生物表现出较强的抗肿瘤活性。近几年来,多个二硫吡咯酮类抗生素的生物合成基因簇相继被报道,其生物合成机理也逐步被阐明。本文将针对目前国内外二硫吡咯酮类抗生素的生物合成研究进展,以及在组合生物合成与代谢工程领域所取得的成果进行综述,旨在为通过合成生物学的方法创造结构新颖、高效低毒的“非天然”二硫吡咯酮类化合物提供理论借鉴。
关键词: 二硫吡咯酮代谢工程组合生物合成生物合成
Biosynthesis and metabolic engineering of dithiolopyrrolone-A review
Sheng Huang1, Yi Yu2


1. Medical College, Hubei University for Nationalities, Enshi 445000, Hubei Province, China;
2. Key Laboratory of Combinatory Biosynthesis and Drug Discovery, Ministry of Education, School of Pharmaceutical Sciences, Wuhan University, Wuhan 430072, Hubei Province, China
Abstract:Dithiolopyrrolones are a family of antibiotics that possess the unique pyrrolinonodithiole (4H-[1, 2] dithiolo [4, 3-b] pyrrol-5-one) skeleton. This family of natural products can be divided into three subfamilies: N-methyl-Nacylpyrrothine, N-acylpyrrothine and thiomarinols. So far, more than 27 members of this group of natural products have been reported including the well-known antibiotics holomycin, thiolutin, aureothricin and recently isolated thiomarinols. Dithiolopyrrolones exhibit relatively broad-spectrum antibiotic activities against many Gram-positive, Gram-negative bacteria and parasites. Some dithiolopyrrolones even have antitumor activities. In recent years, several dithiolopyrrolone biosynthetic gene clusters have been reported and their biosynthetic mechanisms have also been intensively studied. This review will give an overview about the biosynthesis and metabolic engineering of the dithiolopyrrolone natural products, and provides references to guide the creation of hybrid "unnatural" dithiolopyrrolones with better bioactivity and low toxicity by synthetic biology.
Key words: dithiolopyrrolonemetabolic engineeringcombinatorial biosynthesisbiosynthesis
在过去的几个世纪里,天然产物一直倍受大家关注,随着多种抗生素发现策略的成功运用,20世纪40-60年代成为天然产物发现的“黄金时期”,期间发现的不同结构骨架特征的天然产物中,超过50%的已经应用于临床[1]。随着细菌抗药性问题日益突出,新一代抗生素的研究开发迫在眉睫,而已经发现的抗生素骨架为医药化学家改造提供了原始材料[2]。除了对已知的抗生素改造外,发现新作用靶点的抗生素,也是解决抗药性的一个重要途径。近年来,代谢工程和合成生物学的兴起为快速、高效开发新型低毒的抗生素提供了强有力技术保障。
二硫吡咯酮类化合物是一类具有很大潜在应用价值的天然抗生素,在革兰氏阴性和革兰氏阳性菌的代谢产物中都有发现,具有独特的吡咯酮-二硫杂环戊二烯(4氢-[1, 2]二硫[4,3-b] 吡咯-5-酮)发色团结构(图 1)[2]。65年前该家族的第一个成员金霉素(aureothricin,见图 1)从土壤细菌Streptomyces sp. 26A的代谢产物中分离,之后这类化合物吸引了众多研究课题组的注意[3]。不仅因为它们结构独特,抗细菌、抗真菌活性突出,更因为它们生物合成和调控机制非常独特。这个家族的很多成员表现出很强的广谱抗性,对革兰氏阳性、革兰氏阴性、酵母菌、真菌甚至是寄生虫都有较好的抑制活性[1]。二硫吡咯酮类天然产物按照其结构不同,可分为3个亚家族:N-methyl-N-acylpyrrothine 类二硫吡咯酮、N-acylpyrrothine类二硫吡咯酮和thiomarinols类二硫吡咯酮[2]。
 |
| 图 1. 常见的天然二硫吡咯酮类化合物 Figure 1. The structure of common dithiolopyrrolones. |
| 图选项 |
近年来,在众多科学家的努力下,在二硫吡咯酮类化合物的生物合成和代谢工程改造方面取得了较大的进展。本文针对近年来在这两方面的进展进行综述。
1 二硫吡咯酮类抗生素的生物合成机理研究 1.1 N-acylpyrrothine类二硫吡咯酮的生物合成研究全霉素(Holomycin,图 1)是N-acylpyrrothine类二硫吡咯酮的典型代表,所以这一类化合物也被称为全霉素类二硫吡咯酮[2]。Holomycin最早是Ernst等1961年从灰色链霉菌(Streptomyces griseus)的代谢产物中分离得到的一种黄色晶体物质[4]。1969年,Daehne从一株放线菌的代谢产物中分离了vD844,其与holomycin有着相同的化学组成和分子量,但结构却明显不同[5]。随后Okamura和Kenig等分别从Streptomyces sp. P662 和Streptomyces clavuligerus的突变株的代谢产物中分离到了holomycin[6, 7]。此外,从分离自中国胶州湾海洋沉积物样品中的海洋链霉菌Streptomyces sp. M095的代谢产物中也检测到了holomycin[8]。除了革兰氏阳性菌以外,革兰氏阴性菌中也有很多能产生holomycin类二硫吡咯酮。共生细菌Xenorhabdus. nematophilus XQ1能产生3种全霉素的衍生物:xenorhabdin 1、xenorhabdin 2和xenorhabdin 3[9](图 1)。通过生物活性测定介导的天然产物分离的方法发现海洋来源的革兰氏阴性菌Photobacterium halotolerans S2753也是holomycin产生菌[10]。通过基因组挖掘和代谢分析,鱼的病原菌Yersinia ruckeri的代谢产物中也发现了holomycin[11]。最近,在Pseudoalteromonas SANK73390突变株的代谢产物中检测到6个新的含有不同链长度脂肪酸的xenorhabdin衍生物[12]。此外,在X. bovienii的代谢产物中还检测到了含砜结构的氧化型二硫吡咯酮类化合物xenorxide 4和5,是迄今为止发现的Xenorhabdus属中唯一能产生氧化xenorhabdin衍生物的种[2, 13]。
虽然不断有新的N-acylpyrrothine类二硫吡咯酮类化合物被发现,但这类化合物生物合成途径的研究一直进展缓慢。2002年,Fuente等通过无细胞实验体系证明holomycin产生菌中存在一种催化N-7位的乙酰化的酰基转移酶,该酶在乙酰辅酶A存在的情况下催化去乙酰全霉素向全霉素不可逆的转化,说明该步反应可能发生在全霉素生物合成的后期[14]。进一步分析发现,该酶的活性在不同全霉素高产棒状链霉菌突变株中是呈比例变化的,随着发酵时间的增加而减少[14]。2010年以后,全霉素的生物合成取得了较大进展。
1.1.1 全霉素生物合成基因簇的克隆:2010年到2011年,Walsh课题组和我们课题组独立的分别报道了棒状链霉菌中holomycin的生物合成基因簇。他们都通过生物信息学分析和遗传学分析在棒状链霉菌的基因组定位了holomycin的生物合成基因簇,并分别被命名为hlm[15, 16]和hom[17](图 2)。2013年,Robles-Reglero等通过转录分析和蛋白组学分析也定位了全霉素的生物合成基因簇,并命名为hml[18]。该基因簇命名混乱,为了方便叙述,下文笔者统一使用hlm。hlm基因簇由13个基因组成,包括4个非核糖体多肽合成酶(NRPS)基因(hlmC、hmlE、hlmK和hlmL)、珠蛋白类似蛋白基因(hlmG)、酰基辅酶A脱氢酶基因(hlmB)、3个黄素依赖的氧化还原酶基因(hlmI、hlmD和hlmF)、 1个酰基转移酶基因(hlmA)、2个调节基因(hlmJ 和hlmM)和1个编码MFS转运蛋白的基因(hlmH)[15, 17]。 4个NRPS蛋白中,HlmC和HlmK是硫酯酶同源蛋白、HlmE是一个含有环化结构域-腺苷化结构域-肽酰载体蛋白结构域(Cy-A-T)的多酶复合体、HlmL是一个游离的缩合结构域[15, 17]。转录分析发现,该基因簇由7个转录单元构成,最大的转录单元从hlmA到hlmE[18]。
 |
| 图 2 已报道的二硫吡咯酮类化合物的生物合成基因簇比较 Figure 2. Comparison of characterized dithiolopyrrolone pathways. Shaded areas are conserved region. |
| 图选项 |
Li等通过对该基因簇中的功能基因hlmE和hlmF的失活实验和生物化学实验对其生物合成进行研究,发现基因失活菌株丧失了产生holomycin 的能力,说明hlm基因簇确实参与全霉素的生物合成[15]。他们对HlmE的腺苷化结构域活性进行测定,发现其只能活化L-半胱氨酸,而对其它氨基酸如L-甲硫氨酸等没有活性[15]。他们还在反应体系中检测到了L-Cys-L-Cys二肽,暗示HlmE可能有催化形成肽键的功能[15]。与此同时,我们课题组也通过生物信息学分析、对hlmI的基因敲除实验以及异源表达实验确定了holomycin的生物合成基因簇[17]。2013年,Robles-Reglero通过对棒状链霉菌ccaR::aph突变株的蛋白组学研究发现,相对于野生菌株而言,突变菌株中HlmD、HlmF和HlmG呈现出差异表达,并通过蛋白质质谱测定了它们的末端蛋白序列,接着通过生物信息学分析和对高产菌株中hlmF、hlmE和hlmA的基因失活实验定位了holomycin的生物合成基因簇[18]。hlm基因簇中没有发现与自我抗性相关的基因,但该基因簇在白色链霉菌和天蓝色链霉菌中成功的异源表达说明基因簇中负责holomycin生物合成的结构基因是完整的[17, 18]。
2013年,Qin等通过同源搜索,在holomycin 的产生菌Y. ruckeri的基因组发现了另一个holomycin 生物合成基因簇,并将其命名为hom[11]。hom基因簇中含有多个与棒状链霉菌中全霉素生物合成途径同源的蛋白(图 2)。对HlmD的同源蛋白编码基因hom6的基因失活实验证明hom基因簇负责全霉素的生物合成[11]。然而,hom基因簇中缺少hlm基因簇hlmI和hlmL编码蛋白的同源蛋白,他们可能分别负责催化形成二硫键和2个半胱氨酸的缩 合[11]。暗示革兰氏阴性菌中二硫键的形成机制可能与革兰氏阳性菌中不一样。
1.1.2 全霉素生物合成途径中部分酶的功能研究:2010年以来,Li等对holomycin生物合成过程中的关键酶进行了较深入的研究。2010年,他们通过体外生化实验发现,将重组的HlmA与乙酰辅酶A和去乙酰全霉(Holothin)素温育以后可以检测到holomycin的生成,说明HlmA是一个乙酰辅酶A转移酶[15]。同时他们还发现,HlmA也可以对还原型的二氢holothin进行乙酰化[15]。为了考察HlmA的底物识别性,他们还利用长链的酰基辅酶A作为底物进行测试,发现HlmA可以识别部分长链酰基辅酶A(如己酰、辛酰和棕榈酰辅酶A)并进行转化,只是效率相对来说要低一些[15]。然而,长链酰基化的holothin在棒状链霉菌的发酵液中并没有检测到,却能在其它产生菌的代谢产物中检测到,可能是由于这些长链脂肪酸或者酰基辅酶A在棒状链霉菌的底物池里面浓度很低,不利于HlmA的利用[15]。
2013年,Li等对hlm基因簇中的hlmI进行了研究。生物信息学的分析显示HlmI是一个黄素蛋白,与硫氧还蛋白氧化还原酶DepH和GliT同源,可能催化还原型的全霉素或去乙酰全霉素生成holomycin或holothin[16]。当HlmI与FADH2和还原型的holomycin或holothin温育时,在氧气存在的情况下可以检测到holomycin或holothin的快速合成[16]。说明HlmI是一个FAD依赖的二硫醇氧化酶,利用氧气作为氧化试剂,在全霉素生物合成后期催化分子内二硫键的形成[16]。随后他们构建了hlmI的缺失突变体,发现突变体对全霉素更加敏感。在缺失突变体的代谢产物中发现了含有单个或者双硫醇甲基化的产物以及分子间形成二硫键的产物[16]。说明ΔhlmI突变株产生的硫醇中间体可能活性很高甚至有毒性,宿主采取对硫醇基团的失活来保护自己[16],而hlmI的存在也可能作为一种自我保护机制。
1.1.3 N-acylpyrrothine类二硫吡咯酮的生物合成途径:基于对全霉素生物合成基因簇的生物信息学分析和其生物合成途径中关键酶的表征,我们推导了N-acylpyrrothine类二硫吡咯酮的生物合成途径(图 3)。
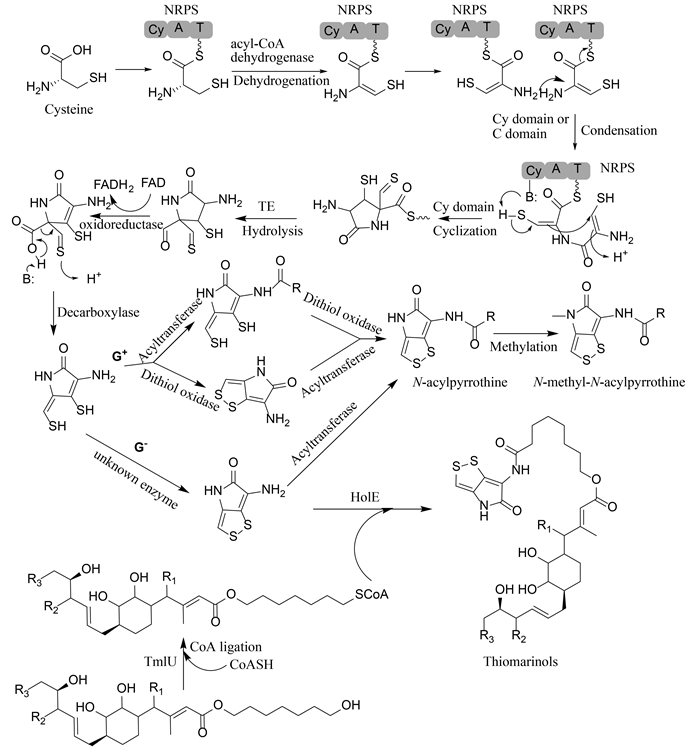 |
| 图 3 二硫吡咯酮类化合物的生物合成途径汇总 Figure 3. The overall biosynthetic pathways of dithiolopyrrolones. G+: Gram-positive bacteria; G–: Gram-negative bacteria. |
| 图选项 |
N-acylpyrrothine类二硫吡咯酮生物合成的第一步是L-Cys结合到HlmE的腺苷化结构域上,并被活化成L-Cys-AMP的形式,HlmE是一个含有三结构域的单模块NRPS,其缺乏对全霉素生物合成所必需的第2个L-Cys活化的腺苷化结构域,暗示HlmE可能是一个反复使用的酶,或者以二聚体的形式发挥功能[1, 2, 19]。接着,结合在HlmE的PCP 上的Cys可能先经过hlmB编码的酰基辅酶A脱氢酶从Cys上消除2个H原子形成双键、再由游离的C结构域(HlmL)催化两分子脱氢的Cys形成肽键、随后HlmE的环化(Cy)结构域催化形成全霉素的第一个环,这一步环化也可能是自发进行的。接着修饰后的二肽可能由硫酯酶HlmC从HlmE-二肽中间体上水解释放下来。水解下来的中间体经过HlmF催化发生脱羧反应。HlmF是一个黄素蛋白,属于黄素酶超家族,能催化脱羧反应,和那些参与羊毛硫氨酸抗生素生物合成的酶一样[1, 2, 19]。 然而,这一步脱羧反应还没得到体外生化实验的证实。脱羧中间体接下来经过氧化还原酶HlmD还原和自发的修饰反应生成还原型holothin[1, 2, 19]。最后还原型holothin经氧化形成二硫键和酰基化加载酰基侧链[1, 2, 19](图 3)。但在革兰氏阳性菌和革兰氏阴性菌中二硫键的形成机制不一样。在革兰氏阳性菌中,还原型的去乙酰全霉素在经过HlmI的催化形成二硫键[16],在革兰氏阴性菌中,由于缺少HlmI的同源蛋白,二硫键的形成机制还有待进一步研究。
1.2 N-methyl-N-acylpyrrothine类二硫吡咯酮的生物合成研究硫藤黄菌素(Thiolutin,图 1)是N-methyl-N-acylpyrrothine类二硫吡咯酮的典型代表之一,所以这类化合物也被称为硫藤黄菌素类[2]。1948 年,Umezawa等从Streptomyces sp. 26A的代谢产物分离了第1个二硫吡咯酮类抗生素——金霉 素[3]。两年后,辉瑞集团的一个研究团队从白色链霉菌(Streptomyces albus)的代谢产物中分离得到了thiolutin[20]。结构对比发现金霉素是硫藤黄菌素的3-丙酰胺的衍生物(图 1)[21]。此后,研究者们发现多种放线菌菌株都可以产生thiolutin和金霉 素[2]。2002年,Lamari等从稀有放线菌阿尔及利亚糖丝菌Saccharothrix algeriensis的代谢产物中分离了至少5种二硫吡咯酮类抗生素,包括thiolutin和它的4种衍生物:isobutanoylpyrrothine (ISP)、 butanoylpyrrothine (BUP)、 senecioylpyrrothine (SEP)和 tigloylpyrrothine (TIP)[22](图 1)。链霉菌Streptomyces pimprina也是ISP的产生菌[23]。革兰氏阴性菌如共生细菌Xenorhabdus属也能够产生thiolutin类二硫吡咯酮天然产物。1991年,Mcinerney 和他的同事从X. nematophilus XQ1的发酵液中发现了2种新的N-甲基二硫吡咯酮类化合物xenorhabdin 4和xenorhabidin 5[9](图 1)。
N-methyl-N-acylpyrrothine类二硫吡咯酮是二硫吡咯酮家族成员最丰富的一类,前期研究者们对其生物合成做出了初步的探索,但直到最近thiolutin的生物合成基因簇才被克隆。
1.2.1 Thiolutin生物合成基因簇的克隆:2014 年,Saker等利用无细胞提取物体系证实了Sa. algeriensis体内含有2个酰基转移酶分别负责乙酰基和苯甲酰基的加载[24]。接着他们通过蛋白分级分离提纯得到了2个酰基转移酶蛋白,通过蛋白质末端测序最后在Sa. algeriensis的基因组中定位相应的基因并命名为actA和actB[24]。随后,我们课题组通过基因组挖掘和异源表达克隆了来源于Sa. algeriensis的thiolutin生物合成基因簇,并命名为dtp (图 2)。对dtp基因簇的生物信息学分析发现,基因簇中包含holothin核心合成所需的全部基因,以及其它一些修饰酶基因[25],其中也包含Saker发现的actA。而actB不在基因簇内部或者基因簇的侧翼。生物信息学分析显示基因簇中的orf-4编码一个甲基转移酶,我们将其在大肠杆菌BL21里面进行超量表达并纯化,纯化后蛋白与holomycin和SAM反应,反应体系中没有检测到thioluthin的产生[25]。进而将该基因簇导入白色链霉菌里面进行异源表达时我们惊奇地发现,在发酵提取物中只检测到了全霉素[25]。将全霉素和SAM (S-adenosyl-L-methionine)与Sa. algeriensis的无细胞提取物进行温育时,可以检测到thiolutin的快速产生,当以pyrrothine (图 1)作为底物时没有检测到转化,说明N-4甲基来源于SAM和负责N-4甲基化可能处于thiolutin类二硫吡咯酮化合物生物合成的后期[25]。以上结果同时也说明编码负责N-4甲基化和N-7位其它酰基化酶的基因位于基因簇外部。
基于这种修饰基因位于基因簇以外的现象,我们提出了二硫吡咯酮类化合物生物合成基因簇水平转移的假说。我们认为,thiolutin 类二硫吡咯酮类化合物的生物合成基因簇来源于holomycin 产生菌基因簇的水平转移。转移后的宿主可能含有一个底物特异性广泛的N-甲基转移酶,例如酰胺N-甲基转移酶,可以识别holomycin或者其衍生物作为非特异性的底物进行甲基化从而产生thiolutin类的二硫吡咯酮类化合物[25]。支持我们这种进化观点现象有2个:(1) 二硫吡咯酮类化合物的酰胺甲基化修饰可以增强其膜通透性从而降低对宿主的毒性,这种修饰结果在gliotoxin和N-methylated peptides的生物合成中已有报道[25, 26];(2) X. nematophilus XQ1既可以产生硫藤黄菌素类化合物也可以产生全霉素类化合物[9],说明X. nematophilus XQ1中甲基转移酶的特异性不是很强,只能对部分进行修饰。
1.2.2 N-methyl-N-acylpyrrothine类二硫吡咯酮的生物合成途径: 根据基因簇里基因的生物信息学分析以及不同课题组的研究结果,我们对N-methyl-N-acylpyrrothine类二硫吡咯酮的生物合成途径进行了推导和总结。我们认为,N-methyl-N-acylpyrrothine类二硫吡咯酮生物合成的前面步骤和N-acylpyrrothine的生物合成是一致的。随着一系列的反应首先形成N-acylpyrrothine,然后经甲基化形成N-methyl-N-acylpyrrothine (图 3)。
1.3 Thiomarinols类二硫吡咯酮的生物合成研究Thiomarinols是二硫吡咯酮天然产物比较独特的一个亚类,它是一类高效的NRPS/PKS杂合天然抗生素,由二硫吡咯酮核心结构通过酰胺键和假单胞菌酸同系物连接。组成假单胞菌酸的单孢酸也是聚酮抗生素莫匹罗星的重要组成部分[27]。
1993年,日本课题组首次报道了从海水里分离的革兰氏阴性菌Pseudoalteromonas sp. nov. SANK 73390 代谢产物thiomarinol A的发酵和分离[27]。通过NMR分析确认thiomarinol A的结构是假单胞菌酸和去乙酰全霉素的杂合(图 1)[27]。随后,thiomarinol A的6个新的同系物thiomarinol B–G也从该菌株的代谢产物中得到分离[28, 29](图 1)。
1.3.1 Thiomarinols的生物合成基因簇:2010年,Fukuda等在一个新型的质粒pTML1发现了2个独立的基因簇:二硫吡咯酮类化合物的生物合成基因簇和假单胞菌酸的生物合成基因簇。假单胞菌酸生物合成基因簇编码一个典型的反式-AT聚酮合酶(Trans-AT PKS)装配线[12]。pTML1上的另一个基因簇与Y. ruckeri中负责holothin的结构单元合成的基因很相似,作者将其命名为hol[12](图 2)。hol基因簇由7个基因组成,编码的蛋白有三结构域的NRPS(HolA,Cy-A-T)、氧化还原酶HolB、硫酯酶HolC、酰基辅酶A脱氢酶HolD、酰基转移酶HolE、黄素依赖的氧化酶HolF和脱羧酶 HolG[12]。holA的失活突变株完全丧失了thiomarinol 的产生能力,只产生假单胞菌酸的类似物marinolic acid,说明hol基因簇负责thiomarinol结构中holothin母核的生物合成。同时,中断假单胞菌酸的生物合成途径以后,突变株只产生xenorhabdins 的衍生物,说明pTML1上的2个基因簇共同负责thiomarinol的生物合成[12]。也既thiomarinol的生物合成途径是2个途径的杂合。
1.3.2 Thiomarinols生物合成途径:在S. clavuligerus 和 Y. ruckeri中,全霉素生物合成后期,乙酰转 移酶利用乙酰辅酶A作为底物介导酰胺键的合 成[11, 15]。在thiomarinol生物合成途径中,推测TmlU是一种ATP依赖的连接酶,tmlU的失活完全终止了thiomarinols的生物合成,同时导致了xenorhabdins 和 marinolic acids的积累[12, 30]。给假 单胞菌酸途径失活的突变株喂养anhydroornithine 和anhydrolysine,或者将假单胞菌酸途径失活的突变株和hol基因簇失活的突变菌株共培养也可以产生thiomarinol H和thiomarinol J以及其它衍生 物[30]。暗示在thiomarinols的生物合成过程中TmlU的功能是连接假单胞菌酸和去乙酰全霉素形成thiomarinols[5, 30]。Xenorhabdins及其衍生物的产生暗示HlmE的同源蛋白HolE是酰胺键形成酶的第二个拷贝,负责酰基辅酶A和holothin之间酰胺键的形成以产生xenorhabdins[5, 30]。
2015年,Li等的研究结果与上面的推测相反,他们发现tmlU编码的蛋白是一个辅酶A连接酶,其将假单胞菌酸活化为硫酯形式进而作为酰基转移酶HolE酰胺化的底物[31]。TmlU倾向于识别复杂的酰基酸为底物,而HolE的底物识别范围相对很广阔,可以接受一系列的酰基辅酶A和胺基作为底物[31]。
综上,我们可以看出thiomarinols的生物合成途径由两部分组成:假单孢酸途径和holothin途径。两条途径首先各自合成各自的母核,假单胞菌酸合成以后被TmlU活化成辅酶A的形式,然后在酰基转移酶HolE的作用下将其和Holothin母核通过酰胺键连接起来(图 3)。
2 二硫吡咯酮类抗生素的代谢工程应用 科学家们认为具有RNA酶抑制剂活性的二硫吡咯酮类化合物,如全霉素和硫藤黄菌素,是新 一代RNA聚合酶相关的药物设计的前体。Yakushiji 等最近通过将全霉素掺入到几个myxopyronin骨架,开发了一系列新的细菌RNA聚合酶抑制 剂[32]。其中的一个对革兰氏阳性菌有良好的抗菌活性,这意味着使用吡咯啉作为一个组成部分来制造杂合型药物是新药开发的重要方向[32]。而对二硫吡咯酮类化合物的生物合成途径及其调控机制研究的不断深入,为通过组合生物合成和代谢工程手段对原始产生菌株进行导向性改造获得高效低毒的新的杂合化合物提供了坚实的理论基础。
2.1 复杂调控系统的研究为代谢工程改造提供依据研究发现,全霉素产生菌株Streptomyces sp. P662和S. clavuligerus中全霉素生物合成途径和头孢霉素C的生物合成途径存在底物竞争[6, 7]。而且在棒状链霉菌中,全霉素的生物合成途径和克拉维酸的生物合成途径存在交叉对话[14]。棒状链霉菌的一个全霉素高产突变株中一个硫氰酸蛋白酶类似的蛋白过量表达,中断编码这个蛋白的基因以后全霉素产量大幅度减小[33]。argR是一个在精氨酸生物合成中普遍保守的阻遏基因,S. clavuligerus 的argR中断突变株S. clavuligerus CZR全霉素的产量大幅提高[34]。通过适应性进化也可以筛选到全霉素高产的突变株[35]。2012年,Charusanti等对适应性进化获得的全霉素高产突变株进行基因型分析发现其丢失了1.8 Mb的大质粒pSCL4[35]。他们的进一步研究发现,pSCL4对全霉素的产量有很大的影响,全霉素产量的提高与pSCL4有极大的关系。进而他们推测pSCL4上存在全霉素生物合成途径的阻遏物[19]。afsR和afsA编码的蛋白是众所周知的抗生素生物合成的激活因子,向野生型的S. clavuligerus中再导入一个拷贝的afsR 基因可以激活正常情况下沉默的全霉素的生物合成基因簇的表达[36]。以上这些研究结果为我们对全霉素产生菌进行合理的改造获得产量更高的突变菌株提供了理论依据。我们可以综合以上各方面结果,通过代谢工程手段,中断克拉维酸的生物合成途径以减少竞争途径,增加半胱氨酸前体的供应,中断精氨酸生物合成途径中的argR基因,提高asfR等正调控因子的拷贝数,减少或除去产生菌株中pSCL4质粒的拷贝数以除去全霉素生物合成途径的阻遏因子等,获得产量更高的产生菌株,为进一步的途径改造提供好的底盘细胞。
2.2 前体介导天然产物的合成(Precursor-Directed Biosynthesis,PDB)为代谢工程改造提供方向前体介导天然产物的合成是一种高效的用于天然产物类似物生物合成的工具[37]。前体介导天然产物的合成利用天然生物合成途径的灵活性,接受天然底物的类似物用于产物的合成[38]。天然产物同系物的产生对天然产物活性的微调有着十分重要的作用同时也可以提高天然产物作为药物的可能性。因而,通过PDB产生新的二硫吡咯酮类化合物的类似物以寻找到人们所需要的化合物是获得有用新化合物的一个重要途径。
在对全霉素产量控制机制研究的同时,科学家们也对通过各种途径获得新的二硫吡咯酮类化合物做出了多方面尝试。Bouras等通过改变Sa. algeriensis培养基中氨基酸的成分和浓度研究氨基酸对各种二硫吡咯酮类化合物产量的影响开启了这类化合物前体介导的天然产物合成策略的先河[39]。2007-2011年,Merrouche等发现,向Sa. algeriensis的培养基中加入有机酸可以诱导新的二硫吡咯酮类化合物的合成。他们向培养基中分别加入苯甲酸、戊酸、肉桂酸和山梨醇,最后获得了formylpyrrothine、valerylpyrrothine、isovalerylpyrrothine、crotonylpyrrothine、sorbylpyrrothine、2-hexenylpyrrothine和2-methyl-3-pentenylpyrrothine等7种新的带有不同酰基侧链的二硫吡咯酮类化合物[40, 41, 42](图 4)。
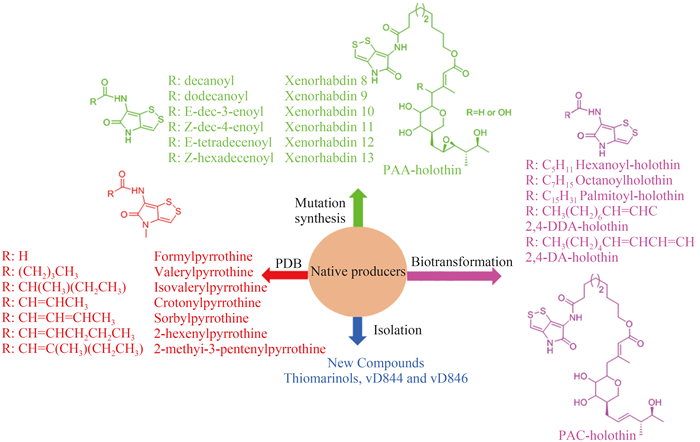 |
| 图 4 通过代谢工程获得的“非天然”的天然抗生素 Figure 4. Metabolic engineering obtained “unnatural” natural antibiotics. PDB: Precursor-Directed Biosynthesis. |
| 图选项 |
这些结果说明Sa. algeriensis中二硫吡咯酮类化合物生物合成途径的高度灵活性,通过PDB可以有效的利用这种灵活性。同时,这种灵活性也指导我们通过代谢工程手段对其合成途径进行改造,在发酵过程中加入更多种类的含有羧基的化合物,获得更多新型衍生物。
2.3 突变合成是获得新的二硫吡咯酮类化合物的重要方法突变合成(mutational biosynthesis)是指通过基因失活的方法得到的突变株将丧失产生原始抗生素的能力,然后在突变株发酵液中加入天然的或化学合成的前提化合物从而在突变株的发酵过程中产生新的结构类似的抗生素或者突变株积累一些野生型菌株所不能积累的抗生素的中间体化合物[43]。
2011年,Fukuda等在研究thiomarinols的生物合成时发现,在假单胞菌酸的生物合成途径中断突变株的代谢产物中检测到了6种新的二硫吡咯酮类化合物:xenorhabdin 8、9、10、11、12和13[12, 30](图 4)。接着他们通过向突变株喂养anhydroornithine和anhydrolysine获得了新的thiomarinol衍生物thiomarinol H和thiomarinol J [12, 30]。同时他们还发现将假单胞菌酸途径失活的突变株和hol基因簇失活的菌株共培养也可以产生thiomarinols或其衍生物[12, 30]。这些结果显示,突变合成是一种有效的产生新的二硫吡咯酮类化合物的方法,利用基因失活突变株结合PDB的方法,可能获得更多的衍生物。
2.4 体外生物转化是体内代谢工程的延伸体外生物转化是研究酶的性质的重要手段也是合成化合物衍生物的重要方法。2010年,Li等采用生物转化的策略在体外利用全霉素生物合成途径中的酰基转移酶HlmA合成了3种新的长链全霉素衍生物:hexanoyl-holothin、octanoyl-holothin和palmitoyl-holothin[15](图 4)。这是首次通过体外生物转化的方式获得新的二硫吡咯酮类化合物。2014年,Saker等通过无形细胞体系反应合成了苯甲酰-pyrrolthine[24]。2015年,Dunn等在研究thiomarinols生物合成途径的关键酶TmlU和HolE的活性及相互关系时,通过体外生物转化的方式合成了3种新的二硫吡咯酮类化合物 2,4-DDA-holothin、2,4-DA-holothin和PCA-holothin[31](图 4)。所以可以利用TmlU/HolE酶系作为工程手段去产生新的杂合分子。
TmlU和HolE作用关系的阐明为我们进一步利用代谢工程,以hol基因簇及tmlU作为载体,与多种含有羧基官能团的抗生素的生物合成途径进行杂合,产生更多的杂合抗生素,实现组合生物合成。除此之外,通过定点突变、体外分子进化等重要分子生物学手段对TmlU和HolE进行改造,通过筛选获得底物特异性强的或者底物特异性很宽广的组合,以提高体外生物转化效率和产物的多样性。
3 展 望研究显示二硫吡咯酮类化合物的生物合成途径可以通过代谢进行改造。不论是利用前体介导的生物合成策略还是利用突变合成策略,或者采用生物转化的途径都可以获得新的二硫吡咯酮类化合物。这些研究一方面增加了二硫吡咯酮类化合物家族成员的数目,另一方面也增加了我们对这个家族化合物生物合成的认识,为我们进一步通过组合生物合成和代谢工程的方法获得更多的“杂合”天然产物提供理论依据。
对二硫吡咯酮类天然产物的进一步研究可以集中在吡咯发色团的形成、TmlU/HolE酶系的工程改造等方面,只有在深入了解这一类天然产物生物合成机理的基础上,才可以更好地利用代谢工程和合成生物学手段制造多种高效、低毒的新型的杂合抗生素。Thiomarinol的生物合成途径给了我们很多启发,二硫吡咯酮生物合成基因簇较小,易于操作,可以将其置于适于天然产物生产的底盘微生物中,通过增加其他来源的化学基团编码生物元件,来合成结构上更丰富的“非天然”天然二硫吡咯酮类似物。另外,还可以优化TmlU/HolE酶系,通过体外生物转化的方法获得更多衍生物。当然,对代谢途径的改造和灵活运用还需要更多的理论支持,需要广大科研工作者共同努力。
参考文献
| [1] | Li B, Wever WJ, Walsh CT, Bowers AA. Dithiolopyrrolones: biosynthesis, synthesis, and activity of a unique class of disulfide-containing antibiotics. Natural Product Reports, 2014, 31: 905-923. |
| [2] | Qin ZW, Huang S, Yu Y, Deng H. Dithiolopyrrolone natural products: isolation, synthesis and biosynthesis. Marine Drugs, 2013, 11: 3970-3997. |
| [3] | Umezaw H, Maeda K, Kosaka H. Isolation of a new antibiotic substance, aureothricin from a strain of Strepomyces. The Japanese Medical Journal, 1948, 1: 512-517. |
| [4] | Ernst G, Vladimi P, Ernst V. (1961). Holothin and derivatives thereof. USA: US3014922. 1959. |
| [5] | Daehne WV, Godtfredsen W, Tybring L, Schaumburg K. New antibiotics containing the 1,2-dithiolo [4, 3-b] pyrrole ring system. The Journal of Antibiotics, 1969, 22: 233-236. |
| [6] | Okamura K, Soga K, Shimauchi Y, Ishikura T, Lein J. Holomycin and N-propionylholothin, antibiotics produced by a cephamycin C producer. The Journal of Antibiotics, 1977, 30: 334-336. |
| [7] | Kenig M, Reading C. Holomycin and an antibiotic (MM 19290) related to tunicamycin, metabolites of Streptomyces clavuligerus. The Journal of Antibiotics, 1979, 32: 549-554. |
| [8] | Hou YH, Li FC, Wang SJ, Qin S, Wang QF. Intergeneric conjugation in holomycin-producing marine Streptomyces sp. strain M095. Microbiological Research, 2008, 163: 96-104. |
| [9] | McInerney BV, Gregson RP, Lacey MJ, Akhurst RJ, Lyons GR, Rhodes SH, Smith DR, Engelhardt LM, White AH. Biologically active metabolites from Xenorhabdus spp., Part 1. Dithiolopyrrolone derivatives with antibiotic activity. Journal of Natural Products, 1991, 54: 774-784. |
| [10] | Wietz M, Mansson M, Gotfredsen CH, Larsen TO, Gram L. Antibacterial compounds from marine Vibrionaceae isolated on a global expedition. Marine Drugs, 2010, 8: 2946-2960. |
| [11] | Qin ZW, Baker AT, Raab A, Huang S, Wang T, Yu Y, Jaspars M, Secombes CJ, Deng H. The fish pathogen Yersinia ruckeri produces holomycin and uses an RNA methyltransferase for self-resistance. Journal of Biological Chemistry, 2013, 288: 14688-14697. |
| [12] | Fukuda D, Haines AS, Song Z, Murphy AC, Hothersall J, Stephens ER, Gurney R, Cox RJ, Crosby J, Willis CL. A natural plasmid uniquely encodes two biosynthetic pathways creating a potent anti-MRSA antibiotic. PLoS One, 2011, 6(3): e18031. |
| [13] | Brachmann, AO. Isolation and identification of natural products and biosynthetic pathways from Photorhabdus and Xenorhabdus. Saarland University, Ph.D. Thesis. 2009. |
| [14] | Fuente A, Lorenzana LM, Martín JF, Liras P. Mutants of Streptomyces clavuligerus with disruptions in different genes for clavulanic acid biosynthesis produce large amounts of holomycin: possible cross-regulation of two unrelated secondary metabolic pathways. Journal of Bacteriology, 2002, 184: 6559-6565. |
| [15] | Li B, Walsh CT. Identification of the gene cluster for the dithiolopyrrolone antibiotic holomycin in Streptomyces clavuligerus. Proceedings of the National Academy of Sciences, 2010, 107: 19731-19735. |
| [16] | Li B, Walsh CT. Streptomyces clavuligerus HlmI is an intramolecular disulfide-forming dithiol oxidase in holomycin biosynthesis. Biochemistry, 2011, 50: 4615-4622. |
| [17] | Huang S, Zhao Y, Qin Z, Wang X, Onega M, Chen L, He J, Yu Y, Deng H. Identification and heterologous expression of the biosynthetic gene cluster for holomycin produced by Streptomyces clavuligerus. Process Biochemistry, 2011, 46: 811-816. |
| [18] | Robles-Reglero V, Santamarta I, álvarez-álvarez R, Martín J, Liras P. Transcriptional analysis and proteomics of the holomycin gene cluster in overproducer mutants of Streptomyces clavuligerus. Journal of Biotechnology, 2013, 163: 69-76. |
| [19] | Liras P. Holomycin, a dithiolopyrrolone compound produced by Streptomyces clavuligerus. Applied Microbiology and Biotechnology, 2014, 98: 1023-1030. |
| [20] | Tanner F, Means J, Davisson J, English A. Thiolutin, an antibiotic produced by certain strains of Streptomyces albus. In Proceedings of the 118th Meeting of the American Chemical Society, 1950, Chicago, IL, USA. |
| [21] | Celmer WD, Solomons I. The structures of thiolutin and aureothricin, antibiotics containing a unique pyrrolinonodithiole nucleus. Journal of the American Chemical Society, 1955, 77: 2861-2865. |
| [22] | Lamari L, Zitouni A, Boudjella H, Badji B, Sabaou N, Lebrihi A, Lefebvre G, Seguin E, Tillequin F. New dithiolopyrrolone antibiotics from Saccharothrix sp. SA 233. I. Taxonomy, fermentation, isolation and biological activities. The Journal of Antibiotics, 2002, 55: 696-701. |
| [23] | Bhate D, Hulyalkar R, Menon S. Isolation of iso-butyropyrrothine along with thiolutin and aureothricin from a Streptomyces sp. Cellular and Molecular Life Sciences, 1960, 16: 504-505. |
| [24] | Saker S, Almousa Almaksour Z, Chorin AC, Lebrihi A, Mathieu F. Enzymatic synthesis of dithiolopyrrolone antibiotics using cell-free extract of Saccharothrix algeriensis NRRL B-24137 and biochemical characterization of two pyrrothine N-acyltransferases in this extract. Journal of Microbiology and Biotechnology, 2014, 24: 26-35. |
| [25] | Huang S, Him Tong M, Qin ZW, Deng Z, Deng H, Yu Y. Identification and characterization of the biosynthetic gene cluster of thiolutin, a tumor angiogenesis inhibitor, in Saccharothrix algeriensis NRRL B-24137. Anti-Cancer Agents in Medicinal Chemistry , 2015, 15: 277-284. |
| [26] | Scharf DH, Habel A, Heinekamp T, Brakhage AA, Hertweck C. Opposed effects of enzymatic gliotoxin N-and S-methylations. Journal of the American Chemical Society, 2014, 136: 11674-11679. |
| [27] | Shiozawa H, Fukuoka T, Fujimoto K, Kodama K. Thiomarinols: discovery from a marine bacterium, structure-activity relationship, and efficacy as topical antibacterial agents. Annual Report-Sankyo Research Laboratories, 1999, 51: 45-72. |
| [28] | Shiozawa H, Kagasaki T, Torikata A, Tanaka N, Fujimoto K, Hata T, Furukawa Y, Takahashi S. Thiomarinols B and C, new antimicrobial antibiotics produced by a marine bacterium. The Journal of Antibiotics, 1995, 48: 907-909. |
| [29] | Shiozawa H, Shimada A, Takahashi S. Thiomarinols D, E, F and G, new hybrid antimicrobial antibiotics produced by a marine bacterium; isolation, structure and antimicrobial activity. The Journal of Antibiotics, 1997, 50: 449-452. |
| [30] | Murphy AC, Fukuda D, Song Z, Hothersall J, Cox RJ, Willis CL, Thomas CM, Simpson TJ. Engineered thiomarinol antibiotics active against MRSA are generated by mutagenesis and mutasynthesis of Pseudoalteromonas SANK73390. Angewandte Chemie, 2011, 123: 3329-3332. |
| [31] | Dunn ZD, Wever WJ, Economou NJ, Bowers AA, Li B. Enzymatic basis of “hybridity” in thiomarinol biosynthesis. Angewandte Chemie, 2015, 127: 5226-5230. |
| [32] | Yakushiji F, Miyamoto Y, Kunoh Y, Okamoto R, Nakaminami H, Yamazaki Y, Noguchi N, Hayashi Y. Novel hybrid-type antimicrobial agents targeting the switch region of bacterial RNA polymerase. ACS Medicinal Chemistry Letters, 2013, 4: 220-224. |
| [33] | Nárdiz N, Santamarta I, Lorenzana LM, Martín JF, Liras P. A rhodanese-like protein is highly overrepresented in the mutant S. clavuligerus oppA2:: aph: effect on holomycin and other secondary metabolites production. Microbial Biotechnology, 2011, 4: 216-225. |
| [34] | Yin H, Xiang S, Zheng J, Fan K, Yu T, Yang X, Peng Y, Wang H, Feng D, Luo Y. Induction of holomycin production and complex metabolic changes by the argR mutation in Streptomyces clavuligerus NP1. Applied and Environmental Microbiology, 2012, 78: 3431-3441. |
| [35] | Charusanti P, Fong NL, Nagarajan H, Pereira AR, Li HJ, Abate EA, Su Y, Gerwick WH, Palsson BO. Exploiting adaptive laboratory evolution of Streptomyces clavuligerus for antibiotic discovery and overproduction. PLoS One, 2012, 7: e33727. |
| [36] | Chen L, Wang Y, Guo H, Xu M, Deng Z, Tao M. High-throughput screening for Streptomyces antibiotic biosynthesis activators. Applied and Environmental Microbiology, 2012, 78: 4526-4528. |
| [37] | Goss RJ, Shankar S, Fayad AA. The generation of “unnatural” products: synthetic biology meets synthetic chemistry. Natural Product Reports, 2012, 29: 870-889. |
| [38] | Cane DE, Kudo F, Kinoshita K, Khosla C. Precursor-directed biosynthesis: biochemical basis of the remarkable selectivity of the erythromycin polyketide synthase toward unsaturated triketides. Chemistry & Biology, 2002, 9: 131-142. |
| [39] | Bouras N, Mathieu F, Sabaou N, Lebrihi A. Nutritional requirements for the production of dithiolopyrrolone antibiotics by Saccharothrix algeriensis NRRL B-24137. Enzyme and Microbial Technology, 2006, 39: 1423-1429. |
| [40] | Bouras N, Mathieu F, Sabaou N, Lebrihi A. Influence on dithiolopyrrolone antibiotic production by organic acids in Saccharothrix algeriensis NRRL B-24137. Process Biochemistry, 2007, 42: 925-933. |
| [41] | Merrouche R, Bouras N, Coppel Y, Mathieu F, Monje MC, Sabaou N, Lebrihi A. Dithiolopyrrolone antibiotic formation induced by adding valeric acid to the culture broth of Saccharothrix algeriensis. Journal of Natural Products, 2010, 73: 1164-1166. |
| [42] | Merrouche R, Bouras N, Coppel Y, Mathieu F, Sabaou N, Lebrihi A. New dithiolopyrrolone antibiotics induced by adding sorbic acid to the culture medium of Saccharothrix algeriensis NRRL B-24137. FEMS Microbiology Letters, 2011, 318: 41-46. |
| [43] | Weist S, Süssmuth R. Mutational biosynthesis-a tool for the generation of structural diversity in the biosynthesis of antibiotics. Applied Microbiology and Biotechnology, 2005, 68: 141-150. |
