霍雨佳1, 卢乔楠2, 郑晓伟1, 马远方1, 卢锋1


1. 河南大学医学院, 抗体药物河南省工程实验室, 河南 开封 475001;
2. 西北农林科技大学林学系, 陕西 杨凌 712100
收稿日期: 2015-05-26; 修回日期: 2015-07-08; 网络出版日期: 2015-08-31
基金项目: 国家自然科学基金(81372147);河南省教育厅自然科学研究项目(2010B320001);河南大学省部共建项目(SBGJ090713)
通信作者: 卢锋, Tel/Fax: +86-10-23880698; E-mail: lufeng@henu.edu.cn
摘要: [目的] 探索大肠埃希氏菌Escherichia coli FtsZ突变体FtsZE75A、FtsZR78G和ftsZD82A对FtsZ自身组装和FtsZ-MreB相互作用的影响。[方法] 利用常规分子克隆和定点突变技术,构建FtsZ及其突变体表达载体,亲和纯化得到相应的目标蛋白;通过同源重组构建QN6 (ftsZ::yfp-cat)、QN7 (ftsZE75A::yfp-cat)、QN8 (ftsZR78G::yfp-cat)和QN9 (ftsZD82A::yfp-cat)菌株;利用活细胞成像技术观察FtsZ及其突变体的胞内定位模式;免疫沉淀和细菌双杂交实验检测FtsZ/FtsZ*-FtsZ*或FtsZ/FtsZ*-MreB间的相互作用;光扫描检测定点突变对FtsZ组装特性的影响。[结果] FtsZE75A、FtsZR78G和ftsZD82A突变体的功能活性降低、各突变体在E. coli内不能正确的定位和形成功能性Z环;FtsZ/FtsZ*-FtsZ*单体间的相互作用减弱或消失,FtsZ*-MreB相互作用破坏;FtsZ突变体体外聚合效率降低。[结论] FtsZ E75、R78和D82是影响FtsZ正确组装和功能及FtsZ-MreB相互作用的重要氨基酸。
关键词: 大肠埃希氏菌FtsZ 组装MreB细胞内定位
E75, R78 and D82 of Escherichia coli FtsZ are key residues for FtsZ cellular self-assembly and FtsZ-MreB interaction
Yujia Huo1, Qiaonan Lu2, Xiaowei Zheng1, Yuanfang Ma1, Feng Lu1


1. Henan Engineering Laboratory of Antibody Medicine, Medical School of Henan University, Kaifeng 475001, Henan Province, China;
2. College of Forestry, Northwest A&F University, Yangling 712100, Shaanxi Province, China
Received: 26 May 2015; Revised: 8 July 2015; Published online: 31 August 2015
Foundation Item: Supported by the National Natural Science Foundation of China (81372147),by the Education Department of Henan Province Natural Science Foundation (2010B320001) and by the Henan University Fund co-sponsored by Province and Ministry (SBGJ090713)
Corresponding author: Feng Lu, Tel/Fax: +86-10-23880698; E-mail: lufeng@henu.edu.cn
Abstract:[Objective] To explore effects of FtsZ mutants FtsZE75A, FtsZR78G and ftsZD82A on FtsZ self-assembly and interaction of FtsZ with MreB in Escherichia coli strains. [Methods] We constructed FtsZ and its mutant's plasmids by molecular clone and site-directed mutagenesis methods, and purified targeted proteins by affinity chromatography. QN6(ftsZ::yfp-cat), QN7(ftsZE75A::yfp-cat), QN8(ftsZR78G::yfp-cat) and QN9(ftsZD82A::yfp-cat) strains were constructed by linear DNA homologous recombination. We observed cellular localization pattern of FtsZ and its mutants in E. coli by living cell imaging experiments, examined interaction of FtsZ/FtsZ*-FtsZ* and FtsZ/FtsZ*-MreB by Co-immunoprecipita-tion and bacteria two hybrid, and analyzed assembly characteristics of FtsZ mutants by Light scattering. [Results] The Yfp-labeled FtsZE75A, FtsZR78G and ftsZD82A mutant proteins failed to assemble into functional Z-ring structure and localize correctly in E. coli strains. Interaction of FtsZ with its mutants, or FtsZ*-FtsZ* and FtsZ*-MreB interaction were weakened or completely disappeared. In addition, in vitro experiments show that E75A, R78G and D82A mutations decreased the polymerization efficiency of FtsZ monomer. [Conclusion] FtsZ E75, R78 and D82 are critical amino acids in the assembly, function of FtsZ protein and FtsZ-MreB interaction in E. coli strains.
Key words: Escherichia coliFtsZ assemblyMreBcellular localization
细菌细胞的分裂和特定形态的维持受多种蛋白的调节,其中微管蛋白类似物FtsZ和肌动蛋白类似物MreB起核心作用[1, 2],但具体的机制目前未完全清楚,这限制了以FtsZ为靶点进行新一代抗生素的开发和利用。FtsZ是大肠埃希氏菌Escherichia coli等细菌分裂所必需的蛋白,在原核生物中高度保守。其单体通过纵向和横向(侧面)相互作用形成束状聚合物,最后在菌体中部形成Z环。Z环的稳定需要FtsA、ZipA、ZapA和ZapB等多种蛋白的参与,其最终起骨架作用,通过招募下游与细胞分裂有关的多种蛋白,在即将分裂的菌体细胞中部形成分裂体(divisome),调控细菌的分裂[3, 4]。MreB是目前公认的控制E. coli形态的主要蛋白,其基因敲除可导致E. coli菌体由杆状变为球状,但当菌体内FtsZ高水平表达,可抑制MreB缺陷所引起的形态改变[5, 6, 7]。近年来有研究者发现,FtsZ特殊位点氨基酸的突变可致菌体呈马蹄形、新月形和鹿角形等[8, 9, 10, 11];另外也有研究者发现,MreB的某些突变体可致细菌的异常分裂[12],说明FtsZ与MreB间存在某种协调关系。为进一步探讨FtsZ和MreB是否共同调控细菌的生长和分裂,本研究通过分析FtsZ的三维结构,对位于FtsZ侧面H3结构域的特定氨基酸位点进行突变,观察其对FtsZ胞内组装和定位、FtsZ-MreB相互作用的影响及相关的分子机制和意义。
1 材料和方法 1.1 材料1.1.1 菌株、载体和质粒:E. coli MC 1000 [F-araD139△(araABC-leu)7679 galU galK △(lac)X74 rpsL thi]和BTH101菌,载体pKT25、pUT18C、pMLB1113和pET30a(+) (Invitrogen),质粒pKD3、pKD119、pKT25-zip、pUT18C-zip和pFL1由实验室保存[13, 14, 15, 16, 17]。详细信息见表1。
表 1. 研究中所使用的菌种和质粒Table 1. Strains and plasmids used in this study
| Strains or plasmids | Genotype and/or features | Resistance marker | Source |
| MC1000 | F -,△ ( araA-leu ) 7697, [ara139] B/r,△(codB-lacI)3, galk16, galE15(GalS), λ - , e14-, relA1, rpsL150(strR), spoT1, mcrB1 | - | (13) |
| BTH101 | F, - cya-99, araD139, galE15, galK16, rpsL1, hsdR2, mcrA1, mcrB1 | - | (14) |
| pFL1 | P lac-yfp::rnb | Ampicillin | (15) |
| pMLB1113 | Low copy number vector (18 per cell) | Ampicillin | (13) |
| pUT18C | P lac-T18- | Ampicillin | (16) |
| pKT25 | P lac-T25- | Kanamycin | (16) |
| pUT18C-zip | P lac-T18-leucine zipper region from yeast GCN4 | Ampicillin | (16) |
| pKT25-zip | P lac-T25-leucine zipper region from yeast GCN4 | Kanamycin | (16) |
| pKD119 | Red recombinase expression plasmid | Tetracycline | (17) |
| pKD3 | Cat marker for gene deletion constructs | Chloramphenicol | (17) |
| pQN1 | P lac-ftsZ | Ampicillin | This study |
| pQN2 | P lac-ftsZ E75A | Ampicillin | This study |
| pQN3 | P lac-ftsZ R78G | Ampicillin | This study |
| pQN4 | P lac-ftsZ D82A | Ampicillin | This study |
| pQN5 | P lac-ftsZ::his 6 | Ampicillin | This study |
| pQN6 | P lac-ftsZ E75A:: his 6 | Ampicillin | This study |
| pQN7 | P lac-ftsZ R78G:: his 6 | Ampicillin | This study |
| pQN8 | P lac-ftsZ D82A:: his 6 | Ampicillin | This study |
| pQN9 | P lac-T18-ftsZ E75A | Ampicillin | This study |
| pQN10 | P lac-T18-ftsZ R75G | Ampicillin | This study |
| pQN11 | P lac-T18-ftsZ D82A | Ampicillin | This study |
| pUT18C-ftsZ | P lac-T18-ftsZ | Ampicillin | This study |
| pKT25-mreB | P lac-T25-mreB | Kanamycin | This study |
| QN6 | MC1000:ftsZ::yfp-Cat | Chloramphenicol | This study |
| QN7 | MC1000: ftsZ E75A::yfp-Cat | Chloramphenicol | This study |
| QN8 | MC1000: ftsZ R78G::yfp-Cat | Chloramphenicol | This study |
| QN9 | MC1000: ftsZ D82A::yfp-Cat | Chloramphenicol | This study |
表选项
1.1.2 主要试剂:ES-Tag DNA聚合酶、T4 DNA 连接酶、限制性核酸内切酶XbaⅠ、HindⅢ、BamHⅠ和EcoRⅠ均购自宝生物工程(大连)有限公司;抗MreB抗体由Lawrence Rothfield提供[18],抗FtsZ抗体(AS10715)为美国Agrisera公司产品;基因组DNA提取试剂盒(CW0552)和抗GFP标签抗体(CW0087)购自北京康为世纪,二抗-HRP (羊抗兔BA1054)为博士德生物产品;基因定点突变试剂盒(Cat#200518 )为美国Stratagene公司产品;BAC TH细菌双杂交系统(Cat#Euk001)为Euromedex公司产品;蛋白亲和纯化柱(HisTrap HP,17-5247-01)为GE公司产品;免疫共沉淀试剂盒(Cat#46147) 为Thermo公司产品。
1.2 质粒构建1.2.1 FtsZ和FtsZ突变体(FtsZ*)表达质粒的构建:以E. coli MC1000基因组DNA为模板,使用pMLB1113载体,常规构建FtsZ表达质粒pQN1 (Plac-ftsZ)并测序证明。以pQN1质粒DNA为模板,按基因定点突变试剂盒(Stratagene,Cat#200518) 操作手册对目的基因进行定点突变,构建pQN2 (Plac-ftsZE75A)、pQN3 (Plac-ftsZR78G)和pQN4 (Plac-ftsZD82A)表达质粒;使用pET30a(+)载体,分别构建C端带His标签的FtsZ及其突变体表达质粒pQN5 (Plac-FtsZ::his6)、pQN6 (Plac-FtsZE75A::his6)、pQN7 (Plac-FtsZR78G::his6)和pQN8 (Plac-FtsZD82A::his6),经双向测序验证序列。
1.2.2 FtsZ/FtsZ*-MreB细菌双杂交质粒的构建:以E. coli基因组DNA为模板,扩增FtsZ (XbaⅠ/ BamHⅠ)和MreB (XbaⅠ/ EcoRⅠ)基因。产物经双酶切后,与同样双酶切载体pUT18C (XbaⅠ/ BamHⅠ)和pKT25 (XbaⅠ/ EcoRⅠ)相连结,获得表达质粒pUT18C-ftsZ (Plac-T18-ftsZ)和pKT25-mreB (Plac-T25-mreB)。另外用同样的分子克隆手段,以pQN2、 pQN3和pQN4为模板,分别构建pQN9 (Plac-T18-ftsZE75A)、pQN10 (Plac-T18-ftsZR78G)和pQN11 (Plac-T18-ftsZD82A)表达质粒。最后,构建的表达质粒皆经测序进行验证。
1.3 同源重组构建ftsZ/ftsZ*::yfp菌株根据文献[17]报道的方法,通过λ同源重组构建QN6 (ftsZ::yfp-cat)菌株。简述如下:分别以pKD3 和pFL1质粒DNA为模板,扩增氯霉素抗性基因(Cat)和yfp基因;通过Overlap PCR将两基因融合。设计同源重组引物,扩增yfp-cat融合基因,通过电穿孔方式将yfp-cat cassette DNA片段(约100 ng)导入MC1000/pKD119感受态菌中,经平板(含30 μg/mL氯霉素)筛选初步获得重组菌株QN6 (ftsZ::yfp-cat),然后经克隆PCR和基因测序进一步证明。后续再通过定点突变和同源重组方法,分别构建QN7 (ftsZE75A::yfp-cat)、QN8 (ftsZR78G::yfp-cat)和QN9 (ftsZD82A::yfp-cat)菌株。
1.4 荧光显微镜观察FtsZ及其突变体在E. coli中的定位模式为了观察FtsZ及其突变体在E. coli中的定位情况,挑取QN6、QN7、QN8和QN9单菌落接种于5 mL LB培养基(包含30 μg/mL氯霉素)中37 °C过夜培养,次日按1%比例于相同培养基中重新活化至菌液OD600≈0.8。取10 μL的菌液滴于载玻片上并盖上盖玻片,在荧光显微镜(Olympus BX60;fil-ters:Chroma Set ID 41029)下观察实验结果和获得相应的图像(Openlab image acquisition program)(物镜×100,目镜×10)。
1.5 细菌双杂交技术检测FtsZ/FtsZ*与MreB间的相互作用参照BATCH system kit产品说明书检测FtsZ/ FtsZ*-MreB相互作用。pUT18C-ftsZ和pKT25-mreB表达质粒共转染BTH101菌细胞,经双抗琼脂平板(LB/Kan+Amp)筛选后获得相应的阳性克隆。将阳性克隆接种于5 mL LB培养基中(含100 μg/mL Amp、50 μg/mL Kan和 0.4% glucose) 37 °C过夜培养,次日按1%比例稀释后重新活化菌液至OD600≈0.5,然后取适量菌液进行系列稀释(10-3、10-4、10-5、10-6)。用微量移液器分别吸取1.5 μL的不同稀释度的菌液,接种于LB选择性琼脂培养板上(含有100 μg/mL Amp、50 μg/mL Kan、0.5 mmol/L IPTG和40 μg/mL X-gal),于30 °C培养48 h后观察每个接种点菌体颜色的变化。同时使用pUT18C-zip和pKT25-zip质粒共转染BTH101作为阳性对照,pUT18C和pKT25作为阴性对照组。另外检测FtsZ突变体FtsZE75A、FtsZR78G和FtsZD82A与MreB的相互作用方法与上述相同。
1.6 免疫共沉淀分析(Co-immunoprecipitation,Co-IP)1.6.1 验证FtsZ/FtsZ*-MreB间的相互作用关系: QN6、QN7、QN8和QN9菌细胞裂解液的制备参照前期研究工作[15]。最后用5-10 mL的Buffer A [10 mmol/L Tris-HCl (pH 7.5),5% glycerol,0.5% Tween-20,1 mmol/L EDTA,0.1 mmol/L DTT,0.1 mmol/L PMSF]溶解蛋白沉淀,定量其浓度后分装,-80 °C保存。Co-IP分析过程按免疫共沉淀试剂盒说明书(Cat#46147,Thermo)和文献报道进行[15]。每个反应管使用10 μg抗-Yfp抗体(CW 0087,康为世纪)、1000 μg菌体细胞裂解液蛋白和30 μL protein A/G plus agarose beads,Co-IP蛋白洗脱液体积为50 μL。
1.6.2 检测野生型FtsZ与其定点突变体及同种突变体蛋白间的相互作用: 制备携带有pQN1表达质粒的QN6、QN7、QN8和QN9菌细胞和QN7/ pQN2、QN8/pQN3和QN9/pQN4菌细胞裂解液及Co-IP分析过程与1.6.1相同。
1.7 FtsZ及其突变体的表达纯化及光扫描实验分别将质粒pQN5(Plac-FtsZ::his6)、pQN6(Plac-FtsZE75A::his6)、pQN7(Plac-FtsZR78G::his6)和pQN8(Plac-FtsZD82A::his6) 转化到BL21(DE3)中并诱导表达目标蛋白。具体制备和纯化过程参照文献[7]方法进行。使用LB培养基、0.3 mmol/L IPTG、30 °C条件下诱导培养5 h,离心收菌(4696×g,10 min);在50 mmol/L Tris (pH 7.9)缓冲液中反复冻融及超声破碎菌体细胞;超速离心(23480×g,40 min,4 °C)得上清液,使用 HisTrap HP 亲和纯化目标蛋白并定量。
使用荧光分光光度仪(LS50,Perkin Elmer),通过光扫描检测E. coli FtsZ及其突变体蛋白的聚合能力[9]。将纯化的FtsZ或其突变体蛋白在比色杯中用聚合Buffer (50 mmol/L Mes pH 6.5,50 mmol/L KCl,5 mmol/L MgCl2,10 mmol/L CaCl2) 稀释到0.015 mmol/L,30 °C孵育3 min,然后加入GTP至终浓度1 mmol/L,轻轻搅动混匀液体。反应体系总体积为300 μL。发射光和激发光波长为350 nm,狭缝宽为5 nm。每次光扫描的时间间隔为2 min。
1.8 Western blot 分析具体方法参照文献[15]。菌体蛋白裂解液上样量是根据OD600进行计算,以使每个泳道上样总蛋白尽量相同。抗-MreB、抗-FtsZ、抗-YFP和羊抗兔二抗的稀释度分别为1:500、1:1000、1:2000和1:20000,显色方法为ECL (P1020,北京普丽莱)。
1.9 FtsZ突变体功能检测QN6、QN7、QN8和QN9 37 °C过夜培养的菌液,次日重新活化(按1%比例)于相同培养基中至OD600≈0.5。系列稀释(10-1-10-5)各组菌液,每组样品每个稀释度取1 μL轻轻接种于LB琼脂板上(含30 μg/mL Cat),37 °C培养24 h。细菌生长曲线的绘制从OD600≈0.05开始,第一个检测时间点为活化开始后1.5 h,以后每间隔1 h测1次菌液OD600,最后使用Excel软件处理数据。
2 结果和分析 2.1 FtsZ突变体位点选择与设计使用3D-JIGSAM软件对E. coli FtsZ的三维结构进行预测[19] (图1-A),发现其H3结构域位于FtsZ蛋白的外侧,对其结构进一步分析发现,FtsZ (74-82)区域可形成两性螺旋结构(图1-B);E75、R78和D82不仅为极性氨基酸,而且位于两性螺旋的一侧和FtsZ蛋白表面。推测E75、R78和D82可能是影响H3结构和FtsZ胞内组装的重要氨基酸。
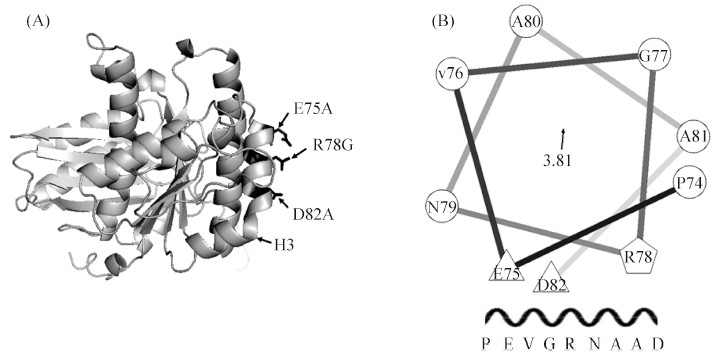 |
| 图 1. FtsZ三维结构预测(A)和FtsZ (74–82)结构域轮状图(B) Figure 1. 3D structure prediction of FtsZ (A) and helical wheel projection of FtsZ (74–82) domain (B). |
| 图选项 |
2.2 E75、R78和D82是影响FtsZ在E. coli中形成Z环的重要氨基酸活细胞成像观察到,带YFP标签的野生型FtsZ在E. coli内形成规则的Z环(图2-A1),菌体呈规则的杆状(图2-A2);突变体FtsZE75A::Yfp和FtsZD82A::Yfp在菌体内呈横跨细胞两极的螺旋状(图2-B1,D1),并且细胞明显变长,呈丝状(图2-B2,D2);FtsZR78G::Yfp突变体则在菌体内呈极化现象,即FtsZR78G突变体融合蛋白主要定位在细胞的两极(图2-C1),细胞的形态与野生型相似(图2-C2)。为了排除FtsZ及其突变体的表达水平可能对FtsZ的胞内组装影响,以MreB蛋白作为内参对照,通过Western blot分析观察到,FtsZ及其突变体融合蛋白的表达水平大致相同且与MC1000的FtsZ表达相似(图2-E)。
 |
| 图 2. FtsZ及其定点突变体 ftsZE75A、ftsZR78G和ftsZD82A在E.coli中的荧光定位(A-D)和表达水平分析(E) Figure 2. The Fluorescence localization patterns (A-D) and expression levels (E) of FtsZ and its point mutants FtsZ ftsZE75A, ftsZR78G and ftsZD82A in Escherichia coli strains. A: QN6, ftsZ::yfp-cat; B: QN7, ftsZE75A::yfp-cat; C: QN8, ftsZR78G::yfp-cat; D: QN9, ftsZD82A::yfp-cat; E: MC1000, wild type. |
| 图选项 |
2.3 补偿实验验证FtsZ突变体功能由于FtsZ是细菌分裂和生存所必需的蛋白,能通过同源重组获得QN6、QN7、QN8和QN9菌株,本身证明FtsZ定点突变体具有一定功能,但LB琼脂糖平板补偿实验发现,与野生型FtsZ相比,E75A、R78G和D82A位点突变体功能皆有所降低,且以FtsZD82A降低最为显著(图3-A)。这些结果与图3-B的生长曲线基本一致。
 |
| 图 3. FtsZ及其定点突变体的平板补偿实验(A)和生长曲线分析(B) Figure 3. Plate complement assays(A) and growth-curve analysis(B) of FtsZ and its point mutants. MC1000, wild type; QN6, ftsZ::yfp-cat; QN7, ftsZE75A::yfp-cat; QN8, ftsZR78G::yfp-cat; QN9, ftsZD82A::yfp-cat. |
| 图选项 |
2.4 E75、R78和D82是影响FtsZ自身组装的重要氨基酸以FtsZ/FtsZ*::Yfp为靶分子,使用抗Yfp抗体和免疫共沉淀试剂盒(Cat#46147,Thermo)进行免疫共沉淀分析发现,将FtsZ E75、R78和D82位点氨基酸突变为性质不同的氨基酸后,FtsZE75A和FtsZR78G与FtsZ相互作用明显减弱,而FtsZD82A-FtsZ相互作用未能检测到;另外,相同的FtsZ突变体单体间相互作用与对照组相比也发生明显的改变,FtsZE75A- FtsZE75A和FtsZR78G-FtsZR78G相互作用变弱,FtsZD82A突变体单体间相互作用消失(图4),显示E75、R78和D82位点是影响FtsZ单体间相互作用的重要氨基酸。
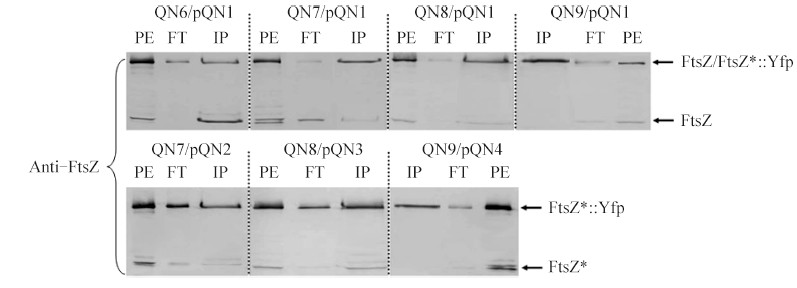 |
| 图 4. FtsZ*-FtsZ/FtsZ*相互作用的免疫共沉淀分析 Figure 4. Co-immunoprecipitation analysis of FtsZ*-FtsZ/FtsZ* interaction. PE, protein extract; FT, flow through; IP, immunoprecipitate. QN6/pQN1, ftsZ::yfp-cat/Plac-ftsZ; QN7/pQN1, ftsZE75A::yfp-cat/Plac-ftsZ; QN8/pQN1, ftsZR78G::yfp-cat/Plac-ftsZ); QN9/pQN1, ftsZD82A::yfp-cat/Plac-ftsZ; QN7/pQN2, ftsZE75A::yfp-cat/ Plac-ftsZE75A; QN8/pQN3, ftsZR78G::yfp-cat/Plac-ftsZR78G; QN9/pQN4, ftsZD82A::yfp-cat/Plac-ftsZD82A. |
| 图选项 |
为了进一步证明上述实验结果,我们表达纯化了C端带His标签的FtsZ及其突变体融合蛋白,经考染(图5-A1)及WB分析(图5-A2)验证后,利用光扫描技术,在体外研究FtsZ各突变体聚合特性的变化。当E75、R78和D82突变为性质不同的氨基酸时,与野生型FtsZ相比,突变体蛋白聚合能力降低,单体间聚合延迟,其中以FtsZD82A突变体最为显著,表现为聚合曲线(S曲线)明显右移(图5-B)。
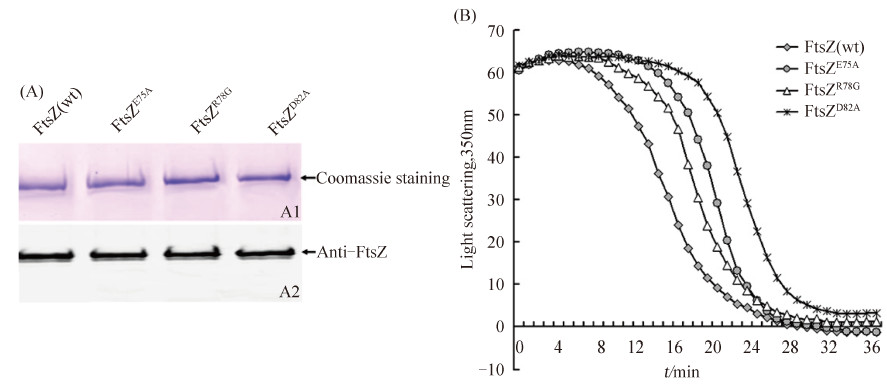 |
| 图 5. FtsZ(wt)及其突变体FtsZE75A、FtsZR78G和FtsZD82A的表达纯化 (A) 和体外聚合分析(B) Figure 5. Expression, purification (A) and in vitro polymerization analysis (B) of FtsZ (wt) and its mutants FtsZE75A, FtsZR78G and FtsZD82A. |
| 图选项 |
2.5 E75、R78和D82位点氨基酸极性改变影响FtsZ-MreB相互作用细菌双杂交实验发现,与对照组相比,FtsZ E75、R78和D82突变为性质不同的氨基酸,使FtsZ-MreB相互作用明显减弱或消失(图6-A)。另外常规制备QN6、QN7、QN8和QN9菌细胞裂解液,以FtsZ/FtsZ*::Yfp为靶分子,使用抗YFP抗体,通过免疫共沉淀和Western blot分析观察到,FtsZE75A、FtsZR78G和FtsZD82A突变体蛋白不能与MreB相互作用,该结果与细菌双杂交实验一致(图6-B)。显示E75、R78和D82位点氨基酸是影响FtsZ-MreB相互作用的重要氨基酸。
 |
| 图 6. FtsZ和其位点突变体与MreB相互作用的细菌双杂交(A)和免疫共沉淀分析(B) Figure 6. Analysis of FtsZ and its mutants interaction with MreB by bacterial two hybrid(A) and Co-immunoprecipitation assay(B). A: +ve, positive control, BTH101/pKT25-zip/pUT18C-zip; -ve, negative control, BTH101/pKT25/pUT18C; B: PE, protein extract; FT, flow through; IP, immunoprecipitate; QN6, ftsZ::yfp-cat; QN7, ftsZE75A::yfp-cat; QN8, ftsZR78G::yfp-cat; QN9, ftsZD82A::yfp-cat. |
| 图选项 |
3 讨论FtsZ和MreB是调控细菌分裂的两个重要蛋白,但二者协调的分子机制目前知之甚少。我们通过对FtsZ的三维结构进行分析,观察其H3结构域位于FtsZ侧面,轮状图显示FtsZ (74-82)区域可形成两性螺旋结构。另外发现E75、R78和D82氨基酸不仅位于FtsZ表面,而且位于FtsZ (74-82)区域两性螺旋的一个侧面(图1)。近年来有文献报道,蛋白的两性螺旋结构域是目标蛋白的重要结构,当这种结构被破坏后,蛋白的组装、细胞内定位或功能往往会受到重要的影响,如MinD(270-255)区域,FtsA(406-420)区域[20, 21]和我们前期研究中发现的RNaseⅡ(3-19)区域[15]。本研究将E75、R78和D82突变为性质不同的氨基酸,导致FtsZE75A、FtsZR78G和FtsZD82A突变体在细胞内的定位模式改变(图2),提示FtsZ*引起FtsZ (74-82)区域的两性螺旋结构改变,进而影响FtsZ在菌体内的组装。与野生型FtsZ相比,FtsZ突变体FtsZE75A、FtsZR78G和FtsZD82A虽具有一定的的补偿能力,但3个突变体所引起的功能降低改变不完全一致,以FtsZD82A突变体最为明显(图3),这可能与D82在H3结构域的位置有关,具体机制有待进一步探讨。另外我们通过Western blot分析,检测了FtsZ及其突变体蛋白在菌体内的表达水平,排除了由于蛋白水平的异常表达而产生的假象。
为了进一步探索FtsZ突变体所引起的菌体内异常组装和定位的分子机制,我们推测FtsZ的位点突变可能改变了其单体间的相互作用,进而影响了FtsZ单体能正确组装成复合体和最终形成Z环的能力。虽然本次研究选择的3个氨基酸位点位于FtsZ蛋白的侧表面,理论上讲不应该影响FtsZ单体的纵向组装,即对FtsZ-FtsZ的相互作用影响较小,但免疫共沉淀(Co-IP)结果显示FtsZ与FtsZE75A和FtsZR78G相互作用明显减弱,而FtsZ-FtsZD82A相互作用完全破坏;同一种突变体蛋白单体间的相互作用分析结果与上述相同。另外,我们对表达纯化的突变体蛋白进行体外聚合能力测试,发现FtsZE75A、FtsZR78G和FtsZD82A聚合能力明显降低,而且以FtsZD82A突变体蛋白最为显著,证明E75、R78和D82对维持FtsZ单体正确空间构象和单体间相互作用非常重要性。我们的研究结果提示FtsZ的横向相互作用可能对维持单体间的纵向相互作用具有重要的协同作用。
近年来有研究者发现,在E. coli中,FstZ与MreB间存在直接的相互作用,而且这种相互作用对E. coli Z环的收缩、分裂体的成熟和菌体细胞壁肽聚糖的形成等起重要的调控作用[22],那么引起FtsZ菌体内异常组装和定位的突变体是否也影响FtsZ-MreB间的相互作用?我们通过多次细菌双杂交实验发现,与对照组相比,FtsZ*-MreB的相互作用显著减弱或消失,显示FtsZE75A、FtsZR78G和FtsZD82A突变体所引起单体构象和胞内组装模式的改变对FtsZ-MreB相互作用的影响。另外,为进一步证实细菌双杂交结果,我们以FtsZ/FtsZ*::Yfp为靶分子,通过免疫共沉淀分析观察到,E75、R78和D82位点氨基酸所维持的FtsZ空间构象是FtsZ-MreB相互作用的重要分子基础。
本研究发现,FtsZ E75、R78和D82是影响E. coli内功能性Z环的正确组装和MreB-FtsZ相互作用的重要氨基酸。研究观察到FtsZE75A和FtsZD82A突变体所引起的菌体细胞呈长丝状及FtsZR78G突变体在菌体内呈极化定位模式的机制还在进一步研究之中,而这些机制的阐明将从另一角度证明了MreB功能的多样性和FtsZ与MreB功能的协调对E. coli分裂与生长的意义。
参考文献
| [1] | Bi E, Lutkenhaus J. FtsZ ring structure associated with division in Escherichia coli. Nature, 1991, 354(6349): 161-164. |
| [2] | Figge RM, Divakaruni AV, Gober JW. MreB, the cell shape-determining bacterial actin homologue, co-ordinates cell wall morphogenesis in Caulobacter crescentus. Molecular Microbiology, 2004, 51(5): 1321-1332. |
| [3] | Randich AM, Brun YV. Molecular mechanisms for the evolution of bacterial morphologies and growth modes. Frontier in Microbiology, 2015, 9(6): 580-593. |
| [4] | Galli E, Gerdes K. FtsZ-ZapA-ZapB interactome of Escherichia coli. Journal of Bacteriology, 2012, 194(2): 292-302. |
| [5] | Domínguez-Escobar J, Chastanet A, Crevenna AH, Frominon V, Wedlich-Söldner R, Carballido-López R. Processive movement of MreB-associated cell wall biosynthetic complexes in bacteria. Science, 2011, 333(6039): 225-228. |
| [6] | Garner EC, Bernard R, Wang WQ, Zhuang XW, Rudner DZ, Mitchison T. Coupled, circumferential motions of the cell wall synthesis machinery and MreB filaments in B. subtilis. Science, 2011, 333(6039): 222-225. |
| [7] | Aaron M, Charbon G, Lam H, Schwarz H, Vollmer W, Jacobs-Wagner C. The tubulin homologue FtsZ contributes to cell elongation by guiding cell wall precursor synthesis in Caulobacter crescentus. Molecular Microbiology, 2007, 64(4): 938-952. |
| [8] | Fletcher DA, Mullins RD. Cell mechanics and the cytoskeleton. Nature, 2010, 463(7280): 485-492. |
| [9] | Garrido T, Sánchez M, Palacios P, Aldea M, Vicente M. Transcription of FtsZ oscillates during the cell cycle of Escherichia coli. European Molecular Biology Organization Journal, 1993, 12(10): 3957-3965. |
| [10] | Typas A, Banzhaf M, Gross CA, Vollmer W. From the regulation of peptidoglycan synthesis to bacterial growth and morphology. Nature Reviews Microbiology, 2011, 10(2): 123-136. |
| [11] | Van der Ploeg R, Verheul J, Vischer NO, Alexeeva S, Hoogendoorn E, Postma M, Banzhaf M, Vollmer W, den Blaauwen T. Colocalization and interaction between elongasome and divisome during a preparative cell division phase in Escherichia coli. Molecular Microbiology, 2013, 87(5): 1074-1087. |
| [12] | Kruse T, Møller-Jensen J, Løbner-Olesen A, Gerdes K. Dysfunctional MreB inhibits chromosome segregation in Escherichia coli. European Molecular Biology Organization Journal, 2003, 22(19): 5283-5292. |
| [13] | De Boer PAJ, Crossley RE, Rothfield LI. A division inhibitor and a topological specificity factor coded for by the minicell locus determine proper placement of the division septum in E. coli. Cell, 1989, 56(4): 641-649. |
| [14] | Karimova G, Dautin N, Ladant D. Interaction network among Escherichia coli membrane proteins involved in cell division as revealed by bacterial two-hybrid analysis. Journal of Bacterial, 2005, 187(7): 2233-2243. |
| [15] | Lu F, Taghalout A. Membrane association via an amino-terminal amphipathic helix is required for the cellular organization and function of RNase Ⅱ. Journal of Biological Chemistry, 2013, 288(10): 2741-2751. |
| [16] | Karimova G, Ullmann A, Ladant D. Protein-protein interaction between Bacillus stearothermophilus tyrosyl-tRNA synthetase subdomains revealed by a bacterial two-hybrid system. Journal of Molecular Microbiology and Biotechnology, 2001, 3(1): 73-82. |
| [17] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceeding of the National Academy of Sciences of the United States of America, 2000, 97(12): 6640-6645. |
| [18] | Vats P, Rothfield L. Duplication and segregation of the actin (MreB) cytoskeleton during the prokaryotic cell cycle. Proceeding of the National Academy of Sciences of the United States of America, 2007, 104(45): 17795-17800. |
| [19] | Bates PA, Kelley LA, MacCallum RM, Sternbery MJE. Enhancement of protein modeling by human intervention in applying the automatic programs 3D-JIGSAW and 3D-PSSM. Proteins, 2001, 5(S5): 39-46. |
| [20] | Szeto TH, Rowland SL, Rothfield LI, King GF. Membrane localization of MinD is mediated by a C-terminal motif that is conserved across eubacteria, archaea, and chloroplasts. Proceeding of the National Academy of Sciences of the United States of America, 2005, 99(24): 15693-15698. |
| [21] | Pichoff S, Lutkenhaus J. Tethering the Z ring to the membrane through a conserved membrane targeting sequence in FtsA. Molecular Microbiology, 2005, 55(6): 1722-1734. |
| [22] | Fenton AK, Gerdes K. Direct interaction of FtsZ and MreB is required for septum synthesis and cell division in Escherichia coli. European Molecular Biology Organization Journal, 2013, 32(13): 1953-1965. |
