CRISPR-Cas系统在微生物基因组中稳定性维持是其抗病毒功能实现的关键基础。一方面,CRISPR-Cas系统具有自我免疫的风险,并可能阻碍有益外源基因的获取,因此可对宿主细胞造成适合度代价(fitness cost)而可能在进化过程中频繁丢失。另一方面,微生物宿主与其病毒的“军备竞赛”中,CRISPR-Cas系统也会成为病毒反攻(Anti-CRISPR)的目标而丧失功能。面对多重的进化压力和适应性挑战,CRISPR-Cas系统为何能在微生物中广泛存在(存在于约90%的古菌和40%细菌中)并发挥其功能?在微生物宿主基因组中是否存在一类保护CRISPR-Cas功能但至今尚未被揭示的“暗物质”?这是科学家长期关注而又未能充分认识的前沿科学问题。

2021年4月30日,国际顶级期刊Science以长文形式(Research Article)在线发表了中国科学院微生物研究所向华/李明团队的最新研究成果“Toxin-antitoxin RNA pairs safeguard CRISPR-Cas systems”(Li M. et al., Science 372, eabe5601,DOI: 10.1126/science.abe5601)。他们首次在自然界分布最广泛的I型CRISPR-Cas基因簇内部发现了一类特殊的RNA“暗物质”:一类前所未见的对其偶联的CRISPR-Cas系统具护卫功能的一对RNA的毒素-抗毒素(CreTA)系统。由于CRISPR-Cas系统可利用RNA抗毒素CreA控制RNA毒素CreT的表达,使宿主菌无法丢失其CRISPR-Cas系统(对其“上瘾”)。因为一旦CRISPR-Cas组分被破坏,就会诱导CreT毒素的表达,从而抑制甚或杀死该宿主菌(图1),从而保护了CRISPR-Cas系统在细胞群体中的稳定存在。这一 “成瘾”机制的发现为理解CRISPR-Cas系统的稳定性维持和广泛性分布提供了全新视角,同时该文还揭示了一大类前所未知、功能多样的小RNA(曾被称为基因组中的“暗物质”-Nat Rev Microbiol 12(2014):647),开辟了一个全新的研究领域。
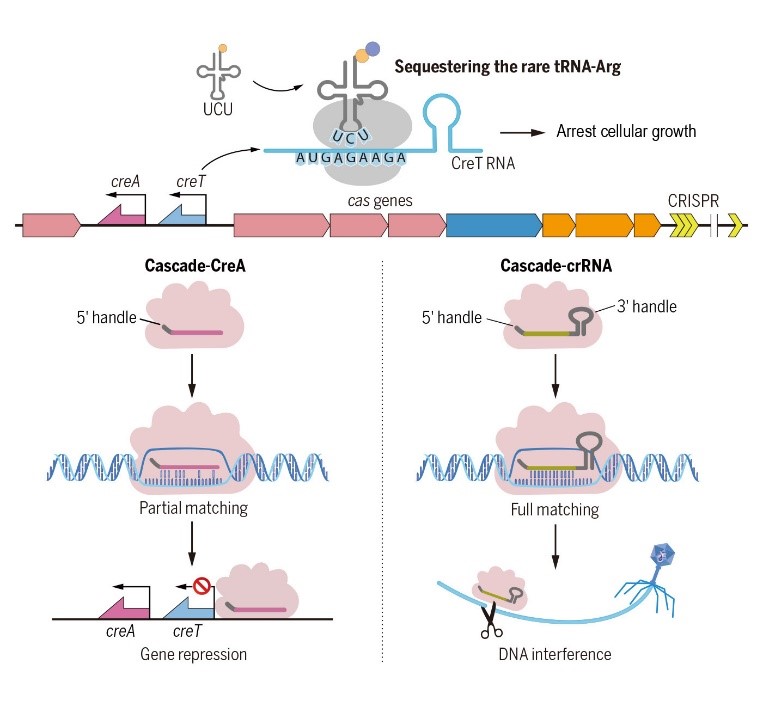
图1. CRISPR-Cas系统与CreTA毒素-抗毒素系统的互作机制
早在2014年,向华/李明团队即利用西班牙盐盒菌(Haloarcula hispanica)及其病毒在国际上建立了第一个I型CRISPR系统的高效适应模型,揭示了CRISPR系统对病毒高效适应需要引发的规律,并深入解析了“引发适应”过程精细的分子机制,包括Cascade与crRNA的可塑性。在这一研究过程中,他们发现了一个奇怪的现象: 4个成簇的编码 CRISPR效应复合物Cascade的基因(cas6-cas8-cas7-cas5)无法单独敲除,但可以作为整体一起敲除,从而推测这个基因簇内部可能隐藏了一个未知的“细胞成瘾”元件。经过近7年的探索,他们最终在cas6与cas8之间一段仅311 bp的基因间区内发现了一类全新的小RNA毒素-抗毒素系统,分别命名为CreT(RNA毒素)和CreA(RNA抗毒素)。CreTA通过与CRISPR效应复合物4个编码基因的结构与功能的偶联,守护了CRISPR-Cas系统的稳定性(图1)。
主要创新性发现:
1、首次发现受Cascade蛋白控制的小RNA毒素
该团队首先通过敲除cas6和cas8基因间区的311 bp序列获得?TA菌株,然后基于该菌株成功获得了Cascade各单基因缺失菌株。接着,他们利用携带这一段311 bp基因间区的质粒(pTA)转化各突变株,发现在Cascade基因缺失(?cas5-8)或其单基因缺失(?TA?cas5/6/7/8)的菌株中,pTA均表现出细胞毒性(转化效率降低约4个数量级),而在Cascade基因完整表达的?TA菌株中,pTA的细胞毒性则被完全抑制。上述系列实验表明,311 bp的基因间区产生了某种未知毒素,命名为Cascade抑制型毒素(Cascade-repressed Toxin),即CreT(图2A和2B)。
为确定CreT的具体序列,该团队通过截短实验发现CreT毒性来自于一段132bp的序列(图2A中-54到+78),其中有一个典型的启动子序列,可起始转录产生了一个78nt的小RNA。突变分析进一步表明,该启动子及小RNA内部一段茎环结构的突变均导致CreT毒性的消失(图2C),说明CreT是一种具有细胞毒性的小RNA分子。CreT 的诱导表达实验进一步证明了其抑菌(bacteriostatic)而非杀菌 (bactericidal)活性(图2D和2E)。
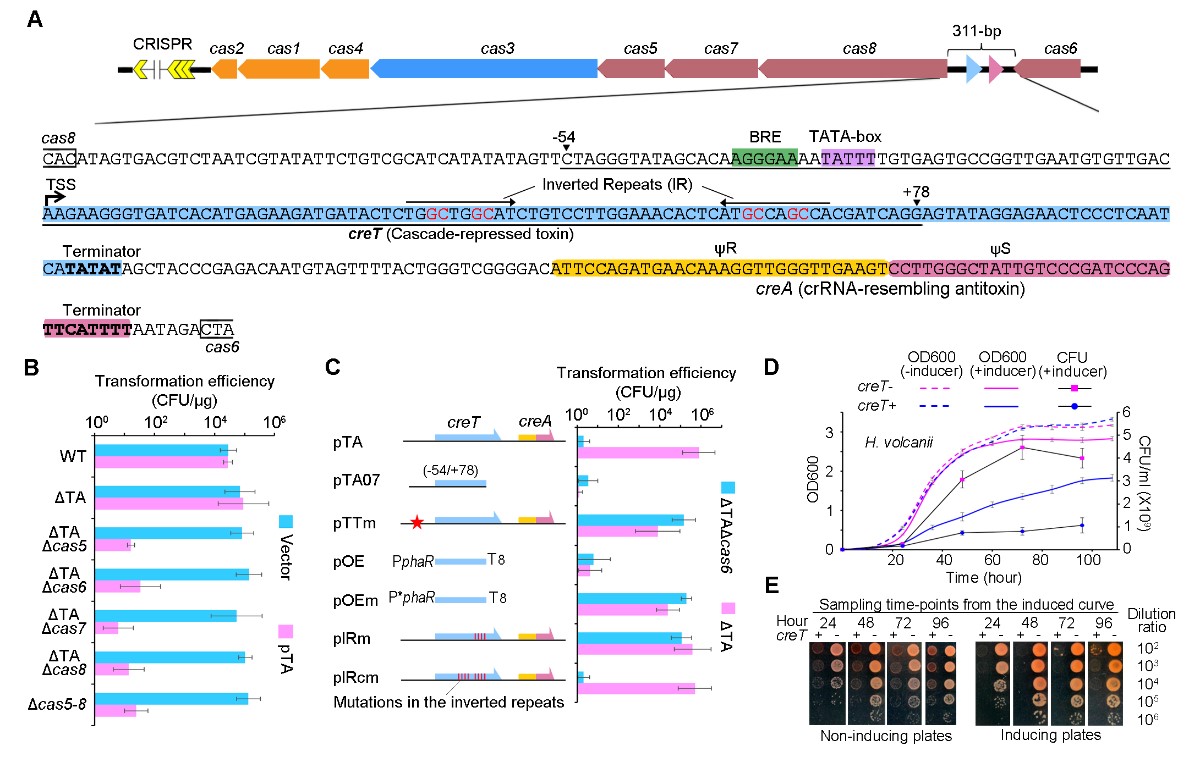
图2 . creTA在CRISPR-cas基因簇中的位置及功能分析
2、解析了小分子RNA毒素CreT独特的抑菌机制
对CreT RNA的精细序列和结构分析表明,其5'端具有强效的翻译信号SD(Shine-Dalgarno)序列和AUG起始密码,紧接其后的是两个精氨酸稀有密码子AGA, 随后是终止密码子UGA和一个茎环结构(图3A)。系列突变实验表明,上述关键序列和结构都是CreT毒性所必需。基于此,该团队推断了一个全新的分子回路,即CreT RNA借助强效的翻译起始信号定位在核糖体上,然后通过两个串联的稀有密码子AGA进一步劫持胞内稀有的精氨酸tRNAUCU,从而抑制了胞内其它重要蛋白的翻译合成和细胞生长。基于这一猜想,他们通过过表达tRNAUCU成功解除了CreT的细胞毒性(图3B),证实了这一全新的毒性机制,即CreT 是通过其RNA劫持胞内稀有的精氨酸tRNA发挥毒性,而非翻译产生含二精氨酸的小肽发挥作用,因此CreT是一类小RNA毒素。
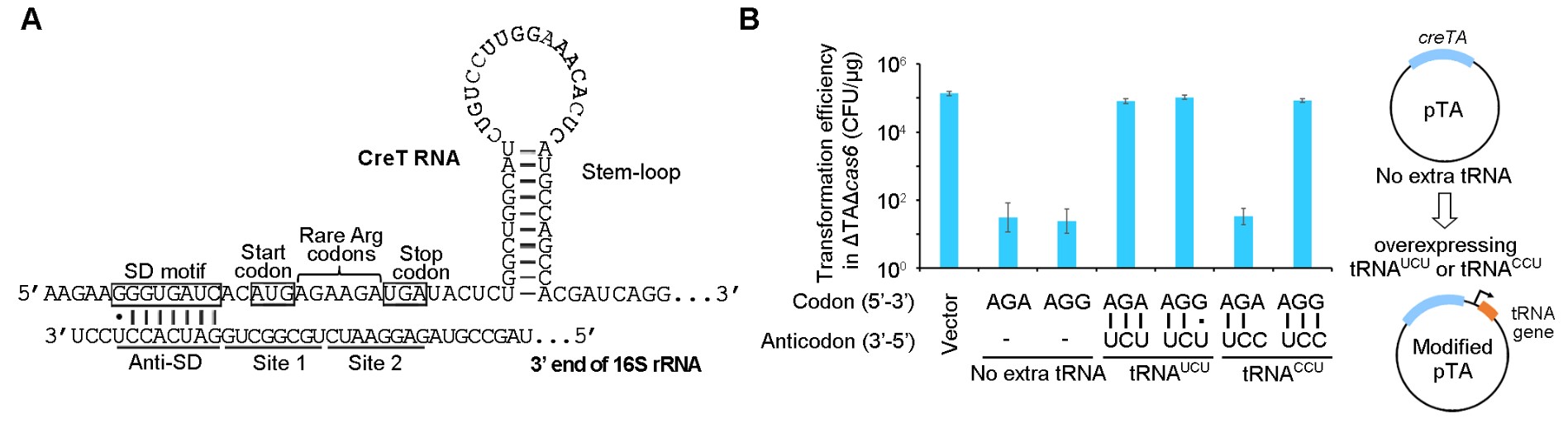
图3. CreT毒素通过劫持胞内稀有的精氨酸tRNAUCU抑制菌体生长
3、发现CreA抗毒素——类似crRNA的小分子RNA
为揭示Cascade蛋白抑制CreT毒性的分子机制,该团队首先敲除了胞内唯一的CRISPR array结构,从而排除了CRISPR array来源的crRNA参与毒性抑制的可能性。对311bp基因间区的截短与突变实验表明,creT下游序列为毒性抑制所必需(图2C)。精细的序列分析和Northern印迹等实验表明,creT下游有一段与CRISPR repeat高度相似的序列(图2A),该序列及其下游序列可独立转录产生一个约90nt的前体RNA,并被Cas6加工为一个的成熟的小RNA(图4A)。该小RNA约41nt,含有与crRNA一样的完整的5' handle序列(8 nt),特定的spacer序列(约33nt),但缺乏crRNA常见的3' handle,因此命名为“类crRNA抗毒素”(CrRNA-resembling Antitoxin),简称CreA。CreA作为crRNA类似分子,即可能联合Cascade蛋白对CreT毒素活性发挥抑制作用。

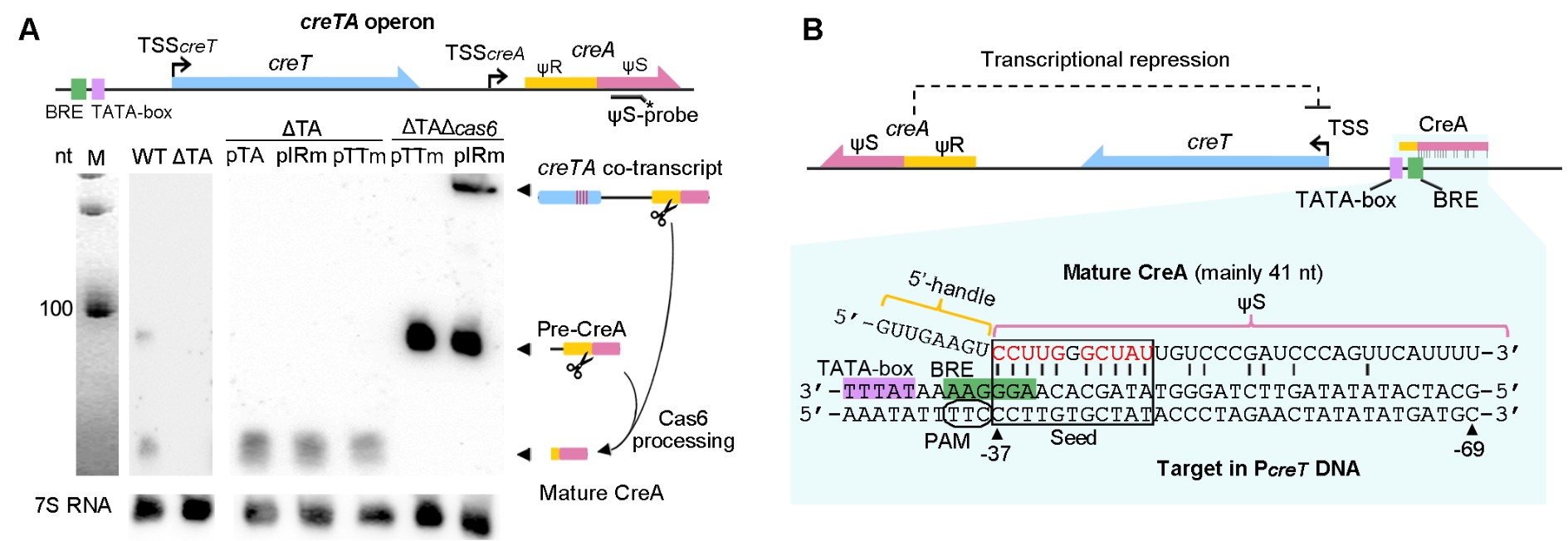
图4. CreA模拟crRNA指导Cascade特异性抑制creT启动子
4、揭示CreA RNA联合Cascade发挥抗毒素活性的分子机制
该团队进一步揭示,与crRNA介导Cascade复合物识别外源靶序列一样,CreA介导Cascade精确识别creT启动子(图4B)。CreA种子序列区(5' handle后约11nt)与creT启动子完全匹配(除不参与碱基匹配的第6个核苷酸外),而其余大部分序列不匹配。对CreA的5' handle序列和种子序列区的突变、以及对保守PAM(protospacer adjacent motif)序列的突变,都将导致CreA对 creT启动子抑制活性的丧失,进一步证明CreA联合Cascade实现了对CreT的转录水平调控,从而在正常情况下可抑制CreT的表达及毒性。
特别需要指出的是,该团队早期实验已经揭示了crRNA的高度可塑性,其结构(如是否含有3' handle序列)及spacer与靶序列匹配长度的不同,可以指导Cascade对外源靶标DNA实现干扰或引发适应两种不同的生理效应。本研究进一步发现CreA与creT基因启动子序列之间的不完全匹配性指引了多亚基Cascade效应物结合并抑制毒素启动子的活性,从而实现对毒素基因的转录水平调控,这在国际上首次揭示了多亚基CRISPR效应物固有的基因调控生理功能。
5、揭示CreTA对CRISPR-Cas系统的护卫功能
通过研究在有无CreTA存在时,活跃的可移动元件IS(insertion element)对cascade基因的破坏行为,该团队进一步证实了CreTA作为一个极简的“成瘾”元件保护CRISPR-Cas系统的生理功能(图5)。当CreTA存在时,可以保证Cascade基因(cas6-cas8-cas7-cas5)不被可移动元件插入失活,而当CreTA丢失后,由于CRISPR-Cas系统固有的适合度代价,Cascade基因被IS元件频繁破坏。

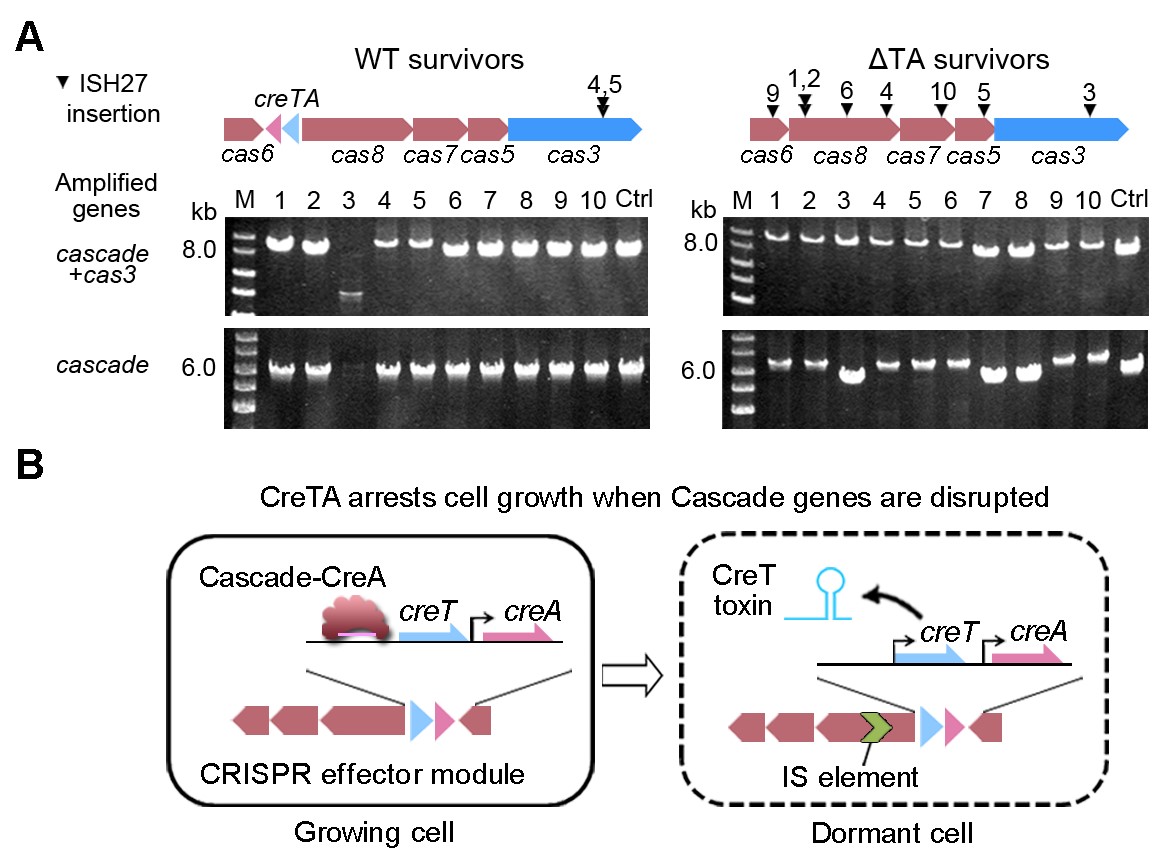
图5. CreTA保护Cascade基因簇的遗传稳定性
6、揭示CreTA 同源或类似系统在不同微生物和不同CRISPR亚型中的普遍存在
该团队还通过深入挖掘现有的微生物基因组数据,并与美国NCBI生物信息领域知名专家Eugene V Koonin教授团队合作,进一步发现多种古菌/细菌的不同类型CRISPR-Cas系统中潜藏着CreTA类似物,暗示CRISPR-Cas可能普遍利用CRISPR效应物固有的基因调控功能(如抑制一个毒性RNA的转录)对冲其适合度代价。这一全新机制的发现从新的视角解释了CRISPR-Cas在微生物中的广泛分布。
值得特别强调的是,已知的毒素-抗毒素(TA)系统都编码一个毒素蛋白,而该工作发现的CreTA利用了一个极简的小RNA(CreT)作为毒素组分,而且其抗毒素(CreA)也是一个小RNA,并依赖于Cascade复合物发挥功能,因此,CreTA或可定义一个新的TA分类单元。更重要的是,这类系统全新的分子机制为基因工程和基因编辑等应用提供了重要的元件和启示,例如,该团队一方面已利用CreT开发了可在细菌和古菌中通用的极简的反向筛选标记,为基因工程提供了新元件;另一方面基于CreA调控creT转录的分子回路开发了同步实现基因编辑和基因调控的新技术(均已申请专利)。
该发现不仅在CRISPR-Cas及其偶联系统生物学研究中具有里程碑意义,而且为研究原核微生物“非编码RNA暗物质世界”敞开了一道门。该论文指出在不同古菌和细菌不同类型的 CRISPR-Cas系统中发现的CreTA类似物在序列上多样性丰富,可能蕴藏了大量未知的毒性机制和功能元件。因此,这类丰富多样的“暗物质”的深入发掘将进一步推动生物技术的发展,包括对未来小RNA药物的研制或将具有启发意义。
中国科学院微生物研究所向华研究员和李明研究员为该论文的共同通讯作者,李明研究员、向华研究组博士后龚路遥和博士生程飞跃为并列第一作者。美国国立卫生研究院(NIH)生物技术信息中心(NCBI)Eugene Koonin教授及其团队给予了重要帮助。
向华研究员自2009年获得国家****科学基金项目资助开始CRISPR-Cas系统的前沿研究,现为微生物资源前期开发国家重点实验室主任、微生物研究所副所长、国家重大研究计划重点项目负责人、国家重点研发计划项目首席科学家。李明于2009年进入向华实验室硕博连读并专注于CRISPR研究,于2020年获得国家优秀青年项目资助,现为中国科学院微生物生理与代谢工程重点实验室青年课题组长。该工作得到了中科院战略先导研究计划、国家重点研发计划、国家自然科学基金、国家转基因重大科技专项、中国科协青年人才托举工程、中国科学院青年创新促进会等项目的支持。
论文链接:
https://science.sciencemag.org/content/372/6541/eabe5601
