 , 文湘华1
, 文湘华1
 , 张冰1, 秦文韬1,2, 陈湛1, 邱勇1
, 张冰1, 秦文韬1,2, 陈湛1, 邱勇11. 清华大学环境学院, 环境模拟与污染控制国家重点联合实验室, 北京 100084;
2. 北京市农林科学院植物与环境保护研究所, 北京 100097
收稿日期: 2020-05-26; 修回日期: 2020-06-17; 录用日期: 2020-06-17
基金项目: 中国国家自然科学基金项目(No.51678335);环境模拟与污染控制国家重点联合实验室项目(No.18L02ESPC)
作者简介: 陈慧钰(1995-), 女, E-mail:chenhuiyu726@163.com
通讯作者(责任作者): 文湘华, E-mail:xhwen@tsinghua.edu.cn
摘要:原后生动物是污水生物处理系统中的常见生物,了解它们是否能够摄食与去除抗生素抗性菌(ARB)和抗生素抗性基因(ARGs),对于建立藉捕食作用的ARB和ARGs去除技术具有重要意义.本研究以大型溞作为代表生物,探究了其对ARB和ARGs的摄食、去除效果与机制.使用荧光抗性菌直接观察其摄食过程,证实大型溞对ARB的摄食与富集.分析了溶液中和肠道中的ARB数目及drfA基因丰度变化,发现大型溞对ARB的摄食过程符合一级动力学;不同ARB浓度会影响大型溞的摄食及去除效果,在高ARB浓度下具有较高的过滤速率和滤食速率;大型溞的摄食减少了试验体系中的drfA基因丰度,检测到大型溞肠道内的DNA酶,其可能是导致其丰度减少的原因.
关键词:大型溞抗生素抗性菌(ARB)抗生素抗性基因(ARG)摄食过程消化酶
Ingestion and digestion of antibiotic resistance bacteria and genes by Daphnia in water
CHEN Huiyu1
 , WEN Xianghua1
, WEN Xianghua1
 , ZHANG Bing1, QIN Wentao1,2, CHEN Zhan1, QIU Yong1
, ZHANG Bing1, QIN Wentao1,2, CHEN Zhan1, QIU Yong11. Environmental Simulation and Pollution Control State Key Joint Laboratory, School of Environment, Tsinghua University, Beijing 100084;
2. Institute of Plant and Environment Protection, Beijing Academy of Agriculture and Forestry Sciences, Beijing 100097
Received 26 May 2020; received in revised from 17 June 2020; accepted 17 June 2020
Abstract: Zooplankton is commonly found in biological wastewater treatment process. It is very important to know if they can ingest and reduce the antibiotic resistance genes (ARGs) and antibiotic resistance bacteria (ARB) in order to develop biological predation and removal method of ARB and ARGs. So that,Daphnia magna (D. magna) was used as a representative species and its predatory process and removal mechanism for ARB and ARGs were studied. The genetically modified ARB E. coli MG1655 with a fluorescent protein gene which allows directly microscopic observation of feeding process was used. Hence the uptake of ARB by D. magna was evidenced and confirmed. Changes of ARB concentrations and drfA gene abundance were analyzed to determine the grazing kinetics. It was found that the ARB concentrations affected the filter-feeding process which follows first-order kinetics. At high ARB concentration,D. magna has higher filtering rate and grazing rate of ARB. The abundance of drfA gene decreased by the grazing process of D. magna. The DNase activity in the gut matters of D. magna was detected which probably catalyzed the digestion of ARGs.
Keywords: Daphnia magnaantibiotic resistance bacteria (ARB)antibiotic resistance genes(ARGs)filtering feedingdigestive enzyme
1 引言(Introduction)抗生素自问世以来, 在畜牧养殖、病虫害防治和临床医学等多个领域中得到了广泛的应用, 被视为人类最伟大的发现之一(Larsson, 2014; Van Boeckel et al., 2014).但近年来, 由于抗生素的滥用, 种种不良后果开始浮现.不仅影响环境中生物的生存和繁殖, 改变生态系统的群落结构和生态功能, 也导致了环境中抗生素抗性基因(Antibiotic resistance genes, ARGs)和抗生素抗性菌(Antibiotic resistance bacteria, ARB)的日益增加和广泛传播, 甚至导致了“超级细菌”的出现, 对公共健康和环境安全产生严重威胁(Livermore, 2003;Alanis, 2005;Rizzo et al., 2013; Van Boeckel et al., 2014; Rodriguez-Mozaz et al., 2015).已有研究提出, 应将ARGs视为一种新兴环境污染物(Pruden et al., 2006; Rodriguez-Mozaz et al., 2015).
污水处理系统, 被认为是ARGs重要的源和汇(Auerbach et al., 2007; Rizzo et al., 2013).作为新的研究热点, 许多研究都对污水处理厂中的ARGs和ARB进行了检测.研究表明, 在污水处理系统中, ARGs和ARB的丰度高、种类多(Schwartz et al., 2003;Auerbach et al., 2007; Rodriguez-Mozaz et al., 2015), 并且, 在污水处理厂中发生ARGs在细菌之间水平转移的可能性更高(Walsh, 2000;Schluter et al., 2007).而这些ARB随出水进入环境后, 会进一步加剧ARG的扩散和转移(Rizzo et al., 2013).
原后生动物可以以细菌、藻类、悬浮有机颗粒等为食, 在污水处理厂生物处理系统中起着重要的生态作用(Madoni et al., 1996; Mara et al., 2003; Snelling et al., 2006; Papadimitriou et al., 2010).在污水处理系统中, 原后生动物能够捕食活性污泥颗粒, 减少曝气池中的细菌总数(Pau et al., 2013; Serra et al., 2016).利用原后生动物的减量化作用可以减少出水的颗粒物浓度, 提高出水水质(Shiny et al., 2005;Serra et al., 2014).但原后生动物的吞噬作用对ARB和ARGs会产生何种影响, 相关研究很少.
大型溞(Daphnia magna)是一种枝角类淡水浮游动物, 在多种环境水体中均有分布, 是研究中常用的模式生物(Juergens, 1994).Eckert等发现在实验室条件下, 大型溞能够摄食四环素抗性细菌, 并且摄食结束后, 在大型溞肠道内的微生物基因组中能够检测到四环素抗性基因(Eckert et al., 2016).目前的研究主要关注了摄食压力的存在引起接合转移频率的提高(Olanrewaju et al., 2019), 并未关注其对于环境中ARB和ARGs的消除效果.因此, 拟使用大型溞作为微型后生动物的代表生物, 研究其①如何摄食水中ARB; ②不同ARB浓度下, 大型溞对ARB的摄食动力学与去除效果; ③大型溞的摄食活动对ARGs的影响.目标是揭示水环境中原后生动物与ARGs、ARB之间的关系, 为认识原后生动物对ARGs的作用乃至建立ARB与ARGs控制技术提供理论依据和支持.
2 材料与方法(Materials and methods)2.1 大型溞的培养大型溞采集于山东烟台的湖泊中, 经过实验室6个月以上的传代培养, 置于含有30 L人工水溞培养基(Artificial Daphnia medium, ADaM)(Manakul et al., 2017)的水箱中, 缓慢匀速曝气.温度(21 ± 1) ℃, 光暗比16 h:8 h.每隔一日喂食螺旋藻粉与酵母混合物, 每周更换一次1/3体积的培养基.试验开始前收集体长1.5~2.5 mm的大型溞.所有受试生物均为健康、有活力、体型均匀的个体.使用前清洗3次受试溞, 喂食交联葡聚糖凝珠(Sephadex G-25)以充分清空肠道内容物(Schallenberg et al., 2005), 并放置于黑暗环境24 h.
2.2 ARB的培养2.2.1 试验用Escherichia coliMG1655(MGS)试验用ARB为Escherichia coli MG1655(MGS), 来源于丹麦技术大学Bath Smets教授所赠予.E.coli MG1655中含有携带甲氧苄啶抗菌基因(drfA)的pKJK5质粒, 染色体上具有编码红色荧光蛋白(RFP)的基因(Musovic et al., 2006;Klumper et al., 2015).荧光显微镜下可观察到该菌株产生红色荧光.激发波长为561 nm, 发射波长为600~620 nm.
2.2.2 菌培养与菌液配制试验菌种的培养.取0.4 mL E.coli MG1655菌液加入含有30 mg·mL-1甲氧苄啶的营养肉汤培养基中, 37 ℃, 150 r·min-1振荡培养24 h.
试验菌液的配置.收集过夜培养后的菌液, 10000 r·min-1离心15 min, 弃去上清液, 收集沉淀.使用无菌ADaM培养基重新稀释至菌液OD600 = 1(≈109 CFU·mL-1)后, 再稀释成相应浓度的菌液.
2.3 试验体系2.3.1 大型溞对ARB的摄食试验试验探究大型溞能否摄食ARB.试验体系见表 1.温度、光照等条件同2.1节中的培养条件.试验时间结束后, 收集大型溞, 仔细清洗.使用激光共聚焦显微镜对大型溞肠道进行观察以确定摄食情况.
表 1(Table 1)
| 表 1 摄食试验条件 Table 1 The predatory test design | ||||||||||||||||||||
表 1 摄食试验条件 Table 1 The predatory test design
| ||||||||||||||||||||
2.3.2 大型溞摄食及去除ARB和ARG的特性试验体系见表 2.试验分为2个阶段:摄食阶段(stage 1)和去除阶段(stage 2).选用不含有成熟卵的大型溞, 试验阶段不会产生新的幼体.在摄食阶段, 研究不同初始ARB浓度下, 大型溞对ARB的摄食情况及体内富集的ARB变化情况; 去除阶段, 研究停止从外界摄食ARB后, 大型溞对已摄食的ARB的去除情况.摄食阶段和去除阶段的试验均在1000 mL的玻璃烧杯中进行, 温度(21 ± 1) ℃, 光暗比16 h:8 h.每个试验进行3个平行重复.
表 2(Table 2)
| 表 2 大型溞摄食及去除ARB和ARG的试验体系 Table 2 Experimental setup for ingestion and digestion of ARB and ARG by D. magna | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 2 大型溞摄食及去除ARB和ARG的试验体系 Table 2 Experimental setup for ingestion and digestion of ARB and ARG by D. magna
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
在摄食阶段(stage 1)中, 共设置了4个不同的菌液浓度(107、106、105、104 CFU·mL-1), 每个浓度包含一个试验组(ES1)与两个对照组(CAS1、CBS1).均设置6个重复.其中, 摄食阶段的试验只取其中的3个重复进行平行试验, 保留剩余3个重复组进行去除阶段的研究.
在去除阶段(stage 2)中, 将摄食阶段(stage 1)中ES1和CAS1组的3个重复命名为ES2和CAS2.将其中的大型溞取出, 快速清洗后, 置于无菌ADaM培养液中.其余条件与摄食阶段相同.
2.4 采样方法① 水中ARB的数目(NS).从液面下方1/3吸取水样.平板计数法检测ARB数目.
② 大型溞体内的ARB数目(ND).取5只大型溞, 仔细清洗, 分离外壳, 收集肠道部分.将肠道充分匀浆破碎.平板计数法检测ARB数目.
③ 水中drfA基因丰度(QS).使用0.45 μm醋酸纤维膜过滤烧杯中溶液, -20 ℃存储备用.使用实时定量PCR(Realtime-quantitative PCR, RT-qPCR)检测样品中drfA基因的丰度.
④ 大型溞体内drfA基因丰度(QD).取5只大型溞, 清洗后收集肠道部分.-20 ℃存储备用.使用RT-qPCR检测drfA基因丰度.
⑤ 大型溞体内脱氧核糖核酸酶活力(UD).试验结束后, 取20只大型溞, 收集肠道部分, 进行脱氧核糖核酸酶活力测定.
2.5 检测方法2.5.1 细菌计数样品倍比稀释后, 涂布于含有30 mg·mL-1甲氧苄啶抗生素的伊红美蓝培养平板上, 37 ℃培养18~24 h后计数.
2.5.2 实时定量qPCR(RT-qPCR)DNA提取.根据美国MOBIO DNA isolation kit试剂盒的操作指南提取DNA.使用微量分光光度计(NanoDropTM One, ThermoFisher, 美国)对DNA样品进行浓度和纯度的测定.样品-20 ℃存储.
菌种E.coli MG1655所含质粒pKJK5已经过测序, 针对目的基因dfrA使用的引物为(5′-CAATGGCTGTTGGTTGGACGACGACCGCATACTTT CGGTT -3′).使用PowerUpTM SYBRTM Green的操作说明配置反应体系.扩增条件如下:UDG酶激活阶段, 50 ℃, 2 min; 预变性阶段, 95 ℃, 2 min; 变性, 95 ℃, 15 s; 退火与延伸, 60 ℃, 1 min, 共40个循环.对比标准曲线, 确定丰度.
2.5.3 脱氧核糖核酸酶(DNA酶)活力测定粗酶液的制备.收集的肠道样品加入200 μL磷酸缓冲液, 使用微量组织匀浆器充分研磨.4 ℃, 10000 r·min-1下离心5 min, 弃去沉淀, 保留上清液.
脱氧核糖核酸酶活力的测定.根据中华人民共和国国家标准GB/T34801—2017(国家质量监督检验检疫总局, 2017), 使用分光光度法测定DNA酶活力.一个酶活力单位(U)的定义为:21 ℃、pH 5.0条件下, 溶液的A260在1 min内增加0.001所对应的酶量.
2.6 数据分析过滤速率与滤食速率.过滤速率(F, Filtering rate)为单位时间内单个动物可滤食干净的水的体积, 滤食速率(G, Grazing rate)为单位时间内单个动物滤食的细胞数目.
过滤速率与滤食速率依照式(1)~(2)进行计算(Makoto and Tsutomu, 1984):
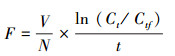 | (1) |
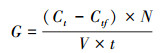 | (2) |
摄食动力学分析.以往的研究表明, 大型溞对藻类和有机颗粒物等食物颗粒的摄食过程符合一级反应动力学(Burnet et al., 2017; Serra et al., 2019):
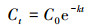 | (3) |
试验数据Microsoft Excel 2019软件进行数据处理和统计分析, 利用Origin 2019进行绘图.对滤食速率、酶活力等数据进行单因素方差分析(ANOVA).p < 0.05表示显著性差异, p < 0.01表示极显著差异.
3 结果与讨论(Results and discussion)3.1 大型溞对ARB的摄食在共聚焦显微镜下观察纯培养的E.coli MG1655细菌, 呈现分散的红色颗粒状(图 1a).试验所使用的大型溞如图 1b所示.
图 1(Fig. 1)
 |
| 图 1 显微镜观察试验生物 Fig. 1Microscopic observation of experimental organisms |
在摄食试验中, 分别使用共聚焦显微镜和荧光显微镜对摄食了ARB菌液的大型溞肠道进行了观察(图 1c、1d).肠道中充满红色荧光ARB, 透明的甲壳部分几乎不存在红色荧光.大型溞能够在短时间内摄食水中的ARB并富集在肠道中.
试验选用荧光菌E.coli MG1655作为试验生物, 可肉眼观察到大型溞对ARB的摄食和富集, 提供直接证据.通常的观察方法需要使用化学染料或荧光试剂, 这些试剂具有毒性, 且需要通过杀灭试验生物进行固定和染色.该方法避免了传统显微染色技术的缺点, 能够观察ARB从体外进入大型溞的动态摄食过程.
3.2 大型溞对ARB的摄食与去除过程3.2.1 不同ARB浓度对摄食动力学的影响摄食阶段(stage 1)中, ARB数目随时间变化的情况如图 2所示.在摄食过程中, 不含有大型溞的对照组CB中的ARB数目基本保持稳定, 而不同浓度的试验组中的ARB数目均明显下降.
图 2(Fig. 2)
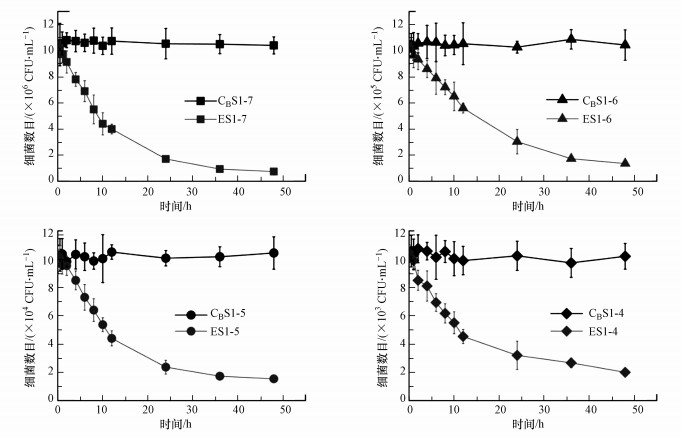 |
| 图 2 摄食阶段(stage1)不同浓度试验组中溶液ARB数目变化 Fig. 2Changes of ARB concentrations in water solution at different concentrations during stage 1 |
参照式(2)~(3)对各组大型溞摄食ARB的过程进行了反应动力学拟合.结果表明, 大型溞对ARB的摄食过程符合一级反应动力学, 参数见表 3.
表 3(Table 3)
| 表 3 不同ARB浓度下摄食动力学拟合参数 Table 3 Grazing kinetics fitting results at different ARB concentrations | |||||||||||||||
表 3 不同ARB浓度下摄食动力学拟合参数 Table 3 Grazing kinetics fitting results at different ARB concentrations
| |||||||||||||||
试验初始ARB浓度C0为107 CFU·mL-1时, ARB减少的速率最快, 速率常数k最大, 为0.071;C0= 104 CFU·mL-1时, k最小, 为0.039.Burnet等使用k值来表征大型溞对水环境中大肠杆菌的去除速率(loss rate)(Burnet et al., 2017), 发现在高细菌密度下, 细菌的去除速率要显著高于低细菌密度下的去除速率, 与本试验结果一致.
3.2.2 不同ARB浓度对摄食动力学的影响过滤速率和滤食速率是衡量滤食性浮游动物摄食过程的重要指标.摄食阶段中, 随着初始ARB浓度的增加, 大型溞的滤食速率和过滤速率增加(图 3).ARB浓度为~107 CFU·mL-1时, 过滤速率和滤食速率最大, 为1.38 mL·h-1·ind-1, 相应的单位时间所摄食的细菌数目也最多.试验结果表明, 在较高的起始ARB浓度下大型溞具有较高的过滤速率和滤食速率, 单位时间能够摄食更多的ARB, 具有更强的净水能力.
图 3(Fig. 3)
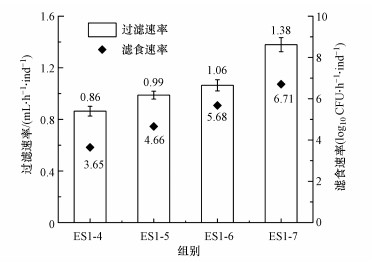 |
| 图 3 摄食阶段(stage 1)大型溞对不同浓度ARB的过滤速率和滤食速率 Fig. 3Filtering rate and grazing rate of D. magna at different initial ARB concentrations in stage 1 |
在自然条件下, 大型溞的过滤速率为0.1~2.8 mL·h-1·ind-1 (Jurgens et al., 1994; Schallenberg et al., 2005), 与本试验结果相符.造成不同过滤速率和滤食速率的因素有多种, 例如动物的体长、食物的种类及食物颗粒粒径等.
研究结果表明, 不同起始的ARB浓度对大型溞的过滤速率和滤食速率产生了影响.在高ARB浓度组中检测到了较高的过滤速率和滤食速率, 而低浓度下反之.一个原因是, 高ARB浓度下, 细菌密度大, 大型溞接触到细菌的概率较大.大型溞的滤食是通过附肢搅动微小范围中的水流并截留其中的食物颗粒, 因此单位时间内, 大型溞能够同时摄食更多的细菌.此外, 有研究提出, 在食物密度较大的情况下, 大型溞的摄食行为会产生“背负式吸收(piggybacking)”现象(Porter et al., 1983), 即食物颗粒团聚成块或较大的颗粒堵塞了水蚤的过滤附肢时, 散布在水环境中的细菌或颗粒会通过附着在较大的颗粒或者胸腔附肢上后被大型溞摄食进入体内, 从而大大提高了摄食量(Rosenkranz et al., 2009).
3.2.3 摄食阶段大型溞肠道内ARB的变化摄食阶段中大型溞体内的ARB数目随时间变化如图 4a所示.在摄食阶段中, 大型溞摄入ARB, 体内数目在开始时逐渐增加, 一段时间后趋于稳定.不同ARB浓度的试验组中, 其增加的速率不同.当起始ARB浓度为~107 CFU·mL-1时, 大型溞体内的ARB最快达到稳定数目, 并且体内的ARB数目最高.当起始ARB浓度较低, 为~105 CFU·mL-1和~104 CFU·mL-1时, 平衡时刻其体内ARB数目均较低.
图 4(Fig. 4)
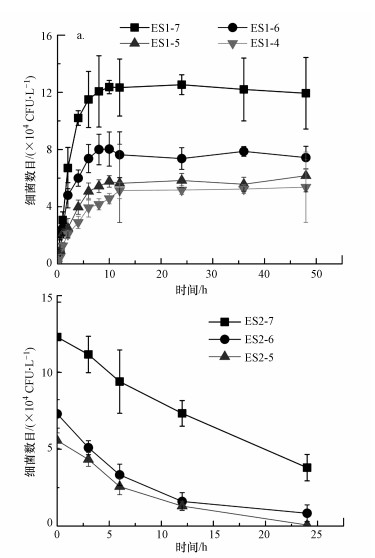 |
| 图 4 摄食阶段(stage1)(a)及去除阶段(stage2)(b)大型溞体内ARB数目变化 Fig. 4Changes of ARB concentrations in the gut of D. magna in stage Ⅰ (a) and stage Ⅱ(b) |
试验所选用的大型溞具有相似的大小、体长和活力, 因此大型溞肠道容量基本相同.理论上讲, 如果给予足够的时间, 最终达到平衡状态时, 大型溞体内的细菌数目应当十分接近.然而, 试验发现, 在起始浓度较低时, 平衡状态下大型溞体内的ARB数目相较高浓度组的数目要少.当ARB被大型溞摄食进入体内后, 一部分ARB在大型溞肠道内的消化酶和微生物群的共同作用下被破坏分解, 另一部分随着粪便排泄进入环境.当摄食的ARB数目与大型溞肠道内消化量相等时, 体内的ARB数目达到平衡.因此, 影响大型溞摄食富集ARB数目的原因主要是摄入数量和消化能力的差异.试验结果表明, 不同初始浓度下, 过滤速率和滤食速率存在差异, 导致了摄入数量的不同.对于消化能力而言, 食物颗粒的浓度会影响大型溞的消化效率(Hebert, 1978).当食物颗粒浓度较低时, 细菌在肠道中通过的时间会增加, 因此大部分的细菌能够被充分分解消化(Searle et al., 2013).而在高浓度的食物颗粒条件下, 肠道中的细菌密度更高, 在团聚块或食物颗粒内部的细菌对不利环境和消化酶的抵抗能力更强, 因此能在肠道中生存更长的时间(King et al., 1991; Olanrewaju et al., 2019), 能够在平板上生长并被检出.因此高ARB浓度试验组中的数目明显高于低ARB浓度组.
3.2.4 去除阶段中体系内ARB的变化为了探究ARB进入大型溞体内后的情况, 分析了大型溞体内的ARB数目随时间变化(图 4b).随着时间的增加, 大型溞体内的ARB数量逐渐下降, ARB起始浓度为~107 CFU·mL-1的试验组中大型溞体内的ARB数目下降速率最快, 而起始浓度为~106 CFU·mL-1和~105 CFU·mL-1的试验组中, 大型溞体内的ARB下降速率没有显著差异.
在去除阶段开始一段时间后, 在原本无菌的水环境中检测到了ARB的存在(图 5).在较短时间内水溶液中的ARB数目增加, 达到最高值后, 又随时间逐渐降低.这一试验结果表明, 大型溞在向体外排出的肠道内容物中, 依然存在能在抗性平板上生长的ARB.
图 5(Fig. 5)
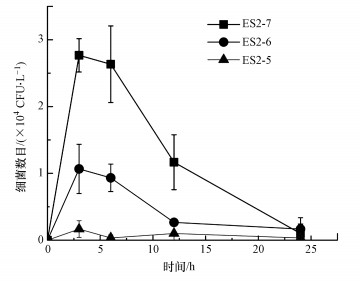 |
| 图 5 去除阶段(stage2)水环境中ARB数目变化 Fig. 5Changes of ARB concentrations in water solutionin stage Ⅱ |
在去除阶段中, 大型溞肠道中一部分的ARB被排泄到体外, 进入水环境中, 其中一些细菌仍然具有活力.而进入水环境中的ARB数目在试验开始时先增加, 然后又减少, 这可能是由于大型溞循环摄食体外细菌所致(Schallenberg et al., 2005).有研究表明, 大型溞能够反复摄取外部环境中的食物颗粒, 甚至是自己排泄出的粪便颗粒(“食粪性”)(Pilati et al., 2004;Burnet et al., 2017).尽管无法简单将大型溞肠道内的ARB浓度与水环境中的ARB浓度简单相加得到总量, 但两者均有不同程度的降低, 反映整个体系中的ARB总量随着时间减少.这表明, 大型溞对于ARB的消化和分解速率要高于对ARB的排泄速率和ARB本身的增殖速率.整体上看, 大型溞的摄食与消化有助于减少环境中的ARB.
3.3 大型溞对ARG的去除摄食阶段结束后, 对水样中的drfA基因进行定量分析(图 6).对比对照组和试验组的drfA基因丰度发现, 不同初始ARB浓度的试验组中, 经过大型溞的摄食活动后, drfA基因的丰度均显著降低.
图 6(Fig. 6)
 |
| 图 6 摄食阶段(stage1)结束后试验组和对照组drfA丰度对比 Fig. 6Comparison of the abundance of drfA between the control groups and the treatment groups at the end of stage Ⅰ |
去除阶段大型溞体内drfA基因的变化见图 7.不同起始ARB浓度的试验组中, 大型溞体内的drfA丰度均随时间减少.
图 7(Fig. 7)
 |
| 图 7 去除阶段(stage2)大型溞体内drfA丰度变化 Fig. 7Changes of the abundance ofdrfA in the gut of D. magna in stage Ⅱ |
环境中的ARB被大型溞摄入后, 在大型溞体内通过酶解消化, 这些过程与消化酶的作用息息相关.例如, 大型溞消化道中的蛋白酶、淀粉酶和脂肪酶等在食物消化分解中起着重要作用(Hebert, 1973; 1978).因此, 可以合理推测drfA基因丰度的降低可能是由肠道中的核酸酶引起的.为了验证这一推测, 对大型溞肠道内的DNA酶活力进行检测, 结果如表 4所示.
表 4(Table 4)
| 表 4 大型溞体内DNA酶活力 Table 4 DNase activity in the gut of D. magna | ||||||||||
表 4 大型溞体内DNA酶活力 Table 4 DNase activity in the gut of D. magna
| ||||||||||
对照组和试验组的大型溞肠道中均检测到DNA酶的活力, 对照组的酶活力最低.浓度为~107 CFU·mL-1组中的酶活力最高, 但差异不显著(p>0.05).在肠道中检测出的DNA酶的存在, 其对DNA的分解作用可能是导致试验组中drfA基因的丰度下降的原因.在已有报道中, 未发现对大型溞体内的DNA酶活力开展的测定和研究.本试验首次对大型溞肠道内该酶的活力进行了测定, 但对于该酶的具体来源, 例如是大型溞肠道细胞分泌的, 或是共生微生物群所产生的, 有待进一步的鉴定和分析.
本研究验证了大型溞对抗生素抗性细菌的摄食, 研究不同起始浓度下, 摄食阶段与去除阶段水环境与大型溞体内的ARB数目与抗性基因drfA丰度随时间的变化情况, 分析了大型溞的摄食、消化等活动对ARB和drfA的去除效果.试验结果表明, 大型溞能够有效减少环境中的ARB数目及其所携带的抗性基因drfA的丰度, 提高出水质量.试验所使用的是人工配置的溶液, 在今后的研究中, 可使用实际出水或环境水体进行进一步的研究, 评估应用大型溞进行生物处理减少ARB和ARGs的可能性并探索适宜条件.
4 结论(Conclusions)1) 浮游微型后生动物大型溞摄食并富集水环境中的ARB.大型溞对ARB的摄食过程符合一级反应动力学, 起始ARB浓度对反应动力学的参数有影响.高ARB浓度下, 大型溞的滤食速率常数、过滤速率和滤食速率较高, 单位时间内能够滤食清除更多细菌.当抗性菌浓度为~107 CFU·mL-1时, 过滤速率最大, 为1.38 mL·h-1·ind-1, 当抗性菌浓度较低(~104 CFU·mL-1)时, 过滤速率仅为0.86 mL·h-1·ind-1.
2) 大型溞摄食ARB后, 体内ARB数目先增加, 一段时间后趋于稳定.在去除阶段中, 大型溞体内的ARB数目逐渐下降, 而原本无菌的水环境中, 短时间先出现细菌数目的上升, 随后又降低.大型溞对ARB的消化和分解速率高于对ARB的排泄和ARB的增殖速率.大型溞的摄食与消化有助于减少环境中的ARB.
3) 大型溞的摄食减少了试验体系中的抗性基因drfA丰度.体内的drfA丰度随时间明显减少, 可能是其体内存在的DNA酶分解了该抗性基因.
参考文献
| Alanis Alfonso J. 2005. Resistance to antibiotics:are we in the post- antibiotic era?[J]. Archives of Medical Research, 36(6): 697-705. DOI:10.1016/j.arcmed.2005.06.009 |
| Auerbach Eric A, Seyfried Erin E, McMahon Katherine D. 2007. Tetracycline resistance genes in activated sludge wastewater treatment plants[J]. Water Research, 41(5): 1143-1151. DOI:10.1016/j.watres.2006.11.045 |
| Burnet J B, Faraj T, Cauchie H M, et al. 2017. How does the cladoceran Daphniapulex affect the fate of Escherichia coli in water?[J]. PLoS One, 12(2): e0171705. DOI:10.1371/journal.pone.0171705 |
| Eckert E M, Di Cesare A, Stenzel B, et al. 2016. Daphnia as a refuge for an antibiotic resistance gene in an experimental freshwater community[J]. Science of the Total Environment, 571: 77-81. DOI:10.1016/j.scitotenv.2016.07.141 |
| 国家质量监督检验检疫总局. 2017. GB/T 34801-2017脱氧核糖核酸酶活力检测方法[S].北京: 中国标准出版社 |
| Hebert P D. 1978. The population bilogy of Daphnia (Crustacea, Daphnidae)[J]. Biological Reviews, 53(3): 387-426. DOI:10.1111/j.1469-185X.1978.tb00860.x |
| Herbert P D. 1973. Phenotype variability of lactate dehydrogenase in Daphniamagna[J]. The Journal of Experimental Zoology, 186(1): 33-38. DOI:10.1002/jez.1401860106 |
| Juergens K. 1994. Impact of Daphnia on planktonic microbial food webs:A review[J]. Marine Microbial Food Webs, 8(1/2): 295-324. |
| Jurgens K, Gasol J M, Massana R, et al. 1994. Control of heterotrophic bacteria and protozoans by daphnia-pulex in the epilimnion of lake ciso[J]. Archiv Fur Hydrobiologie, 131(1): 55-78. |
| King C H, Sanders R W, Shot E B, et al. 1991. Differential survival of bacteria ingested by zooplankton from a stratified eutrophic lake[J]. Limnology and Oceanography, 36(5): 829-844. DOI:10.4319/lo.1991.36.5.0829 |
| Klumper U, Riber L, Dechesne A, et al. 2015. Broad host range plasmids can invade an unexpectedly diverse fraction of a soil bacterial community[J]. ISME Journal, 9(4): 934-945. DOI:10.1038/ismej.2014.191 |
| Larsson D G. 2014. Antibiotics in the environment[J]. Upsala Journal of Medical Sciences, 119(2): 108-112. DOI:10.3109/03009734.2014.896438 |
| Livermore D M. 2003. Bacterial resistance:origins, epidemiology, and impact[J]. Clinical Infectious Diseases, 36(Suppl 1): S11-23. |
| Madoni P, Davoli D, Gorbi G, et al. 1996. Toxic effect of heavy metals on the activated sludge protozoan community[J]. Water Research, 30(1): 135-141. DOI:10.1016/0043-1354(95)00124-4 |
| Makoto O, Tsutomu I. 1984. Methods in marine zooplankton ecology[M]. New York: John Wiley & Sons. 162-169 |
| Manakul P, Peerakietkhajorn S, Matsuura T, et al. 2017. Effects of symbiotic bacteria on chemical sensitivity of Daphniamagna[J]. Marine Environmental Research, 128: 70-75. DOI:10.1016/j.marenvres.2017.03.001 |
| Mara D, Horan N J. 2003. Handbook of water and wastewater microbiology[M]. California: Academic press. 69-76 |
| Musovic S, Oregaard G, Kroer N. 2006. Cultivation- independent examination of horizontal transfer and host range of an IncP-1 plasmid among gram- positive and gram-negative bacteria indigenous to the barley rhizosphere[J]. Applied and Environmental Microbiology, 72(10): 6687-6692. DOI:10.1128/AEM.00013-06 |
| Olanrewaju T O, McCarron M, Dooley J S G, et al. 2019. Transfer of antibiotic resistance genes between Enterococcus faecalis strains in filter feeding zooplankton Daphnia magna and Daphnia pulex[J]. Science of the Total Environment, 659: 1168-1175. DOI:10.1016/j.scitotenv.2018.12.314 |
| Papadimitriou C A, Papatheodoulou A, Takavakoglou V, et al. 2010. Investigation of protozoa as indicators of wastewater treatment efficiency in constructed wetlands[J]. Desalination, 250(1): 378-1869. DOI:10.1016/j.desal.2009.09.060 |
| Pau C, Serra T, Colomer J, et al. 2013. Filtering capacity of Daphnia magna on sludge particles in treated wastewater[J]. Water Research, 47(1): 181-186. DOI:10.1016/j.watres.2012.09.047 |
| Pilati A, Wurtsbaugh W A, Brindza N R. 2004. Evidence of coprophagy in freshwater zooplankton[J]. Freshwater Biology, 49(7): 913-918. DOI:10.1111/j.1365-2427.2004.01234.x |
| Porter K G, Feig Y S, Vetter E F. 1983. Morphology, flow regimes, and filtering rates of daphnia, ceriodaphnia, and bosmina fed natural bacteria[J]. Oecologia, 58(2): 156-163. DOI:10.1007/BF00399211 |
| Pruden A, Pei R, Storteboom H, et al. 2006. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 40(23): 7445-7450. |
| Rizzo L, Manaia C, Merlin C, et al. 2013. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment:a review[J]. Science of the Total Environment, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| Rodriguez-Mozaz S, Chamorro S, Marti E, et al. 2015. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 69: 234-242. DOI:10.1016/j.watres.2014.11.021 |
| Rosenkranz P, Chaudhry Q, Stone V, et al. 2009. A comparison of nanoparticle and fine particle uptake by Daphnia magna[J]. Environmental Toxicology and Chemistry, 28(10): 2142-2149. DOI:10.1897/08-559.1 |
| Schallenberg M, Bremer P J, Henkel S, et al. 2005. Survival of Campylobacter jejuni in water:Effect of grazing by the freshwater crustacean Daphnia carinata (Cladocera)[J]. Applied and Environmental Microbiology, 71(9): 5085-5088. DOI:10.1128/AEM.71.9.5085-5088.2005 |
| Schluter A, Szczepanowski R, Puhler A, et al. 2007. Genomics of IncP-1 antibiotic resistance plasmids isolated from wastewater treatment plants provides evidence for a widely accessible drug resistance gene pool[J]. FEMS Microbiology Reviews, 31(4): 449-477. DOI:10.1111/j.1574-6976.2007.00074.x |
| Schwartz T, Kohnen W, Jansen B, et al. 2003. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms[J]. FEMS Microbiology Ecology, 43(3): 325-335. DOI:10.1111/j.1574-6941.2003.tb01073.x |
| Searle C L, Mendelson J R, Green L E, et al. 2013. Daphnia predation on the amphibian chytrid fungus and its impacts on disease risk in tadpoles[J]. Ecology and Evolution, 3(12): 4129-4138. DOI:10.1002/ece3.777 |
| Serra T, Colomer J. 2016. The hydraulic retention time on the particle removal efficiency by Daphniamagna filtration on treated wastewater[J]. International Journal of Environmental Science and Technology, 13(6): 1433-1442. DOI:10.1007/s13762-016-0985-4 |
| Serra T, Colomer J, Pau C, et al. 2014. Tertiary treatment for wastewater reuse based on the Daphnia magna filtration - comparison with conventional tertiary treatments[J]. Water Science Technology, 70(4): 705-711. DOI:10.2166/wst.2014.284 |
| Serra T, Müller M F, Barcelona A, et al. 2019. Optimal light conditions for Daphnia filtration[J]. Science of the Total Environment, 686: 151-157. DOI:10.1016/j.scitotenv.2019.05.482 |
| Shiny K J, Remani K N, Nirmala E, et al. 2005. Biotreatment of wastewater using aquatic invertebrates, Daphnia magna and Paramecium caudatum[J]. Bioresource Technology, 96(1): 55-58. DOI:10.1016/j.biortech.2004.01.008 |
| Snelling W J, Moore J E, McKenna J P, et al. 2006. Bacterial-protozoa interactions; an update on the role these phenomena play towards human illness[J]. Microbes and Infection, 8(2): 578-587. DOI:10.1016/j.micinf.2005.09.001 |
| Van Boeckel T P, Gandra S, Ashok A, et al. 2014. Global antibiotic consumption 2000 to 2010:an analysis of national pharmaceutical sales data[J]. The Lancet Infectious Diseases, 14(8): 742-750. DOI:10.1016/S1473-3099(14)70780-7 |
| Walsh C. 2000. Molecular mechanisms that confer antibacterial drug resistance[J]. Nature, 406(6797): 775-781. DOI:10.1038/35021219 |
