 , 邱勇2, 李冰3, 王硕1, 李激1
, 邱勇2, 李冰3, 王硕1, 李激1
1. 江南大学环境与土木工程学院, 无锡 214122;
2. 清华大学环境学院, 北京 100084;
3. 北京科技大学能源与环境工程学院, 北京 100083
收稿日期: 2018-12-06; 修回日期: 2019-02-23; 录用日期: 2019-02-23
基金项目: 国家水体污染控制与治理科技重大专项(No.2017ZX07202001-004);国家自然科学基金(No.51778325,51708023)
作者简介: 朱引(1993-), 男, E-mail:zyfreely@126.com;
李激, E-mail:liji@jiangnan.edu.cn
摘要: 应用活细胞三维成像技术,实时和原位观测了活性污泥菌群中抗生素抗性基因转移动态特征.微流控芯片是一种细菌生长的良好载体,活细胞成像系统低光毒性不损伤细菌活性,两者结合能有效实现基因转移的长时间原位观测,能够区分抗性基因的水平转移和垂直转移.结果表明:垂直转移是污泥菌群中抗性基因的主要转移方式,菌群纵向转移速率与其厚度有关,菌群内部转移率较高,基因转移速率为0.24~0.29 h-1.
关键词:微流控芯片活细胞成像抗生素抗性基因菌群
Transfer dynamics of antibiotic resistance genes in sludge bacterial community using live cell 3D imaging technology
ZHU Yin1
 , QIU Yong2, LI Bing3, WANG Shuo1, LI Ji1
, QIU Yong2, LI Bing3, WANG Shuo1, LI Ji1
1. School of Environment and Civil Engineering, Jiangnan University, Wuxi 214122;
2. School of Environment, Tsinghua University, Beijing 100084;
3. School of Energy and Environmental Engineering, University of Science and Technology Beijing, Beijing 100083
Received 6 December 2018; received in revised from 23 February 2019; accepted 23 February 2019
Abstract: The transfer dynamics of antibiotic resistance genes (ARGs) in activated sludge bacterial community was observed in situ and real time, using a microfluidic chip coupled with live-cell three-dimensional (3D) imaging system. The microfluidic chip was designed for cultivating bacteria, and the live-cell 3D imaging system could record gene transfer process without lose bacteria activity due to its low phototoxicity. By combining the two systems, long time in-situ observation of gene transfer in bacterial community was achieved, meanwhile, horizontal and vertical gene transfer could be distinguish. The results show that vertical gene transfer was the major way for transfer of antibiotic resistance genes in sludge bacterial community. The distribution of transconjugants in the vertical direction was related with the community thickness. The transfer rate inside the community was higher than the surface, which varied in the range of 0.24~0.29 h-1.
Keywords: microfluidic chiplive-cell imaging systemantibiotic resistance genebacterial community
1 引言(Introduction)近年来, 随着抗生素在医药、畜牧等行业的大规模使用, 造成了抗生素在环境中大量残留(Pruden et al., 2013).大部分残留的抗生素在环境中可存在较长时间, 易诱导产生抗生素抗性菌(Gao et al., 2012), 抗生素抗性菌携带的抗生素抗性基因(Antibiotic resistance gene, ARG)可通过水平转移与垂直转移两种方式在细菌间转移扩散, 从而增加环境中的抗生素耐药菌数量(Shakibaie et al., 2009).研究表明, ARG的转移扩散会给人类健康及生态环境造成严重的危害(S?rensen et al., 2005), 有必要引起重视.
城市污水处理厂接受各种污染源排放的抗性基因, 并通过不同途径排放到自然环境中, 是环境中抗性基因的主要排放源(窦春玲等, 2013).目前污水处理工艺主要设计用来降低有机污染物, 对ARG去除效果有限, 大量研究发现在污水处理系统出水中仍可以检测出多种ARG(Heuer et al., 2002; B?rjesson et al., 2009; Munir et al., 2011).为了更好地理解污水处理过程中ARG的扩散行为、控制相关过程产生的风险, 有必要深入研究ARG在污泥菌群中转移的行为特征.
目前对菌群中ARG转移的研究较多(Angles et al., 1993;李蒙英等, 2006; Palyada et al., 2009), 一般采用培养和分子生物学的研究手段.这些方法都是终点法, 即在转移全部结束后对结果进行观测.终点法不能提供菌群形成过程中ARG转移的动态信息, 不利于对其转移过程的理解和控制.本研究基于微流控芯片培养方法和高分辨率活细胞成像技术, 构建了一个可实时原位观察菌群形成过程中ARG转移特征的系统, 得到了ARG在污泥菌群间转移过程中的动态变化和空间特征.
2 材料与方法(Materials and methods)2.1 供受体细菌及其培养方法供体菌为大肠杆菌(Escherichia coli)MG1655, 来自丹麦科技大学Barth教授课题组.供体菌携带具有甲氧苄胺嘧啶抗性的PKJK5质粒, 该抗性质粒用绿色荧光蛋白(GFP)标记, 同时供体菌染色体上携带了编码红色荧光蛋白(RFP)和LacI蛋白的基因.在供体菌中GFP受到抑制无法表达, 而在接合子中因缺乏抑制子而得到表达.因此, 供体菌可表达RFP而抑制GFP, 转移接合子表达GFP, 从而抗性质粒转移的结果得以可视化.采用甲氧苄胺嘧啶的质量浓度为30 μg·mL-1的LB培养基(胰蛋白胨10 g·L-1, 酵母提取物5 g·L-1, 氯化钠10 g·L-1), 在转速为150 r·min-1的摇床中恒温30 ℃过夜培养供体菌.
受体菌为提取自无锡市某污水处理厂好氧池的活性污泥, 提取过程如下:用高速破碎仪(BilonJJ-2, 中国上海Bilon公司)间歇破碎污泥60 min, 破碎后的污泥在4 ℃冰箱静置过夜.取200 μL上清液接种于20 mL模拟污水培养基, 并于30 ℃、150 r·min-1条件下摇床过夜培养.模拟污水成分:蛋白胨8 g·L-1, 尿素1.5 g·L-1, 牛肉膏5.5 g·L-1, 氯化钠0.35 g·L-1, 七水合硫酸镁0.1 g·L-1, 二水合氯化钙0.2 g·L-1, 磷酸氢二钾1.4 g·L-1.
2.2 微流控芯片实验采用如图 1所示的微流控芯片进行细胞培养, 多层结构包括PDMS(聚二甲基硅氧烷)芯片、琼脂糖层和玻片.PDMS芯片(24 mm×24 mm×6 mm)制取、琼脂糖层制备方法可参考文献(Wong et al., 2010).实验时分别取1 mL的供体菌和受体菌液于离心管中, 反复3次高速离心和PBS清洗.用模拟污水培养基将菌液稀释至OD600为0.3左右, 再将供体菌和受体菌按1:1等体积混合.取5 μL供体菌和受体菌的混合液接种在琼脂糖层和下盖玻片之间, 盖上PDMS芯片.然后, 将模拟污水培养基注射到PDMS的小孔内, 通过琼脂糖层扩散供给细菌利用.最后, 将上盖玻片边缘贴合在芯片上表面, 防止培养基蒸发和外界污染.
图 1(Fig. 1)
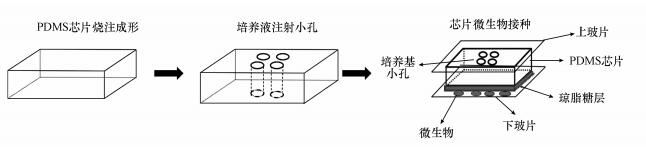 |
| 图 1 多孔PDMS芯片实验示意图 Fig. 1Porous PDMS chip production process |
2.3 活细胞成像与实时原位观测使用活细胞成像系统平台(型号DeltaVision Elite, 美国)观测多孔PDMS芯片上混合菌群的形成过程.除了实时获得明场的信号, 还同时使用561 nm激发波长观测供体菌表达RFP的红色荧光信号, 以及使用488 nm激发波长观测受体菌表达GFP的绿色荧光信号.
在供受体菌接种至芯片上培养2 h后, 在芯片上选3个已出现GFP信号的位置进行拍摄观察.实验拍摄间隔为1 h, 总时长为23 h.拍摄的总厚度设置为26 μm, 厚度间隔设置为1 μm, 实验平台温度控制在27 ℃.同时开启自动追焦, 以避免焦平面错位.
2.4 实验数据处理2.4.1 图像处理方法采用Imaris图像处理软件(Imaris 9.0, 瑞士)对实验图像数据进行分析.首先, 根据荧光图像的三维像素构建三维表面视觉图像(3D surface), 使荧光信号图像表面完整包覆三维像素, 将实际三维图像的体积与厚度关联起来.然后, 利用软件的“biofilm analysis”功能, 计算出不同表面位置对应的菌群厚度(单位μm)与生物量(单位μm3).接种细菌在芯片上形成局部细胞团块前处于单细胞状态, 因此可用ImageJ软件对Imaris导出的图片进行处理和计数, 根据荧光表达情况, 计算不同时间的供体菌、受体菌和污泥菌细菌个数, 可拟合出细菌生长曲线.
2.4.2 抗性基因扩散速率的计算方法抗性基因在菌群中的扩散包括水平与垂直方向上的扩散, 垂直方向的扩散与菌群厚度相关(Li et al., 2018), 而菌群的厚度在菌群成熟稳定后处于平衡状态(Brann et al., 2017), 因此水平方向上的扩散速率更能反映抗性基因在菌群中的扩散速度.抗性基因可以通过转移子增长过程进行扩散, 即垂直转移.描述转移子生长过程的数学模型包括以下重要参数:比生长速率、延迟期和最大生长量.使用转移子的荧光面积近似表征细菌的数量, 可以利用指数生长模型描述转移子生长过程.
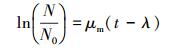 | (1) |
3 结果与讨论(Results and discussion)3.1 污泥菌群中ARG的转移过程图 2A显示了接种细菌在芯片上生长形成污泥菌群的过程, 依次经历细菌散落附着在玻片上(早期定殖阶段)、局部细菌团块(集聚阶段)、致密膜样结构(成熟阶段).在接种后第10 h, 污泥菌群基本覆盖观测区域.随后, 污泥菌群随着细菌增殖而不断增厚.实验终点时, 如图 2C污泥菌群三维结构图所示, 所形成污泥菌群呈现出不同厚度, 表面变得很不平整, 污泥菌群中细菌接触十分紧密, 供体菌与转移子随机分布在污泥菌群中.
图 2(Fig. 2)
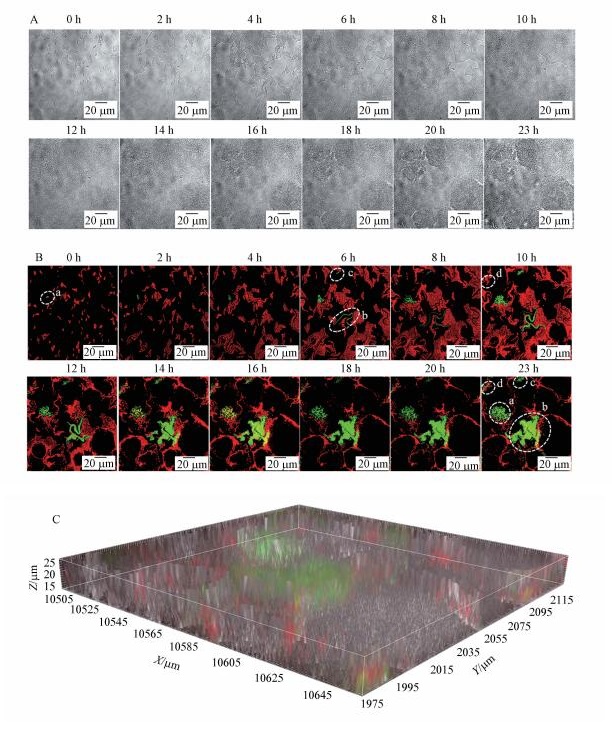 |
| 图 2 芯片上污泥菌群形成时序图 (A为明场图; B为荧光图; C为明场与荧光通道三维层扫图, 红色荧光微生物为供体菌Escherichia coli MG1655, 绿色荧光微生物为转移子, 灰色为明场信号) Fig. 2Time-lapse images of bacteria community formation on chip (A (brightfield), B(fluorescence); C: image of 3-D structure of the biofilm. Escherichia coli MG1655 emit red fluorescence, while sludge bacteria transconjugants emit green fluorescence. gray is the brightfield sign) |
图 2B的荧光时序图显示了抗生素抗性基因从供体菌水平转移到污泥受体菌的动态过程.接合转移是抗性基因水平转移的途径之一, 即抗性质粒通过供体菌和受体菌的菌毛形成通道进行水平转移, 因此转移子可能在接触的初期就出现, 比如图中a点; 更大的可能性是在菌群形成后再出现, 比如图中的b、c、d点.菌群中细菌之间接触更加紧密, 使得供受体菌发生水平转移的几率更大(S?rensen et al., 2005).
图 3显示了芯片上各细菌的增长曲线, 根据式(1)进行拟合可得:供体菌Escherichia coli MG1655的比增长速率为0.38 h-1、污泥菌的比增长速率为0.51 h-1、转移子的比增长速率为0.56 h-1.供体菌Escherichia coli MG1655与活性污泥菌比增长速率存在较大差异, 所以活性污泥菌会逐渐占据芯片上的主要生长空间.污泥菌与转移子的比增长速率基本一致, 即转移接合子的增加主要来源于细菌分裂生长, 表明除了水平转移之外, 垂直转移也是抗生素抗性基因在菌群中扩散转移的重要方式.
图 3(Fig. 3)
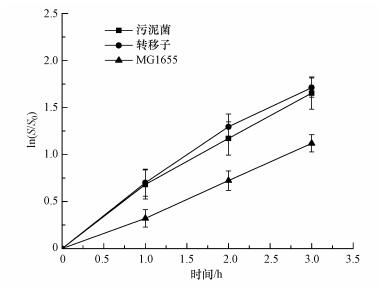 |
| 图 3 芯片上供受体菌及转移接合子的增长曲线 Fig. 3The growth curves of Escherichia coli MG1655, transconjugants and the activated sludge bacterial on the chip |
3.2 ARG在污泥菌群中的空间结构图 4a显示了转移子在菌群中的空间结构分布情况.利用Imaris图像处理软件对转移子区域进行三维重构和计算分析, 结果表明成熟的菌群表面高低起伏、细菌之间接触十分紧密.在水平方向上, 转移子分布具有一定的随机性, 大部分以菌落的形式镶嵌在菌群中.在垂直方向上, 转移子范围和菌群厚度相似, 扩散趋势与菌群形成厚度同步.
图 4(Fig. 4)
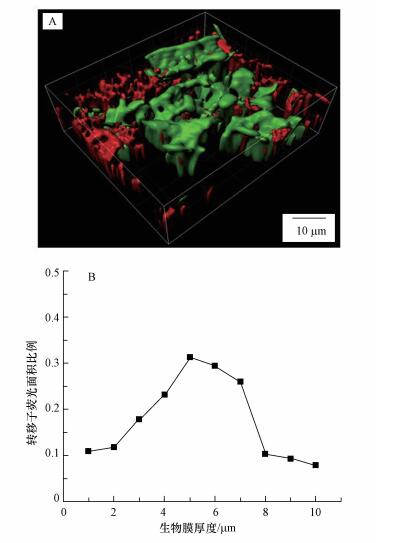 |
| 图 4 污泥菌群中转移接合子的三维分析 (a.荧光三维重构图-23 h; b.转移子在菌群中的分布-23 h) Fig. 4Three- dimensional analysis of transconjugants in sludge bacterial community (A.3D reconstruction model of 23 hour time-point; b. Green fluorescence area ratio of transconjugants in bacterial community -23 hour time-point |
计算某区域三维视图在不同水平面上的绿色荧光比例(图 4b), 可以看出菌群内部的转移子密度要明显高于底面和表面.这可能是因为菌群内部菌落结构稳定, 细菌之间接触程度更加紧密, 发生基因转移的几率更大.在生物膜法污水处理工艺中, 菌群厚度往往超过100 μm(王荣昌等, 2006), 且伴随表面细菌脱落与再定植过程, 因此容易增加抗生素抗性基因的内部转移和外界扩散, 需要引起重视.
3.3 ARG在污泥菌群中的转移速率污泥菌的比增长速率与转移子的比增长速率较为一致, 表明垂直转移是抗性基因在菌群中扩散转移的主要方式, 因此可以用转移子的生长模型来近似描述抗性基因在菌群中的扩散动力学.图 5显示了3个观测位置(P1~P3)的转移子相对数量(荧光面积占百分比)随时间变化的趋势.从图中可以看出, 首次水平转移出现的时间有明显区别, 如P2在0 h即出现首次水平转移, P1约为2 h, 而P3的首次转移晚于5 h.比较实验终点时转移子面积比例的最大值, 可以发现P3最大、P2最小.由于首次水平转移后, 还会多次出现水平转移和垂直转移, 这使得对整体趋势的预测变得比较困难.
图 5(Fig. 5)
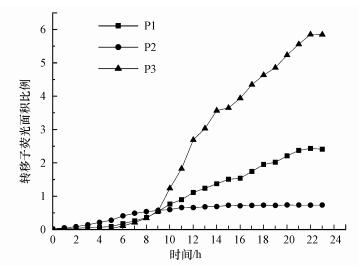 |
| 图 5 不同区域转移子绿色荧光面积比例变化 Fig. 5Green fluorescence area ratio of transconjugants in different position on chip during the experiment |
虽然转移子绿色荧光面积的最大值有明显差异, 但增长趋势体现出一定的相似性.可以看到, 转移子的增长趋势与典型的细菌生长曲线相似.采用式(1)的指数模型拟合增长数据, 可以得到P1~P3的比增长速率μ分别为0.27、0.29和0.24 h-1, 呈现出很好的一致性.基于荧光面积的比增长速率相对基于细胞数量的比增长速率值较小, 这是因为细胞数量增加主要在细胞分裂时出现变化, 而细胞分裂后引起的体积变化具有一定滞后性.但相比细菌数量变化, 采用荧光面积占百分比的比增长速率可以更加准确的反映抗性基因在菌群中的转移速率.
4 结论(Conclusions)1) 水平转移是抗性基因转移的重要机制, 垂直转移是抗性基因在污泥菌群中的主要转移方式.
2) 三维成像技术解释了转移子在污泥菌群中的空间分布.在平面上, 转移子呈现分散和菌落状分布; 在纵向上, 转移子分布连续均匀, 内部转移率高于表面.
3) 分析了污泥菌群中不同位置的转移子随时间变化趋势, 其增长曲线与细菌生长曲线相似.不同位置转移子产生时间和荧光面积最大值差异较大, 但比增长速率比较一致, 在0.24~0.29 h-1之间.
参考文献
| Angles M L, Marshall K C, Goodman A E. 1993. Plasmid transfer between marine bacteria in the aqueous phase and biofilms in reactor microcosms[J]. Applied & Environmental Microbiology, 59(3): 843. |
| B?rjesson S, Melin S, Matussek A, et al. 2009. Erratum to A seasonal study of the mecA gene and Staphylococcus aureus including methicillin-resistant S. aureus in a municipal wastewater treatment plant[J]. Water Research, 43(4): 925–932.DOI:10.1016/j.watres.2008.11.036 |
| Brann M, Suter J D, Addleman R S, et al. 2017. Monitoring bacterial biofilms with a microfluidic flow chip designed for imaging with white-light interferometry[J]. Biomicrofluidics, 11(4): 44113.DOI:10.1063/1.4985773 |
| 窦春玲, 郭雪萍, 尹大强. 2013. 污水处理厂抗生素抗性基因分布和去除研究进展[J]. 环境化学, 2013, 32(10): 1885–1893.DOI:10.7524/j.issn.0254-6108.2013.10.010 |
| Gao P, Munir M, Xagoraraki I. 2012. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Science of the Total Environment, s 421-422(3): 173–183. |
| Heuer H, Kr?gerrecklenfort E, Wellington E M H, et al. 2002. Gentamicin resistance genes in environmental bacteria:prevalence and transfer[J]. Fems Microbiology Ecology, 42(2): 289–302.DOI:10.1111/fem.2002.42.issue-2 |
| Li B, Qiu Y, Zhang J, et al. 2018. Real-Time study of rapid spread of antibiotic resistance plasmid in biofilm using microfluidics[J]. Environmental Science & Technology, 52(19): 11132–11141. |
| 李蒙英, 沈标, 李顺鹏. 2006. 生物膜中水平基因转移及其促成的生物强化作用[J]. 应用与环境生物学报, 2006, 12(3): 441–444.DOI:10.3321/j.issn:1006-687X.2006.03.034 |
| Munir M, Wong K, Xagoraraki I. 2011. Release of antibiotic resistant bacteria and genes in the effluent and biosolids of five wastewater utilities in Michigan[J]. Water Research, 45(2): 681–693.DOI:10.1016/j.watres.2010.08.033 |
| Palyada K, Sun Y Q, Flint A, et al. 2009. Characterization of the oxidative stress stimulon and PerR regulon of Campylobacter jejuni[J]. Bmc Genomics, 10(1): 481.DOI:10.1186/1471-2164-10-481 |
| Pruden A, Larsson D G, Amézquita A, et al. 2013. Management options for reducing the release of antibiotics and antibiotic resistance genes to the environment[J]. Environmental Health Perspectives, 121(8): 878–885.DOI:10.1289/ehp.1206446 |
| Shakibaie M R, Jalilzadeh K A, Yamakanamardi S M. 2009. Horizontal transfer of antibiotic resistance genes among gram negative bacteria in sewage and lake water and influence of some physico-chemical parameters of water on conjugation process[J]. Journal of Environmental Biology, 30(1): 45–49. |
| S?rensen S J, Bailey M, Hansen L H, et al. 2005. Studying plasmid horizontal transfer in situ:A critical review[J]. Nature Reviews Microbiology, 3(9): 700–710.DOI:10.1038/nrmicro1232 |
| Wong I, Atsumi S, Huang W, et al. 2010. An agar gel membrane-PDMS hybrid microfluidic device for long term single cell dynamic study[J]. Lab on a Chip, 10(20): 2710–2719.DOI:10.1039/c004719h |
| 王荣昌, 文湘华, 钱易. 2006. 悬浮载体生物膜内硝化菌群空间分布规律[J]. 环境科学, 2006(11): 2358–2362.DOI:10.3321/j.issn:0250-3301.2006.11.041 |
