 , 石伟, 张红, 刘勇, 李君剑
, 石伟, 张红, 刘勇, 李君剑

山西大学黄土高原研究所, 太原 030006
收稿日期: 2018-09-29; 修回日期: 2018-11-28; 录用日期: 2018-11-28
基金项目: 国家自然科学基金(No.41771548)
作者简介: 杜宏宇(1993-), 女, E-mail:Duhongyu1114@163.com
通讯作者(责任作者): 李君剑, E-mail:lijunjian@sxu.edu.cn
摘要: 抗生素抗性基因(Antibiotic resistance genes,ARGs)作为一种新型污染物已经成为国内外研究的热点.为了探究河套灌区不同排水渠(3N、3S、5N、5S和7N)沉积物中ARGs的分布情况,采用定量PCR测定了沉积物中ARGs、整合酶基因(intⅠ1和intⅡ2)和转座酶基因(tnpA)的丰度.结果表明,36种ARGs中检测到31种,在各样地中均能检测到intⅠ1和tnpA基因,而intⅡ2基因未检测到.在各样地中,tetA、tetS、sulⅠ和aadA1基因丰度较高;在5N样地ereA基因丰度最高,为1.65×109 copies·g-1;15种ARGs在7N样地与其它样地之间有显著差异,其中,aadA1基因在7N样地丰度最高,为1.37×109 copies·g-1.氮含量与sulⅢ、ereB、blaampC基因丰度显著正相关,而pH与tetX、tetO和tetM基因丰度呈负相关关系.综上所述,抗生素抗性基因广泛存在于河套灌区的沉积物中.
关键词:抗生素抗性基因河套灌区排水渠沉积物基因丰度
Distribution of antibiotic resistance genes in sediments of Hetao irrigation district
DU Hongyu
 , SHI Wei, ZHANG Hong, LIU Yong, LI Junjian
, SHI Wei, ZHANG Hong, LIU Yong, LI Junjian

Institute of Loess Plateau, Shanxi University, Taiyuan 030006
Received 29 September 2018; received in revised from 28 November 2018; accepted 28 November 2018
Abstract: Antibiotic resistance genes (ARGs), as an emerging environmental contaminant, has been a research hotspot in world. In this study, we collected sediments from different drainage channel (3N, 3S, 5N, 5S, 7N) in Hetao irrigation district. The abundance of 36 ARGs, integrase genes (intⅠ1 and 2) and transposase genes(tnpA) were determined quantitative real time PCR. The results showed that 31 ARGs, the intⅠ1 and tnpA genes were detected in all the sites, but the intⅡ2 gene was not detected. The abundances of tetA, tetS, sulⅠ and aadA1 were high in each plot. The highest abundance ARGs was the ereA gene with 1.65×109 copies·g-1 in the 5N plot, and aadA1 gene (1.37×109 copies·g-1) was the highest one in the 7N plot. The abundances of 15 ARGs in the 7N plot were significantly different from those in other plots (p < 0.05). Nitrogen demonstrated strongly positive correlations with the abundances of sulⅢ、ereB and blaampC genes. pH negatively correlated with the abundances of tetX, tetO, and tetM genes. In summary, ARGs are widely present in sediments in the Hetao irrigation district.
Keywords: antibiotic resistance genesHetao irrigation districtdrainage channelssedimentsabundance
1 引言(Introduction)抗生素的发现和使用是人类医学史上的巨大进步, 对人类健康及在畜牧养殖方面有重要作用.目前, 抗生素已被广泛用于治疗人类和动物的传染病, 并且还可以促进牲畜和水产养殖中动物的生长(Zhou et al., 2012).但由于抗生素的滥用, 且进入生物体内的抗生素难以被全部吸收, 都以原药或者代谢产物的形式随排泄物进入环境中, 进而对环境微生物产生选择压力, 导致细菌产生了各种抗生素抗性基因(Antibiotic Resistance Genes, ARGs), 使得细菌的耐药性增加(Martinez, 2009).在2006年, ARGs已经被世界健康组织(WHO)定义为一种新型的环境污染物, 在环境污染研究领域越来越受到关注(Pruden et al., 2006).
ARGs主要由移动基因元件(Mobile genetic elements, MGEs)(如质粒、整合子、转座子等)通过基因水平转移在不同个体之间, 以及不同环境水平中迁移、转化和传播扩散(Marti et al., 2014; 徐晨, 2017).一旦携带ARGs的微生物死亡后, 携带ARGs的DNA仍然可以在环境中长期存在(Crecchio et al., 2005; Hill et al., 1998).研究表明, ARGs具有持久性和足够的移动性, 并且比抗生素本身残留对环境和人类健康的危害更大(Martínez, 2008; 罗义等, 2008; Szekeres et al., 2018).
目前很多研究表明, ARGs已经在各种水体中(如河流、湖泊、饮用水和医院污水等)被检测到(Jiang et al., 2013; Ouyang et al., 2015; Li et al., 2016). Jiang等(2013)对黄浦江和上海饮用水来源中的ARGs和抗生素之间的关系进行了研究, 发现在郊区采集到的水样中ARGs的多样性和丰度普遍比城市地区的高, 饮用水源也在郊区地区或附近存在较高的ARGs污染.Lu等通过qPCR方法对黄河流域处理过程中的饮用水中17种ARGs和2种MGEs的变化研究发现, 在整个流域尺度上, 饮用水处理后ARGs和MGEs的绝对丰度下降, 但与以前的研究相比, 其平均水平比黄浦江要低(Jiang et al., 2013; Lu et al., 2018).而针对沉积物的研究也有一些报道, Luo等(2010)通过对天津海河流域中ARGs的存在情况进行了研究, 发现沉积物中磺胺类ARGs的丰度要比水样中高出120~2000倍, 由此认为沉积物是ARGs更稳定的储存库.Purden等(2012)研究了南佩雷特河上游人类活动对河水和沉积物中抗性基因丰度的影响, 结果表明, 四环素类和磺胺类抗生素抗性基因丰度与动物养殖场和污水处理厂的分布显著相关, 并且与它们之间的距离直接相关, 表明人类活动对抗性基因的传播有重要影响.
基于此, 本文以河套灌区排水渠沉积物为研究对象, 通过对沉积物理化性质的测定, 使用PCR和Q-PCR等方法来检测ARGs和MGEs基因的丰度和多样性, 探究其中ARGs的污染水平.同时, 通过分析不同沉积物的理化性质、ARGs、MGEs和16S rRNA之间的差异性, 以及它们之间的相关性, 来分析该地区与其他地区沉积物中污染物的分布差异, 以期为河套灌区ARGs的生态风险评价及污染防治提供数据支持和理论依据.
2 材料与方法(Materials and methods)2.1 研究区概况河套灌区位于内蒙古自治区西部, 地理坐标为40°19′~41°18′N, 106°20′~109°19′E, 是我国面积最大的自流灌区, 总面积达1.12×104 km2, 平均海拔为1005~1060 m.河套灌区地处我国干旱的西北高原, 常年降水量为145~216 mm, 且集中在7—8月, 年平均气温为5~9 ℃, 总日照时数为3085~3320 h, 平均风速为1.9~5.4 m·s-1.区域主要的农作物为小麦、向日葵、玉米和蜜瓜(冯兆忠等, 2003; Luan et al., 2018).
2.2 样品采集沉积物样品于2017年11月在河套灌区采集, 具体位置如图 1所示, 其中, 红色线条为灌渠, 蓝色线条为排水渠, 灌渠水来自黄河, 灌溉过农田之后从排水渠流经总排干进入乌梁素海, 最后汇入黄河.根据农业、工业和城市人口分布, 分别对第三、第五、第七排干的南端和北端进行采样, 共采集了5个样地, 分别为三排干北(3N)、三排干南(3S)、五排干北(5N)、五排干南(5S)、七排干北(7N).使用抓泥斗采集每个排干表层5 cm的沉积物为子样品, 将3个子样品合并混匀为一个独立样品, 装入无菌塑封袋中在4 ℃下保存, 每个样地采集3个样品作为重复.
图 1(Fig. 1)
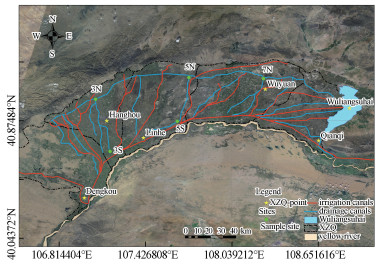 |
| 图 1 样地分布示意图 Fig. 1The sample sites |
2.3 样品分析2.3.1 沉积物理化性质的测定沉积物样品烘干称重后计算含水量, pH值以水土比2:1浸提后用pH计测定;土壤氮、碳和硫使用元素分析仪测定.
2.3.2 沉积物DNA的提取称取沉积物样品0.25 g, 根据PowerSoil DNA Isolation Kit(Mo-Bio, America)试剂盒说明书提取沉积物中总DNA, 提取的DNA采用多功能读版机Infinite 200 PRO(TECAN, Sweden)检测含量及提取纯度.
2.3.3 沉积物中ARGs的检测和定量分析对样品DNA进行抗生素抗性基因的定性PCR检测, 为下一步ARGs的定量检测做好条件预算的准备.PCR反应体系:25 μL扩增体系, 包括模板DNA 1.0 μL, Taq PCR Master Mix 12.5 μL, 上/下游引物各1.0 μL, ddH2O 9.5 μL.反应条件:94 ℃预变性5 min, 94 ℃变性30 s, 45~60 ℃退火30 s, 72 ℃延伸7 min.所得扩增产物用1.0%的琼脂糖凝胶电泳在120 V条件下进行检测.
本研究使用Bio-Rad CFX96 iCycler iQ5 thermocycler(CA, USA)进行实时荧光定量PCR, 选择了细菌16S rRNA基因、36种ARGs、1类和2类整合酶基因(intⅠ1和intⅡ2)和1种转座酶基因(tnpA)的引物(Luo et al., 2010; Zhu et al., 2013; Wang et al. 2014)进行检测, ARGs包括15种常见的四环素类抗生素抗性基因(tetA、tetC、tetE、tetG、tetK、tetL、tetA/P、tetS、tetX、tetM、tetO、tetQ、tetT、tetW、tetB/P)、3种磺胺类抗生素抗性基因(sulⅠ、sulⅡ、sulⅢ)、3种喹诺酮类抗生素抗性基因(qnrA、qnrB、qnrS)、3种大环内酯类抗生素抗性基因(ereA、ereB、mphA)、4种β-内酰胺类抗生素抗性基因(blaCTX-M、blaTEM、blaSHV、blaampC)和8种氨基糖苷类抗生素抗性基因(aac、aacC、aadD、aph、str、spcN-01、aadA1、strB).
反应体系10 μL:模板DNA 2 μL, iTaq Universal SYBR Green Supermix 5 μL, 上/下游引物各0.5 μL, ddH2O 2 μL.反应条件:95 ℃预变性3 min, 95 ℃变性30 s, 45~60 ℃退火30 s, 72 ℃延伸1 min, 最后在60~90 ℃之间每隔0.3 ℃停留5 s, 采集一次荧光以生成溶解曲线, 根据溶解曲线的变化检测扩增结果的特异性.
2.4 数据分析采用Microsoft Excel 2016和SPSS 19.0对数据进行处理和分析, 采用Microsoft Excel 2016和R 3.3.1做图, 使用单因素方差分析(one-way ANOVA)对不同样品之间的差异性进行分析, 用Spearman相关分析来研究沉积物理化性质、MGEs和ARGs之间的相关性.
3 结果(Results)3.1 沉积物理化性质从表 1可以看出, 各样地之间的pH、氮、碳和硫含量均有显著差异, pH在3S、5N和5S样地之间有显著差异, 氮在5N、5S和7N样地之间有显著差异, 碳在5S和7N样地之间有显著差异, 硫在3N和5N样地之间有显著差异.
表 1(Table 1)
| 表 1 各样地沉积物的pH、氮、碳和硫 Table 1 Sediment pH, nitrogen, carbon, sulfur from sample sites | |||||||||||||||||||||||||||||||||||
表 1 各样地沉积物的pH、氮、碳和硫 Table 1 Sediment pH, nitrogen, carbon, sulfur from sample sites
| |||||||||||||||||||||||||||||||||||
3.2 沉积物中16S rRNA基因、整合酶基因和转座酶基因的丰度图 2为沉积物中16S rRNA、intⅠ1和tnpA基因的丰度, 而intⅡ2未检测到, 其中, tnpA和16S rRNA的检出率为100%, intⅠ1的检出率为93.33%.7N样地沉积物中16S rRNA基因的丰度最高, 为3.91×1010 copies·g-1, 且与其他样地之间有显著差异(p < 0.05);5S样地沉积物中16S rRNA基因的丰度最低, 为4.28×109 copies·g-1;各样地沉积物之间的intⅠ1和tnpA基因的丰度没有显著差异, 丰度最高的样地分别为7N和5S.
图 2(Fig. 2)
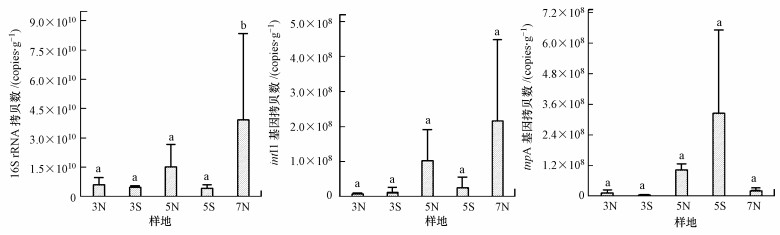 |
| 图 2 各样地沉积物16S rRNA、intⅠ1和tnpA基因的丰度 Fig. 2The abundances of 16S rRNA, intⅠ1 and tnpA genes in sample sites |
3.3 沉积物中各种ARGs的种类和丰度本研究共检测到ARGs 31种, 未检测到的5种, 分别为四环素类抗生素抗性基因tetA/P、喹诺酮类抗生素抗性基因qnrA和qnrB, 以及β-内酰胺类抗性基因blaCTX-M和blaTEM.
从图 3可以看出, 在所有检出的基因中, 5N样地中ereA基因相对丰度最高, 并且5N样地中检出的基因种类最多;而7N样地中检测到aadA1基因的相对丰度最高, 其次是sulⅠ基因, 而其他基因的占比相对较少.在各样地中, 相对丰度较高的为tetA、aadA1、sulⅠ、sulⅢ、tetS和ereA基因.在5个样地中, 7N样地检出的ARGs种类最少(26种), 3N和5S样地均为27种, 3S样地检出28种, 而5N样地检出最多, 为31种.按照分子机制将检测到的ARGs分为3类, 其中, 抗生素失活机制所占比例最大, 其次是细胞保护机制, 而外排泵机制的基因丰度较高.
图 3(Fig. 3)
 |
| 图 3 各样地中检测到的ARGs相对丰度(a)和作用机制(b) Fig. 3The relative abundance of ARGs (a) and the mechanism of resistance (b) for the ARGs were detected in sample sites |
3.3.1 四环素类抗生素抗性基因的丰度表 2中6种四环素类抗生素抗性基因(tetC、tetK、tetX、tetM、tetQ和tetT)在7N与其它样地之间有显著差异, 而其它7种基因在各样地之间无显著差异.tetA基因是四环素中检测到丰度最高的基因, tetS和tetC是丰度相对较高的两个基因.
表 2(Table 2)
| 表 2 各样地沉积物中四环素类抗生素抗性基因的丰度 Table 2 The abundance of tetracycline ARGs in sediments in sample sites | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 2 各样地沉积物中四环素类抗生素抗性基因的丰度 Table 2 The abundance of tetracycline ARGs in sediments in sample sites
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3.3.2 磺胺类抗生素抗性基因的丰度如图 4所示, 3种磺胺类ARGs都被检测到, 其中, sulⅠ和sulⅡ基因的检出率都为100%, 而sulⅢ的检出率为73.33%. sulⅠ基因丰度在7N样地最高, 为6.76×108 copies·g-1;sulⅡ基因在3N和3S样地与其他样地之间有显著差异, 但其丰度比磺胺类其他两个ARGs的丰度低;sulⅢ基因的丰度在7N样地比其他样地明显偏高, 且与其他样地之间有显著差异.
图 4(Fig. 4)
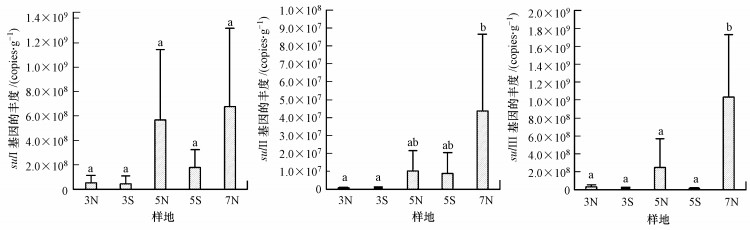 |
| 图 4 各样地沉积物中磺胺类抗生素抗性基因的丰度 Fig. 4The abundance of sulfonamide ARGs in sediments in sample sites |
3.3.3 氨基糖苷类抗生素抗性基因的丰度氨基糖苷类ARGs中aadA1基因丰度在7N样地最高, 为1.37×109 copies·g-1, 且与其它样地之间有显著差异;aacC、strB和str基因在7N与其它样地之间也有显著差异;aac基因在5N样地最高, 为3.53×106 copies·g-1, 与3N样地之间无差异, 而与3S、5S和7N样地之间有显著差异;aadD在5S和7N样地均未检出, aph基因只在5N样地有检出, 且丰度较低.
表 3(Table 3)
| 表 3 各样地沉积物氨基糖苷类抗生素抗性基因的丰度 Table 3 The abundance of aminoglycoside ARGs in sediments in sample sites | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 3 各样地沉积物氨基糖苷类抗生素抗性基因的丰度 Table 3 The abundance of aminoglycoside ARGs in sediments in sample sites
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||
3.3.4 大环内酯类抗生素抗性基因丰度大环内酯类ARGs中ereA基因丰度在5N样地最高, 为1.65×109 copies·g-1, 与3S和5S样地之间无显著差异, 但检出率较低, 为40%;而ereB和mphA基因丰度较低, 检出率分别为66.67%和93.33%, mphA基因在7N样地与其它样地之间有显著差异(p < 0.05).
图 5(Fig. 5)
 |
| 图 5 各样地中大环内酯类抗生素抗性基因的丰度 Fig. 5The abundance of macrolide ARGs in sediments in sample sites |
3.3.5 其它类抗生素抗性基因的丰度β-内酰胺类ARGs中blaampC丰度较其它类ARGs的低;而在7N样地blaSHV基因丰度显著高于其它样地(p < 0.05), 约为3S和5N样地的100倍, 而在其它样地丰度较低, 或未检出.喹诺酮类抗生素抗性基因的丰度和多样性都较低, 其中, qnrA和qnrB基因未检测到;qnrS基因检出率较低, 为53.3%, 其丰度在7N样地与5N样之间有显著差异(p < 0.05), 而与5S样地之间无显著差异.
表 4(Table 4)
| 表 4 各样地沉积物中其它类抗生素抗性基因的丰度 Table 4 The abundance of other ARGs in sediments in sample sites | ||||||||||||||||||||||||||||||||
表 4 各样地沉积物中其它类抗生素抗性基因的丰度 Table 4 The abundance of other ARGs in sediments in sample sites
| ||||||||||||||||||||||||||||||||
3.4 沉积物中理化性质、MGEs和ARGs的相关关系沉积物中ARGs与各理化性质和MGEs之间的相关关系如表 5所示.intⅠ1和tnpA基因丰度分别与四环素类ARGs中tetQ和tetB/P基因丰度之间有显著相关性;而β-内酰胺类ARGs中blaampC丰度与细菌丰度和氮含量之间显著相关(p < 0.01), blaSHV基因的丰度与碳含量之间呈显著相关(p < 0.01), 而氨基糖苷类ARGs中aadD丰度与硫含量呈显著相关(p < 0.01).氮含量与sulⅢ、ereB和blaampC基因丰度有相关性, 与细菌丰度显著正相关(p < 0.01);硫与四环素类tetG和tetK, 以及intⅠ1基因丰度有显著负相关, pH与四环素类ARGs中tetX、tetM和tetO基因丰度之间呈显著负相关.
表 5(Table 5)
| 表 5 各样地沉积物ARGs、理化性质和MGEs之间的相关关系 Table 5 The correlations among the physicochemical characteristics, MGEs, ARGs of sediments in sample sites | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 5 各样地沉积物ARGs、理化性质和MGEs之间的相关关系 Table 5 The correlations among the physicochemical characteristics, MGEs, ARGs of sediments in sample sites
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
4 讨论(Discussion)从ARGs的定量分析和分布研究得出, 16S rRNA基因在7N样地的丰度最高, 与其它样地之间有显著差异.研究表明, 微生物的量(如细菌的含量)与土壤的总氮含量之间显著正相关, 即氮含量越高, 细菌丰度越高(赵溪等, 2010).而本研究实验结果也印证了这一说法, 氮含量同样在7N样地显著高于其它样地, 且氮含量与16S rRNA基因丰度呈显著正相关(p < 0.01, 表 5).这可能与河套灌区排水渠的农业活动有关, 如农田施氮肥可能会对排水渠中沉积物的总氮含量有影响.
本研究中磺胺类ARGs的sulⅠ、sulⅡ、sulⅢ基因均被检测到, 其中, sulⅠ基因的丰度较高(图 4).Stoll等(2012)对德国的莱茵河和澳大利亚的布里斯班河的研究发现, sulⅠ基因比sulⅡ基因检测到的丰度更高, 且都高于四环素类ARGs丰度, 这也与黄浦江、长江流域的研究结果一致(沈群辉等, 2012; 江月等, 2018), 但与海河和东南沿海地区的研究结果不一致(张宏杰, 2011; 陈超琼, 2017).本研究中sulⅠ和sulⅡ基因丰度比黄埔江的磺胺类ARGs丰度低(沈群辉等, 2012), 与长江流域的丰度差别不大.图 4结果与美国科罗拉多州北部河流研究结果相似, sulⅠ基因相对丰度均在0~10-3范围内, sulⅡ基因相对丰度均在0~10-4范围内(Pruden et al., 2006).许多研究结果都表明, 整合酶基因与ARGs(如sulⅠ基因)之间有相关关系;对海河沉积物的研究发现, intⅠ1基因与sulⅠ基因相对丰度之间有显著相关性, 并且intⅠ1参与了环境中sulⅠ的传播(Luo et al., 2011).而表 5中磺胺类ARGs与intⅠ1丰度之间无显著相关, 而四环素类ARGs tetQ与intⅠ1基因丰度显著正相关(p < 0.05), 这与Yang等(2017a; 2017b)的研究结果类似, 该研究发现intⅠ1基因与水体和沉积物中的几种四环素类抗性基因(tetA、tetB、tetO、tetG)显著相关;并且表 5结果显示, tnpA基因与tetB/P基因丰度之间显著正相关, 这些可能是四环素类ARGs在环境中普遍存在的原因.
Xu等(2016)对北京地区城市河流温榆河等水体和沉积物进行研究, 发现沉积物中的tetA基因丰度比其它四环素类ARGs高, 图 2中tetA基因丰度在各样地中较其它四环素类ARGs高;但与Chen等(2013)在广东珠江区域的研究结果有区别, 在其样品中未检测到tetA基因.tetA、tetC、tetG和tetS基因丰度比其它四环素类ARGs的丰度较高, 可能是因为它们由可迁移的载体如转座子和较小的质粒携带, 移动性较强, 可以在细菌之间传播;王凤花等的研究表明, 编码外排泵的基因具有更高的丰度(Wang et al., 2015), tetA、tetC和tetG基因的分子机制都为泵外排机制(图 3), 与本研究结果相似.而四环素类其它基因存在于不可移动的大的共轭质粒或染色体中, 所以这些基因转移和传播的可能性较低(Schwartz et al., 2003).表 5中pH与tetX、tetM和tetO基因丰度之间显著负相关, 而本研究中的pH较高, 因此, tetX、tetM和tetO基因丰度较低.
氨基糖苷类抗性基因在7N样地有显著差异, 且其检出率也较高, 但与长江下游相比, 其污染水平较低(江月等, 2018), 只有aadA1基因在个别样点比较高.这可能与氨基糖苷类抗生素的广泛使用有关, 有研究显示, 氨基糖苷类抗生素是临床使用量最多的五大抗生素之一, 并且广泛应用于农业和养殖业(张红等, 2018);相对于其他类基因, 氨基糖苷类ARGs更容易被富集(Zhu et al., 2013).与三峡库区沉积物中喹诺酮类ARGs的丰度和检出率相比, 本研究结果中qnrS基因的检出率较低, 且qnrA和qnrB基因未检出, 说明河套灌区的喹诺酮类ARGs的污染情况没有三峡水库地区严重(徐晨, 2017).大环内酯类抗性基因ereA基因在5N样地中检测到的丰度最高, 并且是全部基因中相对丰度最高的, 而在其它研究中没有相近的结果(Rahube et al., 2016).有研究表明, 大环内酯类抗生素容易被水解并且常吸附在沉积物中(Luo et al., 2011).在5N样地中, 除了ereA基因, 其它基因的相对丰度都较低, 但ARGs的多样性在5N样地中是最高的, 很多研究都表明人类活动是ARGs污染和传播的重要原因(Chen et al., 2013; Purden et al., 2012).出现上述现象可能的原因是5N样地所在的五排干是巴彦淖尔市最大的人口活动区临河区的主要排水渠, 经过生活污水废水、工农业排污之后, 随着水体流动从南到北富集在排水渠沉积物中.
β-内酰胺类抗生素具有低毒性的特点, 在人类医疗和养殖业中被广泛使用, 人类经常使用的抗生素药物如青霉素、头孢菌素等都为这一类(Stoll et al., 2012).因此, 该类ARGs的检出可能是由于医疗废水、养殖废水排放进入环境所导致.β-内酰胺类ARGs在本研究中检出率较低, 且基因丰度也较低, 与对水体的研究结果不一致(江月等, 2018);而在黄浦江流域的研究中β-内酰胺类ARGs的丰度与本研究结果较接近(Jiang et al., 2013), 可能的原因是β-内酰胺类抗生素在水体中比较容易被水解, 并且在碱性条件下更容易水解(贾励瑛, 2012), 所以沉积物相比水体中检测出的丰度较低.并且β-内酰胺类ARGs可能位于松弛型质粒上, 相对于位于紧密型质粒上的ARGs更容易丢失.Li等(2017)的研究也证实了这一观点, 抗头孢匹胺的菌株半衰期最短, 对应的blaSHV基因丢失速率最快.
5 结论(Conclusions)1) 河套灌区排水渠沉积物中普遍存在四环素类、磺胺类、氨基糖苷类、大环内酯类、β-内酰胺类ARGs的污染.其中, 四环素类和磺胺类ARGs的检出率和丰度都较高, 氨基糖苷类和大环内酯类ARGs中aadA1和ereA的丰度也较高.但是, 河套灌区沉积物中ARGs的整体水平较其它地区的低或与之持平, 即该地区的ARGs的污染水平处于中下等水平.
2) 沉积物中intⅠ1基因和tnpA基因的检出率和丰度也较高, 它与四环素类ARGs之间有显著正相关关系, 可以促进ARGs的水平转移.
3) 沉积物的理化性质中, 氮和pH在各样地之间有显著差异, 且与细菌丰度和ARGs丰度有关.
参考文献
| 陈超琼.2017.抗生素在中国东南沿海沉积物中的分布初探[D].上海: 华东师范大学http://cdmd.cnki.com.cn/Article/CDMD-10269-1017062631.htm |
| Chen B W, Liang X M, Huang X P, et al. 2013. Differentiating anthropogenic impacts on ARGs in the Pearl River Estuary by using suitable gene indicators[J]. Water Research, 47(8): 2811–2820.DOI:10.1016/j.watres.2013.02.042 |
| Crecchio C, Ruggiero P, Curci M, et al. 2005. Binding of DNA from Bacillus subtilis on montmorillonite-humic acids-aluminum or iron hydroxypolymers:Effects on transformation and protection against DNase[J]. Soil Science Society of America Journal, 69(3): 834–841.DOI:10.2136/sssaj2004.0166 |
| 冯兆忠, 王效科, 冯宗炜, 等. 2003. 河套灌区秋浇对不同类型农田土壤氮素淋失的影响[J]. 生态学报, 2003, 23(10): 2027–2032.DOI:10.3321/j.issn:1000-0933.2003.10.010 |
| Hill K E, Top E M. 1998. Gene transfer in soil systems using microcosms[J]. FEMS Microbiology Ecology, 25(4): 319–329.DOI:10.1111/fem.1998.25.issue-4 |
| 贾励瑛.2014.β-内酰胺类抗生素在污水处理厂的分布与迁移转化[D].邯郸: 河北工程大学http://cdmd.cnki.com.cn/Article/CDMD-10076-1013126366.htm |
| 江月, 陈奕涵, 何义亮. 2018. 长江下游某水源型水库抗生素抗性基因污染研究[J]. 环境科学学报, 2018, 38(3): 884–891. |
| Jiang L, Hu X, Xu T, et al. 2013. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 458: 267–272. |
| Jiang L, Hu X L, Xu T, et al. 2013. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 4(38): 267–272. |
| Li C, Lu J J, Liu J, et al. 2016. Exploring the correlations between antibiotics and antibiotic resistance genes in the wastewater treatment plants of hospitals in Xinjiang, China[J]. Environmental Science and Pollution Research, 23(15): 15111–15121.DOI:10.1007/s11356-016-6688-z |
| Li J J, Xin Z H, Zhang Y Z, et al. 2017. Long-term manure application increased the levels of antibiotics and antibiotic resistance genes in a greenhouse soil[J]. Applied Soil Ecology, 121: 193–200.DOI:10.1016/j.apsoil.2017.10.007 |
| Luan X B, Wu P T, Sun S K, et al. 2018. Impact of land use change on hydrologic processes in a large plain irrigation district[J]. Water Resources Management, 32(9): 3203–3217.DOI:10.1007/s11269-018-1986-5 |
| 罗义, 周启星. 2008. 抗生素抗性基因(ARGs):一种新型环境污染物[J]. 环境科学学报, 2008, 28(8): 1499–1505.DOI:10.3321/j.issn:0253-2468.2008.08.002 |
| Luo Y, Mao D Q, Rysz M, et al. 2010. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science and Technology, 44(19): 7220–7225.DOI:10.1021/es100233w |
| Luo Y, Xu L, Rysz M, et al. 2011. Occurrence and transport of tetracycline, sulfonamide, quinolone, and macrolide antibiotics in the Haihe River Basin, China[J]. Environmental Science and Technology, 45(5): 1827–1833.DOI:10.1021/es104009s |
| Marti E, Variatza E, Balcazar J L. 2014. The role of aquatic ecosystems as reservoirs of antibiotic resistance[J]. Trends in Microbiology, 22(1): 36–41. |
| Martinez J L. 2009. Environmental pollution by antibiotics and by antibiotic resistance determinants[J]. Environmental Pollution, 157(11): 2893–2902.DOI:10.1016/j.envpol.2009.05.051 |
| Martínez J L. 2008. Antibiotics and antibiotic resistance genes in natural environments[J]. Science, 321(5887): 365–367.DOI:10.1126/science.1159483 |
| Ouyang W Y, Huang F Y, Zhao Y, et al. 2015. Increased levels of antibiotic resistance in urban stream of Jiulongjiang River, China[J]. Applied Microbiology and Biotechnology, 99(13): 5697–5707.DOI:10.1007/s00253-015-6416-5 |
| Pruden A, Arabi M, Storteboom H N. 2012. Correlation between upstream human activities and riverine antibiotic resistance genes[J]. Environmental Science and Technology, 46(21): 11541–11549.DOI:10.1021/es302657r |
| Pruden A, Pei R, Storteboom H, et al. 2006. Antibiotic resistance genes as emerging contaminants:Studies in Northern Colorado?[J]. Environment Science and Technology, 40(23): 7445–7450.DOI:10.1021/es060413l |
| Rahube T O, Marti R, Scott A, et al. 2016. Persistence of antibiotic resistance and plasmid-associated genes in soil following application of sewage sludge and abundance on vegetables at harvest[J]. Canadian Journal of Microbiology, 62(7): 600–607.DOI:10.1139/cjm-2016-0034 |
| 沈群辉, 冀秀玲, 傅淑珺, 等. 2012. 黄浦江水域抗生素及抗性基因污染初步研究[J]. 生态环境学报, 2012, 21(10): 1717–1723.DOI:10.3969/j.issn.1674-5906.2012.10.015 |
| Schwartz T, Kohnen W, Jansen B, et al. 2003. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms[J]. FEMS Microbiology Ecology, 43(3): 325–335.DOI:10.1111/fem.2003.43.issue-3 |
| Stoll C, Sidhu J P S, Tiehm A, et al. 2012. Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia[J]. Environmental Science and Technology, 46(17): 9716–9726.DOI:10.1021/es302020s |
| Szekeres E, Chiriac C M, Baricz A, et al. 2018. Investigating antibiotics, antibiotic resistance genes, and microbial contaminants in groundwater in relation to the proximity of urban areas[J]. Environmental Pollution, 236: 734–744.DOI:10.1016/j.envpol.2018.01.107 |
| Wang F H, Qiao M, Chen Z, et al. 2015. Antibiotic resistance genes in manure-amended soil and vegetables at harvest[J]. Journal of Hazardous Materials, 299: 215–221.DOI:10.1016/j.jhazmat.2015.05.028 |
| Wang F H, Qiao M, Lv Z E, et al. 2014. Impact of reclaimed water irrigation on antibiotic resistance in public parks, Beijing, China[J]. Environmental Pollution, 184: 247–253.DOI:10.1016/j.envpol.2013.08.038 |
| 徐晨.2017.三峡库区水体、土壤和沉积物中抗生素与抗生素抗性基因的污染特征[D].武汉: 中国科学院武汉植物园http://cdmd.cnki.com.cn/Article/CDMD-80108-1017056075.htm |
| Xu Y, Guo C S, Luo Y, et al. 2016. Occurrence and distribution of antibiotics, antibiotic resistance genes in the urban rivers in Beijing, China[J]. Environmental Pollution, 213: 833–840.DOI:10.1016/j.envpol.2016.03.054 |
| Yang Y, Xu C, Cao X, et al. 2017a. Antibiotic resistance genes in surface water of eutrophic urban lakes are related to heavy metals, antibiotics, lake morphology and anthropic impact[J]. Ecotoxicology, 26(6): 831–840.DOI:10.1007/s10646-017-1814-3 |
| Yang Y Y, Liu W Z, Xu C, et al. 2017b. Antibiotic resistance genes in lakes from middle and lower reaches of the Yangtze River, China:effect of land use and sediment characteristics[J]. Chemosphere, 178: 19–25.DOI:10.1016/j.chemosphere.2017.03.041 |
| 张红, 程寒飞. 2018. 水环境中氨基糖苷类抗性基因污染及研究进展[J]. 环境科学与技术, 2018, 41(10): 1–10. |
| 张宏杰, 2011.海河流域中抗生素抗性基因分布及有关抗性基因的进化分析[D].沈阳: 沈阳药科大学http://cdmd.cnki.com.cn/Article/CDMD-10163-2010183911.htm |
| 赵溪, 李君剑, 李洪建. 2010. 关帝山不同植被恢复类型对土壤碳、氮含量及微生物数量的影响[J]. 生态学杂志, 2010, 29(11): 2102–2110. |
| Zhou L, Ying G, Liu S, et al. 2013. Excretion masses and environmental occurrence of antibiotics in typical swine and dairy cattle farms in China[J]. Science of the Total Environment, 444: 183–195.DOI:10.1016/j.scitotenv.2012.11.087 |
| Zhu Y G, Johnson T A, Su J Q, et al. 2013. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 110(9): 3435–3440.DOI:10.1073/pnas.1222743110 |
| Zhu Y G, Johnson T A, Su J Q., et al. 2013. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. PNAS, 110(9): 3435–3440.DOI:10.1073/pnas.1222743110 |
