A genetic sub-structure study of the Tibetan population in Southwest China
Xiaojuan Wang1,2, Enfang Qian1, Yue Li2, Zhengyang Song1,2, Hui Zhao2, Hexin Xie2, Caixia Li1,2, Jiang Huang1, Li Jiang2通讯作者: 黄江,博士,教授,研究方向:法医物证学。E-mail:mmm_hj@gmc.edu.cn江丽,博士,副主任法医师,研究方向:法医遗传学。E-mail:jl@mail.bnu.edu.cn
编委: 谢小冬
收稿日期:2019-12-26修回日期:2020-04-14网络出版日期:2020-06-20
| 基金资助: |
Editorial board:
Received:2019-12-26Revised:2020-04-14Online:2020-06-20
| Fund supported: |
作者简介 About authors
王小娟,硕士研究生,专业方向:法医物证学。E-mail:757164796@qq.com。

摘要
藏族为中国西南地区典型的少数民族,分为卫藏、康巴、安多和嘉绒等多个支系。然而,对藏族支系人群的遗传结构,特别是各分支人群的父系、母系遗传结构却缺乏深度解析。本研究基于个体水平的常染色体、父系来源的Y染色体和母系来源的线粒体3个类别遗传信息,对西藏地区卫藏藏族、四川甘孜地区康巴藏族、青海地区安多藏族和四川阿坝地区嘉绒藏族共4个藏族群体进行研究,以揭示其遗传亚结构关系。采用微测序技术检测各位点分型,利用PowerPlex ?Y23和DNATyper TM Y26试剂盒检测Y-STRs基因座分型,通过热图和主成分分析、祖先成分分析、单倍群频率统计、网络图及多维尺度分析等探讨其遗传亚结构。结果表明,常染色体和Y染色体遗传标记可将4个藏族人群分为3类:青藏高原的卫藏藏族为一类,高原周边地区的康巴藏族和安多藏族的遗传结构类似分为一类,“藏彝走廊”中嘉绒藏族的遗传结构与其他藏族人群差异显著而为一类。不同藏族分支人群在线粒体遗传信息方面无明显差异性。上述多类别遗传信息的分析结果为深入了解藏族不同分支人群的遗传亚结构提供了新视角。
关键词:
Abstract
Tibetan is a typical ethnic minority population in Southwest China, which can be divided into U-Tsang, Kham, Amdo, Jiarong and other sub-populations. However, the genetic structure of these sub-populations has not been comprehensively analyzed, especially from the perspective of paternal and maternal lineages. Based on genetic markers of autosomes, the Y chromosome and mitochondria, we studied four Tibetan populations (the U-Tsang population in Tibet Autonomous Region; the Kham population in Garze, Sichuan province; the Amdo population in Qinghai province and the Jiarong population in Aba, Sichuan province) to interpret their genetic structure. The mini-sequencing technology was used to detect the genotype of each maker. Meanwhile, the PowerPlex ?Y23 and DNA Typer TM Y26 kit were applied to genotype Y-STRs. Subsequently, the genetic structure was analyzed by heatmap and principal component analysis, ancestry component, haplogroup frequency, network map and multi-dimensional scaling analysis. The results showed that the four Tibetan populations could be divided into three sets based on the autosomal and Y-chromosomal genetic markers, in which set 1 was the U-Tsang population in the Tibetan Plateau, set 2 comprised of the Kham and Amdo populations in the surrounding areas of the plateau, and set 3 was the Jiarong population that resided in the Tibetan and Yi Corridor. No significant difference was observed in mitochondrial genetic markers among four Tibetan populations. In general, multi-category genetic information provides a new comprehensive insight into the Tibetan sub-population.
Keywords:
PDF (1031KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
王小娟, 钱恩芳, 李悦, 宋正阳, 赵慧, 谢何鑫, 李彩霞, 黄江, 江丽. 中国西南地区藏族人群遗传亚结构研究. 遗传[J], 2020, 42(6): 565-576 doi:10.16288/j.yczz.19-330
Xiaojuan Wang.
藏族是中国古老的民族之一,人数达628.2万(2010年全国第6次人口普查数据),其中43.2%分布于西藏自治区,其余主要分布于青海和四川等地(
藏族人群长期居住于海拔3000~5000 m的青藏高原,经长期自然选择,表现出对高原低氧环境较强的适应能力[3,4]。研究表明,藏族人群的高原适应(high-altitude adaptation, HAA)具有明显的遗传学基础。全基因组关联研究发现,在缺氧诱导因子(hypoxia-inducible factor, HIF)通路上存在EGLN1、EPAS1等多个候选基因与藏族人群的HAA相关[5,6]。候选基因上多个单核苷酸多态性(single nucleotide polymorphisms, SNPs)位点的等位基因频率分布在藏族人群与平原人群间具有显著差异,如EPAS1基因上5个SNPs位点组成的单倍型AGGAA (rs115321619、rs73926263、rs73926264、rs73926265和rs55981512)高频分布于藏族人和丹尼索瓦人[7,8];EGLN1基因上也检测出rs18699510和rs12097901等多个分布差异位点[9]。除常染色体外,Y染色体的非重组区域以单倍型形式呈父系遗传,有效群体小,对漂变敏感,包含人群更多的父系历史信息[10]。Y-SNPs单倍群在世界人群的分布呈现明显的地理特异性,其中单倍群D-M174主要分布于中国西藏、日本和安达曼群岛[11,12,13]。与Y染色体类似,线粒体DNA呈母系遗传,是反映人群母系遗传信息的遗 传标记,其单倍群的支系分布同样具有地域差异(http://www.phytree.org/tree/index.htm)。目前也有研究结合父系和母系遗传信息探索藏族人群的遗传结构及源流[14]。但是,不同地理位置的藏族分支人群之间的遗传结构特点有待进一步探究。
本研究基于常染色体、Y染色体及线粒体多类别基因组遗传标记,对西藏地区卫藏藏族、四川甘孜地区康巴藏族、青海地区安多藏族和四川阿坝地区嘉绒藏族等4个藏族人群共297份男性样本进行检测,分析其遗传结构,探讨西南地区不同藏族分支人群的遗传亚结构。
1 材料与方法
1.1 样本信息
采集中国西南地区297份男性藏族无关个体静脉血样本,其中西藏地区卫藏藏族89例、四川甘孜地区康巴藏族88例、青海地区安多藏族82例、四川阿坝地区嘉绒藏族38例。样本信息及人群简称见表1。本研究已通过公安部物证鉴定中心伦理委员会审查,上述样本供者均签署知情同意书。根据试剂盒说明书,采用QIAamp® DNA Blood Midi Kit (德国QIAGEN公司)进行DNA提取。使用NanoDrop 2000c (美国Thermo Fisher Scientific公司)进行DNA定量并稀释至5 ng/μL待检。1.2 位点筛选
基于多类别基因组遗传信息,共选择134个位点,位点信息见附表1。其中包括:(1)通过文献调研[15]选择藏族特异性常染色体位点25个;(2)依据Y染色体谱系树2018版(Table 1
表1
表14个藏族人群的样本信息
Table 1
| 序号 | 群体 | 所属支系 | 英文全称 | 英文简称 | 检测样本量(例) | 海拔高度(m) |
|---|---|---|---|---|---|---|
| 1 | 西藏藏族 | 卫藏 | Chinese Tibetan in Tibetan Plateau | CTT | 89 | 3650 |
| 2 | 四川甘孜藏族 | 康巴 | Chinese Tibetan in Garze Sichuan, China | CTG | 88 | 3300 |
| 3 | 青海藏族 | 安多 | Chinese Tibetan in Qinghai, China | CTQ | 82 | 2420 |
| 4 | 四川阿坝藏族 | 嘉绒 | Chinese Tibetan in Aba Sichuan, China | CTA | 38 | 2130 |
新窗口打开|下载CSV
1.3 分型检测
1.3.1 筛选位点分型检测使用Primer Premier 5.0软件设计PCR扩增及延伸引物并由生工生物工程(上海)股份有限公司合成。利用Applied BiosystemsTM SNaPshotTM Multiplex试剂盒(美国Thermo Fisher Scientific公司)进行位点检测。应用Mastercycler ProS热循环仪(德国Eppendorf公司)进行复合扩增,扩增产物使用ABI 3130xl遗传分析仪(美国Thermo Fisher Scientific公司)进行毛细管电泳检测,利用GeneMapper ID v3.2软件分析样本分型。
1.3.2 Y-STRs分型检测
选取16个单拷贝Y-STRs基因座(DYS19、DYS389I、DYS389II、DYS390、DYS391、DYS392、DYS393、DYS437、DYS438、DYS439、DYS448、DYS456、DYS458、DYS533、DYS635和GATA-H4)和1个多拷贝基因座DYS385ab,使用试剂盒检测西藏藏族(CTT)、四川甘孜藏族(CTG)、青海藏族(CTQ)和四川阿坝藏族(CTA)4个人群的分型数据,其中西藏藏族(CTT)、四川甘孜藏族(CTG)和青海藏族(CTQ)人群样本使用PowerPlex® Y23荧光标记复合扩增系统(美国Promega公司)试剂盒检测;四川阿坝藏族(CTA)人群样本使用DNATyperTM Y26试剂盒(中国公安部物证鉴定中心)检测。扩增产物用ABI 3500xl遗传分析仪(美国Thermo Fisher Scientific公司)进行毛细管电泳检测并使用GeneMapper® ID-X软件进行Y-STRs分型分析。
1.4 数据分析
1.4.1 常染色体遗传标记采用Genepop v4.2软件(
1.4.2 Y染色体SNPs及STRs遗传标记
基于国际Y染色体协会(Y chromosome consortium, YCC)的单倍群命名原则[17],根据Y-SNPs位点的分型情况划分样本所属单倍群,采用直接计数法统计各单倍群频率。基于16个单拷贝Y-STRs基因座分型使用公式D= [n(1-ΣPi2)]/(n-1)计算每个藏族分支人群的基因/单倍型多样性,其中Pi为第i个等位基因/单倍型频率,n为样本数。对Y-SNPs单倍群相同的样本,基于16个单拷贝Y-STRs基因座分型使用Network v5.0软件进行网络图分析[18]。根据Y-STRs基因座的基因多样性和突变率综合评估设定权重值,基因多样性和突变率越高,权重值越低。其中Y-STRs基因座突变率参考YHRD数据库(https://yhrd.org/pages/resources/mutation_rates)。为评估藏族分支人群的遗传关系,比较了其与6个邻近人群(四川成都/泸州汉族[19,20,21]、四川彝族[22]、甘肃藏族(YA004043)、昌都康巴藏族[23]、青海回族[24,25]、青海撒拉族(YA003302))和南方汉族(广西汉族(YA004218))、北方汉族(北京汉族[26,27,28])的17个Y-filer基因座组成的单倍型。所有参考人群的分型数据来自YHRD数据库。利用YHRD数据库在线软件AMOVA & MDS工具计算群体间遗传距离Rst矩阵,并构建多维尺度(MDS)散点图进行可视化。
1.3.3 线粒体SNPs遗传标记
根据PhyloTree 17版谱系树,结合各样本在mt-SNPs位点的分型判断其单倍群类型,采用直接计数法统计各单倍群频率。
2 结果与分析
2.1 个体水平遗传结构
2.1.1 群体聚类分析25个位点在4个藏族人群的等位基因频率分布热图如图1A所示。基于突变型等位基因频率可将4个藏族人群分为3类,除rs10206434、rs375554942、rs76347095、rs6544888和rs1562453位点外,西藏藏族(CTT)在其他各位点的突变型等位基因频率最高,四川甘孜藏族(CTG)和青海藏族(CTQ)的突变型等位基因频率类似且次于西藏藏族(CTT),四川阿坝藏族(CTA)的突变型等位基因频率最低,与其他藏族分支人群具有较大差异性。根据各位点在人群的等位基因频率进行主成分分析,结果见图1B。PC1和PC2成分的累积贡献率为100%,在PC1维度(99.7%)上,4个藏族人群可分为3类且彼此距离较远而区分开:西藏藏族(CTT);四川甘孜藏族(CTG)和青海藏族(CTQ);四川阿坝藏族(CTA)。
图1
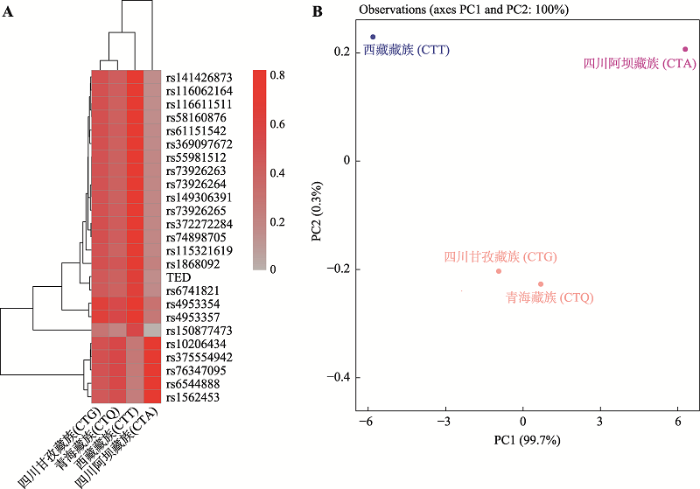 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图14个藏族人群聚类分析
A:基于25个常染色体位点等位基因频率的4个藏族人群热图分析。颜色深浅表示各位点在人群中的突变型等位基因频率大小,树状分枝表示人群和位点的聚类关系。B:基于25个常染色体位点等位基因频率的4个藏族人群主成分分析。
Fig. 1Cluster analysis of four Tibetan populations
2.1.2 祖先成分
STRUCTURE分析可直观反映族群间的聚类关系。4个藏族人群STRUCTURE分析在K=2时的结果见图2,黄色和红色分别代表一种祖先成分(以下分别称为成分1和成分2),颜色比例的构成代表祖先成分比例的构成。结果显示,基于祖先成分比例可将4个藏族人群分为3类:(1)西藏藏族(CTT),群体水平上成分1和成分2分别为0.719和0.281,个体水平上56.2%个体成分1大于0.75,34.8%个体介于0.25~0.75之间,9.0%个体小于0.25;(2)四川甘孜藏族(CTG)和青海藏族(CTQ),群体水平上成分1和成分2分别约为0.45和0.55,个体水平上约25.0%个体成分1大于0.75,45.0%个体介于0.25~0.75之间,30.0%个体小于0.25;(3)四川阿坝藏族(CTA),群体水平上成分1和成分2分别为0.175和0.825,个体水平上无成分1大于0.75的个体,36.8%个体介于0.25~0.75之间,63.2%个体小于0.25。
图2
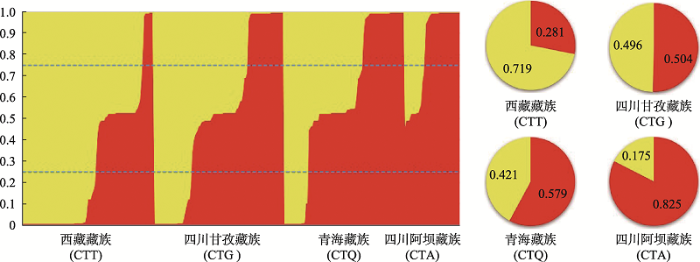 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基于25个常染色体位点分型的4个藏族人群STRUCTURE分析
个体水平祖先成分见柱状图,其中每个柱形代表一个个体,每种颜色代表一种祖先成分,颜色比例代表该个体祖先成分比例;群体水平祖先成分见饼图,其中每种颜色代表的祖先成分同柱状图,颜色比例代表该群体祖先成分比例。K=2,Ln=-5201.9。
Fig. 2STRUCTURE analysis of four Tibetan populations based on the genotype of 25 autosomal markers
2.2 父系遗传结构
2.2.1 Y-SNPs单倍群在藏族人群中的分布藏族人群Y染色体单倍群类型及分布频率见表2。各单倍群频率在藏族人群之间存在显著差异,其中以单倍群D-M174 (34.21%~71.91%)和O-M175 (16.85%~60.53%)为主要类型。在下游分支单倍群中,以单倍群D1a2-P99 (44.44%)和O2-M122 (22.90%)为主。基于单倍群D-M174类型,可将4个藏族人
群分为3类:(1)西藏藏族(CTT),71.91%个体属于单倍群D-M174,下游分支以D1a2-P99为主,分布频率为53.93%;(2)四川甘孜藏族(CTG)和青海藏族(CTQ),约60%个体属于单倍群D-M174,下游分支以D1a2-P99为主,分布频率约为47%;(3)四川阿坝藏族(CTA),34.21%个体属于单倍群D-M174,下游分支以D1a1-M15为主,分布频率为26.32%。
Table 2
表2
表2藏族人群Y-SNPs单倍群谱系树及频率分布
Table 2
| 单倍群谱系树 | 西藏藏族 (CTT) | 四川甘孜藏族 (CTG) | 青海藏族 (CTQ) | 四川阿坝藏族 (CTA) | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| n | % | n | % | n | % | n | % | ||||||||||||||
| M168 | CT | 89 | 100 | 88 | 100 | 82 | 100 | 38 | 100 | ||||||||||||
| M130 | C | 2 | 2.25 | 2 | 2.27 | 2 | 2.44 | 1 | 2.63 | ||||||||||||
| M174 | D | 64 | 71.91 | 55 | 62.50 | 48 | 58.54 | 13 | 34.21 | ||||||||||||
| M15 | D1a1 | 16 | 17.98 | 14 | 15.91 | 8 | 9.76 | 10 | 26.32 | ||||||||||||
| P99 | D1a2 | 48 | 53.93 | 41 | 46.59 | 40 | 48.78 | 3 | 7.89 | ||||||||||||
| M96 | E | 1 | 1.12 | 1 | 1.14 | 1 | 1.22 | ||||||||||||||
| M89 | F | 22 | 24.72 | 30 | 34.09 | 31 | 37.80 | 24 | 63.16 | ||||||||||||
| M201 | G | 1 | 1.22 | ||||||||||||||||||
| M304 | J | 4 | 4.88 | ||||||||||||||||||
| M9 | K | 22 | 24.72 | 30 | 34.09 | 26 | 31.71 | 24 | 63.16 | ||||||||||||
| M231 | N | 5 | 5.62 | 6 | 6.82 | 4 | 4.88 | ||||||||||||||
| M242 | Q | 2 | 2.25 | 1 | 1.22 | ||||||||||||||||
| M207 | R | 1 | 1.14 | 5 | 6.10 | 1 | 2.63 | ||||||||||||||
| M175 | O | 15 | 16.85 | 23 | 26.14 | 16 | 19.51 | 23 | 60.53 | ||||||||||||
| M122 | O2 | 15 | 16.85 | 21 | 23.86 | 12 | 14.63 | 20 | 52.63 | ||||||||||||
| M324 | O2a | 15 | 16.85 | 21 | 23.86 | 11 | 13.41 | 20 | 52.63 | ||||||||||||
| L127.1 | O2a1 | 1 | 1.12 | 3 | 3.41 | 5 | 6.10 | 5 | 13.16 | ||||||||||||
| P201 | O2a2 | 14 | 15.73 | 18 | 20.45 | 6 | 7.32 | 15 | 39.47 | ||||||||||||
| M117 | O2a2b1a1 | 12 | 13.48 | 10 | 11.36 | 4 | 4.88 | 8 | 21.05 | ||||||||||||
新窗口打开|下载CSV
2.2.2 Y-STRs基因座的单倍型多样性和基因多样性分析
16个单拷贝Y-STRs基因座组成的单倍型中,在西藏藏族(CTT,89例)中共检出82种单倍型,单倍型多样性(haplotype diversity, HD)为0.9972;四川甘孜藏族(CTG,88例)中共检出81种单倍型,HD为0.9976;青海藏族(CTQ,82例)中共检出78种单倍型,HD为0.9985;四川阿坝藏族(CTA,38例)中共检出36种单倍型,HD为0.9972。各Y-STRs基因座在4个藏族人群的基因多样性(genetic diversity, GD)如图3所示,GD值在0.2611~0.8350之间。尽管藏族各分支人群间HD无显著差异,但DYS392、DYS389 II、DYS635、DYS448、DYS456、DYS533、DYS393、DYS19和DYS438在西藏藏族(CTT)中的GD值最低,而DYS458、DYS392、DYS439、DYS635、DYS448、DYS456、DYS533、DYS437和DYS391在四川阿坝藏族(CTA)中的GD值最高。
图3
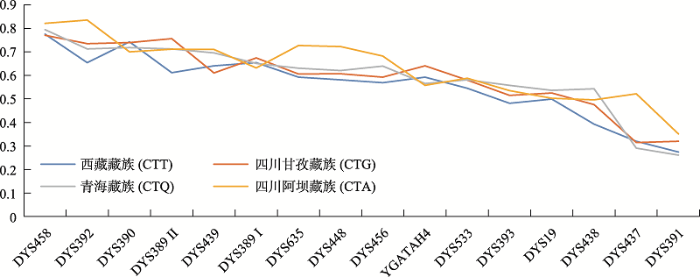 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图316个Y-STRs基因座在4个藏族人群中的基因多样性
Fig. 3Genetic diversity of four Tibetan populations at 16 Y-STRs loci
2.2.3 藏族分支人群遗传关系网络图分析
为进一步观察不同藏族分支人群的遗传关系,选择在4个藏族人群中分布频率较高的3个单倍群D1a1-M15、D1a2-P99和O-M175,根据16个单拷贝Y-STRs基因座组成的单倍型绘制网络图,权重值设置见附表2,网络图如图4所示。结果显示,各单倍群中每一分支人群均未发现明显聚类。西藏藏族(CTT)、四川甘孜藏族(CTG)和青海藏族(CTQ)在单倍群D1a2-P99中的分布较多;四川阿坝藏族(CTA)中单倍群O-M175分布较多。各单倍群均存在不同人群共享Y-STRs单倍型。
图4
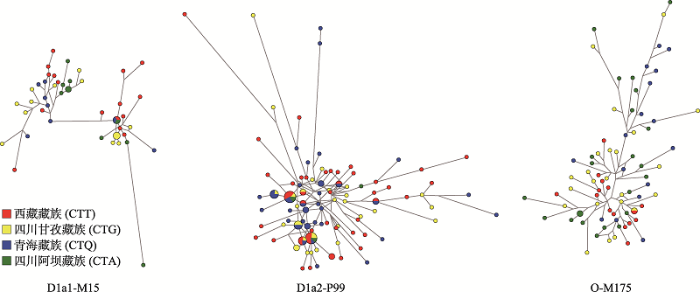 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4基于16个Y-STRs基因座分型的4个藏族人群中D1a1-M15、D1a2-P99和O-M175单倍群网络图分析结果
Fig. 4The network analysis of four Tibetan populations with D1a1-M15, D1a2-P99 and O-M175 haplogroups based on 16 Y-STRs genotypes
2.2.4 藏族分支人群与其他群体间遗传关系
遗传距离是反映群体间遗传差异的重要指标。基于遗传距离的4个藏族人群与其他8个人群的MDS散点图如图5所示。结果表明,西藏藏族(CTT)、四川甘孜藏族(CTG)和青海藏族(CTQ)聚为一簇,与昌都康巴藏族距离较近,与其他群体相距较远;四川阿坝藏族(CTA)与四川彝族距离最近,与其他藏族人群相距较远。
图5
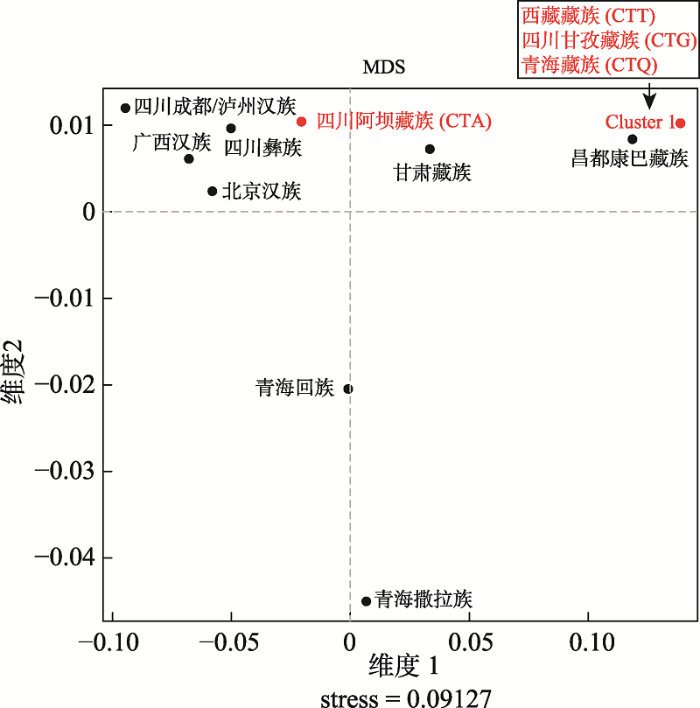 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5基于遗传距离的人群多维尺度分析
红色字体为本研究的4个藏族人群,其他人群来源于YHRD数据库;Cluster 1表示西藏藏族(CTT)、四川甘孜藏族(CTG)和青海藏族(CTQ)因遗传距离近而聚为一簇。
Fig. 5Multi-dimensional scaling (MDS) plot for populations based on Rst values
2.3 母系遗传结构
4个藏族人群线粒体单倍群类型及分布频率见表3,各单倍群的频率分布在藏族人群之间无显著差异性。各藏族人群均检出M、N两支单倍群,其中单倍群M的频率(56.76%~67.05%)高于单倍群N (32.95~43.24%)。单倍群M以M8、M9、G和D单倍群4个下游分支为主,其中单倍群D的分布频率约为25%。单倍群N以A和R两类分支单倍群为主,其中单倍群R的分布频率约为30%。Table 3
表3
表3藏族人群mt-SNPs单倍群谱系树及频率分布
Table 3
单倍群谱系树 | 西藏藏族 (CTT) | 四川甘孜藏族 (CTG) | 青海藏族 (CTQ) | 四川阿坝藏族 (CTA) | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| n | % | n | % | n | % | n | % | ||||||||
| A1018G | L3 | 88 | 100.00 | 87 | 100.00 | 79 | 100.00 | 37 | 100.00 | ||||||
| G10400A | M | 59 | 67.05 | 54 | 62.07 | 48 | 60.76 | 21 | 56.76 | ||||||
| T4715C | M8 | 5 | 5.68 | 9 | 10.34 | 3 | 3.80 | 6 | 16.22 | ||||||
| A13263G | C | 3 | 3.41 | 8 | 9.20 | 2 | 2.53 | 3 | 8.11 | ||||||
| G4491A | M9 | 19 | 21.59 | 13 | 14.94 | 11 | 13.92 | 1 | 2.70 | ||||||
| A3394G | M9a'b | 16 | 18.18 | 9 | 10.34 | 8 | 10.13 | 1 | 2.70 | ||||||
| C7697T | M9a1a1c1b | 11 | 12.50 | 5 | 5.75 | 2 | 2.53 | ||||||||
| G14569A | M12'G | 13 | 14.77 | 10 | 11.49 | 7 | 8.86 | 4 | 10.81 | ||||||
| A4833G | G | 13 | 14.77 | 10 | 11.49 | 7 | 8.86 | 4 | 10.81 | ||||||
| T4561C | M62'68 | 1 | 1.27 | ||||||||||||
| G2735A | M62 | 1 | 1.27 | ||||||||||||
| C4883T | M80'D | 15 | 17.05 | 19 | 21.84 | 24 | 30.38 | 9 | 24.32 | ||||||
| C5178A | D | 15 | 17.05 | 19 | 21.84 | 24 | 30.38 | 9 | 24.32 | ||||||
| C3010T | D4 | 10 | 11.36 | 14 | 16.09 | 16 | 20.25 | 7 | 18.92 | ||||||
| C5262T | D4j1 | 4 | 4.55 | 3 | 3.45 | 2 | 2.53 | ||||||||
| C10873T | N | 29 | 32.95 | 33 | 37.93 | 31 | 39.24 | 16 | 43.24 | ||||||
| A663G | A | 10 | 11.36 | 5 | 5.75 | 10 | 12.66 | 3 | 8.11 | ||||||
| T12705 | R | 19 | 21.59 | 26 | 29.89 | 21 | 26.58 | 12 | 32.43 | ||||||
| C10310T | F | 11 | 12.50 | 14 | 16.09 | 10 | 12.66 | 3 | 8.11 | ||||||
新窗口打开|下载CSV
3 讨论
本研究结合个体水平的常染色体、父系Y染色体和母系线粒体多类别基因组信息,对中国西南地区4个藏族人群的遗传亚结构进行了研究,常染色体位点和Y染色体SNPs遗传结构分析结果表明4个藏族人群可分为3类:海拔最高的西藏藏族(CTT)为一类;海拔次之四川甘孜藏族(CTG)和青海藏族(CTQ)为一类;海拔最低的四川阿坝藏族(CTA)为一类。线粒体SNPs遗传标记角度的分析结果表明不同海拔的藏族人群的遗传结构无显著差异。常染色体遗传标记可从个体水平了解群体遗传结构。藏族人群在长期与环境相适应的进化过程中形成HAA机制,其HAA的生物学基础中,HIF通路作为机体调控氧平衡的主要信号通路,是藏族人群HAA的关键,其分布的HAA相关基因(如EGLN1、EPAS1、PPARA、EDNRA、ATP6V1E2、HMOX2和HBG等)中多个位点在高原藏族和平原人群的频率具有差异性[7,9,29]。使用一组较少的在人群中等位基因频率差异显著的位点可实现人群遗传结构的初步解析。因此,本研究基于文献调研后最终筛选出在藏族人群与平原人群间具有显著差异的25个藏族特异性位点,分别位于2号染色体HIF通路的EPAS1和ATP6V1E2等HAA相关基因。各位点等位基因在不同藏族分支人群间的频率分布也存在地域特异性,与前人的相关研究符合[5,8,30]。同时,各位点的等位基因频率分布差异与海拔呈正相关,同柯金坤[31]的研究结果类似,进一步证明了高海拔环境对HAA相关基因存在适应性选择。当然,藏族人群的HAA机制研究不局限于热点研究的基因,如EPAS1下游的TMEM247基因中rs116983452的突变型等位基因在藏族人群和其他人群中表现出显著的频率差异[32];MKL1基因中rs59098711的等位基因C高频分布于藏族人群[33],此外该基因上163 bp片段缺失和SCUBE2基因中662 bp片段插入也在藏族人群中富集[34]。为增加信息覆盖度,未来可进一步筛选藏族特异性位点对藏族不同分支人群进行研究,探索其遗传亚结构。
Y染色体遗传标记是研究群体父系遗传信息的重要手段。根据Y-SNPs的发生时间可将男性人群分为不同单倍群,同一父系祖先的男性后代享有相同单倍群。东亚人群以O-M175、C-M130、D-M174和N-M231为主要单倍群,其中O-M175为优势单倍群,约75%的中国人群归于该单倍群;D-M174在国内主要分布于藏族人群[35]。本研究的4个藏族人群主要为D-M174和O-M175两种核心单倍群(79.80%),结果与文献[36]报道一致。藏族人群特异性单倍群D-M174及其下游分支在不同藏族分支人群中的分布频率具有差异性。其中D1a2-P99在西 藏藏族(CTT)、四川甘孜藏族(CTG)和青海藏族 (CTQ)的频率明显高于D1a1-M15,印证了前人研究结果[35,36,37]。而四川阿坝藏族(CTA)以下游分支D1a1-M15为主,区别于其他藏族人群。此外,青海藏族(CTQ)中J-M304和R-M207的分布频率显著高于其他藏族人群,该两类单倍群在国内主要分布于回族人群(分别为9.69%和11.85%)[25],提示两人群间存在历史上的基因交流或部分共同父系祖源。Y-STRs突变率较高,可反映人群近期父系基因交流。4个藏族人群的HD均高于0.99,表明藏族人群的Y-STRs遗传多态性较高,其多态性分布的地区差异性较小。在网络图和MDS中,西藏藏族(CTT)、四川甘孜藏族(CTG)和青海藏族(CTQ)紧密聚集,也提示3者的父系遗传差异较小。值得注意的是,四川阿坝藏族(CTA)在MDS中与其他藏族人群相距较远,这与其常染色体遗传结构差异一致,不过也不排除其样本量较少导致的偏差。
线粒体SNPs是解析群体母系遗传结构的理想标记,其单倍群的支系分布具有族群地域差异[38,39,40]。本研究的不同藏族分支人群中以主干单倍群M9a'b、G、D、F的分布频率较高,均为东亚起源支系且在藏、汉群体中均广泛分布[41,42,43,44]。其中高频分布于中国西南地区的单倍群D是4个藏族人群最普遍的单倍群类型(17.05%~30.38%),其次为F。而单倍群M9a'b广泛分布于东亚大陆[45],国内人群中其频率及多样性在西藏藏族人群中达到最高[36,42]。本研究中除四川阿坝藏族(CTA)外,M9a'b在其他藏族人群中均大于10%。单倍群G在4个藏族人群的分布频率约为10%,类似于前人研究的那曲、拉萨、甘孜等地藏族人群的频率分布[36]。然而藏族人群特异单倍群M62仅在青海藏族(CTQ)检出1例,低于文献报道的其在藏族人群中的分布频率(14/680=2.1%)[42],可能为样本采集范围局限和样本量较少所致。
不同藏族分支人群的常染色体位点和Y染色体SNPs遗传信息具有显著差异性,且与海拔具有相关性。从地理位置分析,西南地区汇聚濮系、氐系、羌系及夷系民族群体[46]。西藏藏族(CTT)居住于以藏族为主体民族(90.5%)的高海拔青藏高原地区,逶迤的山脉(如喜马拉雅山脉)和陡峭的峡谷(如雅鲁藏布大峡谷)等复杂多样的地貌形成地理隔离[47],可能阻碍了西藏藏族(CTT)与其他人群的基因交流,导致其遗传结构更具有高原代表性。四川甘孜藏族(CTG)和青海藏族(CTQ)居于青藏高原周边地区,所在的四川甘孜处于青藏高原东南源,包含藏族(78.3%)和汉族(18.2%)等民族以大范围聚居小范围杂居形式分布[48];青海位于青藏高原东北部,容纳汉族(53.0%)、藏族(24.4%)、回族(14.8%)等民族聚居(http://www. stats.gov.cn)。多民族混居的生活环境使得四川甘孜藏族(CTG)和青海藏族(CTQ)的遗传结构与西藏藏族(CTT)的差异显著,提示其与周围民族之间存在基因交流。四川阿坝藏族(CTA)虽同样居于高原周边地区,但其所隶属的嘉绒藏族是夷系民族在唐代吐蕃东进后融合羌人和吐蕃人基因而形成的[2,49],其所在的“藏彝走廊”位于横断山脉-河谷交错地区,是西北、西南各民族流动迁徙的重要场所。特殊的历史民族交流和地理位置,使四川阿坝藏族(CTA)呈现明显混融特征而区别于其他藏族人群。然而,线粒体SNPs遗传信息在藏族分支人群间无明显差异性,从社会学角度分析可能与藏族人群的婚配制度有关。藏族作为中国典型的少数民族之一,是氐羌族群、游牧民族和西藏土著居民融合的民族[50],可与其他民族通婚。在公元6世纪,西藏松赞干布与唐朝文成公主的联姻打开了西部与中原地带的经济文化等方面的交流新格局[51]。此后中原地区王朝对藏区设置行政管理机构使民族融合趋势加强,推进了藏汉通婚的进程[50]。民族间基因交流尤其是母系基因的渗入使藏族人群的母系遗传多态性增加。
综上所述,本研究基于多类别基因组信息探讨了4个不同藏族分支人群的遗传亚结构,结果表明4个藏族人群间具有遗传差异性,获得的数据可为群体遗传学和法医学实践研究提供数据支持。但涉及的样本量少,且对甘肃、云南等地的藏族人群遗传亚结构尚未探索,未来可增加样本量及更多藏族分支人群开展研究,以进一步探索藏族不同分支的遗传相似性和差异性。
附录:
附表详见文章电子版Supplementary Table 1
附表1
附表1位点信息
Supplementary Table 1
 |
新窗口打开|下载CSV
Supplementary Table 2
附表2
附表2网络图分析中各Y-STR基因座的权重值信息
Supplementary Table 2
| 基因座 | 权重 | 突变率 | CTT | CTG | CTQ | CTA |
|---|---|---|---|---|---|---|
| DYS458 | 1 | 0.006 | 0.7750 | 0.7704 | 0.7943 | 0.8208 |
| DYS392 | 1 | 0.001 | 0.6545 | 0.7348 | 0.7124 | 0.8350 |
| DYS390 | 1 | 0.002 | 0.7421 | 0.7396 | 0.7185 | 0.6999 |
| DYS389 II | 2 | 0.004 | 0.6117 | 0.7565 | 0.7127 | 0.7112 |
| DYS439 | 2 | 0.005 | 0.6411 | 0.6108 | 0.6960 | 0.7098 |
| DYS389 I | 2 | 0.003 | 0.6540 | 0.6742 | 0.6501 | 0.6316 |
| DYS635 | 2 | 0.004 | 0.5924 | 0.6063 | 0.6308 | 0.7269 |
| DYS448 | 2 | 0.002 | 0.5812 | 0.6074 | 0.6208 | 0.7226 |
| DYS456 | 2 | 0.004 | 0.5687 | 0.5927 | 0.6399 | 0.6821 |
| GATA-H4 | 3 | 0.003 | 0.5927 | 0.6408 | 0.5652 | 0.5576 |
| DYS533 | 3 | 0.004 | 0.5457 | 0.5798 | 0.5815 | 0.5875 |
| DYS393 | 4 | 0.001 | 0.4814 | 0.5154 | 0.5580 | 0.5349 |
| DYS19 | 4 | 0.002 | 0.4990 | 0.5256 | 0.5363 | 0.5021 |
| DYS438 | 5 | 0.001 | 0.3933 | 0.4762 | 0.5434 | 0.4964 |
| DYS437 | 6 | 0.001 | 0.3202 | 0.3145 | 0.2906 | 0.5220 |
| DYS391 | 6 | 0.002 | 0.2743 | 0.3195 | 0.2611 | 0.3514 |
新窗口打开|下载CSV
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
