 ,福建医科大学基础医学院,医学生物信息学福建省高校重点实验室,消化道恶性肿瘤教育部重点实验室,福州 350122
,福建医科大学基础医学院,医学生物信息学福建省高校重点实验室,消化道恶性肿瘤教育部重点实验室,福州 350122The development of a general drug resistance score model based on MIC50 related gene pairs in colorectal cancer cell lines
Huxing Chen, Lei Xu, Jing Li, Zheng Guo, Lu Ao ,Fujian Key Laboratory of Medical Bioinformatics, Key Laboratory of Ministry of Education for Gastrointestinal Cancer, School of Basic Medical Sciences, Fujian Medical University, Fuzhou 350122, China
,Fujian Key Laboratory of Medical Bioinformatics, Key Laboratory of Ministry of Education for Gastrointestinal Cancer, School of Basic Medical Sciences, Fujian Medical University, Fuzhou 350122, China通讯作者: 敖露,博士,副教授,硕士生导师,研究方向:肿瘤生物信息学。E-mail:lukey@fjmu.edu.cn
编委: 卢大儒
收稿日期:2020-01-6修回日期:2020-04-29网络出版日期:2020-06-20
| 基金资助: |
Editorial board:
Received:2020-01-6Revised:2020-04-29Online:2020-06-20
| Fund supported: |
作者简介 About authors
陈湖星,硕士,助理实验师,研究方向:肿瘤生物信息学。E-mail:huxingchen@fjmu.edu.cn。

摘要
癌细胞系模型已广泛用于药物敏感性测试及耐药标志筛选。然而研究者常忽视癌细胞系的基础耐药性,导致许多药效标志物难以应用于临床实践。为评估肿瘤细胞系的基础耐药性水平及其与临床药效的相关性,本研究以48种结直肠癌(colorectal cancer, CRC)细胞系为例,通过计算265种药物IC50值之间的相关性系数,以每种CRC 细胞系对上述药物IC50的中值构建可反映基础耐药性水平的评估指标MIC50,并识别出表达值与MIC50显著正相关的基因。然后基于样本内基因表达值的相对大小秩序关系构建临床CRC组织样本的基础耐药评分模型。结果表明:在药物两两间IC50的相关系数中,有99%以上呈显著正相关(FDR < 0.05),证明CRC细胞系存在基础耐药性特征;与MIC50显著相关602个基因,富集到4个与肿瘤耐药密切相关的功能模块。根据5368个MIC50相关基因对,筛选出一个由21个基因对组成的5-FU联合用药评分模型,卡方检验结果表明患者基础耐药性水平的高低与用药后的响应信息显著相关,生存分析显示低分组患者比高分组患者预后更好。本研究结果为CRC联合化疗耐药机制的研究及临床个体化用药提供了一定的理论依据。
关键词:
Abstract
Cancer cell line models are widely used for testing drug sensitivity and in screening for drug resistance markers. However, the general level of drug resistance in cancer cell lines is often ignored by researchers, making it difficult to apply many drug efficacy markers in clinical practice. In this study, we examined 48 colorectal cancer (CRC) cell lines to calculate the correlation coefficients between the IC50 values for 265 drugs. The general drug resistance evaluation index MIC50 was constructed using the median value of 265 drugs’ IC50 values. Genes with positively correlated expression values and a MIC50 which rose to significance were selected for further study. To analyze the effect of general drug resistance on the response status and prognosis in CRC patients, the general drug resistance scoring model was established based on within-sample relative expression orderings of gene pairs. The results demonstrate that more than 99% of the IC50 correlation coefficients of 265 drugs were significantly positive (FDR<0.05), indicating that CRC cell lines possessed general drug resistance characteristics. Furthermore, we identified 602 general drug resistance related genes, and by using Metascape, we identified four functional modules closely related to tumor resistance. A scoring model of 5-FU-based general drug resistance levels consisting of 21 gene pairs was built. After performing χ 2 test, we found that the general drug resistance level in CRC patients was significantly correlated with the response information after accepting 5-FU-based combination drug therapy. Survival analysis showed that the low scoring cohort of patients had a better prognosis than the higher scoring cohort, indicating that the level of basic drug resistance was closely related to the prognosis and drug response status in these patients. These results provide basic theoretical support for further research on the mechanism of combined chemotherapy resistance and the individualized regimen of clinical drug use in patients with CRC.
Keywords:
PDF (851KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
陈湖星, 徐蕾, 李静, 郭政, 敖露. 基于结直肠癌细胞系MIC50相关基因对的基础耐药
评分模型构建 . 遗传[J], 2020, 42(6): 577-585 doi:10.16288/j.yczz.19-336
Huxing Chen.
癌细胞系模型被广泛用于耐药标志物的筛选及耐药机制的研究。然而,有研究发现癌细胞系通常会呈现对多种药物普遍敏感或者普遍耐药的现象,即癌细胞存在与药物作用机制无关的一般药物敏感性水平(general levels of drug sensitivity, GLDS)[1],本文称之为基础耐药性水平(general levels of drug resistance, GLDR)。同时在临床上,肿瘤细胞对多种化学结构不同、作用机制各异的药物产生的交叉耐受现象,即多药耐药性(multidrug resistance, MDR),是造成化疗失败的重要原因 [2]。以中晚期结直肠癌为例,目前采用5-氟尿嘧啶(5-fluorouracil, 5-FU)为基础的多种药物联合化疗是临床一线治疗手段,然而因个体差异,约有50%患者对联合化疗不敏感,进而出现MDR现象[3,4]。因此研究者在利用细胞系模型识别耐药标志或研究耐药机制之前,有必要评估肿瘤细胞系的基础耐药性水平,并判断其与临床药效的相关性。
随着基因芯片与二代测序等高通量检测技术的迅猛发展,产生了大量的基因表达谱数据。利用这些数据,国内外研究者已识别出一些耐药标志物[4,5,6]。但上述基于基因表达值的药效评分标志物容易受实验室条件、试剂及实验操作人员等因素所造成的批次效应影响,可重现性较差,难以被应用于临床[7]。但基于样本内基因表达值的相对大小秩序关系(relative gene expression orderings, REO)的基因对标志物不易受到批次效应的影响,具有很强的稳健性,在癌的分子诊断与预后评估中应用价值明显[8]。
本研究将根据CRC细胞系对不同药物IC50值的中值,构建一个基础耐药性水平评估指标MIC50,并识别出MIC50相关基因;然后基于MIC50相关基因对的REO构建CRC组织的基础耐药性评分模型,以评估基础耐药性水平与临床上以5-FU基础的联合化疗药效的相关性,以期为CRC联合化疗耐药机制的研究及临床个体化用药提供一定的理论依据。
1 材料与方法
1.1 材料
本研究所用到的细胞系数据来自Genomics of Drug Sensitivity in Cancer (GDSC,Table 1
表1
表1CRC组织样本数据描述
Table 1
| 数据集 | 平台 | 样本数 | 联合用药样本 | 用药类型 | 文献 |
|---|---|---|---|---|---|
| GSE72968 | GPL570 | 68 | 68a | FUFOL/FOLFOX/FOLFIRI | [9] |
| GSE72969 | GPL570 | 56 | 28b | FOLFOX/FOLFIRI/FOLFIRINOX | [10] |
| GSE28702 | GPL570 | 83 | 56 c | FOLFOX | [11] |
新窗口打开|下载CSV
1.2 评估细胞系基础耐药性水平
构建指标MIC50表示细胞系对所有药物的基础耐药性水平(公式1):其中i代表第i个癌细胞系,j代表第j种药物,IC50(i,j)为第i个癌细胞系经药物j处理的IC50值,利用Spearman相关性分析计算CRC细胞系中265种药物中任意两种药物间IC50值的相关系数。
1.3 MIC50相关基因的筛选及蛋白质互作分析
在CRC细胞系的基因表达谱中,利用Spearman相关识别表达值与MIC50显著相关的基因(P<0.05),其中将相关系数大于0.3的基因定义为基础耐药相关基因。使用基因注释工具Metascape (1.4 基于MIC50相关基因对构建基础耐药评分模型
将上述基础耐药相关基因进行两两组合,构成基因对(gene pair, GP)。假设某一相关基因对由基因a和基因b组成,Ea和Eb分别表示该基因的表达值,根据每个GP的REO,将48种CRC细胞系分为1或0的两组。例如,Ea > Eb,分类标签为1;反之,分类标签为0。控制FDR < 0.2,采用T检验判断两组细胞系的MIC50是否存在显著的差异。若存在显著的差异,则判定该基因对的REO与MIC50相关。定义所有MIC50相关基因对为基础耐药相关基因对,并将其方向调整一致(即以Ga > Gb表示基础耐药模式,支持结直肠癌细胞系具有较高基础耐药水平)。以GSE72968为训练集,评价基础耐药相关基因对的REO与临床CRC组织样本联合用药响应状态间的对应关系。若Ga > Gb,则支持CRC组织具有相对较高的基础耐药水平,将样本标记为不响应;反之,则标记为响应。将判定的分类标签与CRC组织样本真实的响应状态进行比较,分别计算每个基础耐药相关基因对的敏感性(sensitivity)、特异性(specificity)和F-score (公式2),判断其对CRC组织样本联合化疗响应状态的分类效能。
提取F-score > 0.5的基础耐药相关基因对,采用顺序前向搜索(sequential forward selection, SFS)获得具有最优分类效能的特征基因对组合,定义为5-FU-based基础耐药相关基因对(具体流程详见图1)。然后计算上述所有5-FU-based基础耐药相关基因对的基础耐药模式在组织样本中的保持频率,即为CRC组织样本的5-FU-based基础耐药评分。
图1
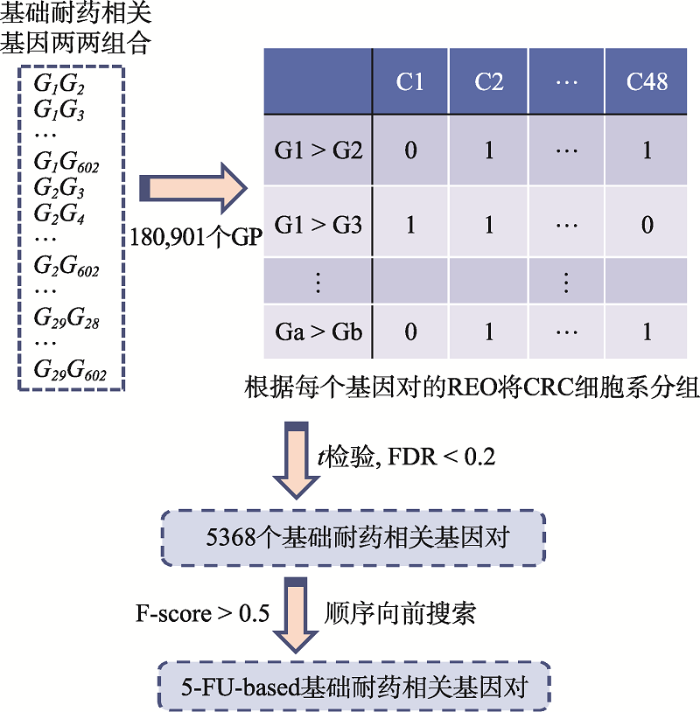 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图15-FU-based基础耐药相关基因对筛选流程图
Fig. 1The flow chart of screening 5-Fu-based general drug resistance related genes
1.5 统计分析
采用Benjamini-Hochberg(BH)法计算多重假设检验的假阳性发生率(false discovery rate, FDR)[13]。以每个数据集中所有组织样本基础耐药评分的算术平均值为阈值,将高于阈值的样本归为高基础耐药组(高分组),否则归为低基础耐药组(低分组)。通过χ2检验判断两组CRC组织样本对5-FU基础化疗方案的响应信息是否存在显著差异。采用Kaplan- Meier方法评估两类患者的生存率,绘制患者的生存曲线图;log-rank检验判断两组患者的生存率是否存在显著差异;单变量Cox比例风险回归模型用于计算风险比(hazard rate, HR)以及HR的95%置信区间(confidence intervals, CIs)。使用R软件3.5.1版进行上述统计分析。2 结果与分析
2.1 CRC细胞系基础耐药水平评估
采用Spearman相关性分析,控制FDR<0.05,计算CRC细胞系中任意两种药物的IC50值的相关性,结果发现全部药物中的8204对药物组合的IC50值存在显著相关性,其中99.80% (8185对)呈显著正相关;靶向药物中6770对药物组合的IC50值存在显著相关性,占比99.76%;化疗药物中药物间的IC50值相关系数则100%呈正相关(表2)。结果表明,若CRC细胞系对某种药物的IC50值高(或低),则对其他药物的IC50值也倾向于高(或低)。药物之间IC50值普遍存在的正相关关系,支持CRC细胞系存在基础耐药性水平的假设。Table 2
表2
表2CRC细胞系药物两两间IC50的相关系数
Table 2
| 药物类型 | 总相关系数 数目 | 正相关系数 数目 | 正相关系数 比例(%) |
|---|---|---|---|
| 全部药物 | 8204 | 8185 | 99.80 |
| 靶向药物 | 6770 | 6754 | 99.76 |
| 化疗药物 | 107 | 107 | 100 |
新窗口打开|下载CSV
2.2 MIC50相关基因的筛选与蛋白质互作分析
控制P < 0.05,采用Spearman相关分析得到1576个表达与CRC细胞系MIC50显著相关的基因。将相关系数大于0.3的602个基因定义为基础耐药相关基因。通过基因注释工具Metascape进行蛋白质互作分析,并利用分子复合体(MCODE) 算法识别密切联系的网络组分[14],从602个基因中识别出12个功能模块(图2)。图2
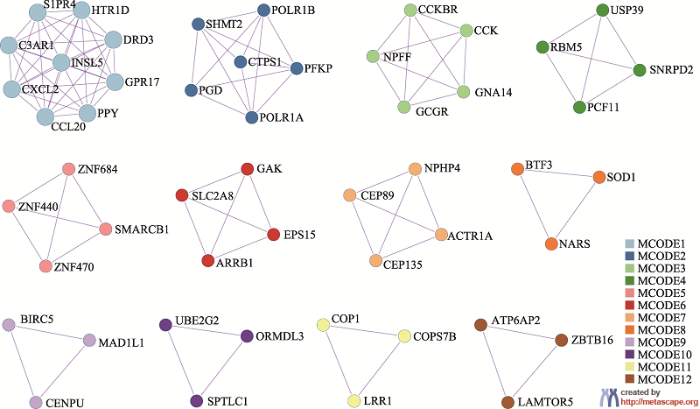 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基础耐药相关基因的蛋白质互作与MCODE识别
Fig. 2Protein interaction and MCODE recognition of the general drug resistance related genes
根据网络连通度排名,前4个MCODE包含的基因主要涉及的功能通路如表3所示。其中,MCODE1和MCODE3模块中的基因与G蛋白偶联受体(G-protein-coupled receptor, GPCR)关系密切;MCODE2模块中的基因涉及嘧啶(pyrimidine)、碳、磷酸核糖等代谢功能,即与细胞自身代谢相关;而MCODE4则主要与mRNA 剪接相关。已有研究表明上述功能与肿瘤耐药有密切联系,如:激活Gli分子可通过平滑受体(smoothened, SMO)介导的GPCR样信号引起肿瘤细胞获得性耐药的产生[15];嘧啶代谢通路能够促进化疗药物5-FU的代谢,进而导致耐药[16];一些特定基因(CD19、BIM和BRAF)的可变剪接可导致相应治疗的失败[17]。
Table 3
表3
表3每个MCODE涉及基因的通路富集
Table 3
| MCODE | 通路ID | 通路名称 | Log10(P)值 |
|---|---|---|---|
| 1 | R-HSA-373076 | 视紫红质样受体(class A/1 (rhodopsin-like receptors)) | -16.9 |
| 1 | R-HSA-418594 | G alpha (i)信号事件(G alpha (i) signalling events) | -16 |
| 1 | R-HSA-500792 | GPCR配体结合(GPCR ligand binding) | -15.5 |
| 2 | hsa00240 | 嘧啶代谢(pyrimidine metabolism) | -5.8 |
| 2 | hsa01200 | 碳代谢(carbon metabolism) | -5.7 |
| 2 | GO:0019693 | 磷酸核糖代谢过程(ribose phosphate metabolic process) | -5.3 |
| 3 | R-HSA-416476 | G alpha (q)信号事件(G alpha (q) signalling events) | -10.3 |
| 3 | R-HSA-500792 | GPCR配体结合(GPCR ligand binding) | -6.2 |
| 3 | R-HSA-375276 | 肽配体结合受体(peptide ligand-binding receptors) | -5.3 |
| 4 | R-HSA-72163 | mRNA剪接-主要途径(mRNA Splicing-Major Pathway) | -8.5 |
| 4 | R-HSA-72172 | mRNA剪接(mRNA Splicing) | -8.4 |
| 4 | R-HSA-72203 | mRNA前体加工(processing of capped intron-containing pre-mRNA) | -8.0 |
新窗口打开|下载CSV
2.3 CRC组织样本的基础耐药水平与临床药效的相关性评价
将602个基础耐药相关基因组合,共得到180 901个基因对。利用t检验,控制FDR < 0.2,从中得到了5368对基础耐药相关基因对。以GSE72968为训练集,从基础耐药基因对中筛选出21对5-FU-based基础耐药基因对,建立临床CRC组织样本的5-FU-based基础耐药评分模型(表4)。Table 4
表4
表45-FU-based基础耐药评分模型包含的基因对
Table 4
| 序号 | 基因对(Ga > Gb) | 序号 | 基因对(Ga > Gb) | 序号 | 基因对(Ga > Gb) |
|---|---|---|---|---|---|
| 1 | BATF2> HDDC2 | 8 | LRP4> ASMTL | 15 | TBC1D2B> ESPNL |
| 2 | BATF2> TAZ | 9 | SIM2> MBD2 | 16 | TNFAIP8> KCNH2 |
| 3 | BATF2> TULP3 | 10 | SPRR2G > LMTK3 | 17 | TMEM38B> SLC26A3 |
| 4 | CELSR3> AGTRAP | 11 | WFS1> GOLT1A | 18 | TMEM38B> PCF11 |
| 5 | EPHX2> KCNH2 | 12 | IL1RL2> OR1D2 | 19 | RHOU > PCF11 |
| 6 | GSTM3> NID1 | 13 | IL1RL2> ZDHHC14 | 20 | HMGB4> SMIM10 |
| 7 | GSTM3> NOD1 | 14 | PREB > TMEM60 | 21 | CCDC17> FAM110B |
新窗口打开|下载CSV
根据基础耐药评分模型,将训练集GSE72968的患者划分为基础耐药低分组(25例)和基础耐药高分组(43例),其中低分组患者表现为用药响应的有21例,高分组患者表现为用药响应的仅有16例。χ2检验结果表明基础耐药高、低分两组患者对基于5-Fu联合用药的响应信息率有显著差别(P = 0.00019),即基础耐药高分组的患者对5-FU联合用药的响应率显著低于基础耐药低分组的患者(表5)。这一结果在数据集GSE72969与GSE28702中得到了验证(表5),即基础耐药低分组患者对5-FU联合用药的响应率也显著高于基础耐药高分组(χ2检验,P < 0.05)。以上结果说明患者基础耐药水平的高低与5-FU联合用药的临床响应信息显著相关。
Table 5
表5
表5基础耐药性水平与样本响应信息的相关性评价
Table 5
| 数据集 | 样本数 | 低分组响应率 | 高分组响应率 | P值 |
|---|---|---|---|---|
| GSE72968 | 68 | 84% (21/25) | 37.2% (16/43) | 0.00019 |
| GSE72969 | 28 | 71.4% (10/14) | 28.5% (4/14) | 0.02334 |
| GSE28702 | 56 | 76% (19/25) | 38.7% (12/31) | 0.00526 |
新窗口打开|下载CSV
此外,上述数据集中包含96例有生存信息的样本,其中基础耐药低分组39例、高分组57例。生存分析结果表明:低分组患者的无进展生存时间(progression-free survival, PFS)显著长于高分组患者(10个月的无进展生存率66.7% vs. 28%, HR = 1.86, P = 0.00423,图3A)。另外,低分组患者的总体生存时间(overall survival,OS)也显著长于高分组患者(30个月的总体生存率48.7% vs. 21 %, HR = 1.78, P = 0.0182,图3B)。以上说明基础耐药水平的高低与患者联合用药的获益也显著相关,且低基础耐药水平的患者倾向于预后良好。
图3
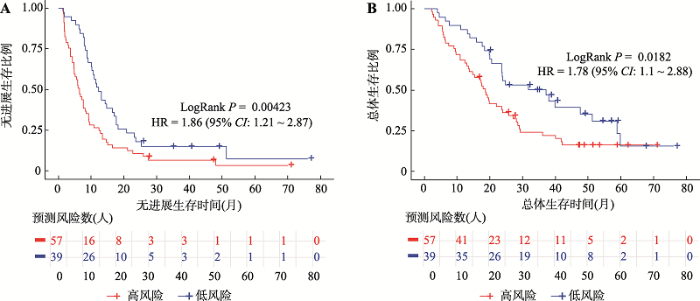 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3组织样本的5-FU-based基础耐药评分对患者生存时间的预测
A:无进展生存时间(PFS)预测;B:总体生存时间(OS)预测。
Fig. 3Prediction of survival time of patients with 5FU-based general drug resistance scores in tissue samples
3 讨论
癌细胞系实验常用于药物敏感性测试与药效标志的筛选。目前研究者已构建了许多大型药物敏感性数据库,如Genomics of Drug Sensitivity in Cancer (GDSC)、Cancer Cell Line Encyclopedia (CCLE)和Cancer Therapeutics Response Portal (CTRP)数据库等,旨在通过这些数据库来实现精准医学的研究。Geeleher等[1]通过对CCLE、CTRP和CRP等数据库进行分析,发现不同药物的IC50值之间普遍存在正相关关系,但是该研究未单独针对某特定癌种的细胞系进行系统分析。本研究通过计算GDSC数据库中48例CRC细胞系对应265种药物彼此间IC50值的Spearman相关系数,结果表明不同药物的IC50值之间的正相关比例高达99.80%。值得注意的是,265种药物包括氟尿嘧啶、顺铂(cisplatin)和紫杉醇(paclitaxel)等24种传统化疗药物,及吉非替尼(gefitinib)、索拉菲尼(sorafenib)和阿法替尼(afatinib)等241种靶向作用药物,其作用机制各有不同,说明CRC细胞系存在某种与药物作用机制无关的基础耐药特征。绝大多数以癌细胞系为基础的临床前药效模型,虽然经过大量的实验验证,但仍在3期临床试验中失败[18]。研究者分析了诸多可能导致药效模型的失败原因,如:细胞系在培养过程中发生未知的遗传变异[19],或者培养未能包含肿瘤微环境的关键组分,再或者是丢失了在原发组织中的肿瘤异质性特征[18]。然而,癌细胞系的基础耐药性水平(GLDR)并未受到广大研究者重视。本研究初步证实一种癌细胞系对所有药物的IC50值的中值(即MIC50)可以较好地代表其基础耐药性水平,并根据48种CRC细胞系的MIC50值,识别出602个CRC基础耐药相关基因。通过蛋白质网络互作与功能富集发现,4个主要的基因功能模块(MOCODE)都与肿瘤耐药相关。另外,Tong等[20]根据6对基因组成预后标志,可将NCI-60中的58细胞系分为耐药或敏感细胞系,其中7种结直肠癌细胞系(HCT-116、COL205、SW620、HT29、KM12、HCC2998和HCT-15)均被判定为敏感。而事实上,若根据MIC50值对本研究中的48种CRC细胞系进行高低排序,将排序前50%的细胞系归为低基础耐药组,Tong等[20]报道的7种结直肠癌细胞系都被分为低分组,且有5种排序在前20% (图4),说明CRC癌细胞系的MIC50值与临床药效标志的识别关系紧密。因此,在使用癌细胞系进行药敏测试或耐药标志的筛选时,有必要预先评估癌细胞系的GLDR。
图4
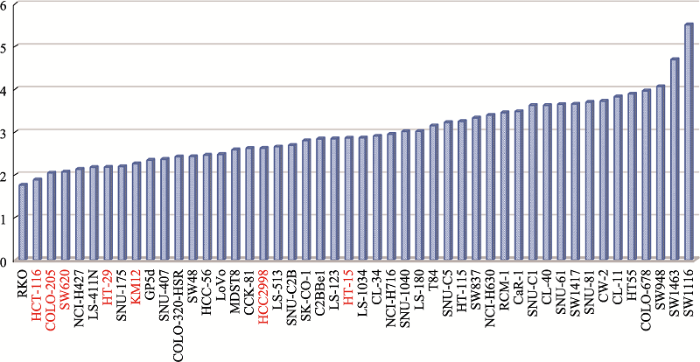 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4CRC细胞系的MIC50值分布图
Tong等[20]报道的7种结直肠癌细胞系名称已标为红色。
Fig. 4Distribution of MIC50 values of CRC cell lines
此外,Geeleher等[1]发现通过主成分分析的方法校正细胞药物敏感性的一般水平(GLDS),可显著改进药物特异性药效标志的识别。Marc等[21]发现校正细胞周期因素也能够显著改进药效标志的识别,并建立一个判断药物敏感性的新指标GR50。但上述研究建立的校正方法需要遵循严格的实验方案,难以实现跨数据集的应用,也难以对临床组织样本进行个体化分析。相比之下,本研究提出的基于MIC50相关基因对(基础耐药相关基因对) REO的评分模型可以对临床组织样本的基础耐药水平进行评估,具有一定的临床应用价值。通过对联合用药响应信息的分析,表明该评分模型能够有效评价CRC患者对基于5FU的联合用药的基础耐药水平。同时,经文献检索发现该评分模型中的大多数基因都与癌的发生发展、预后或者辅助化疗的耐药相关,如:TULP3基因在CRC组织中的表达显著高于癌旁组织,且该基因的表达与血管及淋巴结侵袭相关,是一个潜在的CRC诊断与预后生物标志[22];AGTRAP基因的过表达与结肠癌及黑色素瘤患者的预后呈负相关[23];TNFAIP8基因编码的SCC-S2蛋白是结肠癌组织中的一种重要的癌蛋白,在维持结肠癌细胞的恶性增生中发挥重要作用[24],并且Wang等[25]发现根据该蛋白的表达情况可预测卵巢癌患者新辅助化疗的耐药及预后;BATF2作为一种肿瘤抑制因子,在肺腺癌、结直肠癌、食管癌中均得到了验证,近来有研究者发现BATF2可以通过抑制Wnt/β-catenin信号逆转胃癌细胞的多药耐药[26];LMTK3是一种致癌激酶,涉及乳腺癌、肺癌和结直肠癌等多种癌症,其过表达能够降低乳腺癌细胞系对化疗药物的敏感性[27]。但上述基因均尚未在CRC中进行较为系统的耐药研究,其更深层次的遗传机制值得进一步关注。
综上所述,CRC细胞系存在基础耐药特征,且组织样本的基础耐药性水平与5-FU联合用药患者的预后及药物响应状态密切相关。但根据MIC50值校正基础耐药性水平后,CRC细胞系对药物特异的耐药机制分析及与临床的相关性评价尚待后续研究。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
