 ,1
,1Transcriptome profiling of high oleic peanut under low temperatureduring germination
Gao-hua Zhang1, Shutao Yu2, He Wang1, Xuda Wang ,1
,1通讯作者:
编委: 张根发
收稿日期:2019-04-4修回日期:2019-09-5网络出版日期:2019-11-20
| 基金资助: |
Editorial board:
Received:2019-04-4Revised:2019-09-5Online:2019-11-20
| Fund supported: |
作者简介 About authors
张高华,博士,研究方向:植物抗逆基因工程E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (1079KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张高华, 于树涛, 王鹤, 王旭达. 高油酸花生发芽期低温胁迫转录组及差异表达基因分析[J]. 遗传, 2019, 41(11): 1050-1059 doi:10.16288/j.yczz.19-097
Gao-hua Zhang, Shutao Yu, He Wang, Xuda Wang.
低温伤害是影响作物生长和产量的重要环境因素之一,引起植物细胞发生一系列生理生化改变,包括细胞膜脂相变、结构变化和各种酶活性的改变,进而导致膜脂过氧化、胞壁黏连、细胞脱水,甚至蛋白质变性,最终致使植物部分或整体死亡[1]。但是也有一些植物在低温胁迫下可以通过自身生理和生化的调整,提高对冷冻温度的耐受能力[2]。研究表明,植物对低温的耐受性涉及到细胞结构的重建和基因表达的改变,通过触发一系列抵御寒冷损伤的保护机制,如细胞内可溶性物质的增加、低温保护剂和抗氧化剂的积累以及防冻蛋白的诱导来提高耐寒性[3,4,5]。近年来,人们对植物的低温应答机制进行了广泛的研究。随着高通量测序技术的飞速发展,通过基因组、转录组等生物信息学分析可以更加全面地认识作物抗逆性的遗传基础,了解植物的基因表达调控模式和代谢途径。
花生(Arachishypogaea L.)是世界上第四大油料作物,也是我国最重要的经济作物和油料作物之一[6]。随着花生育种研究的发展,高油酸花生受到越来越多的关注。高油酸花生中油酸含量高,不仅能降低人体有害胆固醇,减缓动脉粥样硬化,有效预防心血管疾病的发生[7];同时,由于油酸的稳定性高,可以延长高油酸花生及其制品的货架期[7,8]。因此,提高花生油酸含量已成为国内外花生品质改良育种的研究热点之一。很多国家已经开展了高油酸花生育种,并利用高油酸材料F435培育出不同植物类型的新品种,并在生产上得到应用[9]。我国越来越重视高油酸花生的育种研究,目前也取得了很多突破性进展,研究成果显著[10]。
辽宁省花生以品质优良、黄曲霉污染率低、出口量大而著称,是省内仅次于玉米和水稻的第三大粮油作物[11]。辽宁花生主要生产于干旱半干旱的辽西北地区,年度间气温波动大。一直以来,受到地域环境和地理分布的影响,高油酸花生在辽宁地区的引种和研发受到很大的限制[12]。从当地花生品种中筛选油酸含量高而且抗寒性好的花生品种,是高油酸花生在低温地带引种和育种成功较为有效快速的方法。辽宁省沙地治理与利用研究所选用40余种待选花生高油酸品种,在辽宁阜新花生试验田进行田间抗寒能力和适应性评价,筛选出几种耐寒表现较好的高油酸花生品种[13]。为了解高油酸花生在分子水平上对低温胁迫的响应机制,本研究选择田间试验中4种耐寒性不同的高油酸花生品种,构建了花生发芽期低温胁迫后转录组测序文库,采用转录组高通量测序技术获得转录组基因序列,并对不同的花生品种之间的差异表达基因进行功能分类和代谢通路分析,筛选并鉴定出几种在低温条件下起重要调节作用的基因,为利用基因工程技术改良花生抗逆能力提供理论基础。
1 材料与方法
1.1 植物材料
高油酸花生品种S51、S53、花育661 (HY661)和L126的种子由辽宁省沙地治理与利用研究所提供,均为3年以内收获的种子。1.2 花生低温冷浸发芽
选择田间耐寒表现较好的辽宁花生高油酸品种S51和S53及相对不耐寒的高油酸花生品种HY661和L126,选取品相一致的花生种子各50粒,低温2℃黑暗浸泡处理72 h,然后置于培养皿里,在无菌水浸泡的滤纸上萌芽,22℃黑暗培养,3 d后统计露白率和芽长/种长。取相同数目的花生种子作为对照 组,常温22℃浸泡黑暗处理72 h。每个实验设3次生物学重复,并进行ANOVA测验和Tukey多重比较检验。将低温浸泡组和对照组放入25℃人工气候箱中,16 h/8 h光周期持续培养14 d后观察其生长状况。1.3 花生低温胁迫及转录组测序
选取耐寒的高油酸花生品种S51和S53,以不耐寒品种HY661和L126作为对照,进行低温胁迫转录组测序。发芽3 d的花生种子经过0℃处理24 h后,经液氮冷冻,采用RNeasy Plant Mini Kit (Qiagen, Hilden,德国)提取花生种子总RNA,经NanoDrop ND-1000分光光度计(NanoDrop,德国)进行RNA纯度与质量检验,由南京诺唯赞生物科技公司通过Illumina Hiseq2000平台测序组装。1.4 抗寒基因生物信息学分析
对测序获得的转录组数据进行生物信息学分析,使用BLAST软件将Unigene序列在NR、Swiss-Prot、GO、COG、KOG和KEGG数据库中比对,预测Unigene氨基酸序列;利用HMMER软件与Pfam数据库比对,获得Unigene的注释信息。根据NR注释信息,使用Blast2GO软件得到Unigene的GO注释信息,采用WEGO软件对所有Unigene进行GO功能分类统计。将Unigene和COG数据库进行比对,预测Unigene功能并对其进行功能分类统计。将S53与HY661、S51和L126分别进行差异基因比较,统计差异表达量大于2倍的基因数目,并对差异表达基因进行GO功能注释和KEGG Pathway富集分析。1.5 抗寒基因筛选及功能分析
根据转录组测序结果,筛选7种低温诱导基因进行qRT-PCR验证。采用发芽3 d的高油酸花生品种S51,低温0℃黑暗处理24 h,分别在处理后0 h、3 h、12 h、24 h采集样品,提取发芽种子的总RNA。用DNaseⅠ消化DNA并检验RNA浓度及质量。利用 Primer 5.0 软件,根据转录组获得的基因序列信息,设计qRT-PCR扩增引物(引物序列见附表1),并进行引物熔解曲线分析,扩增效率达到95%以上。采用SYBRPremixExTaqTM(TaKaRa,日本)试剂盒在ABI 7500快速实时定量系统(ABI,美国)上进行扩增。反应条件:95℃30 s;95℃ 10 s,56℃ 25 s,72℃ 25 s,40个循环。以花生Actin为内参基因,采用2-ΔΔCt法计算相对表达量,每个目的基因分别进行3次生物学重复和3次技术重复。2 结果与分析
2.1 花生发芽期耐寒性检测
对43份高油酸花生材料进行田间种植及初步耐寒性筛选,选择各方面生长状况良好的耐寒品种18个,由中国农业科学院油料作物研究所进行高油酸含量测试。结果显示,这18个耐寒品种花生的油酸含量均高于75% (数据未发表)。选择田间耐寒能力较好的花生品种S51、S53和耐寒性一般的HY661、L126进行实验室发芽耐寒能力检测。经低温2℃浸泡处理72 h,22℃萌发3 d后统计花生的发芽率(图1A)和平均芽长/种长(图1B)。结果显示,HY661和L126的萌发率明显低于S51和S53,其平均芽长/种长也相应低于S53和S51。进一步在25℃常温条件下持续培养14 d后,观察花生的生长状态。结果显示,经低温处理的花生生长状态都弱于对照组,但是S51和S53的生长状态要明显好于HY661和L126 (图2)。图1
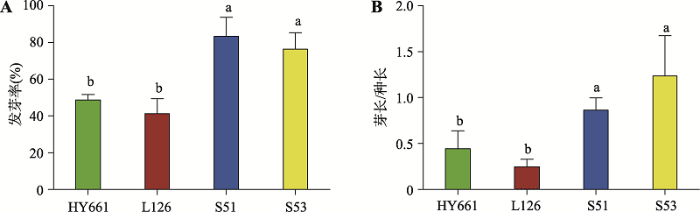 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图14个品种花生经2℃冷浸处理72 h后发芽状态检测
A:发芽率;B;平均芽长/种长。统计结果为3次重复实验数据的平均值±标准误差;小写字母不同表示差异显著(P≤0.05)。
Fig. 1Determing the germination status of different peanut varieties under 2℃ cold stress for 72 h
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图24个品种花生经低温处理72 h后生长14 d时表型观察
Fig. 2The phenotypes of four different peanut varieties Growing 14 d after 72 h of cold stress
2.2 花生转录组数据分析
2.2.1 花生转录组注释结果统计经过田间低温筛选和室内发芽实验,选取较耐寒的高油酸花生品种S51、S53,以不耐寒品种HY661和L126作为对照,用于低温胁迫转录组测序。发芽3 d的花生种子经过0℃处理24 h后,提取总RNA并进行测序。测序结果表明,共获得139 429条Unigene,总长为175 715 359nt,平均长
度1260nt,N50为1968nt。将获得的Unigene进行功能注释,注释到NR、NT、Swiss-Prot、KEGG、COG和GO数据库的Unigene数目分别为96 398、95 976、63 984、59 614、39 213和76 800条。通过预测编码蛋白框(coding sequence, CDS),比对到蛋白库的CDS为96 306个,预测出的CDS为2285个。
2.2.2 Unigene的GO和COG功能分类GO功能分类可以从宏观上认识该物种的基因功能分布特征。由于存在单个Unigene可以与多种基因功能条目相对应的关系,本研究最终有76 800个Unigene与GO注释条目建立了对应关系。所有的Unigene分别注释到3个功能大类,即基因的分子功能(molecular function)、细胞组分(cellular component)和参与的生物过程(biological process) (图3A)。在生物过程中涉及基因最多的是代谢过程和细胞过程;此外,涉及应激反应的基因数目为21 895条,其中响应低温刺激(GO:0009409)的基因数为764条。细胞组分中涉及功能最多的是细胞、细胞部分以及细胞器,高达5万条以上。分子功能中基因数目最多的是结合反应和催化反应功能的基因。
图3
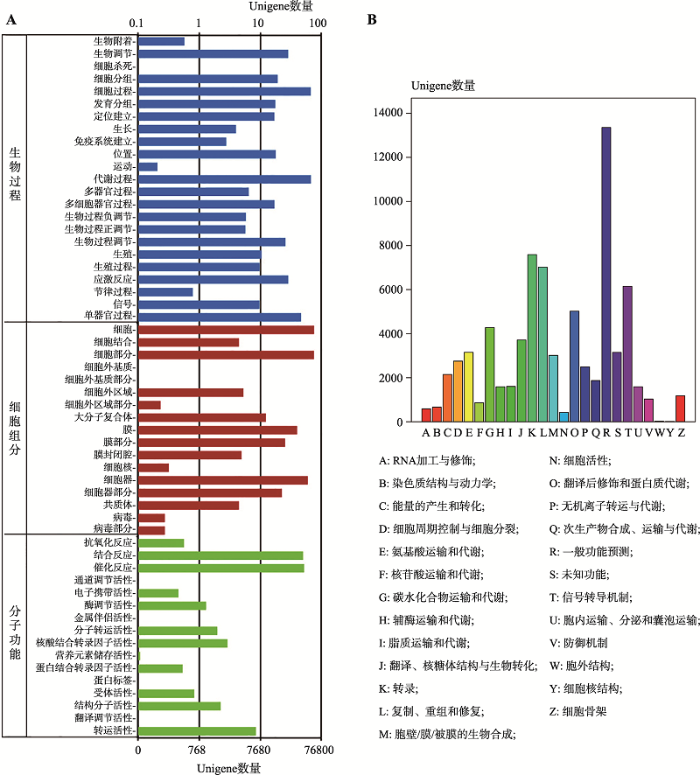 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3花生种子发芽期转录组Unigene GO和COG功能分类
A:GO功能分类;B:COG功能分类。
Fig. 3GO and COG functional classification of unigenes of peanut under cold stress at germination stage
为了解花生发芽期转录基因产物的功能分布特征,本研究将测序获得的全部Unigene与COG数据库进行比对,对转录表达的蛋白功能进行预测和统计归类。数据分析显示,共有39 213条Unigene与COG数据库中的功能分类项目匹配。根据功能分类,初步将花生种子转录组中的基因归为25类,其中数目最多的是一般功能预测(R)的基因,有13 354条之多,其次是涉及转录(K)和复制、重组、修复(L)以及信号转导机制(T)的基因,分别达到5000条以上。另外还有3147条未知功能(S)类别的基因(图3B)。
2.2.3 高油酸花生发芽期低温诱导转录组差异表达基因分析
本研究对不同品种高油酸花生低温处理后的转录组进行差异表达分析,找出在不同样本间差异表达基因(DGE)。结果显示,共有21 665个基因存在显著性差异。分别将品种S53与HY661、S51与L126进行比较,结果显示两组耐寒性不同花生品种之间的DGE分别是12 744和8920个,共同的DGE为3520个(图4A)。而两个耐寒品种相对于不耐寒品种,上调表达的DGE数目远远大于下调表达的DGE数目(图4B)。
图4
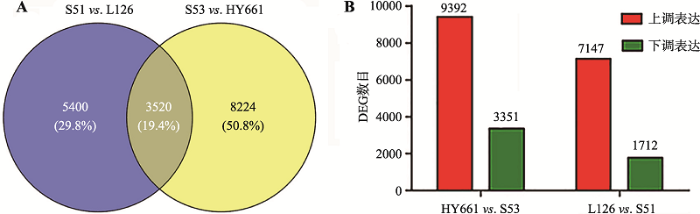 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图44个花生品种经低温处理后差异表达基因数目
A:DGE维恩图;B:DGE数目统计。
Fig. 4Statistics of DEG in four peanut varieties under cold stress
对两组耐寒和不耐寒高油酸花生之间差异表达基因进行GO功能分类比对,品种S51与L126之间的比对结果显示,生物过程和细胞成分中差异基因数目较多,其中细胞成分中数量较多的是细胞膜和细胞外周功能,而在生物过程中差异基因数目较多的是单器官发育过程、发育过程、碳水化合物代谢过程和解剖结构发育,但是分子功能中差异基因数目并不多(图5A)。进一步通过KEGG通路分析显示,S51与L126差异基因中数量较多集中在植物病菌互作、植物激素信号转导、淀粉和糖类代谢、剪切体和RNA转运功能(图5B)。品种S53与HY661的GO功能分类和KEGG通路分析结果与品种S51与L126类似,说明这两组耐寒和不耐寒高油酸花生之间具有相似的基因表达模式。
图5
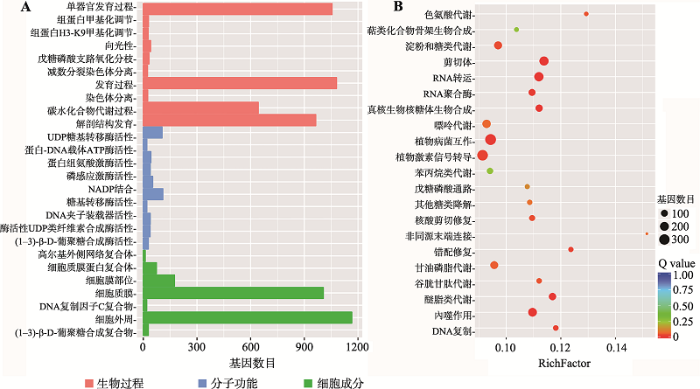 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5花生品种S51和L126差异表达基因的GO功能分类及KEGG通路分析
A:差异表达基因的GO功能分类;B:显著富集的差异基因的KEGG通路分类。
Fig. 5GO and KEGG pathway of peanuts varieties S51 and L126
2.2.5 花生低温诱导基因的表达分析
表观遗传调控、糖酵解、细胞壁重构、蛋白激酶、转录因子、脱落酸(abscisic acid, ABA)信号途径等对种子萌发至关重要[14,15]。根据转录组测序结果中的差异表达基因的分析及功能分类结果,本研究筛选出在上述调控途径中表达量上调的关键基因,其中Unigene44032_All是苜蓿(Medicagotruncatula)时钟调控蛋白TIME FOR COFFEE (TIC)的同源基因,在ABA信号途径中发挥作用[16]。Unigene22874_All和Unigene5925_All分别是大豆(Glycine max)组蛋白赖氨酸N甲基转移酶ATX3和AGO4蛋白的同源基因,参与表观遗传调控[17,18]。CL243.Contig3_All是FERONIA类受体蛋白激酶(FER)基因,与细胞性状、伸长及环境胁迫有关[19]。此外还筛选出3种转录因子基因:bHLH49-like (CL13529.Contig29_All)、3R-1MYB (CL13161.Contig1_All)和EREB (CL13161. Contig1_All)。为了验证转录组测序结果的准确性,本研究对发芽3 d的高油酸花生品种S51,经低温0℃黑暗处理,通过qRT-PCR检测这些基因在0 h、3 h、12 h、24 h时的表达量。结果显示,随着低温胁迫时间的延长,TIC、ATX3和AGO4的表达量在胁迫3 h后明显升高,并且TIC和ATX3在胁迫12 h后的表达水平达到10倍以上,而FER在胁迫12 h后才有明显升高。转录因子基因bHLH、MYB、EREB在受到低温诱导3 h后表达量都显著升高,但bHLH在12h后表达量有所降低(图6),表明这些基因在花生发芽期低温胁迫时均明显上调,响应并参与了转录调控,这与转录组数据分析结果一致。
图6
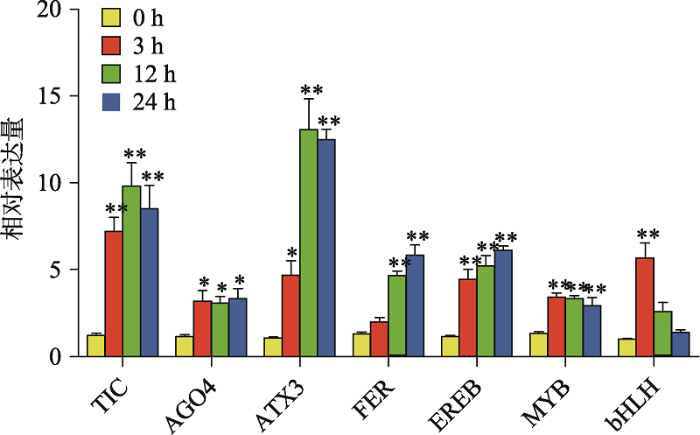 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6S51花生低温诱导基因表达量分析
基因表达量为3次重复的平均值±标准误,采用ANOVA测验,进行LSD/SNK检验。*P<0.05,表示差异显著;**P<0.01,表示差异极显著。
Fig. 6The expression patterns of cold-induced genes in peanut S51
3 讨论
本研究对耐寒性不同的高油酸花生品种在低温胁迫下的转录组文库进行了比较分析,初步了解在发芽期低温诱导基因的转录表达机制。分析结果显示,两组耐寒性不同花生品种之间的DGE分别是12 744和8920个,共同DGE为3520个。而相对于不耐寒品种,两个耐寒品种上调表达的DGE数目远远大于下调表达的数目(图4)。该研究结果表明在耐寒和低温敏感花生品种之间存在大量的差异表达基因,共同的差异表达基因也显示了在耐寒花生品种间具有很多相同的基因调节特征和途径,并且大多是通过正调节的方式来响应低温胁迫。GO功能分类分析显示,差异基因数量较多的集中在器官发育、碳水化合物合成、细胞外周和细胞膜方面(图5A),这表明在花生萌发期,其器官形态发育及能量合成代谢极为活跃,而低温胁迫容易造成细胞结构、细胞质膜和细胞外周组织受损伤,所以诱导大量调控基因激活耐寒花生中的细胞膜修复机制,重建细胞膜内外结构的调控网络,可能是耐寒高油酸花生具有更好的适应性和保护性的主要原因之一。KEGG通路分析显示,耐寒花生与不耐寒花生的差异基因的数量较多集中在植物病原体互作、植物激素信号转导通路中(图5B),说明在受到低温胁迫时,植物对逆境的响应与病害入侵的途径有共同的模式。另外,很多植物在受到逆境胁迫时会积累ABA等植物激素来调节相关基因的表达[2,3],因此差异基因在植物激素信号转导途径的富集证明植物激素在低温胁迫下起重要调控作用,激活和诱导相关代谢途径中的基因表达,提高花生对低温的适应性。研究表明在种子萌发初期,细胞壁重构以及植物激素信号途径等方面的早期激活对种子萌发至关重要。本研究挑选了相关基因进行了表达模式分析,其中TIC编码的时钟调控蛋白是植物体内平衡的核心元素,它整合和协调发育、代谢和环境信号。在拟南芥(Arabidopsis thaliana)中,tic-2突变体转录组分析显示TIC和ABA响应基因之间存在极大的关联性。在ABA处理下,tic-2突变体的种子萌发率下降20%,表明TIC表达下调会导致ABA信号调节基因增强[16]。在本研究中,耐寒花生S53/S51种子低温萌发率高于不耐寒花生品种HY661和L126,同时对低温诱导基因表达量进行检测,结果显示S51的TIC表达显著升高,推测TIC表达升高抑制了ABA信号途径,从而提高了萌发率。另一个转录表达明显升高的是FERONIA类受体蛋白激酶基因(FER),其编码蛋白是植物信号转导中的重要载体,参与植物生长发育、胚珠受精、细胞壁完整性和病原体反应。最新的研究显示,拟南芥质膜的FER类蛋白对于高盐胁迫后恢复根系生长非常必要。FER具有细胞壁感受功能,在FER突变体中,根细胞在生长恢复过程中会突然爆裂,表明FER在受到渗透胁迫后能够维持细胞壁的完整性[19]。在本研究中,耐寒花生S51和S53种子中FER基因在低温胁迫下表达量升高,猜测其对细胞的完整性有保护作用。
最近的研究表明,表观遗传调控在种子休眠和萌发进程中发挥了重要作用。转录组差异表达分析发现耐寒花生种子中AGO4和ATX3同源基因在低温处理下表达水平显著升高,这两个基因分别与DNA甲基化[18]和组蛋白修饰[17]相关。Singh等[20,21,22]通过分析AGO4在大麦(Hordeumvulgare)和小麦(Triticumaestivum)中的表达模式以及对5S rDNA的甲基化作用,表明AGO4对种子休眠起负调控作用。本研究耐寒花生种子中AGO4在萌发过程中表达量升高,可能打破了种子的休眠过程,促进了种子的萌发。拟南芥Trithorax (ATX)蛋白家族负责组蛋白赖氨酸的双/三甲基化。已有的研究表明,atx4和atx5突变体种子在ABA处理下的萌发率显著降低,表明ATX4和ATX5参与了ABA信号通路的调控[23]。此外,ATX3的表达量在种子休眠到萌发过程中逐渐升高,使种子萌发相关基因的三甲基化水平升高,相应地提高了它们的表达水平[17]。另有研究表明,atx3/4/5三突变体会引起严重的植物生长缺陷,因此认为ATX3、ATX4和ATX5基因亚家族很有可能共同通过调控ABA信号调节基因的双/三甲基化来参与种子的萌发过程[24]。在本研究中,耐寒花生种子中ATX3同源基因在低温处理下表达量升高,很可能负调控了ABA信号途径,从而提高了种子在低温条件下的萌发率。
在低温条件下,植物进化出一系列精密的调控网络来响应低温胁迫,提高植物对低温的适应性,转录因子就是其中重要的环节。目前研究最全面的低温响应途径是由CBF/DREB(C-repeat-binding factors/dehydration response element)转录因子家族调控[4]。这些转录因子可以结合到CRT/DRE反应元件上,在低温驯化过程中对下游低温调节基因(cold regulated, COR)的表达进行正调控[25]。同时,CBF/DREB又可以被CBF诱导基因(inducer of CBF expression, ICE)通过特异性识别MYC顺式元件激活,形成低温诱导调节途径ICE-CBF-COR[26,27,28]。近年来,bHLH转录因子在非生物胁迫方面的作用也越来越引起重视,其中ICE属于MYC类bHLH转录因子家族[29],它可以特异性地提高CBF3的表达[30]。另有研究显示,拟南芥中的bHLH122和bHLH92、烟草(Nicotianatabacum)bHLH123以及小麦bHLH13在低温、干旱、渗透胁迫中表达量升高,显示bHLH类转录因子基因在逆境胁迫下起调控作用[30]。本研究在耐寒性较好的花生品种中筛选出几种转录因子基因,在S51花生品种中表达量相对较高。低温胁迫实验结果显示,S51在低温诱导3 h后bHLH、EREB、MYB基因表达量显著升高,说明这些基因在花生萌发早期与低温胁迫转录调控相关。相对于冷敏感花生,耐寒花生品种在受到低温胁迫时,各种转录因子表达活跃,积极激活下游的低温诱导基因表达,对低温伤害起一定的保护和修复作用。
本研究对高油酸花生在发芽期转录水平上的耐寒机制进行了初步探索。随着野生种和栽培种花生全基因组测序信息的全面释放,将有大量的基因和调控因子被克隆,探明这些基因的功能和调控途径是提高作物对低温胁迫耐受性的关键。今后将通过进一步的生物信息学分析、基因表达调控及功能验证来了解花生在转录水平上的低温应答机制,为高油酸花生在低温地区的引进或改良提供更多的参考。
附录:
附表1见文章电子版(Supplementary Table 1
附表1
附表1 qRT-PCR引物序列
Supplementary Table 1
| 引物名称 | 序列(5′→3′) |
|---|---|
| Actin-F | CGGAGCGTGGATACTCCTTC |
| Actin-R | ACCTTTCCGCCCCTATTGTG |
| TIC-F | GTCCCATAGTTCAGGTGCCA |
| TIC-R | TGATTGCTGCATTAGCCCCA |
| AGO4-F | TCGTTACCCAGTGCATTGCT |
| AGO4-R | GGGACACATCCATGCCAAGA |
| ATX3-F | ACGTGGTAATCGACGCAACT |
| ATX3-R | TCATACCCGCAGAAAGATCGG |
| FER-F | TTGGGGTCCCTTACCTTCCA |
| FER-R | AAGTTCGCCGGATAAGGCAT |
| bHLH-F | AAAGGGGGAGCAACAACCAA |
| bHLH-R | GCTGCATCCAGGTACAAGGT |
| EREB-F | TGTGGCTGAGACACTGCAAA |
| EREB-R | TCCCCATGTCCTCTGCCTAA |
| MYB-F | GCGAACAAACTGCTGCTCAA |
| MYB-R | TGATTTGCTGCTGTCACCCT |
新窗口打开|下载CSV
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 3]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
