0 引言
【研究意义】转基因动物技术是一种行之有效的快速动物育种新技术,可用于高附加值[1,2]、环境友好[3]、生产性能[4]或产品品质改良[5]以及抗病[6]等的动物新品种培育。目前转基因动物制作所获得的转基因动物,外源基因多以多拷贝串联方式随机整合到受体动物基因组中。这就使得目前转基因动物制备中存在导致内源基因表达异常[7]、外源基因异位表达[8,9]、外源基因表达及遗传稳定性差[10]且外源基因的表达水平缺乏可预见性和可控性[11]等问题。这些因素一直制约着转基因动物的研究与应用,探讨解决上述问题的有效方法是推动转基因动物技术进步并有效服务于人类的重要前提。基因打靶技术[12]能够将外源基因精确的插入到受体基因组中的特定位点。探讨通过基因打靶技术将外源基因定点插入到内源基因的开放阅读框的上游或者下游,通过融合肽2A序列[13]连接,使外源基因与内源基因融合表达,是解决上述问题的思路之一。早期的基因打靶技术因为效率低下 [14],无法实现上述目标。对基因打靶机理的研究表明,在特定位点高效实现DNA双链断裂能大大提高了基因打靶的效率[15]。近年来一些人工核酸酶技术如:锌指核酸酶[16,17]、类转录激活因子效应物核酸酶 [18,19],和CRISPR/Cas 系统[20,21]等的出现。使得人们可以在基因组的任意位点高效产生DNA双链断裂而使得基因打靶效率得到了显著提高[22],这使得上述思路的实现成为可能。然而这些新的技术的出现与应用虽然使得基因打靶效率获得了显著提高但仍然较低。在大多数情况下利用这些技术进行基因打靶仍然需要使用报告基因来有效富集阳性克隆以提高筛选效率[23,24]。而从生物安全角度考虑在获得了同源重组的转基因细胞后需要将报告基因表达结构删除。Cre/Loxp系统是目前应用较为广泛的报告基因删除系统[25,26,27]。【前人研究进展】Cre/loxp系统是1981年人们从P1噬菌体及其宿主细胞中发现的重组酶系统[28]。loxp序列是由34个碱基对构成的一个反向重复序列。Cre酶能够对该序列进行识别和加工。如果在一段序列的两侧各有一个loxp序列且方向相同,则在Cre酶作用下位于两个loxp序列之间的DNA就会被从原来的DNA分子中切下,也就是删除。人们应用这一原理建立了基于Cre/loxp的删除系统,并应用于报告基因的删除以及在基因功能研究中用于条件敲除内源基因,来阐明该基因在特定组织细胞类型或特定发育时期的功能[29,30]。Cre/loxp系统用于报告基因或作为条件敲除内源基因时loxp序列都是位于报告基因或欲敲除基因完整表达结构的两侧,已有的研究表明这种结构对报告基因或欲敲除的内源基因的表达没有影响。【本研究切入点】而上述希望通过基因打靶技术将外源基因与内源基因通过2A序列链接实现共表达的设想中会将loxp序列留置于基因开放阅读框的上游或下游的非翻译区中。而loxp序列已如前述是一种反向重复序列,其出现在基因开放阅读框的上游或下游的非翻译区中时,将在转录产生的mRNA上游或下游非翻译区中形成发卡型二级结构,而这种二级结构的形成有可能对mRNA的翻译效率产生影响,而且loxp位点的位置不同可能影响程度有所不同,阐明这一效应对开展上述思路的研究中打靶位点的选择具有重要意义,而目前还缺乏这方面的相关研究报道。【拟解决的关键问题】以增强的绿色荧光蛋白(egfp)基因为试验基因,探究loxp序列在egfp基因开放阅读框的上游或下游的非翻译区序列中对基因表达的影响,并进一步以植酸酶基因为试验基因对以egfp基因为试验基因得到的结果进行进一步验证,为上述思路的研究中打靶位点的选择提供参考依据。1 材料与方法
1.1 材料
1.1.1 细胞系、菌株、载体 猪肾细胞系PK15(ATCC,美国);大肠杆菌(Escherichia coli)DH5α(TaKaRa, 中国大连);质粒pEGFP-N2(Clonth,美国)、pIREsNeo(Clonth,美国)、pGL4.13[luc2/SV40](Promaga, 美国)、pGEM-5zf-loxp(河北农业大学动物科技学院实验室构建)和pDsRed2-N1(TaKaRa, 中国大连)等由河北农业大学动物科技学院实验室保存。
1.1.2 试剂 无内毒质粒DNA提取试剂盒QIAGEN Plasmid Mini Kit购自QIAGEN(德国);限制性内切酶、DNA 分子量标准 1kb ladder marker、DNA连接酶等购自Fermentas(美国);LipofectamineTM 2000脂质体转染试剂购自Invitrogen(美国);胎牛血清、DMEM、0.25%胰酶购自GIBCO(美国);无水乙酸钠、植酸钠购自Sigma公司(德国),其他试剂均为国产,萤火虫萤光素酶报告基因检测试剂盒购自碧云天生物技术公司(中国);DNA测序工作由上海生工生物工程有限公司完成。
1.2 试验方法
1.2.1 ploxp-EGFP、pEGFP-loxp、ploxp-EGFP--loxp质粒的构建 试验于2017年3月于河北农业大学动物遗传育种与繁殖实验室进行。以pEGFP-N2、pGEM-5zf-loxp质粒为骨架质粒,通过一系列酶切连接反应构建所需质粒。这些质粒的结构见图1。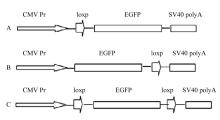 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1loxp位于试验基因1(egfp基因)开放阅读框的不同位置的试验质粒结构模式图
A为ploxp-EGFP(loxp序列位于egfp基因开放阅读框中kozak上游的5′非翻译区); B为pEGFP-loxp(loxp序列位于egfp基因开放阅读框终止密码子下游3′非翻译区);C为ploxp-EGFP—loxp(egfp基因开放阅读框的Kozak序列上游5′非翻译区和终止密码子下游3′非翻译区各有一个loxp序列)
-->Fig. 1The map of the experimental plasmid with different location of the loxp sequence at the open reading frame of the experimental gene1 (egfp gene)
A is the plasmid ploxp-EGFP (The loxp sequence located at the upstream of the Kozak sequence in the 5′ un-translation region of egfp gene); B is the plasmid pEGFP-loxp (The loxp sequence located at the downstream of the termination codon in the 3′un-translation region of egfp gene); C is the plasmid ploxp-EGFP—loxp (There is one loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region and another downstream of the termination codon in the 3′un-translation region of egfp gene)
-->
1.2.2 pSV40-luciferase-CMV-loxp-phytase、pSV40- luciferase-CMV-loxp-phytase-lox、p-SV40- luciferase-CMV-phytase-loxp和pSV40-luciferase- CMV-phytase质粒的构建 试验于2018年1月于河北农业大学动物遗传育种与繁殖实验室进行。以pIREsNeo、pGEM-5zf-loxp、pT-phytase、pGL4.13[luc2/ SV40]为基础通过一系列酶切连接反应构建了这些质粒。这些质粒的结构见图2。
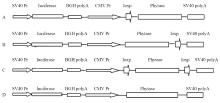 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2loxp位于试验基因2(phytase基因)开放阅读框的不同位置的试验质粒结构模式图
A为pSV40-luciferase-CMV-loxp-phytase(loxp序列位于phytase基因开放阅读框中kozak上游的5′非翻译区); B为p-SV40-luciferase-CMV-phytase-loxp(loxp序列位于phytase基因开放阅读框终止密码子下游3′非翻译区);C为pSV40-luciferase-CMV-loxp-phytase-lox(phytase基因开放阅读框的Kozak序列上游5′非翻译区和终止密码子下游3′非翻译区各有一个loxp序列);D为pSV40-luciferase-CMV-phytase(phytase基因上下游非翻译区序列中均添加loxp序列)
-->Fig. 2The map of the experimental plasmid with different location of the loxp sequence at the open reading frame of the experimental gene2 (phytase gene)
A is the plasmid pSV40-luciferase-CMV-loxp-phytase (The loxp sequence located at the upstream of the Kozak sequence in the 5′ un-translation region of phytase gene); B is the plasmid p-SV40-luciferase-CMV-phytase-loxp (The loxp sequence located at the downstream of the termination codon in the 3′un-translation region of phytase gene); C is the plasmid pSV40-luciferase-CMV-loxp-phytase-lox (There is one loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region and another downstream of the termination codon in the 3′un-translation region of phytase gene); D is the plasmid pSV40-luciferase-CMV-phytase (There is no loxp sequence located at the up and downstream of the5′ and 3′-UTR of phytase gene)
-->
1.2.3 转染用质粒DNA提取 各质粒DNA提取按QIAGEN Plasmid Mini Kit 质粒提取试剂盒说明书进行,所提取质粒电泳结果见图3.
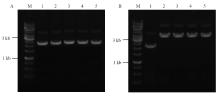 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3转染用质粒电泳检查
A 为试验基因1(egfp基因)系列质粒电泳结果 M为1kb ladder 分子量标准;1为pEGFP-N2质粒;2为ploxp-EGFP质粒, 3为ploxp-EGFP-loxp质粒,4为pEGFP-loxp质粒, 5为pDsRed2-N1质粒;B为试验基因2(phytase基因)系列质粒电泳结果,其中M为1kb ladder 分子量标准,1为pEGFP-N2质粒, 2为pSV40-luciferase-CMV-loxp-phytase质粒, 3为pSV40-luciferase-CMV-loxp-phytase-loxp质粒,4为pSV40-luciferase-CMV- phytase-loxp质粒, 5为pSV40-luciferase-CMV-phytase质粒
-->Fig. 3The electrophoresis results of the plasmid used in the transfection experiment
A is the electrophoresis results of the set of plasmids of the experimental gene 1( egfp gene). M is the 1kb ladder DNA molecular weight marker, 1 is the plasmid of pEGFP-N2;2 is the plasmid of ploxp-EGFP; 3 is the plasmid of ploxp-EGFP-loxp;4 is the plasmid of pEGFP-loxp; 5 is the plasmid of pDsRed2-N1; B is the electrophoresis results of the set of plasmids of experimental gene 2(phytase gene). M is the 1kb ladder DNA molecular weight marker, 1 is the plasmid of pEGFP-N2;2 is the plasmid of pSV40-luciferase-CMV-loxp-phytase; 3 is the plasmid of pSV40-luciferase-CMV-loxp-phytase-loxp; 4 is the plasmid of pSV40-luciferase-CMV -phytase-loxp; 5 is the plasmid of pSV40-luciferase-CMV -phytase
-->
1.2.4 细胞培养与转染 猪肾细胞系PK15在37℃,5% CO2条件下和含10%新生牛血清的DMEM培养基中常规方法进行培养和传代。
试验基因1 (egfp基因)相关结构转染试验:试验于2017年4月于河北农业大学动物遗传育种与繁殖实验室进行。在转染前24 h接种1×105个/mL指数生长期的细胞于24孔板中。培养24 h后细胞汇合度达90%时进行脂质体转染。每孔转染质粒800 ng,其中包括对照质粒pEGFP-N2或每种构建的试验质粒(plxop-EGFP、pEGFP-loxp、ploxp-EGFP-loxp)400 ng和pDsRed2-N1内参质粒400 ng,每组转染设置3个重复,转染24 h后将各转染组置于倒置显微镜下观察各孔内参基因和试验基因1表达情况。
试验基因2 phytase基因相关转染试验:试验于2018年3月于河北农业大学动物遗传育种与繁殖试验室进行。在转染前24 h接种1×105个/mL指数生长期的细胞于24孔板中。培养24 h后细胞汇合度达90%时进行脂质体转染。每孔转染质粒800 ng,其中每种构建的试验质粒(pSV40-luciferase-CMV-loxp-phytase、pSV40- luciferase-CMV-loxp-phytase-lox、p-SV40-luciferase-CMV- phytase-loxp和pSV40-luciferase-CMV- phytase)各400 ng和pEGFP-N2内参质粒400 ng,另以400 ng pDsRed2- N1+400 ng pEGFP-N2的共转染细胞为酶活性空白对照,每组转染设置3个重复,转染48 h后将各转染组置于倒置显微镜下观察各孔转染内参egfp基因表达情况,并制备酶粗体液做酶活性测定。
1.2.5 试验基因1 egfp基因转染结果检测与分析 试验于2017年4月于河北农业大学动物遗传育种与繁殖实验室进行。
1.2.5.1 荧光显微镜观察荧光蛋白基因表达情况 转染细胞前在细胞培养板上用记号笔对每个孔进行划线均分为6个区域,转染24 h后在荧光显微镜下观察并记录所分区域,对各区域egfp与dsred2基因表达情况以及明视野中细胞照相并用Image J软件分析光密度值。整理数据,进行统计学分析。
1.2.5.2 结果的统计与分析 提取每孔中不同区域的红色荧光和绿色荧光的光密度值后,依据所得数据应用SPSS 19.0 统计软件进行一维方差分析,分析方法采用Tukey法,对每转染组的三个重复间以及不同转染组间的两种荧光强度的差异显著性进行分析,来确认loxp序列的位置对外源基因表达的影响。
1.2.6 试验基因2(phytase基因)转染结果检测与分析 本试验于2018年3月于河北农业大学动物遗传育种与繁殖实验室进行。
1.2.6.1 荧光显微镜观察绿色荧光蛋白基因表达情况 转染细胞前在细胞培养板上用记号笔对每个孔进行划线均分为6个区域,转染48 h后在荧光显微镜下观察并记录所分区域,对各区域egfp基因表达情况以及明视野中细胞照相并用Image J软件分析光密度值。整理数据,进行统计学分析。
提取每孔中不同区域的绿色荧光光密度值后,依据所得数据应用SPSS 19.0 统计软件进行一维方差分析,分析方法采用Tukey法,对每转染组的三个重复间以及不同转染组间的绿色荧光强度的差异显著性进行分析,来确定各转染间是否存在因转染质粒纯度、细胞活性以及转染操作带来的误差。
1.2.6.2 表达内参基因和试验基因2(phytase基因)表达情况水平分析 转染48 h后,将细胞版中的待测细胞用PBS(pH7.3)洗一次,去除残余液体,加入细胞裂解液200 µL,室温裂解20 min,将细胞裂解物移至1.5 mL离心管中,12 000 r/min 4℃离心5 min去除细胞碎片,称为酶粗提液,然后用酶粗提液测定荧光素酶和植酸酶的活性。
荧光素酶活性测定:参考试剂盒说明书进行,大致过程为:取20 µL酶粗提液,加入底物100 µL,使用LKB公司的化学发光仪Wallac 1250 Luminoneter,输出值为毫伏(mV),测定10秒的发光强度(代表试样的荧光素酶活性)。
植酸酶基因活性测定:采用钼酸显色法进行测定,主要参照程海娜等[31,32]方法进行。颜色/终点混合液:250 mL钼酸铵(100 g·L-1)+ 250 mL钒酸铵(2.35 g·L-1)+ 165 mL 65%硝酸+335 mL水。取100 µL酶粗提液加入100 µL 1. 25 mol·L-1乙酸缓冲液(pH 5. 5)进行稀释,同时设置一个空白对照,即200 µL 1. 25 mol·L-1乙酸缓冲液(pH 5.5)替代酶粗提液稀释液,与其他试样同时进入后续步骤。向稀释的酶粗提液和对照样中加入0.6 mL 1.25 mol·L-1乙酸缓冲液(pH 5.5),混匀,在37℃保温5 min,然后加入1.6 mL的8.4 g·L-1植酸钠,继续保温 30 min,加入颜色/终点混合液 1.6 mL,以空白对照校正,采用分光光度计在415 nm波长下测定吸光值,记录每试样的吸光值(代表试样植酸酶活性)。
2 结果
2.1 转染用质粒DNA提取
图3显示所提取的转染用质粒质量一致性很好,质粒质量引起的各转染间产生的差异基本可以忽略。2.2 试验基因1(egfp基因)试验
2.2.1 定性分析 图4为对照质粒及不同结构的试验基因1质粒与内参质粒共转染后绿色荧光蛋白和红色荧光蛋白基因表达情况。结果显示,不同转染组中红色荧光蛋白表达基本一致,表明各不同转染组间因细胞活性、转染质粒纯度及转染操作产生的误差不显著,在此情况下,不同loxp位置的质粒的绿色荧光蛋白表达情况不完全一致,pEGFP-loxp与pEGFP-N2转染组的绿色荧光蛋白表达强度基本一致,ploxp- EGFP、ploxp-EGFP-loxp转染组绿色荧光蛋白表达要比pEGFP-N2转染组绿色荧光蛋白表达强度明显低,这一结果表明,loxp序列在基因开放阅读框的下游对基因表达基本无影响,而loxp序列在基因开放阅读框上游时对基因表达呈显著负影响。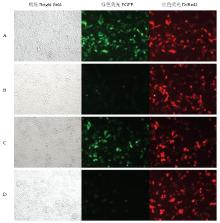 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4对照质粒及不同结构的试验基因1(egfp基因)试验质粒与内参质粒共转染后代表性区域绿色荧光蛋白和红色荧光蛋白基因表达
A.为对照质粒pEGFP-N2与内参质粒pDsRed2-N1共转染;B为试验质粒ploxp-EGFP与内参质粒pDsRed2-N1共转染;C为试验质粒pEGFP-loxp与内参质粒pDsRed2-N1共转染;D为试验质粒ploxp-EGFP-loxp与内参质粒pDsRed2-N1质粒共转染
-->Fig. 4The expression of the egfp gene and dsred2 gene in the representative area after co-transfection of the control plasmid or the constructed experimental gene 1 (egfp gene) experimental plasmids with the internal reference plasmid pDsRed2-N1
A is the result of co-transfection of control plasmid pEGFP-N2 and internal reference plasmid pDsRed2-N1;B is the result of co-transfection of experimental plasmid ploxp-EGFP and internal reference pDsRed2-N1; C is the result of co-transfection of experimental plasmid pEGFP-loxp and internal reference pDsRed2-N1;D is the result of co-transfection of experimental plasmid ploxp-EGFP-loxp and internal reference pDsRed2-N1
-->
2.2.2 egfp基因转染结果分析 图4结果只是对转染结果的定性分析,为了得到更为确切的结论,本研究应用Image J软件对所拍照片的荧光强度进行了提取,得到了每个区域的两种荧光的光密度值(表1)。
Table 1
表1
表1不同转染三个重复试验的不同区域的两种荧光光密度值
Table 1The density of the two fluorescent in different regions of the three repeat of different transfection
| 序号 Serial number | pEGFP-N2+Re | pEGFP-loxp+Re | ploxp-EGFP+Re | ploxp-EGFP-loxp+Re | ||||
|---|---|---|---|---|---|---|---|---|
| RP | GP | RP | GP | RP | GP | RP | GP | |
| 1.1 | 0.0533 | 0.0524 | 0.0541 | 0.0164 | 0.0535 | 0.0514 | 0.0522 | 0.0178 |
| 1.2 | 0.0515 | 0.0505 | 0.0526 | 0.0224 | 0.0479 | 0.0532 | 0.0500 | 0.0193 |
| 1.3 | 0.0494 | 0.0526 | 0.0500 | 0.0206 | 0.0486 | 0.0525 | 0.0474 | 0.0181 |
| 1.4 | 0.0492 | 0.0511 | 0.049 | 0.0206 | 0.0523 | 0.0544 | 0.0481 | 0.0178 |
| 1.5 | 0.0507 | 0.0497 | 0.0487 | 0.0164 | 0.0526 | 0.0518 | 0.0504 | 0.0171 |
| 1.6 | 0.0502 | 0.0515 | 0.0505 | 0.0175 | 0.0498 | 0.0487 | 0.0531 | 0.0209 |
| 2.1 | 0.0500 | 0.0520 | 0.0485 | 0.0183 | 0.0533 | 0.0530 | 0.0534 | 0.0170 |
| 2.2 | 0.0540 | 0.0526 | 0.0507 | 0.0162 | 0.0520 | 0.0470 | 0.0516 | 0.0181 |
| 2.3 | 0.0501 | 0.0492 | 0.0525 | 0.0204 | 0.0493 | 0.0525 | 0.0509 | 0.0165 |
| 2.4 | 0.0495 | 0.0508 | 0.0531 | 0.0209 | 0.0510 | 0.0505 | 0.0540 | 0.0186 |
| 2.5 | 0.0526 | 0.0530 | 0.0518 | 0.0166 | 0.0494 | 0.0485 | 0.0511 | 0.0170 |
| 2.6 | 0.0506 | 0.0495 | 0.0497 | 0.0162 | 0.0540 | 0.0557 | 0.0534 | 0.0207 |
| 3.1 | 0.0490 | 0.0514 | 0.0519 | 0.0183 | 0.0512 | 0.0489 | 0.0527 | 0.0168 |
| 3.2 | 0.0481 | 0.0530 | 0.049 | 0.0164 | 0.0499 | 0.0516 | 0.0490 | 0.0196 |
| 3.3 | 0.0514 | 0.0518 | 0.0498 | 0.0171 | 0.0481 | 0.0543 | 0.0481 | 0.0172 |
| 3.4 | 0.0544 | 0.0525 | 0.0519 | 0.0214 | 0.0530 | 0.0502 | 0.0517 | 0.0175 |
| 3.5 | 0.0542 | 0.0496 | 0.0528 | 0.0166 | 0.0509 | 0.0513 | 0.0486 | 0.0186 |
| 3.6 | 0.0501 | 0.0500 | 0.0538 | 0.0180 | 0.0523 | 0.0499 | 0.0553 | 0.0199 |
新窗口打开
依据表1中的数据应用SPSS 19.0 统计软件进一步对每组转染三个重复间及不同转染组间绿色荧光蛋白和红色荧光蛋白表达的差异显著性进行分析(表2)。
Table 2
表2
表2荧光蛋白平均光密度值差异显著性分析
Table 2The results of significance test of the average fluorescent density value of fluorescent protein
| 转染质粒 Transfection plasmid | 平行1 Parallel 1 | 平行2 Parallel 2 | 平行3 Parallel 3 | |||
|---|---|---|---|---|---|---|
| GP | RP | GP | RP | GP | RP | |
| pEGFP-N2+Re | 0.05±0.001Aa | 0.05±0.002Aa | 0.05±0.002Aa | 0.05±0.002Aa | 0.05±0.001Aa | 0.05±0.003Aa |
| pEGFP-loxp+Re | 0.02±0.003Bb | 0.05±0.002Aa | 0.02±0.002Bb | 0.05±0.002Aa | 0.02±0.002Bb | 0.05±0.002Aa |
| ploxp-EGFP+Re | 0.05±0.002Aa | 0.05±0.002Aa | 0.05±0.003Aa | 0.05±0.002Aa | 0.05±0.002Aa | 0.05±0.002Aa |
| ploxp-EGFP-loxp+Re | 0.02±0.001Bb | 0.05±0.002Aa | 0.02±0.002Bb | 0.05±0.001Aa | 0.02±0.001Bb | 0.05±0.003Aa |
新窗口打开
由表2可看出,转染孔中红色荧光蛋白平均光密度值组内与组间均差异不显著,排除了转染过程中试验操作误差或不同质粒纯度以及细胞活性差异等对试验结果造成的影响。而转染构建质粒表达的绿色荧光蛋白平均光密度值在4个转染组中不完全相同,转染pEGFP- loxp与pEGFP-N2质粒中绿色荧光蛋白平均光密度值差异不显著(P<0.05,P<0.01),转染ploxp-EGFP、ploxp-EGFP-loxp质粒中绿色荧光蛋白平均光密度值与转染pEGFP-N2质粒中绿色荧光蛋白平均光密度值有极显著差异(P>0.01),表明在转染条件一致的情况下,loxp序列在基因下游位置对基因表达影响不显著,loxp序列在基因上游对基因表达影响极显著,且为负影响。
2.3 试验基因2(phytase基因)转染分析
2.3.1 转染内参基因egfp表达分析 从图5的定性分析结果显示各转染间转染内参egfp基因表达水平差异并不明显,初步显示质粒纯度、细胞活性以及转染操作等误差对试验结果没有显著影响。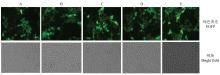 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5不同结构的phytase基因试验质粒与转染内参质粒pEGFP-N2共转染后代表性区域绿色荧光蛋白表达情况
A.为表达对照质粒pSV40-luciferase-CMV -phytase与转染内参质粒pEGFP-N2共转染结果;B为试验质粒pSV40-luciferase-CMV-loxp-phytase与内参质粒pEGFP-N2共转染结果;C为试验质粒pSV40-luciferase-CMV -phytase-loxp与内参质粒pEGFP-N2共转染结果;D为试验质粒pSV40-luciferase-CMV-loxp-phytase-loxp与内参质粒pEGFP-N2质粒共转染结果;E为pDsRed2-N1与pEGFP-N2共转染作为试验基因phytase和表达内参基因luciferase的阴性对照
-->Fig. 5The expression of the egfp gene in the representative area after co-transfection of the constructed phytase gene experimental plasmids with the transfection internal reference plasmid pEGFP-N2
A is the result of co-transfection of expression control plasmid pSV40-luciferase-CMV -phytase and transfection internal reference plasmid pEGFP-N2;B is the result of co-transfection of experimental plasmid pSV40-luciferase-CMV-loxp-phytase and internal reference pEGFP-N2; C is the result of co-transfection of experimental plasmid pSV40-luciferase-CMV-phytase-loxp and internal reference pEGFP-N2;D is the result of co-transfection of experimental plasmid pSV40-luciferase-CMV-loxp-phytase-loxp and internal reference pEGFP-N2; E is the result of co-transfection of plasmid pDsRed2-N1 and pEGFP-N2 as experimental gene (phytase) and expression interal reference gene (luciferase) expression negative control
-->
依据表3中的数据应用SPSS 19.0 统计软件进一步对每组转染三个重复间及不同转染组间绿色荧光蛋白表达的差异显著性进行分析(表4)。表3和表4的分析表明不同转染间绿色荧光蛋白表达水平差异不显著,从这一分析结果可以认为各转染间由于质粒的纯度、细胞活性差异以及转染操作带来的误差很小,不影响最终得出结论的准确性。
Table 3
表3
表3不同转染三个重复试验的不同区域的绿色荧光光密度值
Table 3The density of the green fluorescent in different regions of the three repeat of different transfection
| 序号 Serial number | A | B | C | D | E |
|---|---|---|---|---|---|
| 1.1 | 0.0633 | 0.0621 | 0.0597 | 0.0612 | 0.0642 |
| 1.2 | 0.0595 | 0.0636 | 0.0612 | 0.0631 | 0.0575 |
| 1.3 | 0.0594 | 0.0609 | 0.0623 | 0.0597 | 0.0624 |
| 1.4 | 0.0622 | 0.0619 | 0.0598 | 0.0593 | 0.0622 |
| 1.5 | 0.0607 | 0.0592 | 0.0602 | 0.0623 | 0.0615 |
| 1.6 | 0.0602 | 0.0603 | 0.0652 | 0.0622 | 0.0602 |
| 2.1 | 0.0597 | 0.0587 | 0.0593 | 0.0631 | 0.0621 |
| 2.2 | 0.0613 | 0.0597 | 0.0578 | 0.0620 | 0.0623 |
| 2.3 | 0.0621 | 0.0625 | 0.0602 | 0.0602 | 0.0631 |
| 2.4 | 0.0595 | 0.0621 | 0.0617 | 0.0611 | 0.0612 |
| 2.5 | 0.0626 | 0.0638 | 0.0597 | 0.0631 | 0.0596 |
| 2.6 | 0.0606 | 0.0594 | 0.0623 | 0.0643 | 0.0622 |
| 3.1 | 0.0596 | 0.0619 | 0.0611 | 0.0621 | 0.0611 |
| 3.2 | 0.0611 | 0.0627 | 0.0592 | 0.0592 | 0.0598 |
| 3.3 | 0.0634 | 0.0618 | 0.0597 | 0.0587 | 0.0602 |
| 3.4 | 0.0614 | 0.0599 | 0.0621 | 0.0613 | 0.0631 |
| 3.5 | 0.0588 | 0.0578 | 0.0641 | 0.0593 | 0.0604 |
| 3.6 | 0.0621 | 0.0612 | 0.0593 | 0.0653 | 0.0610 |
新窗口打开
Table 4
表4
表4绿色荧光蛋白平均光密度值差异显著性分析
Table 4The results of significance test of the average green fluorescent density value of green fluorescent protein
| 转染质粒 Transfection plasmid | 平行1 Parallel 1 | 平行2 Parallel 2 | 平行3 Parallel 3 |
|---|---|---|---|
| pEGFP-N2+pSV40-luciferase-CMV-phytase | 0.06±0.002Aa | 0.06±0.001Aa | 0.06±0.002Aa |
| pEGFP-N2+pSV40-luciferase-CMV-loxp-phytase | 0.06±0.002Aa | 0.06±0.002Aa | 0.06±0.002Aa |
| pEGFP-N2+:pSV40-luciferase-CMV-phytase-loxp | 0.06±0.002Aa | 0.06±0.002Aa | 0.06±0.002Aa |
| pEGFP-N2+ pSV40-luciferase-CMV-loxp-phytase-loxp | 0.06±0.002Aa | 0.06±0.001Aa | 0.06±0.002Aa |
新窗口打开
2.3.2 表达内参基因luciferase和试验基因phytase表达分析
2.3.2.1 表达内参基因luciferase表达测定结果从表5的分析可以看出,除了空白对照组(pDsRed2-N1+ pEGFP-N2)外其他各转染的不同转染间荧光素酶的活性差异不显著(P>0.05),而空白对照的三个重复间差异也不显著(P>0.05),这一结果进一步说明各转染间因质粒质量、细胞活性以及和转染误差以及酶活测定中的操作误差造成的差异很小可以忽略。
Table 5
表5
表5不同转染三个重复试验的荧光素酶活性(mV)测定及统计分析
Table 5The density of the activity of luciferase(mV) of the three repeat of every transfection
| 序号Serial number | A | B | C | D | E |
|---|---|---|---|---|---|
| 1. | 1006.34 | 995.33 | 995.66 | 1019.03 | 30.11 |
| 2 | 996.89 | 1020.11 | 1016.22 | 998.77 | 32.13 |
| 3 | 1017.26 | 1008.32 | 1003.12 | 1006.02 | 29.22 |
| 均值 Average | 1011.80±7.72Aa | 1007.92±12.39Aa | 1005.00±10.41Aa | 1007.94±10.27Aa | 30.49±1.49Bb |
新窗口打开
2.3.2.2 试验基因1 phytase表达测定结果 表6的结果显示试验质粒的转染间植酸酶活性存在差异,试验质粒pSV40-luciferase-CMV-phytase和试验质粒pSV40-luciferase-CMV-phytase-loxp之间植酸酶活性差异不显著,试验质粒pSV40-luciferase-CMV-loxp -phytase和试验质粒pSV40-luciferase-CMV-loxp- phytase-loxp之间差异不显著,而试验质粒pSV40- luciferase-CMV-phytase、pSV40-luciferase-CMV-phytase- loxp与试验质粒pSV40-luciferase-CMV-loxp-phytase、pSV40- luciferase-CMV-loxp-phytase-loxp两两间差极异显著(P<0.01),而所有试验质粒植酸酶活性与空白对照间差异都极显著(P<0.01)综合表5和表6的结果进一步证实了试验基因1(egfp基因)试验所得出的结论,即loxp序列位于外源基因Kozak序列上游5′非翻译区时对外源基因表达具有负影响,而只在外源基因的3′端终止密码子下游时对外源基因的表达无显著影响。
Table 6
表6
表6不同转染三个重复试验的植酸酶活性(吸光值)测定及统计分析
Table 6The density of the activity of phytase (absorbance value) of the three repeat of every transfection
| 序号Serial number | A | B | C | D | E |
|---|---|---|---|---|---|
| 1. | 0.39 | 0.13 | 0.41 | 0.14 | 0.02 |
| 2 | 0,44 | 0.15 | 0.36 | 0.10 | 0.03 |
| 3 | 0.36 | 0.11 | 0.39 | 0.12 | 0.02 |
| 均值 Average | 0.40±0.0016Ab | 0.13±0.0004aB | 0.39±0.0006Ab | 0.12±0.0004aB | 0.02±0.01ab |
新窗口打开
3 讨论
mRNA作为蛋白质合成的模板指导蛋白质的合成。虽然mRNA为单链分子,但由于其局部互补配对在生理条件下可互补配对形成二级结构,这种二级结构的形成对mRNA 指导蛋白质的合成效率的影响是多方面的。首先,在mRNA指导蛋白质合成过程中需要打开这些二级结构才能阅读密码子进行正常翻译,因此大量的二级结构的存在势必会减慢蛋白质合成的速度。尤其是在mRNA的5′非翻译区大量的RNA二级结构的存在会影响蛋白质合成的起始,5′非翻译区的二级结构对蛋白质合成效率的影响可能更为强烈[33]。对酵母全基因组水平的研究表明,起始密码子周围-40—+40 nt范围内高含量的二级结构对基因翻译的确存在一定的抑制作用,同时mRNA 5′非翻译区及起始密码子下游40nt的序列中二级结构的存在不利于mRNA出核[34],而成熟的mRNA出核是其进行蛋白质合成前提条件。这一研究似乎表明mRNA 5′非翻译区中RNA二级结构的存在对蛋白质具有更多不利的影响,人们对小鼠[35]、斑马鱼[36]和拟南芥[37]的研究也发现了同样的规律,即多数基因5′非翻译区二级结构相对含量比较低,这表明这一现象在进化上具有一定的保守性,而mRNA3′非翻译区在酵母和拟南芥中二级结构含量相对较低,而在小鼠、斑马鱼中却相对较高这似乎说明mRNA 3′非翻译区的二级结构含量保守性较低,因此对基因表达效率的影响并不重要。然而对小鼠胚胎干细胞mRNA二级结构的研究[38],却发现这种细胞类型中mRNA的5′非翻译区和3′非翻译区的二级结构含量都相对较高,这可能与小鼠胚胎干细胞中表达的有些基因5′非翻译区中某些二级结构有特殊的生物学功能如屏蔽micoRNA作用位点等有关。因此从上述分析中可知在一个基因的5′或(和)3′非翻译区添加一个反向重复序列其效应需要进行评价。Cre/loxp删除系统中loxp序列基本结构为长34 bp,中间是8 bp的核心序列,两边是13 bp的反向重复序列。当Cre 重组酶识别 loxp 位点后,可以在该位置切断 DNA,并在断端形成中间体,在 Cre 重组酶催化下可重组连接。因此,同一条 DNA 分子上若有两个loxp位点,且方向相同,重组后则loxp位点间的核酸序列被特异性删除,并在原位点留置一个loxp序列,如果以Cre/loxp系统作为报告基因删除系统通过基因打靶将外源基因定点整合到内源基因的上游或下游并通过2A序列链接形成共表达结构,会在融合表达结构的上游或(和)下游非翻译区留置一个loxp序列。当基因表达时转录产生的mRNA分子中上游或(和)下游会存在一个loxp序列,该序列会形成一个13 bp长的发卡型二级结构,该二级结构对外源基因的影响需要进行评估以便为上述设想的研究中打靶位点的选择提供依据。
本研究先选择egfp基因作为试验基因1,开展了研究结果初步显示loxp序列位于外源基因上游时对基因表达形成了强烈的抑制作用,而在外源基因下游时对外源基因的表达没有显著的影响,为了进一步证实试验基因1(egfp基因)的结果,笔者选用phytase基因作为试验基因2进一步验证试验基因1的结果,试验基因2(phytase基因)的结果和试验基因1(egfp基因)的结果基本一致。据此可以认为loxp序列位于基因上游时对基因表达有强烈的抑制作用,而在外源基因终止密码子下游时对外源基因的表达没有显著的影响。
分析产生这一现象的原因在于Cre/loxp结构是感染大肠杆菌的P1噬菌体的基因和序列,loxp序列在真核生物细胞基因组中并不存在,因此真核细胞中不会有针对loxp序列的相应的调控因子,因此loxp经转录出现在成熟mRNA的5′非翻译区时,不会有类似屏蔽micoRNA结合位点或被其他反式调控因子识别增强蛋白质翻译的作用,其自身形成的发卡结构会使核糖体的翻译效率下降甚至提前脱落,而因此其作用只有抑制了核糖体的翻译起始过程,从而导致外源基因表达下降;而loxp序列在基因开放阅读框下游的3′非翻译区内时其序列形成的发卡结构在外源基因终止密码子之后,即核糖体在翻译完成后才遇到相应的二级结构,因此该二级结构对蛋白翻译效率没有明显影响。这一结果也和前人对真核生物mRNA二级结构对基因表达的影响的研究结果基本一致。
4 结论
在开展通过基因打靶技术将外源基因与内源基因通过2A序列连接,实现外源基因和内源基因共表达的转基因动物制作研究中,以Cre/loxp系统作为报告基因删除工具时,则内源基因的下游为最佳的打靶位点。The authors have declared that no competing interests exist.
