0 引言
【研究意义】桃(Prunus persica)原产中国,是我国栽培最为广泛的落叶果树之一,仅次于苹果、梨和葡萄[1]。桃基因组较小,染色体数量少,单基因控制性状较多,被认为是多年生果树研究的模式植物[2]。随着分子标记技术在桃育种工作中的应用,大规模样本DNA提取操作过程复杂、耗时长、成本高等问题突出,而基因分型、分子标记辅助选择、品种鉴定等工作对DNA的浓度和产量要求不高[3],不需要DNA样品长时间保存。因此,寻找一种简单、快速、高通量、成本低的DNA提取方法非常必要。【前人研究进展】全基因组测序的完成为桃优良性状基因定位、遗传多样性分析、资源评价及分子辅助选种体系的建立等奠定了基础[4,5,6,7]。随着植物科学向分子水平进一步发展,利用分子标记进行定位和克隆控制桃优良性状的基因、分子标记选择育种、大规模杂交后代单株基因型鉴定等成为桃分子育种的重要环节[8],而该类分子生物学和遗传学研究都需大批量DNA以应用于下游生物学反应[9,10,11]。目前,快速高通量提取DNA的方法在作物中的应用已有许多报道,RANDHAWA等[12]将小麦DNA高通量提取方法成功应用于分子标记辅助选择育种;XIN等[13]结合CTAB与DNA提取试剂盒的方法提取高粱叶片与种子中的基因组DNA;DEVI等[14]报道了适用于PCR扩增等下游生物学研究的生姜基因组DNA快速提取方法。在木本果树中高通量提取DNA的研究与应用较少,KIM等[15]最先通过改良已有的4种DNA提取方法,报道了苹果、梨、葡萄及柿子的DNA快速提取方法,可获得高质量的DNA;CHENG等[16]以柑橘为研究材料,报道了可应用于20多种热带和亚热带果树作物基因组DNA快速提取的方法。关于桃DNA快速DNA提取的研究虽已有报道,但仅提高DNA提取过程中的研磨速度,操作过程复杂,无法满足高通量的需求[17]。【本研究切入点】实验室中常用的传统DNA提取方法,如CTAB或SDS等,操作步骤复杂,耗时耗力,难以适应科研快速发展的需求[18]。虽然已经有专用的设备可以实现高通量大规模的DNA提取,但是对于普通实验室而言,构建一种简单、快速且低成本的中小通量的植物基因组DNA提取方法很有必要。【拟解决的关键问题】采用改良CTAB法,以1.2 mL八联排管代替2 mL离心管,建立一种高通量提取桃基因组DNA的有效方法,成功应用于基因分型,提高桃大群体基因型筛选和分子标记辅助选种的效率。1 材料与方法
试验于2017年在农业部果树育种技术重点实验室进行。1.1 材料
以‘中油桃8号’(standard type,ST)为母本,基因型为tssdtssd;‘09-1-112’ (temperature-sensitive semi-dwarf in Prunus persica,PpTssd)为父本,来源于‘SD9238’,基因型为TSSDtssd。手工去雄后人工授粉,获得F1杂交后代500株实生苗,其中PpTssd型246株,普通生长型254株,P=0.800,χ2=0.064,比例约为1﹕1,符合孟德尔遗传规律,采集各单株幼嫩叶片用于DNA提取。1.2 基因组DNA提取
采用CTAB[19]法提取桃叶片基因组DNA,略作修改(图1),具体如下:(1)取桃幼嫩叶片约30 mg,用镊子放入1.2 mL八联排离心管底部,每孔各加入1个5 mm钢珠(图1-A),放入96孔底座,为防止液氮进入离心管,确保离心管盖高于液氮液面,并充分冷冻;(2)手动摇晃数次,保证钢珠充分打碎样品,DNA自动研磨仪(上海领成生物科技有限公司)进行研磨,频率30 Hz,时间90 s(图1-B);(3)立即用镊子轻轻敲打离心管盖,使附着在管盖内壁的样品掉落,待离心管盖充分解冻后,于通风橱中开盖,各离心管盖朝上分别放于干净实验纸上;(4)用300 mL量程8通道移液器加入配制好的CTAB液600 μL(图1-C),放入60℃电热恒温鼓风干燥箱加热30 min,其间约每10 min轻摇匀;(4)称量配平,放入冷冻离心机(Eppendorf 5810 R)4℃条件下,4 000 r/min瞬时离心,将管盖及内壁液体离心至底部;加入氯仿和异戊醇混合液,体积比为24﹕1,直至1.2 mL的八联排离心管满载线,后缓慢颠倒混匀5 min,配平后,放入冷冻离心机(Eppendorf 5810 R)4℃条件下,4 000 r/min,离心10 min;(5)分别吸取上清液100 µL于两个200 µL PCR板中,其中一板加入等体积的无水乙醇,于-20℃冰箱1 h,称量配平,4℃条件下,4 000 r/min,离心10 min,板孔朝下,迅速弃上清液;另一板放入4℃冰箱,保存备用;(6)在带有沉淀的96孔PCR板中加入150 μL的70%的乙醇,4 000 r/min瞬时离心,洗涤沉淀两次,加入无水乙醇洗涤沉淀一次,用100 μL的8通道移液器(Eppendorf)吸除离心管底部剩余无水乙醇,更换吸头,自然晾干;(7)在室温下自然风干沉淀后,加入150 μL的0.1×TE溶解,同时加入0.5 μL的RNase(图1-D),37℃放置1 h,祛除RNA污染(长期保存在-20℃冰箱,常用则存于4℃冰箱),以用于后续研究。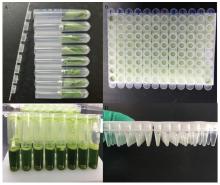 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1DNA提取主要步骤
A:取幼嫩叶片放入1.2 mL八联排管;B:研磨后的DNA样品;C:加入预热的CTAB;D:150 μL TE溶解DNA沉淀;
-->Fig. 1The major steps of DNA isolation protocol
A:Young leaves sampled into 1.2 mL thin-wall 8 strip polypropylene PCR tubes; B:Samples after grinding; C:DNA samples with preheated extraction buffer; D:DNA dissolved in 150 μL TE
-->
1.3 DNA质量和浓度检测
用1%琼脂糖凝胶电泳对提取的DNA片段大小和完整度进行检测,在紫外凝胶成像仪上观察并拍照。吸取1 μL DNA样品,采用NanoDrop 1000 spectrophotometer(Themo)紫外分光光度计检测DNA浓度和纯度(OD260mm/OD280mm),稀释成工作液浓度(约25 ng·μL-1),以用于后续研究。1.4 基于高分辨率熔解曲线(high resolution melting,HRM)的SNP基因分型
基于重测序数据开发与双亲基因型和表型紧密连锁的SNP标记,采用Primer 3.0软件(http://primer3. ut.ee/)设计引物,设置引物的退火温度60—63℃,扩增片段长度100—200 bp。采用HRM master mix(Roche)进行PCR扩增,反应总体积为15 μL,利用LightCycler 480II定量PCR仪(Roche)进行PCR扩增和HRM分析[20]。1.5 PCR扩增验证
参考桃基因组(Version 2.0)数据在亲本(‘中油桃8号’ב09-1-112’)中开发InDel标记。采用Primer 3.0软件(http://primer3. ut.ee/)设计引物,引物的退火温度60℃左右,扩增片段长度150—200 bp。采用TaKaRa EX Taq在F1群体中进行PCR扩增,反应总体积为15 μL,其中含DNA模板1 μL、酶0.075 μL、dNTP 1.2 μL、10×buffer(含Mg2+)1.5 μL、正/反向引物和ddH2O。扩增程序为95℃ 2 min;94℃ 15 s,56.5℃ 15 s,72℃ 20 s,30个循环;72℃ 4 min,4℃冷却10 min。取1.2 μL PCR产物进行8%聚丙烯酰胺凝胶电泳检测,硝酸银染色,氢氧化钠脱色后观察。2 结果
2.1 DNA浓度和纯度
经紫外分光光度计检测提取的DNA浓度和纯度,部分结果见表1。由表1 可知,OD260mm/OD280mm介于1.81—1.98,浓度介于25—200 ng·μL-1。RNA酶处理后,经琼脂糖凝胶电泳检测(图2),结果表明条带明亮清晰,整齐单一,DNA较完整。说明所提取的DNA 较纯,符合进一步研究分析要求。Table 1
表1
表1部分样品DNA纯度和浓度
Table 1UV absorbance ratios and DNA concentrations from partial peach samples
| 样品名称 Sample name | OD260nm/OD280nm | DNA浓度 DNA concentration (ng·μL-1) | 样品名称 Sample name | OD260nm/OD280nm | DNA浓度 DNA concentration (ng·μL-1) | |
|---|---|---|---|---|---|---|
| SD-A1 | 1.86 | 31.6 | SD-C1 | 1.84 | 61.2 | |
| SD-A2 | 1.92 | 35.8 | SD-C2 | 1.97 | 56.5 | |
| SD-A3 | 1.96 | 44.6 | SD-C3 | 1.87 | 66.7 | |
| SD-A4 | 1.83 | 29.5 | SD-C4 | 1.91 | 69.4 | |
| SD-A5 | 1.89 | 28.6 | SD-C5 | 1.96 | 63.6 | |
| SD-A6 | 1.91 | 44.9 | SD-C6 | 1.89 | 42.2 | |
| SD-A7 | 1.96 | 32.7 | SD-C7 | 1.84 | 100.7 | |
| SD-A8 | 1.91 | 28.1 | SD-C8 | 1.98 | 58.4 | |
| SD-A9 | 1.86 | 83.8 | SD-C9 | 1.89 | 25.5 | |
| SD-A10 | 1.98 | 32.7 | SD-C10 | 1.97 | 32.1 | |
| SD-A11 | 1.92 | 25.6 | SD-C11 | 1.87 | 25.3 | |
| SD-A12 | 1.87 | 26.8 | SD-C12 | 1.81 | 31.7 | |
| SD-B1 | 1.94 | 57.5 | SD-D1 | 1.97 | 118 | |
| SD-B2 | 1.92 | 38.4 | SD-D2 | 1.85 | 99.1 | |
| SD-B3 | 1.89 | 30.8 | SD-D3 | 1.89 | 73.5 | |
| SD-B4 | 1.87 | 25.3 | SD-D4 | 1.89 | 68.9 | |
| SD-B5 | 1.97 | 41.2 | SD-D5 | 1.87 | 86.0 | |
| SD-B6 | 1.91 | 27.7 | SD-D6 | 1.94 | 32.7 | |
| SD-B7 | 1.98 | 34.0 | SD-D7 | 1.85 | 67.2 | |
| SD-B8 | 1.82 | 95.0 | SD-D8 | 1.81 | 112.1 | |
| SD-B9 | 1.99 | 59.1 | SD-D9 | 1.84 | 100.5 | |
| SD-B10 | 1.93 | 25.6 | SD-D10 | 1.92 | 53.5 | |
| SD-B11 | 1.85 | 54.1 | SD-D11 | 1.90 | 80.8 | |
| SD-B12 | 1.84 | 36.8 | SD-D12 | 1.92 | 29.3 |
新窗口打开
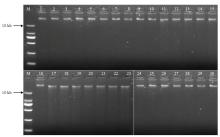 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2部分DNA样品琼脂糖凝胶电泳图
-->Fig. 2The 1% agarose gel electrophoresis of partial DNA samples
M: DNA Marker
-->
2.2 SNP基因分型检测
参考桃基因组(Version 2.0),基于双亲重测序数据获得引物SNP_Pp03_3758620,正向引物5′-GTGAA GTCCCACCAGTGCAG-3′,反向引物5′-TGGAGTCA GAGAGGATCGTCAA-3′,扩增片段长度171 bp;PpTssd型在该位点的基因型为A/G,普通生长型的基因型为G/G。基于HRM的基因分型结果发现,96个样品全部扩增成功,经计算后,PpTssd型与普通生长型呈现明显不同的峰型曲线(图3)。表明该方法提取的桃基因组DNA质量好、纯度高,适用于HRM基因分型。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3基于HRM分析PpTssd型A/G(蓝色)和普通型G/G(红色)位点的SNP鉴定
-->Fig. 3HRM analysis profile genotyping PpTssd type A/G (blue curve) and standard type G/G (red curve)
-->
2.3 PCR电泳检测
基于双亲‘中油桃8号’ב09-1-112’基因型开发InDel标记InDel_Pp03_3829009,缺失大小4 bp,采用Primer 3.0软件(http://primer3. ut.ee/)设计引物,正向引物:5′-AGCCCTGTATTGGTTCCATCCT-3′, 反向引物5′-AGAAGGTAGCGACTCCTTTTCCT-3′,扩增片段大小201 bp。从聚丙烯凝胶电泳分析图谱结果显示(图4),目的条带整齐清晰、分辨率高,PCR扩增结果良好,PpTssd型与ST型具有明显的多态性,基因型与表型相符,证明PCR扩增稳定,提取的DNA可用于基于InDel标记的基因型分析。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4PCR扩增片段聚丙烯酰胺凝胶电泳图
-->Fig. 4PCR amplification fragment profiling of polyacrylamide gel electrophoresis
-->
3 讨论
目前,已报道的植物DNA提取方法有几十种,如CTAB法、碱裂解法、高低盐pH法、苯酚法、试剂盒提取DNA法等。传统的DNA提取方法经过改良后,分离的DNA质量与纯度较高,满足了一般分子生物学实验所需DNA的质量要求[21],但其操作步骤繁琐,且需消耗大量人力和时间,达不到高通量的要求。一些简易DNA提取方法,虽简化了提取步骤,提取速度快,但分离DNA纯度不高,且扩增效果不稳定[22,23,24];许多DNA提取试剂盒虽然操作简单,但其成本较高,产量低,不适宜大规模高通量DNA制备[25,26,27,28]。本研究采用1.2 mL的八联排管结合改良CTAB法快速提取大批量桃叶片基因组DNA的方法,可以在3 min之内研磨192个样品,八联排管与8通道移液枪相结合,操作简便,每人每天可提取1 000个样品,节约时间,每个样品提取成本约为DNA提取试剂盒的1/10,极大提高了工作效率;分离的DNA产率大、纯度高,经检测可以满足InDel分析、PCR扩增及SNP基因分型等大规模高通量分子生物学研究。DNA提取过程中,首先应对1.2 mL的八联排离心管和管盖进行位置和方向标记,防止打乱样品顺序。用20 cm枪状镊子取幼嫩叶片,利于将叶片放入离心管底部,取样完成后,每个1.2 ml八联排深孔管中各放一个5 mm钢珠,液氮冷冻后,研磨前手工摇动96孔板,保证钢珠充分晃动,研磨时间仅需90 s,避免样品研磨不充分或在研磨过程中组织褐变;研磨后立即用镊子轻轻敲打离心管盖,使附着在内壁的样品掉落,待管盖充分解冻后,再开盖,防止因液氮挥发造成样品溅飞,造成交叉污染;另外,实验过程中每次开盖前,称量配平,瞬时离心,八通道移液枪悬空加样,吸头污染时及时更换可有效防止样品间交叉污染。传统CTAB法所需的样品量约为0.5 g,且需要研钵手动研磨,采用磨样仪磨样时一次只能磨几十个样品,且磨样时间较长,本研究所需样品组织量仅为30 mg,不会影响幼苗的生长及试验材料的其他研究工作,且幼嫩叶片DNA含量多,糖类、酚类、蛋白质等次生代谢产物积累少[11,29],有利于高质量DNA的分离。实验中采用氯仿、异戊醇混合液仅抽提一次,简化了实验步骤,但获得的DNA样品OD260mm/OD280mm仍介于1.81—1.98,与普通提取方法所获得的DNA纯度几乎无差异。DNA沉淀采用150 μL的0.1×TE溶解,样品浓度最低约为25 ng·μL-1,虽无法达到CTAB法或高成本试剂盒高浓度要求,但类似于普通DNA提取试剂盒浓度,产量和浓度均可充分保证PCR检测、HRM分型、聚丙烯酰胺凝胶电泳等基础研究的进行。
利用提取的DNA进行HRM基因分型和聚丙烯酰胺凝胶电泳分析,两种研究方法灵敏度高,能够准确直观反应DNA模板的质量,有效对不同基因型进行分析;且所需模板量约为1 μL,一次提取的DNA量可进行100次SNP基因分型、InDel标记分析等生物学研究,可保证后续研究进行。
本研究提出的DNA提取方法快速高效,节约时间和成本,应用于大规模群体样本DNA的制备,PCR效果和重复性好,虽简化了提取步骤,但兼具CTAB法提取DNA的高质量[30]和简易提取DNA高通量的优点,克服了传统DNA提取方法程序复杂、耗时长的缺点,适用于大规模的基因型检测、自交系和品种纯度检测等研究工作。
4 结论
建立了一种简便、有效、低成本的桃基因组DNA提取方法,可以满足基因分型、品种鉴定及遗传分析等分子生物学研究,实现了大批量不同样本基因组DNA的同时提取,具有较高的应用价值。The authors have declared that no competing interests exist.
