
*通讯作者(Corresponding author): 赵涵, E-mail: zhaohan@jaas.ac.cn 第一作者联系方式: E-mail: gemin8614@163.com
收稿日期:2016-02-22 接受日期:2016-06-20网络出版日期:2016-07-28基金:
摘要
关键词:玉米; 氮素; 氮胁迫; 转录组分析; 氮素利用率
Global Transcriptome Analysis in High- and Low-Nitrogen Responsive Inbred Lines of Maize
GE Min, LYU Yuan-Da, ZHANG Ti-Fu, ZHOU Ling, LIN Feng, ZHAO Han

Fund:
Abstract
Keyword:Maize; Nitrogen; Nitrogen stress; Transcriptome profiling; Nitrogen use efficiency (NUE)
Show Figures
Show Figures






氮素作为必需的大量营养元素对植物的生长发育具有至关重要的作用, 氮素的缺乏将严重制约作物产量乃至整个生态系统的初级生产量[1, 2]。玉米作为世界三大粮食作物之一, 其产量的高低很大程度上依赖于氮肥施用量的多少。据统计, 每年约1.2亿吨的氮肥用于全球粮食生产, 被作物利用的氮肥却不超过40%[3], 氮肥有效利用率很低。因此, 如何提高作物对氮素的高效利用已成为农业可持续发展亟待解决的重要课题[4, 5]。
作物氮素利用率(NUE)的广义定义是生物产量与土壤氮肥水平的比值[6], 既反映植物对氮素的获得能力, 同时反映植物对体内氮素的分配和利用能力。该性状极为复杂, 涉及氮素吸收、转运、同化利用等多个生理过程。水稻中曾发现多个QTL对NUE有显著贡献, 并对主效QTL精细定位[7, 8, 9]。Sun等[10]利用水稻氮敏感材料NJ6和钝感材料QZL2构建的重组自交系及其回交群体鉴定出一个控制水稻NUE的主效QTL (qNGR9), 经定点克隆和遗传互补实验发现qNGR9为早期已鉴定的DEP1基因[11], 该基因位点的改变可提高水稻籽粒产量。Sun等[10]后续试验还发现DEP1蛋白可以与G蛋白亚基(RGA1和RGB1)互作, 通过减小RGA1或增加RGB1的活性来抑制氮信号的感知和响应, 最终通过综合分析推断异三聚体G蛋白调控水稻的氮素利用率。此外, 一系列研究也显示玉米的多条染色体上都存在决定NUE的QTL[12, 13, 14]。因此, NUE是一个由多基因控制的数量遗传性状。
提高作物氮素利用率, 实际上就是挖掘作物氮高效利用的潜力。氮高效品种理论上可分为 “ 低氮高效” 和“ 高氮高效” 两类, 前者可以在相近产量水平下通过高效吸收环境氮素而达到节省氮肥的目的, 而后者则可在高产的基础上随氮肥投入的增加进一步提高其产量[15]。然而, 面对国内耕地土壤大规模养分贫瘠的现状, 如何培育出适应低氮贫瘠环境的“ 低氮高效” 玉米品种显得尤为重要。已报道的表达谱研究结果表明植物对氮素的利用率高度依赖于环境和遗传变异的互作和由其所对应的表型性状。Chen等[16]通过对玉米自交系郑58和昌7-2转录组分析发现氮环境和基因型显著互作的96个基因, 这些基因富集于转录因子特别是AP2/EREBP 和WRKY转录因子家族, 推测转录因子在氮环境与基因型互作中发挥重要作用。Gelli等[17]筛选出3种氮敏感型和4种钝感型高粱材料, 比较其低氮环境中的转录组变化发现, 氮敏感型材料的差异表达基因富集于应激反应途径, 例如氧化应激和刺激应激反应; 氮钝感型材料中高亲和氮转运蛋白(NRT2.2、NRT2.3、NRT2.5和NRT2.6)和赖氨酸组氨酸转运蛋白(LHT1)基因的表达量高, 并且钝感型材料还可以通过提高根部的生物量来增加对氮素的摄入。Gelli等[17]研究发现, 在低氮环境下氮敏感和钝感型高粱材料基因的表达模式存在差异, 钝感型材料差异表达基因富集在氮高效吸收等途径。然而, 玉米中这方面的较少。鉴于此, 本研究选取对氮素环境敏感性存在显著差异的玉米自交系B73和Mo17, 借助于RNA-seq二代测序技术对足氮(SN)和低氮(LN)条件下V3期(3片完全展开叶)叶组织的转录组测序, 鉴定不同氮环境下的差异表达基因, 并对差异基因进行双尾方差、聚类分析和基因富集分析(Gene set enrichment analysis), 寻找响应氮环境的关键基因, 为玉米氮高效育种提供一定的理论基础。
1 材料与方法1.1 植物材料与处理方法将玉米自交系B73和Mo17种植于江苏省农业科学院温室(江苏南京)。取饱满的玉米种子置25℃培养箱催芽48 h, 挑选生长状态一致的材料种植于2.5 L的盆钵中, 盆钵内装有充分拌匀的沙土(含氮量为87.5 ± 2.7 mg kg-1)。从玉米V1期(一片完全展开叶)开始进行足氮(SN)和低氮(LN)两种处理, 直至玉米V3期, 每种处理各3盆, 每盆2株。处理溶液为改良的 Hoagland营养液[18](SN: 15.00 mmol L-1 KNO3, LN: 0.15 mmol L-1 KNO3), 每3 d施加一次, 每次每盆200 mL。处理期间利用凯氏定氮法实时监控沙土氮含量, 并将SN和LN处理下沙土氮含量的比值控制在2~4范围内。待玉米生长至V3期, 取植株叶片利用凯氏定氮法测定叶片总氮含量。
1.2 玉米叶片转录组分析于玉米V3期取样(B73-SN、B73-LN和Mo17-SN、Mo17-LN, 生物学重复混合池取样)。利用Promega RNA Isolation System试剂盒提取叶片组织总RNA, 并用TaKaRa公司的RNase-Free DNase I处理除去残留的基因组DNA。所提取的总RNA经质量检测后用于文库构建及Illumina paired-end测序(Illumina Hiseq2500 V4试剂测序平台, 测序模式为125 PE, 测序公司为贝瑞和康)。
1.3 主成分分析首先对玉米叶片转录组测序结果进行预处理, 经低质量Q20清理、Reads读长过滤、rRNA及病毒序列过滤, 获得清理后的高质量序列。随后利用Tophat2程序将这些序列比对到玉米参考基因组, 并利用eXpress程序解析比对结果, 统计分析获得4个样品转录组表达量的FPKM值(fragments per kilobase of exon per million fragments mapped)[19]。最后依据Patterson等所描述的操作流程对4组样品的转录组数据进行主成分分析(Principal Component Analysis, PCA), 利用EIGENSOFT软件提供的 twstats 程序进行Tracey-Widom检验, 得到特征向量的显著性分析[20]。
1.4 基因表达差异的分析为解析氮素对玉米材料转录组的影响, 分析样品的转录组基因差异表达。差异表达基因的筛选条件为玉米材料在低氮(LN)和足氮(SN)处理下基因相对表达量(FPKM值)的比值达2倍以上, 即log2LN/SN ≥ 1或≤ -1。
1.5 差异表达基因的聚类分析利用R软件包, 根据 Widiez等[21]研究方法, 对B73和Mo17中的差异表达基因(differentially expressed genes, DEGs)进行双尾方差分析(因素A为氮环境; 因素B为基因型; P ≤ 0.05)。利用MeV4.9 (http://www.tm4.org/mev.html)软件包K均值聚类方法, 对DEGs进行表达模式的聚类分析。利用KOBAS 2.0 (http://kobas.cbi.pku.edu.cn)[22]网站, 对DEGs进行功能注释和序列相似性映射, 通过Fisher测验找出基因显著性富集的代谢途径(P≤ 0.05)。
1.6 氮标记基因(nitrogen biomarker gene)的表达模式分析首先在NCBI数据库中获取Yang等[23]研究中N标记基因的探针序列GSE32361 (http://www.ncbi. nlm.nih.gov/geo/query/acc.cgi?acc=GSE32361), 然后利用生物信息学软件将探针序列比对到玉米V3版基因组(AGPv3; http://www.maizegdb.org/)上, 获得氮标记基因。如方法1.4所述对氮标记基因进行基因表达差异分析(即log2LN/SN ≥ 1或≤ -1)。最后利用MEV4.9软件包绘制氮标记基因的Heatmap图, 并分析其表达模式。
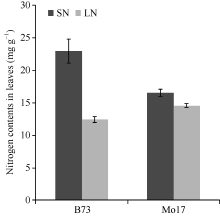
2 结果与分析2.1 氮处理下玉米叶片氮含量分析如图1所示, B73足氮(SN)处理下叶片总氮含量为(22.9 ± 1.9) mg g-1, 低氮(LN)处理下叶片总氮含量为(12.5± 0.4) mg g-1(图1)。经单因素方差分析, 不同氮处理下B73叶片总氮含量差异显著(表1), LN处理下总氮含量仅为SN处理的54.6% (12.5/22.9), 下降明显。LN和SN处理下Mo17叶片总氮含量分别为(14.6± 0.3) mg g-1和(16.5± 0.6) mg g-1, 施氮量减少后叶片总氮含量有所下降但两者差异并不显著(表1), LN和SN处理下总氮含量的比值为0.9, 接近一致。此外, LN处理下Mo17叶片总氮含量高于B73, 为其1.3倍(图1)。以上表明, 玉米自交系B73对环境中氮浓度敏感性强, 植株体内的氮积累量极大依赖于外界氮的施加量, 而玉米自交系Mo17对外界氮依赖程度较低。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 玉米叶片总氮含量SN: 足氮; LN: 低氮Fig. 1 Nitrogen contents in maize leavesSN: sufficient nitrogen; LN: limiting nitrogen |
表1
Table 1
表1(Table 1)
| 表1 氮处理下玉米叶片氮含量方差分析 Table 1 ANOVA analysis of nitrogen contents between different treatments |
2.2 玉米叶片转录组分析为了系统分析氮敏感和钝感型材料在不同氮素环境下转录组的变化情况, 对玉米自交系B73和Mo17在2种氮处理下V3期叶片进行取样(B73-SN、B73-LN和Mo17-SN、Mo17-LN)和转录组测序分析。测序后获得高质量数据, B73-SN为14.3 Gb, B73-LN为12.5 Gb, Mo17-SN为14.0 Gb, Mo17-LN为17.1 Gb。4组数据经清理过滤及标准化处理后, 分别获得62 777、64 251、58 418和84 702个转录本(表达量FPKM值> 0)。上述结果显示敏感型材料B73在LN处理下转录本数目比SN多1474个, 而钝感型材料Mo17在LN处理下比SN多26 184个转录本, 数目远高于B73。
2.3 转录组主成分分析上述4组样品的转录组数据(B73-SN、B73-LN和Mo17-SN、Mo17-LN)经主成分分析后共获得2个主成分, 第一主成分(PC1)的贡献率为87.6%, 第二主成分(PC2)的贡献率为12.4%, 清楚地说明了本研究数据的变化趋势。如图2所示, 基因型(B73和Mo17)间完全分开, 氮环境(SN和LN)间也完全分开, 并且Mo17在2种氮环境下的距离较B73远。表明本研究氮浓度的试验设计合理, 能够使试验材料在两种不同氮浓度环境下转录组明显变化, 并且为了更好适应低氮环境, 钝感型材料Mo17转录组发生较大变化, 该变化大于敏感型材料B73。
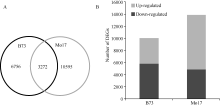
2.4 基因表达差异的分析氮敏感型材料B73在两种氮处理下, 差异表达基因(DEGs, log2LN/SN ≥ 1或≤ -1)数目为10 028个, 其中包含氮代谢相关转录因子Dof等; 氮钝感型材料Mo17在不同氮环境下DEGs数目为13 867个, 高于B73, 约为其数目的1.4倍, 其中包含氮转运蛋白基因(NRT)、氨基酸转运蛋白(AAT)、丝氨酸苏氨酸蛋白激酶(HT1)、氮代谢相关转录因子Dof和Myb。两材料共有DEGs数目3272个(图3-A)。相对于足氮(SN)环境, B73在低氮(LN)条件下约42.2% (4233/10028) DEGs的表达上调, 约57.8% (5795/ 10028) DEGs的表达下调, 基因上调的比例低于下调的比例。然而, 在低氮(LN)环境, Mo17约65.2% (9044/13867) DEGs的表达上调, 约34.8% (4823/ 13867) DEGs的表达下调, 基因上调的比例高于下调的比例, 为其1.9倍(图3-B)。上述结果表明, 相对于B73, 氮钝感型材料Mo17较多基因发生差异表达, 且大部分DEGs表达上调, 推测与其适应氮胁迫有关。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 转录组主成分分析Fig. 2 Principal component analysis (PCA) for experimental samples |
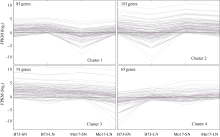
2.5 表达差异基因的聚类分析受氮环境差异直接影响的DEGs为716个, 受基因型差异直接影响的DEGs为545个, 受氮环境和基因型共同影响的DEGs为342个。其中受两者共同影响的基因数目与受基因型影响的数目重叠较大, 为62.8% (342/545), 远高于随机分布。为了对受氮环境和基因型共同影响的DEGs建立更为系统的了解, 我们对上述基因进行了聚类分析。首先, 根据基因的表达模式利用 MEV 4.9软件包K均值聚类方法将342个基因分为4个类群(Cluster1-4), 如图4所示, 4个类群B73和Mo17在两种氮环境下表达模式均有所不同, 基因上调或下调的幅度差异较大。此外, 各类群基因富集分析(P≤ 0.05)表明, Cluster1中聚集的基因数目为83个, 此类群中B73和Mo17的DEGs基因在LN环境下表达量均上调, 上调幅度Mo17高于B73, 此类基因富集在色氨酸代谢(Tryptophan metabolism, zma00380)和醚脂代谢(Ether lipid metabolism, zma00565)途径; Cluster2中基因数目为103个, 在LN环境下基因表达量均下调, 整体表达量水平Mo17高于B73, 此类基因富集在赖氨酸降解(Lysine degradation, zma00310)、类胡萝卜素生物合成(Carotenoid biosynthesis, zma00906)和次生代谢产物的生物合成(Biosynthesis of secondary metabolites, zma01110)途径; Cluster3中基因数目为91个, 在LN环境下该类基因表达量均下调, 下调幅度Mo17高于B73, 此类基因富集在缬氨酸、亮氨酸和异亮氨酸的生物合成(Valine, leucine and isoleucine biosynthesis, zma00290)途径; Cluster4中基因数目为65个, 在LN环境下基因的表达量均上调, 上调幅度B73高于 Mo17, 此类基因富集在碱基切除修复(Base excision repair, zma03410)、真核生物核糖体合成(Ribosome biogenesis in eukaryotes, zma03008)。整体而言, 低氮胁迫环境下受环境和基因型共同影响的DEGs中, 钝感型Mo17基因表达上调或下调的幅度高于敏感型B73, 即对氮缺乏的响应更为剧烈(图4), 且此类基因富集在与氨基酸代谢、光合作用、次级代谢及基因复制表达相关途径, 表明上述途径极可能与低氮环境下氮素的高效利用有关。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 基因表达差异分析Fig. 3 Analysis of differentially expressed genes (DEG)A: B73和Mo17重叠差异表达基因; B: 差异表达基因上调或下调的数目。A: overlap of DEGs in B73 and Mo17; B: number of DEGs down-regulated or up-regulated in N treatment |
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 基因聚类分析Fig. 4 Clusters of DEGs affected by N treatment and genotypes |
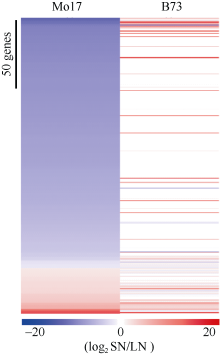
2.6 氮标记基因的表达模式分析为了探究前人报道的氮响应标记基因在本研究材料中的表达情况, 我们对Yang等[23]研究中的氮标记基因分析。发现, 2种氮处理下B73和Mo17中差异表达(log2LN/SN ≥ 1或≤ -1)的氮标记基因[23]数目分别为69个和201个, Mo17远高于B73 为其数目的2.9倍。在LN条件下, B73约44.9% (31/69)氮标记DEG的表达下调, 约55.1% (38/69)的基因表达上调, 基因上调和下调比例接近一致; Mo17氮标记DEG上调和下调比例分别为79.1% (159/ 201)和20.9% (42/201), 基因上调比例远高于下调比例。此外, 在低氮环境下Mo17中约88.1% (177/ 201)的氮标记DEGs在B73中表现出相反或较弱的氮响应模式(图5, 蓝色为下调, 白色为无变化, 红色为上调)。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 两种氮环境下氮标记基因的表达模式分析Fig. 5 Differential expression of N biomarker genes in nitrate-induced Mo17 and B73 |
3 讨论基于已知的氮代谢生理途径, 许多研究对一些关键基因过量表达以期显著提高作物对氮素的利用率。例如, Liu等[24]研究发现拟南芥中过量表达根系氮转运蛋白基因(NRT1.1)可以提高氮的吸收率。Martin等[25]通过在玉米中过量表达胞质谷氨酰胺合成酶基因(cytosolic glutamine synthetase)发现, 温室中种植的转基因玉米植株在籽粒数和籽粒产量上均比对照增加30%。但在随后的大田试验增产效果不显著。Brauer等[26]则认为过量表达胞质谷氨酰胺合成酶基因并不能提高低氮条件下转基因水稻的NUE水平。目前尚未有足够的证据表明过表达氮代谢途径中的单个基因可以显著提高植物NUE, 说明氮素利用率是一个受多基因控制的复杂性状并且和外界环境存在紧密的互作关系。本研究基于二代测序技术对不同氮环境下玉米氮敏感和钝感型自交系V3期叶片组织转录组进行分析, 挖掘出苗期氮环境和基因型互作的关键基因324个, 基因的功能集中于氨基酸代谢、光合作用、次级代谢及基因复制表达等相关的不同代谢途径, 这与前人研究结果一致, 玉米氮素利用率与环境存在紧密互作, 为多基因调控多个代谢途径共同作用的复杂性状。
通过转录组主成分分析验证了本研究氮浓度设计的合理性, 分析结果显示玉米材料间的确存在氮敏感性的差异, 钝感型材料Mo17为了更好适应低氮环境转录组发生较大变化。Mo17在两种氮环境下差异基因的数目为13 867个, 远高于B73差异基因的数目, 并且与B73相反, Mo17在低氮环境下基因上调比例高于下调比例, 表明氮钝感型材料Mo17在低氮环境下较敏感型材料B73激活更多的基因表达以提高植株对氮的吸收和同化能力。Yang等[23]研究中的氮标记基因在本研究中也得到较好的验证, 低氮处理下Mo17差异表达的氮标记基因数目高于B73, 为其数目的2.9倍, 且79.1%的氮标记DEGs表达量上调, 表明钝感型材料在低氮环境下可激发更多的氮响应标记基因表达, 使氮代谢途径中的关键基因处于活跃状态, 尽可能多地吸收利用有限的氮素以适应低氮贫瘠环境。
本研究所鉴定的环境和基因型互作基因的功能涉及范围较广, 覆盖上游基因的表达调控。前人曾发现在外界氮信号刺激下, 拟南芥转录因子AtNLP7[2]作为初期氮响应的重要元件, 能迅速响应并对氮代谢关键基因(NRT、NIA、GS等)的表达有直接调控作用[27, 28]。Chen等[16]也推测转录因子在氮环境与基因型互作中发挥重要作用。表明转录因子作为基因表达的调控元件, 对外界环境中氮浓度感知和氮信号转导及氮代谢途径的调控有重要作用。本研究鉴定的互作基因功能涉及基因的表达调控, 可能其中也含有一些重要的转录因子基因, 后期可作更为详尽的研究。此外, 互作基因的功能还直接涉及氨基酸代谢、光合作用等与氮代谢直接相关的代谢途径。Bi等[3]对玉米自交系SRG100和SRG200, 及其杂交种SRG150表达谱分析发现, 氮响应基因富集在氮复合物代谢、氨基酸代谢等途径。Humbert等[4]对干旱和氮胁迫下玉米材料的表达谱分析发现, 光合作用和氨基酸代谢途径与适应低氮胁迫有关。从上述研究结果可以推测, 氮钝感型材料是通过积极的氮响应机制、转录因子的表达调控、氮代谢相关途径直接调节共同作用以适应氮胁迫环境。然而, 本研究所鉴定的氮环境和基因型互作基因是否对氮素利用率存在贡献, 后期还需利用基因定点突变或转基因技术手段对基因的功能进行更为深入的解析, 以期获得调控玉米氮素利用率的候选基因。此外本研究主要针对玉米苗期表达谱, 该时期为氮素吸收同化储备时期, 鉴于NUE性状的复杂性, 今后还可选择玉米生长的其他关键时期例如籽粒灌浆期进行研究。
4 结论本研究鉴定的玉米自交系氮敏感型B73和迟钝型Mo17在不同氮环境下转录组存在较大差异。发现342个氮环境和基因型互作基因, 低氮环境下钝感型材料中这些基因大部分表达量上调, 这些基因的功能与氨基酸代谢、光合作用、次级代谢及基因复制表达等途径相关。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] | |
| [25] | |
| [26] | |
| [27] | |
| [28] |
