
 , 刘奕1
, 刘奕1

1. 湖北大学 生命科学学院 省部共建生物催化与酶工程国家重点实验室,湖北 武汉 430062;
2. 湖北大学 知行学院,湖北 武汉 430011
收稿日期:2020-10-28;接收日期:2021-02-18;网络出版时间:2021-03-11
基金项目:国家自然科学基金(Nos. 21907027,22007030),中国博士后科学基金(No. 2019M662573),湖北省自然科学基金(No. 2020CFB253) 资助
摘要:研究不同基因、染色体以及基因与染色体之间的时空关系在遗传学、发育生物学和生物医学等领域具有重要意义。CRISPR/Cas9基因编辑技术具有优异的靶向性,已经成为应用最广泛的基因编辑工具。近年来,研究人员基于Cas9的核酸酶失活突变体dCas9发展了一系列先进的活细胞成像技术,为染色质、基因组特定位点的高分辨成像提供了快速、方便的研究工具。文中从细胞递送方式、荧光信号优化以及正交多色成像3个方面对CRISPR/dCas9系统在活细胞成像中的研究进展进行了综述,并对该领域的发展趋势进行了展望。
关键词:基因编辑CRISPR/dCas9基因组成像染色体成像
Advances of CRISPR/dCas9 system in live cell imaging
Siyu Lin1, Xing Zhong2, Lixin Ma1, Jie Qiao1

 , Yi Liu1
, Yi Liu1

1. State Key Laboratory of Biocatalysis and Enzyme Engineering, School of Life Sciences, Hubei University, Wuhan 430062, Hubei, China;
2. Zhixing College, Hubei University, Wuhan 430011, Hubei, China
Received: October 28, 2020; Accepted: February 18, 2021; Published: March 11, 2021
Supported by: National Natural Science Foundation of China (Nos. 21907027, 22007030), China Postdoctoral Science Foundation (No. 2019M662573), Natural Science Foundation of Hubei Province, China (No. 2020CFB253)
Corresponding author: Jie Qiao. Tel: +86-27-88661237-6349; E-mail: jieqiao@hubu.edu.cn;
Yi Liu. Tel: +86-27-88661237-6349; E-mail: yiliu0825@hubu.edu.cn.
Abstract: The study of distinct genes, chromosomes and the spatio-temporal relationships between them is of great significance in genetics, developmental biology and biomedicine. CRISPR/Cas9 has become the most widely used gene editing tool due to its excellent targeting ability. Recently, researchers have developed a series of advanced live cell imaging techniques based on the nuclease-inactivated mutant of Cas9 (dCas9), providing rapid and convenient tools for high-resolution imaging of specific sites in the chromatin and genome. This review summarizes the advances of CRISPR/dCas9 system in live cell imaging from three aspects, including the strategies of cell delivery, optimization of the fluorescence signals, as well as orthogonal and multicolor imaging. Furthermore, we shed light on the development trends and prospects of this field.
Keywords: gene editingCRISPR/dCas9genome imagingchromosome imaging
CRISPR/Cas (Clustered regulatory interspaced short palindromic repeats/CRISPR-associated system) 系统是细菌和古菌在长期演化过程中形成的一种适应性免疫防御系统,可用来对抗入侵的病毒及外源DNA[1-3]。该系统由Cas核酸蛋白酶以及单引导RNA (Single guide RNA,sgRNA) 结合形成核酸蛋白复合物(Ribonucleoprotein,RNP),然后靶向切割原核或真核生物的基因组[4]。近年来,以CRISPR/Cas9为代表的新一代基因编辑技术已经成功应用于生物学的各个领域。此外,研究人员还基于Cas9的核酸酶失活突变体dCas9 (Nuclease-deficient Cas9) 发展了一系列生物成像技术,为研究染色质活性、生理机能以及疾病发生机制之间的关系提供了有效的研究工具[5-6]。本文对CRISPR/dCas9系统和活细胞成像技术进行简介,并从细胞递送方式、荧光信号优化以及正交多色成像3个方面综述了CRISPR/dCas9系统在活细胞成像上的最新研究进展,以期为该领域的发展方向提供参考意见。
1 CRISPR/dCas9系统以化脓性链球菌Streptococcus pyogenes来源的Cas9 (spCas9) 为代表的CRISPR家族蛋白已经广泛应用于遗传育种[7-8]和生物医学[9-11]等领域。通过sgRNA与基因序列的特异性结合,Cas9可在特定的基因位点完成切割,造成DNA双链的断裂。Cas9核酸内切酶的活性依赖于HNH与RuvC结构域,其中HNH结构域负责切割与sgRNA互补的靶标DNA单链(Target DNA strand,tDNA)[12],而RuvC结构域介导非靶标DNA链(Non-target DNA strand,ntDNA) 的切割[13]。如图 1所示,通过突变Cas9第10位的天冬氨酸(Asp) 与第840位的组氨酸(His),可获得核酸酶失活的突变体dCas9 (D10A/H840A)[14-15]。该突变体不具有切割DNA的能力,但保留了结合目标基因序列的功能。研究者基于CRISPR/dCas9系统开发了一系列基因组标记和成像工具,实现了对细胞内基因组特定位点的高分辨成像[6, 16-18]。
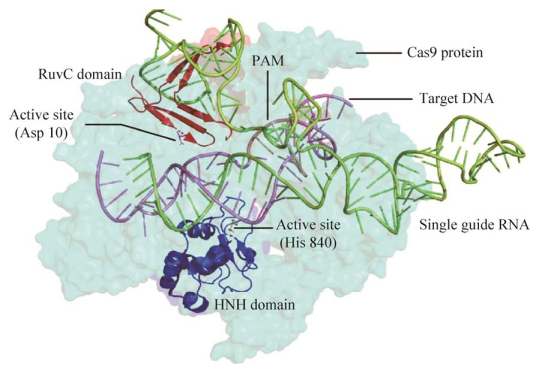 |
| 图 1 Cas9/sgRNA蛋白复合物与靶DNA结合的三维结构示意图 Fig. 1 Schematic diagram of the three-dimensional structure of Cas9/sgRNA protein complex binding to the target DNA. |
| 图选项 |
2 染色体成像技术染色质是基因组的基本调节单元。在细胞核的三维空间范围内,染色质的进一步包装可以直接影响基因的活性。因此,研究染色质在时空上的动态变化对理解基因组功能非常重要[19]。标记基因组的特定序列,然后用显微镜直接观察细胞核内基因组的结构和动态变化是一种经典的染色体成像方法,例如荧光原位杂交(Fluorescent in situ hybridization,FISH) 技术是目前绘制基因图谱[20-23]和医学遗传诊断[24]的常用手段。FISH要求将dsDNA部分解链,然后解链的DNA与外源荧光探针通过碱基互补配对形成杂交链。因此,用于FISH的生物材料往往需要经过筛选、固定化等苛刻的处理步骤,极大地限制了该技术在活细胞成像上的应用[25-26]。利用DNA操纵子/结合蛋白标记系统,如Lac O/阻遏蛋白标记系统,可直接观察活细胞染色质的结构和动力学变化[27],但该方法依赖于人工插入外源的重复基因序列,无法用于基因组内源性序列的标记。因此,开发更有效的活细胞染色体成像工具十分必要。
3 CRISPR/dCas9活细胞成像技术2013年,Chen等首次将CRISPR/dCas9系统应用于活细胞成像[28]。研究者首先将dCas9和荧光蛋白的编码基因整合入人源细胞的基因组,构建了dCas9-荧光蛋白融合表达的细胞系;然后将sgRNA的转录质粒转染至上述细胞,实现了对染色体基因组序列的定位成像。最近,基于CRISPR/dCas9系统的活细胞成像技术的不断发展,如利用相关成像工具对染色体端粒和等位基因的动态变化机制进行了深入研究[17]。
活细胞成像技术通常需要提供适合细胞生长与动力学研究的环境,维持较高的荧光信号信噪比。CRISPR/dCas9活细胞成像技术的基本原理是将dCas9与荧光基团偶联,在细胞核定位序列(Nuclear localization sequence,NLS) 的引导下穿过细胞器膜,特异性地标记染色体基因序列。本文将从递送方式、信号优化以及多色成像3个方面综述CRISPR/dCas9系统在活细胞成像领域的研究进展。
3.1 CRISPR/dCas9的递送方式用于生物成像所用的细胞材料一般不具备编码CRISPR/dCas9系统的功能。因此,如何将dCas9蛋白与sgRNA成功地递送入细胞或在细胞中表达成为影响成像效果的关键因素。如图 2所示,主要有3种方式在细胞内产生用于活细胞成像的dCas9/sgRNA复合物,即胞内稳定表达、胞内瞬时表达和体外组装RNP。
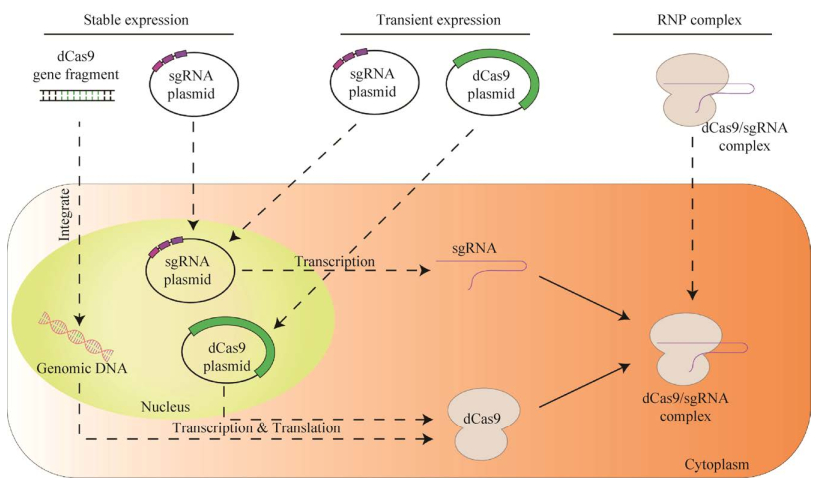 |
| 图 2 三种CRISPR/dCas9系统的递送方式 Fig. 2 Three strategies for delivering CRISPR/dCas9 system. |
| 图选项 |
3.1.1 基于胞内稳定表达的递送方式首先,研究者通过慢病毒感染的方式将dCas9和荧光蛋白的编码序列整合入细胞的基因组。然后,利用选择性标记筛选获得稳定表达dCas9融合荧光蛋白的细胞系。接着,将sgRNA转录质粒导入上述细胞系,实现对染色体基因组特定序列的定位成像[29]。Duan等利用胞内稳定表达的dCas9-EGFP标记小鼠染色体端粒等内源性高度重复序列,用于观察多种组织细胞中的基因组动态变化[30]。该方法的优势在于细胞系中的每一个细胞均表达融合蛋白,可提供均一的荧光强度。此外,通过外源导入质粒的方式可同时转录多条针对内源性非重复基因序列的sgRNA[30-31],适用于长期追踪基因组上的目标序列,以及研究固定模型下基因序列空间位置的动态变化。但是,该方法的成像效果会受到病毒质粒转染水平和sgRNA转录水平的限制。例如,sgRNA的不足会造成dCas9-融合荧光蛋白在细胞内呈现弥散分布状态,造成强荧光背景或聚集成荧光灶,阻碍目标位置荧光信号的分辨[32]。
3.1.2 基于胞内瞬时表达的递送方式通过电转或化学试剂转染等方式,可将dCas9融合荧光蛋白的表达质粒和sgRNA的转录质粒导入细胞中,实现细胞内dCas9/sgRNA复合物的高水平瞬时表达。Anton等通过共转染融合了GFP标签的dCas9表达质粒与sgRNA转录质粒,成功实现了对小鼠胚胎干细胞的端粒(Telomeres,Tel) 重复序列、主要卫星(Major satellite,MaS) 和次要卫星(Minor Satellite,Mis) 重复序列等内源基因的标记[33]。采用胞内瞬时表达方式产生的蛋白表达水平依赖于质粒转染量、启动子强度与表达时间等因素,不同细胞之间的表达水平差异较大。通过优化实验条件可获得合适的融合蛋白表达量,减少因融合蛋白过度表达或sgRNA转录水平不足造成的荧光背景。
3.1.3 基于体外组装RNP复合物的递送方式研究表明,未与Cas9蛋白和目标DNA形成三元复合物的sgRNA在胞内非常容易降解[34]。在体外,孵育重组表达的dCas9、合成或体外转录制备的sgRNA,可制备完整组装的dCas9/sgRNA复合物。Zhang等将RNP复合物递送入细胞,发现可显著缩短形成三元复合物所需的时间[35],具有开发快速成像工具的潜力。该方法的另一大优势是能够对sgRNA方便地进行化学修饰。Wang等在sgRNA的5′端标记荧光素,并与等量的dCas9蛋白孵育以制备荧光核糖核蛋白复合物(Fluorescent ribonucleoproteins,fRNPs),发展了名为CRISPR LiveFISH的成像技术[36]。此时,没有和dCas9结合的sgRNA会被降解,从而极大地降低了背景荧光,实现了对活细胞内源性基因时空变化的动态监测。
由于dCas9体积较大,将大量的RNP复合物递送入细胞仍极具挑战[32]。除了常规的电穿孔(电转) 方式以外,研究人员相继报道了膜变形[37]、金纳米组装体[38]及壳聚糖纳米粒子包裹[39]等递送方法,未来有望更好地用于RNP的活细胞成像。采用直接递送RNP进行活细胞成像的关键在于获得高纯度、高活性的核酸蛋白复合物。近期,笔者课题组建立了“一步法”制备Cas9 RNP的方法,将含有Cas9蛋白表达元件和sgRNA转录元件的质粒转入大肠杆菌中自组装形成RNP复合物,利用IM7/CL7强相互作用系统进行蛋白纯化,可快速获取具有超高稳定性的RNP复合物,有望实现RNP活细胞成像工具的大规模、低成本制备[40]。
3.2 荧光信号优化用于活细胞成像的荧光信号通常来自荧光素或荧光蛋白,需要在适宜的细胞环境下具有可被检测的荧光亮度与荧光稳定性。一般而言,单个荧光标签能提供的荧光信号有限。为了能清晰地分辨目标基因,研究人员陆续开发出多种基于RNA或蛋白支架的方法来富集荧光信号和提高信噪比。
3.2.1 RNA支架2016年,多篇以CRISPR/dCas9系统为基础的活细胞成像工作采用了RNA支架偶联荧光蛋白的优化方法[32, 41-43],即利用RNA茎环重复序列与对应蛋白特异性结合的特性,将荧光蛋白与RNA关联,例如,MS2-MCP、PP7-PCP等系统的应用。图 3A展示了MS2-MCP系统的设计思路:在sgRNA的3′端加入MS2茎环重复序列以构建RNA支架,可招募多个与荧光蛋白融合的MCP蛋白(MS2 coat protein),实现荧光信号的富集。Shao等对RNA茎环重复序列的插入位置以及茎环序列进行了研究,避免茎环支架的插入影响dCas9/ sgRNA复合物与目标位点结合[42]。后来,Qin等[44]设计了整合16个MS2的sgRNA,减少针对内源性非重复基因组成像时sgRNA的设计和使用数量。此外,Cheng等则开发了可串联多达25拷贝的直链RNA支架的Casilio系统[45]。如图 3B所示,融合荧光蛋白的PUF蛋白与PBS (PUF-binding site) 序列结合,能够对端粒和着丝粒等基因组位点进行选择性标记。与环状的茎环支架相比,一单位dCas9/sgRNA直链支架复合物可招募更多的荧光蛋白。此外,直链RNA更容易被转录,并与dCas9形成RNP复合物。通过更改仅有8-mer大小的PBS碱基序列,可形成4种正交的PUF蛋白-PBS结合对,为后续多色成像工具的开发奠定了基础。
 |
| 图 3 构建sgRNA茎环支架[42] (A) 或直链支架[45] (B) 用于增强荧光信号 Fig. 3 Construction of sgRNA stem-loop scaffold (A)[42] or linear scaffold (B)[45] to enhance the fluorescence signal. |
| 图选项 |
3.2.2 蛋白支架提高荧光信号的信噪比的常见方式包括增强目标位点的荧光信号,或者降低体系的荧光背景。前者一般通过富集更多的荧光蛋白得以实现。例如,一单位dCas9蛋白可被多个荧光蛋白标记,在提高荧光强度的同时还可减少sgRNA的使用种类,增加了CRISPR/dCas9系统标记的灵活性[46]。但是,受重组蛋白在细胞内表达量的影响,融合表达多拷贝荧光蛋白标签具有一定的难度,因此串联标签一般选择小分子多肽[47-48]。如图 4所示,Tanenbaum等[47]开发了Suntag系统:在dCas9蛋白的C端融合了24拷贝的抗原决定簇(短肽),然后共表达与抗原决定簇特异性结合的抗体-GFP融合蛋白,利用抗原-抗体的特异性结合间接实现多拷贝GFP蛋白的标记。相比于dCas9融合单拷贝GFP,Suntag系统的信噪比有5.5倍的提高。降低成像体系的荧光背景要求荧光来源物在非成像位置显现出较低的荧光信号。例如,将完整的荧光蛋白一分为二,当两个基团独立存在时不发荧光,只有互补重组后才能恢复荧光,称为自补体分裂荧光蛋白(Split fluorescence protein,FP)[49]。如图 5A、B所示,将GFP在第10和第11个β-折叠片处进行分割,可得到分裂的荧光蛋白元件FP1-10以及FP11。dCas9蛋白融合多个FP11标签,可招募多个FP1-10片段,在靶位点处产生高荧光信号[50]。基于上述原理,Hong等将分裂荧光蛋白与Suntag系统和sgRNA支架关联,开发了一种名为双分子荧光互补的技术(Bimolecular fluorescent complimentary,BiFC)[51]。如图 5C所示,dCas9融合Suntag系统招募分裂黄色荧光蛋白Venus的N端区域,sgRNA支架招募C端区域。近距离的富集使分裂荧光蛋白自发组装成具有完整功能的荧光蛋白。使用该方法,游离的分裂荧光蛋白没有荧光信号,从而极大地降低了背景荧光,并提高了信噪比,实现了对端粒和MUC4e基因座的高分辨活细胞成像。
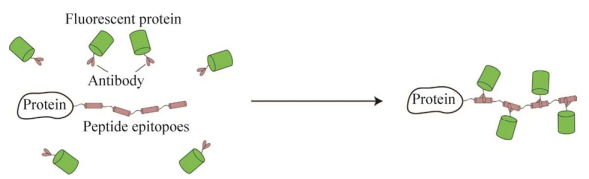 |
| 图 4 Suntag蛋白支架示意图[47] Fig. 4 Schematic diagram of Suntag protein scaffold[47]. |
| 图选项 |
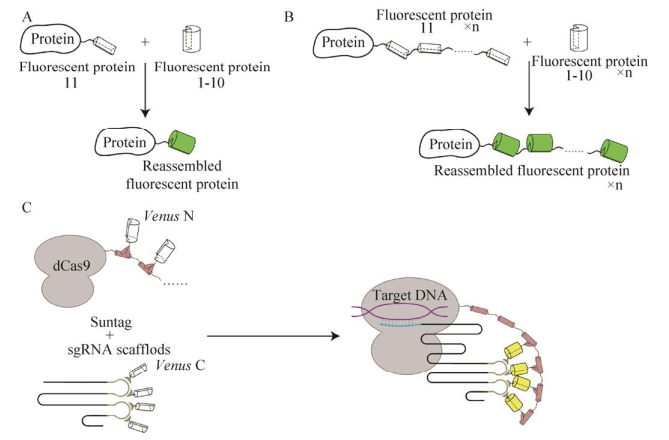 |
| 图 5 自补体分裂荧光蛋白(A、B)[50]与双分子荧光互补技术(C)[51]示意图 Fig. 5 Schematic of the split fluorescence protein (A, B)[50] and BiFC (C)[51]. |
| 图选项 |
3.3 正交多色成像活细胞内同时追踪两个或以上基因的动态变化,需要将不同基因的“定位”与“颜色”一一对应。在不改动目标基因拷贝数的情况下,对细胞内基因多色、多点同时追踪的关键在于使用正交系统,即利用正交对互不干扰的特性,分别标记不同颜色的荧光因子,以达到在细胞内同步成像多基因位点的目的。如利用化脓性链球菌Streptococcus pyogenes (Sp)、脑膜炎双球菌Neisseria meningitidis (Nm)、嗜热链球菌Streptococcus thermophilus (St1) 和金黄色葡萄球菌Staphylococcus aureus (Sa)等不同种属来源的Cas9蛋白具有不同PAM识别位点和互不干扰的特性,相互间作为正交对蛋白,实现对活细胞内多条染色体重复序列的多色成像[46, 52]。除SpCas9蛋白外,其他来源的dCas9蛋白对PAM位点的要求较为严苛,目标基因序列的选择有限。因此,更多的研究将目光投向荧光信号分子与sgRNA的关联。如前文所述的CRISPR LiveFISH技术,Wang等分别采用绿色荧光素Atto488和红色荧光素Atto565修饰多条sgRNA,形成正交系统,实现了对等位基因易位与活细胞转录的同时监测[36]。此外,Ma等开发的CRISPRainbow技术(图 6) 在MS2-MCP和PP7-PCP的RNA支架基础上引入了第3种boxB茎环基序和N22蛋白作为第3种支架体系,然后通过MCP、PCP或N22分别融合蓝色、绿色和红色荧光蛋白用来标记sgRNA,在细胞内形成正交标记[41]。同时,利用多色叠加效应可以产生青、黄、紫、白等颜色,充分发挥了sgRNA支架在多色成像上的优势。
 |
| 图 6 CRISPRainbow多色成像技术[41] Fig. 6 CRISPRainbow multicolor imaging technology[41]. |
| 图选项 |
4 总结与展望CRISPR/Cas9系统因其独特的sgRNA靶向性、使用的简便性以及可融合不同蛋白因子的多样性,已经被广泛应用于基因编辑、转录调控和染色体成像等领域。由Cas9的核酸内切酶失活突变体dCas9与FISH的共定位证明了CRISPR/ dCas9系统能够精确地定位和成像目标基因。近年来,从单色到多色、从单位点到多位点、从定位染色体基因组到追踪染色体基因间易位事件,CRISPR/dCas9系统逐渐展示了在活细胞成像上的巨大应用潜力。当然,目前该领域仍然存在很多亟待解决的问题。例如,针对基因组内源性非重复序列进行高分辨成像仍极具挑战。开发具有更高荧光强度的新型荧光标记多肽,用于与dCas9蛋白偶联荧光标记是解决上述问题的有效途径之一。
CRISPR/dCas9成像技术不可避免地会受到CRISPR/Cas9基因编辑系统本身缺陷的制约。例如,Cas9的脱靶效应有可能造成假阳性的结果。因此,发展新方法,设计和筛选更有效的sgRNA用于目标基因的精准定位成像十分必要。此外,由于受PAM位点的限制,采用CRISPR/dCas9成像系统会有一定的序列局限性。通过基因挖掘、人工改造等方法获得PAM自由度更高的Cas9蛋白变体,是解决上述问题的有效途径。总之,CRISPR/Cas是一个非常庞大的蛋白超家族,Cas9只是其中最为人熟知的一类蛋白。近年来,Cas 12、Cas 13以及Cas14等家族蛋白被相继报道[53-55]。其中,Cas13类蛋白能够特异性地编辑RNA而非DNA。基于CRISPR/dCas13系统,可开发出针对活细胞内RNA进行实时成像的工具。未来,结合CRISPR/dCas9与CRISPR/dCas13成像技术,可同时追踪活细胞甚至活体内DNA与RNA在时空上的动态变化,为理解更复杂的生物调控网络以及遗传疾病的发生机制提供可靠的研究工具。
参考文献
| [1] | Jiang FG, Doudna JA. CRISPR-Cas9 structures and mechanisms. Annu Rev Biophys, 2017, 46: 505-529. DOI:10.1146/annurev-biophys-062215-010822 |
| [2] | Zhang F, Wen Y, Guo X. CRISPR/Cas9 for genome editing: progress, implications and challenges. Hum Mol Genet, 2014, 23(R1): R40-R46. DOI:10.1093/hmg/ddu125 |
| [3] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [4] | Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. DOI:10.1126/science.1258096 |
| [5] | Ma YX, Wang MX, Li W, et al. Live visualization of HIV-1 proviral DNA using a dual-color-labeled CRISPR system. Anal Chem, 2017, 89(23): 12896-12901. DOI:10.1021/acs.analchem.7b03584 |
| [6] | Knight SC, Xie LQ, Deng WL, et al. Dynamics of CRISPR-Cas9 genome interrogation in living cells. Science, 2015, 350(6262): 823-826. DOI:10.1126/science.aac6572 |
| [7] | Chen KL, Wang YP, Zhang R, et al. CRISPR/Cas genome editing and precision plant breeding in agriculture. Annu Rev Plant Biol, 2019, 70: 667-697. DOI:10.1146/annurev-arplant-050718-100049 |
| [8] | Belhaj K, Chaparro-Garcia A, Kamoun S, et al. Editing plant genomes with CRISPR/Cas9. Curr Opin Biotechnol, 2015, 32: 76-84. DOI:10.1016/j.copbio.2014.11.007 |
| [9] | Liu C, Zhang L, Liu H, et al. Delivery strategies of the CRISPR-Cas9 gene-editing system for therapeutic applications. J Control Release, 2017, 266: 17-26. DOI:10.1016/j.jconrel.2017.09.012 |
| [10] | Lu XJ, Xue HY, Ke ZP, et al. CRISPR-Cas9: a new and promising player in gene therapy. J Med Genet, 2015, 52(5): 289-296. DOI:10.1136/jmedgenet-2014-102968 |
| [11] | Saayman S, Ali SA, Morris KV, et al. The therapeutic application of CRISPR/Cas9 technologies for HIV. Exp Opin Biol Ther, 2015, 15(6): 819-830. DOI:10.1517/14712598.2015.1036736 |
| [12] | Zuo Z, Liu J. Structure and dynamics of Cas9 HNH domain catalytic state. Sci Rep, 2017, 7: 17271. DOI:10.1038/s41598-017-17578-6 |
| [13] | Zuo Z, Liu J. Cas9-catalyzed DNA cleavage generates staggered ends: evidence from molecular dynamics simulations. Sci Rep, 2016, 5: 37584. |
| [14] | Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [15] | Anders C, Niewoehner O, Duerst A, et al. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 2014, 513(7519): 569-573. DOI:10.1038/nature13579 |
| [16] | Dreissig S, Schiml S, Schindele P, et al. Live-cell CRISPR imaging in plants reveals dynamic telomere movements. Plant J, 2017, 91(4): 565-573. DOI:10.1111/tpj.13601 |
| [17] | Guo DG, Wang DB, Liu C, et al. CRISPR-based genomic loci labeling revealed ordered spatial organization of chromatin in living diploid human cells. Biochim Biophys Acta Mol Cell Res, 2019, 1866(12): 118518. DOI:10.1016/j.bbamcr.2019.07.013 |
| [18] | Maass PG, Barutcu AR, Shechner DM, et al. Spatiotemporal allele organization by allele-specific CRISPR live-cell imaging (SNP-CLING). Nat Struct Mol Biol, 2018, 25(2): 176-184. DOI:10.1038/s41594-017-0015-3 |
| [19] | Fraser J, Williamson I, Bickmore WA, et al. An overview of genome organization and how we got there: from FISH to Hi-C. Microbiol Mol Biol Rev, 2015, 79(3): 347-372. DOI:10.1128/MMBR.00006-15 |
| [20] | Amarillo FIE, Bass HW. A transgenomic cytogenetic sorghum (Sorghum propinquum) bacterial artificial chromosome fluorescence in situ hybridization map of maize (Zea mays L.) pachytene chromosome 9, evidence for regions of genome hyperexpansion. Genetics, 2007, 177(3): 1509-1526. DOI:10.1534/genetics.107.080846 |
| [21] | Wang CJR, Harper L, Cande WZ. High-resolution single-copy gene fluorescence in situ hybridization and its use in the construction of a cytogenetic map of maize chromosome 9. Plant Cell, 2006, 18(3): 529-544. DOI:10.1105/tpc.105.037838 |
| [22] | Brenna-Hansen S, Li JY, Kent MP, et al. Chromosomal differences between European and North American Atlantic salmon discovered by linkage mapping and supported by fluorescence in situ hybridization analysis. BMC Genomics, 2012, 13: 432. DOI:10.1186/1471-2164-13-432 |
| [23] | Lowenstein MG, Goddard TD, Sedat JW. Long-range interphase chromosome organization in Drosophila: a study using color barcoded fluorescence in situ hybridization and structural clustering analysis. Mol Biol Cell, 2004, 15(12): 5678-5692. DOI:10.1091/mbc.e04-04-0289 |
| [24] | Ratan ZA, Bin Zaman S, Mehta V, et al. Application of fluorescence in Situ hybridization (FISH) technique for the detection of genetic aberration in medical science. Cureus, 2017, 9(6): e1325. |
| [25] | Heng HHQ, Tsui LC. High resolution free chromatin/DNA fiber fluorescent in situ hybridization. J Chromatogr A, 1998, 806(1): 219-229. DOI:10.1016/S0021-9673(98)00150-2 |
| [26] | Vaandrager JW, Schuuring E, Zwikstra E, et al. Direct visualization of dispersed 11q13 chromosomal transloc ations in mantle cell lymphoma by multicolor DNA fiber fluorescence in situ hybridization. Blood, 1996, 88(4): 1177-1182. DOI:10.1182/blood.V88.4.1177.bloodjournal8841177 |
| [27] | Roukos V, Voss TC, Schmidt CK, et al. Spatial dynamics of chromosome translocations in living cells. Science, 2013, 341(6146): 660-664. DOI:10.1126/science.1237150 |
| [28] | Chen BH, Gilbert LA, Cimini BA, et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell, 2013, 155(7): 1479-1491. DOI:10.1016/j.cell.2013.12.001 |
| [29] | Chen BH, Huang B. Imaging genomic elements in living cells using CRISPR/Cas9. Methods Enzymol, 2014, 546: 337-354. |
| [30] | Duan JZ, Lu GQ, Hong Y, et al. Live imaging and tracking of genome regions in CRISPR/dCas9 knock-in mice. Genome Biol, 2018, 19: 192. DOI:10.1186/s13059-018-1530-1 |
| [31] | Xue Y, Acar MJI. Live-cell imaging of chromatin condensation dynamics by CRISPR. iScience, 2018, 4: 216-235. DOI:10.1016/j.isci.2018.06.001 |
| [32] | Fu Y, Rocha PP, Luo VM, et al. CRISPR-dCas9 and sgRNA scaffolds enable dual-colour live imaging of satellite sequences and repeat-enriched individual loci. Nat Commun, 2016, 7: 11707. DOI:10.1038/ncomms11707 |
| [33] | Anton T, Bultmann S, Leonhardt H, et al. Visualization of specific DNA sequences in living mouse embryonic stem cells with a programmable fluorescent CRISPR/Cas system. Nucleus, 2014, 5(2): 163-172. DOI:10.4161/nucl.28488 |
| [34] | Ma HH, Tu LC, Naseri A, et al. CRISPR-Cas9 nuclear dynamics and target recognition in living cells. J Cell Biol, 2016, 214(5): 529-537. DOI:10.1083/jcb.201604115 |
| [35] | Zhang KX, Deng RJ, Teng XC, et al. Direct visualization of single-nucleotide variation in mtDNA Using a CRISPR/Cas9-mediated proximity ligation assay. J Am Chem Soc, 2018, 140(36): 11293-11301. DOI:10.1021/jacs.8b05309 |
| [36] | Wang HF, Nakamura M, Abbott TR, et al. CRISPR-mediated live imaging of genome editing and transcription. Science, 2019, 365(6459): 1301-1305. DOI:10.1126/science.aax7852 |
| [37] | Han X, Liu ZB, Jo MC, et al. CRISPR-Cas9 delivery to hard-to-transfect cells via membrane deformation. Sci Adv, 2015, 1(7): e1500454. DOI:10.1126/sciadv.1500454 |
| [38] | Mout R, Ray M, Tonga GY, et al. Direct cytosolic delivery of CRISPR/Cas9-ribonucleoprotein for efficient gene editing. ACS Nano, 2017, 11(3): 2452-2458. DOI:10.1021/acsnano.6b07600 |
| [39] | Qiao J, Sun WL, Lin SY, et al. Cytosolic delivery of CRISPR/Cas9 ribonucleoproteins for genome editing using chitosan-coated red fluorescent protein. Chem Commun, 2019, 55(32): 4707-4710. DOI:10.1039/C9CC00010K |
| [40] | Qiao J, Li WQ, Lin SY, et al. Co-expression of Cas9 and single-guided RNAs in Escherichia coli streamlines production of Cas9 ribonucleoproteins. Commun Biol, 2019, 2: 161. DOI:10.1038/s42003-019-0402-x |
| [41] | Ma HH, Tu LC, Naseri A, et al. Multiplexed labeling of genomic loci with dCas9 and engineered sgRNAs using CRISPRainbow. Nat Biotechnol, 2016, 34(5): 528-530. DOI:10.1038/nbt.3526 |
| [42] | Shao SP, Zhang WW, Hu H, et al. Long-term dual-color tracking of genomic loci by modified sgRNAs of the CRISPR/Cas9 system. Nucleic Acids Res, 2016, 44(9): e86. DOI:10.1093/nar/gkw066 |
| [43] | Wang SY, Su JH, Zhang F, et al. An RNA-aptamer-based two-color CRISPR labeling system. Sci Rep, 2016, 6: 26857. DOI:10.1038/srep26857 |
| [44] | Qin PW, Parlak M, Kuscu C, et al. Live cell imaging of low- and non-repetitive chromosome loci using CRISPR-Cas9. Nat Commun, 2017, 8: 14725. DOI:10.1038/ncomms14725 |
| [45] | Cheng AW, Jillette N, Lee P, et al. Casilio: a versatile CRISPR-Cas9-Pumilio hybrid for gene regulation and genomic labeling. Cell Res, 2016, 26(2): 254-257. DOI:10.1038/cr.2016.3 |
| [46] | Ma HH, Naseri A, Reyes-Gutierrez P, et al. Multicolor CRISPR labeling of chromosomal loci in human cells. Proc Natl Acad Sci USA, 2015, 112(10): 3002-3007. DOI:10.1073/pnas.1420024112 |
| [47] | Tanenbaum ME, Gilbert LA, Qi LS, et al. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 2014, 159(3): 635-646. DOI:10.1016/j.cell.2014.09.039 |
| [48] | Neguembor MV, Sebastian-Perez R, Aulicino F, et al. (Po)STAC (Polycistronic SunTAg modified CRISPR) enables live-cell and fixed-cell super-resolution imaging of multiple genes. Nucleic Acids Res, 2018, 46(5): 30-40. DOI:10.1093/nar/gkx1271 |
| [49] | Romei MG, Boxer SG. Split green fluorescent proteins: scope, limitations, and outlook. Annu Rev Biophys, 2019, 48: 19-44. DOI:10.1146/annurev-biophys-051013-022846 |
| [50] | Kamiyama D, Sekine S, Barsi-Rhyne B, et al. Versatile protein tagging in cells with split fluorescent protein. Nat Commun, 2016, 7: 11046. DOI:10.1038/ncomms11046 |
| [51] | Hong Y, Lu GQ, Duan JZ, et al. Comparison and optimization of CRISPR/dCas9/gRNA genome-labeling systems for live cell imaging. Genome Biol, 2018, 19: 39. DOI:10.1186/s13059-018-1413-5 |
| [52] | Chen BH, Hu J, Almeida R, et al. Expanding the CRISPR imaging toolset with Staphylococcus aureus Cas9 for simultaneous imaging of multiple genomic loci. Nucleic Acids Res, 2016, 44(8): e75-e87. DOI:10.1093/nar/gkv1533 |
| [53] | Li TW, Zhu LW, Xiao BX, et al. CRISPR-Cpf1-mediated genome editing and gene regulation in human cells. Biotechnol Adv, 2019, 37(1): 21-27. DOI:10.1016/j.biotechadv.2018.10.013 |
| [54] | Ali Z, Mahas A, Mahfouz M. CRISPR/Cas13 as a tool for RNA interference. Trends Plant Sci, 2018, 23(5): 374-378. DOI:10.1016/j.tplants.2018.03.003 |
| [55] | Harrington LB, Burstein D, Chen JS, et al. Programmed DNA destruction by miniature CRISPR- Cas14 enzymes. Science, 2018, 362(6416): 839-842. DOI:10.1126/science.aav4294 |
