

1. 常州大学 药学院,江苏 常州 213164;
2. 常州大学 城乡矿山研究院,江苏 常州 213164;
3. 广州市公安局 广州市刑事科学技术研究所,广东 广州 510030
收稿日期:2020-11-06;接收日期:2021-02-18
基金项目:国家自然科学基金(No. 32002210),常州大学(Nos. 2019K1238,2019K1237,2018K0948,2018K0947) 资助
摘要:大环内酯类抗生素是一类以大环内酯为母核的广谱抗生素。近些年,由于人们对其不规范的生产和使用,抗生素污染成为了重要的环境问题。大量研究表明,微生物降解是现阶段处理抗生素污染的最理想方法。为进一步推动大环内酯类抗生素生物降解的研究,文中概述了大环内酯类抗生素的环境污染现状、微生物降解菌株、降解酶、降解途径和降解大环内酯类抗生素的微生物处理方法,并对大环内酯类抗生素生物降解亟待解决的瓶颈问题进行了讨论,以期为微生物降解后续研究提供参考。
关键词:大环内酯类抗生素微生物降解抗生素菌渣
Advances in biodegradation of macrolide antibiotics
Yulong Yuan1, Dongmei Liu2, Rongcheng Xiang1, Zhenzhen Li1, Meng Zhang1, Jian Zhao3, Bo Fan1, Chunyu Li2, Dongze Niu2, Jianjun Ren2


1. School of Pharmacy, Changzhou University, Changzhou 213614, Jiangsu, China;
2. Urban and Rural Mine Research Institute, Changzhou University, Changzhou 213614, Jiangsu, China;
3. Guangzhou Institute of Criminal Science and Technology, Public Security Bureau of Guangzhou Municipality, Guangzhou 510030, Guangdong, China
Received: November 6, 2020; Accepted: February 18, 2021
Supported by: National Natural Science Foundation of China (No. 32002210), Changzhou University (Nos. 2019K1238, 2019K1237, 2018K0948, 2018K0947)
Corresponding author: Jianjun Ren. Tel/Fax: +86-519-85516132; E-mail: rjj666@aliyun.com.
Abstract: Macrolide antibiotics are a class of broad-spectrum antibiotics with the macrolide as core nucleus. Recently, antibiotic pollution has become an important environmental problem due to the irregular production and abuse of macrolide antibiotics. Microbial degradation is one of the most effective methods to deal with antibiotic pollution. This review summarizes the current status of environmental pollution caused by macrolide antibiotics, the degradation strains, the degradation enzymes, the degradation pathways and the microbial processes for degrading macrolide antibiotics. Moreover, the critical challenges on the biodegradation of macrolide antibiotics were also discussed.
Keywords: macrolide antibioticsmicroorganismsdegradationantibiotic residues
自从1928年弗莱明发现了青霉素后,各类抗生素在医疗、生物科学研究、农业和畜牧业等方面都作出了巨大贡献[1]。抗生素按照化学结构不同可分为β-内酰胺类(Beta-lactam)、氟喹诺酮类(Fluoroquinolones)、大环内酯类(Macrolides)、氨基糖苷类(Aminoglycosides)、四环素类(Tetracyclines)和磺胺类(Sulfonamides) 等6大类,能按不同的作用机制起到抑菌或杀菌作用[2]。其中,大环内酯类抗生素是一类具有12–16个碳内酯母核的广谱抗生素,它与细菌核糖体蛋白的50S亚单位相结合,通过抑制细菌蛋白质合成,从而抑制细菌繁殖[3]。常用的大环内酯类抗生素包括14元环的红霉素(Erythromycin)、克拉霉素(Clarithromycin) 和罗红霉素(Roxithromycin),15元环的阿奇霉素(Azithromycin) 等。其中红霉素是发现最早并被广泛应用于临床的大环内酯类抗生素。
目前,大环内酯类抗生素主要通过发酵法生产。抗生素生产会带来大量含抗生素的菌渣及废液,带来环境风险,这限制了我国抗生素工业的发展。以红霉素为例,我国年产能超万吨,但因为环保问题,实际年产量仅6 000余t。
同时,在抗生素使用过程中,也会造成环境污染。约有30%–90%的抗生素以母体化合物或代谢物形式由人和畜禽的粪、尿排入环境,不仅对环境中的动植物产生毒害作用,还可以改变原有环境的菌群结构,最终导致耐药性细菌(Antibiotic resistance bacteria,ARB) 的产生和耐药性基因(Antibiotic resistance genes,ARGs) 的传播[2]。近年来,大环内酯类抗生素在水生环境中的检出率逐渐增加,李文最等在闽江流域福州段水体中检测出浓度为14.1 ng/L的红霉素[4],方昊等在江苏高淳中华绒蟹养殖塘水体中检测出高达198.53 ng/L的螺旋霉素[5],魏志雄等在东江东莞城区水体中检测出高达265.1 ng/L的阿奇霉素[6]。全球范围内,已在污水处理厂的污水、淡水和海水系统、生物胶体、沉积物及土壤中检测到ng/L级别的红霉素和其他抗生素[7]。
抗生素污染已经成为重要的环境问题。本文介绍了大环内酯类抗生素的降解菌株、降解机制、降解酶以及微生物降解大环内酯类抗生素的应用,旨在为后续微生物降解大环内酯类抗生素研究提供参考。
1 大环内酯类抗生素的微生物降解微生物降解法是降解抗生素的重要方法。相比于物理法、化学法,它具有成本低、效能高、环境污染小等优点,相比于抗生素在环境中自发地水解、光降解,它具有能降解化学稳定结构片段的优势,因而微生物降解法是现阶段降解抗生素最理想的方法[8]。据统计,具有抗生素降解作用的细菌有80%属于厚壁菌门和变形菌门,少部分属于栖热菌门、拟杆菌门、放线菌门和浮霉菌门等[8]。除细菌外,还有一些真菌也被证明具有降解抗生素的能力。与细菌相比,真菌对抗生素有更强的耐受能力,并且可以同时降解多种污染物。
1.1 大环内酯类抗生素降解菌研究者们陆续从环境中筛选分离出能够降解大环内酯类抗生素的菌株(表 1)[9-19]。由表 1可以看出,有些菌株,如乳酸片球菌Pediococcus acidilactici P3-4,具有降解多种大环内酯类抗生素的能力,有很好的应用潜力。但目前利用抗生素降解细菌处理环境中抗生素的实例不多,主要原因是担心细菌中大环内酯类耐药基因的扩散。
表 1 降解大环内酯类抗生素的菌株Table 1 Microbial strains capable of degrading macrolide antibiotics
| Antibiotics | Strains | Concentration | Removal rate (%) | References |
| Bacteria | ||||
| Erythromycin | Pseudomonas putida Ery-E | 30 mg/L | 83.9% (5 d) | [14] |
| Pediococcus acidilactici P3-4 | 2 mg/L | 92.1% (1 d) | [16] | |
| Ochrobactrum sp. WX-J1 | 100 mg/L | 97% (3 d) | [19] | |
| Stenotrophomonas maltophilia ZJB-14120 | – | – | [25] | |
| Bacillus thuringiensis | 53% | 53% (24 h) | [26] | |
| Roxithromycin | Pediococcus acidilactici P3-4 | 8 mg/L | 68.9% (5 d) | [16] |
| Spiramycin | Pediococcus acidilactici P3-4 | 8 mg/L | 82.8% (5 d) | [16] |
| Stenotrophomonas maltophilia ZJB-14120 | – | – | [25] | |
| Jiasamycin | Nocardiaceae | 50 mg/L | 40%–60% (21 d) | [27] |
| Tylosin | Citrobacter amalonaticus | 50 mg/L | 95.2% (2 d) | [9] |
| Burkholderia vietnamiensis | 50–500 mg/L | 99% (7 d) | [15] | |
| Achromobacter | 50 mg/kg soil | 96.08% (7 d) | [17] | |
| Avermectin | Bacillus subtilis G1 | 100 mg/L | 90% (15 d) | [10] |
| Serratia mnarcescens G6 | 150 mg/L | 80% (15 d) | [10] | |
| Bacillus cereus G10 | 150 mg/L | 70% (15 d) | [10] | |
| Acinetobacter tandoiia AW1-18 | 100 mg/L | 75% (6 d) | [11] | |
| Ochrobactrum haematophilum AW1-12 | 100 mg/L | 80% (9 d) | [12] | |
| Bacillus stearothermophilus AZ11 | 100 mg/L | 77.6% (3 d) | [13] | |
| Burkholderia cepacia | 100 mg/L | 90% (36 h) | [18] | |
| Stenotrophomonas maltophilia ZJB-14120 | 200 mg/L | 84.82% (48 h) | [25] | |
| Fungi | ||||
| Erythromycin | Trametes versicolor | – | – | [21] |
| Bjerkandera adusta | – | – | [21] | |
| Rhodotorula mucilaginosa | 100 mg/L | 100% (2 d) | [28] |
表选项
大量的研究表明,利用真菌来降解环境中残留抗生素的方法是可行的[8]。相对于其他种类抗生素,应用真菌处理含大环内酯类抗生素废水或被大环内酯类抗生素污染土地的案例较少。董金霞等利用筛选到的白腐真菌处理阿维菌素生产厂家排放的废水,60 h能将废水的化学耗氧量(Chemical oxygen demand,COD)从1 300 g/L降至182 g/L,降解效率最高可达86 %[20]。Aydin将分离自活性污泥中的真菌变色栓菌Trametes versicolor和烟管菌Bjerkandera adusta进行固定化,用于污泥中红霉素的降解[21]。近年来分离得到的具有抗生素降解能力的真菌,多以白腐真菌为主。Llorca等在实验室条件下用源自白腐真菌的漆酶降解红霉素,去除率达到78%[22]。本课题组已筛选出3株高效的红霉素降解真菌,经鉴定分别属于青霉菌属Penicillium、曲霉属Aspergillus、弯孢菌属Curvularia,并对其形态特征、生理生化和降解性能等进行了进一步研究。研究发现青霉属菌株Penicillium oxalicum RJJ-2,96 h内可以去除84.88%的红霉素[23];弯孢菌属菌株Curvularia sp. RJJ-5通过酶催化断裂糖苷键可以将红霉素降解[24]。目前,获得高效降解大环内酯类抗生素的真菌是应用微生物降解大环内酯类抗生素的重要前提。获得菌株后从分子水平对降解机制进行分析,并挖掘关键酶的作用,可更好地应用于工艺实践。
1.2 大环内酯类抗生素的生物降解途径抗生素的微生物降解是指在微生物通过破坏或修饰抗生素结构使抗生素的分子结构发生改变,使其失活并最终达到无害化。通常包含水解、基团转移和氧化还原3种方式。微生物降解抗生素的某些代谢中间产物及代谢产物,仍具有一定的生物活性和毒性作用。因此,研究抗生素的微生物降解途径十分重要。
目前已有微生物降解泰乐菌素、阿维菌素、红霉素等大环内酯抗生素降解途径的报道。例如越南伯克霍尔德氏菌Burkholderia vietnamiensis对泰乐菌素的降解途径如图 1所示,泰乐菌素A首先脱去1个碳霉糖转化成泰乐菌素B,泰乐菌素B的内酯键水解生成降解产物2,或者泰乐菌素B内酯键水解后醛基也被还原成醇生成产物3[15]。简而言之,微生物对泰乐菌素的作用位点有3处,即泰乐菌素A的糖基、内酯键和醛基。嗜麦芽窄食单胞菌Stenotrophomonas maltophilia对阿维菌素B1a的降解途径如图 2所示,其C24位氧化引入羟基形成代谢产物A。代谢产物A经过C-O键、C-C键、内酯键的断裂和去甲基化等作用分别生成产物B、C和D[25]。苍白杆菌Ochrobactrum WX-J1对红霉素A的降解如图 3所示。通过去糖基化、内酯键断裂等一系列作用,最终降解为丙酸乃至二氧化碳[19]。本课题组利用液质联用色谱(High performance liquid chromatography-mass spectrometry,HPLC-MS)对弯孢菌属菌株Curvularia sp. RJJ-5降解红霉素的产物进行了分析,红霉素A在初始反应中被糖苷水解酶降解为3-氨基氧基红霉素A,初步明确了其对红霉素的降解主要是水解了分子中的糖苷[24]。
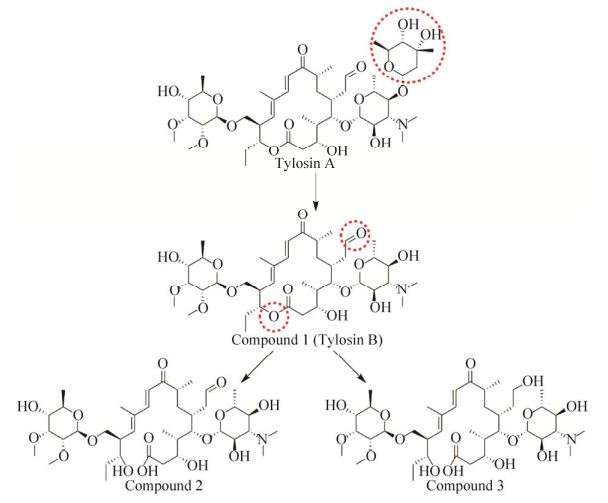 |
| 图 1 泰乐菌素的微生物降解途径[15] Fig. 1 Microbial degradation pathway of tylosin[15]. |
| 图选项 |
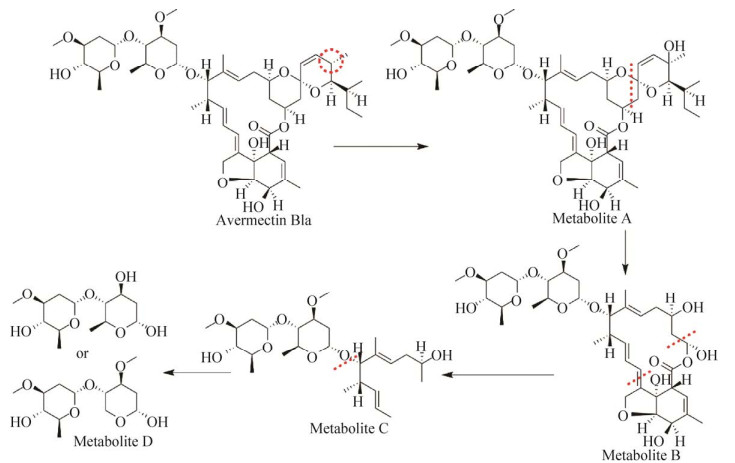 |
| 图 2 阿维菌素的微生物降解途径[25] Fig. 2 Microbial degradation pathway of avermectin[25]. |
| 图选项 |
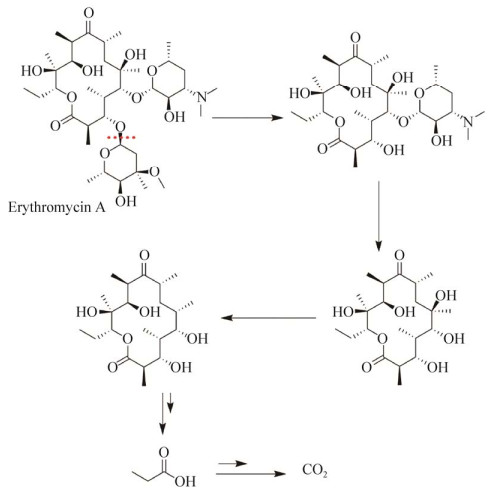 |
| 图 3 红霉素A的微生物降解途径[19] Fig. 3 Microbial degradation pathway of erythromycin A[19]. |
| 图选项 |
研究表明,大环内酯类抗生素的降解是一个复杂的过程,其降解途径主要包括水解、氧化还原、基团转移等方式。不同种属的微生物因为降解酶系存在差异,对不同结构的大环内酯类抗生素,它们的降解途径也存在差异。多种降解产物的检出为大环内酯类抗生素的微生物降解途径研究提供了参考信息。
1.3 降解基因和降解酶目前研究的大环内酯类抗生素降解基因主要来源于抗生素耐药菌。挖掘高效的降解基因及降解酶对大环内酯类抗生素的生物降解具有极其重要的意义。
细菌中常见大环内酯类抗生素的降解酶和基因见表 2。可以利用含有相关基因的野生菌、工程菌或直接利用对应的酶用于抗生素的降解。总体上看,这些基因编码的酶主要有大环内酯酯酶、2'-磷酸转移酶、糖基转移酶。这3类酶分别通过水解、磷酸化和糖基化作用,改变抗生素的结构,使其失去活性[29]。
表 2 大环内酯类抗生素的降解酶和基因Table 2 Enzymes and genes involved in the degradation of macrolide antibiotics
| Degrading enzyme names | Gene names | Drug-resistant strains | References |
| Erythromycin esterase | ereA | Escherichia coli | [30] |
| Providencia stuartii | [41] | ||
| Staphylococcus carnosus | [42] | ||
| Staphylococcus aureus | [43] | ||
| ereB | Escherichia coli | [31] | |
| Staphylococcus aureus | [44] | ||
| Staphylococcus carnosus | [42] | ||
| Lactobacillus plantarum | [42] | ||
| ereC | Klebsiella pneumonia | [32] | |
| ereD | Riemerella anatipestifer | [33] | |
| 2'-phosphotransferase | mphA | Escherichia coli | [45] |
| Shigella sonnei | [46] | ||
| Vibrio fluvialis | [47] | ||
| Bacillus cereus | [48] | ||
| mphB | Escherichia coli | [49] | |
| Staphylococcus aureus | [50] | ||
| Bacillus cereus | [48] | ||
| mphC | Staphylococcus aureus | [51] | |
| Staphylococcus lentus | [52] | ||
| Staphylococcus sciuri | [52] | ||
| Staphylococcus cohnii | [52] | ||
| Staphylococcus xylosus | [53] | ||
| mphD | Pseudomonas aeruginosa | [37] | |
| mphE | Mannheimia haemolytica | [54] | |
| Pasteurella multocida | [36] | ||
| Acinetobacter baumannii | [55] | ||
| Glycosyltransferase | mgt | Streptomyces lividans | [38] |
| oleD | Streptomyces antibioticus | [56] | |
| Streptomyces lividans | [57] | ||
| oleI | Streptomyces antibioticus | [57] |
表选项
第一类是酯酶。文献报道,基因ereA、ereB、ereC和ereD编码的红霉素酯酶能够水解大环内酯类抗生素的内酯键,从而使抗生素失去活性[30-33]。其中ereA和ereB的氨基酸序列有43%的相似性和23%的相同性[31]。Liu等利用表面展示技术在大肠杆菌中表达红霉素酯酶,并用其有效地降解了动物粪便中残留的红霉素[34]。第二类是磷酸转移酶。大环内酯2'-磷酸转移酶将ATP上的γ-磷酸转移到大环内酯的2'-OH,即磷酸化共价修饰大环内酯类抗生素,从而抑制抗生素与核糖体结合,使其失去抗菌活力[25, 35-36]。在大肠杆菌、志贺氏菌、弧菌、巴斯德菌等阴性菌和芽孢杆菌、葡萄球菌等阳性菌中发现此类酶。已知能够编码2'-磷酸转移酶的基因有mphA、mphB、mphC、mphD、mphE等[37]。第三类是糖基转移酶。菌株通过糖基转移酶催化的糖基化反应将抗生素与核糖体结合的重要位点糖基化修饰,使其失活。第一个大环内酯糖基转移酶是从链霉菌Streptomyces lividans中克隆得到的,被命名为mgt[38]。随后陆续发现了一些编码大环内酯糖基转移酶的基因,如oleI和oleD,其中oleI基因位于菌株合成竹桃霉素Oleandomycin的基因簇中,而oleD基因位于该基因簇之外[39-40]。位于基因簇中的糖基转移酶可专一性地糖基化竹桃霉素,而位于合成基因簇之外的糖基转移酶显示出更广泛的大环内酯类化合物的糖基化能力,能使其他大环内酯类抗生素失活从而保护菌株。
2 微生物降解大环内酯类抗生素的应用目前,大环内酯类抗生素的生产方法主要是生物发酵法或以红霉素为原料药经化学半合成法制得。大环内酯类抗生素生产过程中会产生大量含抗生素的固体、液体废物。例如抗生素菌渣就是抗生素发酵生产中的主要固体废物,是较难处置的副产品。抗生素菌渣包含发酵培养基、发酵菌丝、残留抗生素和重金属(铜、铁、锌) 等。研究发现,抗生素菌渣的不当处置会导致其中残留的抗生素进入环境,引起耐药性细菌的产生和耐药性基因的传播,威胁人类健康。因此,在2016年抗生素发酵废物被列入中国危险废物国家目录[58]。在处理抗生素菌渣的过程中,不仅要考虑对其进行充分的无害化处理,还要尽可能地利用其中的营养成分。菌渣减量化的处置方法如焚烧、填埋、用作肥料和动物饲料等普遍存在成本高、二次污染、残留药物积累等隐患。而微生物降解法则能够避免以上问题的出现,是现阶段处理抗生素污染最理想的方法。根据应用微生物的需氧情况又可分为好氧法和厌氧法。
2.1 微生物好氧堆肥法好氧堆肥法,是在合适的湿度、pH和碳氮比情况下,好氧菌将不稳定的有机固体废弃物转化为高度腐殖、卫生无害的有机肥料的微生物处理方法[59-61]。抗生素菌渣通过好氧堆肥,不仅可以减少菌渣对环境的危险,还可以产生有机肥[62]。
好氧堆肥作为处理抗生素菌渣的有效方法,受到研究者的广泛关注。抗生素菌渣中菌群种类单一,抗生素菌渣单独进行堆肥难以达到良好的处理效率,因此一般与其他废弃物共同堆肥[63]。Zhang等通过探究不同浓度铜离子对泰乐菌素菌渣与污泥堆肥中大环内酯类抗性基因丰度的影响,提出ARGs丰度的增加可能与重金属共选择作用有关[64]。菌渣与动物粪便混合进行好氧堆肥是其中一种重要的方法。传统的堆肥方法40 d只能去除猪粪中39%的泰乐菌素残留,效果不理想[65]。超高温堆肥技术处理泰乐菌素抗生素发酵菌渣,去除了95.0%的泰乐菌素、75.8%的ARGs和98.5%的可移动遗传元件。通过降低抗生素抗性质粒和相关宿主细菌的丰度,超高温堆肥可将抗生素发酵废物中的ARGs高效地去除[66]。
文献表明在堆肥过程中细菌、真菌和放线菌均存在,其中细菌占绝对优势且多为革兰氏阳性菌[67]。由于堆肥过程中自然存在的微生物菌群不固定,除了适宜的外界环境条件外,加入特定的抗生素降解菌株或抗生素降解酶可以提高抗生素降解效率。但加入具有降解能力的细菌容易引起ARGs的传播,因此菌渣堆肥过程中加入真菌或者相关酶制品可以降低抗性基因传播的风险。提高真菌在堆肥中的定植能力,对去除菌渣中残留抗生素具有重要意义。菌渣堆肥中ARGs的降解效果主要受温度影响。此外可以通过添加沸石、过磷酸钙等提高ARGs的降解率。
2.2 微生物厌氧发酵法厌氧发酵法是指微生物在缺乏氧气条件下进行生物降解的过程,有时伴有甲烷和二氧化碳产生。抗生素菌渣含有丰富的生物质,因此可以采用厌氧发酵达到减量化和资源化的目的,既减少了污染又增加了经济效益[68]。
厌氧消化技术处理红霉素菌渣既可消除污染又可获得沼气,具有良好效益[69]。蒋明烨等研究表明,红霉素在厌氧发酵过程中的降解主要受发酵液pH的影响,pH越低降解越快,pH越接近中性降解越缓慢[70]。
厌氧消化已被证明可有效减少ARGs[71]。然而,在抗生素菌渣处理中应用常规厌氧消化技术,残留的高浓度抗生素会抑制甲烷的生成,还会导致耐药菌的产生[72]。残留的抗生素可通过降解菌或者降解酶预处理。处理后低浓度的残留抗生素对产甲烷菌可起到一定的驯化作用。利用被红霉素驯化的甲烷菌可在一定程度上提高厌氧发酵效果[68]。高温消化已成为一种有效的生物固体废弃物预处理技术,可改善厌氧消化性能。高温消化可降低螺旋霉素菌渣中抗生素的浓度并去除ARGs,对螺旋霉素菌渣和菌渣与污泥混合物中螺旋霉素的去除率分别为55.3%和99.0%[73]。抗生素菌渣中菌丝体的细胞壁是生物质高效利用的障碍,如果不进行破壁处理,细胞内的有机物质很难释放出来。因此结合电离辐射处理菌丝体,然后通过高温消化菌渣,对菌渣中抗生素和ARGs的去除具有实际的意义。
好氧和厌氧处理是微生物无害化处理抗生素菌渣的重要手段。本课题组在前期的实验中利用好氧高温发酵的方法来调控菌群的丰度,达到降低ARGs水平转移的概率[74],实验表明,通过酶法和发酵工艺技术集成是抗生素菌渣无害化的重要途径。
3 总结与展望大环内酯类抗生素的不当生产和使用对环境造成严重污染。应用微生物降解大环内酯类抗生素是目前研究的热点。已报道的高效降解大环内酯类抗生素的微生物菌株主要是细菌,真菌较少。由于真菌不易传播抗性基因,并具有高效降解抗生素的能力。因此,挖掘更多高效的降解大环内酯类抗生素的真菌菌株具有重要意义。其次,目前大量的研究主要集中于微生物对抗生素的降解能力上,对于利用多组学手段挖掘新的高效的降解酶、构建工程菌、明确代谢途径等方面研究较少,未来应该加强分子层面对抗生素降解机制的研究。包括通过改变代谢通路或者过表达降解途径中的关键酶来提高菌株对大环内酯类抗生素的降解能力;在大环内酯类抗生素高效降解菌株中导入其他抗生素降解基因;对抗生素经过微生物降解后的产物的降解途径及其毒性进行分析。加强这些方面的研究将为正确利用微生物进行抗生素的降解奠定基础。最后,在抗生素菌渣肥料化发酵体系中,直接添加外源的降解细菌容易造成ARGs污染。因此,未来的研究可以探讨添加降解酶进行预处理,或者加入具有降解作用且能分泌胞外酶的真菌进行发酵;还可以从提高真菌在菌渣堆肥中的定植能力、限制负载ARGs水平转移载体的路径、对发酵体系的参数的改变等方面来调节菌群丰度,从而达到降低ARGs水平传播的可能。
目前,关于大环内酯类抗生素生物降解的研究越来越多,随着研究的深入和机理的解析,相信在不久的将来会有更多新型的抗生素降解菌株和降解酶被挖掘出来并应用于实践,抗生素菌渣无害化资源化利用的技术将逐步成熟,为我国的环保事业作出巨大贡献。
参考文献
| [1] | Tan SY, Tatsumura Y. Alexander fleming (1881-1955): discoverer of penicillin. Singap Med J, 2015, 56(7): 366-367. DOI:10.11622/smedj.2015105 |
| [2] | Liu XH, Guo XC, Liu Y, et al. A review on removing antibiotics and antibiotic resistance genes from wastewater by constructed wetlands: performance and microbial response. Environ Pollut, 2019, 254: 112-996. |
| [3] | Retsema J, Fu WC. Macrolides: structures and microbial targets. Int J Antimicrob Agents, 2001, 18(S1): 3-10. |
| [4] | 李文最, 陈高水, 郑艳影, 等. 闽江流域福州段水体中抗生素残留污染调查. 实用预防医学, 2018, 25(12): 1455-1457. Li WZ, Chen GS, Zheng YY, et al. Contamination profiles of antibiotics residues in water bodies of the Fuzhou section of the Minjiang river. Pract Prev Med, 2018, 25(12): 1455-1457 (in Chinese). DOI:10.3969/j.issn.1006-3110.2018.12.013 |
| [5] | 方昊, 余军楠, 王智峰, 等. 江苏典型中华绒螯蟹养殖区抗生素污染特征与生态风险评估. 生态与农村环境学报, 2019, 35(11): 1436-1444. Fang H, Yu JN, Wang ZF, et al. Contamination characteristics and ecological risk assessment of antibiotics in typical Eriocheir sinensis aquaculture environments of Jiangsu province. J Ecol Rural Environ, 2019, 35(11): 1436-1444 (in Chinese). |
| [6] | 魏志雄, 刘丹丹, 蓝明雄, 等. 东江东莞城区段水体中抗生素的含量与污染特征研究. 实用预防医学, 2020, 27(1): 42-45. Wei ZX, Liu DD, Lan MX, et al. Concentration and pollution characteristics of antibiotics in Dongguan urban section of the Dongjiang river. Pract Prev Med, 2020, 27(1): 42-45 (in Chinese). DOI:10.3969/j.issn.1006-3110.2020.01.011 |
| [7] | Schafhauser BH, Kristofco LA, De Oliveira CMR, et al. Global review and analysis of erythromycin in the environment: occurrence, bioaccumulation and antibiotic resistance hazards. Environ Pollut, 2018, 238: 440-451. DOI:10.1016/j.envpol.2018.03.052 |
| [8] | 吴迎, 冯朋雅, 李荣, 等. 环境抗生素污染的微生物修复进展. 生物工程学报, 2019, 35(11): 2133-2150. Wu Y, Feng PY, Li R, et al. Progress in microbial remediation of antibiotic-residue contaminated environment. Chin J Biotech, 2019, 35(11): 2133-2150 (in Chinese). |
| [9] | 刘力嘉, 谢丽, 张作义, 等. 泰乐菌素高效降解菌的筛选及降解特性研究. 农业环境科学学报, 2011, 30(5): 1027-1030. Liu LJ, Xie L, Zhang ZY, et al. Isolation and degradation characteristics of a tylosin-degrading strain. J Agro-Environ Sci, 2011, 30(5): 1027-1030 (in Chinese). |
| [10] | 闫彩虹. 三株阿维菌素高效降解菌的筛选、鉴定及降解特性研究[D]. 扬州: 扬州大学, 2011. Yan CH. Screening and identification of three efficient degradation bacteria of avermectin and their study of degradation characteristic[D]. Yangzhou: Yangzhou University, 2011 (in Chinese). |
| [11] | 胡秀虹, 李景壮, 叶胜蓝, 等. 一株阿维菌素降解菌AW1-18的筛选与分类鉴定. 生物技术通报, 2012, 26(10): 223-228. Hu XH, Li JZ, Ye SL, et al. Screening and identification of avermectin-degrading bacterium strain AW1-18. Biotech Bull, 2012, 26(10): 223-228 (in Chinese). |
| [12] | 胡秀虹, 黄剑. 阿维菌素降解菌AW1-12的筛选与分类鉴定. 西南农业学报, 2013, 26(2): 583-586. Hu XH, Huang J. Screening and identification of avermectin-degrading bacterium strain AW1-12. South Chin J Agric Sci, 2013, 26(2): 583-586 (in Chinese). DOI:10.3969/j.issn.1001-4829.2013.02.038 |
| [13] | 魏艳丽, 李纪顺, 扈进冬, 等. 降解阿维菌素耐高温菌株AZ11的分离及降解特性. 山东科学, 2013(4): 16-19. Wei YL, Li JS, Hu JD, et al. Isolation and degradation characteristics of an abamectin-degrading and thermophilic bacterial strain AZ11. Shandong Sci, 2013(4): 16-19 (in Chinese). |
| [14] | 毛菲菲, 刘畅, 何梦琦, 等. 红霉素降解菌的筛分及其降解特性的研究. 环境科学与技术, 2013, 36(7): 912. Mao FF, Liu C, He MQ, et al. Isolation and identification of an erythromycin degradation bacterium and study on its biodegradation characteristics. Environ Sci Technol, 2013, 36(7): 912 (in Chinese). |
| [15] | 王艳, 马玉龙, 马琳, 等. 泰乐菌素的微生物降解途径及其降解产物研究. 环境科学学报, 2015, 35(2): 491-498. Wang Y, Ma YL, Ma L, et al. Study on microbial degradation pathway and products of tylosin residue in pharmaceutical waste. Acta Sci Circum, 2015, 35(2): 491-498 (in Chinese). |
| [16] | 郝莹, 孙军, 曾金金, 等. 降解多种抗生素残留的乳酸菌的筛选鉴定及特性研究. 中国酿造, 2017, 36(10): 104-108. Hao Y, Sun J, Zeng JJ, et al. Screening and identification of multi-antibiotics-degrading lactic acid bacteria and study on its characteristics. Chin Brew, 2017, 36(10): 104-108 (in Chinese). DOI:10.11882/j.issn.0254-5071.2017.10.022 |
| [17] | 章程, 冯瑶, 刘元望, 等. 菠菜土壤中典型抗生素的微生物降解及细菌多样性. 中国农业科学, 2018, 51(19): 3736-3749. Zhang C, Feng Y, Liu YW, et al. The degradation of typical antibiotics and their effects on soil bacterial diversity in spinach soil. Sci Agric Sin, 2018, 51(19): 3736-3749 (in Chinese). DOI:10.3864/j.issn.0578-1752.2018.19.011 |
| [18] | Ali SW, Li R, Zhou WY, et al. Isolation and characterization of an abamectin-degrading Burkholderia cepacia-like GB-01 strain. Biodegradation, 2010, 21(3): 441-452. DOI:10.1007/s10532-009-9314-7 |
| [19] | Zhang WW, Qiu LN, Gong AJ, et al. Isolation and characterization of a high-efficiency erythromycin A-degrading Ochrobactrum sp. strain. Mar Pollut Bull, 2017, 114(2): 896-902. DOI:10.1016/j.marpolbul.2016.10.076 |
| [20] | 董金霞, 许伟, 罗人明, 等. 白腐真菌处理阿维菌素废水优势菌种筛选研究. 河北化工, 2006(10): 65-67. Dong JX, Xu W, Luo RM, et al. Study on the screening of dominant white-rot fungi degrading avermect in wastewater. J Hebei Chemical Industry, 2006(10): 65-67 (in Chinese). |
| [21] | Aydin S. Enhanced biodegradation of antibiotic combinations via the sequential treatment of the sludge resulting from pharmaceutical wastewater treatment using white-rot fungi Trametes versicolor and Bjerkandera adusta. Appl Microbiol Biotechnol, 2016, 100(14): 6491-6499. DOI:10.1007/s00253-016-7473-0 |
| [22] | Llorca M, Rodríguez-Mozaz S, Couillerot O, et al. Identification of new transformation products during enzymatic treatment of tetracycline and erythromycin antibiotics at laboratory scale by an on-line turbulent flow liquid-chromatography coupled to a high resolution mass spectrometer LTQ-Orbitrap. Chemosphere, 2015, 119: 90-98. DOI:10.1016/j.chemosphere.2014.05.072 |
| [23] | Ren JJ, Wang ZZ, Deng LJ, et al. Degradation of erythromycin by a novel fungus, Penicillium oxalicum RJJ-2, and the degradation pathway. Waste Biomass Valor, 2021, 12: 4513-4523. DOI:10.1007/s12649-021-01343-y |
| [24] | Ren JJ, Deng LJ, Niu DZ, et al. Isolation and identification of a novel erythromycin-degrading fungus, Curvularia sp. RJJ-5, and its degradation pathway. FEMS Microbiology Lett, 2021, 368(1): fnaa215. DOI:10.1093/femsle/fnaa215 |
| [25] | Wang YS, Zheng XC, Hu QW, et al. Degradation of abamectin by newly isolated Stenotrophomonas maltophilia ZJB-14120 and characterization of its abamectin-tolerance mechanism. Res Microbiol, 2015, 166(5): 408-418. DOI:10.1016/j.resmic.2015.04.002 |
| [26] | Zhou PL, Chen Y, Lu QY, et al. Cellular metabolism network of Bacillus thuringiensis related to erythromycin stress and degradation. Ecotox Environ Safe, 2018, 160: 328-341. DOI:10.1016/j.ecoenv.2018.05.048 |
| [27] | Maki T, Hasegawa H, Kitami H, et al. Bacterial degradation of antibiotic residues in marine fish farm sediments of Uranouchi bay and phylogenetic analysis of antibiotic-degrading bacteria using 16S rDNA sequences. Fish Sci, 2006, 72(4): 811-820. DOI:10.1111/j.1444-2906.2006.01222.x |
| [28] | 王敏奇, 许晓玲, 李卫芬, 等. 一株红霉素降解菌红圆酵母的选育及其降解特性. 农业生物技术学报, 2009, 17(2): 341-346. Wang MQ, Xu XL, Li WF, et al. Screening of an erythromycin-degrading strain rhodotorula and its degradation characteristics. J Agric Biotechnol, 2009, 17(2): 341-346 (in Chinese). DOI:10.3969/j.issn.1674-7968.2009.02.027 |
| [29] | 刘元望, 李兆君, 冯瑶, 等. 微生物降解抗生素的研究进展. 农业环境科学学报, 2016, 35(2): 212-224. Liu YW, Li ZJ, Feng Y, et al. Research progress in microbial degradation of antibiotics. J Agro-Environ Sci, 2016, 35(2): 212-224 (in Chinese). |
| [30] | Ounissi H, Courvalin P. Nucleotide sequence of the gene ereA encoding the erythromycin esterase in Escherichia coli. Gene, 1985, 35(3): 271-278. DOI:10.1016/0378-1119(85)90005-8 |
| [31] | Arthur M, Autissier D, Courvalin P. Analysis of the nucleotide sequence of the ereB gene encoding the erythromydn esterase type Ⅱ. Nucleic Acids Res, 1986, 14(12): 4987-4999. DOI:10.1093/nar/14.12.4987 |
| [32] | Yong D, Toleman MA, Giske CG, et al. Characterization of a new metallo-β-lactamase gene, blaNDM-1, and a novel erythromycin esterase gene carried on a unique genetic structure in Klebsiella pneumoniae sequence type 14 from India. Antimicrob Agents Chemother, 2009, 53(12): 5046-5054. DOI:10.1128/AAC.00774-09 |
| [33] | Xing LL, Yu H, Qi JJ, et al. ErmF and ereD are responsible for erythromycin resistance in Riemerella anatipestifer. PLoS ONE, 2015, 10(6): e0131078. DOI:10.1371/journal.pone.0131078 |
| [34] | Liu MR, Feng PY, Kakade A, et al. Reducing residual antibiotic levels in animal feces using intestinal Escherichia coli with surface-displayed erythromycin esterase. J Hazard Mater, 2020, 388: 122-032. |
| [35] | Noguchi N, Katayama J, Sasatsu M. A transposon carrying the gene mphB for macrolide 2'-phosphotransferase Ⅱ. FEMS Microbiol Lett, 2000, 192(2): 175-178. |
| [36] | Rose S, Desmolaize B, Jaju P, et al. Multiplex PCR to identify macrolide resistance determinants in Mannheimia haemolytica and Pasteurella multocida. Antimicrob Agents Chemother, 2012, 56(7): 3664-3669. DOI:10.1128/AAC.00266-12 |
| [37] | Van Hoek AHAM, Mevius D, Guerra B, et al. Acquired antibiotic resistance genes: an overview. Front Microbiol, 2011, 2: 203. |
| [38] | Jenkins G, Cundliffe E. Cloning and characterization of two genes from Streptomyces lividans that confer inducible resistance to lincomycin and macrolide antibiotics. Gene, 1991, 108(1): 5562. |
| [39] | 梁冬梅, 乔建军. 大环内酯糖基转移酶研究进展. 药学学报, 2007, 42(5): 455-462. Liang DM, Qiao JJ. Recent advances in the study of macrolide glycosyltransferases. Acta Pharm Sin, 2007, 42(5): 455-462 (in Chinese). DOI:10.3321/j.issn:0513-4870.2007.05.001 |
| [40] | Quirós LM, Aguirrezabalaga I, Olano C, et al. Two glycosyltransferases and a glycosidase are involved in oleandomycin modification during its biosynthesis by Streptomyces antibioticus. Mol Microbiol, 1998, 28(6): 1177-1185. DOI:10.1046/j.1365-2958.1998.00880.x |
| [41] | Plante I, Centrón D, Roy PH. An integron cassette encoding erythromycin esterase, ere (A), from Providencia stuartii. J Antimicrob Chemother, 2003, 51(4): 787-790. DOI:10.1093/jac/dkg169 |
| [42] | Wang J, Li MY, Wang J, et al. Antibiotic resistance of coagulase-negative staphylococci and lactic acid bacteria isolated from naturally fermented Chinese cured beef. J Food Prot, 2018, 81(12): 2054-2063. DOI:10.4315/0362-028X.JFP-18-195 |
| [43] | Khodabandeh M, Mohammadi M, Abdolsalehi MR, et al. Analysis of resistance to Macrolide-lincosamide-streptogramin B among mecA-positive Staphylococcus Aureus isolates. Osong Public Health Res Perspect, 2019, 10(1): 25-31. DOI:10.24171/j.phrp.2019.10.1.06 |
| [44] | Fe?ler AT, Wang Y, Wu CM, et al. Mobile macrolide resistance genes in staphylococci. Plasmid, 2018, 99: 210. |
| [45] | Noguchi N, Emura A, Matsuyama H, et al. Nucleotide sequence and characterization of erythromycin resistance determinant that encodes macrolide 2'-phosphotransferase Ⅰ in Escherichia coli. Antimicrob Agents Chemother, 1995, 39(10): 2359-2363. DOI:10.1128/AAC.39.10.2359 |
| [46] | Howie RL, Folster JP, Bowen A, et al. Reduced azithromycin susceptibility in Shigella sonnei, United States. Microb Drug Resist, 2010, 16(4): 245-248. DOI:10.1089/mdr.2010.0028 |
| [47] | Chowdhury G, Ramamurthy T, Ghosh A, et al. Emergence of azithromycin resistance mediated by phosphotransferase-encoding mph (A) in diarrheagenic Vibrio fluvialis. mSphere, 2019, 4(3): e0021519. |
| [48] | Wang C, Sui ZH, Leclercq SO, et al. Functional characterization and phylogenetic analysis of acquired and intrinsic macrolide phosphotransferases in the Bacillus cereus group. Environ Microbiol, 2015, 17(5): 1560-1573. DOI:10.1111/1462-2920.12578 |
| [49] | Noguchi N, Katayama J, O'Hara K. Cloning and nucleotide sequence of the mphB gene for macrolide 2'-phosphotransferase Ⅱ in Escherichia coli. FEMS Microbiol Lett, 1996, 144(2/3): 197-202. |
| [50] | Noguchi N, Tamura Y, Katayama J, et al. Expression of the mphB gene for macrolide 2'-phosphotransferase Ⅱ from Escherichia coli in Staphylococcus aureus. FEMS Microbiol Lett, 1998, 159(2): 337-342. |
| [51] | Matsuoka M, Inoue M, Endo Y, et al. Characteristic expression of three genes, msr (A), mph (C) and erm (Y), that confer resistance to macrolide antibiotics on Staphylococcus aureus. FEMS Microbiol Lett, 2003, 220(2): 287-293. DOI:10.1016/S0378-1097(03)00134-4 |
| [52] | Hauschild T, Schwarz S. Macrolide resistance in Staphylococcus spp. from free-living small mammals. Vet Microbiol, 2010, 144(3/4): 530-531. |
| [53] | Papagiannitsis CC, Malli E, Tsilipounidaki K, et al. First description in Greece of mphC-positive Staphylococci causing subclinical mastitis in ewes. Microb Drug Resist, 2018, 24(7): 1050-1053. DOI:10.1089/mdr.2017.0425 |
| [54] | Desmolaize B, Rose S, Wilhelm C, et al. Combinations of macrolide resistance determinants in field isolates of Mannheimia haemolytica and Pasteurella multocida. Antimicrob Agents Chemother, 2011, 55(9): 4128-4133. DOI:10.1128/AAC.00450-11 |
| [55] | Blackwell GA, Hall RM. The tet39 determinant and the msrE-mphE genes in Acinetobacter plasmids are each part of discrete modules flanked by inversely oriented p dif (XerC-XerD) sites. Antimicrob Agents Chemother, 2017, 61(8): e00780-17. |
| [56] | Hernández C, Olano C, Méndez C, et al. Characterization of a Streptomyces antibioticus gene cluster encoding a glycosyltransferase involved in oleandomycin inactivation. Gene, 1993, 134(1): 139-140. DOI:10.1016/0378-1119(93)90189-A |
| [57] | Quirós LM, Carbajo RJ, Salas JA. Inversion of the anomeric configuration of the transferred sugar during inactivation of the macrolide antibiotic oleandomycin catalyzed by a macrolide glycosyltransferase. FEBS Lett, 2000, 476(3): 186-189. DOI:10.1016/S0014-5793(00)01721-X |
| [58] | Chu LB, Chen D, Wang JL, et al. Degradation of antibiotics and antibiotic resistance genes in erythromycin fermentation residues using radiation coupled with peroxymonosulfate oxidation. Waste Manage, 2019, 96: 190-197. DOI:10.1016/j.wasman.2019.07.031 |
| [59] | Gou M, Hu HW, Zhang YJ, et al. Aerobic composting reduces antibiotic resistance genes in cattle manure and the resistome dissemination in agricultural soils. Sci Total Environ, 2018, 612: 1300-1310. DOI:10.1016/j.scitotenv.2017.09.028 |
| [60] | Corbala-Robles L, Sastafiana WND, Van Volcke E, et al. Life cycle assessment of biological pig manure treatment versus direct land application— a trade-off story. Resour Conserv Recycl, 2018, 131: 86-98. DOI:10.1016/j.resconrec.2017.12.010 |
| [61] | Zhang HB, Zhou Y, Huang YJ, et al. Residues and risks of veterinary antibiotics in protected vegetable soils following application of different manures. Chemosphere, 2016, 152: 229-237. DOI:10.1016/j.chemosphere.2016.02.111 |
| [62] | Qian X, Sun W, Gu J, et al. Reducing antibiotic resistance genes, integrons, and pathogens in dairy manure by continuous thermophilic composting. Bioresour Technol, 2016, 220: 425-432. DOI:10.1016/j.biortech.2016.08.101 |
| [63] | 温沁雪, 陈希, 张诗华, 等. 城市污泥混合青霉素菌渣堆肥实验. 哈尔滨工业大学学报, 2014, 46(4): 43-49. Wen QX, Chen X, Zhang SH, et al. Experimental study on co-composting of municipal sewage sludge and penicillin fermentation dregs. J Harbin Inst Technol, 2014, 46(4): 43-49 (in Chinese). |
| [64] | Zhang B, Wang MM, Wang B, et al. The effects of bio-available copper on macrolide antibiotic resistance genes and mobile elements during tylosin fermentation dregs co-composting. Bioresour Technol, 2018, 251: 230-237. DOI:10.1016/j.biortech.2017.12.051 |
| [65] | Zhang RR, Gu J, Wang XJ, et al. Contributions of the microbial community and environmental variables to antibiotic resistance genes during co-composting with swine manure and cotton stalks. J Hazard Mater, 2018, 358: 8291. |
| [66] | Liao HP, Zhao Q, Cui P, et al. Efficient reduction of antibiotic residues and associated resistance genes in tylosin antibiotic fermentation waste using hyperthermophilic composting. Environ Int, 2019, 133: 105203. DOI:10.1016/j.envint.2019.105203 |
| [67] | 张欣阳, 许旭萍. 微生物技术去除抗生素残留污染的研究进展. 生物技术进展, 2014, 4(5): 355-360. Zhang XY, Xu XP. Progress on microbial technology for removal of antibiotics residues pollution. Curr Biotechnol, 2014, 4(5): 355-360 (in Chinese). DOI:10.3969/j.issn.2095-2341.2014.05.08 |
| [68] | 陈立文, 方森海, 王明兹. 抗生素发酵废菌渣的无害化及资源再利用研究进展. 生物技术通报, 2015, 31(5): 1319. Chen LW, Fang SH, Wang MZ. Research progress on the harmless treatment and resource reuse of antibiotic bacteria residues. Biotechnol Bull, 2015, 31(5): 1319 (in Chinese). |
| [69] | 苏建文, 王俊超, 许尚营, 等. 红霉素菌渣厌氧消化实验研究. 中国沼气, 2013, 31(5): 2528. Su JW, Wang JC, Xu SY, et al. Anaerobic digestion of bacterial residues from erythromycin production. China Biog, 2013, 31(5): 2528 (in Chinese). |
| [70] | 蒋明烨, 宋思奇, 付欢, 等. 厌氧发酵处理红霉素菌渣的效能及红霉素降解规律研究. 环境保护科学, 2017, 43(4): 62-67. Jiang MY, Song SQ, Fu H, et al. Research of feasibility for anaerobic digestion of erythromycin mycelial dreg and degradation rules of erythromycin. Environ Prot Sci, 2017, 43(4): 62-67 (in Chinese). |
| [71] | Tian Z, Zhang Y, Yu B, et al. Changes of resistome, mobilome and potential hosts of antibiotic resistance genes during the transformation of anaerobic digestion from mesophilic to thermophilic. Water Res, 2016, 98: 261-269. DOI:10.1016/j.watres.2016.04.031 |
| [72] | Tian Z, Zhang Y, Yang M. Chronic impacts of oxytetracycline on mesophilic anaerobic digestion of excess sludge: inhibition of hydrolytic acidification and enrichment of antibiotic resistome. Environ Pollut, 2018, 238: 1017-1026. DOI:10.1016/j.envpol.2018.02.023 |
| [73] | Awad M, Tian Z, Gao YX, et al. Pretreatment of spiramycin fermentation residue using hyperthermophilic digestion: quick startup and performance. Water Sci Technol, 2018, 78(9): 1823-1832. DOI:10.2166/wst.2018.256 |
| [74] | Ren JJ, Deng LJ, Li CY, et al. Effects of added thermally treated penicillin fermentation residues on the quality and safety of composts. J. Environ. Manage, 2021, 283: 111984. DOI:10.1016/j.jenvman.2021.111984 |
