

1. 西北农林科技大学 动物医学院,陕西 杨凌 712100;
2. 渭南市动物疫病预防控制中心,陕西 渭南 714000
收稿日期:2020-12-11;接收日期:2021-03-25
基金项目:国家重点研发计划项目(No. 2017YFD0500605),陕西省重点研发计划项目(No. 2019NY-076),陕西高校青年创新团队项目,西北农林科技大学试验示范站科技创新与成果转化项目(No. TGZX2020-24) 资助
摘要:文中旨在利用猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV) S1蛋白生物素化纳米抗体建立一种阻断酶联免疫吸附试验(Blocking enzyme-linked immunosorbent assay,bELISA) 方法,用于检测猪体内PEDV抗体水平及疫苗免疫效果的评估。对PEDV S1蛋白的特异性单域抗体(Single-domain antibodies,sdAb) sdAb3基因进行扩增,并在3′端融合Avitag序列,构建至原核表达载体pET21b,进行sdAb3-Avitag蛋白诱导表达纯化。对纯化的sdAb3-Avitag融合蛋白进行生物素标记并鉴定其活性。以PEDV重组S1蛋白为抗原,通过对各反应条件进行摸索与优化,建立一种可靠灵敏的bELISA方法应用于血清样品检测,并与商品化试剂盒检测进行比价。成功构建重组载体pET21b-sdAb3-Avitag并诱导表达出sdAb3-Avitag蛋白。体外生物素标记的sdAb3 (sdAb3-Biotin) 具有良好的活性。所构建的bELISA方法中,最适参数为:S1蛋白的包被浓度200 ng/孔;血清稀释比例1︰2,血清孵育时间2 h;sdAb3-Biotin稀释比1︰8 000,孵育时间30 min;酶标抗体稀释比例1︰5 000,反应时间30 min。所建立的方法与猪传染性胃肠炎病毒、猪繁殖与呼吸综合征病毒等主要猪源病毒的阳性血清均无交叉反应,具有良好的特异性和重复性。利用建立的bELISA方法对临床54份猪血清样品进行检测,结果显示,该方法与商品化试剂盒检测结果具有92.56%的总体符合率。文中建立了一种省时可靠的bELISA方法,可用于PEDV的临床监测和疫苗免疫效果评估。
关键词:猪流行性腹泻病毒生物素化单域抗体阻断ELSIAS1蛋白
Development of a blocking ELISA based on a single-domain antibody target the S1 protein of porcine epidemic diarrhea virus
Zhiqian Ma1*, Ge Bai2*, Tianyu Wang1, Zhiwei Li1, Yang Li1, Shuqi Xiao1, Shuang Li1


1. College of Veterinary Medicine, Northwest Agricultural and Forestry University, Yangling 712100, Shaanxi, China;
2. Weinan Animal Disease Control Center, Weinan 714000, Shaanxi, China
Received: December 11, 2020; Accepted: March 25, 2021
Supported by: National Key Research and Development Program of China (No. 2017YFD0500605), Key Research and Development Program of Shaanxi Province (No. 2019NY-076), the Youth Innovation Team of Shaanxi Universities and the Science and Technology Extension Project in Northwest A & F University (No. TGZX2020-24)
Corresponding author: Shuang Li. Tel: +86-29-87091117; E-mail: lishuang2006001@126.com.
*These authors contributed equally to this study.
Abstract: The aim of this study was to develop a blocking enzyme-linked immunosorbent assay (bELISA) based on a biotinylated nanobody target the S1 protein of porcine epidemic diarrhea virus (PEDV) for detecting the anti-PEDV antibodies and evaluating the immune effect of the vaccine. The gene encoding the single-domain antibody sdAb3 target the PEDV S1 protein was amplified and the Avitag sequence was fused at its 3′-end. The PCR product was cloned into the expression vector pET-21b for expression and purification of the sdAb3-Avitag protein. The purified sdAb3-Avitag fusion protein was biotinylated and its activity was determined. Using the recombinant S1 protein as a coating antigen, a bELISA was established and optimized. Serum samples were tested in parallel by the bELISA and a commercial kit. The recombinant vector pET21b-sdAb3-Avitag was constructed to express the tagged sdAb3. After induction for expression, the biotin-labeled sdAb3 (sdAb3-Biotin) with high purity and good activity was obtained. For the optimized bELISA, the coating concentration of the S1 protein was 200 ng/well, the serum dilution was 1:2 and incubated for 2 h, the dilution ratio of the biotinylated sdAb3 was 1:8 000 and incubated for 30 min, the dilution of the enzyme-labeled antibody was 1:5 000 and incubated for 30 min. The bELISA had no cross reaction with the sera of major porcine viruses including transmissible gastroenteritis virus, porcine reproductive and respiratory syndrome virus and showed good specificity and reproducibility. For a total of 54 porcine serum samples tested, the overall compliance rate of the bELISA with a commercial kit was 92.56%. This study developed a rapid and reliable bELISA method, which can be used for serosurveillance and vaccine evaluation for PEDV.
Keywords: porcine epidemic diarrhea virusbiotinylated single-domain antibodyblocking ELISAS1 protein
猪流行性腹泻(Porcine epidemic diarrhea,PED) 是由猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV) 引起的一种严重的肠道疾病,主要表现为仔猪出现呕吐、脱水、水样腹泻和高死亡率[1-2]。PEDV S蛋白是一类位于病毒表面的糖蛋白,在入侵宿主细胞、病毒毒力和诱导中和抗体等方面发挥重要作用,根据功能不同可将S蛋白分为S1和S2[3]。在感染PEDV的猪体内,由于S蛋白具有较强的抗原性,血清中针对S蛋白的抗体比N蛋白的抗体持续时间更长[4]。因此,S蛋白是建立诊断方法和研发疫苗的靶蛋白。
由于引起猪腹泻的病原能够诱发猪产生相似的临床症状,因此根据临床症状无法准确诊断PED,只能借助实验室诊断技术[5-6]。目前,已有多种方法用于PEDV的检测,主要分为两大类:病原学检测(核酸和病毒蛋白) 和血清学检测。血清学检测方法通常包括间接免疫荧光、病毒中和试验和酶联免疫吸附试验(Enzyme-linked immunosorbent assay,ELISA),这些方法已被广泛用于检测粪便和血清中的PEDV抗体[7-9]。ELISA由于其灵敏度、简便性和安全性等优势而被广泛应用。检测PEDV抗体常用ELISA方法包括间接ELISA、竞争ELISA和阻断ELSIA,且后两种方法具有更强的特异性[9-10]。
抗体是ELISA的核心试剂,它决定了ELISA方法检测的灵敏性和特异性,目前大多数ELISA试剂盒使用多克隆抗体或者单克隆抗体作为监测试剂,但这些抗体生产工艺烦琐、价格昂贵和储存困难[9, 11-12]。因此,迫切需要开发易于生产且生产成本低的抗体用于ELISA方法的建立。与常规抗体不同,纳米抗体,又称单域抗体,是来自骆驼科动物或者软骨鱼类的重链抗体,仅有一个重链可变区结构域,分子量小,只有约15 kDa [13]。纳米抗体具有良好的热稳定性、构象稳定性和良好的溶解性,且易在多种表达系统进行生产[14]。基于这些优势,纳米抗体受到越来越多的关注,有望被用于生物诊断和疫病治疗中[15-16]。2019年,Sheng等将新城疫病毒(Newcastle disease virus,NDV) 的纳米抗体与辣根过氧化物酶(Horseradish peroxidase,HRP) 融合表达,建立了一种竞争ELISA用于临床NDV抗体的检测[17]。Du等利用生物素化单域抗体建立了一种阻断ELISA检测猪流感病毒(Swine influenza virus,SIV) 抗体[18]。Ma等利用PEDV N蛋白纳米抗体建立了一种快速检测PEDV抗体的bELISA方法[19]。Ji等将NDV的纳米抗体以铁蛋白融合的纳米抗体(Fenobody) 和纳米抗体融合的报告基因(RANbody) 的形式呈现,开发了用于检测不同样品中NDV的夹心ELISA[20]。目前尚无PEDV S1蛋白特异性单域抗体及其在ELISA方法建立中的应用报道。本课题组前期利用噬菌体展示技术筛选出针对PEDV S1蛋白的单域抗体sdAb3,本研究对sdAb3进行体外生物素标记,并利用生物素标记的sdAb3建立一种检测PEDV抗体的bELISA方法,以期为PED监测和免疫效果评估提供一个可靠方法。
1 材料与方法1.1 主要试剂单域抗体sdAb3是由本课题组前期利用噬菌体展示技术筛选的针对PEDV S1蛋白的单域抗体,基因大小为339 bp。pET21b载体和pCANTAB-5E-sdAb3质粒由本实验室保存,大肠杆菌E. coli Trans5α克隆菌株、E. coli BL21(DE3)菌株和Ni-NTA Agarose购自TransGen Biotech,生物素连接试剂盒购自广州易锦生物技术有限公司,HRP标记的链霉亲和素购自美国Thermo Fisher Scientific公司,PEDV抗体检测试剂盒购自加拿大Biovet公司。
1.2 引物设计与合成为了构建重组单域抗体sdAb3的表达载体,根据sdAb3基因序列设计引物,在5′端引入BamHⅠ酶切位点,3′端引入Avitag短肽序列和Hind Ⅲ限制性内切酶位点,由西安擎科泽西生物科技有限责任公司负责引物的合成,序列如下(斜体字分别为限制性内切酶位点BamH Ⅰ和Hind Ⅲ酶切位点,下划线处为Avitag标签):pET21b-sdAb3-Avitag-F:FCGGGATCCGCAGGT CCAACTGCAGGAG;pET21b-sdAb3-Avitag-R:CCCAAGCTTTTCGTGCCATTCGATTTTCTGAGCTTCGAAATATCGTTCAGACCTGAGGAGACGGTGACCTGGGTCC。
1.3 重组单域抗体sdAb3原核表达载体的构建以pCANTAB-5E-sdAb3质粒为模板,进行sdAb3基因片段的扩增,利用相应的酶切位点将其连入原核表达载体pET21b中,得到pET21b-sdAb3-Avitag质粒,测序正确后,用于下一步试验。
1.4 重组单域抗体sdAb3的表达与纯化将pET21b-sdAb3-Avitag质粒转化至E. coli BL21(DE3) 感受态细胞中,挑取阳性克隆菌,接种于LB培养基中,于37 ℃恒温摇床中200 r/min振荡培养过夜,按照1︰100的比例接种于大的摇瓶中,培养至对数期,加入终浓度为1 mmol/L的IPTG进行诱导表达,诱导5 h后收菌。取1 mL诱导后菌液于1.5 mL离心管中,12 000×g离心1 min收取菌液沉淀,加入100 μL 2×上样缓冲液,重悬菌体,置于沸水中煮10 min,12 000×g离心10 min,取上清进行SDS-PAGE分析。对诱导后的菌体进行超声破碎,138 kW超声3 s,暂停3 s,共超声40 min,4 ℃ 12 000×g离心10 min,收集上清,沉淀用8 mol/L尿素溶解,分别对上清和溶解的沉淀进行SDS-PAGE分析,进行sdAb3-Avitag蛋白的可溶性分析。按照Ni-NTA蛋白纯化试剂盒说明书进行重组蛋白sdAb3-Avitag的纯化。
1.5 SdAb3-Avitag融合蛋白的生物素标记将纯化的sdAb3-Avitag融合蛋白透析至10 mmol/L Tris (pH 8.0) 的缓冲液中,测定蛋白浓度后对纳米抗体进行生物素标记,具体操作如下:Buffer A 8.3 μL,Buffer B 8.3 μL,sdAb3-Avitag 50 μg,BirA酶0.84 μL,ddH2O 34.3 μL,混匀后置于30 ℃恒温条件下反应30 min,反应完成后加入等体积的丙三醇,混匀后分装,命名为sdAb3-Biotin,放置-20 ℃保存。
1.6 SdAb3-Biotin活性分析利用间接ELISA方法和Western blotting方法鉴定sdAb3-Biotin的生物学活性。间接ELISA方法:(1) 抗原包被。将截短的PEDV S1蛋白(Asp,aa22-Pro,aa505) 和PEDV N蛋白分别包被于酶标板,100 ng/孔,置于4 ℃包被过夜。(2) 封闭酶标板。将包被液弃去,用PBST洗板4次,200 μL/孔加入1% BSA,37 ℃恒温箱中封闭1 h。(3) 加样。弃去封闭液,用PBST洗板4次,将sdAb3-Biotin分别按1︰100、1︰1 000、1︰2 000、1︰4 000和1︰8 000进行稀释,100 μL/孔加入酶标板中,37 ℃孵育2 h。(4) 孵育酶标抗体。弃去sdAb3-Biotin,用PBST洗板4次,以1︰5 000的比例稀释链霉亲和素-HRP抗体,100 μL/孔加入酶标板中,37 ℃恒温箱中孵育1 h。(5) 显色。弃掉酶标二抗,用PBST洗涤4次,100 μL/孔加入新鲜配制的TMB显色底物,37 ℃恒温箱中避光孵育15 min。(6) 终止。50 μL/孔加入3 mol/L H2SO4终止显色反应,测定OD450。
Western blotting方法:将PEDV S1重组蛋白进行SDS-PAGE后转印至PVDF膜上,转印后的PVDF膜置于5%脱脂奶粉中,25 ℃孵育2 h;随后,将PVDF膜置于用1% BSA按1︰2 500稀释的sdAb3-Biotin中,25 ℃孵育1 h;用PBST洗涤后,对PVDF膜进行孵育于酶标二抗(按照1︰5 000的链霉亲和素-HRP),25 ℃孵育1 h;弃去酶标二抗,用PBST洗涤后曝光,用凝胶成像系统拍照记录。
1.7 抗原包被量与sdAb3-Biotin稀释比例利用棋盘法确定最适抗原包被量与sdAb3-Biotin稀释比例,操作流程如步骤1.6,包被不同量的PEDV S1蛋白,分别为25 ng/孔、50 ng/孔、100 ng/孔和200 ng/孔,sdAb3-Biotin的稀释比例为:1︰500、1︰1 000、1︰2 000、1︰4 000、1︰6 000、1︰8 000和1︰10 000,确定出最适抗原包被量和sdAb3-Biotin的稀释比例。
1.8 最适血清稀释比例采用最适抗原包被量进行包被且封闭后进行bELISA:对酶标板进行封闭,弃去封闭液,用PBST洗涤后,将PEDV阴阳性血清分别按照1︰1、1︰5、1︰10、1︰20、1︰40和1︰80进行稀释后,100 μL/孔加入酶标板中,37 ℃孵育90 min,用PBST洗涤4次,加入最适比例稀释的sdAb3-Biotin,用PBST洗涤后,加入酶标二抗后进行显色,计算各稀释比例阻断率。阻断率PI=(1–阳性血清OD450/阴性血清OD450)×100%,选取阻断率最高的稀释比例作为最适血清稀释比例。
1.9 最适血清孵育时间采用最适包被抗原和最适血清稀释比例的条件下,按照步骤1.8进行bELISA,不同之处将血清分别孵育30 min、60 min、90 min和120 min,计算各组的阻断率,选取阻断率最高的组作为血清最适孵育时间。
1.10 SdAb3-Biotin最适孵育时间采用上述筛选条件进行SdAb3-Biotin的最适孵育时间摸索,将最适稀释比例的SdAb3-Biotin分别孵育30 min、60 min、90 min和120 min,计算各组的阻断率,阻断率最高的作为sdAb3-Biotin最适孵育时间。
1.11 最适链霉亲和素-HRP最佳孵育时间按照上述已确定的条件进行链霉亲和素-HRP的最适孵育时间的筛选,将链霉亲和素-HRP按照1︰5 000稀释后,分别孵育30 min、60 min、90 min和120 min,计算各组的阻断率,阻断率最高的孵育时间为链霉亲和素-HRP最适孵育时间。
1.12 bELISA临界值的确定对90份PEDV阴性血清按照上述确定的各项最适条件进行bELISA,计算90份阴性血清的阻断率,按照阻断率确定bELISA的临界值:临界值A=阴性血清平均PI值+2×标准差(s);临界值B=阴性血清平均PI值+3×标准差(s),计算出临界值A与临界值B,若样品阻断率PI≥临界值B,则判定样品为PEDV抗体阳性;若临界值A<PI<临界值B,则判定为PEDV抗体可疑,需要对样品进行复检;若复检结果仍为可疑,则判定样品为PEDV抗体阴性;若PI≤临界值A,则判定样品为PEDV抗体阴性。
1.13 重复性试验为验证bELISA方法的重复性,选取不同人员在不同时间按照上述bELISA操作流程对7份猪血清进行4次测试,每个样品进行3个重复孔,对检测样品的阻断率进行统计分析来评估bELISA方法的重复性。
1.14 特异性试验为验证bELISA方法的特异性,利用建立的bELSIA方法对实验室保存的其他病原的阳性血清进行检测,包括TGEV、PRRSV、PCV、PRV、PPV和JEV的阳性血清,计算检测样品的阻断率来评估bELISA方法的特异性。
1.15 符合率试验利用建立的bELISA方法和商品化PEDV抗体检测试剂盒分别对54份血清样品进行检测,计算两者的符合率。
2 结果与分析2.1 重组单域抗体sdAb3的表达与纯化将构建的pET21b-sdAb3-Avitag转化至E. coli BL21(DE3) 感受态细胞,利用终浓度为1 mmol/L的IPTG进行诱导表达,结果如图 1A所示,与预期结果一致,在约15 kDa处可见目的蛋白。此外,sdAb3-Avitag重组蛋白以包涵体的形式存在(图 1B)。用Ni-NTA进行纯化后,得到纯度较高的sdAb3-Avitag重组蛋白(图 1C)。
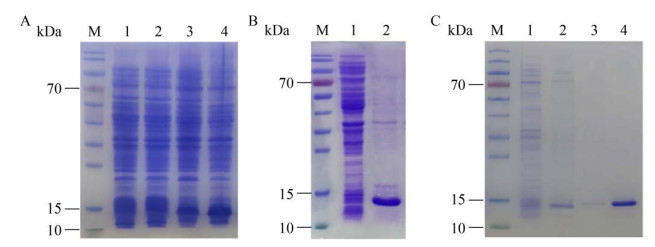 |
| 图 1 SdAb3-Avitag重组蛋白的表达与纯化 Fig. 1 Expression and purification of sdAb3-Avitag recombinant protein. SDS-PAGE showing the expression (A), solubility (B), and purity (C) of the sdAb3-Avitag recombinant protein. |
| 图选项 |
2.2 SdAb3-Biotin活性分析利用间接ELISA和Western blotting对sdAb3生物素标记效果进行了验证。Western blotting结果显示,sdAb3-Biotin能够特异性地检测到S1重组蛋白(图 2A),间接ELISA结果显示,当sdAb3-Biotin稀释到1︰8 000仍能够与S1蛋白进行良好的反应(图 2B),以上表明sdAb3-Biotin具有良好的活性。
 |
| 图 2 SdAb3-Biotin活性分析 Fig. 2 Activity of the sdAb3-Biotin. (A) The PEDV S1 protein was detected with the sdAb3-Biotin by Western-blotting. (B) PEDV S1 protein was detected by the sdAb3-Biotin using indirect ELISA. |
| 图选项 |
2.3 抗原包被量与sdAb3-Biotin稀释比例通过棋盘滴定法进行间接ELISA,结果表明当S1蛋白包被量为200 ng/孔、sdAb3-Biotin稀释比例为1︰8 000时,OD450约为1.0,故将此抗原包被量和sdAb3-Biotin抗体稀释比例作为最适包被量和抗体稀释比例(图 3)。
 |
| 图 3 优化S1蛋白包被量和最适sdAb3-Biotin稀释比例 Fig. 3 Optimization of the S1 coating quantity and the dilution ratio of sdAb3-Biotin. |
| 图选项 |
2.4 最适血清稀释比例将阴阳性对照血清分别按照1︰2、1︰5、1︰10、1︰20、1︰40和1︰80的比例进行稀释后用bELISA进行检测,结果显示,血清在1︰2稀释的情况下具有最高的阻断率(图 4A),故血清最适稀释比例为1︰2。
 |
| 图 4 bELISA最佳条件 Fig. 4 Screening of optimal conditions for bELISA. (A) The serum dilution ratio. (B) The serum incubation time. (C) The sdAb3-Biotin incubation time. (D) The incubation time of streptavidin-HRP on the performance of bELISA. |
| 图选项 |
2.5 最适血清孵育时间基于上述最适条件进行bELISA,摸索最适血清孵育时间,结果显示,血清孵育120 min时具有最高的阻断率(图 4B),故将此作为最适血清孵育时间。
2.6 SdAb3-Biotin最适孵育时间基于上述最适条件进行bELISA,摸索最适sdAb3-Biotin孵育时间,结果显示,孵育时间为30 min时具有最高的阻断率(图 4C),故将此作为最适sdAb3-Biotin孵育时间。
2.7 酶标二抗最适孵育时间基于上述最适条件进行bELISA摸索,最适链霉亲和素-HRP的孵育时间,结果显示,链霉亲和素-HRP按照1︰5 000稀释,孵育时间为30 min时具有最高的阻断率(图 4D),故将此作为酶标二抗最适孵育时间。
2.8 bELISA的临界值对90份已知阴性血清进行了bELISA检测,90份阴性血清的平均PI为7.67%,标准差(s) 为8.98%,因此,当样品的PI≥35%,则判定样品为PEDV抗体阳性;若26%<样品的PI<35%,则判定为PEDV抗体可疑,需要进行重新检测,若重新检测结果仍为可疑,则判定为PEDV抗体阴性;若样品PI≤26%,则判定为PEDV抗体阴性(图 5)。
 |
| 图 5 bELISA的临界值 Fig. 5 The cut-off value of the bELSA. |
| 图选项 |
2.9 bELSIA的重复性为验证bELISA方法的重复性,选取不同人员在不同时间按照上述bELISA操作流程,对7份猪血清进行4次测试,每个样品进行3个重复孔,结果表明,在4次重复试验中,不同人员检测同一份血清样品的阴阳性结果一致(图 6),且CV%均在10%以下,表明该bELISA方法具有良好的重复性。
 |
| 图 6 bELISA的重复性 Fig. 6 Reproducibility of the bELISA. |
| 图选项 |
2.10 bELISA特异性利用建立的bELISA方法对TGEV、PRRSV、PCV、PRV、PPV和JEV阳性血清进行检测,结果表明,除PEDV阳性对照血清,其余病原阳性均不与S1蛋白进行结合,从而不能阻断sdAb3-Biotin与S1蛋白结合。结果表明该bELISA方法表现出良好的特异性(图 7)。
 |
| 图 7 bELISA的特异性 Fig. 7 Specificity of the bELISA. |
| 图选项 |
2.11 bELISA与商品化试剂盒符合率应用商品化试剂盒和本研究建立的bELSIA分别对54份临床猪血清进行检测,经计算,二者的总体符合率可达92.59% (表 1)。
表 1 bELISA与商品试剂盒的符合率评价Table 1 The compliance rate between the bELISA and a commercial ELISA kit
| Methods and determination indexes | Blocking ELISA | Coincidence rate (%) | |||
| Positive serum numbers | Negative serum numbers | Total | |||
| Commercialization ELISA | Positive serum numbers | 16 | 2 | 18 | 88.89 |
| Negative serum numbers | 2 | 34 | 36 | 94.44 | |
| Total | 18 | 36 | 54 | 92.59 | |
表选项
3 讨论由于变异毒株的高流行性,PED已成为威胁世界养猪业的最重要的疫病之一,早期快速的诊断对防控PEDV的传播非常重要[21]。S蛋白是PEDV的主要抗原之一,且含有多个抗原表位,是进行PEDV遗传变异分析和建立ELISA方法的主要靶蛋白[22]。抗体在间接ELISA、竞争ELISA和阻断ELISA等不同形式的ELISA检测方法中起着重要的作用。由于这些方法都是基于PEDV特异性单克隆抗体或多克隆抗体,因此制备、纯化和标记抗体的过程复杂且昂贵[23]。然而,纳米抗体的出现克服了这些问题,与传统的抗体相比,纳米抗体可在传统的原核表达系统和酵母表达系统中进行大量的表达。笔者课题组前期报道的基于PEDV N蛋白纳米抗体建立的bELISA方法[19]和本研究基于S1蛋白纳米抗体建立的bELISA与商品化试剂盒的符合率分别是94%和92.56%,这可能与他们检测PEDV感染后不同时间段的抗体以及抗原的保守性有关。PEDV感染早期时,猪体内首先会产生大量针对N蛋白的抗体,而针对S蛋白的抗体持续时间长于N蛋白[5, 24]。此外,几种基于不同病毒纳米抗体建立的ELISA方法已被用于病毒血清学检测,包括NDV和SIV。
生物素-链霉亲和素体系是一种非常有效的生物反应扩增体系,一个链霉亲和素可以特异性结合4个生物素分子,起到放大信号的作用,从而提高反应的灵敏度[25]。生物素-链霉亲和素体系由于其高特异性和强亲和力,已被广泛应用于各种物质的检测。此外,生物素的分子量只有244.31 Da,是HRP分子量的1/160[26]。因此,用生物素标记蛋白时,蛋白的生物活性基本不受影响。考虑到纳米抗体的分子量小,生物素标记可以最大限度地保留其结构和功能。以生物素化的sdAb5为基础,建立了一种特异性、敏感性和重现性良好的检测猪血清中SIV抗体的ELISA方法。考虑到纳米体在诊断开发中的应用日益增多,本研究以S1蛋白的特异性单域抗体sdAb3为基础建立了一种具有良好的特异性、灵敏度、重复性和低成本的bELISA方法,其中,sdAb3可通过原核表达系统进行大量表达,从而能够大幅度降低检测成本,有望用于临床实践中。
总之,本研究通过对PEDV S1蛋白的特异性单域抗体sdAb3进行生物素化标记,以生物素化sdAb3为基础建立了bELISA方法并对临床54份样品进行检测,检测结果与商品化试剂盒检测结果符合率为92.56%。该bELISA方法具有良好的特异性、灵敏性与重复性,具有省时、低成本的优势,在生产实践中具有较高的应用价值。本研究为间接诊断PEDV的感染和监测猪群中PEDV疫苗免疫水平提供了一种新的方法。
参考文献
| [1] | Sun RQ, Cai RJ, Chen YQ, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China. Emerg Infect Dis, 2012, 18(1): 161-163. DOI:10.3201/eid1801.111259 |
| [2] | Ge FF, Yang DQ, Li X, et al. Novel method for isolation of porcine epidemic diarrhea virus with the use of suspension vero cells and immunogenicity analysis. J Clin Microbiol, 2021, 59(2): e02156-20. |
| [3] | Li DL, Feng H, Liu YC, et al. Molecular evolution of porcine epidemic diarrhea virus and porcine deltacoronavirus strains in Central China. Res Vet Sci, 2018, 120: 63-69. DOI:10.1016/j.rvsc.2018.06.001 |
| [4] | Lin HX, Zhou H, Gao L, et al. Development and application of an indirect ELISA for the detection of antibodies to porcine epidemic diarrhea virus based on a recombinant spike protein. BMC Vet Res, 2018, 14(1): 243. DOI:10.1186/s12917-018-1570-5 |
| [5] | Chae C, Kim O, Choi C, et al. Prevalence of porcine epidemic diarrhoea virus and transmissible gastroenteritis virus infection in Korean pigs. Vet Rec, 2000, 147(21): 606-608. DOI:10.1136/vr.147.21.606 |
| [6] | Wood EN. Transmissible gastroenteritis and epidemic diarrhoea of pigs. Br Vet J, 1979, 135(4): 305-314. DOI:10.1016/S0007-1935(17)32831-2 |
| [7] | Diel DG, Lawson S, Okda F, et al. Porcine epidemic diarrhea virus: an overview of current virological and serological diagnostic methods. Virus Res, 2016, 226: 60-70. DOI:10.1016/j.virusres.2016.05.013 |
| [8] | Liu GP, Jiang YH, Opriessnig T, et al. Detection and differentiation of five diarrhea related pig viruses utilizing a multiplex PCR assay. J Virol Methods, 2019, 263: 32-37. DOI:10.1016/j.jviromet.2018.10.009 |
| [9] | Okda F, Liu XD, Singrey A, et al. Development of an indirect ELISA, blocking ELISA, fluorescent microsphere immunoassay and fluorescent focus neutralization assay for serologic evaluation of exposure to North American strains of porcine epidemic diarrhea virus. BMC Vet Res, 2015, 11: 180. DOI:10.1186/s12917-015-0500-z |
| [10] | Fan JH, Zuo YZ, Shen XQ, et al. Development of an enzyme-linked immunosorbent assay for the monitoring and surveillance of antibodies to porcine epidemic diarrhea virus based on a recombinant membrane protein. J Virol Methods, 2015, 225: 90-94. DOI:10.1016/j.jviromet.2015.07.021 |
| [11] | Carvajal A, Lanza I, Diego R, et al. Evaluation of a blocking ELISA using monoclonal antibodies for the detection of porcine epidemic diarrhea virus and its antibodies. J Vet Diagn Invest, 1995, 7(1): 60-64. DOI:10.1177/104063879500700109 |
| [12] | Fan BC, Sun J, Zhu L, et al. Development of a novel double antibody sandwich quantitative enzyme-linked immunosorbent assay for detection of porcine epidemic diarrhea virus antigen. Front Vet Sci, 2020, 7: 540248. DOI:10.3389/fvets.2020.540248 |
| [13] | Hamers-Casterman C, Atarhouch T, Muyldermans S, et al. Naturally occurring antibodies devoid of light chains. Nature, 1993, 363(6428): 446-448. DOI:10.1038/363446a0 |
| [14] | Che T, English J, Krumm BE, et al. Nanobody-enabled monitoring of kappa opioid receptor states. Nat Commun, 2020, 11(1): 1145. DOI:10.1038/s41467-020-14889-7 |
| [15] | Lu QZ, Li XX, Zhao JK, et al. Nanobody- horseradish peroxidase and -EGFP fusions as reagents to detect porcine parvovirus in the immunoassays. J Nanobiotechnol, 2020, 18(1): 7. DOI:10.1186/s12951-019-0568-x |
| [16] | Demine S, Ribeiro RG, Thevenet J, et al. A nanobody-based nuclear imaging tracer targeting dipeptidyl peptidase 6 to determine the mass of human beta cell grafts in mice. Diabetologia, 2020, 63(4): 825-836. DOI:10.1007/s00125-019-05068-5 |
| [17] | Sheng YM, Wang K, Lu QZ, et al. Nanobody-horseradish peroxidase fusion protein as an ultrasensitive probe to detect antibodies against Newcastle disease virus in the immunoassay. J Nanobiotechnol, 2019, 17(1): 35. DOI:10.1186/s12951-019-0468-0 |
| [18] | Du TF, Zhu G, Wu XP, et al. Biotinylated single-domain antibody-based blocking elisa for detection of antibodies against swine influenza virus. Int J Nanomedicine, 2019, 14: 9337-9349. DOI:10.2147/IJN.S218458 |
| [19] | Ma ZQ, Wang TY, Li ZW, et al. A novel biotinylated nanobody-based blocking ELISA for the rapid and sensitive clinical detection of porcine epidemic diarrhea virus. J Nanobiotechnol, 2019, 17(1): 96. DOI:10.1186/s12951-019-0531-x |
| [20] | Ji PP, Zhu JH, Li XX, et al. Fenobody and RANbody-based sandwich enzyme-linked immunosorbent assay to detect Newcastle disease virus. J Nanobiotechnol, 2020, 18(1): 44. DOI:10.1186/s12951-020-00598-2 |
| [21] | Shan Y, Liu Y, Liu Z, et al. Development and application of an indirect enzyme-linked immunosorbent assay using recombinant S1 for serological testing of porcine epidemic diarrhea virus. Can J Microbiol, 2019, 65(5): 343-352. DOI:10.1139/cjm-2018-0240 |
| [22] | Liu JB, Cong GY, Shi HY, et al. Neutralizing monoclonal antibody fails to recognize porcine epidemic diarrhea virus with two regions deleted in spike protein. Veterin Microbiol, 2020, 251: 108917. DOI:10.1016/j.vetmic.2020.108917 |
| [23] | Gerber PF, Lelli D, Zhang JP, et al. Diagnostic evaluation of assays for detection of antibodies against porcine epidemic diarrhea virus (PEDV) in pigs exposed to different PEDV strains. Prev Vet Med, 2016, 135: 87-94. DOI:10.1016/j.prevetmed.2016.11.005 |
| [24] | Knuchel M, Ackermann M, Müller HK, et al. An ELISA for detection of antibodies against porcine epidemic diarrhoea virus (PEDV) based on the specific solubility of the viral surface glycoprotein. Veterin Microbiol, 1992, 32(2): 117-134. DOI:10.1016/0378-1135(92)90100-8 |
| [25] | Gong X, Zhu M, Li GH, et al. Specific determination of influenza H7N2 virus based on biotinylated single-domain antibody from a phage-displayed library. Anal Biochem, 2016, 500: 66-72. DOI:10.1016/j.ab.2015.09.020 |
| [26] | Diamandis EP, Christopoulos TK. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin Chem, 1991, 37(5): 625-636. DOI:10.1093/clinchem/37.5.625 |
