

扬州大学 表观遗传学与表观基因组学研究所,江苏 扬州 225009
收稿日期:2019-05-08;接收日期:2019-07-08;网络出版时间:2019-07-23
基金项目:国家自然科学基金(No. 81773013)资助
摘要:规律成簇间隔的短回文序列(Clustered regularly interspaced short palindromic repeats,CRISPR)是细菌和古菌中的获得性免疫系统,利用该系统能定点进行基因编辑。最近,科学家发现了新的CRISPR-associated (Cas)蛋白,其中由Cas12a介导的基因编辑能显著降低脱靶率。文中对CRISPR/Cas系统的发现历史、组成和分类、工作原理进行概述,并总结了该系统的最新研究进展及在斑马鱼Danio rerio中的应用。
关键词:CRISPR/Cas基因编辑斑马鱼
Research progress and applications of gene editing technology CRISPR/Cas in zebrafish
Juan Ouyang, Songlei Xue, Qiqi Zhou, Hengmi Cui


Institute of Epigenetics & Epigenomics, Yangzhou University, Yangzhou 225009, Jiangsu, China
Received: May 8, 2019; Accepted: July 8, 2019; Published: July 23, 2019
Supported by: National Natural Science Foundation of China (No. 81773013)
Corresponding author: Hengmi Cui. Tel: +86-514-87990309; E-mail: hmcui@yzu.edu.cn.
Abstract: Clustered regularly interspaced short palindromic repeats (CRISPR) are acquired immune system in bacteria and archaea. This system is used in site-directed gene editing. Recently, scientists discovered new CRISPR-associated (Cas) proteins, in which Cas12a-mediated gene editing can significantly reduce the off-target rate. In this article, we review CRISPR/Cas system's discovery of history, composition, classification, and working principle. The latest research progress of the CRISPR/Cas system, and its application in zebrafish are introduced.
Keywords: CRISPR/Casgene editingzebrafish
1970年,DNA重组技术的发现标志着生物学进入新的发展阶段。20年后,科学家们又发现基因组DNA损伤修复可以通过DNA双链断裂而触发。基因组DNA发生损伤后在不同条件下分别以同源重组(Homologous recombination,HR)或非同源末端链接(Non-homologous end joining,NHEJ)进行修复。在基因组修复过程中,可能引起碱基插入或缺失,当存在外源片段时还能引入片段替换。目前存在3种基因编辑技术,包括锌指蛋白核酸酶(Zinc-finger nuclease,ZFN)技术、转录激活样效应因子核酸酶(Transcription activator-like effector nuclease,TALEN)和规律成簇间隔短回文重复序列及相关蛋白(CRISPR/Cas)技术。CRISPR/Cas核酸酶技术由成熟的CRISPR RNA (crRNA)与反式活性的tracrRNA以碱基互补配对的方式,形成拥有二级结构的RNA,它能够指导该系统中相关的Cas9蛋白特异性识别靶标DNA,从而引发DNA双链断裂。在核酸内切酶、两条RNA链的参与下能对特异性DNA链进行程序性编辑[1]。目前CRISPR/Cas技术已经涉及人类疾病研究、模式生物模型构建、微生物和植物等研究领域[2]。本文着重介绍CRISPR/Cas系统的最新研究进展,并简介该系统在斑马鱼研究中的应用情况。
1 CRISPR/Cas系统的发现历史1987年,Ishino等[3]在研究大肠杆菌中与碱性磷酸酶同功酶转换相关的iap基因时,发现该基因的具体位置,及基因两侧序列的特点即下游存在29 nt的重复序列。2000年,Mojica等[4]通过数理统计分析最终将间隔重复序列定义为一类独特重复家族。Ruud等[5]提出了以首字母缩略词CRISPR对微生物基因组进行描述,将系统分为3类(Ⅰ–Ⅲ),发现了不同CRISPR系统之间存在的差异,例如CRISPR介导的Ⅲ-A型系统中Cas酶,最终不是靶向RNA序列[6]。科学家提出CRISPR序列作为免疫记忆的防御机制,可能有防止噬菌体感染等功能[7]。研究表明,CRISPR序列被转录并转化成包含独立间隔子shorter CRISPR RNAs (crRNAs)指导Cas核酸酶活性[8],Cas酶能够获取间隔序列,间隔序列能对噬菌体进行防御[9-10];此外CRISPR的形成涉及DNA的降解过程[11]。Hélène等[12]通过分析20种突变型噬菌体,发现噬菌体突变后,原本细菌Protospacer-adjacent motif (PAM)序列中的NGG就不能定位,从而导致CRISPR系统不能发挥作用。CRISPR系统中通过crRNA的碱基互补配对,不仅能够进行特异性靶向,而且能防止自身染色体被切断[13]。该系统机理研究例如抗噬菌体菌株筛选和培养[14]等,促进了其在生命科学领域的应用。同时,该系统的应用也使更多科研人员对系统进行更为深入的研究。Garneau等[15]发现Cas9是Cas中独特的由酶介导切割靶标DNA序列的基因簇。系统中的trans-activating crRNA (tracrRNA)可以通过广泛保守的核酸内切酶和CRISPR相关蛋白Csn1靶向突变的crRNA[16]。不久,Rimantas等[17]通过实验表明嗜热链球菌中的CRISPR/Cas系统能转移至大肠杆菌中发挥免疫作用,抵御外源噬菌体的入侵,这一机制有利于细菌间交流,增强细菌对噬菌体的防御功能。嗜热链球菌中的CRISPR/Cas系统,除能在细菌间进行转移,其中Cas9-crRNA复合物还能在体外通过DNA特异性位点与crRNAs互补配对从而引发双键断裂,发挥切割目的DNA序列的作用[18]。研究人员根据靶标基因,成功设计多个guide RNA (gRNA),提高了基因编辑效率[19]。此后,CRISPR/Cas9系统逐渐成为研究人员青睐的基因编辑工具。
2 CRISPR基因座结构组成及系统分类与之前的基因编辑技术相比,CRISPR基因座结构较为简单。因CRISPR/Cas系统的信息加工和处理机制上存在一定差异,据此将CRISPR/Cas系统分为3型,分别为TypeⅠ、TypeⅡ和Type Ⅲ。
TypeⅠ系统需要多种蛋白参与才能形成具有切割功能的蛋白复合物[19]。该系统中CRISPR相关的复合物中的碱基序虽相似性不明显,但是其结构相似度高[20]。
TypeⅡ系统中所包含的Cas蛋白数量比TypeⅠ少,发挥作用的主要蛋白质分子是Cas9,Cas9蛋白拥有Ruv和HNH两种核酸酶结构域。Type ⅡA系统中,产脓链球菌Streptococcus pyogenes基因座结构见图 1。
 |
| 图 1 产脓链球菌中CRISPR系统ⅡA型基因座结构[21] Fig. 1 CRISPR locus organization from Streptococcus pyogenes type ⅡA CRISPR systems[21]. |
| 图选项 |
Type Ⅲ系统与TypeⅠ类似,都是多亚基的蛋白。该系统中作用的主要是Cas10蛋白分子,该蛋白同时具有RNA酶活性以及与TypeⅠ中复合物类似的功能[22]。
上述3种CRISPR/Cas系统分别拥有其自身特点,同时也存在相似之处。
3 CRISPR/Cas系统的作用原理TypeⅡ型CRISPR/Cas9系统较其他两种而言,相对简单,主要由重复间隔序列、编码Cas9核酸酶序列和PAM组成[23]。行使功能时主要包括3个阶段:适应阶段、CRISPR/Cas9表达和初级转录阶段以及免疫阶段[15]。
第一阶段,当外界的噬菌体或DNA入侵细菌时,细菌会启动其自身的防御机制。由PAM指导gRNA与目标DNA序列结合,从而促进Cas9蛋白结合[24]。其中,Cas9空间结构的变化有利于Cas9与靶标序列结合,发挥切割功能[25]。
第二阶段,CRISPR/Cas9基因进行表达。CRISPR序列在RNA聚合酶的帮助下进行转录,然后被剪切为成熟crRNAs。最后,成熟crRNAs与tracrRNAs结合,形成双链RNA也称向导RNA,发挥导向功能。
第三阶段,在向导RNA引导Cas9蛋白与靶标序列相互作用,引发切割,产生双链断裂(Double-strand breaks,DSBs),从而引发自身修复,包括同源重组修复和非同源末端连接修复[26]。
以上3个阶段作为一个整体,在原核生物机体中发挥免疫功能。此外,这3个阶段又能分别作为一个独立单元发挥作用。
4 CRISPR/Cas9最新研究进展随着CRISPR系统的广泛应用,科研工作者也继续深入对系统进行研究。2014年,O’Connell等[27]科学家证明Streptococcus pyogenes Cas9 (SpCas9)可以切割单链RNA (ssRNA)。在此之前,大多数科研工作者认为Cas9只能靶向DNA,该研究结果表明CRISPR系统中PAM序列以寡链核苷酸(PAM-presenting oligonucleotides,PAMmer)形式存在时,能提高Cas9与ssRNA亲和力,进而发挥基因编辑功能。相比原来Cas9对基因组DNA进行编辑,这一系统能直接编辑ssRNA,使编辑更具高效性。该系统在应用时需要注意PAMmer序列容易降解[28-29],所以应尽可能缩短操作时间。除上述系统中Cas9外,科研人员还发现TypeⅡ型CRISPR/Cas系统中另一种新型核酸内切酶Cas12a (Cpf1)。Cpf1蛋白具有以下优点:Cpf1较小,更容易进入所研究对象中进行切割;Cpf1通过切割作用,引导crRNA成熟[30];Cpf1有两个功能域,RuvC切割双链DNA,Nuc主要起辅助作用[31];Cpf1不仅能对PAM中富含T的序列进行编辑[31-32],还能对PAM远端序列进行切割[33];Cpf1发挥切割作用后切口处留下互补黏性末端,能提高DNA插入的可控性同时提高插入效率[31];Cpf1能够靶向ssDNA,并将其完全降解[34];Cpf1脱靶率比Cas9蛋白脱靶率低[35]。研究表明,在某些菌株中SpCas9没有功能,Cpf1则能在该菌株中通过同源重组介导高效的基因敲入和敲除,弥补了SpCas9在编辑对象上的局限(FnCas9和FnCpf1的CRISPR基因座结构及结构域的比较如图 2),拓展了CRISPR/Cas系统的应用范围[36]。此外,研究人员还利用Cpf1建立DNA endonuclease-targeted CRISPR trans reporter (DETECTR)系统,用来快速准确地检测人乳头瘤病毒,为临床应用提供新型平台[34]。
上述系统进行基因编辑时都需要通过PAM序列定位识别,研究表明存在一种不依赖PAM序列,且能对靶标RNA发挥识别作用的Francisella novicidaCas9 (FnCas9)蛋白[37]。起初是科学家在研究细菌脂蛋白对抗噬菌体反应时发现该系统能引发天然免疫。这个过程主要是通过small CRISPR/Cas associated RNA (scaRNA)来抑制细菌脂蛋白内源转录物,当时研究人员发现CRISPR/FnCas9系统对于抑制细菌脂蛋白非常重要[38]。之后,有研究结果表明FnCas9蛋白可以通过crRNA和tracrRNA相互作用来识别5′-NGG-3′ PAM结构,切割双链DNA (dsDNA)[39]。Price等[37]对该系统进行改造研究,他们把人肝癌细胞中丙肝病毒基因失活,最后病毒蛋白的表达呈现下降趋势,下降约60%。但系统定点编辑RNA的机制还有待进一步研究[39]。不久,有研究人员将该系统用于植物抗病毒研究,发现FnCas9有利于增强植物对ssDNA、RNA病毒的抵抗力[40-41]。FnCas9在植物抗病领域显现出较大优势,进一步扩展了CRISPR/Cas系统的应用领域。
2016年,Abudayyeh等[42]在研究纤毛菌Leptotrichia shahii时发现在特异性RNA引导下,该细菌中Cas13a (LshCas13a)蛋白能发挥抵御噬菌体入侵的功能。不久,他们又在纤毛菌L. wadei中发现另外一种Cas13a蛋白(LwaCas13a)[43]。LwaCas13a蛋白通过多个靶向crRNA对多个位点进行编辑,它在CRISPR系统属于二类CRISPR/Cas中的typeType Ⅵ-A类型。这种新型Cas13a具有以下特点,Cas13a是在前间隔区侧翼位点(Protospacer flanking site,PFS)的存在下,由crRNA指导行使功能[44];Cas13a蛋白的靶标为单链RNA时,可以直接利用crRNA发挥作用[45];Cas13a蛋白中包含两个HEPN结构域,该结构域中Arg (精氨酸)和His (组氨酸)突变可形成dead Cas13a (dCas13a),该蛋白与dead Cas9 (dCas9)作用类似,起结合靶标作用;Cas13a蛋白还拥有两个RNase活性中心。与之前发现的(LshCas13a)以及其他Cas13a相比,LwaCas13a的切割活性最高。随着对Cas13a基础研究的深入,科研工作者开始将其应用于生命科学领域,例如作为哺乳动物[43]和植物细胞[46]中特异性敲低工具;灵敏监测细胞中的转录产物[47];依靠酶报告解锁的高灵敏监测系统(Specific High-Sensitivity Enzymatic Reporter Unlocking,SHERLOCK)[48]以及后续建立的SHERLOCK version 2 (SHERLOCKv2)[49]系统,以上系统能对病毒进行检测。最近也有研究表明Cas13a能快速准确检测病毒包括人类RNA病毒[50]、埃博拉病毒[51]和H7N9病毒[52]等。
 |
| 图 2 Francisella novicida U112中FnCas9和FnCpf1的CRISPR基因座结构及结构域的比较[31] Fig. 2 Organization of two CRISPR loci found in Francisella novicida U112 (NC_008601)[31]. The domain architectures of FnCas9 and FnCpf1 are compared. |
| 图选项 |
除以上主要几种Cas蛋白外,研究人员还发现几种其他Cas蛋白。例如含有RNA引导的RNA酶Cas13b[53],这种CRISPR/Cas系统缺少Cas1、Cas2,其基本作用机制与Cas13a一致。不过值得注意的是,Cas13b工作时需要靶点处存在PFS序列,这样有利于提高Cas13b特异性切割。CRISPR/Cas13b系统中存在两种蛋白Csx27与Csx28,它们分别起着抑制和加强Cas13b发挥识别和切割作用[53-54]。此外,科学家鉴定出VI-D型dCas13[55-56],它能特异靶向RNA。其中来自黄色瘤胃球菌Ruminococcus flavefaciens XPD3002 (CasRx)的核酸酶效应因子是一种可编程的RNA结合模块,可有效靶向细胞RNA,为转录组工程和未来的治疗开发提供了一个通用平台。另外,研究人员通过构建突变型Bacillus hisashii Cas12b (BhCas12b)从而提高基因编辑效率[57]。
2019年,CRISPR/cas技术在较多方面都取得进展。除传统基因编辑工具,Liu等[58]研究发现一种新型基因编辑工具CasX,它以独特的存在形式结合双链DNA对其进行切割,使用低温电子显微镜发现CasX拥有RNA支架和DNA解旋结构域等特殊结构,不过CasX的具体工作原理还需要科学家继续深入研究。另外,一种新型抗CRISPR蛋白也开始进入研究人员视野。已有研究表明,可通过抑制Cas9的组装来调节抗CRISPR蛋白的活性[59]。传统CRISPR/Cas系统主要是依靠细菌中免疫防御机制来维持外源基因整合,现在研究人员发现了转座子也可以编码CRISPR/Cas系统[60]。另外,研究还发现PAM与Cas9存在多种相互作用机制,这一发现也为系统的应用提供理论基础[61]。研究人员在研究该系统的应用中,发现在果蝇某一类细胞中Cas9蛋白表达受到抑制。这一结构有利于揭示发育神经元的重塑机制[62]。
综上,CRISPR/Cas系统在研究人员的努力探索下,一直在不断发展和完善。主要在以下几个方面有所突破:1)系统的靶向性。刚开始发现该系统的靶向序列是DNA[13],随后发现Cas酶在不同情况下,靶向性存在差异,例如可以靶向dsDNA和ssRNA[39]等。2)系统的定位。从一开始依靠PAM序列中NGG进行定位[13],到不仅能对PAM中富含T的序列进行编辑[31-32],还能对PAM远端序列进行切割[31];在特殊情况下,还存在一种不依赖PAM序列发挥识别作用的蛋白[34];另外还能依赖PFS进行定位[39]。3)系统编辑的效率和脱靶率。主要通过发现新型Cas酶具有不同编辑原理[33]以及对原有的Cas酶进行改造从而提高编辑效率降低脱靶率[57]。4)系统的应用领域。从用于研究基因功能发展至用于植物抵抗病毒[40-41]、临床疾病检测[52]等方面。由此,我们可知CRISPR/Cas系统拥有巨大的发展潜力,在科研工作者共同努力下,能将CRISPR/Cas系统应用于更多的领域。
5 CRISPR/Cas9在斑马鱼中应用随着科研人员对CRISPR/Cas9系统的深入研究,该技术得到大力发展。CRISPR/Cas9技术开始在斑马鱼、小鼠等模式生物中应用。通过对目标物种进行定向基因编辑,提高诱变突变率,获得可遗传的基因编辑个体。斑马鱼是一种模式生物,拥有较小个体、方便饲养、较强的繁殖能力、受精过程和整个发育过程都在体外完成、受精卵透明等优点。1970年,美国遗传学家Streisinger开始对斑马鱼发育和遗传方面进行研究[63]。之后,Westerfield总结斑马鱼相关实验,并整理成书[64]。随着对斑马鱼基础研究的深入,加上测序技术的发展,研究人员发现斑马鱼的基因与人类基因高度相似,开始建立斑马鱼疾病模型。
斑马鱼作为水生生物中的模式生物,科学家开始对其进行研究,在方法优化[65]、细胞实验[66]和模型构建等多个方面已取得预期效果。2013年,Hwang等[67]首次将CRISPR/Cas9技术成功在斑马鱼上应用,通过将Cas9和sgRNA的mRNA混合,注射至一细胞期的斑马鱼胚胎中,后期检测出斑马鱼中存在插入或删除突变。此外,研究中还发现当注射的Cas9和sgRNA处于最佳比例时,能提高突变效率。相比之前的ZFN和TALEN,该技术能够高效进行基因编辑。不过,该技术在应用中也存在脱靶的现象,在后续研究中需要尽可能在降低脱靶率的前提下进行操作。例如在设计靶点时,尽可能选择基因组上特异性高的序列从而降低脱靶率。同年,有研究团队[68]也成功将这一技术应用于斑马鱼基因编辑中(图 3),实验中选取多个基因进行基因编辑,都获得理想的结果。此外,他们对当时基因编辑领域提出的另一论断进行验证,即在哺乳动物和斑马鱼胚胎中DNA双链断裂后,在single-strand DNA (ssDNA)存在的情况下能进行同源重组。他们团队利用斑马鱼这一模式生物进行实验,验证了其具有可行性。不过实验结果表明进行同源重组后,斑马鱼基因组发生同源重组的区域还存在其他非ssDNA的序列,研究人员认为可能是因为斑马鱼体内基因组进行同源重组时存在其他复杂的机制。
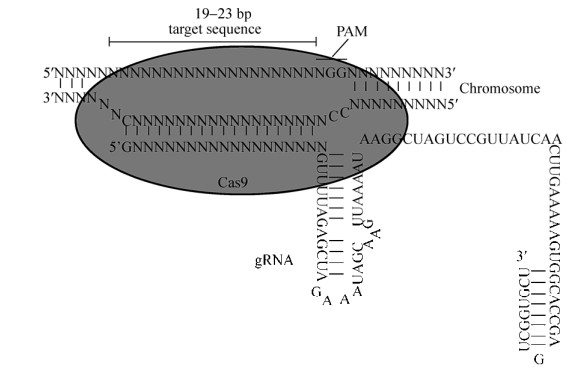 |
| 图 3 用于斑马鱼基因编辑的Cas9和gRNA组成的CRISPR系统[68] Fig. 3 CRISPR system consisting of Cas9 and gRNA for zebrafish gene editing[68]. |
| 图选项 |
该技术相比ZFN和TALEN具有高效、操作方便、经济等优点,但编辑过程中的脱靶效应仍然不可忽视。一方面,可以利用这一新技术对斑马鱼进行基因水平操作,另一方面也需要努力探索降低脱靶的方法。对斑马鱼进行基因编辑时,除脱靶率外,我们还需考虑另外一个提高编辑效率的因素。因为斑马鱼胚胎发育速度快,实验最终目的通常是为了获得生殖系突变的个体,如果对已分裂的斑马鱼胚胎进行基因编辑,CRISPR系统则只能对分裂中部分细胞起作用,所以这一技术对斑马鱼进行基因编辑时,需要确保使用一细胞期斑马鱼胚胎进行实验[69]。
CRISPR/Cas9系统还可以对斑马鱼进行基因敲入操作。研究表明当斑马鱼体内存在DSB位点时,主要的修复方式是NHEJ,这一修复方式能有效促进斑马鱼体内发生同源重组[70]。研究人员[71]起初利用该系统对小鼠进行基因敲入操作,将Cas9和sgRNA的mRNA注射至一细胞期的小鼠胚胎中,成功构建插入外源片段的突变小鼠,降低脱靶率。此外实验设计靶向一个基因的两个靶点进行基因敲除,获得长片段敲除的基因小鼠,为后续该系统的应用提供了方向。使用这一方法对斑马鱼进行基因编辑存在局限,因为基因敲除操作主要是利用Cas9蛋白切割产生DSB,在存在外源供体的情况下,进行同源重组。正如前文中提到斑马鱼胚胎发育快,该系统还未完全发挥作用,斑马鱼胚胎已经开始分裂。小鼠的一细胞期持续时间较长,所以能使用该方法进行基因敲入。之后,有科研人员[72]利用CRISPR/Cas9系统和斑马鱼NHEJ修复原理,成功将含有热休克蛋白启动子的报告基因敲入斑马鱼基因组中。该方法转基因效率高于传统转基因效率,为后续研究斑马鱼基因敲入提供了新思路。但该系统中,基因敲入部位处于顺式作用元件序列附近,会影响内源基因的表达,所以之后的研究应尽可能在减少对内源基因表达的影响的前提下,提高基因敲入效率,此外应当基于实验目的设计实验。例如通过基因插入的方式阻断基因表达,则需要在标签序列后加入终止序列。
曾有研究团队利用格氏嗜盐碱杆菌Natronobacterium gregoryi Argonaute (NgAgo)在斑马鱼体内特异性敲低基因表达,引起表型改变。实验结果表明,该基因的DNA水平并未发生变化,仅mRNA表达降低[73],该系统是否能应用仍然需要进一步研究。另外,有研究人员[74]设计出一种新型基因编辑工具(图 4),即将细菌海床黄杆菌Flavobacterium okeanokoites中的核酸内切酶FokⅠ与片状核酸内切酶(Flap endonucleaseⅠ,FenⅠ)结合,形成一种新型基因编辑工具结构引导的核酸内切酶(Structure-guided endonuclease,SGN),并且将其成功应用于斑马鱼基因编辑中。这一技术的优势在于能进行长片段基因敲除,不足之处则是敲除效率较低,脱靶情况有待进一步研究。
 |
| 图 4 应用于斑马鱼的SNG基因编辑技术[74] Fig. 4 Application of SNG gene editing technology in zebrafish[74]. |
| 图选项 |
现今除构建长片段基因插入的突变模型外,研究人员[75]还能利用改进的CRISPR/Cas9系统在斑马鱼基因组中引入点突变,构建单核苷酸突变疾病模型(图 5)。主要是将CRISPR/Cas9与50 nt的ssDNA模板相结合,共同发挥作用,其中单链DNA模板中包含突变位点和PAM序列。以上系统发挥作用时,该单链不仅能引入特异性突变,而且能够保护斑马鱼基因组中引入的单核苷酸突变。在实验过程中,进行单核苷酸敲入的限制性因素是PAM序列中特异性序列。后续实验中应考虑到Cas9编辑效率、PAM识别兼容性和DNA靶标特异性以及系统脱靶率。
 |
| 图 5 构建斑马鱼单核苷酸突变步骤及各步骤最小时间间隔[75] Fig. 5 Procedure followed to establish the mutation lines and corresponding minimal timeline for each step[75]. |
| 图选项 |
我们实验室的一个研究方向是使用CRISPR/Cas9技术,对斑马鱼基因进行编辑,探究基因功能。2016年,我们团队利用CRISPR/Cas9技术,在Nondret00679基因的启动子序列处设计靶点,通过筛选,获得突变型纯合个体。我们利用该技术,构建能自体表达Cas9蛋白的斑马鱼模型。研究人员利用该模型研究基因功能时,无需注射Cas9表达载体,只需注射靶向目的基因的sgRNA即可达到基因编辑的作用,使特定发育时期组织细胞中能自身表达Cas9,实现基因组织特异性敲除[76]。2018年,我们利用Cas9转基因斑马鱼研究斑马鱼中内源性反转录病毒基因的功能,发现敲除斑马鱼ERV基因可能通过Notch/Delta D信号通路影响斑马鱼发育[77]。目前,我们正利用CRISPR/Cas9系统敲除斑马鱼体内的RNA甲基转移酶。实验中我们发现,可以从F0代中筛选出相同突变类型,将具有相同突变类型的雌雄鱼交配,F1代即可获得纯合突变个体,一定程度上缩短实验时间。此外,有研究人员在鉴定突变的斑马鱼时,根据特定突变类型设计引物,鉴定突变个体[78]。实验过程中,我们也采用此方法简化了鉴定步骤。
6 总结及展望短短30年的时间,从CRISPR序列的发现到将它发展成强大的基因编辑工具,这与科学家孜孜不倦的努力密不可分。水产生物方面,2015年美国FDA允许转基因三文鱼合法出售,它是全球首例获批进入市场进行正常售卖的转基因动物[79]。家畜方面,Qian等[80]研究人员对家猪Susscrofa domestica进行基因编辑,将抑制肌肉生长mstn基因敲除,从而获得一种肌肉含量比基因敲除前更高的家猪。农作物方面,通过基因编辑技术能够更加快速获得高产、抗病能力强的新植株[81]。如果敲除水产生物中抑制肌肉生长的基因,敲入抗病基因,从而获得更高产和抗病性更强的水产品,将极大提高经济效应,促进水产生物的发展。
CRISPR/Cas技术是进行基因水平研究的有利工具,该技术可操作性强,实验投入成本较低,获得突变体频率高,但是该技术的脱靶率问题仍然不容忽视。此外进行基因编辑的生物,如果比野生型更适应环境,一旦该群体进入大自然,可能会对该物种产生影响。另外,2018年11月,“基因编辑婴儿”事件掀起轩然大波,涉及生命伦理[82]、法律[83]等问题。虽然该技术发展十分迅速,但是现阶段仍然处于实验动物阶段,科学工作者不宜将该技术应用于人。不过随着科学研究不断深入,相信研究人员能不断更新完善CRISPR/Cas系统,使该技术能够更加安全、合理、广泛地应用于各个领域。
参考文献
| [1] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [2] | Komor AC, Badran AH, Liu DR. CRISPR-based technologies for the manipulation of eukaryotic genomes. Cell, 2016, 168(1/2): 20-36. |
| [3] | Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol, 1987, 169(12): 5429-5433. DOI:10.1128/JB.169.12.5429-5433.1987 |
| [4] | Mojica FJM, Diez-Villasenor C, Soria E, et al. Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria. Mol Microbiol, 2000, 36(1): 244-246. |
| [5] | Jansen R, Embden JDAV, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol, 2010, 43(6): 1565-1575. |
| [6] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. DOI:10.1126/science.1165771 |
| [7] | Mojica FJM, Díez-Villasenor C, García-Martínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evolut, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [8] | Brouns SJJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964. |
| [9] | Horvath P, Romero DA, Co?té-Monvoisin AC, et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus. J Bacteriol, 2008, 190(4): 1401-1412. DOI:10.1128/JB.01415-07 |
| [10] | Horvath P, Co?té-Monvoisin AC, Romero DA, et al. Comparative analysis of CRISPR loci in lactic acid bacteria genomes. Int J Food Microbiol, 2009, 131(1): 62-70. DOI:10.1016/j.ijfoodmicro.2008.05.030 |
| [11] | Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8): 2551-2561. DOI:10.1099/mic.0.28048-0 |
| [12] | Deveau H, Barrangou R, Garneau JE, et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J Bacteriol, 2008, 190(4): 1390-1400. DOI:10.1128/JB.01412-07 |
| [13] | Marraffini LAM, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity. Nature, 2010, 463(7280): 568-571. DOI:10.1038/nature08703 |
| [14] | Quiberoni A, Moineau S, Rousseau GM, et al. Streptococcus thermophilus bacteriophages. Int Dairy J, 2010, 20(10): 657-664. DOI:10.1016/j.idairyj.2010.03.012 |
| [15] | Garneau JE, Dupuis Mè, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature, 2010, 468(7320): 67-71. DOI:10.1038/nature09523 |
| [16] | Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [17] | Sapranauskas R, Gasiunas G, Fremaux C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Res, 2011, 39(21): 9275-9282. DOI:10.1093/nar/gkr606 |
| [18] | Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci USA, 2012, 109(39): E2579-E2586. DOI:10.1073/pnas.1208507109 |
| [19] | Liu L, Fan XD. CRISPR-Cas system: a powerful tool for genome engineering. Plant Mol Biol, 2014, 85(3): 209-218. DOI:10.1007/s11103-014-0188-7 |
| [20] | Haurwitz RE, Jinek M, Wiedenheft B, et al. Sequence- and structure-specific RNA processing by a CRISPR endonuclease. Science, 2010, 329(5997): 1355-1358. DOI:10.1126/science.1192272 |
| [21] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [22] | Anantharaman V, Iyer LM, Aravind L. Presence of a classical RRM-fold palm domain in Thg1-type 3'- 5'nucleic acid polymerases and the origin of the GGDEF and CRISPR polymerase domains. Biol Direct, 2010, 5: 43. DOI:10.1186/1745-6150-5-43 |
| [23] | Chylinski K, Makarova KS, Charpentier E, et al. Classification and evolution of type Ⅱ CRISPR-Cas systems. Nucleic Acids Res, 2014, 42(10): 6091-6105. DOI:10.1093/nar/gku241 |
| [24] | Shah SA, Erdmann S, Mojica FJM, et al. Protospacer recognition motifs: mixed identities and functional diversity. RNA Biol, 2013, 10(5): 891-899. DOI:10.4161/rna.23764 |
| [25] | Sternberg SH, Redding S, Jinek M, et al. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507(7490): 62-67. DOI:10.1038/nature13011 |
| [26] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [27] | O'Connell MR, Oakes BL, Sternberg SH, et al. Programmable RNA recognition and cleavage by CRISPR/Cas9. Nature, 2014, 516(7530): 263-266. DOI:10.1038/nature13769 |
| [28] | Kleinstiver BP, Tsai SQ, Prew MS, et al. Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells. Nat Biotechnol, 2016, 34(8): 869-874. DOI:10.1038/nbt.3620 |
| [29] | Nelles DA, Fang MY, O'Connell MR, et al. Programmable RNA tracking in live cells with CRISPR/Cas9. Cell, 2016, 165(2): 488-496. DOI:10.1016/j.cell.2016.02.054 |
| [30] | Fonfara I, Richter H, Bratovi? M, et al. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature, 2016, 532(7600): 517-521. DOI:10.1038/nature17945 |
| [31] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a Class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [32] | Shmakov S, Abudayyeh OO, Makarova KS, et al. Discovery and functional characterization of diverse class 2 CRISPR-cas systems. Mol Cell, 2015, 60(3): 385-397. DOI:10.1016/j.molcel.2015.10.008 |
| [33] | Yang H, Gao P, Rajashankar KR, et al. PAM-dependent target DNA recognition and cleavage by C2c1 CRISPR-Cas endonuclease. Cell, 2016, 167(7): 1814-1828. DOI:10.1016/j.cell.2016.11.053 |
| [34] | Chen JS, Ma EB, Harrington LB, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science, 2018, 360(6387): 436-439. DOI:10.1126/science.aar6245 |
| [35] | Kim D, Kim J, Hur JK, et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nat Biotechnol, 2016, 34(8): 863-868. DOI:10.1038/nbt.3609 |
| [36] | Yeo WL, Heng E, Tan LL, et al. Characterization of Cas proteins for CRISPR-Cas editing in streptomycetes. bioRxiv, 2019, 526996. |
| [37] | Price AA, Sampson TR, Ratner HK, et al. Cas9-mediated targeting of viral RNA in eukaryotic cells. Proc Natl Acad Sci USA, 2015, 112(19): 6164-6169. DOI:10.1073/pnas.1422340112 |
| [38] | Sampson TR, Saroj SD, Llewellyn AC, et al. A CRISPR/Cas system mediates bacterial innate immune evasion and virulence. Nature, 2013, 497(7448): 254-257. DOI:10.1038/nature12048 |
| [39] | Hirano H, Gootenberg J, Horii T, et al. Structure and engineering of Francisella novicida Cas9. Cell, 2016, 164(5): 950-961. DOI:10.1016/j.cell.2016.01.039 |
| [40] | Green JC, Hu JS. Editing plants for virus resistance using CRISPR-Cas. Acta Virol, 2017, 61(2): 138-142. DOI:10.4149/av_2017_02_02 |
| [41] | Zhang T, Zheng QF, Yi X, et al. Establishing RNA virus resistance in plants by harnessing CRISPR immune system. Plant Biotechnol J, 2018, 16(8): 1415-1423. DOI:10.1111/pbi.12881 |
| [42] | Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [43] | Abudayyeh OO, Gootenberg JS, Essletzbichler P, et al. RNA targeting with CRISPR-Cas13. Nature, 2017, 550(7675): 280-284. DOI:10.1038/nature24049 |
| [44] | Liu L, Li XY, Ma JQ, et al. The molecular architecture for RNA-guided RNA cleavage by Cas13a. Cell, 2017, 170(4): 714-726. DOI:10.1016/j.cell.2017.06.050 |
| [45] | East-Seletsky A, O'Connell MR, Burstein D, et al. RNA targeting by functionally orthogonal Type Ⅵ-A CRISPR-Cas enzymes. Mol Cell, 2017, 66(3): 373-383. DOI:10.1016/j.molcel.2017.04.008 |
| [46] | Aman R, Ali Z, Butt H, et al. RNA virus interference via CRISPR/Cas13a system in plants. Genome Biol, 2018, 19: 1. DOI:10.1186/s13059-017-1381-1 |
| [47] | East-Seletsky A, O'Connell MR, Knight SC, et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection. Nature, 2016, 538(7624): 270-273. DOI:10.1038/nature19802 |
| [48] | Gootenberg JS, Abudayyeh OO, Lee JW, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321 |
| [49] | Gootenberg JS, Abudayyeh OO, Kellner MJ, et al. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6. Science, 2018, 360(6387): 439-444. DOI:10.1126/science.aaq0179 |
| [50] | Hadidi A. Next-generation sequencing and CRISPR/Cas13 editing in viroid research and molecular diagnostics. Viruses, 2019, 11(2): 120. DOI:10.3390/v11020120 |
| [51] | Qin PW, Park M, Alfson K, et al. Rapid and fully microfluidic ebola virus detection with CRISPR-Cas13a. ACS Sens, 2019, 4(4): 1048-1054. DOI:10.1021/acssensors.9b00239 |
| [52] | Liu YF, Xu HP, Liu C, et al. CRISPR-Cas13a nanomachine based simple technology for avian influenza a (H7N9) virus on-site detection. J Biomed Nanotechnol, 2019, 15(4): 790-798. DOI:10.1166/jbn.2019.2742 |
| [53] | Smargon AA, Cox DBT, Pyzocha NK, et al. Cas13b is a type Type Ⅵ-B CRISPR-associated RNA-guided RNase differentially regulated by accessory proteins Csx27 and Csx28. Mol Cell, 2017, 65(4): 618-630. DOI:10.1016/j.molcel.2016.12.023 |
| [54] | Barrangou R, Gersbach CA. Expanding the CRISPR toolbox: targeting RNA with Cas13b. Mol Cell, 2017, 65(4): 582-584. DOI:10.1016/j.molcel.2017.02.002 |
| [55] | Cox DBT, Gootenberg JS, Abudayyeh OO, et al. RNA editing with CRISPR-Cas13. Science, 2017, 358(6366): 1019-1027. DOI:10.1126/science.aaq0180 |
| [56] | Yan WX, Chong SR, Zhang HB, et al. Cas13d is a compact RNA-targeting type Type Ⅵ-A CRISPR effector positively modulated by a WYL-domain-containing accessory protein. Mol Cell, 2018, 70(2): 327-339. DOI:10.1016/j.molcel.2018.02.028 |
| [57] | Strecker J, Jones S, Koopal B, et al. Engineering of CRISPR-Cas12b for human genome editing. NatCommun, 2019, 10: 212. |
| [58] | Liu JJ, Orlova N, Oakes BL, et al. CasX enzymes comprise a distinct family of RNA-guided genome editors. Nature, 2019, 566(7743): 218-223. DOI:10.1038/s41586-019-0908-x |
| [59] | Thavalingam A, Cheng Z, Garcia B, et al. Inhibition of CRISPR-Cas9 ribonucleoprotein complex assembly by anti-CRISPR AcrⅡC2. Nature Communications, 2019, 10(1): 2806. DOI:10.1038/s41467-019-10577-3 |
| [60] | Klompe SE, Vo PLH, Halpin-Healy TS, et al. Transposon-encoded CRISPR-Cas systems direct RNA-guided DNA integration. Nature, 2019, 1. |
| [61] | Hirano S, Abudayyeh OO, Gootenberg JS, et al. Structural basis for the promiscuous PAM recognition by Corynebacterium diphtheriae Cas9. Nature communications, 2019, 10(1): 1968. DOI:10.1038/s41467-019-09741-6 |
| [62] | Meltzer H, Marom E, Alyagor I, et al. Tissue-specific (ts) CRISPR as an efficient strategy for in vivo screening in Drosophila. Nature communications, 2019, 10(1): 2113. DOI:10.1038/s41467-019-10140-0 |
| [63] | Walker C, Streisinger G. Induction of mutations by γ-rays in pregonial germ cells of zebrafish embryos. Genetics, 1983, 103(1): 125-136. |
| [64] | Westerfield M. The zebrafish book: a guide for the laboratory use of zebrafish. 4th ed. Eugene: University of Oregon Press, 2000. |
| [65] | Vejnar CE, Moreno-Mateos MA, Cifuentes D, et al. Optimized CRISPR-Cas9 system for genome editing in Zebrafish. New York: Cold Spring Harbor Laboratory Press, 2016: 113-132. |
| [66] | Hasani A, Molina C. In vitro knock out of inducible cyclicamp early repressor (ICER) in zebrafish cell lines through the use of the CrispR/Cas9 system (LB530). The FASEB Journal, 2014: 28. |
| [67] | Hwang WY, Fu YF, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(3): 227-229. DOI:10.1038/nbt.2501 |
| [68] | Chang NN, Sun CH, Gao L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos. Cell Res, 2013, 23(4): 465-472. DOI:10.1038/cr.2013.45 |
| [69] | Hruscha A, Krawitz P, Rechenberg A, et al. Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish. Development, 2013, 140(24): 4982-4987. DOI:10.1242/dev.099085 |
| [70] | Liu JG, Gong L, Chang CQ, et al. Development of novel visual-plus quantitative analysis systems for studying DNA double-strand break repairs in zebrafish. J Genet Genom, 2012, 39(9): 489-502. DOI:10.1016/j.jgg.2012.07.009 |
| [71] | Yang H, Wang HY, Shivalila CS, et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell, 2013, 154(6): 1370-1379. DOI:10.1016/j.cell.2013.08.022 |
| [72] | Auer TO, Duroure K, De Cian A, et al. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Res, 2014, 24(1): 142-153. DOI:10.1101/gr.161638.113 |
| [73] | Qi JL, Dong ZJ, Shi YW, et al. NgAgo-based fabp11a gene knockdown causes eye developmental defects in zebrafish. Cell Res, 2016, 26(12): 1349-1352. DOI:10.1038/cr.2016.134 |
| [74] | Xu S, Cao SS, Zou BJ, et al. An alternative novel tool for DNA editing without target sequence limitation: the structure-guided nuclease. Genome Biol, 2016, 17: 186. DOI:10.1186/s13059-016-1038-5 |
| [75] | Tessadori F, Roessler HI, Savelberg SMC, et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Dis Models Mechan, 11(10): dmm035469. |
| [76] | Tian JJ. The application of CRISPR/Cas9 system in zebrafish gene editing[D]. Yangzhou: Yangzhou University, 2016 (in Chinese). 田净净. CRISPR/Cas9系统在斑马鱼基因编辑研究中的应用[D].扬州: 扬州大学, 2016. |
| [77] | Yang Z, Chen SH, Xue SL, et al. Generation of Cas9 transgenic zebrafish and their application in establishing an ERV-deficient animal model. Biotechnol Lett, 2018, 40(11/12): 1507-1518. |
| [78] | Cai SJ, Chen Y, Shang Y, et al. Knockout of zebrafish interleukin 7 receptor (IL7R) by the CRISPR/Cas9 system delays retinal neurodevelopment. Cell Death Dis, 2018, 9(3): 273. DOI:10.1038/s41419-018-0337-z |
| [79] | Lin YC. Genetically modified animal food first listed in the America. Food Safty, 2015, 124(34): 13 (in Chinese). 林雨晨. 动物转基因食品在美国首次上市. 食品安全导刊, 2015, 124(34): 13. |
| [80] | Qian LL, Tang MX, Yang JZ, et al. Targeted mutations in myostatin by zinc-finger nucleases result in double-muscled phenotype in Meishan pigs. Sci Rep, 2015, 5: 14435. DOI:10.1038/srep14435 |
| [81] | Watakabe I, Hashimoto H, Kimura Y, et al. Highly efficient generation of knock-in transgenic medaka by CRISPR/Cas9-mediated genome engineering. Zool Lett, 2018, 4: 3. DOI:10.1186/s40851-017-0086-3 |
| [82] | Liu CQ. The certification of "Event as gene edited babies" and life jurisprudence. Oriental Law, 2019, 67(1): 23-31 (in Chinese). 刘长秋. "基因编辑婴儿事件"与生命法学之证成. 东方法学, 2019, 67(1): 23-31. |
| [83] | Liu RS. Analysis of legal issues related to gene-editing baby event. Med Phil, 2019, 40(2): 21-26 (in Chinese). 刘瑞爽. 基因编辑婴儿事件相关法律问题探析. 医学与哲学, 2019, 40(2): 21-26. |
