

1. 天津科技大学 工业发酵微生物教育部重点实验室,天津 300457;
2. 中国科学院天津工业生物技术研究所 中国科学院系统微生物工程重点实验室,天津 300308;
3. 天津科技大学 生物工程学院,天津 300457
收稿日期:2019-03-22;接收日期:2019-05-10
基金项目:中国科学院重点部署项目(No. KFZD-SW-212-3-1),中国科学院科研仪器设备研制项目(No. YJKYYQ201700233)资助
摘要:对氨基苯甲酸是一种重要的有机合成中间体,广泛应用于医药、染料等行业。近年来对氨基苯甲酸作为一种潜在的高强度共聚物单体越来越受到重视。对氨基苯甲酸作为叶酸合成的前体之一,其合成在大肠杆菌体内由叶酸合成途径的pabA、pabB和pabC三个基因负责,催化分支酸合成对氨基苯甲酸。本研究以实验室构建的酪氨酸高产工程菌TYR002作为出发菌株,首先弱化双功能分支酸突变酶/预苯酸脱氢酶TyrA的表达,以减少酪氨酸积累,然后利用3种不同强度的组成型启动子分别调控pabA、pabB和pabC的表达。摇瓶发酵表明不同的组合调控模式下大肠杆菌发酵培养基中的对氨基苯甲酸积累量存在显著差异,最高可获得0.67 g/L的摇瓶发酵产量。进一步通过发酵条件优化和分批补料发酵,在5 L发酵罐中获得了6.4 g/L的对氨基苯甲酸产量。本研究为改善对氨基苯甲酸生物合成效率提供了重要理论参考。
关键词:对氨基苯甲酸大肠杆菌组合调控代谢工程叶酸合成途径
Improving the production of 4-aminobenzoic in engineered Escherichia coli by combinatorial regulation
Yicheng Xu1,2,3, Fuping Lu1,3, Qinhong Wang2


1. Key Laboratory of Industrial Fermentation Microbiology, Ministry of Education, Tianjin University of Science & Technology, Tianjin 300457, China;
2. CAS Key Laboratory of Systems Microbial Biotechnology, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3. College of Biotechnology, Tianjin University of Science & Technology, Tianjin 300457, China
Received: March 22, 2019; Accepted: May 10, 2019
Supported by: Key Research Program of the Chinese Academy of Sciences (No. KFZD-SW-212-3-1), Research Equipment Program of Chinese Academy of Sciences (No. YJKYYQ201700233)
Corresponding author: Qinhong Wang. Tel/Fax: +86-22-84861950; E-mail: wang_qh@tib.cas.cn.
Abstract: Para-aminobenzoate (PABA) is an important chemical for organic synthesis and extensively used in pharmaceutical and dye industry. In recent years, PABA has received increasing attention as a potential component of high-strength polymer. In Escherichia coli, three genes of pabA, pabB and pabC are responsible for PABA production from chorismate in folate synthetic pathway. However, E. coli does not accumulate or accumulates very few amounts of PABA under normal growth condition. In this study, the tyrosine-producing E. coli TYR002 constructed previously was used as the starting strain for developing PABA-producing strain. First, the activity of bifunctional chorismate mutase/prephenate dehydrogenase TyrA in E. coli TYR002 was weakened to reduce the production of tyrosine. Then, three different constitutive promoters were used to regulate the expression of pabA, pabB and pabC in recombinant plasmid which was transformed into E. coli for improving PABA production. The shake-flask fermentation showed that the different combination of constitutive promoters significantly affected the production of PABA, and the highest shake-flask fermentation titer was 0.67 g/L. After further condition optimization, the engineered E. coli produced 6.4 g/L PABA under 5 L fed-batch fermentation. This study could be a good reference for improving microbial production of PABA.
Keywords: para-aminobenzoateEscherichia colicombinatorial regulationmetabolic engineeringfolate synthetic pathway
对氨基苯甲酸(4-aminobenzoic acid或者para-aminobenzoic acid,PAPA)是一种应用广泛的化工原料,可作为医药和染料的中间体,也是细胞生长和分裂所必需物质叶酸的合成前体[1]。目前对氨基苯甲酸主要由石油中提取的甲苯经过氧化、硝化、最后加氢还原制取。近几年材料学方面的研究显示,对氨基苯甲酸有潜力用于生产包括工程塑料在内的芳香族高分子聚合物以及抗菌材料,同时美国国防部的Living Foundries项目也将对氨基苯甲酸视作一种能够合成高强度高聚物但合成困难的化合物[2]。因此,对氨基苯甲酸的合成受到越来越多的关注。
大多数微生物都具备通过莽草酸和叶酸途径合成对氨基苯甲酸的能力(图 1)。莽草酸途径从4-磷酸赤藓糖(E4P)和磷酸烯醇式丙酮酸(PEP)缩合形成3-脱氧-D-阿拉伯糖-庚酮糖-7-磷酸(DAHP)开始,逐步反应形成的分支酸是一种用于合成芳香族化合物的中间代谢产物[3-5]。叶酸合成途径中的3个酶参与分支酸经4-氨基-4脱氧分支酸(4-ADC)到对氨基苯甲酸的转化过程[6-10]。首先,谷氨酰胺氨基转移酶(pabA编码)获取谷氨酰胺的氨基,随后,4-氨基-4脱氧分支酸合成酶(pabB编码)催化谷氨酰胺提供的氨基与分支酸4位点上的羟基进行置换,使之转化为4-氨基-4-脱氧分支酸。最后一步是由4-氨基-4-脱氧分支酸裂合酶(pabC编码)催化脱去丙酮酸后环化形成对氨基苯甲酸。
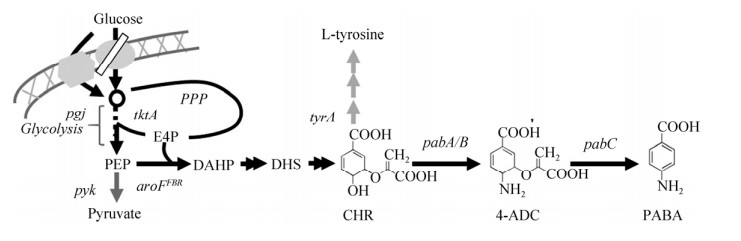 |
| 图 1 大肠杆菌对氨基苯甲酸生物合成途径 Fig. 1 4-aminobenzoic acid biosynthetic pathway in E. coli. PEP: phosphoenolpyruvate; E4P: erythrose-4-phosphate; DAHP: 3-deoxy-D-arabino-heptulosonate-7-phosphate; 3-DHS: 3-dehydroshikimate; CHR: Chorismate; 4-ADC: 4-amino-4-deoxychorismate; PABA: para-aminobenzoate; pgi: phosphoglucose isomerase gene; tktA: transketolase gene; pyk: pyruvate kinase gene; aroFFBR: DAHP synthase mutant (tyrosine feedback-inhibition resistance[16]); tyrA: bifunctional chorismate mutase/prephenate dehydrogenase; pabA: glutamine amidotransferase; pabB: 4-amino-4-deoxychorismate synthase; pabC: 4-amino-4-deoxychorismate lyase. |
| 图选项 |
正常生长情况,微生物几乎不积累或少量积累对氨基苯甲酸。随着合成生物学与代谢工程的快速发展,设计构建微生物工程菌来发酵生产对氨基苯甲酸逐渐受到重视[11-13]。在大肠杆菌中,编码3-脱氧-D-阿拉伯糖-庚酮糖-7-磷酸(DAHP)合成酶反馈抑制解除突变基因aroFFBR、pabAB和pabC分别在诱导型T7lac启动子控制下被整合到基因组中,并且提高pabA和pabB基因拷贝数可以有效提升对氨基苯甲酸合成效率,最后构建的大肠杆菌工程菌在分批补料发酵条件下,最高获得35 mmol/L (约4.8 g/L)的对氨基苯甲酸(每摩尔葡萄糖产率为21%)[11]。通过代谢工程手段改造获得的酿酒酵母和谷氨酸棒杆菌工程菌也实现了对氨基苯甲酸的生物发酵生产[12-13]。
本研究组前期通过代谢工程改造获得了一株高产3-脱氢莽草酸的大肠杆菌工程菌E. coli WJ60,其5 L罐分批补料发酵的3-脱氢莽草酸产量达95 g/L[14-15],该菌株有望提供足够的莽草酸合成途径相关代谢产物,以获取更高产量的对氨基苯甲酸。本研究以E. coli WJ60中恢复aroE基因弱化调控的产酪氨酸工程菌株E. coli TYR002[14]为出发菌株,在弱化双功能分支酸突变酶/预苯酸脱氢酶TyrA减少酪氨酸积累的基础上,通过对重组质粒携带的pabA、pabB和pabC基因进行不同强度组成型启动子控制的组合代谢调控,尝试提高大肠杆菌在没有诱导条件下发酵生产对氨基苯甲酸的能力,为对氨基苯甲酸高效生物合成奠定基础。
1 材料与方法1.1 材料1.1.1 菌株与质粒本试验所用菌株与质粒如表 1和表 2所示。
表 1 本研究的菌株Table 1 Strains used in this study
| Strains | Relative characteristics | Sources |
| E. coli WJ060 E. coli TYR002 | DSM 1576 PM1-12-aroE*, PM1-37-aroFFBR, △tyrR, PM1-12-galP, PM1-93-glk, △ptsI, PM1-12-pykA*, PM1-12-pykF*, PM1-12-pgi* E. coli WJ060 PM1-93-aroE | Lab collection[16] Lab collection[16] |
| E. coli DH5α | F-Φ80lacZΔM15 Δ (lacZYA-argF) U169 recA1 endA1 hsdR17 (rK-, mK+) phoA supE44 λ-thi-1 gyrA96 relA1 | Beijing TransGen This study |
| E. coli PABA 0 | TYR002 P1-TyrA* | This study |
| E. coli Control | E. coli PABA 0 harboring pACYC184 | This study |
| E. coli PABA 1 | E. coli PABA 0 harboring pACYC184-PABA-1 | This study |
| E. coli PABA 2 | E. coli PABA 0 harboring pACYC184-PABA-2 | This study |
| E. coli PABA 3 | E. coli PABA 0 harboring pACYC184-PABA-3 | This study |
| E. coli PABA 4 | E. coli PABA 0 harboring pACYC184-PABA-4 | This study |
| E. coli PABA 5 | E. coli PABA 0 harboring pACYC184-PABA-5 | This study |
| E. coli PABA 6 | E. coli PABA 0 harboring pACYC184-PABA-6 | This study |
| E. coli PABA 7 | E. coli PABA 0 harboring pACYC184-PABA-7 | This study |
| E. coli PABA 8 | E. coli PABA 0 harboring pACYC184-PABA-8 | This study |
| E. coli PABA 9 | E. coli PABA 0 harboring pACYC184-PABA-9 | This study |
| E. coli PABA 10 | E. coli PABA 0 harboring pACYC184-PABA-10 | This study |
| E. coli PABA 11 | E. coli PABA 0 harboring pACYC184-PABA-11 | This study |
| E. coli PABA 12 | E. coli PABA 0 harboring pACYC184-PABA-12 | This study |
| E. coli PABA 13 | E. coli PABA 0 harboring pACYC184-PABA-13 | This study |
| E. coli PABA 14 | E. coli PABA 0 harboring pACYC184-PABA-14 | This study |
| E. coli PABA 15 | E. coli PABA 0 harboring pACYC184-PABA-15 | This study |
| E. coli PABA 16 | E. coli PABA 0 harboring pACYC184-PABA-16 | This study |
| E. coli PABA 17 | E. coli PABA 0 harboring pACYC184-PABA-17 | This study |
| E. coli PABA 18 | E. coli PABA 0 harboring pACYC184-PABA-18 | This study |
| E. coli PABA 19 | E. coli PABA 0 harboring pACYC184-PABA-19 | This study |
| E. coli PABA 20 | E. coli PABA 0 harboring pACYC184-PABA-20 | This study |
| E. coli PABA 21 | E. coli PABA 0 harboring pACYC184-PABA-21 | This study |
| E. coli PABA 22 | E. coli PABA 0 harboring pACYC184-PABA-22 | This study |
| E. coli PABA 23 | E. coli PABA 0 harboring pACYC184-PABA-23 | This study |
| E. coli PABA 24 | E. coli PABA 0 harboring pACYC184-PABA-24 | This study |
| E. coli PABA 25 | E. coli PABA 0 harboring pACYC184-PABA-25 | This study |
| E. coli PABA 26 | E. coli PABA 0 harboring pACYC184-PABA-26 | This study |
| E. coli PABA 27 | E. coli PABA 0 harboring pACYC184-PABA-27 | This study |
| E. coli PABA001 | E. coli TYR002 harboring pACYC184-PABA-4 | This study |
| * An asterisk indicates that the first base in the coding region is changed from A to T. | ||
表选项
表 2 本研究的质粒Table 2 Plasmids used in this study
| Plasmids | Relative characteristics | Sources |
| Crispr-Cas9 system plasmids | ||
| pCas | repA101(Ts) kan Pcas-cas9 ParaB-Red lacIq Ptrc-sgRNA-pMB1 | Lab collection[18] |
| pTargetF | pMB1 aadA sgRNA | Lab collection[18] |
| pTargetT-TyrA↓(with donor DNAs) | pMB1 aadA sgRNA-TyrA↓(636bp) | This study |
| Expression plasmids | ||
| pACYC184-PABA-1 | P1-pabA-T7, P1-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-2 | P2-pabA-T7, P1-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-3 | P3-pabA-T7, P1-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-4 | P1-pabA-T7, P1-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-5 | P2-pabA-T7, P1-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-6 | P3-pabA-T7, P1-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-7 | P1-pabA-T7, P1-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-8 | P2-pabA-T7, P1-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-9 | P3-pabA-T7, P1-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-10 | P1-pabA-T7, P2-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-11 | P2-pabA-T7, P2-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-12 | P3-pabA-T7, P2-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-13 | P1-pabA-T7, P2-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-14 | P2-pabA-T7, P2-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-15 | P3-pabA-T7, P2-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-16 | P1-pabA-T7, P2-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-17 | P2-pabA-T7, P2-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-18 | P3-pabA-T7, P2-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-19 | P1-pabA-T7, P3-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-20 | P2-pabA-T7, P3-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-21 | P3-pabA-T7, P3-pabB-T7, P1-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-22 | P1-pabA-T7, P3-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-23 | P2-pabA-T7, P3-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-24 | P3-pabA-T7, P3-pabB-T7, P2-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-25 | P1-pabA-T7, P3-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-26 | P2-pabA-T7, P3-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
| pACYC184-PABA-27 | P3-pabA-T7, P3-pabB-T7, P3-pabC-T7, p15A, CmR | This study |
表选项
1.1.2 主要试剂氨苄青霉素(工作浓度100 μg/mL)、氯霉素(工作浓度33 μg/mL)、奇霉素(工作浓度50 μg/mL)购自生工生物工程(上海)股份有限公司;TransStart Fast Pfu DNA聚合酶购自北京全式金生物技术有限公司,与参考文献[17]同名试剂一致;质粒小量快速提取试剂盒、2×Taq PCR MasterMix、DNA Marker购自康为世纪公司;对氨基苯甲酸标准品购自国药集团;其他试剂均为分析纯。BamHⅠ、AvaⅠ、T4 DNA连接酶、T4 DNA磷酸化酶(T4 PNK)均来自Thermo公司。Clone ExpressTM Ⅱ一步克隆试剂盒来自诺唯赞公司。
1.1.3 培养基LB培养基(g/L):胰蛋白胨10,酵母浸出物5,氯化钠10(固体培养基加入1.5%琼脂粉),与参考文献[17]同名培养基一致。
摇瓶发酵NBS培养基(g/L):K2HPO4·3H2O 6.5,KH2PO4 3.5,MgSO4·7H2O 0.25,(NH4)2HPO4 3.5,CaCl2·2H2O 0.015。微量元素(mg/L)包括:FeCl3·6H2O 1.6,CoCl2·6H2O 0.2,ZnCl2 0.2,CuCl2·2H2O 0.1,Na2MoO4·2H2O 0.2,H3BO3 0.05,与参考文献[17]同名培养基一致。
发酵罐发酵种子培养基为LB培养基。发酵罐发酵培养基(g/L):MgSO4·7H2O 2,K2HPO4·3H2O 7.5,(NH4)2SO4 1.6,柠檬酸2,FeSO4·7H2O 0.077 5。微量元素(g/L)包括:Na2SO4 0.02,CuSO4·5H2O 0.000 6,ZnSO4 0.0064,CoCl2·6H2O 0.004,与参考文献[17]同名培养基一致。
1.2 方法1.2.1 表达载体的构建整合到表达载体中的由低强度到高强度的组成型启动子P1、P2、P3[19](表 3)与大肠杆菌的pabA、pabB、pabC以及T7终止子串联表达载体构建如下。
表 3 本研究所用的引物Table 3 Primers used in this study
| Primer name | Primer sequence (5'-3') |
| pACYC184-pabC-F | ATACGCGAGCGAACGTGAAGCG |
| pACYC184-pabC-R | TCATTCTGCTAACCAGTAAGGC |
| P1-pabC-F | GGTTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCTTTTGGTGCGTCAGTCAGTTTAAACCAGGAAACAGCT ATGTTCTTAATTA ACGGTTATAAGC |
| P2-pabC-F | GGTTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCTGAGGTGGCTTATTATTCGTTTAAACCAGGAAACAGCTATGTTCTTAATTAACGGTTATAAGC |
| P3-pabC-F | GGTTATCTCTGGCGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCGTATTGTTAGCATGTACGTTTAAACCAGGAAACAGCTATGTTCTTAATTAACGGTTATAAGC |
| pabC-T7-R | CAAAAAACCCCTCAAGACCCGTTTAGAGGCCCCAAGGGGTTATGCTAGTCAGCGATGCAGG |
| BamHⅠ-P1-pabB F | CCGGGATCCTTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCTTTTGGTGCGTCAGTCAGTTTAAACCAGGAAACAGCTATGAAGACGTTATCTCCCGCTGTG |
| BamHⅠ-P2-pabB F | CCGGGATCCTTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCTGAGGTGGCTTATTATTCGTTTAAACCAGGAAACAGCTATGAAGACGTTATCTCCCGCTGTG |
| BamHⅠ-P3-pabB F | CCGGGATCCTTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCGTATTGTTAGCATGTACGTTTAAACCAGGAAACAGCTATGAAGACGTTATCTCCCGCTGTG |
| pabB-T7-AvaⅠ R | GCCCCCGAGCAAAAAACCCCTCAAGACCCGTTTAGAGGCCCCAAGGGGTTATGCTAGTTACTTCTCCAGTTGCTTCAGG |
| P1-pabA-F | CGCTCTAGATTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCTTTTGGTGCGTCAGTCAGTTTAAACCAGGAAACAGCTATGATCCTGCTTATAGATAACTAC |
| P2-pabA-F | CGCTCTAGATTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCTGAGGTGGCTTATTATTCGTTTAAACCAGGAAACAGCTATGATCCTGCTTATAGATAACTAC |
| P3-pabA-F | CGCTCTAGATTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCGTATTGTTAGCATGTACGTTTAAACCAGGAAACAGCTATGATCCTGCTTATAGATAACTAC |
| pabA-T7-R | GCGGGATCCCAAAAAACCCCTCAAGACCCGTTTAGAGGCCCCAAGGGGTTATGCTAGTTAGCGATGCAGGAAATTAGCC |
| pACYC184-pabA-F | GGGGTTTTTTGGGATCCCGCTATATGCGTTGATGCAATTT |
| pACYC184-pabA-R | GCCAGAGATAATCTAGAGCGTACAACTTATATCGTATGGG |
| tyrA promoter sgRNA F | GACGGCTCGCGTGGCTTAAGGTTTTAGAGCTAGAAATAGC |
| tyrA promoter sgRNA R | ACTAGTATTATACCTAGGACTGAGC |
| P1 promoter[19] | 5′-TTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCC TTTTGGTGCGTCAGTCAGTTTAAACCAGGAAACAGCT-3′ |
| P2 promoter[19] | 5′-TTATCTCTGGCGGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCTGAGGTGGCT TATTATTCGTTTAAACCAGGAAACAGCT-3′ |
| P3 promoter[19] | 5′-TTATCTCTGGCGTGTTGACAAGAGATAACAACGTTGATATAATTGAGCCCG TATTGTTAGCATGTACGTTTAAACCAGGAAACAGCT-3′ |
| The underlined is the sequence of synthetic promoter corresponding to the primer name. | |
表选项
1) pACYC184-PX-pabC-T7 (X=1、2或3)质粒的构建:以pACYC184质粒为模板,用引物(pACYC184-pabC-F和pACYC184-pabC-R)进行扩增,获得线性的载体片段。以E. coli TYR002菌株基因组为模板,用引物(P1/P2/P3-pabC-F和pabC-T7-R)进行扩增,获得P1/2/3-pabC-T7片段。用Thermo T4 PNK磷酸化酶对P1/2/3-pabC- T7片段进行磷酸化,然后将线性化的pACYC184载体与磷酸化的P1/2/3-pabC-T7进行连接。将连接产物转化大肠杆菌E. coli DH5α后,挑取单菌落送样测序验证正确的载体进行下一步构建。
2) pACYC184-PX-pabC-T7-PY-pabB-T7 (X或Y=1、2或3)质粒的构建:用BamHⅠ和AvaⅠ限制性内切酶双切pACYC184-PX-pabC-T7 (X=1、2或3)质粒。以TYR002菌株基因组为模板用引物(BamHⅠ-P1/P2/P3-pabB F和PabB-T7-AvaⅠ R)扩增,获得带有BamHⅠ和AvaⅠ酶切位点的P1/2/3-pabB-T7片段。用BamHⅠ和AvaⅠ内切酶双酶切纯化后的P1/2/3-pabB-T7片段。用T4 DNA连接酶连接酶切后的载体和片段。将连接产物转化大肠杆菌E. coli DH5α后,挑取单菌落送样测序验证正确的载体进行下一步构建。
3) pACYC184-PABA-Z (Z=1,2,…,或27)质粒的构建:以pACYC184-PX-pabC-T7-PY-pabB- T7 (X或Y=1、2或3)质粒为模板,用(pACYC184-pabA-F/R)引物进行扩增,获得线性化的载体片段,以TYR002菌株基因组为模板,用引物(P1/P2/P3-pabA-F/R)进行扩增,获得P1/2/3-pabA-T7片段,用诺唯赞克隆重组试剂盒按照生产商建议流程及实验标准进行操作,将连接产物转化大肠杆菌E. coli DH5α后,挑取单菌落送样测序验证获得正确的表达载体。
具体构建片段与整合流程如图 2所示。本研究中所用引物见表 3。
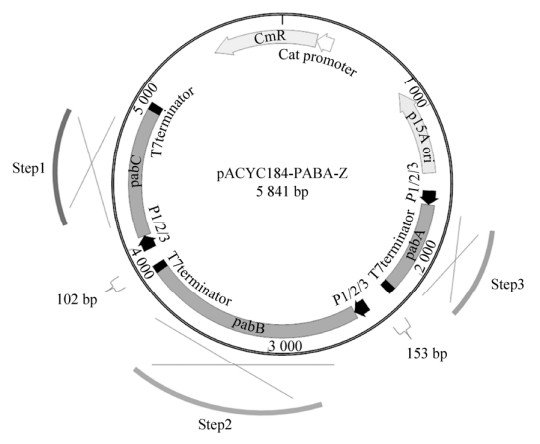 |
| 图 2 重组表达质粒构建示意图 Fig. 2 Schematic diagram of the recombinant expression plasmid pACYC184-PABA-Z (Z=1, 2…, or 27). |
| 图选项 |
1.2.2 tyrA基因弱化载体pTargetT-TyrA↓的构建由华大基因北京合成部合成tyrA弱化用供体DNA片段(整合序列见网络版附件,共计636 bp)亚克隆至pTargetF载体[18]上,根据切割位点序列结合NCBI blast功能设计tyrA弱化用crispr-cas9 N20序列(序列为5′-GACGGCTCGCGTGGCTTA AG-3′),利用引物(tyrA Promoter sgRNA F/R)反扩至pTargetF载体,以thermo T4 PNK磷酸化酶对反扩后片段进行磷酸化,以T4 DNA连接酶进行连接,将连接产物转化E. coli DH5后,挑取单菌落溶于LB液体培养基,送样测序验证。
1.2.3 tyrA基因弱化菌株构建在E. coli TYR002菌株中,aroF与tyrA基因为串联在同一启动子后的多顺反子结构,通过CRISPR-Cas9系统在aroF与tyrA基因间插入T7终止子与P1启动子,同时将tyrA基因的ATG起始密码子改为TTG,实现了tyrA基因的弱化表达。tyrA经由CRISPR-Cas9系统的弱化操作流程参照文献[18]所述。
1.2.4 摇瓶发酵与发酵罐补料发酵摇瓶发酵生产对氨基苯甲酸:挑取单菌落到含有2 mL LB的试管中,30 ℃、200 r/min培养12 h,200 μL接种到10 mL NBS培养基中17 μg/mL氯霉素,35 ℃、250 r/min培养48 h,分别在24 h和48 h取样用于对氨基苯甲酸产量的测定。
发酵罐补料发酵生产对氨基苯甲酸:挑取单菌落到含有3 mL LB中,30 ℃、250 r/min培养12-18 h,再将1 mL培养液接种于200 mL种子培养基中,30 ℃、250 r/min培养12-16 h,然后将200 mL种子液接种到装有1.8 L发酵培养基、氯霉素起始浓度为22.6 mg/L的5 L发酵罐中,在35 ℃、pH 7、溶氧30%的条件下发酵。严格控制培养基中残糖含量低于5 g/L。以氨水调控pH。从16 h开始,每隔4 h取样检测发酵液中的葡萄糖、酪氨酸以及对氨基苯甲酸含量。
1.2.5 代谢产物和菌体量检测利用生物传感检测仪来确定发酵液中葡萄糖消耗量,以高效液相色谱仪检测对氨基苯甲酸和酪氨酸的积累量。取发酵液用0.1 mol/L浓度的盐酸稀释适当倍数后取0.5 mL于12 000 r/min离心5 min,取上清液制样,上液相。检测条件:UVD检测器,Innoval C18色谱柱(4.6 mm×250 mm,5 μm),流动相A为水︰磷酸=1 000︰1 (V/V,占80%),B为甲醇(占20%),柱温30 ℃,流速0.8 mL/min,检测波长分别为266 nm、280 nm。每个待测样品分别有3个平行样,实验结果取自3个平行的平均值。用对氨基苯甲酸以及酪氨酸标准品构建HPLC标准曲线。用分光光度计于600 nm处测定OD600值作为单位时间的菌体量。
2 结果与分析2.1 tyrA弱化表达对对氨基苯甲酸生产的影响前期构建的菌株E. coli TYR002可以在摇瓶发酵过程中积累酪氨酸[14]。根据图 1代谢途径所示,分支酸既是合成对氨基苯甲酸的重要代谢中间产物,也是合成酪氨酸的重要代谢中间产物,对氨基苯甲酸和酪氨酸的合成存在碳流分配竞争的问题。因此,为了增强流向对氨基苯甲酸合成途径的碳流提高其合成能力,需要阻断或弱化酪氨酸的合成途径。在出发菌株E. coli TYR002中,利用CRISPP/Cas9基因编辑的方法,通过在tyrA基因前插入T7终止子和弱启动子P1构建了E. coli PABA0,降低了底盘细胞在发酵过程中的酪氨酸积累量。在E. coli TYR002和E. coli PABA0转入表达对氨基苯甲酸合成基因pabA、pabB和pabC的重组质粒pACYC184- PABA-4 (表 2)进行了摇瓶发酵分析。48 h摇瓶发酵结果表明(图 3),tyrA基因弱化后对氨基苯甲酸产量明显增加,从弱化前的109 mg/L上升到362 mg/L,产量增加超过3倍。同时酪氨酸的积累也明显下降,从弱化前的2.64 g/L下降到2.19 g/L,下降了17%。由于还是有大量的酪氨酸积累,所以进一步弱化调控tyrA应该可以继续提高对氨基苯甲酸的合成能力。
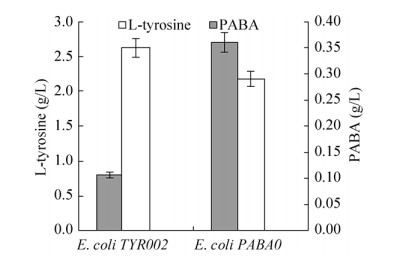 |
| 图 3 携带重组质粒pACYC184-PABA-4的E. coli TYR002(tyrA未弱化菌株)与E. coli PABA0 (tyrA弱化菌株)摇瓶发酵生产结果 Fig. 3 Shake flask fermentation of E. coli TYR002 and E. coli PABA0 harboring pACYC184-PABA-4, respectively, at 48 hours. |
| 图选项 |
2.2 不同强度启动子组合调控pabA、pabB和pabC表达对对氨基苯甲酸合成的影响由图 1对氨基苯甲酸合成途径可知,从分支酸到对氨基苯甲酸涉及pabA、pabB和pabC三个基因。在正常条件下,大肠杆菌只能少量积累对氨基苯甲酸(图 4,对照菌株E. coli Control)。因此,为了提高对氨基苯甲酸的合成能力,需要对pabA、pabB和pabC三个基因进行表达调控。本研究选取了3种表达强度由低到高大致为1︰10︰50的P1、P2和P3 (启动子序列见表 3)[19],分别与pabA、pabB和pabC三个基因进行组合,构建了27种不同组合的重组表达质粒(表 2)。把这27种不同组合的重组表达质粒分别导入tyrA弱化的大肠杆菌E. coli PABA0获得27个工程菌株。然后通过摇瓶发酵分析不同组合表达调控对对氨基苯甲酸生产的影响。由图 4A和B可知,无论在24 h还是48 h发酵条件下,pabA、pabB和pabC三个基因的不同组合表达调控显著影响对氨基苯甲酸的生产,最高产量是最低产量70倍,而且发酵时间的变化对产量没有产生大的影响。在27种不同组合表达中,由P1控制pabA、P1控制pabB和P2控制pabC的组合获得了最高的对氨基苯甲酸产量,达到420 mg/L,表明合适强度组合是保持代谢途径畅通的重要前提。根据图 4的结果进一步分析,在本研究条件下,发现高产对氨基酸苯甲酸的调控组合多不包含P3启动子,这个结果表明高强度表达未必有利于目标代谢物的积累,找到与目标代谢物相关的最佳表达组合才是提升目标产物的关键[20]。
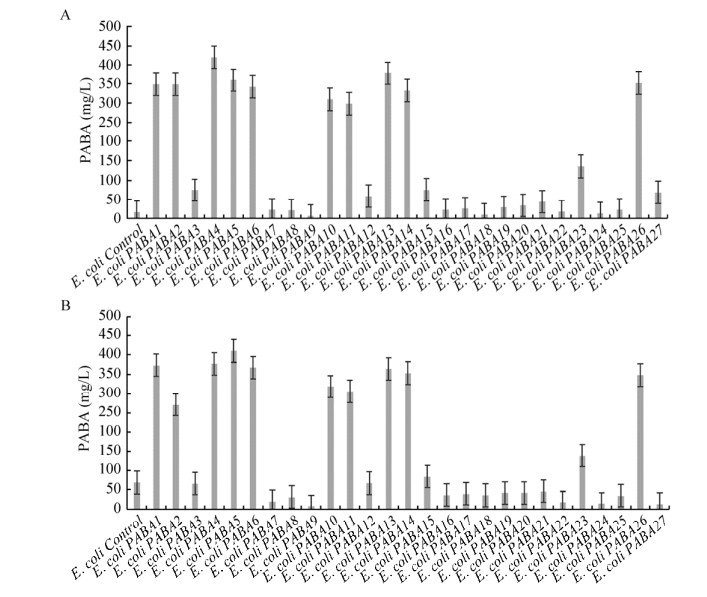 |
| 图 4 不同强度启动子组合调控对对氨基苯甲酸生产的影响 Fig. 4 Effect of different combination of gene expression on PABA production. (A) 24 h shake-flask fermentation. (B) 48 h shake-flask fermentation. |
| 图选项 |
2.3 不同发酵温度对对氨基苯甲酸生产的影响由于含有重组质粒的工程菌株在发酵过程容易出现质粒丢失引起产量减少,而且质粒丢失本身又与发酵温度有关[21],为了寻求产量与发酵温度的关系,本研究探索了不同发酵温度对对氨基苯甲酸生产的影响。在保持培养基、接种量相同的情况下,以不同温度进行摇瓶发酵,分析了发酵24 h和48 h的对氨基苯甲酸生产情况,结果如图 5所示。通过24 h发酵,37 ℃发酵温度可使对氨基苯甲酸生产积累较快,而30-35 ℃的发酵温度则不同程度影响菌株生长,造成耗糖速率减缓,继而导致对氨基苯甲酸积累量低于37 ℃发酵时的积累量。但是继续发酵至48 h时,35 ℃发酵温度获得了最高的对氨基苯甲酸积累量,达到670 mg/L。因此,35 ℃是本实验条件下最适合的对氨基苯甲酸发酵生产温度。
 |
| 图 5 不同发酵温度对对氨基苯甲酸生产的影响 Fig. 5 Effect of different fermentation temperature on PABA production. (A) 24 h shake-flask fermentation. (B) 48 h shake-flask fermentation. |
| 图选项 |
2.4 对氨基酸苯甲酸分批补料发酵生产在上述研究基因表达调控和发酵温度影响对氨基苯甲酸生产的基础上,本研究进一步进行了对氨基酸苯甲酸分批补料发酵生产研究。选择35 ℃作为5 L罐的发酵温度,分别以含有最优启动子组合的tyrA弱化菌株和未弱化菌株进行对氨基酸苯甲酸分批补料发酵。分批补料发酵过程中采用前12 h溶氧与转速联动,以保证溶氧在30%左右;12 h后根据溶氧情况逐渐提高转速,把发酵液中的残余葡萄糖浓度控制在5 g/L以下,以避免乙酸过多积累影响细胞生长和发酵生产[22]。采用上述分批补料发酵生产策略,2株大肠杆菌工程菌株的发酵生产过程如图 6所示。根据发酵结果,含有最优启动子组合的tyrA弱化菌株最高可以积累6.4 g/L对氨基苯甲酸,而同样条件下tyrA未弱化菌株最高只能积累1.95 g/L对氨基苯甲酸,前者比后者增加了约3.3倍。tyrA未弱化菌株可以积累高达约37 g/L的酪氨酸,而tyrA弱化菌株的酪氨酸积累量则大幅降低,最高只仅有8.76 g/L。因此,本研究也证实tyrA弱化调控策略可以大幅降低酪氨酸的积累。
 |
| 图 6 大肠杆菌5L发酵罐分批补料发酵生产对氨基酸苯甲酸 Fig. 6 5 L fed-batch fermentation for PABA production by engineered E. coli. (A) E. coli PABA001. (B) E. coli PABA4. Circle: OD600; square: PABA; diamond: L-tyrosine; triangle: glucose. |
| 图选项 |
3 讨论本研究从前期构建的生产3-脱氢莽草酸大肠杆菌工程菌株出发,通过基因表达调控和发酵优化,在大肠杆菌中显著提升了对氨基苯甲酸的生物合成能力。对氨苯甲酸和酪氨酸的生物合成存在碳流竞争的问题(图 1),但酪氨酸及其合成途径中多个中间代谢产物是维持细胞生长所必需的。为了避免在发酵培养基中额外添加酪氨酸,本研究采用弱化碳流向酪氨酸分配的策略来调整碳流分配,期望更多的碳流向对氨基苯甲酸合成的方向。通过摇瓶发酵,尤其是5 L罐分批补料发酵生产研究表明,通过替换弱启动子的方法有效地降低了酪氨酸的积累。当然,本研究目前没能完全阻止酪氨酸积累,因此进一步的弱化策略仍然需要继续研究。大量的代谢工程研究表明[20],要提高目标产物的合成能力,控制途径的代谢平衡,防止某个代谢中间产物过多积累是重要影响因素。目前的多个研究表明,组合表达调控是实现代谢平衡控制的重要策略[19]。本研究的对氨基苯甲酸合成途径组合表达调控研究也证实这个结论。在本研究中pabA和pabB基因表达量接近,并保持较低的表达水平(使用同样强度的、表达量较低的启动子),同时控制pabC基因的表达量略高于pabA和pabB基因表达量,这样的组合相对于其他组合能获得较高的PABA积累量。而较高的pabA和pabB基因表达水平(使用同样强度的、表达量较高的启动子)则会降低对氨基苯甲酸的积累量。这个结果表明,对于代谢途径关键酶,强表达能力反而不利于目标产物的高效合成。本研究的组合代谢调控策略不仅提高了大肠杆菌合成对氨基苯甲酸的能力,也为相关代谢工程的改造工作提供了重要参考。
参考文献
| [1] | Walsh CT, Haynes SW, Ames BD. Aminobenzoates as building blocks for natural product assembly lines. Nat Prod Rep, 2012, 29(1): 37-59. DOI:10.1039/C1NP00072A |
| [2] | Flores Bueso Y, Tangney M. Synthetic biology in the driving seat of the bioeconomy. Trends Biotechnol, 2017, 35(5): 373-378. DOI:10.1016/j.tibtech.2017.02.002 |
| [3] | Nichols BP, Seibold AM, Doktor SZ. para-aminobenzoate synthesis from chorismate occurs in two steps. J Biol Chem, 1989, 264(15): 8597-8601. |
| [4] | Wegkamp A, van Oorschot W, de Vos WM, et al. Characterization of the role of para-aminobenzoic acid biosynthesis in folate production by Lactococcus lactis. Appl Environ Microbiol, 2007, 73(8): 2673-2681. DOI:10.1128/AEM.02174-06 |
| [5] | Chang Z, Sun Y, He J, et al. p-aminobenzoic acid and chloramphenicol biosynthesis in Streptomyces venezuelae: gene sets for a key enzyme, 4-amino-4-deoxychorismate synthase. Microbiology, 2001, 147(8): 2113-2126. DOI:10.1099/00221287-147-8-2113 |
| [6] | Roux B, Walsh CT. p-aminobenzoate synthesis in Escherichia coli: kinetic and mechanistic characterization of the amidotransferase PabA. Biochemistry, 1992, 31(30): 6904-6910. DOI:10.1021/bi00145a006 |
| [7] | Roux B, Walsh CT. p-aminobenzoate synthesis in Escherichia coli: mutational analysis of three conserved amino acid residues of the amidotransferase PabA. Biochemistry, 1993, 32(14): 3763-3768. DOI:10.1021/bi00065a031 |
| [8] | Ye QZ, Liu J, Walsh CT. p-Aminobenzoate synthesis in Escherichia coli: purification and characterization of PabB as aminodeoxychorismate synthase and enzyme X as aminodeoxychorismate lyase. Proc Natl Acad Sci USA, 1990, 87(23): 9391-9395. DOI:10.1073/pnas.87.23.9391 |
| [9] | Green JM, Merkel WK, Nichols BP. Characterization and sequence of Escherichia coli pabC, the gene encoding aminodeoxychorismate lyase, a pyridoxal phosphate-containing enzyme. J Bacteriol, 1992, 174(16): 5317-5323. DOI:10.1128/jb.174.16.5317-5323.1992 |
| [10] | Green JM, Nichols BP. p-Aminobenzoate biosynthesis in Escherichia coli. purification of aminodeoxychorismate lyase and cloning of pabC. J Biol Chem, 1991, 266(20): 12971-12975. |
| [11] | Koma D, Yamanaka H, Moriyoshi K, et al. Production of p-aminobenzoic acid by metabolically engineered Escherichia coli. Biosci Biotechnol Biochem, 2014, 78(2): 350-357. DOI:10.1080/09168451.2014.878222 |
| [12] | Kubota T, Watanabe A, Suda M, et al. Production of para-aminobenzoate by genetically engineered Corynebacterium glutamicum and non-biological formation of an N-glucosyl byproduct. Metab Eng, 2016, 38: 322-330. DOI:10.1016/j.ymben.2016.07.010 |
| [13] | Averesch NJH, Winter G, Kr mer JO. Production of para-aminobenzoic acid from different carbon-sources in engineered Saccharomyces cerevisiae. Microb Cell Fact, 2016, 15: 89. DOI:10.1186/s12934-016-0485-8 |
| [14] | Cao P. Metabolic Engineering of Escherichia coli to Produce L-Tyrosine and Aromatic Derivatives[D]. Tianjin: Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, 2018 (in Chinese). 曹鹏.代谢工程改造大肠杆菌生产酪氨酸及芳香族衍生物[D].天津: 中国科学院天津工业生物技术研究所, 2018. |
| [15] | Yuan F, Chen WJ, Jia SR, et al. Improving 3-dehydroshikimate production by metabolically engineered Escherichia coli. Chin J Biotech, 2014, 30(10): 1549-1560 (in Chinese). 元飞, 陈五九, 贾士儒, 等. 利用代谢工程改善大肠杆菌的3-脱氢莽草酸生产. 生物工程学报, 2014, 30(10): 1549-1560. |
| [16] | Umbarger HE. Amino acid biosynthesis and its regulation. Annu Rev Biochem, 1978, 47: 533-606. DOI:10.1146/annurev.bi.47.070178.002533 |
| [17] | Song GT, Jiang XL, Chen WJ, et al. Construction and optimization of microbial cell factories for producing cis, cis-muconic acid. Chin J Biotech, 2016, 32(9): 1212-1223 (in Chinese). 宋国田, 江小龙, 陈五九, 等. 产顺, 顺-粘康酸细胞工厂的构建与优化. 生物工程学报, 2016, 32(9): 1212-1223. |
| [18] | Jiang Y, Chen B, Duan CL, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Appl Environ Microbiol, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [19] | Lu J, Tang JL, Liu Y, et al. Combinatorial modulation of galP and glk gene expression for improved alternative glucose utilization. Appl Microbiol Biotechnol, 2012, 93(6): 2455-2462. DOI:10.1007/s00253-011-3752-y |
| [20] | Lee SY, Kim HU. Systems strategies for developing industrial microbial strains. Nat Biotechnol, 2015, 33(10): 1061-1072. DOI:10.1038/nbt.3365 |
| [21] | Park SH, Ryu DDY, Lee SB. Determination of kinetic parameters related to the piasmid instability: for the recombinant fermentation under repressed condition. Biotechnol Bioeng, 1991, 37(5): 404-414. DOI:10.1002/bit.260370503 |
| [22] | van Duuren JBJH, Wijte D, Karge B, et al. pH-stat fed-batch process to enhance the production of cis, cis-muconate from benzoate by Pseudomonas putida KT2440-JD1. Biotechnol Prog, 2012, 28(1): 85-92. |
