
 , 刁爱坡1
, 刁爱坡1

1 天津科技大学 生物工程学院,天津 300457;
2 中国科学院系统微生物工程重点实验室,天津 300308;
3 中国科学院天津工业生物技术研究所,天津 300308
收稿日期:2018-01-29;接收日期:2018-04-26 基金项目:天津市科技计划项目(Nos. 15PTCYSY00020,14ZCZDSY00058),天津市特支计划高层次创新创业团队,国家自然科学基金(No.31700044)资助
摘要:启动子是重要的转录调控元件,广泛用于工业菌株的代谢工程改造。谷氨酸棒杆菌Corynebacterium glutamicum是重要的氨基酸生产菌株,但已报道的组成型强启动子较少。对谷氨酸高产菌Corynebacterium glutamicum SL4发酵过程的10个时间点样品进行转录组测序,筛选在发酵过程中稳定转录并且转录水平最高的10个基因;分别克隆其启动子序列至红色荧光蛋白(RFP)报告系统,通过荧光强度表征启动子在SL4菌株中的强度,再在野生型C. glutamicum ATCC 13869和ATCC 13032中验证部分启动子的通用性;并采用LacZ蛋白进一步评价强启动子的表达效果。结果显示,成功筛选到3个可以通用的组成型启动子PcysK、PgapA和PfumC。其中PcysK的表达强度最高,与诱导型强启动子Ptac对比,在SL4和13869菌株中均达到其2倍(RFP)和4倍(LacZ)以上;在ATCC 13032菌株中,PcysK的表达强度为Ptac的0.3-0.4倍。PcysK首次被报道为强启动子,可用于谷氨酸棒杆菌强化合成途径的代谢工程改造。
关键词:谷氨酸棒杆菌 时间序列转录组 启动子 RFP报告系统
Screening efficient constitutive promoters in Corynebacterium glutamicum based on time-series transcriptome analysis
Yingchun Wang1,2,3, Jiao Liu2,3, Xiaomeng Ni2,3, Yu Lei3, Ping Zheng2,3

 , Aipo Diao1
, Aipo Diao1

1 School of Biological Engineering, Tianjin University of Science and Technology, Tianjin 300457, China;
2 Key Laboratory of Systems Microbial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3 Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
Received: January 29, 2018; Accepted: April 26, 2018
Supported by: Tianjin Science and Technology Program (Nos. 15PTCYSY00020, 14ZCZDSY00058), Tianjin Municipal City, the First "Special Support Plan for Talents Development" and "High-level Innovation and Entrepreneurship Team", National Natural Science Foundation of China (No. 31700044)
Corresponding author:Ping Zheng. Tel/Fax: +86-22-84861943; E-mail: zheng_p@tib.cas.cn
Aipo Diao. Tel/Fax: +86-22-60602948; E-mail: diaoaipo@tust.edu.cn
Abstract: Promoter, an essential regulatory element, is widely used for metabolic engineering of industrial strains. Corynebacterium glutamicum is an important industrial workhorse to produce various amino acids. However, strong constitutive promoters that are applicable to C. glutamicum are rarely reported. In this study, we first performed a time-series transcriptome analysis of a glutamate hyper-producing strain C. glutamicum SL4 by using RNA-Seq. Overall, we picked 10 samples at different time during the fermentation process. By analyzing the time-series transcriptome data, we selected 10 candidate genes with the highest transcriptional level. These genes were all transcribed stably during the fermentation process. We subsequently cloned the promoter sequences and evaluated the promoters' strength in strain SL4 using a red fluorescent protein reporter system. To evaluate the universality of the promoters in different C. glutamicum strains, we further tested the performance of some promoters in wild type C. glutamicum strains, including ATCC 13869 and ATCC 13032. The strongest promoter was further characterized using LacZ as a reporter in all the three C. glutamicum strains. Finally, we successfully obtained three constitutive promoters with universality, PcysK, PgapA and PfumC. PcysK is the most efficient promoter among the three C. glutamicum strains. In strains SL4 and ATCC 13869, the strength of PcysK is 2-fold of the strong inducible promoter Ptac using the red fluorescent protein as a reporter and 4-fold of Ptac using LacZ as a reporter. Moreover, the strength of PcysK reaches 30%-40% of Ptac in strain ATCC 13032. The promoter PcysK is identified as a strong promoter for the first time, which can be used as an efficient biobrick for metabolic engineering of synthesis pathways in C. glutamicum.
Keywords: Corynebacterium glutamicum time-series transcriptome promoter red fluorescent protein reporter system
谷氨酸棒杆菌Corynebacterium glutamicum是重要的氨基酸工业生产菌株,主要用于生产L-谷氨酸、L-赖氨酸和L-异亮氨酸等氨基酸[1]。谷氨酸棒杆菌可以适应简单原料和复杂环境,耐受高浓度碳源[2],并已被美国FDA认证为安全(Generally recognized as safe,GRAS)的微生物[3],近年来又被广泛用于生产生物乙醇和异丁醇等生物燃料[4-6]、琥珀酸和乳酸等有机酸、二元胺和聚羟基丁酸脂等生物材料单体[7-9]以及重组蛋白等生物大分子[10]。代谢工程强化目标产物的合成途径是提升工业菌株性能的常用策略,其中利用强启动子构建高效的转录表达盒已经被证明是非常有效的手段[3, 11-12]。
谷氨酸棒杆菌中关于诱导型启动子的研究较多,例如已经成功地构建了IPTG (Isopropyl- β-dthiogalactopyranoside)诱导型、麦芽糖诱导型、葡萄糖酸诱导型、阿拉伯糖诱导型、鼠李糖诱导型、乙酸诱导型、四环素诱导型和丙酸诱导型等诱导型启动子[13-16]。在代谢工程改造中,诱导型启动子可以通过改变诱导剂浓度实现不同强度表达,使用最多的是IPTG诱导启动子,如Ptrc和Ptac。然而由于其需要添加诱导剂,近年来谷氨酸棒杆菌的代谢工程改造更倾向使用组成型启动子。常用的内源组成型启动子包括PcspB、Ptuf、Psod和PgapA。Tateno等[17]和Vogt等[18]利用PcspB启动子分别提高L-赖氨酸和L-异亮氨酸的产量。Park等[19]和Kim等[20]利用Ptuf启动子分别提高L-精氨酸和L-鸟氨酸的产量。Park等[19]和Jensen等[21]利用Psod启动子分别提高L-精氨酸和L-谷氨酸衍生物的产量。随着合成生物学的发展,研究者开始发展人工合成启动子。Yim等[22]通过人工合成完全随机的70 bp序列,再基于GFP筛选了系列强度启动子,并且应用于木聚糖内切酶等的分泌生产。Rytter等[23]基于谷氨酸棒杆菌启动子–10区共有序列和大肠杆菌启动子–35区共有序列设计并筛选了人工启动子。
目前已经表征的组成型启动子的表达强度大多低于完全诱导的Ptac等启动子。Ravasi等[24]用GFP作为报告系统进行比较,结果显示PcspB和Psod启动子的表达强度分别约为Ptac的0.5和0.1倍。Shang等[25]用LacZ作为报告系统表征部分内源启动子,结果显示Ptuf和Psod启动子的表达强度分别约为Ptac的0.7倍和0.3倍,只有Ppgk启动子的表达强度比Ptac高,约为Ptac的1.35倍。因此依然需要挖掘更多高表达强度的组成型启动子。
转录组和蛋白质组学可以系统地表征基因的表达水平,近年来已经被用于筛选启动子元件。Zhao等[26]从谷氨酸棒杆菌蛋白组数据中显示高蛋白表达的12个基因中筛选到强组成型启动子Ppgk。蛋白组数据只能间接反映基因的转录水平,直接反映基因转录水平的转录组数据更有利于筛选高表达强度的启动子。刘秀霞等[27]通过分析谷氨酸棒杆菌高、中、低溶氧条件下的转录组数据,从不同条件下均高转录的6个候选基因中筛选到组成型强启动子Ptuf。本研究基于谷氨酸高产菌C. glutamicum SL4发酵过程的时间序列转录组数据,筛选发酵过程均稳定转录且转录水平最高的10个基因;通过红色荧光蛋白(RFP)报告系统在SL4菌株中表征这些基因的启动子强度,并采用野生型菌株和LacZ蛋白进一步验证部分启动子的通用性。最终获得了3个通用的组成型启动子,其中PcysK首次被报道为强启动子,这些启动子均可用于谷氨酸棒杆菌的代谢工程改造。
1 材料与方法1.1 材料1.1.1 菌株与质粒本研究所用菌株与质粒见表 1。
表 1 本研究所用菌株与质粒Table 1 Strains and plasmids used in this study
| Strains or plasmids | Description | Reference or source |
| Escherichia coli | ||
| DH5α | General cloning host | TaKaRa |
| Corynebacterium glutamicum | ||
| ATCC 13869 | Wild-type strain | Lab stock |
| ATCC 13032 | Wild-type strain | Lab stock |
| SL4 | A mutant of ATCC 13869, hyper-producing glutamate, temperature-sensitive | Lab stock |
| Plasmids | ||
| pEC-XK99E | Expression vector of C. glutamicum, IPTG-inducible promoter Ptrc, KmR | [28] |
| pSB4K5-rfp | A vector containing rfp gene | Lab stock |
| pZSP1 | A vector containing lacZ gene | Lab stock |
| pXMJ19 | Shuttle vector of C. glutamicum/E. coli, IPTG-inducible promoter Ptac, CmR | [29] |
| pEC-XK99E-rfp | pEC-XK99E derivative, expressing RFP under the control of Ptrc | This study |
| pWYC-1 | pEC-XK99E-rfp derivative, Ptrc was replaced with Ptuf | This study |
| pWYC-2 | pEC-XK99E-rfp derivative, Ptrc was replaced with PgapA | This study |
| pWYC-3 | pEC-XK99E-rfp derivative, Ptrc was replaced with PrpsA | This study |
| pWYC-4 | pEC-XK99E-rfp derivative, Ptrc was replaced with PgltA | This study |
| pWYC-5 | pEC-XK99E-rfp derivative, Ptrc was replaced with PcysK | This study |
| pWYC-6 | pEC-XK99E-rfp derivative, Ptrc was replaced with PgdhA | This study |
| pWYC-7 | pEC-XK99E-rfp derivative, Ptrc was replaced with PctaD | This study |
| pWYC-8 | pEC-XK99E-rfp derivative, Ptrc was replaced with PrpoB | This study |
| pWYC-9 | pEC-XK99E-rfp derivative, Ptrc was replaced with PinfB | This study |
| pWYC-10 | pEC-XK99E-rfp derivative, Ptrc was replaced with PfumC | This study |
| pWYC-11 | pEC-XK99E-rfp derivative, Ptrc was replaced with Ptac | This study |
| pEC-XK99E-lacZ | pEC-XK99E-rfp derivative, rfp was replaced with lacZ | This study |
| pWYC-12 | pWYC-11 derivative, rfp was replaced with lacZ | This study |
| pWYC-13 | pWYC-5 derivative, rfp was replaced with lacZ | This study |
表选项
1.1.2 引物设计与合成本研究所用引物使用Vector软件设计,引物名称及序列如表 2所示,引物均由苏州金唯智公司合成。
表 2 本研究所用引物Table 2 Primers used in thisstudy
| Primer name | Primer sequence (5′–3′) |
| rfp-F | GGATAACAATTTCACACAGGAAAGGAGTTGAGAATGGCTTCCTCCGAAGACGTTATC |
| rfp-R | CCAAGCTTGCATGCCTGCAGTTAAGCACCGGTGGAGTGACGAC |
| pEC-XK99E-F | CTGCAGGCATGCAAGCTTGGC |
| pEC-XK99E-R | CCTGTGTGAAATTGTTATCCGCTC |
| △rfp-F | CTGTTTTGGCGGATGAGAGAAG |
| △rfp-R | TCTCAACTCCTTTGGCCTGTGTGA |
| lacZ-F | ACAGGCCAAAGGAGTTGAGAATGATTACGGATTCACTGGCC |
| lacZ-R | TCTCTCATCCGCCAAAACAGTTACGCGAAATACGGGCAGA |
| Ptac-F | CCTGATGCGGTATTTTCTCCGCCAGGGTGGTTTTTCTTTTC |
| Ptac-R | ATAGCTGTTTCCTGTGTGGGGTGTGAAATTGTTATCCGCTC |
| △trc-F | GAGCGGATAACAATTTCACACAGG |
| △trc-R | GGAGAAAATACCGCATCAGGC |
| Ptuf -F | CCTGATGCGGTATTTTCTCCCATCGGTGACGTGAACTCC |
| Ptuf -R | TGTGAAATTGTTATCCGCTCTGTATGTCCTCCTGGACTTCG |
| PgapA -F | CCTGATGCGGTATTTTCTCCCGCGCCGAAGATCTGAAGAT |
| PgapA -R | TGTGAAATTGTTATCCGCTCCACGCACCAAACCTACTCAC |
| PrpsA -F | CCTGATGCGGTATTTTCTCCATTGGTGGCTTGAGGTTTTC |
| PrpsA -R | TGTGAAATTGTTATCCGCTCATTGCTCCGAAATGGATAGG |
| PgltA -F | CCTGATGCGGTATTTTCTCCGGAAGTCGGTCATGTCTTCG |
| PgltA -R | TGTGAAATTGTTATCCGCTCTTCCGGATTACGGAAGTAGTC |
| PcysK -F | CCTGATGCGGTATTTTCTCCTGAACATAGTCGAGGCGGATC |
| PcysK -R | TGTGAAATTGTTATCCGCTCATTGCCCATGTCGGGTGGT |
| PgdhA -F | CCTGATGCGGTATTTTCTCCGGTCAAATTTCTCCGATGTAGCG |
| PgdhA -R | TGTGAAATTGTTATCCGCTCGTTCCCATCTCGGCTGCATG |
| PctaD -F | CCTGATGCGGTATTTTCTCCGCAGACCGGACTTTTGAATTTGG |
| PctaD -R | TGTGAAATTGTTATCCGCTCAGGTTCCTCCTGACTACGGGA |
| PrpoB -F | CCTGATGCGGTATTTTCTCCCCTGCTGTTCAAATAGATTTTCCC |
| PrpoB -R | TGTGAAATTGTTATCCGCTCCTCACGCGGGTAATTGCAC |
| PinfB -F | CCTGATGCGGTATTTTCTCCAGTCGCTTGAACCGGCATGAA |
| PinfB -R | TGTGAAATTGTTATCCGCTCAGTTTTTGCCAGAGGTTCGAAGG |
| PfumC -F | CCTGATGCGGTATTTTCTCCTGCACCTAGTAGACCATTCAGT |
| PfumC -R | TGTGAAATTGTTATCCGCTCGAGAATTAAATACGGCTGTCAC |
表选项
1.1.3 主要试剂与仪器质粒提取试剂盒、PCR产物纯化试剂盒、胶回收试剂盒均购自北京TIANGEN公司;基因组提取试剂盒购自美国Biomiga公司;RNA提取试剂盒(RNeasy Mini Kit)购自QIAGEN公司;重组克隆试剂盒(ClonExpress? CE Ⅱ)购自南京Vazyme公司;酵母粉和蛋白胨购自英国Oxoid公司;卡那霉素等抗生素购自北京Solarbio公司;其余所有试剂均为国产分析纯。PCR热循环仪,Applied Biosystems公司;分光光度计,Shimadzu公司;Nanodro超微量核酸蛋白测定仪,Thermo Scietific公司;电击转化仪,Mettler Toledo公司;DM5000B荧光显微镜,Leica公司;SpectraMaxM5多功能连续酶标仪,Molecular Devices公司;Illumina MiSeq平台,Illumina公司。
1.1.4 培养基LB培养基:10 g/L NaCl,10 g/L蛋白胨,5 g/L酵母粉,根据需要添加50 μg/mL卡那霉素,用于E. coli培养。LBG培养基:10 g/L NaCl,10 g/L蛋白胨,5 g/L酵母粉,5 g/L葡萄糖,根据需要添加25 μg/mL卡那霉素,用于C. glutamicum ATCC 13869和ATCC 13032培养。SGY培养基:5 g/L葡萄糖,10 g/L酵母粉,18 g/L大豆蛋白胨,1 g/L K2HPO4·3H2O,10 g/L尿素,0.5 g/L丁二酸,10 μg/L生物素,pH调至7.1,根据需要添加25 μg/mL卡那霉素,用于C. glutamicum SL4培养。
1.2 方法1.2.1 构建基于RFP的启动子报告系统以pEC-XK99E质粒为模板,以pEC-XK99E-F和pEC-XK99E-R为引物反向扩增得到线性化载体pEC-XK99E。以pSB4K5-rfp质粒为模板,以rfp-F和rfp-R为引物扩增得到携带RBS序列的rfp片段。由于引物rfp-F和rfp-R携带用于重组克隆的同源臂,使用重组克隆试剂盒,将扩增得到的rfp片段与线性化载体pEC-XK99E进行重组连接,获得重组质粒pEC-XK99E-rfp。采用100倍油镜在绿色激发光下显微镜观察荧光。
1.2.2 C. glutamicum SL4菌株的时间序列转录组测序及目标基因筛选实验室前期在5 L发酵罐,采用温敏工艺(以葡萄糖为碳源,玉米浆、大豆蛋白胨和氨水调pH提供氮源,生物素充足)分批补料发酵C. glutamicum SL4菌株生产谷氨酸,初期发酵温度为33 ℃,8 h开始升温至36.5 ℃,其后逐渐升温至39.5 ℃,发酵周期28 h。本研究直接从发酵罐中取样,取样时间分别为1、3、5.5、8、9.5、11.5、13.5、15.5、18.5、23.5 h,菌体4 ℃离心快速收集,液氮速冻并保存于–80 ℃。液氮研磨菌体成粉末状,再采用QIAGEN RNeasy Mini Kit试剂盒进行RNA的提取及纯化。RNA样品送中国科学院天津工业生物技术研究所技术支撑中心,在Illumina MiSeq平台上进行建库及双端150 bp测序,每个样品获得约1.5 G原始数据。测序数据经过质量过滤后,利用Samtools和HTSeq软件计算每个样本中每个基因的count值(每个基因比对上的reads数),通过Python脚本和FPKM (Fragments Per Kilobase Million)公式计算每个样本中每个基因的FPKM值。计算每个基因10个时间点的FPKM平均值,根据(每个时间点的FPKM值-平均值)/平均值计算每个点的波动,筛选8个以上时间点的上下波动不超过平均值50%的基因(由于10个点波动都不超过50%会去掉大量高转录水平的基因),再选择FPKM平均值最高的10个基因。
1.2.3 启动子的克隆及其RFP报告质粒的构建tuf、gapA、gltA、gdhA基因的启动子序列由查阅文献分析比对菌株C. glutamicum SL4的基因序列获得,rpsA、cysK、ctaD、rpoB、infB、fumC选择全部上游非编码区作为其启动子序列,所有启动子表征时使用相同的5′-UTR和SD序列(下划线):5′-GAGCGGATAACAATTTCACACAGGC CAAAGGAGTTGAGAATG-3′,ATG(加粗)为起始密码子。以SL4菌株的基因组为模板,采用相应的引物(表 2)扩增10个启动子片段。以pXMJ19质粒为模板,采用Ptac-F和Ptac-R引物扩增Ptac启动子片段。以pEC-XK99E-rfp质粒为模板,以△trc-F和△trc-R为引物反向扩增去除质粒中的Ptrc,获得线性化载体片段。使用重组克隆试剂盒,将启动子片段与线性化载体片段分别进行重组连接,获得含有不同启动子的11个报告质粒。所有报告质粒分别转化C. glutamicum SL4、C. glutamicum ATCC 13869和C. glutamicum ATCC 13032菌株,获得表达荧光蛋白的重组菌。
1.2.4 OD600和荧光值的测定挑取平板活化的单克隆接入含20 mL SGY或LBG培养基的100 mL三角瓶中,30 ℃、220 r/min条件下过夜培养,取适量培养物转接入含30 mL SGY或LBG培养基的250 mL三角瓶中,使得初始OD600为0.5,在同样的条件下培养,每隔2-4 h取样检测OD600和荧光强度。每个样品3个平行。包含Ptrc和Ptac启动子的对照菌株在接种时添加0.5 mmol/L IPTG。OD600稀释至合适浓度在酶标仪上测定。荧光强度测定,先用PBS缓冲液洗涤菌体2次,再稀释至合适浓度,在多功能连续酶标仪上分别测定OD600和荧光值,激发波长为560 nm,发射波长为607 nm,荧光强度为测定的荧光值/OD600。
1.2.5 LacZ表达质粒和菌株的构建及β-半乳糖苷酶活性的测定分别以pEC-XK99E-rfp、pWYC-11和pWYC-5质粒为模板,以△rfp-F和△rfp-R为引物反向扩增获得各质粒去除rfp的质粒骨架;以质粒pZSP1为模板,以lacZ-F和lacZ-R为引物扩增得到lacZ片段。采用一步重组克隆试剂盒,将扩增得到的lacZ段与对应的质粒骨架进行重组连接,分别获得pEC-XK99E-lacZ、pWYC-12和pWYC-13重组质粒。所有质粒使用相同的5′-UTR和SD序列(下划线):5′-GAGCGGATAACAATTTCACACAG GCCCACACAGGAAACAGCTATG-3′,ATG(加粗)为起始密码子。所有重组质粒分别电转化至3株谷氨酸棒杆菌中。
检测β-半乳糖苷酶活性:菌株培养与荧光测定相同,每个样品设定3个平行。取适量稳定期菌液冰上孵育20 min,再取0.5 mL菌液于4 ℃、6 000 r/min离心10 min,用0.5 mL预冷的Z缓冲液(0.06 mol/L Na2HPO4·7H2O、0.04 mol/L NaH2PO4·H2O、0.01 mol/L KCl、0.001 mol/L MgSO4、0.05 mol/L β-巯基乙醇,pH 7.0)重悬细胞,测定OD600。取0.1 mL上述样品加入100 μL氯仿和50 μL 0.1% SDS,振荡混匀,30 ℃孵育1 h。加入0.2 mL 30 ℃预热的反应底物ONPG (4 mg/mL),混匀并开始计时,30 ℃孵育一定时间。当反应液充分变为黄色后,加入0.5 mL 1 mol/L Na2CO3溶液,振荡混匀,同时记录时间。在酶标仪中测定OD420和OD550的数值,通过以下公式计算酶活:Miller Units=1000×(OD420–1.75×OD550)/ (OD600×菌液体积(mL)×反应时间(min))。
2 结果与分析2.1 构建基于RFP的启动子报告系统以rfp作为报告基因,根据1.2.1所描述的方法,在大肠杆菌DH5α中构建pEC-XK99E-rfp质粒(图 1),转化子菌落PCR验证和质粒测序都正确,表明重组质粒构建成功。将pEC-XK99E-rfp质粒电转化至C. glutamicum SL4中,获得C. glutamicum SL4 (pEC-XK99E-rfp)菌株,荧光显微镜观察显示其可以表达红色荧光蛋白(图 2),表明RFP报告系统构建成功。
 |
| 图 1 pEC-XK99E-rfp质粒图谱 Figure 1 Plasmid map of pEC-XK99E-rfp. |
| 图选项 |
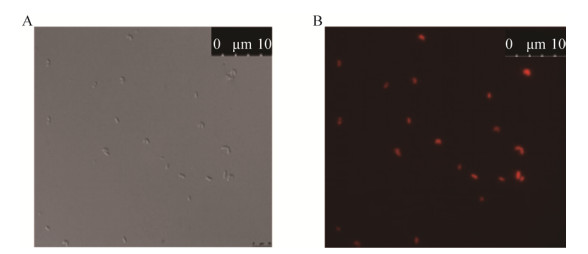 |
| 图 2 C. glutamicum SL4 (pEC-XK99E-rfp)菌株的荧光显微镜观察 Figure 2 Fluorescence microscope observation of C.glutamicum SL4 (pEC-XK99E-rfp). (A) Visible light. (B) Fluorescence. |
| 图选项 |
2.2 C. glutamicum SL4菌株发酵过程的转录组测序及目标基因的筛选根据1.2.2描述的方法,进行C. glutamicum SL4菌株发酵过程10个时间点样品的转录组测序,并通过组学数据分析筛选候选基因。取样时间包括菌体生长和产酸的各个时期,可以全面地分析该菌株发酵过程的基因转录水平。由于常规谷氨酸棒杆菌RNA提取方法中的溶菌酶处理过程会改变胞内真实的RNA水平,本研究采用液氮研磨法提取10个样品的总RNA,所有样品的RNA浓度和完整性都可以达到测序要求。根据转录组的数据分析,筛选出发酵过程均稳定转录且转录水平最高的10个基因,其发酵过程的转录水平如图 3所示,FPKM平均值及基因功能等如表 3所示,其中tuf和gapA基因的整体转录水平更高,其他8个基因转录水平差异较小。
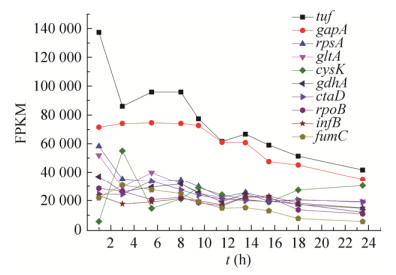 |
| 图 3 10个目标基因在发酵过程中的转录水平 Figure 3 Thetranscriptional expression levels of 10 selected genes in fermentation process. |
| 图选项 |
表 3 10个目标基因的功能、FPKM平均值和启动子长度Table 3 Functions, FPKM averagesand promoter lengths of 10 selected genes
| Gene | Function | FPKM average | Promoter | Length |
| tuf | Elongation factor Tu | 77 355 | Ptuf | 590 |
| gapA | Glyceraldehyde-3-phosphate dehydrogenase | 61 702 | PgapA | 421 |
| rpsA | 30S ribosomal protein S1 | 29 382 | PrpsA | 246 |
| gltA | Citrate synthase 1 | 28 012 | PgltA | 718 |
| cysK | O-acetylserine sulfhydrylase | 24 792 | PcysK | 610 |
| gdhA | NADP-specific glutamate dehydrogenase | 24 372 | PgdhA | 527 |
| ctaD | Cytochrome c oxidase subunit 1 | 23 469 | PctaD | 521 |
| rpoB | DNA-directed RNA polymerase subunit beta | 20 301 | PrpoB | 402 |
| infB | Translation initiation factor IF-2 | 19 880 | PinfB | 543 |
| fumC | Fumarate hydratase class Ⅱ | 16 284 | PfumC | 299 |
表选项
2.3 C. glutamicum SL4菌株中表征启动子的强度按照方法1.2.3成功构建含不同启动子的报告质粒。将含有不同启动子表达RFP的C. glutamicum SL4重组菌接入培养基中,按照1.2.4中的方法进行培养,并使用多功能酶标仪分别检测OD600和荧光强度,以稳定期35 h的荧光强度表征启动子强度。重组菌的生长情况如图 4A和4C所示,与空质粒、Ptrc和Ptac对照相比,所有菌的生长无明显差别,都在12 h即可达到稳定期,OD600约为6,说明这些启动子表达RFP不影响菌株生长。荧光强度检测结果如图 4B和4D所示,前12 h所有启动子的荧光强度都较低,在12–23 h期间荧光强度逐渐增强,培养至23 h时基本达到最高,其后保持稳定。IPTG诱导型对照启动子Ptrc和Ptac的强度和表达模式都基本一致,为了同文献对比,以Ptac启动子的强度进行比较。PcysK、PgapA和PfumC启动子为表达强度最高的3个组成型启动子(图 4B),其强度顺序为PcysK > PgapA > PfumC,分别达到(1 190±31)、(163±3)和(110±4) RFU/ OD600,其强度分别为Ptac的2.2、0.3和0.2倍;其余7个启动子的表达强度都更低。
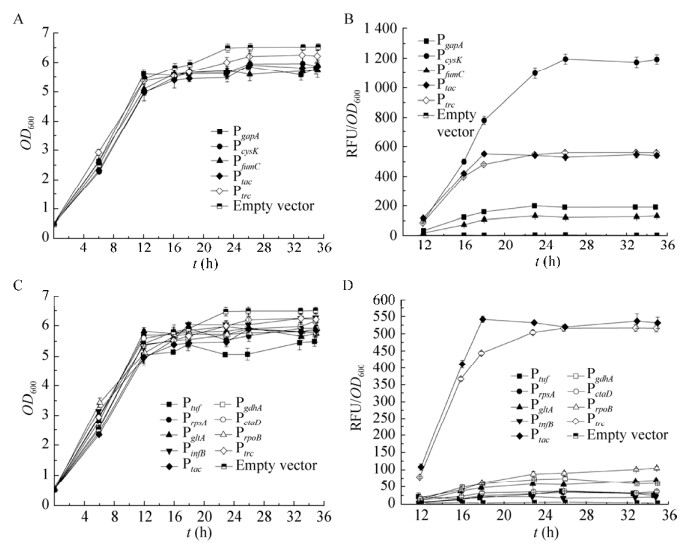 |
| 图 4 不同启动子表达RFP的C. glutamicum SL4系列重组菌的生长曲线及荧光强度 Figure 4 The growth curves and fluorescence intensities of C. glutamicum SL4-based recombinants expressing RFP with different promoters. The growth curves were shown in A and C, and the fluorescence intensities were shown in B and D. (A) and (B) PcysK, PgapA and PfumC; (C) and (D) Ptuf, PrpsA, PgltA, PgdhA, PctaD, PrpoB and PinfB. Ptrc and Ptac were used as 2 control promoters. Error bars indicate the standard deviations from 3 independent cultures. |
| 图选项 |
2.4 野生型谷氨酸棒杆菌中表征启动子强度为了评价3个启动子PcysK、PgapA和PfumC的通用性,将含有这3个启动子的报告质粒及对照质粒分别转入野生型C. glutamicum ATCC 13869和ATCC 13032,并采用更通用的LBG培养基培养,以稳定期23 h的荧光强度表征启动子强度。与对照菌株相比,不同启动子表达RFP的ATCC 13869和ATCC 13032重组菌的生长无明显差别,说明在这2个菌株中不同启动子表达RFP均不影响菌株的生长。荧光强度检测结果如图 5所示,Ptrc和Ptac的强度和表达模式也基本一致,同样以Ptac启动子的强度进行比较,在ATCC 13869菌株中,PcysK、PgapA和PfumC启动子强度分别达到(1 075±21)、(98±5)和(72±5) RFU/OD600,强度及顺序与在SL4菌株中类似,其强度分别是Ptac的2.1、0.2和0.1倍;在ATCC 13032菌株中,PcysK、PgapA和PfumC启动子强度分别达到(154±2)、(77±6)和(48±6) RFU/OD600,强度顺序与在SL4和ATCC 13869菌株中类似,其中PgapA和PfumC 启动子强度变化较小,分别为Ptac的0.2倍和0.1倍;但PcysK启动子强度明显下降,只有Ptac的0.3倍。
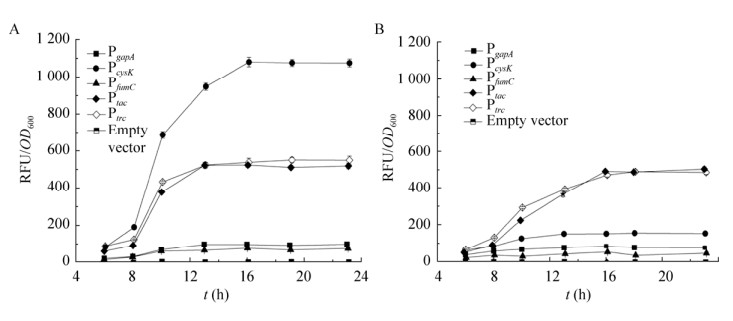 |
| 图 5 PcysK、PgapA和PfumC启动子表达RFP的C. glutamicum ATCC 13869和ATCC 13032系列重组菌的荧光强度 Figure 5 The fluorescence intensities of C. glutamicum ATCC 13869 and ATCC 13032-based recombinants expressing RFP with PcysK, PgapA and PfumC. (A) ATCC 13869. (B) ATCC 13032. Ptrc and Ptac were used as 2 control promoters. Error bars indicate the standard deviations from 3 independent cultures. |
| 图选项 |
2.5 PcysK启动子用于LacZ蛋白表达为了进一步评价强启动子PcysK的蛋白表达效果,分别在C. glutamicum SL4、ATCC 13869和ATCC 13032菌株中验证其表达LacZ的效果,并以Ptrc和Ptac启动子为对照。测定蛋白表达稳定期的β-半乳糖苷酶活性,结果如图 6所示,Ptrc和Ptac启动子在3株菌中的表达效果相似,也以Ptac进行比较,在SL4和ATCC 13869菌株中,PcysK启动子表达的LacZ活性分别约为Ptac的4.1和4.3倍;在ATCC 13032菌株中,PcysK启动子表达的LacZ活性约为Ptac的0.4倍。以上结果表明,PcysK启动子表达LacZ的趋势与RFP一致,但在SL4和ATCC 13869菌株中可以更加高效地表达LacZ。
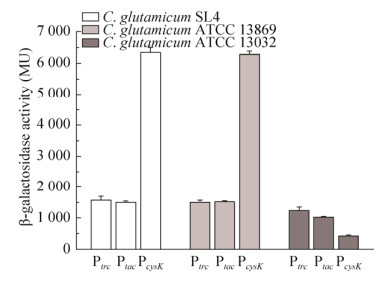 |
| 图 6 PcysK启动子在C. glutamicum SL4、ATCC 13869和ATCC 13032菌株中表达LacZ Figure 6 LacZ were expressed by using the promoter PcysK in C. glutamicum SL4, ATCC 13869 and ATCC 13032, respectively. Ptrc and Ptac were used as 2 control promoters. Error bars indicate the standard deviations from 3 independent cultures. |
| 图选项 |
3 讨论随着谷氨酸棒杆菌在现代生物技术产业中被越来越多地用于生产更多产品,需要挖掘更多的表达元件用于遗传育种改造,从而实现高效生产不同目标产物[10, 30]。相比于诱导型启动子,高效的组成型启动子逐渐展现出优势[31-33]。本研究采用RFP报告系统,从发酵过程均高水平转录的10个基因中筛选到3个内源组成型启动子,其中PcysK启动子首次被报道为强启动子,为谷氨酸棒杆菌的代谢工程改造提供了新的表达元件。
多种条件或多个时间点的转录组数据,可以反映每个基因的转录水平和转录模式,从而可以帮助筛选所需类型的启动子元件。本研究基于发酵过程的时间序列转录组数据筛选的10个发酵过程均高水平转录的基因,与刘秀霞等[27]通过分析不同溶氧条件转录组数据筛选的6个不同溶氧条件均高水平转录的基因相比,只有tuf基因是共有的,其基于EGFP报告系统表征Ptuf启动子为强启动子,而在本研究中采用RFP报告系统表征发现Ptuf的强度很低。与其相比,本研究选取的Ptuf启动子长度更长。Ravasi等[24]发现不同报告基因对相同启动子的评价结果也存在较大差异。因此我们认为启动子序列、报告基因、菌株和培养条件的差异是造成Ptuf启动子表征结果不同的主要原因。此前报道的组成型启动子Psod和PcspB,其起始转录的基因在发酵过程中均可以稳定转录,但平均转录水平没有排在前10位,因此没有入选。另外,本研究中多个基因基于RFP报告系统表征的启动子强度与转录组数据中的转录水平FPKM值并不一致,该现象在其他已报道的类似研究中同样存在。在原核生物中启动子是影响基因转录水平的主要因素,因此一般来说,以全基因组规模的转录组数据筛选内源启动子是一种可行的初筛方法。但是由于基因的转录水平也受基因序列(包括报告基因)、基因的拷贝数、DNA的二级结构、mRNA的稳定性和基因转录的外部环境条件等的影响,所以高转录水平的基因不一定都能通过报告系统表征出强启动子。蛋白质的表达水平还与5′-UTR、SD序列、mRNA的二级结构和密码子使用频率等相关,所以当采用相同的5′-UTR、SD序列和报告基因表征不同启动子时,可以通过RFP等报告蛋白的表达量来表征不同启动子在相同条件的强度,但是如果以上因素发生变化时,启动子强度也可能不同。由此可见,以上因素都可能导致报告系统表征的启动子强度与转录组数据中的转录水平不一致,研究者在使用相关结果时应当全面慎重评估。
为验证本研究发现的3个内源组成启动子的普适性,又分别在ATCC 13869和ATCC 13032菌株中进行了表征和效果评价。在SL4和ATCC 13869菌株中,PcysK启动子强度约为IPTG诱导型启动子Ptac的2.2倍;在ATCC 13032菌株中,其强度约为Ptac的0.3倍,其在3株菌中表达LacZ的效果与RFP趋势一致,该启动子的表达强度受菌株影响较大,因此在不同的谷氨酸棒杆菌菌株中使用时需要注意此点。PgapA和PfumC启动子的强度在3株菌中差异较小,约为Ptac的0.1-0.3倍,其受菌株影响较小。根据Shang等[25]建议的内源组成型强启动子标准,将表达强度大于等于Ptac的0.3倍划分为强启动子,而小于0.3倍为弱启动子。由此可见,PcysK为强启动子,PgapA和PfumC为弱启动子。
4 结论本研究基于谷氨酸高产菌C. glutamicum SL4的时间序列转录组数据,从发酵过程均高水平转录的基因筛选内源的组成型强启动子,通过RFP报告系统表征了10个候选启动子,成功获得3个可以通用的组成型启动子PcysK、PgapA和PfumC。其中PcysK的表达强度最高,也可以高效表达LacZ蛋白,该启动子首次被报道为强启动子,可作为高效元件用于谷氨酸棒杆菌的代谢工程改造。
参考文献
| [1] | Dong XY, Quinn PJ, Wang XY. Metabolic engineering of Escherichia coli and Corynebacterium glutamicum for the production of L-threonine.Biotechnol Adv, 2011, 29(1): 11–23.DOI: 10.1016/j.biotechadv.2010.07.009 |
| [2] | Ohnishi J, Mitsuhashi S, Hayashi M, et al. A novel methodology employing Corynebacterium glutamicum genome information to generate a new L-lysine- producing mutant.Appl Microbiol Biotechnol, 2002, 58(2): 217–223.DOI: 10.1007/s00253-001-0883-6 |
| [3] | Woo HM, Park JB. Recent progress in development of synthetic biology platforms and metabolic engineering of Corynebacterium glutamicum.J Biotechnol, 2014, 180(15): 43–51. |
| [4] | Jojima T, Noburyu R, Sasaki M, et al. Metabolic engineering for improved production of ethanol by Corynebacterium glutamicum.Appl Microbiol Biotechnol, 2015, 99(3): 1165–1172.DOI: 10.1007/s00253-014-6223-4 |
| [5] | Blombach B, Eikmanns BJ. Current knowledge on isobutanol production with Escherichia coli, Bacillus subtilis and Corynebacterium glutamicum.Bioeng Bugs, 2011, 2(6): 346–350.DOI: 10.4161/bbug.2.6.17845 |
| [6] | Yamamoto S, Suda M, Niimi S, et al. Strain optimization for efficient isobutanol production using Corynebacterium glutamicum under oxygen deprivation.Biotechnol Bioeng, 2013, 110(11): 2938–2948.DOI: 10.1002/bit.24961 |
| [7] | Inui M, Murakami S, Okino S, et al. Metabolic analysis of Corynebacterium glutamicum during lactate and succinate productions under oxygen deprivation conditions.J Mol Microbiol Biotechnol, 2004, 7(4): 182–196.DOI: 10.1159/000079827 |
| [8] | Schneider J, Wendisch VF. Putrescine production by engineered Corynebacterium glutamicum.Appl Microbiol Biotechnol, 2010, 88(4): 859–868.DOI: 10.1007/s00253-010-2778-x |
| [9] | Matsumoto K, Kitagawa K, Jo SJ, et al. Production of poly(3-hydroxybutyrate-co-3-hydroxyvalerate) in recombinant Corynebacterium glutamicum using propionate as a precursor.J Biotechnol, 2011, 152(4): 144–146.DOI: 10.1016/j.jbiotec.2010.07.031 |
| [10] | Liu XX, Yang YK, Zhang W, et al. Expression of recombinant protein using Corynebacterium glutamicum: progress, challenges and applications.Crit Rev Biotechnol, 2016, 36(4): 652–664. |
| [11] | Becker J, Wittmann C. Systems and synthetic metabolic engineering for amino acid production-the heartbeat of industrial strain development.Curr Opin Biotechnol, 2012, 23(5): 718–726.DOI: 10.1016/j.copbio.2011.12.025 |
| [12] | Seo SW, Yang J, Min BE, et al. Synthetic biology: tools to design microbes for the production of chemicals and fuels.Biotechnol Adv, 2013, 31(6): 811–817.DOI: 10.1016/j.biotechadv.2013.03.012 |
| [13] | Lee JY, Na YA, Kim E, et al. The actinobacterium Corynebacterium glutamicum, an industrial workhorse.J Microbiol Biotechnol, 2016, 26(5): 807–822.DOI: 10.4014/jmb.1601.01053 |
| [14] | Lausberg F, Chattopadhyay AR, Heyer A, et al. A tetracycline inducible expression vector for Corynebacterium glutamicum allowing tightly regulable gene expression.Plasmid, 2012, 68(2): 142–147.DOI: 10.1016/j.plasmid.2012.05.001 |
| [15] | Fan R, Liu JL, Han WY, et al. Study of the new inducible promoter in Corynebacterium glutamicum.J Hainan Normal Univ: Nat Sci, 2016, 29(2): 154–159.(in Chinese). 范荣, 刘金雷, 韩武洋, 等. 谷氨酸棒状杆菌新型诱导启动子的研究.海南师范大学学报:自然科学版, 2016, 29(2): 154-159. |
| [16] | Chen XX, Chen B, Sun BB, et al. Analysis the intensity regulated by inducible promoters in Corynebacterium glutamicum ATCC 13032.J Shenyang Pharmaceut Univ, 2017, 34(2): 169–175.(in Chinese). 陈晓雪, 陈飚, 孙兵兵, 等. 诱导型启动子在谷氨酸棒杆菌ATCC 13032中调控强度的分析.沈阳药科大学学报, 2017, 34(2): 169-175. |
| [17] | Tateno T, Fukuda H, Kondo A. Direct production of L-lysine from raw corn starch by Corynebacterium glutamicum secreting Streptococcus bovis α-amylase using cspB promoter and signal sequence.Appl Microbiol Biotechnol, 2007, 77(3): 533–541.DOI: 10.1007/s00253-007-1191-6 |
| [18] | Vogt M, Krumbach K, Bang WG, et al. The contest for precursors: channelling L-isoleucine synthesis in Corynebacterium glutamicum without byproduct formation.Appl Microbiol Biotechnol, 2015, 99(2): 791–800.DOI: 10.1007/s00253-014-6109-5 |
| [19] | Park SH, Kim HU, Kim TY, et al. Metabolic engineering of Corynebacterium glutamicum for L-arginine production.Nat Commun, 2014, 5: 4618.DOI: 10.1038/ncomms5618 |
| [20] | Kim SY, Lee J, Lee SY. Metabolic engineering of Corynebacterium glutamicum for the production of L-ornithine.Biotechnol Bioeng, 2015, 112(2): 416–421.DOI: 10.1002/bit.v112.2 |
| [21] | Jensen JVK, Eberhardt D, Wendisch VF. Modular pathway engineering of Corynebacterium glutamicum for production of the glutamate-derived compounds ornithine, proline, putrescine, citrulline, and arginine.J Biotechnol, 2015, 214: 85–94.DOI: 10.1016/j.jbiotec.2015.09.017 |
| [22] | Yim SS, An SJ, Kang M, et al. Isolation of fully synthetic promoters for high-level gene expression in Corynebacterium glutamicum.Biotechnol Bioeng, 2013, 110(11): 2959–2969.DOI: 10.1002/bit.24954 |
| [23] | Rytter JV, Helmark S, Chen J, et al. Synthetic promoter libraries for Corynebacterium glutamicum.Appl Microbiol Biotechnol, 2014, 98(6): 2617–2623.DOI: 10.1007/s00253-013-5481-x |
| [24] | Ravasi P, Peiru S, Gramajo H, et al. Design and testing of a synthetic biology framework for genetic engineering of Corynebacterium glutamicum.Microb Cell Factor, 2012, 11(1): 147.DOI: 10.1186/1475-2859-11-147 |
| [25] | Shang XL, Chai X, Lu XM, et al. Native promoters of Corynebacterium glutamicum and its application in L-lysine production.Biotechnol Lett, 2018, 40(2): 383–391.DOI: 10.1007/s10529-017-2479-y |
| [26] | Zhao ZH, Liu XX, Zhang W, et al. Construction of genetic parts from the Corynebacterium glutamicum genome with high expression activities.Biotechnol Lett, 2016, 38(12): 2119–2126.DOI: 10.1007/s10529-016-2196-y |
| [27] | Liu XX, Zhao ZH, Sun Y, et al. Selection of endogenous expression elements from Corynebactrium glutamicum.Microbiol China, 2016, 43(8): 1671–1678.(in Chinese). 刘秀霞, 赵子豪, 孙杨, 等. 谷氨酸棒杆菌内源表达元件的筛选.微生物学通报, 2016, 43(8): 1671-1678. |
| [28] | Kirchner O, Tauch A. Tools for genetic engineering in the amino acid-producing bacterium Corynebacterium glutamicum.J Biotechnol, 2003, 104(1/3): 287–299. |
| [29] | Jakoby M, Ngouoto-Nkili CE, Burkovski A. Construction and application of new Corynebacterium glutamicum vectors.Biotechnol Technol, 1999, 13(6): 437–441.DOI: 10.1023/A:1008968419217 |
| [30] | Kang MK, Lee J, Um Y, et al. Synthetic biology platform of CoryneBrick vectors for gene expression in Corynebacterium glutamicum and its application to xylose utilization.Appl Microbiol Biotechnol, 2014, 98(13): 5991–6002.DOI: 10.1007/s00253-014-5714-7 |
| [31] | Yang L, Xu JL, Chen XY, et al. Effects of promoter modification on xylose isomerase gene xylA expression in Corynebacterium glutamicum.Adv New Renewable Energy, 2014, 2(5): 353–357.(in Chinese). 杨柳, 许敬亮, 陈小燕, 等. 启动子对木糖异构酶基因在谷氨酸棒杆菌中表达的影响.新能源进展, 2014, 2(5): 353-357.DOI:10.3969/j.issn.2095-560X.2014.05.005 |
| [32] | Choi JW, Yim SS, Lee SH, et al. Enhanced production of gamma-aminobutyrate (GABA) in recombinant Corynebacterium glutamicum by expressing glutamate decarboxylase active in expanded pH range.Microb Cell Factor, 2015, 14(1): 21.DOI: 10.1186/s12934-015-0205-9 |
| [33] | Yim SS, Choi JW, Lee SH, et al. Modular optimization of a hemicellulose-utilizing pathway in Corynebacterium glutamicum for consolidated bioprocessing of hemicellulosic biomass.ACS Synth Biol, 2016, 5(4): 334–343. |
