1 南京师范大学 生命科学学院 江苏省分子医学重点实验室,江苏 南京 210046
2 南京医科大学 康达学院,江苏 连云港 222000
基金项目:国家自然科学基金 (No. 31370401)
摘要: 构建可溶性肿瘤坏死因子相关凋亡诱导配体 (TRAIL) 基因的表达体系,研究其蛋白表达产物对肿瘤细胞凋亡的影响,为以后江豚免疫系统的研究奠定基础。通过RT-PCR 技术从江豚Neophocaena phoconoides血液总RNA 中反转录扩增出肿瘤坏死因子相关凋亡诱导配体 (简称fTRAIL) 的全长cDNA 序列,并将fTRAIL的胞外可溶性 (简称fsTRAIL) 片段连接入表达载体pET43.1a 中,在大肠杆菌BL21 (DE3) 中表达并纯化,Western blotting 对产物Nus-His-fsTRAIL 蛋白进行鉴定。体外用MTT 法、台盼蓝拒染法及流式细胞术检测Nus-His-fsTRAIL 蛋白对Jurkat 细胞和HeLa 细胞的影响。成功构建了fTRAIL 胞外可溶性片段 (简称fsTRAIL)与pET43.1a 组成的表达载体,并获得Nus-His-fsTRAIL 蛋白。体外实验表明,Nus-His-fsTRAIL 蛋白能够以剂量依赖的方式抑制Jurkat 和HeLa 细胞的增殖并诱导其凋亡。Nus-His-fsTRAIL 表达产物具有对Jurkat 和HeLa细胞体外抗肿瘤活性的作用。
关键词: TRAIL 江豚 细胞凋亡 Jurkat 细胞 HeLa 细胞
Molecular cloning, in vitro expression and bioactivity of TRAIL (TNFSF10) gene from finless porpoises
Pei Lili1,2, Zhang Weijing1, Lu Jia1, Huang Fang1, Cao Qianqian1, Ren Wenhua1
1 Jiangsu Province Key Laboratory for Molecular and Medical Biotechnology, College of Life Sciences, Nanjing Normal University,Nanjing 210046, Jiangsu, China;
2 Kangda College of Nanjing Medical University, Lianyungang 222000, Jiangsu, China
Received: October 10, 2015; Accepted: March 14, 2016
Supported by:National Natural Science Foundation of China (No. 31370401)
Abstract: To construct soluble TNF related apoptosis inducing ligand (TRAIL) expression system and investigate theeffect of the expression product on tumor cell. It may provide valuable information for research into the immune system ofthe finless porpoise. The full-length cDNA of TRAIL (designated fTRAIL) was cloned from the total RNA of the finlessporpoises blood using RT–PCR techniques and then the extracellular soluble fragments of fTRAIL (designated fsTRAIL)was ligated into pET43.1a. Recombinant soluble fTRAIL (pET43.1a-fsTRAIL) fused with Nus-his tag was efficientlyexpressed in Escherichia coli BL21 (DE3) and the Nus-His-fsTRAIL protein was purified. The expression ofNus-His-fsTRAIL was verified by Western blotting. In vitro, the effects of the purified Nus-His-fsTRAIL protein on Jurkatand HeLa cells were etected by the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl tetrasodium bromide (MTT) assay,TrypanBlue and Flow Cytometry analysis. The expression system pET43.1a-fsTRAIL was constructed andNus-His-fsTRAIL protein was expressed successfully. In vitro, the Nus-His-fsTRAIL protein was able to inhibit theproliferation and induce apoptosis of Jurkat and HeLa cells in a dose-dependent manner. The Nus-His-fsTRAIL protein hasanti-tumor activity against Jurkat and HeLa cells in vitro.
Key words: TRAIL finless porpoises (Neophocaena phoconoides) apoptosis Jurkat cells HeLa cells
肿瘤坏死因子 (TNF) 是1975 年Carswell 等发现的一种能使肿瘤细胞凋亡、坏死的细胞 因子。细胞凋亡是一个内在基本的细胞程序化 死亡过程,在多细胞有机体的发展和组织稳态 的维持中起着关键的作用[1]。TRAIL 是在其序 列同源性的基础上,于1995 年由Wiley 首次发 现[2],与肿瘤坏死因子配体家族的其他成员一 样,TRAIL 对体外的细胞系包括一些肿瘤细胞 系具有诱导细胞凋亡的作用。与其他TNF 家族 成员相比,TRAIL 具有两个特性:一是TRAIL 在大多数组织中都能表达,而其他TNF 家族成 员的表达受到严格调控并在活化的细胞中暂时 表达[3]。第二,TRAIL 是一个促凋亡配体,目 前发现在一些肿瘤和炎症细胞中能够高表达, 如小儿恶性肿瘤[4]、肾脏疾病[5]、类风湿关节炎、 骨关节炎和脊柱关节病等[6],但其对正常细胞无 明显毒性[7],这意味着,TRAIL 在癌症治疗中 具有潜在的治疗作用[8-11]。TRAIL 在与其受体 结合时形成三聚体,并诱导其受体也形成三聚 体而相互结合,从而选择性地诱导肿瘤细胞发 生凋亡。这些特性预示着TRAIL 在治疗肿瘤方 面有着广泛的应用前景。
1 材料与方法1.1 材料1.1.1 实验动物、细胞和宿主菌江豚新鲜外周血从安徽铜陵繁育中心淡水 长江江豚体检期间获得;实验用细胞系Jurkat细胞与HeLa 细胞由本实验室保存;大肠杆菌 E. coli Top10、DH5α、BL21 和pET-43.1a 由本 实验室保存;pMD19-T 载体购于TaKaRa 公司。
1.1.2 试剂动物血液RNA 提取试剂盒、PrimeScriptTM 1st Strand cDNA 合成试剂盒、pMD19-T 载体试剂盒、Taq DNA 聚合酶、感受态细胞制备试剂 盒、蛋白Marker、荧光定量PCR 试剂盒、rTaq 酶和限制性内切酶等均购自TaKaRa 公司;割胶 回收试剂盒及质粒小提试剂盒均购自Axygen 公 司;T4 DNA 连接酶、碱性磷酸酶、磷酸激酶和 预染蛋白Marker 等均购自Fermentas 公司;鼠 His6 一抗和辣根过氧化酶标记的山羊抗鼠IgG 购自TIANGEN 公司;FITC 标记的羊抗鼠IgG 抗体 (R&D Systems Inc.,USA) 和IPTG 购自 Promega 公司;所有引物合成及测序由上海英骏 公司完成;其他试剂均为国产分析纯。
1.2 方法1.2.1 江豚外周血总RNA 的提取使用动物血液 RNA 提取试剂盒 (RNeasy Mini Kit,Qiagen) 提取江豚外周血总RNA,实 验方法步骤按照试剂盒中的说明书进行。
1.2.2 江豚TRAIL (fTRAIL) 基因的克隆通过 BLAST,在NCBI 数据库中获得牛 (GenBank Accession No. AC_000158.1)、羊 (GenBank Accession No. ADL27922) 和猪 (GenBank Accession No. ABV21385) 的TRAIL 基因[12],根据同源性比对利用Primer 5.0 软件[13] 设计一对简并引物:正义引物fTRAIL F1 和反 义引物fTRAIL F2 (表 1)。用上述简并引物进行 RT-PCR 试验。扩增参数:95 ℃ 5 min;35 个循 环 (94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min);最后72 ℃孵育7 min。RT-PCR 产物用1%的琼脂糖 凝胶电泳分离后,观察并记录结果。按Axygene 公司的DNA胶回收试剂盒操作说明进行割胶回 收约870 bp 的DNA 片段。
表1 引物序列Table1 Primer sequences
| Primer | Direction | Nucleotide sequence (5′–3′) |
| fTRAIL F1 | Forward | ATGGCCAGCTCAGCTTCCCTGC |
| fTRAIL F2 | Reverse | TTAGCCCACGAGGAAGGCTCCA |
| fsTRAIL F1 | Forward | CCGGAATTCACCAACGAGCTGAAGCAGATGCA |
| fsTRAIL F2 | Reverse | CCGCTCGAGGCCAATTAAAAAGGCCCCGA |
表选项
1.2.3 生物信息学分析为克隆fTRAIL cDNA 序列并对所得序列进 行生物信息学分析,使用以下软件和网站:1) 从 NCBI 网站获得TRAIL 基因序列、氨基酸序列, 以及fTRAIL 的开放阅读框 (Open reading frame, ORF),并对相应的氨基酸序列进行预测;2) 使 用3D-JIGSAW和SWISS-MODEL服务器预测三 级结构,并用RasMol 软件查看;3) 使用Clastal W 软件[14]进行多重比对分析;4) 在序列多重比 对的基础上,使用MEGA 5.0 软件中的 Maximum Likelihood 法构建系统树。
1.2.4 融合表达载体pET43.1a-fsTRAIL 的构建研究表明,TRAIL 的可溶型是其功能发挥 的主要形式,根据fTRAIL 的胞外可溶区 (fsTRAIL) 设计一对含有pET43.1a 酶切位点的基 因特异性引物:fsTRAIL F1 (含EocRⅠ位点) 与 fsTRAIL F2 (含XhoⅠ位点) (表 1)。PCR 反应程 序:94 ℃ 5 min;35 个循环 (94 ℃ 30 s;56 ℃ 30s;72 ℃ 1 min);最后 72 ℃延伸 7 min。将PCR 反应产物经电泳、纯化回收PCR 产物(870 bp 左右)。以EcoRⅠ、XhoⅠ酶切PCR 产 物与pET43.1a 载体,然后进行pET43.1a 载体 的去磷酸化与fsTRAIL 目的片段的磷酸化,两 者进行连接反应,构建pET43.1a-fsTRAIL 表达 载体。
1.2.5 连接产物的转化、表达及纯化鉴定原核表达载体pET43.1a-fsTRAIL 在大肠杆 菌中的表达。首先将原核表达载体pET43.1afsTRAIL 转入大肠杆菌BL21 (DE3),挑取含表 达质粒的转化菌株,于LB 培养基 (含Amp+) 中,37 ℃、220 r/min 培养至A600 达0.4-0.6 时, 加入1 mol/L IPTG 至终浓度为0.2 mmol/L, 16℃ 、100 r/min 振荡培养24 h ,诱导pET-43.1a-fsTRAIL 重组蛋白可溶性的表达。培 养液在冰上超声破碎后,分离上清和沉淀,用 SDS-PAGE 检测蛋白的表达。然后用镍柱亲和 层析纯化可溶性重组蛋白Nus-His-fsTRAIL,取 出少量蛋白进行SDS-PAGE 电泳,将其余蛋白 质溶液放于4 ℃冰箱备用。用1× PBS (pH 8.0) 对收集的蛋白液进行透析浓缩后根据需要保存 于-70 ℃。按同样的方法纯化空载体pET-43.1a 的表达产物,即Nus-His 标签蛋白。通过Western blotting 鉴定纯化的蛋白表达产物。
1.2.6 重组可溶性蛋白生物活性测定对纯化的 Nus-His-fsTRAIL 蛋白进行生物 学活性检测,并以Nus-His 标签蛋白作为对照。 采用倒置显微镜及台盼蓝拒染法观察不同浓度 上述两种蛋白在不同时间对白血病T 细胞 (Junkat 细胞) 和人宫颈癌细胞 (HeLa 细胞) 形 态的影响;MTT 法检测在不同浓度作用下,重 组蛋白对肿瘤细胞的抑制作用;经MTT 法和台 盼蓝拒染法检测后,选取适宜浓度的重组蛋白 及适宜的时间梯度,使用流式细胞术检测重组 蛋白对Junkat 细胞的促凋亡作用。
2 结果与分析2.1 江豚全长fTRAIL cDNA 和预测氨基酸序列分析 江豚TRAIL 的开放阅读框为864 bp,编码 一个288 个氨基酸的fTRAIL 蛋白质。克隆所得 全长cDNA序列和预测的氨基酸序列如图 1A 所 示。fTRAIL 的cDNA 序列已提交GenBank 数据 库 (GenBank Accession No. KF151167)。江豚 TRAIL 序列含预测的细胞质结构域,一个跨膜 结构域 (氨基酸18-38) 和一个由250 个氨基酸 组成的胞外结构域。它具有TNF 结构域、一个 保守的半胱氨酸残基和预测的糖基化位点。 fTRAIL 是典型的II 型跨膜蛋白 (图 1B)。在氨 基酸水平上,用Clustal 软件分析显示,江豚和 马、人、狗及牛之间的序列同源性分别为86%、 79%、82%和80% (图 1B)。
 |
| 图1 fTRAIL 基因的cDNA 序列和氨基酸序列 Figure1 The nucleotide and deduced amino acid sequences of fTRAIL.(A) The nucleotide and deduced amino acidsequences of fTRAIL. The initiation codon and the stop codon are boxed, and the stop codon is indicated by anasterisk (*). The putative transmembrane domain is underlined. (B) Amino-acid sequence alignment of TRAIL fromseveral species: finless porpoise, human, dog, cow, and horse were performed using Clustal W software. Identicalamino acids among all sequences are indicated by “*”, whereas those with high or low similarity are indicated by “:”and “.” respectively. The putative transmembrane domain is marked by gray shadings. The TNF superfamilysignature is boxed. The conserved cysteine and asparagine site are marked by red shadings. |
| 图选项 |
2.2 江豚和人TRAIL 胞外区氨基酸序列比对及其蛋白质空间结构分析 人TRAIL (hTRAIL) 胞外区与受体结合部 位为C 端的114-281 位氨基酸,江豚TRAIL (fTRAIL) 胞外区与受体结合部位为C 端的 119-289 位氨基酸,如图 2 所示,hTRAIL(114-281) 与fTRAIL (119-289) 氨基酸序列比 对,相似度为83.3%以上。尽管二者在氨基酸水 平上存在一定的差异,但预测fTRAIL 的胞外功 能区3D 结构 (c,d) 和hTRAIL 胞外区3D 结构 (a,b) 非常相似。这表明江豚的TRAIL 与人 TRAIL 可能具有相似的生物学功能。
 |
| 图2 fsTRAIL (aa119-289) 与hsTRAIL (aa 114-281) 胞外区氨基酸序列比对及其蛋白质空间结构比较 Figure2 Amino acid sequence alignment and homology modelling of the fsTRAIL monomer (aa 119-289) based onthe human structure (aa 114-281).(A) The amino acid sequence alignment of fsTRAIL and human sTRAIL. (B) Thehomology modelling of the fsTRAIL and human sTRAIL. For cartoons display and sphere display of structuremodels: yellow represents β strands, red represents α-helix, blue represents turns and whites represent other residues.(a) and (b) represent the cartoon display and sphere display of the 3D structure of human sTRAIL, respectively. (c)and (d) represent the cartoon and sphere displays of the 3D structure of fsTRAIL, respectively. |
| 图选项 |
2.3 系统进化树分析为确定江豚TRAIL 基因在进化中的位置, 运用Maximum Likelihood 法构建了20 个脊椎 动物的TRAIL 序列的系统进化树。如图 3A 所 示,系统树主要分两个分支,一个是鱼类的分 支,一个是哺乳类和鸟类的分支。江豚TRAIL 与哺乳动物的偶蹄目在进化上接近,而与其他 非哺乳动物的TRAIL 序列较远。为进一步确定 江豚在进化上的位置,我们又用GILT 基因构 建了系统进化树,如图 3B 所示,江豚与偶蹄 目哺乳动物在进化上依然比较接近。以上结果 表明江豚可能与偶蹄目哺乳动物具有共同的原 始祖先。
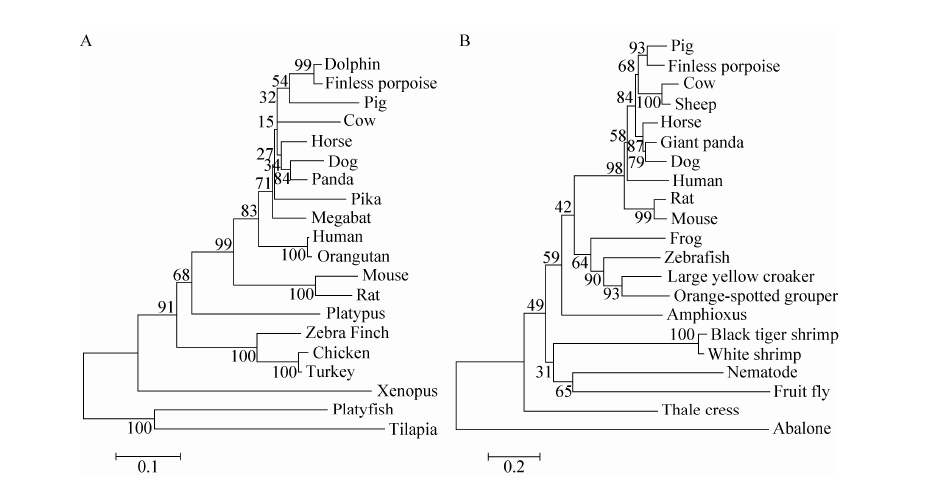 |
| 图3 最大似然法构建系统进化树 Figure3 The phylogenetic tree was constructed by the Maximum Likelihood method using MEGA 5.0 packages and was bootstrapped 1 000 times.(A) Phylogenetic tree showing the relationship between fTRAIL DNA sequences andother identified TRAIL sequences. (B) The phylogenetic tree showing the relationship of GILT sequences fromdifferent species. |
| 图选项 |
2.4 重组蛋白的表达、纯化和鉴定为检测Nus-His-fsTRAIL 蛋白的生物学活性,将重组pET43.1a-fsTRAIL 导入大肠杆菌BL21 (DE3) 中表达。经SDS-PAGE 检测 (图 4)显示,在大约90 kDa 处为目的蛋白,即Nus-HisfsTRAIL蛋白,经IPTG 诱导24 h 达到最大表达量。经Ni2+-NTA 柱纯化后,收集的蛋白经SDS-PAGE 分析和Western blotting 检测,结果见图 4。
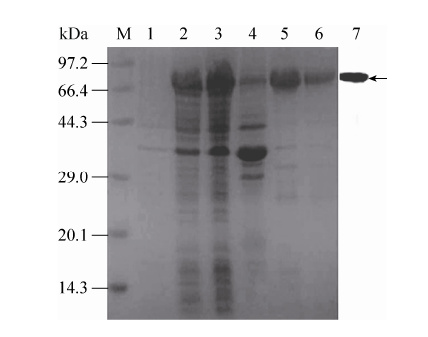 |
| 图4 SDS-PAGE 分析Nus-His-fsTRAIL 蛋白在大肠杆菌中的表达 Figure4 SDS-PAGE analysis of Nus-His-fsTRAIL protein expressed in E. coli BL21 (DE3).M: low molecular weight marker. Lane 1: cell lysates of bacteria transformed with pET43.1a-fsTRAIL without IPTG induction; Lane 2: induced recombinant BL21; Lane 3: the recombinant BL21 supernatant; Lane 4: the recombinant BL21 precipitation; Lane 5: the fusion protein purified by the Ni-IDA affinity chromatography; Lane 6: the secondary chromatography from Lane 5 protein by the Ni-IDA affinity chromatography; Lane 7: the Western blotting analysis of purified Nus-His-fsTRAIL fusion protein using mAb against His6-tag. The arrow represents the location of Nus-His-fsTRAIL fusion protein. |
| 图选项 |
2.5 MTT 法检测Nus-His-fsTRAIL 蛋白采用MTT 法检测Nus-His-fsTRAIL 蛋白对Jurkat 与HeLa 肿瘤细胞的作用。从图 5 中可以看出,以Nus-His 标签蛋白作为对照,在不同浓度下,Nus-His-fsTRAIL 蛋白表现出抑制细胞增殖的作用,而且这种作用呈剂量依赖性。
 |
| 图5 Nus-His-fsTRAIL 蛋白对Jurkat 和HeLa 细胞活性的影响 Figure5 Effect of Nus-His-fsTRAIL protein on the viability of Jurkat and HeLa cell lines by MTT assay.(A) TheJurkat cells were treated with different concentrations of the Nus-His-fsTRAIL or Nus-His tag protein at indicatedamounts. (B) HeLa cells were treated with different concentrations of the Nus-His-fsTRAIL or Nus-His tag protein atindicated amounts. The cell viability was examined after treatment with MTT 24 h. Each treatment was performed intriplicate. Values are x± s of three independent experiments. |
| 图选项 |
2.6 Nus-His-fsTRAIL 蛋白对Jurkat 和HeLa细胞作用的形态观察采用不同浓度梯度,在光学显微镜下观察Nus-His-fsTRAIL 蛋白对Jurkat 和HeLa 细胞作用,并选择反应36 h 后进行台盼蓝拒染实验。从图 6 中可以看出随着时间和浓度的增加,细胞发生了明显的破裂和死亡。
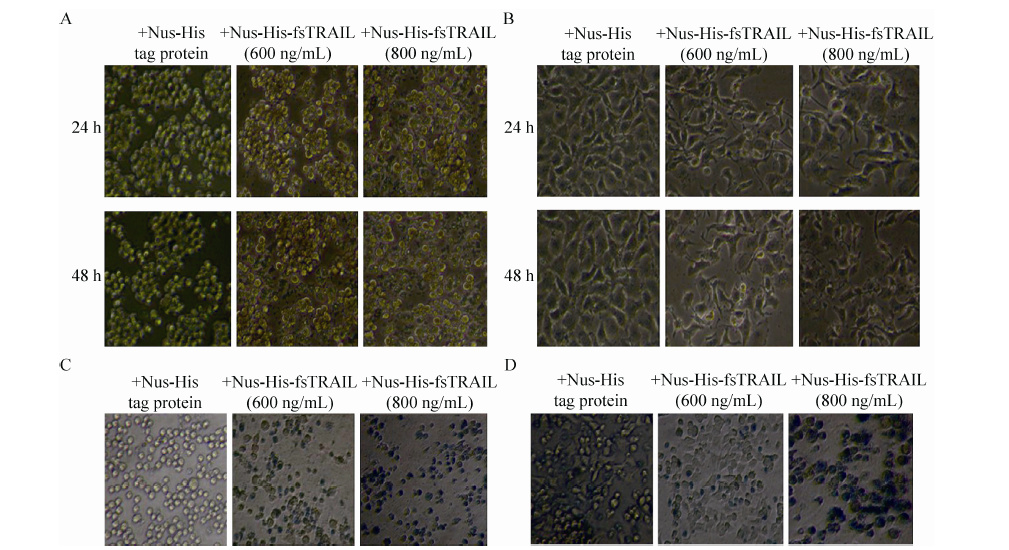 |
| 图6 Nus-His-fsTRAIL 蛋白对Jurkat 和HeLa 细胞作用后形态学的改变 Figure6 Morphology of Jurkat and HeLa cell was visualized under a Nikon Eclipse TE2000-U invertedphase-contrast microscope (200×) equipped with digital camera.(A) and (B) The morphology of Jurkat cells andHeLa cells were photographed under an inverted microscope after treating with different concentrations and differenttime of the Nus-His-fsTRAIL protein or Nus-His tag protein at indicated amounts, respectively. (C) and (D) Jurkatcells and HeLa cells were stained by Trypan Blue after treating with different concentrations at 36 h of theNus-His-fsTRAIL protein or Nus-His tag protein at indicated amounts, respectively. |
| 图选项 |
2.7 流式细胞仪检测Nus-His-fsTRAIL 蛋白的促Jurkat 细胞凋亡作用Nus-His 标签蛋白与Nus-His-fsTRAIL 蛋白分别对Jurkat 细胞作用36 h 后,进行流式细胞仪检测。结果显示,Nus-His 标签蛋白对Jurkat 细胞作用后,在凋亡区FITC (+)/PI(-) 检测到少量细胞(5.67%) (图 7A);Nus-His-fsTRAIL 对Jurkat 细胞作用后,在早期凋亡区FITC (+)/PI(-) 检测到少量细胞 (7.71%),在晚期凋亡区FITC (+)/PI(+) 检测到较多的细胞 (40.97%) (图 7B)。
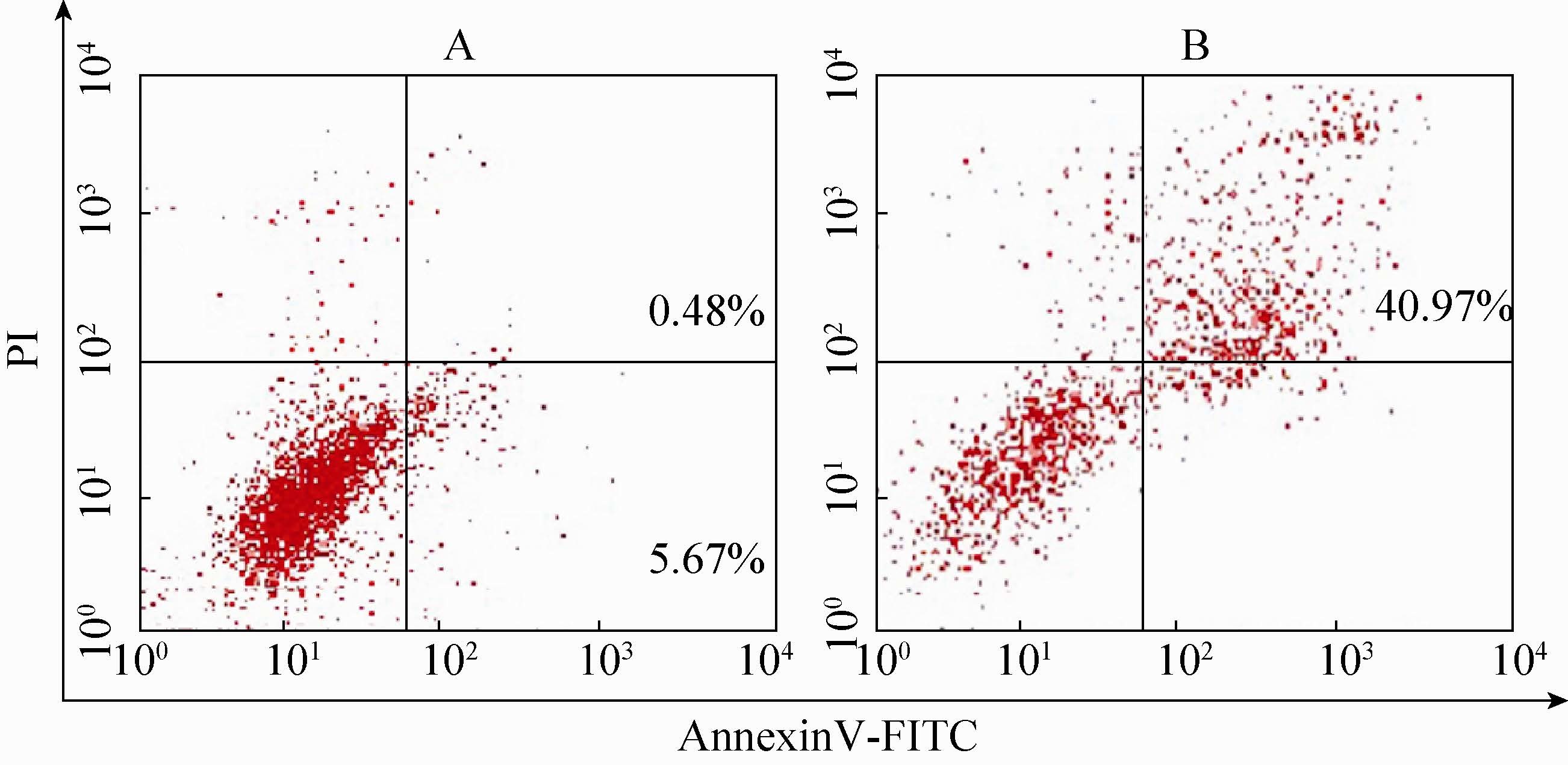 |
| 图7 流式细胞仪检测Nus-His-fsTRAIL 蛋白对Jurkat 细胞的促凋亡作用 Figure7 The apoptotic effect of fsTRAIL on the Jurkat cell was examined by flow cytometry.(A) Jurkat cells weretreated with 800 ng/mL of the Nus-His tag protein. (B) Jurkat cells were treated with 800 ng/mL of theNus-His-fsTRAIL protein. |
| 图选项 |
3 讨论与陆生哺乳动物一样,水生哺乳动物的免 疫系统包括天然免疫和适应性免疫。目前,许 多脊椎动物的肿瘤坏死因子相关凋亡诱导配体 (TRAIL) 的基因已成功克隆[15-16]。然而,水生 哺乳动物TRAIL 仍未见报道。本实验成功克隆 了江豚TRAIL 的CDS 序列并进行了测定。这是 第一个被克隆并鉴定的水生哺乳动物TRAIL 基 因。克隆策略是依据江豚与其他哺乳动物序列 之间的同源性进行的。
江豚TRAIL (fTRAIL) 也具有TRAIL 蛋白 的典型特征。首先,fTRAIL 蛋白也是一个Ⅱ型 跨膜蛋白,有跨膜区且没有启动子;其次, fTRAIL 具有N 端非保守区,而C 端不同物种之 间具有较大的保守性;第三,人TRAIL (hTRAIL) 蛋白的Cys230 是TRAIL 发挥功能所必需的, 通过形成分子间二硫键诱导凋亡[17],而fTRAIL 也具有Cys230。据报道,鲸类与有蹄类动物 (偶 蹄目) 非常相似[18]。我们的研究结果表明,与 其他氨基酸序列相比,江豚TRAIL 与偶蹄目动 物TRAIL 比如猪有88%相似、与牛相似度有 80% (图 2.1b)。但fTRAIL 与非洲爪蟾和岩鸽只有46%的相似性,与其他无脊椎动物 (包括果 蝇、海鞘和鲍鱼) 只有15%-20%的相似性,这 也预示着fTRAIL 可能与偶蹄目动物具有相同 的祖先,可以为鲸类免疫进化的研究提供一定 参考。虽然氨基酸序列比对显示fTRAIL 序列与 偶蹄目序列具有高度相似性,但在某些氨基酸 位点上也存在明显差异,这表明,与陆生哺乳 动物相比,鲸类哺乳动物为了适应水中的环境 可能进行了某些适应性进化。
通过氨基酸序列比对构建系统进化树,获 得了江豚与其他脊椎动物的亲缘关系,结果表 明江豚与偶蹄目哺乳动物的亲缘关系最近。这 一观点同时也被江豚同其他物种的GILT 氨基 酸序列比对所验证[19]。
人TRAIL 与受体结合的胞外区为C 端的114-281 位氨基酸,为TRAIL 的活性部分,有一个 N-糖基化位点并有蛋白酶作用位点,可以 被蛋白酶从膜上剪切下来,形成可溶性分子 sTRAIL[8]。因此,我们将人sTRAIL (114-281) 与江豚的fsTRAIL (119-289) 进行了氨基酸序 列比对,结果表明,它们的相似度在83.3%以上, 即与受体结合部位的氨基酸序列基本一致。通 过蛋白质的3D 结构预测分析,人sTRAIL 与江 豚的fsTRAIL 蛋白具有类似的三维结构。重要 空间结构的氨基酸是非常保守的,这表明 fsTRAIL 可能也具有人sTRAIL 类似的功能,能 够与人肿瘤细胞上的相应受体相结合,这为后 续对江豚TRAIL 胞外可溶片段的表达产物的活 性检测奠定基础。此外,因为我们很难获得江 豚的新鲜组织材料,TRAIL 基因在各种器官组 织 (心脏、肾脏、脾脏、肝脏、肺和肠等) 的表 达水平无法检测,但根据目前的研究表明, TRAIL 基因的mRNA 在脾、肝和肾中有高水平 表达[20]。
为了研究fsTRAIL 的活性,我们曾将其连 接入pET28 表达载体,并在BL21 (DE3) 中表 达。结果蛋白存在于包涵体中,包涵体复性后 没有获得可溶性蛋白。为了增强蛋白的可溶 性,我们将其构建入pET43.1a 表达载体,并进 行低温诱导 (16 ℃),得到了较高水平的可溶 性表达。
文献报道人、 红鳍东方鲀或鳜鱼的 sTRAIL 能够诱导人肿瘤细胞系的凋亡[2, 20-21],因此推测 fsTRAIL 也具有相似作用。为了检测fsTRAIL 诱导肿瘤细胞凋亡的能力,我们首先用MTT 法 检测其对Jurkat 和Hela 细胞作用,实验结果表 明Nus-His-fsTRAIL 蛋白能以剂量依赖的方式 抑制细胞增殖,不难发现Nus-His-fsTRAIL 蛋白 对Jurkat 细胞的抑制能力强于对Hela 细胞的抑 制能力,这可能是由于Nus-His-fsTRAIL 蛋白与 两种细胞上TRAIL 受体结合能力不同所致。此 外,经Nus-His-fsTRAIL 蛋白处理后,Jurkat 和 HeLa 细胞的形态学观察以及台盼蓝染色后的形 态学观察表明fsTRAIL 可以剂量依赖的方式诱 导Jurkat 和HeLa 细胞死亡。为了进一步确定 Nus-His-fsTRAIL 蛋白的肿瘤细胞毒理效用,我 们运用了流式细胞术进一步验证了Nus-HisfsTRAIL 蛋白可以诱导肿瘤细胞凋亡,而对照 Nus-His 标签蛋白则未见此效应。综上所述,我 们可以得出结论,fsTRAIL 可抑制Jurkat 细胞 和HeLa 细胞增殖及诱导凋亡。但fsTRAIL 能 否诱发其他人类肿瘤细胞株的凋亡还有待进一 步研究。
总之,本次实验首次对水生哺乳动物 TRAIL 蛋白进行报道,发现其在进化上与偶蹄 目动物的TRAIL 具有同源性,也发现fTRAIL 能抑制Jurkat 和HeLa 细胞增殖并诱导细胞凋 亡,既为水生哺乳动物从陆生到水生的进化研 究提供了依据,也为江豚甚至是水生哺乳动物 的免疫机制和物种保护的研究奠定了基础。
参考文献
| [1] | Steller H. Mechanisms and genes of cellular suicide.Science, 1995, 267(5203): 1445–1449(in Chinese). |
| [2] | Wiley SR, Schooley K, Smolak PJ, et al. Identification and characterization of a new member of the TNF family that induces apoptosis.Immunity, 1995, 3(6): 673–682(in Chinese). |
| [3] | Goetz FW, Planas JV, MacKenzie S. Tumor necrosis factors.Dev Comp Immunol, 2004, 28(5): 487–497(in Chinese). |
| [4] | Gasparini C, Vecchi Brumatti L, Monasta L, et al. TRAIL-based therapeutic approaches for the treatment of pediatric malignancies.Curr Med Chem, 2013, 20(17): 2254–2271(in Chinese). |
| [5] | Lorz C, Benito A, Ucero AC, et al. Trail and kidney disease.Front Biosci (Landmark Ed), 2009: 3740–3749(in Chinese). |
| [6] | Mustafa MA, Mohamed UK, Bastawisy A. TNF-related apoptosis-inducing ligand levels in rheumatoid arthritis, osteoarthritis, and spondyloarthritis.Egypt J Int Med, 2012, 24(3): 72–78(in Chinese). |
| [7] | Yang SB, Wu XZ. Identification and functional characterization of a human sTRAIL homolog, CasTRAIL, in an invertebrate oyster Crassostrea ariakensis.Dev Comp Immunol, 2010, 34(5): 538–545(in Chinese). |
| [8] | Pitti RM, Marsters SA, Ruppert S, et al. Induction of apoptosis by Apo-2 ligand, a new member of the tumor necrosis factor cytokine family.J Biol Chem, 1996, 271(22): 12687–12690(in Chinese). |
| [9] | Balkwill F. Tumour necrosis factor and cancer.Nat Rev Cancer, 2009, 9(5): 361–371(in Chinese). |
| [10] | Fox NL, Humphreys R, Luster TA, et al. Tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) receptor-1 and receptor-2 agonists for cancer therapy.Expert Opin Biol Ther, 2010, 10(1): 1–18(in Chinese). |
| [11] | Gerspach J, Pfizenmaier K, Wajant H. Therapeutic targeting of CD95 and the TRAIL death receptors.Recent Pat Anticancer Drug Discov, 2011, 6(3): 294–310(in Chinese). |
| [12] | Glenney GW, Wiens GD. Early diversification of the TNF superfamily in teleosts: genomic characterization and expression analysis.JImmunol, 2007, 178(12): 7955–7973(in Chinese). |
| [13] | Lalitha S. Primer premier 5.Biotech Software Internet Rep, 2000, 1(6): 270–272(in Chinese). |
| [14] | Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2.0.Bioinformatics, 2007, 23(21): 2947–2948(in Chinese). |
| [15] | Allen JE, El-Deiry WS. Regulation of the human TRAIL gene.Cancer Biol Ther, 2012, 13(12): 1143–1151(in Chinese). |
| [16] | Chang MX, Nie P, Xie HX, et al. Characterization and expression analysis of TNF-related apoptosis inducing ligand (TRAIL) in grass carp Ctenopharyngodon idella.Vet Immunol Immunopathol, 2006, 110(1/2): 51–63(in Chinese). |
| [17] | Seol DW, Billiar TR. Cysteine 230 modulates tumor necrosis factor-related apoptosis-inducing ligand activity.Cancer Res, 2000, 60(12): 3152–3154(in Chinese). |
| [18] | Gatesy J. Whales and even-toed ungulates (Cetartiodactyla)//Hedges SB, Kumar S, Eds. The Timetree of Life.Oxford, UK: Oxford University Press, 2009: 511-516(in Chinese). |
| [19] | Zhou LD, Yan WL, Yang L, et al. Isolation of gamma-interferon-inducible lysosomal thiol reductase (GILT) from the Yangtze finless porpoise.Dev Comp Immunol, 2013, 41(4): 652–656(in Chinese). |
| [20] | Li JF, Ai HX, Zhang J, et al. Molecular cloning, functional characterization and phylogenetic analysis of TRAIL in Japanese pufferfish Takifugu rubripes.J Fish Biol, 2011, 79(3): 747–760(in Chinese). |
| [21] | Gao Y, Chang MX, Sun BJ, et al. TRAIL in the mandarin fish Siniperca chuatsi: gene and its apoptotic effect in HeLa cells.Fish Shellfish Immunol, 2008, 24(1): 55–66(in Chinese). |
