舒明月1, 付爱思1, 张洁2,3, 陈佳1, 郭静4, 丁琼4, 徐焱成2, 刘天罡1


1. 武汉大学药学院, 组合生物合成与新药发现教育部重点实验室, 湖北 武汉 430071;
2. 武汉大学中南医院内分泌科, 湖北 武汉 430071;
3. 内蒙古呼和浩特第一医院内分泌科, 内蒙古 呼和浩特 010030;
4. 武汉生物技术研究院, 湖北 武汉 430075
收稿日期:2018-12-04;修回日期:2019-01-25;网络出版日期:2019-03-12
基金项目:“****”青年拔尖人才项目;内蒙古自治区自然科学基金(2017MS0872)
作者简介:刘天罡,2010年至今任武汉大学药学院教授,教育部新世纪优秀人才、楚天********,博导。从事天然产物高产机制研究和新化合物高通量挖掘的方法开发。先后获得国家自然科学基金优秀青年基金、中组部****青年拔尖人才计划支持,2017年获湖北省政府专项津贴。从事天然产物高产机制研究和新化合物高通量挖掘的方法开发,同时将该技术体系和研究思路服务于地方经济,实现了维生素E的工艺创新,已为地方新增工业产值20亿,并被评为2018年湖北十大科技事件。目前发表包括Science、PNAS、Metabolic Engineering、Angewandte Chemie等50多篇SCI论文,总SCI引用次数超过1300次,担任Metabolic Engineering Communications的副主编以及ACS Synthetic Biology、Biotechnology Journal等多个杂志的编委.
*通信作者:刘天罡, Tel/Fax:+86-27-68755086, E-mail: liutg@whu.edu.cn.
摘要:[目的] 肠道菌群是位于人体肠道内的微生物菌群,其组成与人类多种疾病相关。例如,已有研究表明肠道菌群的变化与妊娠期糖尿病(gestational diabetes mellitus,GDM)的发病密切相关。因此本研究基于PacBio SMRT测序技术评估及比较了不同民族(汉族和蒙古族)及是否患GDM的孕妇的肠道菌群。[方法] 本研究利用PacBio SMRT测序技术对97例患有GDM及健康的汉族和蒙古族孕妇粪便样本进行了全长16S rRNA测序及分析。[结果] 总体来说,处于相同孕期的4组孕妇肠道菌群的组成相似,不同核心菌群展现出不同强弱的相关性。本研究在种的水平上共鉴定到了44个种。在汉族人中,患有妊娠期糖尿病的孕妇肠道菌群中Akkermansia muciniphila菌的相对丰度要显著低于健康孕妇;而在蒙古族人中,健康孕妇与GDM孕妇间差异并不明显。在健康对照中发现汉族孕妇肠道菌群中Bacteroides uniformis菌的相对丰度显著高于蒙古族孕妇;但在患有GDM组中未找到不同民族分组间的差异。另外,功能预测结果发现四组样本菌群功能组成高度相似,大多数功能基因都与能量代谢有关。汉族GDM患者与健康对照组间没有发现显著差异,但在蒙古族GDM患者中发现无机离子运输等功能相对丰度显著高于与蒙古族正常孕妇。[结论] 在相同的孕期,妊娠期孕妇的核心肠道菌群结构与功能是相对稳定的,而民族差异也不会对妊娠期菌群产生显著性影响。但在四组间可以检测到一些低丰度的差异菌群,如Akkermansia muciniphila,其丰度的变化可能导致了肠道中一些与肠道营养吸收等有关的代谢发生变化,而这些变化可能与妊娠期糖尿病的发生密切相关。本研究将有助于探究肠道菌群在GDM发病机制中的作用。
关键词:PacBio SMRT测序技术妊娠期糖尿病肠道菌群微生物多样性16S rRNA
PacBio SMRT sequencing of the intestinal flora composition in patients with gestational diabetes mellitus
Mingyue Shu1, Aisi Fu1, Jie Zhang2,3, Jia Chen1, Jing Guo4, Qiong Ding4, Yancheng Xu2, Tiangang Liu1


1. Key Laboratory of Combinatorial Biosynthesis and Drug Discovery, Ministry of Education, School of Pharmaceutical Sciences, Wuhan University, Wuhan 430071, Hubei Province, China;
2. Department of Endocrinology, Zhongnan Hospital of Wuhan University, Wuhan 430071, Hubei Province, China;
3. Department of Endocrinology, the First Hospital of Huhhot, Huhhot 010030, Inner Mongolia Autonomous Region, China;
4. Wuhan Institute of Biotechnology, Wuhan 430075, Hubei Province, China
Received: 4 December 2018; Revised: 25 January 2019; Published online: 12 March 2019
*Corresponding author: Tiangang Liu, Tel/Fax:+86-27-68755086, E-mail: liutg@whu.edu.cn.
Foundation item: Supported by the Young Talents Program of National High-level Personnel of Special Support Program (The "Ten Thousand Talent Program") and by the Natural Science Foundation of Inner Mongolia Autonomous Region (2017MS0872)
Abstract: [Objective] The intestinal flora represents the microbial flora located in the human intestine, and its composition has been related to the occurrence of various diseases in humans. For example, the intestinal flora plays an important role in the pathogenesis of gestational diabetes mellitus (GDM). Therefore, in this study, we evaluated and compared the intestinal flora in pregnant women according to ethnicity (Mongolian or Han) and GDM status using PacBio SMRT sequencing. [Methods] Ninety-seven fecal samples from pregnant women with or without GDM and of Han or Mongolian ethnicity were subjected to full-length 16S rRNA sequencing using the PacBio SMRT system. [Results] Overall, the microbial flora compositions of the pregnant women were similar during the same pregnant stage, and various microbes exhibited different degrees of correlation with core flora levels. We identified 44 species at the species level. The relative abundance of Akkermansia muciniphila was significantly higher in the non-GDM pregnant women than in pregnant women with GDM in the Han population, while no differences were found in the Mongolian women with and without GDM. The relative abundance of Bacteroides uniformis in the intestinal flora of healthy Han pregnant women was significantly higher than that in healthy Mongolian pregnant women, while no differences were found in the GDM groups between ethnicities. Moreover, the functional prediction results showed that the functional composition of the four groups of flora was highly similar, and most of the functional genes were related to energy metabolism. There was no significant difference between the GDM patients and controls in the Han population, but the relative abundance of flora related to inorganic ion transport in Mongolian GDM patients was significantly higher than that of healthy Mongolian pregnant women. [Conclusion] The intestinal flora is generally relatively stable during the same pregnant stage, and ethnic differences do not have a significant impact on the flora during pregnancy. However, changes in some low-abundance organisms such as Akkermansia muciniphila may contribute to alterations in some metabolic activities of the intestinal flora, including those related to intestinal nutrient absorption, which could influence the development of GDM. The results of this study can help elucidate the role of intestinal flora in GDM pathogenesis.
Keywords: PacBio SMRT sequencinggestational diabetes mellitusintestinal florabacterial diversity16S rRNA
妊娠期糖尿病(gestational diabetes mellitus,GDM)是孕妇在妊娠中后期最常见的妊娠期并发症之一,是一种由于体内产生的胰岛素水平过低而使血糖升高导致的疾病。随着人们生活质量的提高以及近年来开放了二胎政策,使得妊娠期糖尿病的发病率逐年增加[1]。目前,妊娠前期超重、偏胖或家族有糖尿病史的孕妇是妊娠期糖尿病的高危人群[2]。妊娠期糖尿病多数产后能恢复正常,但对孕妇及胎儿的健康有很大影响[3-4],可能会导致孕妇流产、早产及新生儿低血糖等疾病的发生。并且孩子在儿童期、青春期及成年期容易发生肥胖,是糖尿病的高发人群;孕妇易发生感染、妊娠期高血压等疾病。患有妊娠期糖尿病的孕妇将来患Ⅱ型糖尿病的机会也会增加[5]。在孕妇妊娠过程中,体内会出现一些代谢变化,如血脂异常及高血压等,这些变化有利于胎儿在母体中的发育,且可能与孕妇的肠道菌群密切相关。近年来大量研究表明,肠道菌群在人类免疫、营养和病理过程中发挥着重要的作用。以往研究报道,肠道菌群中的菌群类型及其丰度与人类宿主的遗传信息以及饮食习惯密切相关,与炎症、肿瘤、代谢疾病等多种疾病相关[6-9]。
迄今为止,已有多种方法被应用到妊娠期糖尿病与肠道菌群的相关研究中。如通过荧光原位杂交与流式细胞法相结合的方法(即FCM-FISH,fluorescent in situ hybridization coupled with flow cytometry)以及实时定量PCR的方法(即qPCR,quantitative real-time polymerase chain reaction)对超重和正常体重的孕妇肠道菌群进行分析,发现超重孕妇肠道中的Bacteroides和Staphylococcus明显高于正常体重的孕妇[10]。也有基于高通量测序的方法,Koren等[11]通过对妊娠期正常孕妇的肠道菌群进行研究,发现在怀孕过程中,有超过一半的孕妇肠道菌群中变形菌门和放线菌门的丰度有所增加;而厚壁菌门和拟杆菌门的丰度基本没有发生变化。也有研究[12]对产后3–16个月的GDM患者和健康对照者的粪便进行16S rRNA测序,结果表明,无论GDM患者产后血糖是否正常,其厚壁菌门的相对丰度均少于健康对照者,两组之间α多样性无明显差异。还有研究对GDM和健康孕妇的肠道菌群进行了宏基因组测序分析[13],发现GDM患者中Parabacteroides distasonis、Klebsiella variicola等富集,而正常孕妇肠道中Methanobrevibacter smithii、Alistipes spp.、Bifidobacterium spp.和Eubacterium spp.富集。
目前,基于培养的方法是菌群鉴定的“金标准”,但是由于其培养周期长,难以通过镜检鉴定特殊形态的微生物以及存在一些特异菌难以培养等缺点,无法应用于大规模的研究。而基于高通量测序的方法克服了传统培养方法的缺点。目前基于高通量测序方法来分析菌群主要包括两种方法:扩增子测序,即根据PCR引物对样本中的Marker基因(如16S rRNA基因)进行扩增测序;或者是不进行扩增直接进行宏基因组测序。相比于宏基因组测序而言,基于扩增子测序的方法更经济,分析周期相对较短。16S rRNA是细菌分类学研究中最常用的“分子钟”,总长约1.5 kb,其中包括9个可变区和10个保守区。不同菌的可变区是不同的,且其变异的程度与细菌的系统发育密切相关。通过对16S rRNA基因的可变区序列进行测序,即可对样本中的菌群进行物种鉴定以及多样性分析。目前常用的高通量测序的方法主要包括Sanger测序、Illumina测序以及PacBio测序等。Sanger测序方法能够提供接近全长的16S rDNA片段,但是这种方法成本很高并且通量很低,难以满足复杂环境以及高通量样本的研究需求。以Illumina为代表的二代测序技术,成本大大降低,更适合大规模的测序,但是受到其测序长度的限制,往往只能选择1到3个可变区作为扩增片段,从而导致大部分数据很难有效鉴定到种水平,使得分类的准确性和一致性存在潜在的问题。而以PacBio SMRT为代表的实时单分子测序技术因其超长的读长优势(平均读长可达到10–15 kb),完全可以覆盖16S rRNA基因全长,无需任何组装即可得到完整的全长16S rDNA序列[14]。因此相比于Sanger和Illumina测序而言,利用PacBio SMRT测序技术来进行高质量全长16S rDNA序列测序具有更经济、更高通量、更完整及物种鉴定更准确的优势[15-17],目前已有很多应用[18-19]。PacBio SMRT测序技术能够为微生物群多样性和结构研究提供一种经济高效的研究方式。
本研究首次采用三代PacBio SMRT测序技术基于全长16S rDNA测序的方法对97例患有妊娠期糖尿病和正常孕妇的肠道菌群进行测序分析,并根据样本是否患病以及民族差异分成了4个组(表 1),并研究了4个分组中样本肠道菌群的分布及其核心共有微生物,并在种的水平上以及预测的功能上对四组样本进行了比较分析。这些结果将为妊娠期糖尿病的临床诊断、治疗等相关研究提供重要的参考信息。
表 1. 4个分组临床信息表 Table 1. Clinical information of the four groups
| Group name | Numbera | OGTT test/(mmol/L) | ||
| Fasting blood sugar | 1 h after glucose load | 2 hrs after glucose load | ||
| GDM+_Meng | 17 | 5.56±1.24 | 9.95±2.71 | 8.31±1.39 |
| GDM+_Han | 27 | 5.92±0.95 | 10.28±1.68 | 8.52±2.03 |
| GDM–_Meng | 27 | 4.37±0.45 | 7.30±0.84 | 6.52±0.93 |
| GDM–_Han | 26 | 4.50±0.30 | 6.75±1.49 | 6.86±1.02 |
| GDM+_Meng, Mongolian pregnant woman with gestational diabetes mellitus; GDM+_Han, Han pregnant woman with gestational diabetes mellitus; GDM–_Meng, healthy Mongolian pregnant woman; GDM–_Han, healthy Han pregnant woman. aNumber represents the number of samples for a given group. Values are mean±standard deviation. | ||||
表选项
1 材料和方法 1.1 临床样本 本研究获得了内蒙古呼和浩特第一医院的伦理批准。所有研究参与者均获得书面知情同意,研究方案经呼和浩特第一医院伦理委员会批准。
1.2 参与者招募和研究设计 本研究中的受试者来自2015年11月至2016年12月期间在中国内蒙古呼和浩特市第一医院接受检查的孕妇,妊娠期糖尿病孕妇和正常血糖的孕妇以1:1的比例进行招募。孕妇在妊娠第23周后,GDM的诊断是基于标准化口服葡萄糖耐量试验(OGTT),详细的临床信息如表 1所示。酗酒或药物滥用、胰岛素自身抗体阳性以及需要药物治疗的慢性疾病(甲状腺功能退化除外)的受试者均被排除(n=13)。所有受试者均未患有任何胃肠道疾病,并且在临床评估和粪便采集前至少1个月内未服用过任何抗生素或酸奶。我们根据是否患有GDM和民族差异将97名符合条件的受试者分为四组(表 1),其中有17名蒙古族和27名汉族孕妇患有GDM,27名蒙古族和26名汉族为健康孕妇。
1.3 粪便样本采集和DNA提取 每位受试者在医院均是使用预先填充有粪便DNA稳定剂的纸制粪便收集器进行粪便采集。收集的样品立即被送到内蒙古呼和浩特市第一医院内分泌科,储存于–80 ℃,后干冰低温寄至武汉生物技术研究院–80 ℃处保存,直至进行DNA提取。细菌DNA是按照PowerFecal? DNA Isolation kit (MO BIO)试剂盒标准流程从粪便样本中提取。
1.4 全长16S rDNA序列扩增及PacBio SMRT测序 全长16S rDNA扩增使用正向27F引物(5′-GA GAGTTTGATCCTGGCTCAG-3′)和反向1541R引物(5′-AAGGAGGTGATCCAGCCGCA-3′)。扩增产物利用AMpure XP磁珠(Beckman coulter Gmbh, krefeld, Germany)进行纯化。使用SMRTbell barcode Adapter Complete Prep-96 kit (Pacific Bioscience)试剂盒将每个扩增子连接特定的barcode序列后等质量混合。扩增子混合物使用Pacific Biosciences SMRTbellTM Template Prep kit 1.0试剂盒构建测序文库并在PacBio RS Ⅱ上进行测序。
1.5 数据处理和分析 下机后的原始数据首先基于SMRT Portal version 2.3.0中的RS_ReadsOfinsert.1 protocol进行相应的barcode拆分,并得到每个样本的高质量的CCS (circular consensus sequencing)序列。然后我们按照以下条件进行数据过滤:(1)片段长度范围:1400–1600 bp;(2)去除包含“N”碱基的reads;(3)去除包含超过6 bp homopolymers的reads;(4)去除平均质量 < 90的序列。
过滤后的高质量数据使用Usearch软件(version=6.1.544)[20]进行OUT (operational taxonomic units)聚类及嵌合体去除。Usearch使用的聚类identity参数设为97%。所有过滤后的原始数据会重新比对上OTU代表序列,用于生成OTU丰度表。每个OTU代表序列比对到RDP数据库(Ribosomal Database Project Ⅱ database)得到其物种分类信息。α多样性稀释曲线由QIIME[21]和R软件绘制得到。原始的OTU表基于自行编写的脚本得到,并按照不同的分类层级进行统计(无法分类到某一层级的数据归为“Others”)。
基于16S rDNA测序数据的宏基因组功能预测分析是使用QIIME和PICRUSt软件[22]完成的。预测的功能主要包括KEGG (Kyoto Encyclopedia of Genes and Genomes pathways)和COG (Clusters of Orthologous Groups of proteins)数据库中的基因。
本研究中绘图使用的软件为R和GraphPad Prism6。
1.6 统计分析 本研究使用STAMP和R软件进行组间差异分析。R stats包中的wilcox.test函数用于两组间差异分析,kruskal.test函数用于多组间差异分析。STAMP软件中的ANOVA方法用于分析多组之间的差异,Welch’s t-test用于分析两组之间的差异。P值< 0.05被认为差异有统计学意义。核心肠道微生物的Spearman相关分析采用R软件的corrplot软件包。
2 结果和分析 2.1 组间临床信息差异分析 四组间的年龄、怀孕次数、生产次数均无差异,体重有显著差异(P=0.0024 < 0.01) (表 2)。同时患有妊娠期糖尿病的分组(GDM+,包括GDM+_Han和GDM+_Meng)的孕妇体重比正常孕妇的体重显著升高(P < 0.01)。BMI (body mass index)指数在四组间没有明显差异,而在GDM+ (包括GDM+_Han和GDM+_Meng)和GDM– (包括GDM–_Han和GDM–_Meng)两大组间表现出有显著差异(P < 0.01)。上述结果说明妊娠期高体重和高BMI的孕妇更容易患有妊娠期糖尿病,这也与之前的文献报道一致[23-24]。
表 2. 四组间的临床信息差异分析 Table 2. Differential analysis of clinical characteristics of the four groups
| Variable | GDM–_Han | GDM–_Meng | GDM+_Han | GDM+_Meng | P-value |
| Age/years | 27±3 | 29±4 | 29±4 | 29±4 | 0.1400 |
| Weight/kg | 66±15 | 65±12 | 73±13 | 72±8 | 0.0024 |
| BMIa/(kg/m2) | 0.0680 | ||||
| Healthy | 18 | 17 | 10 | 5 | |
| Overweight | 6 | 8 | 12 | 7 | |
| Obese | 2 | 2 | 5 | 5 | |
| Pregnancyb | 0.1742 | ||||
| N1 | 20 | 14 | 15 | 6 | |
| N2 | 3 | 9 | 9 | 7 | |
| N3 | 3 | 2 | 2 | 1 | |
| N4 | 0 | 1 | 1 | 2 | |
| N5 | 0 | 1 | 0 | 1 | |
| Birthc | 0.1696 | ||||
| P0 | 22 | 19 | 20 | 9 | |
| P1 | 4 | 8 | 6 | 8 | |
| P2 | 0 | 0 | 1 | 0 | |
| aBMI (body mass index) includes three levels: Healthy (BMI < 25), Overweight (25≤BMI≤30), and Obese (BMI > 30). bPregnancy indicates the number of pregnancies (N1, N2, N3, N4, N5 indicate the first, second, third, fourth and fifth pregnancy, respectively); and cBirth indicates the number of children (P0, P1, P2 indicate no child, first child and second child, respectively). | |||||
表选项
2.2 序列丰度和多样性统计分析 本研究基于PacBio SMRT测序技术对妊娠期孕妇的肠道菌群结构进行了解析,序列统计信息如表 3所示。经过数据过滤,97个样本共获得415139条reads,reads平均长度为1519 bp,平均每个样本的含有4280条reads,所有样本中reads数最少的含有1707条,最多的含有13250条,标准差为1622.403。
表 3. 序列统计信息 Table 3. Statistics of sequences
| Samples | Number of Raw reads | Number of Clean reads | Bases/bp | Average length/bp |
| 97 | 692792 | 415139 | 630514337 | 1519 |
表选项
α多样性反映了样本中微生物菌群的丰度、均匀性及多样性的情况。本文通过计算4个分组所有样本的Shannon指数、Simpson指数、Chao1指数和Observed species指数等α多样性指数(表 4),发现大多数样本含有较高的物种多样性。
表 4. 细菌α多样性 Table 4. Bacterial α-diversity
| Group names | Number of reads | Number of OTUs | Shannon index | Simpson index | Chao1 index | Observed species index |
| GDM+_Meng | 92529.00 | 508 | 5.96±0.52 | 0.96±0.02 | 278.36±39.35 | 241.47±40.56 |
| GDM+_Han | 93602.00 | 434 | 5.29±0.80 | 0.93±0.05 | 197.99±49.39 | 167.52±44.84 |
| GDM–_Meng | 106716.00 | 515 | 5.73±0.68 | 0.95±0.03 | 228.63±56.01 | 196.50±49.95 |
| GDM–_Han | 122292.00 | 522 | 5.78±0.51 | 0.96±0.02 | 243.98±54.15 | 211.48±47.45 |
表选项
从图 1可知,各个样本Shannon及Observed species多样性稀释曲线随着数据量的增加趋于平稳,表明各个样本的测序数据深度都已足够。为避免因样本数据大小不同而造成分析时的偏差,本研究对每个样本的测序数据进行随机抽平处理,选择reads抽平数目为1707。随机抽平处理后的所有样本共有576个OTUs (operational taxonomic units),平均每个样本的OTU数目为162。
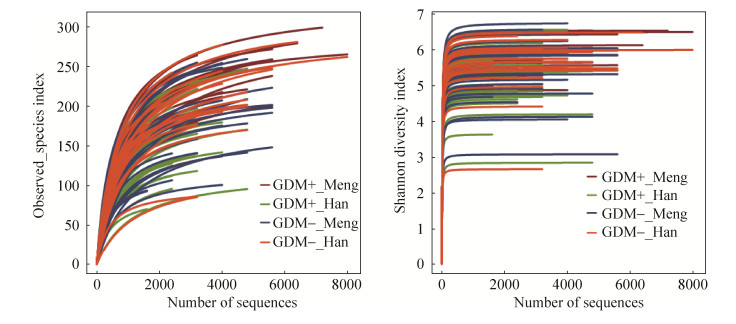 |
| 图 1 Shannon及observed species多样性稀释曲线 Figure 1 Shannon diversity index curves and observed species index curves for the sequenced sample. |
| 图选项 |
本研究发现不同分组间的α多样性存在一定的差异(图 2)。汉族患有GDM的孕妇和健康孕妇之间的Observed species多样性和Shannon多样性均有显著差异(P < 0.05);蒙古族患有GDM的孕妇和健康孕妇之间Observed species多样性有差异(P < 0.05)。这一结果说明患有GDM的孕妇和健康孕妇,无论是蒙古族还是汉族,菌群结构上均有一定的差异。另外,在GDM组中,蒙古族和汉族孕妇肠道菌群中Observed species多样性和Shannon多样性均有显著差异(P < 0.01);但在健康对照组中,不同民族间没有找到显著差异。并且四组间Observed species多样性和Shannon多样性均有显著差异(P < 0.05),说明四组样本间的肠道菌群结构存在一定差异。
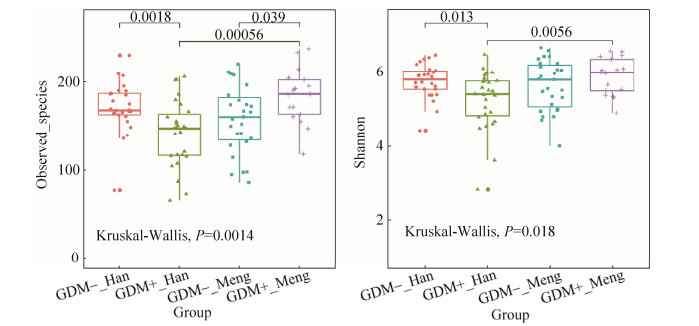 |
| 图 2 组间α多样性指数比较分析 Figure 2 Comparison of α-diversity indexes among groups. |
| 图选项 |
2.3 肠道菌群的物种组成分析 本研究分别从界、门、纲、目、科、属、种这几个水平上对菌群组成进行鉴定及分析,最终共鉴定到了9个门、48个属、44个种。如图 3-A所示,在门的水平上,主要包括拟杆菌门(52.82%)、厚壁菌门(40.44%)和变形菌门(5.39%)这3个门(占所有数据的98.65%)。还有一些低微量的门被鉴定到,如广古菌门、放线菌门、黏胶球形菌门、柔壁菌门和疣微菌门等。在属的水平上,本文共鉴定到48个属,其中平均相对丰度大于1%的优势属有Bacteroides (38.02%)、Prevotella (6.44%)、Faecalibacterium (4.88%)、Lachnospira (4.70%)、Sutterella (4.10%)、Roseburia (3.74%)、Coprococcus (2.99%)、Parabacteroides (2.89%)、Ruminococcus (2.44%)、Dialister (1.68%)、Phascolarctobacterium (1.62%)、Oscillospira (1.37%)、Blautia (1.29%)等。图 3-B显示了各个分组菌群在种的水平上的分布情况,共鉴定到44个不同的种,分别有Bacteroides uniformis (5.47%)、Prevotella copri (5.46%)、Faecalibacterium prausnitzii (4.87%)、Bacteroides plebeius (4.31%)、Parabacteroides distasonis (1.38%)、Bacteroides ovatus (1.33%)等等,也包括一些低微量的菌,如Akkermansia muciniphila (0.64%)等。
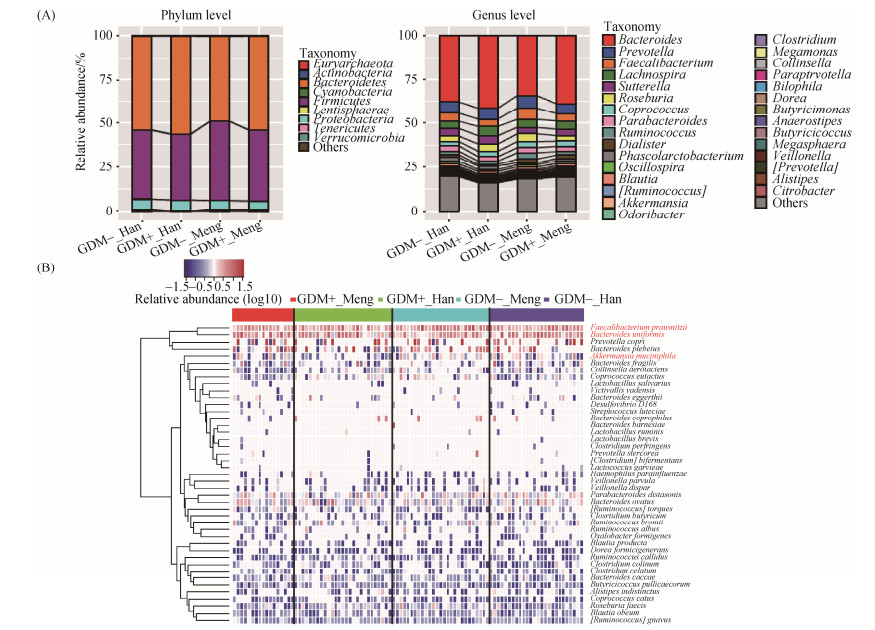 |
| 图 3 四组间菌群门、属水平相对丰度柱状图(A)及种水平热图(B) Figure 3 Bar plots of the relative abundances of intestinal flora at the phylum and genus level (A) and heatmap at the species level (B) for the four groups. |
| 图选项 |
2.4 妊娠期孕妇核心肠道菌群分析 基于OTU水平的Rank Abundance曲线显示,4个分组中所有样本在排序最靠前的200个OTUs上的曲线高度重合,表明所有分组中优势OTUs高度相似(图 4-B)。如图 4-A韦恩图所示,本研究发现4个分组间共有398个OTUs,占所有OTU数目的69.3% (398/574),说明4个分组间存在核心OTUs。这些OTUs可能代表了样本的核心肠道微生物,对孕妇的健康状态可能有着重要的作用。本研究对这398个核心OTUs进行筛选,挑选出可以分类到属水平和种水平的OTUs,并对这些OTUs代表的核心微生物进行了Spearman相关性系数分析。如图 4-C所示,核心微生物之间的相关性大小各有差异,如Bacteroides属与Prevotella属的呈强负相关,而与Akkermansia属呈弱负相关,说明这些核心微生物之间的相互关系十分复杂,这种复杂的关系网络可能对肠道菌群的动态变化起着重要的作用。
 |
| 图 4 妊娠期孕妇核心肠道菌群分析 Figure 4 Analysis of the core microbiota of pregnant women. A: Venn diagram of operational taxonomic units (OTUs) among the four groups; B: Rank abundance curves of OTUs; C: Spearman correlation analysis between the core microbiota at the genus and species levels. |
| 图选项 |
2.5 妊娠期糖尿病人与正常人肠道菌群的结构与功能比较 本研究基于主成分坐标分析(principle coordinate analysis,PCoA),探究不同健康状况以及不同民族样本的肠道菌群之间的关系。Weighted和Unweighted UniFrac距离矩阵均被用于PCoA分析。结果如图 5所示,Unweighted UniFrac主成分1 (PC1)、2 (PC2)和3 (PC3)分别为17.3%、6.4%和5.0%;Weighted UniFrac主成分1、2和3分别为33.2%、15.6%和9.4%。但无论基于哪种距离算法,从PC1和PC2、PC1和PC3、PC2和PC3维度上均没有发现明显的聚类。
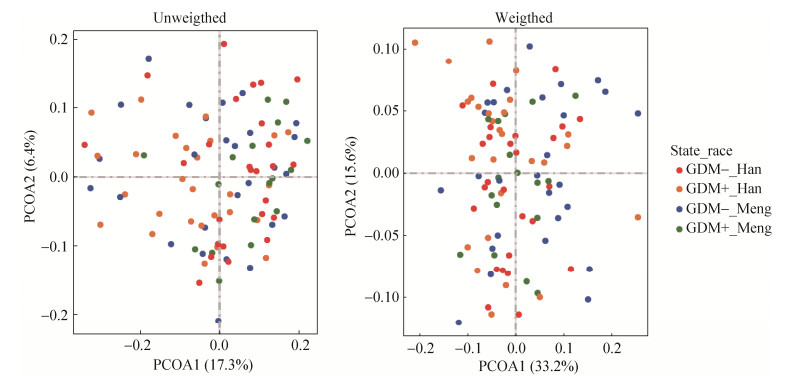 |
| 图 5 基于主成分1和2的UniFrac unweighted和weighted PCoA图 Figure 5 UniFrac unweighted and weighted principal coordinate analysis (PCoA) scores plot based on principal components 1 and 2. |
| 图选项 |
虽然聚类结果显示4组样本肠道菌群的主要构成没有明显差异,但本研究接着利用STAMP软件在种的水平上找到了不同分组间的一些差异菌株,如图 6-A所示。GDM–_Han组疣微菌门Akkermansia属的Akkermansia muciniphila菌的相对丰度要显著高于GDM+_Han组(P < 0.05);GDM–_Meng组中Blautia obeum菌的相对丰度要高于GDM+_Meng组,但P值接近0.05,差异并不极显著。GDM+_Han和GDM–_Meng分组间A. muciniphila菌和Faecalibacterium prausnitzii菌有差异,且这2个菌的相对丰度在GDM–_Meng分组中高于GDM+_Han组。F. prausnitzii菌大约占了肠道菌群的5%,是一种常见的人类肠道共生菌。
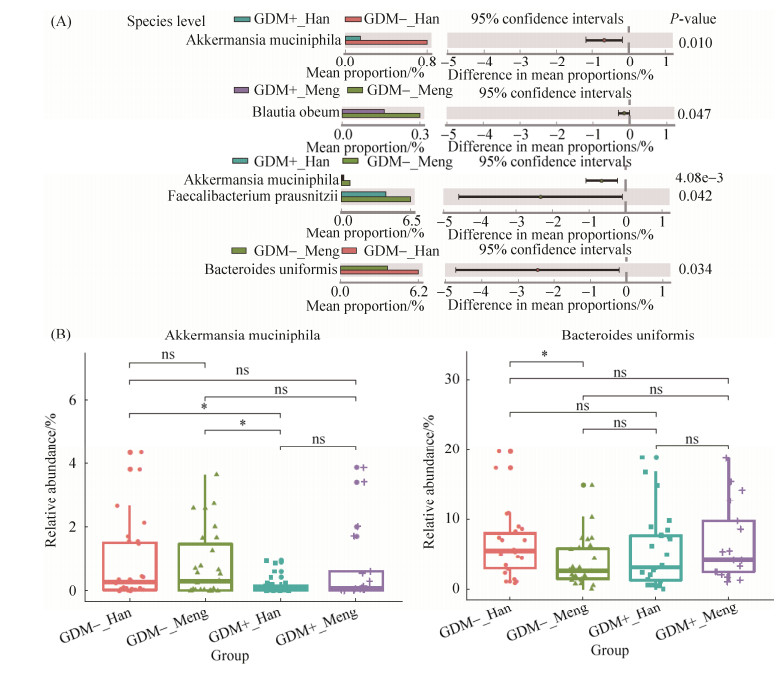 |
| 图 6 四组间种水平上菌群结构的比较 Figure 6 Comparison of bacterial composition among the four groups at the species level. |
| 图选项 |
此外,在不同民族人群的肠道菌群间也能鉴定到一些差异菌株。在健康对照组中,健康汉族人群中拟杆菌门Bacteroides uniformis菌(Bacteroides uniformis菌对应的门水平为拟杆菌门)的相对丰度要高于健康蒙古族人组(P=0.034 < 0.05);但在患有GDM的蒙古族和汉族孕妇组间没有找到显著差异。蒙古族人和汉族人间厚壁菌门、拟杆菌门以及Ruminococcus属存在差异,并且汉族人的拟杆菌门/厚壁菌门比例(即Bacteroidetes/Firmicutes,B/F ratio)要大于蒙古族人。以往研究表明,肠道菌群的B/F比例可能与肥胖相关[25-26]。
为了了解妊娠期肠道菌群的代谢情况,本研究同时也利用PICRUSt软件对肠道菌群进行了功能预测分析。与菌群结构的结果相同,不同分组间菌群的功能组成也高度相似,在整体水平上没有表现出显著差异,碳水化合物代谢是最主要的功能模块。基于三代PacBio SMRT测序得到的高质量数据,本研究利用STAMP软件对4组间菌群功能进行了差异分析,在4组间共鉴定到266个差异KOs (KEGG Orthology)基因和332个COGs基因(P < 0.05,ANOVA test analysis)。在汉族人中,患病与正常人之间没有找到显著功能差异;但在蒙古族人中,GDM患者肠道菌群中无机离子的运输和代谢([P] Inorganic ion transport and metabolism)及其他氨基酸代谢(metabolism of other amino acids)、细胞过程和信号(cellular processes and signaling)等功能丰度显著高于正常人(图 7),但是均非极显著(0.01 < P < 0.05)。同时我们也比较了不同民族间肠道菌群的功能差异,发现在健康对照组中,汉族和蒙古族间其他氨基酸代谢(metabolism of other amino acids)、新陈代谢(metabolism)、运输和分解代谢(transport and catabolism)及信号分子及相互作用(signaling molecules and interaction)有差异;但在患有GDM的分组中,不同种族间没有发现显著差异。
 |
| 图 7 组间功能预测比较分析 Figure 7 Comparison of functional predictions among the four groups. A: COG functional prediction; B: KEGG module prediction. |
| 图选项 |
3 讨论 以往研究发现[11]妊娠期孕妇的肠道菌群在不同的孕期是会发生变化的,如变形菌门和放线菌门的丰度均会有所增加;但也有一些研究[27]表明怀孕早期和晚期孕妇的肠道菌群差异并不明显。而目前关于GDM孕妇肠道菌群的相关研究较少,仅有少量研究报道超重孕妇肠道菌群中Bacteroides和Staphylococcus明显高于正常体重的孕妇[10]。关于GDM与健康孕妇的肠道菌群是否存在明显差异尚未有定论,有待进一步研究。本研究首次基于三代测序技术测得了妊娠期孕妇肠道菌群的全长16S rRNA序列,其高准确性可以更精确地将菌群鉴定到种水平,从而有助于更灵敏地检测到各个分组间一些低丰度菌的差异。
3.1 妊娠期孕妇核心肠道菌群分析 本研究根据不同的健康状态及不同民族将97个孕妇分成了4个组(表 1)。通过对4个分组的临床信息进行差异分析,本研究发现妊娠期高体重和BMI的孕妇更容易患有妊娠期糖尿病,这也与之前的文献报道一致[23-24]。另外本研究也发现不同分组间的α多样性有一定的差异,说明不同分组间肠道菌群的结构有一定的差异。但整体上来看,4个分组间样本的肠道菌群的相似性很高,而核心肠道菌群能够反映妊娠期孕妇肠道菌群的大体状态,因此我们首先对所有妊娠期孕妇的核心肠道菌群进行了分析,结果显示四组样本有相似的核心菌群,共鉴定到398个核心OTUs,占所有OTU数目的69.3%。其中拟杆菌门、厚壁菌门和变形菌门是最主要的3个门,这与以往的GDM肠道菌群的相关研究中报道的一致[28]。在种的水平上,本研究总共鉴定到了44个种,大多数都是人类肠道菌群中的常见菌,如Bacteroides uniformis、Faecalibacterium prausnitzii及Prevotella copri等[29-32]。本研究同时也对不同种属的核心菌进行了Spearman相关性分析,发现不同核心菌之间的相关性强弱不同,说明核心菌群间的相互关系十分复杂,这种复杂的关系网络可能对肠道菌群的动态变化起着重要的作用,有待进一步的研究。
3.2 组间差异菌株与妊娠期糖尿病的潜在关系 基于PCoA分析发现4个分组在各个主成分上均没有发现明显聚类,说明4个分组整体上是高度相似的。但基于三代PacBio SMRT测序技术能更准确地将菌群鉴定到种水平的优势,本文首次在种的水平上发现了GDM孕妇和健康孕妇肠道菌群的一些低微量菌群的差异。GDM–_Han与GDM+_Han组间A. muciniphila菌有显著差异,并且健康对照组中A. muciniphila菌的相对丰度要略高于GDM组。而在以往的一项GDM相关研究中[33],只是在属水平发现Akkermansia属与较低的胰岛素敏感性有一定的关系。上述结果说明基于三代测序进行菌群分析的方法能够更精确地鉴定到不同分组间种水平上的差异。以往研究发现A. muciniphila菌可能与肥胖和二型糖尿病有关[34-35]。A. muciniphila菌的丰度与胰岛素敏感性有一定的联系,A. muciniphila菌与肠道内壁具有一定的共生关系,可以影响肠道吸收[36]。膳食中的膳食脂肪可以影响A. muciniphilia菌的生长,从而影响其在肠道菌群的含量,使得肠道的微环境发生变化[36]。同时本文也发现GDM–_Meng组中Blautia obeum菌的相对丰度要略高于GDM+_Meng组,但是P值接近0.05,并不极显著。另外,我们在GDM–_Meng和GDM+_Han组间发现Faecalibacterium prausnitzii菌的丰度有显著差异。以往的医学相关的研究发现,肠道内如果F. prausnitzii菌丰度低于正常水平可能与克罗恩氏病、肥胖、哮喘、重度抑郁症等相关;丰度高于正常水平可能与牛皮癣等疾病相关[37-39]。低丰度的丁酸盐和丙酸盐产生菌,如F. prausnitzii菌和A. muciniphila菌,是炎症性肠病(inflammatory bowel disease,IBD)的普遍特征[40-41]。综上,我们推测A. muciniphilia菌和F. prausnitzii菌丰度的变化在GDM的发病过程中可能有着一定的作用。
3.3 民族与妊娠期孕妇肠道菌群的关联 本研究通过对不同民族孕妇的肠道菌群进行差异分析发现,在健康对照组中,汉族人中B. uniformis菌的相对丰度显著高于蒙古族人,B. uniformis菌与人类肠道菌群异黄酮的降解有关[29];而在GDM组中,汉族和蒙古族人间没有找到显著差异。并且在蒙古族人(即Meng,包括GDM+_Meng和GDM–_Meng组)以及汉族人(即Han,包括GDM+_Han和GDM–_Han组)组间也没有找到类似的差异。并且,本研究发现在同一分组中B. uniformis的相对丰度存在较大差别,因此,本研究推测在健康人不同民族间找到的差异可能是由于分组中存在极值导致的。以往的一些研究中认为,由于其地理位置以及饮食差异导致不同民族间的肠道菌群存在一定的差异[42-43],而在本研究中,所有孕妇均居住在市区,蒙古族人与汉族人的饮食习惯等差异较小,另一方面,由于妊娠期的孕妇在妊娠期的饮食差异上均较为控制,进一步缩小了饮食上的差异。这也从另一个角度反映了在GDM的发病过程中,孕妇的遗传多样性可能不会对菌群结构变化产生极大影响。
3.4 组间差异功能与妊娠期糖尿病的潜在关系 基于功能预测的结果,本研究发现4个分组的主要功能是高度相似的,大多数都是和能量代谢相关,这也与我们分析得到4个分组间菌群结构高度相似的结果相吻合。而基于PacBio SMRT测序技术得到的高准确性的数据,本研究发现了不同分组间功能组成上的一些差异。在汉族人中,患病与健康对照组间没有找到差异;而在蒙古族人中,患有GDM孕妇肠道菌群中无机离子运输等代谢丰度要显著高于健康孕妇,但均非极显著(0.01 < P < 0.05)。人体肠道菌群中无机离子运输及代谢变化可能会影响人体内无机离子的代谢平衡,而无机离子在人体内有着极其重要的作用[44]。临床研究表明,某些特定无机盐的早期失衡也会扰乱人体内正常的葡萄糖和胰岛素的代谢。目前临床上报道了很多单一无机盐或者某些无机盐的组合对糖尿病的影响[45-47]。但目前肠道菌群中的无机离子代谢与人体内无机盐代谢及糖尿病的相互关系仍需进一步的研究。同时本研究发现在健康对照组中,汉族和蒙古族孕妇肠道菌群中其他氨基酸代谢(metabolism of other amino acids)、新陈代谢(metabolism)、运输和分解代谢(transport and catabolism)及信号分子及相互作用(signaling molecules and interaction)有差异;但在患有GDM的分组中没有发现显著差异。但本研究发现同一分组中不同样本个体间肠道菌群代谢功能丰度差异较大,说明不同个体间肠道菌群代谢相差较大。而在组间找到的这些差异很可能是由于一些低微量菌群的丰度变化导致的,说明不同类型的微生物在核心菌群中所处的功能定位以及与人体产生相互作用的方式可能并不完全相同。
3.5 本研究推论 在怀孕的整个过程中,孕妇的肠道菌群会逐渐发生变化[48],以适应怀孕阶段的生理变化。本研究的结果显示,无论是汉/蒙古族还是GDM与否,在特定的怀孕阶段的孕妇的肠道菌群,尤其是核心菌群,都是处于相对稳态的,而GDM的发生可能是与一些低微量的菌群,如A. muciniphilia菌的丰度变化存在关联。虽然这些低微量的菌在整个菌群中的绝对比例并不高,但很可能会导致肠道中一些与肠道营养吸收等有关的代谢发生变化,从而导致孕妇血糖升高,进而诱发糖尿病。同时由于每个人体质的不同,其中一些人的代谢平衡被打破,从而诱发GDM或者其他疾病。但是这一结论还需要进一步的研究加以证明。
References
| [1] | Ferrara A. Increasing prevalence of gestational diabetes mellitus. Diabetes Care, 2007, 30(S2): S141-S146. |
| [2] | Nelson SM, Matthews P, Poston L. Maternal metabolism and obesity:modifiable determinants of pregnancy outcome. Human Reproduction Update, 2010, 16(3): 255-275. DOI:10.1093/humupd/dmp050 |
| [3] | The HAPO Study Cooperative Research Group. Hyperglycemia and adverse pregnancy outcomes. New England Journal of Medicine, 2008, 358(19): 1991-2002. DOI:10.1056/NEJMoa0707943 |
| [4] | Landon MB, Mele L, Spong CY, Carpenter MW, Ramin SM, Casey B, Wapner RJ, Varner MW, Rouse DJ, Thorp Jr JM, Sciscione A, Catalano P, Harper M, Saade G, Caritis SN, Sorokin Y, Peaceman AM, Tolosa JE, Anderson GD. Eunice Kennedy Shriver National Institute of Child Health, and Human Development (NICHD) Maternal-Fetal Medicine Units (MFMU) Network. The relationship between maternal glycemia and perinatal outcome. Obstetrics and Gynecology, 2011, 117: 218-224. DOI:10.1097/AOG.0b013e318203ebe0 |
| [5] | Bellamy L, Casas JP, Hingorani AD, Williams D. Type 2 diabetes mellitus after gestational diabetes:a systematic review and meta-analysis. The Lancet, 2009, 373(9677): 1773-1779. DOI:10.1016/S0140-6736(09)60731-5 |
| [6] | Gomez-Arango LF, Barrett HL, McIntyre HD, Callaway LK, Morrison M, Dekker Nitert M, SPRING Trial Group. Connections between the gut microbiome and metabolic hormones in early pregnancy in overweight and obese women. Diabetes, 2016, 65(8): 2214-2223. DOI:10.2337/db16-0278 |
| [7] | Vrieze A, Holleman F, Zoetendal EG, de Vos WM, Hoekstra JBL, Nieuwdorp M. The environment within:how gut microbiota may influence metabolism and body composition. Diabetologia, 2010, 53(4): 606-613. DOI:10.1007/s00125-010-1662-7 |
| [8] | Musso G, Gambino R, Cassader M. Interactions between gut microbiota and host metabolism predisposing to obesity and diabetes. Annual Review of Medicine, 2011, 62: 361-380. DOI:10.1146/annurev-med-012510-175505 |
| [9] | Vijay-Kumar M, Aitken JD, Carvalho FA, Cullender TC, Mwangi S, Srinivasan S, Sitaraman SV, Knight R, Ley RE, Gewirtz AT. Metabolic syndrome and altered gut microbiota in mice lacking toll-like receptor 5. Science, 2010, 328(5975): 228-231. DOI:10.1126/science.1179721 |
| [10] | Collado MC, Isolauri E, Laitinen K, Salminen S. Distinct composition of gut microbiota during pregnancy in overweight and normal-weight women. The American Journal of Clinical Nutrition, 2008, 88(4): 894-899. DOI:10.1093/ajcn/88.4.894 |
| [11] | Koren O, Goodrich JK, Cullender TC, Spor A, Laitinen K, Kling B?ckhed H, Gonzalez A, Werner JJ, Angenent LT, Knight R, B?ckhed F, Isolauri E, Salminen S, Ley RE. Host remodeling of the gut microbiome and metabolic changes during pregnancy. Cell, 2012, 150(3): 470-480. DOI:10.1016/j.cell.2012.07.008 |
| [12] | Fugmann M, Breier M, Rottenkolber M, Banning F, Ferrari U, Sacco V, Grallert H, Parhofer KG, Seissler J, Clavel T, Lechner A. The stool microbiota of insulin resistant women with recent gestational diabetes, a high risk group for type 2 diabetes. Scientific Reports, 2015, 5: 13212. DOI:10.1038/srep13212 |
| [13] | Kuang YS, Lu JH, Li SH, Li JH, Yuan MY, He JR, Chen NN, Xiao WQ, Shen SY, Qiu L, Wu YF, Hu CY, Wu YY, Li WD, Chen QZ, Deng HW, Papasian CJ, Xia HM, Qiu X. Connections between the human gut microbiome and gestational diabetes mellitus. Gigascience, 2017, 6(8): 1-12. |
| [14] | Schloss PD, Jenior ML, Koumpouras CC, Westcott SL, Highlander SK. Sequencing 16S rRNA gene fragments using the PacBio SMRT DNA sequencing system. PeerJ, 2016, 4: e1869. DOI:10.7717/peerj.1869 |
| [15] | Wagner J, Coupland P, Browne HP, Lawley TD, Francis SC, Parkhill J. Evaluation of PacBio sequencing for full-length bacterial 16S rRNA gene classification. BMC Microbiology, 2016, 16: 274. DOI:10.1186/s12866-016-0891-4 |
| [16] | Singer E, Bushnell B, Coleman-Derr D, Bowman B, Bowers RM, Levy A, Gies EA, Cheng JF, Copeland A, Klenk HP, Hallam SJ, Hugenholtz P, Tringe SG, Woyke T. High-resolution phylogenetic microbial community profiling. The ISME Journal, 2016, 10(8): 2020-2032. DOI:10.1038/ismej.2015.249 |
| [17] | Mosher JJ, Bowman B, Bernberg EL, Shevchenko O, Kan JJ, Korlach J, Kaplan LA. Improved performance of the PacBio SMRT technology for 16S rDNA sequencing. Journal of Microbiological Methods, 2014, 104: 59-60. DOI:10.1016/j.mimet.2014.06.012 |
| [18] | Li J, Xu HY, Sun ZH, Hou QC, Kwok LY, Laga W, Wang YJ, Ma HM, Yu ZJ, Menghe BLG, Zhang HP. Effect of dietary interventions on the intestinal microbiota of Mongolian hosts. Science Bulletin, 2016, 61(20): 1605-1614. DOI:10.1007/s11434-016-1173-0 |
| [19] | Wang Y, Zhang J, Chen X, Jiang W, Wang S, Xu L, Tu Y, Zheng P, Wang Y, Lin XL, Chen H. Profiling of oral microbiota in early childhood caries using single-molecule real-time sequencing. Frontiers in Microbiology, 2017, 8: 2244. DOI:10.3389/fmicb.2017.02244 |
| [20] | Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [21] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Pe?a AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [22] | Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, Reyes JA, Clemente JC, Burkepile DE, Vega Thurber RL, Knight R, Beiko RG, Huttenhower C. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [23] | Hedderson MM, Williams MA, Holt VL, Weiss NS, Ferrara A. Body mass index and weight gain prior to pregnancy and risk of gestational diabetes mellitus. American Journal of Obstetrics and Gynecology, 2008, 198(4): 409.e1-409.e7. DOI:10.1016/j.ajog.2007.09.028 |
| [24] | Chu SY, Callaghan WM, Kim SY, Schmid CH, Lau J, England LJ, Dietz PM. Maternal obesity and risk of gestational diabetes mellitus. Diabetes Care, 2007, 30(8): 2070-2076. DOI:10.2337/dc06-2559a |
| [25] | Ley RE, Turnbaugh PJ, Klein S, Gordon JI. Human gut microbes associated with obesity. Nature, 2006, 444(7122): 1022-1023. DOI:10.1038/4441022a |
| [26] | Verdam FJ, Fuentes S, de Jonge C, Zoetendal EG, Erbil R, Greve JW, Buurman WA, de Vos WM, Rensen SS. Human intestinal microbiota composition is associated with local and systemic inflammation in obesity. Obesity, 2013, 21(12): E607-E615. DOI:10.1002/oby.20466 |
| [27] | Avershina E, Storro O, ?ien T, Johnsen R, Pope P, Rudi K. Major faecal microbiota shifts in composition and diversity with age in a geographically restricted cohort of mothers and their children. FEMS Microbiology Ecology, 2014, 87(1): 280-290. DOI:10.1111/1574-6941.12223 |
| [28] | Wang JF, Zheng JY, Shi WY, Du N, Xu XM, Zhang YM, Ji PF, Zhang FY, Jia Z, Wang YP, Zheng Z, Zhang HP, Zhao FQ. Dysbiosis of maternal and neonatal microbiota associated with gestational diabetes mellitus. Gut, 2018, 67(9): 1614-1625. DOI:10.1136/gutjnl-2018-315988 |
| [29] | Renouf M, Hendrich S. Bacteroides uniformis is a putative bacterial species associated with the degradation of the isoflavone genistein in human feces. The Journal of Nutrition, 2011, 141(6): 1120-1126. DOI:10.3945/jn.111.140988 |
| [30] | Gauffin Cano P, Santacruz A, Moya á, Sanz Y. Bacteroides uniformis CECT 7771 ameliorates metabolic and immunological dysfunction in mice with high-fat-diet induced obesity. PLoS One, 2012, 7(7): e41079. DOI:10.1371/journal.pone.0041079 |
| [31] | Miquel S, Martín R, Rossi O, Bermúdez-Humarán LG, Chatel JM, Sokol H, Thomas M, Wells JM, Langella P. Faecalibacterium prausnitzii and human intestinal health. Current Opinion in Microbiology, 2013, 16(3): 255-261. DOI:10.1016/j.mib.2013.06.003 |
| [32] | Pianta A, Arvikar S, Strle K, Drouin EE, Wang Q, Costello CE, Steere AC. Evidence of the immune relevance of Prevotella copri, a gut microbe, in patients with rheumatoid arthritis. Arthritis & Rheumatology, 2017, 69(5): 964-975. |
| [33] | Crusell MKW, Hansen TH, Nielsen T, Allin KH, Rühlemann MC, Damm P, Vestergaard H, R rbye C, J rgensen NR, Christiansen OB, Heinsen FA, Franke A, Hansen T, Lauenborg J, Pedersen O. Gestational diabetes is associated with change in the gut microbiota composition in third trimester of pregnancy and postpartum. Microbiome, 2018, 6: 89. DOI:10.1186/s40168-018-0472-x |
| [34] | Everard A, Belzer C, Geurts L, Ouwerkerk JP, Druart C, Bindels LB, Guiot Y, Derrien M, Muccioli GG, Delzenne NM, de Vos WM, Cani PD. Cross-talk between Akkermansia muciniphila and intestinal epithelium controls diet-induced obesity. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(22): 9066-9071. DOI:10.1073/pnas.1219451110 |
| [35] | Plovier H, Everard A, Druart C, Depommier C, Van Hul M, Geurts L, Chilloux J, Ottman N, Duparc T, Lichtenstein L, Myridakis A, Delzenne NM, Klievink J, Bhattacharjee A, van der Ark KCH, Aalvink S, Martinez LO, Dumas ME, Maiter D, Loumaye A, Hermans MP, Thissen JP, Belzer C, de Vos WM, Cani PD. A purified membrane protein from Akkermansia muciniphila or the pasteurized bacterium improves metabolism in obese and diabetic mice. Nature Medicine, 2016, 23(1): 107-113. |
| [36] | Dao MC, Everard A, Aron-Wisnewsky J, Sokolovska N, Prifti E, Verger EO, Kayser BD, Levenez F, Chilloux J, Hoyles L, Dumas ME, Rizkalla SW, Doré J, Cani PD, Clément K. Akkermansia muciniphila and improved metabolic health during a dietary intervention in obesity:relationship with gut microbiome richness and ecology. Gut, 2016, 65(3): 426-436. DOI:10.1136/gutjnl-2014-308778 |
| [37] | Jiang HY, Ling ZX, Zhang YH, Mao HJ, Ma ZP, Yin Y, Wang WH, Tang WX, Tan ZL, Shi JF, Li LJ, Ruan B. Altered fecal microbiota composition in patients with major depressive disorder. Brain, Behavior, and Immunity, 2015, 48: 186-194. DOI:10.1016/j.bbi.2015.03.016 |
| [38] | Sokol H, Pigneur B, Watterlot L, Lakhdari O, Bermúdez-Humarán LG, Gratadoux JJ, Blugeon S, Bridonneau C, Furet JP, Corthier G, Grangette C, Vasquez N, Pochart P, Trugnan G, Thomas G, Blottière HM, Doré J, Marteau P, Seksik P, Langella P. Faecalibacterium prausnitzii is an anti-inflammatory commensal bacterium identified by gut microbiota analysis of Crohn disease patients. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(43): 16731-16736. DOI:10.1073/pnas.0804812105 |
| [39] | Codo?er FM, Ramírez-Bosca A, Climent E, Carrión-Gutierrez M, Guerrero M, Pérez-Orquín JM, de la Parte JH, Genovés S, Ramón D, Navarro-López V, Chenoll E. Gut microbial composition in patients with psoriasis. Scientific Reports, 2018, 8: 3812. DOI:10.1038/s41598-018-22125-y |
| [40] | Sokol H, Seksik P, Furet JP, Firmesse O, Nion-Larmurier I, Beaugerie L, Cosnes J, Corthier G, Marteau P, Doré J. Low counts of Faecalibacterium prausnitzii in colitis microbiota. Inflammatory Bowel Diseases, 2009, 15(8): 1183-1189. DOI:10.1002/ibd.20903 |
| [41] | Png CW, Lindén SK, Gilshenan KS, Zoetendal EG, McSweeney CS, Sly LI, McGuckin MA, Florin THJ. Mucolytic bacteria with increased prevalence in ibd mucosa augment in vitro utilization of mucin by other bacteria. The American Journal of Gastroenterology, 2010, 105(11): 2420-2428. DOI:10.1038/ajg.2010.281 |
| [42] | Yatsunenko T, Rey FE, Manary MJ, Trehan I, Dominguez-Bello MG, Contreras M, Magris M, Hidalgo G, Baldassano RN, Anokhin AP, Heath AC, Warner B, Reeder J, Kuczynski J, Caporaso JG, Lozupone CA, Lauber C, Clemente JC, Knights D, Knight R, Gordon JI. Human gut microbiome viewed across age and geography. Nature, 2012, 486(7402): 222-227. DOI:10.1038/nature11053 |
| [43] | Zhang JC, Guo Z, Xue ZS, Sun ZH, Zhang MH, Wang LF, Wang GY, Wang F, Xu J, Cao HF, Xu HY, Lv Q, Zhong Z, Chen YF, Qimuge S, Menghe BLG, Zheng Y, Zhao LP, Chen W, Zhang HP. A phylo-functional core of gut microbiota in healthy young Chinese cohorts across lifestyles, geography and ethnicities. The ISME Journal, 2015, 9(9): 1979-1990. DOI:10.1038/ismej.2015.11 |
| [44] | Khurshid SJ, Qureshi S, H I. The role of inorganic elements in human body. Nucleus, 1984, 21(4): 3-23. |
| [45] | Kazi TG, Afridi HI, Kazi N, Jamali MK, Arain MB, Jalbani N, Kandhro GA. Copper, chromium, manganese, iron, nickel, and zinc levels in biological samples of diabetes mellitus patients. Biological Trace Element Research, 2008, 122(1): 1-18. DOI:10.1007/s12011-007-8062-y |
| [46] | Carlstr?m M, Larsen FJ, Nystr?m T, Hezel M, Borniquel S, Weitzberg E, Lundberg JO. Dietary inorganic nitrate reverses features of metabolic syndrome in endothelial nitric oxide synthase-deficient mice. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(41): 17716-17720. DOI:10.1073/pnas.1008872107 |
| [47] | Maull EA, Ahsan H, Edwards J, Longnecker MP, Navas-Acien A, Pi JB, Silbergeld EK, Styblo M, Tseng CH, Thayer K A, Loomis D. Evaluation of the association between arsenic and diabetes:a national toxicology program workshop review. Environmental Health Perspectives, 2012, 120(12): 1658-1670. DOI:10.1289/ehp.1104579 |
| [48] | Newbern D, Freemark M. Placental hormones and the control of maternal metabolism and fetal growth. Current Opinion in Endocrinology, Diabetes, and Obesity, 2011, 18(6): 409-416. DOI:10.1097/MED.0b013e32834c800d |
