张晓钗, 李亮

 , 何宁芳, 龚雪晴, 主朋月, 王晓阳
, 何宁芳, 龚雪晴, 主朋月, 王晓阳 河北工业大学化学工程与技术学院, 天津 300130
收稿日期:2018-09-20;修回日期:2019-01-02;网络出版日期:2019-01-10
基金项目:国金自然科学基金(31801948);河北省教育厅项目(QN2018014);河北工业大学大学生创新创业项目(201810080013)
*通信作者:李亮, Tel:+86-22-60204976, Fax:+86-22-60204976, E-mail:liangli@hebut.edu.cn.
摘要:[目的] 通过对杜氏盐藻的转录组进行测序和基因功能分析,阐明不同浓度盐胁迫对杜氏盐藻生长发育以及不同信号途径的影响。[方法] 分别获取9% NaCl浓度和24% NaCl浓度培养下的杜氏盐藻转录组并通过Illumina平台进行测序。将所得的序列进行拼接、去冗余处理。[结果] 获得40682个unigenes,其中注释到NR数据库的10905个,注释到NT数据库的2768个,注释到SWISS-PROT数据库的7261个,注释到COG/KOG数据库的6499个。受到高盐胁迫的杜氏盐藻细胞相比低盐环境下,有717个基因表达上调,1012个基因表达下调。进一步对60个显著差异基因进行了功能聚类,发现盐胁迫诱导了光合作用途径的基因表达。[结论] 杜氏盐藻通过提高光合作用基因表达增强耐盐性。该研究最大范围上挖掘了杜氏盐藻在高盐和低盐环境的基因转录水平,为深入揭示杜氏盐藻盐胁迫下基因差异表达提供了平台,并为进一步研究杜氏盐藻耐盐机理提供理论依据。
关键词:盐胁迫杜氏盐藻转录组基因注释耐盐
Gene expression profiling of Dunaliella salina under different salinity stress
Xiaochai Zhang, Liang Li

 , Ningfang He, Xueqing Gong, Pengyue Zhu, Xiaoyang Wang
, Ningfang He, Xueqing Gong, Pengyue Zhu, Xiaoyang Wang School of Chemical Engineering and Technology, Hebei University of Technology, Tianjin 300130, China
*Corresponding author: Liang Li, Tel:+86-22-60204976, Fax:+86-22-60204976, E-mail:liangli@hebut.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31801948), by the Scientific Research Fund of Hebei Provincial Education Department (QN2018014) and by the Innovative Entrepreneurial Training Plan of College Students in Hebei University of Technology (201810080013)
Abstract: [Objective] Through sequencing and genetic function analysis of the transcripts of Dunaliella salina, we studied its response to salt stress. [Methods] The transcriptome of Dunaliella salina under 9% and 24% NaCl concentration was obtained and sequenced by Illumina platform. All the unigenes were finally obtained by sequence splicing, redundancy removing based on the sequence clustering software. Functional notation and cluster analysis of unigenes were performed by comparing the unigenes to the database. [Results] A total of 40682 unigenes were obtained, including 10905 in the NR database, 2768 in the NT database, 7261 in the Swiss-prot database, and 6499 in the COG/KOG database. By comparing cells grown under hyper-and hyposaline conditions, 717 genes (41.46%) increased expression and 1012 genes (58.5%) decreased expression. Through the data integration analysis, 60 different expression genes of Dunaliella salina under salt stress were excavated. [Conclusion] Salinity stress up-regulated key genes in photosynthesis. Our findings provide reference for dissecting molecular mechanisms of salinity tolerance in algae and higher plants.
Keywords: salt stressDunaliella salinatranscriptomeunigenesalt-tolerance
杜氏盐藻(Dunaliella salina)是无细胞壁的单细胞绿藻,归属于绿藻门,绿藻纲,团藻目,杜氏藻科。该藻是迄今为止发现的极度耐盐的真核生物[1]。当盐浓度发生剧烈变化时,这种微藻能够迅速调节渗透压,甚至能耐受7倍的盐浓度变化[2],但其耐盐机制尚不明确。植物对盐胁迫的反应机制是一个复杂的过程,涉及多个信号和代谢途径,参与的基因数量也非常多。研究人员期望通过生理学、形态学以及生态学等手段以阐释植物耐盐机理[3]。基于Illumina平台的转录组研究能够获得特定物种,特定状态下的全部转录本,揭示特定生物学过程及疾病发生过程的分子机理[4],并能进行差异基因表达分析,从整体水平阐释基因结构和基因功能[5-6]。转录组测序技术已广泛应用于动植物细胞[7-9]和人[10]的RNA测序中。在对于抗逆基因的挖掘上,Xu等[11]利用Illumina测序平台对在200 mmol/L NaCl胁迫下的旱地棉进行了cDNA文库测序分析,挖掘出在盐胁迫处理后基因的差异表达;Wang等[12]利用转录组测序技术鉴定出野茶树抗寒过程中的差异表达基因。利用转录组测序已经在山葡萄[13]、番茄[14-15]、卷丹百合[15]、红三叶[17]等植物中挖掘出大量的胁迫导致的差异表达基因,并进行了分析。刘军力等通过杜氏盐藻转录组测序分析了脂质及淀粉代谢路径中的关键酶的作用[18]。朱立强等已利用测序平台对杜氏盐藻的不同信号路径进行了注释[19]。本研究利用Illumina技术平台,研究盐胁迫下杜氏盐藻转录组在各个信号途径上的不同表达,通过杜氏盐藻转录组的功能注释及聚类分析,得到盐胁迫下杜氏盐藻的差异表达基因,同时利用以上数据结果,重点分析和挖掘盐胁迫对于杜氏盐藻细胞中参与光合作用基因的影响,为下一步实验工作提供理论基础和实验依据。
1 材料和方法 1.1 杜氏盐藻培养 杜氏盐藻由天津科技大学海洋与环境学院徐仰仓教授惠赠。本次研究目的是分析杜氏盐藻基因受高盐和低盐胁迫影响下的表达差异,选取9%和24%两个盐浓度,使用500 mL锥形瓶放置于光照培养箱中培养,培养温度为28 ℃,光照强度为3000 lux,光暗比为12 h/12 h,用人工海水混合f/2培养基[20]静置培养。
1.2 显微镜观察 在不同盐浓度处理的盐藻,当其生长至对数期时,高倍显微镜下(奥林巴斯IX83,日本)观察不同盐浓度下的杜氏盐藻生理形态并记录其特征。
1.3 无参转录组的构建和生物信息分析
1.3.1 RNA提取: 通过TRIzol试剂盒(北京,天根生化科技)提取组织总RNA,随后进行RNA质量检测。
1.3.2 样品检测: 在进行文库构建之前,对RNA样品进行纯度和长度严格检测,以确保符合文库构建的标准。
1.3.3 文库构建: 杜氏盐藻的富集是使用带有Oligo (dT)磁珠;富集完成后,加入分裂缓冲液裂解mRNA为小片段;以mRNA为模板反转录出cDNA,并对cDNA进行纯化、修饰和完善;通过PCR构建cDNA文库。
1.3.4 文库检测: 利用Agilent 2100检测片段大小;如果片段大小符合预期,则利用Q-PCR仪精准定量文库的有效浓度。
1.3.5 上机测序及数据质控: 上机测序技数据控制参考测序公司(奥维森)标准统一执行。质控指标见表 1。
表 1. 质控指标 Table 1. Quality indicators
| Quality value of Phred | Error ratio of base pair recognition | Correct ratio of base pair recognition/% | Value of quality |
| 10 | 1/10 | 90 | Q10 |
| 20 | 1/100 | 99 | Q20 |
| 30 | 1/1000 | 99.9 | Q30 |
| 40 | 1/10000 | 99.99 | Q40 |
表选项
1.3.6 碱基含量分布、数据过滤、转录本拼接: 碱基含量分布、数据过滤、转录本拼接原理均由测序公司(奥维森)统一处理。
1.4 RT-qPCR 基因差异表达采用荧光定量PCR技术。荧光染料采用SYBR Green,方法流程见[20]。简言之,荧光定量PCR采用的仪器为ABI 7500 Fast real-time PCR system (Applied Biosystems)。反应体系包含:1× Super Real PreMix (Tiangen),100 ng cDNA模板,0.3 mol/L引物和1× ROX荧光染料。内参为Actin。反应程序为:95 ℃ 5 min,35个循环(95 ℃ 20 s,58 ℃ 35 s,72 ℃ 40 s,65 ℃ 20 s),最后溶解曲线温度设定保证能扩增出单一的PCR产物。PCR引物见表 2。
表 2. 荧光定量PCR所需引物 Table 2. Primers required for real-time PCR
| ID | Annotation | Sequence (5'→3') | Product length/bp | |
| AF163669.2 | actin | Forward primer | ATGTGGGTTCTGTCTCTGCG | 243 |
| Reverse primer | TGTCATCCCAGTTGGACACG | |||
| EU352601.1 | ChmdH | Forward primer | CATCCGCTCCCTCATCAC | 218 |
| Reverse primer | CACGCTCCTGAATCAACTCC | |||
| AY820754.1 | psaB | Forward primer | TAGCCGTGTTCTGGGTGT | 211 |
| Reverse primer | CAAGCCCAAACTGAAAGA | |||
| EF030488.2 | LhcII-3 | Forward primer | AAGCCCACCCTGAAGACC | 195 |
| Reverse primer | GGTATCCCAGCCGTAGTCG | |||
| DQ074450.1 | psbD | Forward primer | CTACCCTTTAGGACAATC | 142 |
| Reverse primer | ACCTAATACACCAGCAAC | |||
| M23531.1 | MchB | Forward primer | GGATGGCATCAAGTTCGGTG | 166 |
| Reverse primer | GTAACCCTCAACCAGACCCA | |||
| DQ414847.1 | Lhch a\b | Forward primer | CGCTGCCGTGGAGTTCTA | 280 |
| Reverse primer | AGCACCAGCCTTGAACCA | |||
表选项
2 结果和分析 2.1 生长曲线 为了明确不同盐浓度对杜氏盐藻生长速率的影响,分别设置了不同盐浓度的培养基,并对其进行2周的观察,观察发现杜氏盐藻在9%盐浓度下生长状态最佳,而高于此浓度时杜氏盐藻生长变慢,且发现24%是高盐的一个胁迫最佳点,盐度若高于24%,盐藻细胞虽可保持生长但速度极慢,状态几近崩溃。因此,实验过程中选取了9%和24%两个盐浓度梯度,从而比较高盐胁迫对杜氏盐藻的影响。通过血球计数板计数,对9%和24%两个盐浓度下的盐藻进行了为期两周左右的生长曲线绘制,见图 1。杜氏盐藻在24%浓度条件下细胞数量明显低于9%盐浓度下的细胞数量,且可以看出在第7天和15天左右各有一个转折点,接种后一周内杜氏盐藻细胞生长较为缓慢,一周之后基数变大数量增长变快,两周左右数量趋于稳定,基本符合细胞的“S”型生长曲线。
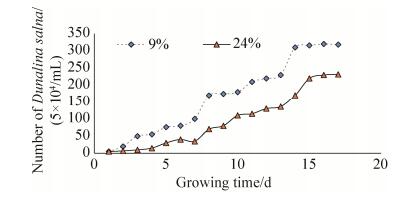 |
| 图 1 杜氏盐藻生长曲线 Figure 1 Growth curve of Dunaliella salina. |
| 图选项 |
2.2 形态结构 显微镜下观察不同盐浓度培养下的杜氏盐藻细胞形态。由图 2-A、B可知,在9%的低盐浓度下,杜氏盐藻细胞为梨形,细胞体积较大,细胞质充盈,颜色明亮,细胞壁位置明显,有两条鞭毛,十分活跃,生长状态较好;在24%的高盐浓度下(图 2-C、D),杜氏盐藻细胞为球形,体积较小,失去鞭毛,细胞停止运动,细胞内胞质变得暗淡。
 |
| 图 2 杜氏盐藻在不同盐浓度条件下的细胞形态变化 Figure 2 Cell morphology variation under different salt concentrations. A, C: Dunaliella salina cells after 48 h of low salt stress(×40 times mirror observation). The length of the legend in picture A, C is 20 μm. B, D: Dunaliella salina cells after 48 h of high salt stress(Olympus inverted microscope). The length of the legend inpicture B, D is 20 μm). |
| 图选项 |
2.3 功能注释 将9%盐浓度和24%盐浓度培养下的杜氏盐藻分别进行转录组测序,将所获得的数据经软件进行处理后获得40682个unigene,将获得的unigene与数据库对比,从而对unigene进行功能注释,其中注释到NR数据库的10905个,注释到NT数据库的2768个,注释到SWISS-PROT数据库的7261个,注释到COG/KOG数据库的6499个。
2.4 物种分类 将获得的转录组数据进行同源性比对,结果如图 3所示。注释的unigenes与盘藻、团藻、莱茵衣藻、单针藻的同源性比较高,杜氏盐藻基因与盘藻(Gonium pectorale)同源性最高为22.77%,与强壮团藻(Volvox carteri f. nagariensis)的同源性次之为21.15%。这些unigenes还比对到了盐藻的其他近源物种如单针藻(Monoraphidium neglectum)、胶球藻属(Coccomyxa subellipsoidea C-169)上。
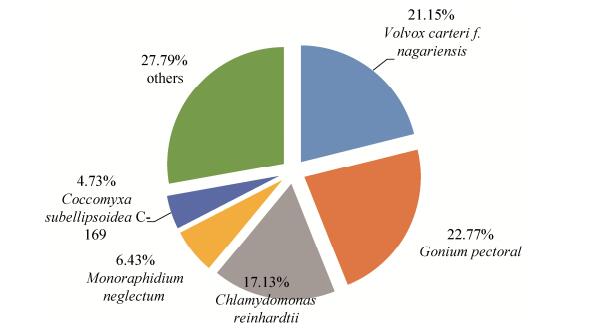 |
| 图 3 物种同源性比对 Figure 3 Species classification. |
| 图选项 |
2.5 GO功能分类 通过与GO数据库的比对和搜索,对杜氏盐藻参与生物学过程(biological process)和分子功能(molecular function)的差异表达基因进行了注释(图 4-A)。
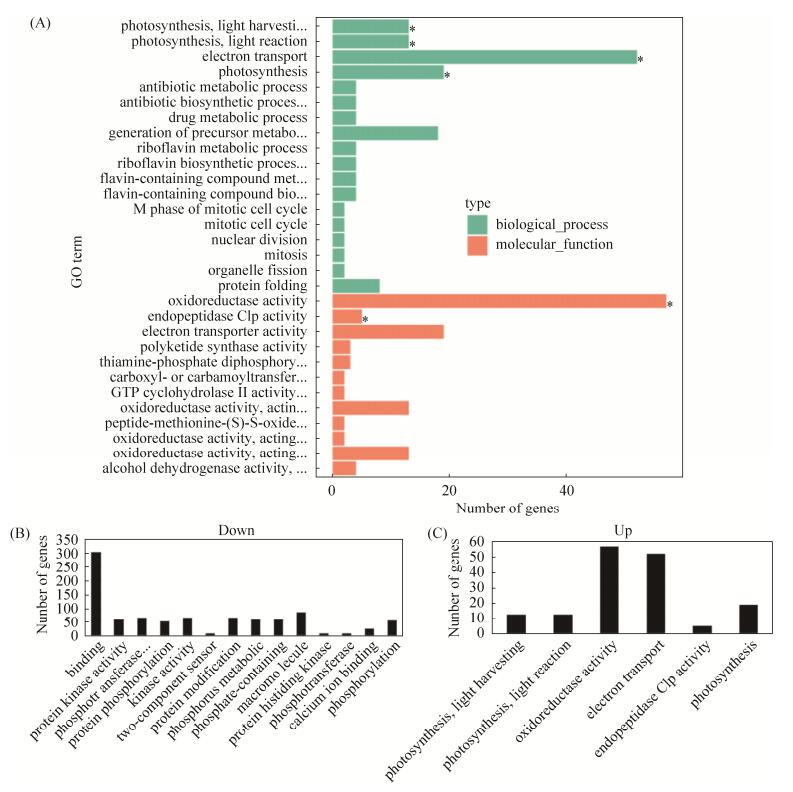 |
| 图 4 差异基因表达 Figure 4 Differential genes expression (DEGs). A: all DEGs; B: up-regulation expression; C: down-regulation expression. The ordinate is the GO term of the enrichment, and the x-coordinate is the number of genes expressed in the term. Different colors are used to distinguish different functions, and the "*" is a significantly enriched GO term. |
| 图选项 |
结果显示,低盐浓度和高盐浓度下,杜氏盐藻的差异表达基因主要富集在生物学过程(biological process)光合作用的光捕获(photosynthesis,light harvesting)以及光反应(photosynthesis,light reaction)两个方面。24%盐浓度培养相对于9%盐浓度培养的杜氏盐藻的显著下调基因富集在生物学过程的蛋白质磷酸化(protein phosphorylation)、蛋白质改性过程(protein modification process)、磷代谢过程(phosphorus metabolic process)、含磷酸盐化合物(phosphate-containing compound)、高分子改性(macromolecule modification)以及磷酸化(phosphorylation);分子功能方面的差异主要表现在结合(binding)、蛋白激酶活性(protein kinase activity)、磷酸转移酶活性-醇组为受体(phosphotransferase activity,alcohol group as acceptor)、激酶活性(kinase activity)、双组分传感器活动(two-component sensor activity)、组氨酸蛋白激酶活性(protein histidine kinase activity)、磷酸转移酶活性-含氮基团为受体(phosphotransferase activity,nitrogenous group as acceptor)、钙离子结合(calcium ion binding)上(图 4-C)。另外,24%盐浓度培养的盐藻相对于9%盐浓度培养的盐藻在涉及生物学过程(biological process)、分子功能(molecular function)方面的基因出现表达上调现象(图 4-C)。显著上调基因富集在生物学过程的光合作用的光收获(photosynthesis, light harvesting)和光反应(photosynthesis, light reaction)、电子传递(electron transport)和分子功能的氧化还原酶活性(oxidoreductase activity)、肽链内切酶中电活动(endopeptidase Clp activity)方面。
2.6 不同盐浓度下杜氏盐藻基因表达统计和对比 我们对高盐和低盐条件下,杜氏盐藻的差异表达基因进行了深入的挖掘。发现24%盐浓度高渗环境对比9%盐浓度低渗环境条件下,显著上调的基因有717个,显著下调的有1012个,另有一部分基因则差异不显著。由此我们可以得到结论,高盐胁迫既能诱导基因的表达也能特异性地抑制基因的表达,但是受到抑制的基因数量要明显高于受到诱导表达的基因数量。
2.7 Unigene在KOG数据库中的分类 通过与KOG数据库进行比对和搜索,对Unigene进行基因功能分类。将基因功能、数量以及所占百分比进行了详细说明(表 3)。可以看出,占比重最大的前6类基因功能分别为:功能预测(General function prediction only),转录后修饰(Posttranslational modification),蛋白伴侣(protein turnover,chaperones),信号转导机制(Signal transduction mechanisms),转录、核糖体结构和生物转化(Translation,ribosomal structure and biogenesis),RNA的加工和修饰(RNA processing and Modification)。
表 3. KOG基因库注释的基因功能分类 Table 3. Gene function classification of the KOG GenBank note
| Classification | Function | Number of unigene | Percent/% |
| A | RNA processing and Modification | 395 | 5.44 |
| B | Chromatin structure and dynamics | 178 | 2.45 |
| C | Energy production and conversion | 292 | 4.02 |
| D | Cell cycle control, cell division, chromosome partitioning | 197 | 2.71 |
| E | Amino acid transport and metabolism | 259 | 3.57 |
| F | Nucleotide transport and metabolism | 109 | 1.50 |
| G | Carbohydrate transport and metabolism | 258 | 3.55 |
| H | Coenzyme transport and metabolism | 123 | 1.69 |
| I | Lipid transport and metabolism | 255 | 3.51 |
| J | Translation, ribosomal structure and biogenesis | 425 | 5.85 |
| K | Transcription | 342 | 4.71 |
| L | Replication, recombination and repair | 329 | 4.53 |
| M | Cell wall/membrane/envelope biogenesis | 104 | 1.43 |
| N | Cell motility | 10 | 0.14 |
| O | Posttranslational modification, protein turnover, chaperones | 788 | 10.85 |
| P | Inorganic ion transport and metabolism | 191 | 2.63 |
| Q | Secondary metabolites biosynthesis, transport and catabolism | 133 | 1.83 |
| R | General function prediction only | 1101 | 15.16 |
| S | Function unknown | 476 | 6.55 |
| T | Signal transduction mechanisms | 617 | 8.49 |
| U | Intracellular trafficking, secretion, and vesicular transport | 362 | 4.98 |
| V | Defense mechanisms | 44 | 0.61 |
| W | Extracellular structures | 23 | 0.32 |
| X | Unamed protein | 0 | 0.00 |
| Y | Nuclear structure | 41 | 0.56 |
| Z | Cytoskeleton | 212 | 2.92 |
| Number of gene | 7264 | 100.00 |
表选项
2.8 差异基因聚类图 FPKM密度分布图:横坐标为基因的log10 (FPKM+1)值,纵坐标为对应log10 (FPKM+1)的相对密度。红色部分代表 9%盐浓度下杜氏盐藻基因表达水平,蓝色代表 24%盐浓度下基因表达水平,见图 5-A。由图可知,低盐条件下杜氏盐藻差异基因的密度分布显著大于高盐条件。
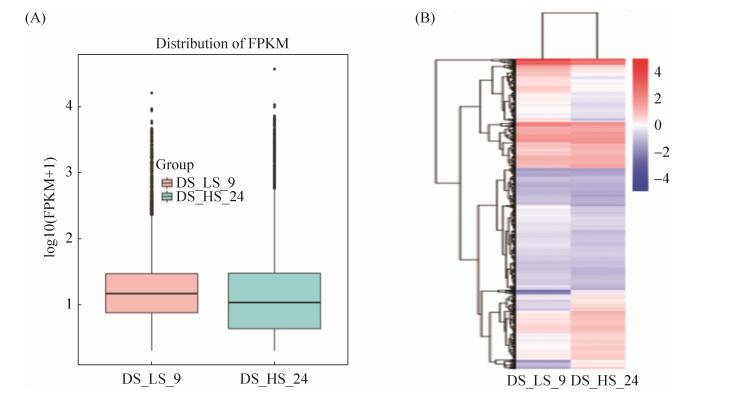 |
| 图 5 差异基因表达模式分析 Figure 5 Differential gene expression pattern analysis. A: FPKM density profile; B: Cluster analysis of differential expressed genes. FPKM is the number of fragments per kilobase length of a certain gene per million fragments. It also takes into account the effect of sequencing depth and gene length on the number of fragments. It is currently the most commonly used gene expression level estimation method. |
| 图选项 |
该分析可判断杜氏盐藻基因在不同生长环境下的表达情况,并将基因进行聚集和分类,以识别已知或未知基因的功能。每一行表示一个基因,每一列表示一个样本。颜色从蓝到红,表示log10 (FPKM+1)从小到大,见图 5-B。通过基因ID在数据库中查找其功能,一部分基因并未找到与之匹配的序列的蛋白质功能,分析可能是未注释过的新基因。在可查到基因中,发现与蛋白质结合、氧化还原酶活性、细胞凋亡过程、应激反应、电子传递、离子结合等有关的基因表达显著上调,参与核酸结合、磷代谢及产能过程等的基因表达显著下调。
2.9 基因表达韦恩(Venn)图 当比较组合数为1个时,绘制2个group共同表达基因的venn图,以fpkm > 1作为判断基因表达的标准。该图展示了各group表达的基因个数,以及其重叠关系。其中圆圈交叉部分为表达出的共有基因,见图 6。由图可知,有7917个基因在9%低渗下特异性表达,3056个基因在24%高渗下特异性表达,有20132个基因在9%和24%盐浓度下同时表达。
 |
| 图 6 基因共表达维恩图 Figure 6 Gene co-expression Venn diagram. |
| 图选项 |
2.10 荧光定量PCR检测光合作用路径基因的表达 为了进一步验证转录组测序结果,对参与光合作用途径的6个基因Lhch a/b、MchB、ChmdH、psaB、LhcII-3、psbD (表 5)进行了荧光定量PCR检测。实验结果显示,当细胞处在高盐胁迫环境下(24%),随着盐害胁迫时间的不断增加(48-120 h),Lhch a/b、MchB、psaB、LhcII-3、psbD 基因的表达量逐渐升高;而只有chloroplast malate dehydrogenase (ChmdH)基因的表达量没有发生显著变化(图 7)。推测ChmdH基因表现为组成型表达,而Lhch a/b、MchB、psaB、LhcII-3、psbD基因受到盐害胁迫诱导表达。
表 5. 参与光合作用基因的功能及相关信息 Table 5. Function and related information of the genes involved in photosynthesis
| Gene name | Accession No. | Function description |
| Lhch a/b | DQ414847.1 | Light-harvesting chlorophyll a/b protein |
| MchB | M23531.1 | Major chlorophyll binding protein |
| ChmdH | EU352601.1 | Chloroplast malate dehydrogenase, nuclear gene for chloroplast product |
| psaB | AY820754.1 | A2photosystem I P700 chlorophyll A apoprotein A2 |
| LhcII-3 | EF030488.2 | Major light-harvesting chlorophyll a/b protein 3 |
| psbD | DQ074450.1 | Dunaliella salina photosystem II D2 protein |
表选项
 |
| 图 7 光合作用途径基因表达 Figure 7 Expression pattern of genes involved in photosynthesis process under hyper-and hyposaline conditions. A: Relative expression of Lhcha/b gene quantification by qRT-PCR; B: Relative expression of MchB gene quantification by qRT-PCR; C: Relative expression of ChmdH gene quantification by qRT-PCR; D: Relative expression of psaB gene quantification by qRT-PCR; E: Relative expression of LhcII-3 gene quantification by qRT-PCR; F: Relative expression of psbD gene quantification by qRT-PCR. The asterisk in the columns indicates significant difference according to Duncan's Multiple Range Test (P < 0.01). |
| 图选项 |
3 讨论 植物耐盐机制一直是生物学研究的热点,但不同生物的巨大差异使得对其机制的阐明一直不够深入,成为生命科学研究的难点问题。杜氏盐藻是目前已知的最耐盐的真核生物,其耐盐机制备受人们关注。我们通过实验发现,杜氏盐藻为了适应高盐胁迫,从形态、生长、生理上均表现出高度的适应和变化。例如,失去鞭毛,细胞密度下降和体积变小。这是一种内在调节作用的结果。有研究显示,杜氏盐藻能够适应高渗环境,其甘油含量随盐浓度升高而增加[21];在基因层面,韩冬梅等分析了杜氏盐藻的磷脂酶C基因在盐胁迫下的诱导表达[22];Fisher等[23-24]从杜氏盐藻中发现了碳酸酐酶基因与转铁样蛋白两个耐盐基因。但关于盐胁迫对于杜氏盐藻基因在不同信号路径上的影响以及其耐盐机制仍不明确。本研究从植物转录组[25]水平着手,以期获得转录组与生物胁迫之间的关系。
本研究针对杜氏盐藻代谢过程的未知基因,并将重点放在光合作用路径,旨在深入挖掘其功能及盐胁迫对参与该信号路径基因的影响。该研究利用Illumina平台,通过对杜氏盐藻全部的转录组进行测序、功能注释和聚类分析,最终得到40682个unigenes,其中有1729个unigenes表现出差异表达:717个基因表现为表达上调,1012个基因表现为表达下调。通过和各公共数据库比对,并用Blast软件进行基因的分析,总结归纳了盐胁迫对于杜氏盐藻相关基因的表达的影响以及基因在高盐和低盐条件的表达差异。经过数据分析,我们发现,盐胁迫下基因表达差异主要体现在光合作用的光捕获和光反应路径以及产能代谢过程。利用荧光定量PCR技术,对光合作用途径的基因进行了表达水平的鉴定,实验结果显示盐胁迫诱导Lhch a/b、MchB、psaB、LhcII-3、psbD基因的表达,说明该类基因参与盐害胁迫的调控。光合作用增强有助于提高盐藻的耐盐性。在盐害胁迫下,盐藻这种不断提高对CO2的吸收和能量生产的特点与高等植物截然不同,高等植物在受到高盐胁迫的时候,表现出光合作用的抑制和光呼吸作用的增强[26]。
在对差异基因分析发现,93.5%的差异基因已经进行了功能预测和鉴定,然而仍然有6.5%的差异基因功能未知,这也意味着这些未知基因在盐藻耐盐方面发挥重要的作用,更有待于深入挖掘。基于目前结果,在下一步实验中,本研究将会针对杜氏盐藻光合路径进行深入剖析,进一步挖掘盐胁迫下与光合作用有关的杜氏盐藻基因,进行基因表达检测,研究并确定具体的基因表达性状及盐胁迫对其表达的影响。该研究为探索新基因、挖掘未知基因功能、揭示盐藻耐盐机制研究提供了理论基础实验依据。
References
| [1] | Geng DG, Han Y, Wang YQ, Li WB, Sun YR. Progress in studies on salt-resistant mechanism and prospects of gene engineering of Dunaliella salina. Chinese Bulletin of Botany, 2002, 19(3): 290-295. (in Chinese) 耿德贵, 韩燕, 王义琴, 李文彬, 孙勇如. 杜氏盐藻的耐盐机制研究进展和基因工程研究的展望. 植物学通报, 2002, 19(3): 290-295. DOI:10.3969/j.issn.1674-3466.2002.03.005 |
| [2] | Sadka A, Himmelhoch S, Zamir A. A 150 kilodalton cell surface protein is induced by salt in the halotolerant green alga Dunaliella salina. Plant Physiology, 1991, 95(3): 822-831. DOI:10.1104/pp.95.3.822 |
| [3] | Parida AK, Das AB. Salt tolerance and salinity effects on plants: a review. Ecotoxicology and Environmental Safety, 2005, 60(3): 324-349. DOI:10.1016/j.ecoenv.2004.06.010 |
| [4] | Liu HL, Zheng LM, Liu QQ, Quan FS, Zhang Y. Studies on the transcriptomes of non-model organisms. Hereditas, 2013, 35(8): 955-970. (in Chinese) 刘红亮, 郑丽明, 刘青青, 权富生, 张涌. 非模式生物转录组研究. 遗传, 2013, 35(8): 955-970. |
| [5] | Wu Q, Zhang L, Huang ZP, Wang DG, Hu GY. Transcription sequencing and its application on discovering the gene resources of wild soybean. Soybean Science, 2013, 32(6): 845-851. (in Chinese) 吴倩, 张磊, 黄志平, 王大刚, 胡国玉. 转录组测序及其在野生大豆基因资源发掘中的应用. 大豆科学, 2013, 32(6): 845-851. |
| [6] | Shu SH, Chen B, Zhou MC, Zhao XM, Xia HY, Wang M. De novo sequencing and transcriptome analysis of Wolfiporia cocos to reveal genes related to biosynthesis of triterpenoids. PLoS One, 2013, 8(8): e71350. DOI:10.1371/journal.pone.0071350 |
| [7] | Nagalakshmi U, Wang Z, Waern K, Shou C, Raha D, Gerstein M, Snyder M. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science, 2008, 320(5881): 1344-1349. DOI:10.1126/science.1158441 |
| [8] | Lister R, O'Malley RC, Tonti-Filippini J, Gregory BD, Berry CC, Millar AH, Ecker JR. Highly integrated single-base resolution maps of the epigenome in Arabidopsis. Cell, 2008, 133(3): 523-536. DOI:10.1016/j.cell.2008.03.029 |
| [9] | Mortazavi A, Williams BA, Mccue K, Schaeffer L, Wold B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods, 2008, 5(7): 621-628. DOI:10.1038/nmeth.1226 |
| [10] | Morin RD, Bainbridge M, Fejes A, Hirst M, Krzywinski M, Pugh TJ, McDonald H, Varhol R, Jones SJM, Marra MA. Profiling the HeLa S3 transcriptome using randomly primed cDNA and massively parallel short-read sequencing. Biotechniques, 2008, 45(1): 81-94. DOI:10.2144/000112900 |
| [11] | Xu P, Liu ZW, Fan XQ, Gao J, Zhang X, Zhang XG, Shen XL. De novo transcriptome sequencing and comparative analysis of differentially expressed genes in Gossypium aridum under salt stress. Gene, 2013, 525(1): 26-34. DOI:10.1016/j.gene.2013.04.066 |
| [12] | Wang XC, Zhao QY, Ma CL, Zhang ZH, Gao HL, Kong YM, Yue C, Hao XY, Chen L, Ma JQ, Li X, Yang YJ. Global transcriptome profiles of Camellia sinensis during cold acclimation. BMC Genomics, 2013, 14: 415. DOI:10.1186/1471-2164-14-415 |
| [13] | Xu WR, Li RM, Zhang NB, Ma FL, Jiao YT, Wang ZP. Transcriptome profiling of Vitis amurensis, an extremely cold-tolerant Chinese wild Vitis species, reveals candidate genes and events that potentially connected to cold stress. Plant Molecular Biology, 2014, 86(4/5): 527-541. |
| [14] | Liu H, Ouyang B, Zhang JH, Wang TT, Li HX, Zhang YY, Yu CY, Ye ZB. Differential modulation of photosynthesis, signaling, and transcriptional regulation between tolerant and sensitive tomato genotypes under cold stress. PLoS One, 2012, 7(11): e50785. DOI:10.1371/journal.pone.0050785 |
| [15] | Chen HY, Chen XL, Chen D, Li JF, Zhang Y, Wang A. A comparison of the low temperature transcriptomes of two tomato genotypes that differ in freezing tolerance: Solanum lycopersicum and Solanum habrochaites. BMC Plant Biology, 2015, 15: 132. DOI:10.1186/s12870-015-0521-6 |
| [16] | Wang JM, Yang Y, Liu XH, Li J, Zhang Q, Gu JH, Lu YM. Transcriptome profiling of the cold response and signaling pathways in Lilium lancifolium. BMC Genomics, 2014, 15: 203. DOI:10.1186/1471-2164-15-203 |
| [17] | Yate SA, Swain MT, Hegarty MJ, Chernukin I, Lowe M, Allison GG, Ruttink T, Abberton MT, Jenkins G, Sk?t L. De novo assembly of red clover transcriptome based on RNA-Seq data provides insight into drought response, gene discovery and marker identification. BMC Genomics, 2014, 15: 453. DOI:10.1186/1471-2164-15-453 |
| [18] | Liu JL, Hong L. Energy metabolic pathways in Dunaleilla salina using the transcriptome data. Geomatics and Information Science of Wuhan University, 2018, 64(1): 84-94. (in Chinese) 刘军立, 红凌. 基于转录组数据分析杜氏盐藻能量代谢途径. 武汉大学学报, 2018, 64(1): 84-94. |
| [19] | Zhu LQ, Li QH, Zhang YT, Zhu XZ, Guan FX, Xue LX. Transcriptome sequencing and analysis of Dunaliella salina based on RNA-seq. Journal of Zhengzhou University (Medical Sciences), 2016, 51(3): 289-293. (in Chinese) 朱立强, 李庆华, 张彦婷, 朱相展, 关方霞, 薛乐勋. 基于RNA-seq的杜氏盐藻全转录组测序与分析. 郑州大学学报(医学版), 2016, 51(3): 289-293. |
| [20] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2T-ΔΔC method. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [21] | Zhou L, Meng XH, Liu CS, Yu LJ, Chen XG. Effects of osmotic stress on intracellular glycerol content and enzyme activity in Dunaliella salina. Chinese Bulletin of Botany, 2006, 23(2): 145-151. (in Chinese) 周丽, 孟祥红, 刘成圣, 于乐军, 陈西广. 渗透胁迫对杜氏盐藻胞内甘油含量及相关酶活性影响. 植物学通报, 2006, 23(2): 145-151. DOI:10.3969/j.issn.1674-3466.2006.02.003 |
| [22] | Han DM, Chai XJ, Wang YY, Liu SC, Yue WJ. Cloning and expression analysis of DsPLC under salt stress from Dunaliella salina. Journal of Nuclear Agricultural Sciences, 2014, 28(10): 1773-1780. (in Chinese) 韩冬梅, 柴晓杰, 王逸云, 刘世才, 岳文静. 杜氏盐藻磷脂酶C基因DsPLC的克隆及盐胁迫下的表达分析. 核农学报, 2014, 28(10): 1773-1780. DOI:10.11869/j.issn.100-8551.2014.10.1773 |
| [23] | Fisher M, Pick U, Zamir A. A salt-induced 60-kilodalton plasma membrane protein plays a potential role in the extreme halotolerance of the alga Dunaliella. Plant Physiology, 1994, 106(4): 1359-1365. DOI:10.1104/pp.106.4.1359 |
| [24] | Fisher M, Gokhman I, Pick U, Zamir A. A Salt-resistant plasma membrane carbonic anhydrase is induced by salt in Dunaliella salina. Journal of Biological Chemistry, 1996, 271(30): 17718-17723. DOI:10.1074/jbc.271.30.17718 |
| [25] | Zhang C, Tang CC, Wang JY, Guo LM, Wang LL, Li WK. Research progress of transcriptomics in plant response to stress. Journal of Biology, 2017, 34(2): 86-90. (in Chinese) 张纯, 唐承晨, 王吉永, 郭龙妹, 王莉莉, 黎万奎. 转录组学在植物应答逆境胁迫中的研究进展. 生物学杂志, 2017, 34(2): 86-90. DOI:10.3969/j.issn.2095-1736.2017.02.086 |
| [26] | Osmond CB, Foyer CH, Bock G, Wingler A, Lea PJ, Quick WP, Leegood RC. Photorespiration: metabolic pathways and their role in stress protection. Philosophical Transactions of the Royal of London Society. Series B: Biological Sciences, 2000, 355(1402): 1517-1529. DOI:10.1098/rstb.2000.0712 |
