习慧君, 臧睿, 刘闯, 赵莹

 , 文才艺
, 文才艺

河南农业大学植物保护学院, 河南 郑州 450002
收稿日期:2018-05-08;修回日期:2018-09-01;网络出版日期:2018-11-20
基金项目:河南省自然科学基金(162300410136)
*通信作者:赵莹, E-mail:nying2009@126.com;
文才艺, E-mail:wencaiyi1965@163.com.
摘要:[目的] 探究河南黄河湿地放线菌多样性,筛选对植物病原菌有拮抗活性的放线菌菌株。[方法] 基于Illumina HiSeq技术的高通量测序分析了河南黄河湿地放线菌物种多样性及其分布特点;利用8种分离培养基对采集自三门峡黄河湿地、郑州黄河湿地和开封黄河湿地21份土壤样品中的放线菌进行了分离纯化,通过16S rRNA基因序列分析对分离株进行了初步的分类鉴定和系统学研究;以7种植物病原菌为靶标筛选有拮抗活性的分离株,并对活性菌株进行了聚酮合酶、非核糖体多肽合成酶和安莎类化合物等基因筛查。[结果] 高通量测序结果表明,河南黄河湿地放线菌物种多样性丰富,且不同湿地区域以及同一湿地不同生境间放线菌多样性存在显著差异,放线菌优势属物种依次为Nocardioides、Streptomyces、CL500-29 marine group、Fodinicola、Mycobacterium和Micromonospora,此外,还具有大量的未知类群;通过分离培养共获得了261株纯培养菌株,分布于放线菌门中的7个目9个科9个属,包含97个可能的已知物种和8个潜在新物种,对植物病原菌有拮抗活性的放线菌86株,其中74株中至少含有一种活性代谢物质合成相关基因。[结论] 河南黄河湿地放线菌物种多样性丰富,具有发掘新物种和新型植物病害生防资源的潜力。
关键词:黄河湿地放线菌多样性生物活性
Diversity of actinobacteria in Henan Yellow River wetland and screening of cultivable actinobacteria with biological control on plant disease
Huijun Xi, Rui Zang, Chuang Liu, Ying Zhao

 , Caiyi Wen
, Caiyi Wen

College of Plant Protection, Henan Agricultural University, Zhengzhou 450002, Henan Province, China
*Corresponding author: Ying Zhao, E-mail:nying2009@126.com;
Caiyi Wen, E-mail:wencaiyi1965@163.com.
Foundation item: Supported by the Henan Natural Science Foundation (162300410136)
Abstract: [Objective] This study is aimed to analyze the diversity of actinobacteria in soils of the Henan Yellow River wetland and screen actinobacteria strains which have antagonistic activity against plant pathogens. [Methods] High-throughput sequencing based on Illumina HiSeq technology was used to analyze the actinobacterial diversity and distribution in the Henan Yellow River wetland. We used eight selective media to isolate actinobacteria from 21 soil samples collected from Sanmenxia, Zhengzhou and Kaifeng Yellow River wetland. The cultivable actinobacteria were identified and phylogeny analyzed based on the 16S rRNA gene sequences. The active isolates were screened using seven plant pathogens as targets, and 5 functional genes were assayed, including polyketide synthases gene (PKS-Ⅰ, PKS-Ⅱ), nonribosomal peptide synthetases gene (NRPS), 3-amino-5-hydroxybenzoic acid synthases gene (AHBA) and 3-hydroxy-3-methylglutaryl coenzyme A gene (HMG CoA). [Results] The high-throughput sequencing results indicated that the actinobacteria were abundant in Henan Yellow River wetland and there were significant differences among different wetland areas and different habitats in the same wetland. Nocardioides was the most frequently isolated genus, followed by the genera Streptomyces, CL500-29 marine group, Fodinicola, Mycobacterium and Micromonospora. In addition, there are still a lot of unknown groups. We isolated a total of 261 dereplicated strains, belonging to 7 orders, 9 families and 9 genera of the phylum Actinobacteria. They were including 97 putative species and eight unknown species. Among them, 86 actinobacteria strains have antagonistic activity against plant pathogens; 74 strains contain at least one gene which involved active metabolites synthesis. [Conclusion] Actinobacteria species diversity was abundant in Henan Yellow River wetland and new species have potential as biocontrol agents against plant diseases.
Keywords: Yellow River wetlandactinobacteriadiversitybiological activity
放线菌是一类重要的微生物资源。目前,临床和农业上应用的150多种抗生素,约2/3来自放线菌[1]。在农业领域,放线菌因产生种类丰富的抗生素和其他生物活性代谢产物而成为植物病虫害生物防治的重要生防因子,此外,放线菌除了可以产生高效的次级代谢活性化合物外,其生防机制复杂多样,显示出巨大的生物防治潜力[2-3]。我国在农用放线菌生防资源开发利用方面,自尹莘耘(1953)从陕西泾阳地区分离到5406菌株(Streptomyces microflavus)以来,先后在广东(Streptomyces griseochromogenes,1959)、江西(Actinomyces microanueus,1964;Streptomyces hygroscopicus var. jinggangensis,1973;Streptomyces nanchangensis,1981)、吉林(Streptomyces ahygroscopicus var. gongzhulingensis,1971)、福建(Streptomyces ahygroscopicus var. wuyiensis,1979)、湖南(Streptomyces griseus var. liuyangensis,1983)、四川(Streptomyces noursei var xichangensis,1995)、海南(Streptomyces lavendulae var. hainanensis,1996)、辽宁(Streptomyces achygruscopicus,2002)和云南(Nocardiopsis sp. YIM DT266,2010)等地区筛选到许多具有应用前景的放线菌菌株,并研发出多种农用抗生素和微生物接种剂(microbial inoculant)产品,如5406、阿维菌素、井冈霉素、宁南霉素、中生菌素、嘧肽霉素、多杀菌素等,在我国植物病虫害生物防治中发挥了重要作用。因此,放线菌也是重要的植物病虫害生物防治资源。
河南黄河湿地分布于黄河中、下游过渡地带,是我国湿地资源的重要组成部分。黄河自三门峡进入河南以后,河床加宽,河水游荡不定,在两岸形成了灌丛、草甸、激流、岛屿和沙洲等各种类型的湿地,以及完整的植被类型谱系,同时还形成了各具特色的湿地景观,如滩地、稻田、滩涂等,因此,河南黄河湿地不仅具有河流湿地的特征,还具有库塘湿地和沼泽湿地的特征,地质、地貌和生物多样性独特,是发掘放线菌新物种和新用途放线菌资源的理想生境。目前已在黄河上游的高原湿地、草原湿地、河套平原区湿地和下游黄河三角洲滨海湿地等生境开展了放线菌多样性、生态分布及生态学功能等方面的研究,并取得一定进展[4-5],但是,至今尚未见黄河中下游湿地生境放线菌资源方面的研究报道。我们分别从三门峡、郑州和开封黄河沿岸湿地的不同生境采集土壤样品21份,基于Illumina HiSeq高通量测序的生物信息学分析和可培养放线菌分离培养及系统学分析,初步研究河南黄河湿地放线菌多样性及其分布特点;以多种植物病原菌为靶标,筛选出有拮抗活性的放线菌菌株,并对活性菌株的活性化合物合成相关基因进行了筛查,为河南黄河湿地放线菌资源的保护和开发利用提供了参考。
1 材料和方法 1.1 材料
1.1.1 样品采集: 2017年5月,分别从三门峡、郑州和开封黄河沿岸湿地的芦苇地和滩涂等生境采集土壤样品21份,取样深度为5-20 cm。样品采集后保存于无菌采样袋中,于-20 ℃冰箱中保藏备用。采集详情及分组见表 1。
表 1. 土壤样品采集信息 Table 1. Information of sampling sites
| Sampling site | Number | Group | Longitude/E | Latitude/N | Distribution characteristics of physical and chemical factors in soil samples | ||||
| pH | TOC/(g/kg) | TP/% | TK/% | TN/% | |||||
| Sanmenxia beach | SMSDA, SMSDB, SMSDC | A | 111.15° | 34.78° | 7.24 | 6.75 | 0.029 | 1.01 | 1.10 |
| Sanmenxia reed | SMLWA, SMLWB, SMLWC | B | 111.16° | 34.78° | 7.31 | 15.20 | 0.082 | 1.21 | 1.37 |
| Zhengzhou beach | ZHSDA, ZHSDB, ZHSDC, ZHSDD, ZHSDE, ZHSDF | C | 113.39° | 34.55° | 7.17 | 2.46 | 0.009 | 0.38 | 0.31 |
| Zhengzhou reed | ZHLWA, ZHLWB, ZHLWC | D | 114.39° | 34.54° | 7.16 | 14.40 | 0.096 | 1.44 | 1.18 |
| Kaifeng beach | KFDBA, KFDBB, KFDBC, KFDBD, KFDBE, KFDBF | E | 114.46° | 34.55° | 7.31 | 6.37 | 0.041 | 0.53 | 0.92 |
表选项
1.1.2 主要试剂和仪器: 细菌基因组提取试剂盒和2×PCR mix (含Taq DNA聚合酶、dNTPs、MgCl2)购自生工生物工程(上海)股份有限公司。
1.1.3 培养基的配制: 培养基M1-M8参考文献配制[6-7]:M1 (淀粉甘油脯氨酸培养基);M2 (组氨酸棉籽糖培养基);M3 (纤维素培养基);M4 (木糖酪蛋白培养基);M5 (腐殖酸培养基);M6 (高氏一号培养基);M7 (丙酸钠酪蛋白培养基);M8 (稀释的葡萄糖酵母麦芽汁培养基)。各培养基添加15%的琼脂制成固体培养基(除M8)。培养基组成与含量见表 2。
表 2. 分离培养基及其组分 Table 2. The isolation media and their compositions
| Media | Components/(g/L) | Antibiotics/(mg/L) |
| M1 | Starch 5.0, Glycerol 5.0, Proline 1.0, (NH4)2SO4 1.0, NaCl 1.0, CaCl2 1.0, K2HPO4 1.0, MgSO4·7H2O 1.0 | Nystatin 50.0, Aztreonam 25.0 |
| M2 | Histidine 0.10, Raffinose 1.00, Na2HPO4 0.50, KCl 1.70, MgSO4·7H2O 0.05, FeSO4·7H2O 0.01, CaCO3 0.02 | Novobiocin 50.0, Rifampicin 20.0, Nalidixic acid 50.0 |
| M3 | Amorphous cellulose 5.00, KH2PO4 0.50, KNO3 0.20, KCl 0.10, MgSO4·7H2O 0.30, NaCl 0.10, FeCl3 0.01, NaNO3 2.50 | Nystatin 50.0, Nalidixic acid 50.0, Aztreonam 20.0 |
| M4 | Xylan 5.00, Casein 0.30, KNO3 0.20, KH2PO4 0.50, KCl 0.10, MgSO4·7H2O 0.30, NaCl 0.10, FeCl3 0.01, NaNO3 2.50 | Nystatin 50.0, Nalidixic acid 50.0 |
| M5 | Humic acid 1.00, Na2HPO4 0.50, KCl 1.70, MgSO4·7H2O 0.05, FeSO4·7H2O 0.01, CaCl2 1.00 | Nystatin 50.0, Aztreonam 50.0 |
| M6 | Starch 20.0, KNO3 1.0, Na2HPO4 0.5, MgSO4·7H2O 0.5, NaCl 0.5, 10% FeSO4·4H2O 2 drops | - |
| M7 | Sodium propionate 2.0, Casein hydrolysate 0.3, KNO3 0.1 | Nystatin 50.0, Nalidixic acid 50.0, Aztreonam 20.0 |
| M8 | Glucose 1.0, Yeast extract 1.0, Malt extract 2.5, CaCO3 0.5 | Cycloheximide 50.0, Novobiocin 50.0, Aztreonam 25.0 |
表选项
1.1.4 植物病原菌: 苹果轮纹病菌(Botryosphaeria dothidea)、西瓜炭疽病菌(Colleetotrichum orbiculare)、玉米大斑病菌(Exserohilum turcicum)、苦瓜枯萎病菌(Fusarium oxysporum f. sp. momodicae)、小麦茎基腐病菌(Fusarium pseudograminearum)、小麦全蚀病菌(Gaeumannnomyces graminis var. tritici)、辣椒疫霉病菌(Phytophthora capsici),均由河南农业大学植物病害生物防治研究室分离、保存。
1.2 免培养放线菌多样性分析 建库测序:提取样品DNA后,按照Zhai等(2016)的方法基于16S rRNA基因序列的设计引物进行PCR扩增,第一步扩增使用放线菌特异性引物[8] 243F (5′-GGATGAGCCCGCGGCCTA-3′)和A3R (5′-CCAGCCCCACCTTCGAC-3′);第二步扩增引物为S-D-Bact-0341-b-S-17 (5′-CCTACGGGN GGCWGCAG-3′)和S-D-Bact-0785-a-A-21 (5′-GAC TACHVGGGTATCTAATCC-3′);PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测,检测合格后对PCR产物检测定量,根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物,使用胶回收试剂盒回收目的产物。使用Illumina NEB建库试剂盒进行文库构建,建好的文库先进行文库质检,质检合格的文库用Illumina HiSeq 2500进行测序。高通量测序得到的原始图像数据文件,经碱基识别(base calling)分析转化为原始测序序列(sequenced reads)。数据预处理:(1)使用FLASH v1.2.7软件,通过overlap对每个样品的reads进行拼接,得到的拼接序列即原始Tags数据(raw tags);(2)使用Trimmomatic v0.33软件,对拼接得到的Raw Tags进行过滤,得到高质量的Tags数据(clean tags);(3)使用UCHIME v4.2软件,鉴定并去除嵌合体序列,得到最终有效数据(effective tags)。结果分析:(1) OTU分析:使用QIIME (version 1.8.0)软件中的UCLUST对Tags在97%的相似度水平下进行聚类、获得OTU,并基于Silva (细菌)分类学数据库对OTU进行分类学注释;(2) Alpha多样性分析:使用Mothur (version v.1.30)软件,对样品Alpha多样性指数进行评估。为比较样品间的多样性指数,分析时将样品所含序列数进行标准化;(3) Beta多样性组间差异分析:使用QIIME软件进行Beta多样性(beta diversity)分析,比较不同样品在物种多样性方面存在的相似程度。
1.3 可培养放线菌的分离及鉴定
1.3.1 样品预处理: 土壤样品进行菌种分离前碾碎过筛,放置无菌通风处自然风干1-2周。取2.0 g风干的粉末状样品加入含18.0 mL无菌水的锥形瓶中,180 r/min振荡1 h。
1.3.2 可培养放线菌的分离、纯化及鉴定保存: 将预处理后的土壤悬浮液进行梯度稀释,吸取10-3和10-4稀释度样品200 μL于分离培养基平板,涂布至干燥,每个稀释度设3个重复。于28 ℃生化培养箱中倒置培养1周后观察菌落生长情况,根据菌落形态、气生菌丝特点等挑选放线菌菌落,分别接种至M6培养基上纯化。根据纯化菌落的生物特征和样品来源进行去重复,去除重复后的菌株保存于25%甘油中,-80 ℃保藏。
1.3.3 菌株DNA提取、扩增及序列分析: 采用CTAB法提取菌株DNA;16S rRNA基因的PCR扩增和测序引物均为特异性引物243F和A3R[8]。25 μL PCR反应体系:2×Easy Taq Master Mix 12.5 μL (含有Taq DNA聚合酶、dNTPs、缓冲液、MgCl2、溴酚蓝),10 μmol/L引物各0.5 μL,模板DNA 1 μL,ddH2O 10.5 μL。PCR扩增程序为:94 ℃ 5 min;95 ℃ 1 min,59 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃ 10 min;PCR产物使用1%琼脂糖凝胶电泳。261株可培养放线菌菌株的16S rRNA基因测序工作由生工生物工程(上海)股份有限公司完成。将所有获得的16S rRNA基因序列在NCBI数据库中分别进行BLAST比对,确定与之亲缘关系最近的种属。从数据库获得相关种属的16S rRNA基因序列,使用MEGA 6.0软件进行比对剔除重复序列后以ML法构建系统发育树。所有分离菌株的16S rRNA基因序列均已提交GenBank数据库,并获得了相应的序列登录号。
1.4 拮抗活性菌株的筛选 采用对峙培养法检测放线菌分离物对植物病原菌的抑制作用。分别将放线菌与靶标病原菌活化之后,用直径5 mm的打孔器打取靶标菌菌饼,倒置接种于直径9 cm的PDA平板中央,距病原菌边缘3 cm处接种目标放线菌,28 ℃恒温培养箱中倒置培养,以不接种放线菌的病原菌平板培养为对照,1周后观察抑菌效果,或当空白对照的病原菌菌落直径为8 cm时,记录实验组抑菌带大小。
1.5 活性化合物合成相关基因筛查 本研究使用5套PCR引物(表 3)对5种功能基因进行扩增。20 μL PCR反应体系包括:2×Easy Taq Master Mix 10 μL,10 μmol/L引物各0.5 μL,模板DNA 1 μL,ddH2O 8 μL。PCR产物用1%琼脂糖凝胶电泳检测。
表 3. 功能基因的PCR引物 Table 3. PCR primers for amplifying the biosynthetic genes used in this study
| Target genes | Primers | Sequences (5'→3') |
| PKS Ⅰ[9] | K1F | TSAAGTCSAACATCGGBCA |
| M6R | CGCAGGTTSCSGTACCAGTA | |
| PKS Ⅱ[9] | KSα F | TSGCSTGCTTGGAYGCSATC |
| KSβ R | TGGAANCCGCCGAABCCTCT | |
| NRPS[9] | A3F | GCSTACSYSATSTACACSTCSGG |
| A7R | SASGTCVCCSGTSCGGTAS | |
| AHBA[10] | P1 | AGAGGATCCTTCGAGCRSGAGTTCGC |
| P2 | GCAGGATCCGGAMCATSGCCATGTAG | |
| HMGA[11] | HMGF | GGGCATCGCCGCGACCCTCGTCGACGAGCG |
| HMGR | GCGATGACGGCGAGGCGGCGGGCGTTCTC |
表选项
2 结果和分析 2.1 黄河湿地免培养放线菌多样性分析 测序结果表明,21个样品测序共获得1679624对Reads,双端Reads拼接、过滤后共产生1292948条Clean tags,平均每个样品产生61569条Clean tags,鉴定并去除嵌合体序列后,平均每个样品产生的最终有效序列(Effective Tags)为58544条。根据不同的相似度水平,对有效序列进行OTU划分,使用QIIME (version 1.8.0)软件中的UCLUST[12]对Tags在97%的相似度水平下进行聚类,获得OTU,并基于Silva (细菌)分类学数据库对OTU进行分类学注释。结果如图 1-a所示,开封黄河湿地和郑州黄河湿地的放线菌种群基本一致,均高于三门峡湿地的放线菌种群数,芦苇湿地生境的放线菌种群分布较滩涂生境的少;不同湿地生境的放线菌物种分布图如图 1-b所示,图中只显示丰度水平前10的物种,其他物种合并为Others,Unknown代表未得到分类学注释的物种。由此可见,河南黄河湿地无论是芦苇生境还是滩涂生境,放线菌优势物种依次为Nocardioides、Streptomyces、CL500-29 marine group、Fodinicola、Mycobacterium和Micromonospora,此外,还具有大量的未知类群,表明河南黄河湿地稀有放线菌物种丰富,且具有一定的新物种发掘潜力。
 |
| 图 1 不同样品中OTU数分布图(A)和在属水平上的分布图(B) Figure 1 OTUs (a) and genus-level distribution (b) of actinobacteria in different samples. A: Sanmenxia wetland (Beach); B: Sanmenxia wetland (Reeds); C: Zhengzhou wetland (Beach); D: Zhengzhou wetland (Reeds); E: Kaifeng wetland (Beach). |
| 图选项 |
Alpha多样性(Alpha diversity)分析结果如表 4所示。表中Chao1和Ace指数表示物种丰度即物种数量的多少,Shannon指数值越大,Simpson指数值越小,说明样品的物种多样性越高,由此可见,河南黄河湿地放线菌物种多样性丰富。Coverage表示OTU覆盖率,其数值越高,则样本中物种被测出的概率越高,而没有被测出的概率越低,从表中可知,各样品中OTU覆盖率均在99%以上,表明本研究中测序文库的构建质量高,放线菌物种多样性分析结果可靠。
表 4. Alpha多样性分析结果 Table 4. Alpha diversity index
| Sample ID | OTU | Ace | Chao1 | Simpson | Shannon | Coverage |
| KFDBA | 1054 | 1127.2448 | 1118.5076 | 0.0166 | 5.2507 | 0.9968 |
| KFDBB | 1130 | 1177.7445 | 1194.4624 | 0.0123 | 5.4854 | 0.9974 |
| KFDBC | 1138 | 1195.8428 | 1229.1765 | 0.0102 | 5.5592 | 0.9970 |
| KFDBD | 1131 | 1175.0714 | 1175.4206 | 0.0088 | 5.6045 | 0.9976 |
| KFDBE | 1123 | 1183.7162 | 1187.0496 | 0.0113 | 5.3876 | 0.9970 |
| KFDBF | 1171 | 1213.4685 | 1218.6944 | 0.0101 | 5.5493 | 0.9976 |
| SMLWA | 944 | 995.3716 | 1001.8673 | 0.0209 | 4.9584 | 0.9974 |
| SMLWB | 936 | 987.3723 | 989.5294 | 0.0172 | 5.0675 | 0.9975 |
| SMLWC | 935 | 987.4610 | 1014.2857 | 0.0194 | 4.9913 | 0.9973 |
| SMSDA | 852 | 922.9842 | 943.8987 | 0.0162 | 5.1312 | 0.9967 |
| SMSDB | 896 | 967.4408 | 987.1765 | 0.0125 | 5.2785 | 0.9964 |
| SMSDC | 954 | 994.5600 | 1007.1548 | 0.0181 | 5.0794 | 0.9977 |
| ZHLWA | 1096 | 1166.2384 | 1181.1622 | 0.0252 | 5.0825 | 0.9967 |
| ZHLWB | 844 | 929.4481 | 1001.5500 | 0.0371 | 4.6831 | 0.9966 |
| ZHLWC | 1010 | 1086.7318 | 1090.5966 | 0.0394 | 4.6658 | 0.9968 |
| ZHSDA | 1091 | 1152.8325 | 1160.0789 | 0.0317 | 4.8929 | 0.9971 |
| ZHSDB | 896 | 945.9618 | 947.5625 | 0.0441 | 4.6512 | 0.9976 |
| ZHSDC | 1111 | 1170.8314 | 1197.4433 | 0.0230 | 5.0992 | 0.9969 |
| ZHSDD | 1092 | 1146.4547 | 1141.2339 | 0.0131 | 5.3734 | 0.9974 |
| ZHSDE | 1124 | 1180.8065 | 1201.5618 | 0.0102 | 5.5532 | 0.9971 |
| ZHSDF | 1034 | 1162.2319 | 1169.0082 | 0.0150 | 5.1595 | 0.9956 |
表选项
在微生物多样性分析中,由于环境中的微生物复杂多样,不同生境间物种的组成差异更为显著,所以通常采用非加权方法进行分析。基于unweight_unifrac的非度量多维标定法(non-metric multi-dimensional scaling,NMDS)是一种适用于生态学研究的排序方法,主要是将多维空间的研究对象简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法。分析结果如图 2所示。结果表明,河南黄河湿地放线菌多样性分布具有明显的差异性,即三门峡湿地与郑州和开封两个湿地之间的放线菌多样性存在明显的差异;同时,黄河湿地中放线菌多样性存在地域连续性,即郑州湿地与开封湿地的放线菌多样性存在部分相似性,郑州湿地与三门峡湿地放线菌的差异性低于开封湿地与三门峡湿地的差异性。Beta多样性差异分析结果如图 3所示,同一地域湿地内,不同生境之间放线菌多样性存在明显的差异,即三门峡湿地中,滩涂和芦苇地的放线菌多样性存在明显的差异;此外,不同地域湿地中,同种生境中放线菌多样性也存在明显的差异,即三门峡芦苇湿地中与郑州芦苇湿地中的放线菌多样性存在显著的差异。
 |
| 图 2 基于unweight_unifrace的NMDS分析 Figure 2 NMDS analysis by unweight_unifrace. KaiF: Kaifeng wetland; SMX: Sanmenxia wetland; ZZhou: Zhengzhou wetland. |
| 图选项 |
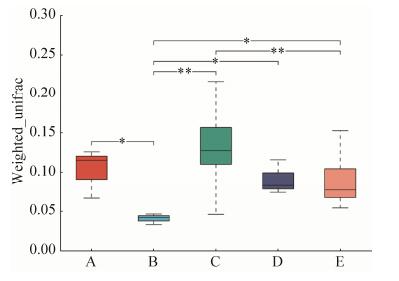 |
| 图 3 基于weight_unifrace的Beta多样性差异分析 Figure 3 Beta diversity analysis based on the weight_unifrace. A: Sanmenxia wetland (Beach); B: Sanmenxia wetland (Reeds); C: Zhengzhou wetland (Beach); D: Zhengzhou wetland (Reeds); E: Kaifeng wetland (Beach). The abscissa represents the grouping; The vertical represents the distance; The different color boxes represent the groups. * represents a significant difference; ** represents a extremely significant difference. |
| 图选项 |
2.2 黄河湿地可培养放线菌分离鉴定结果 用分离培养基M1-M8对21份土壤样品进行了放线菌分离培养,纯化去重复后获得放线菌纯培养261株(表 5),将获得的261株放线菌16S rRNA基因序列分别在NCBI数据库中进行BLAST比对分析可知,261株放线菌分布于放线菌7个目,分别是放线菌目(Actinomycetales)、假诺卡氏菌目(Pseudonocardineae)、丙酸杆菌目(Propionibacterineae)、小单孢菌目(Micromonosporales)、棒杆菌目(Corynebacteriales)、链孢囊菌目(Streptosporangiales)和微球菌目(Micrococcales);9个科,分别是链霉菌科(Sterptomycetaceae)、放线菌科(Actinomycetaceae)、假诺卡氏菌科(Pseudonocardiaceae)、束丝放线菌科(Actinosynnemataceae)、类诺卡氏菌科(Nocardioidaceae)、小单孢菌科(Micromonosporaceae)、诺卡氏菌科(Nocardiaceae)、链孢囊菌科(Streptosporangiaceae)和原小单孢菌科(Promicromonosporaceae);9个属,分别是链霉菌属(Streptomyces)、放线菌属(Actinomyces)、拟无枝酸菌属(Amycolatopsis)、列舍瓦列氏菌属(Lechevalieria)、类诺卡氏菌属(Nocardioides)、小单孢菌属(Micromonospora)、诺卡氏菌属(Nocardia)、野野村氏菌属(Nonomuraea)和原小单孢菌属(Promicromonospora),各属代表菌株的系统进化树如图 4所示。
表 5. 黄河湿地可培养放线菌分离结果 Table 5. Culturable actinobacteria isolated from the Yellow River wetland
| Order | Family | Genus | No. of isolates | No. of OTUs |
| Actinomycetales | Sterptomycetaceae | Streptomyces | 220 | 79 |
| Actinomycetaceae | Actinomyces | 2 | 2 | |
| Pseudonocardineae | Pseudonocardiaceae | Amycolatopsis | 3 | 2 |
| Actinosynnemataceae | Lechevalieria | 2 | 2 | |
| Propionibacterineae | Nocardioidaceae | Nocardioides | 11 | 3 |
| Micromonosporales | Micromonosporaceae | Micromonospora | 10 | 9 |
| Corynebacteriales | Nocardiaceae | Nocardia | 8 | 5 |
| Streptosporangiales | Streptosporangiaceae | Nonomuraea | 4 | 2 |
| Micrococcales | Promicromonosporaceae | Promicromonospora | 1 | 1 |
| Total | 261 | 105 |
表选项
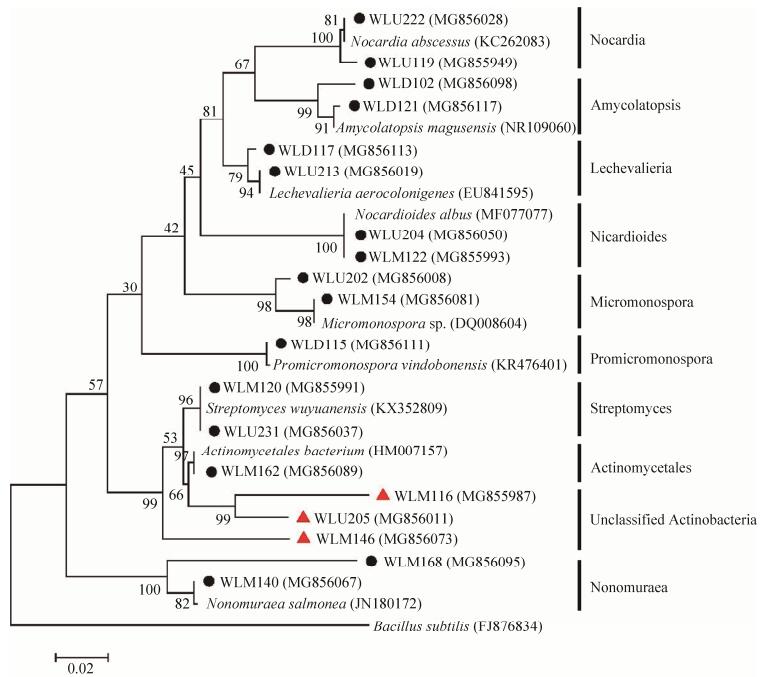 |
| 图 4 基于16S rRNA基因序列构建的代表菌株与相关菌株的ML树 Figure 4 Maximum-Likelihood phylogenetic tree based on 16S rRNA gene sequences between representative actinobacteria and related strains. The dots in the figure are represented the actinobacteria strains isolated from soil samples, the triangles are represented unknown species. Bootstrap values are shown (for 1000 iterations). Bar: 0.02 substitutions per site. |
| 图选项 |
各属及不同湿地样品中的分离株数和分离频率如图 5所示,分离频率较高的物种依次为Streptomyces spp.、Nocardioides spp.、Micromonospora spp.和Nocardia spp.,该结果与免培养放线菌多样性分析结果基本一致。在可培养分离株中,链霉菌属菌株220株,占总分离株的84.3%,其次是类诺卡氏菌属、小单孢菌属和诺卡氏菌属,分别为11株、10株和8株,其他属放线菌分离菌株较少。按照放线菌16S rRNA基因序列相似性99%为阈值[13],261株分离物可被划分为105个OTUs,可能代表 105个物种,其中,链霉菌物种79个,小单孢菌物种9个,诺卡氏菌物种5个。表明河南黄河湿地可培养放线菌物种丰富。与免培养分析结果相比,可培养放线菌多样性偏低,可培养放线菌中链霉菌占绝对优势,而在免培养中,链霉菌的丰富度远低于类诺卡氏菌,甚至低于CL500-29 marine group,且免培养中丰度较高的物种如CL500-29 marine group、Fodinicola、Ilumatobacter、Mycobacterium、Iamia、Aeromicrobium、Corynebacterium和Actinomadura等并没有分离获得可培养菌株,因此,针对黄河湿地放线菌的分离培养条件和技术还有待进一步研究。
 |
| 图 5 所有土壤样品中放线菌在属水平上的分布图(a)和不同黄河湿地放线菌在属水平上的株数图(b) Figure 5 The percentage of the numbers of actinobacteria in genera level isolated from all soil samples (a) and the numbers of actinobacteria in genera level isolated from different yellow river wetlands (b). A: Sanmenxia wetland (Beach); B: Sanmenxia wetland (Reeds); C: Zhengzhou wetland (Beach); D: Zhengzhou wetland (Reeds); E: Kaifeng wetland (Beach). |
| 图选项 |
此外,分离菌株中有8株的16S rRNA基因序列与GenBank中已知序列的比对结果低于97%,可能属于新属(种)(表 6)。表明黄河湿地可培养放线菌种群丰富,且可能存在一些未被分离鉴定的新种。
表 6. 与模式菌株的16S rRNA基因序列相似性低于97%的8株分离菌株 Table 6. Isolates that shared < 97% 16S rRNA gene sequence similarity with the nearest type strains
| Strain No. | GenBank No. | The closest typical strain | Sequence similarity/% |
| WLU244 | MG856133 | Streptomyces coeruleorubidus (EU841625.1) | 92.3 |
| WLU145 | MG856134 | Streptomyces wuyuanensis (KX352809.1) | 94.1 |
| WLM116 | MG855987 | Streptomyces coeruleorubidus (KY213674.1) | 93.4 |
| WLU205 | MG856011 | Streptomyces griseorubens (KJ020693.1) | 94.6 |
| WLM137 | MG856064 | Streptomyces aurantiogriseus (JN180195.1) | 96.5 |
| WLU111 | MG855941 | Streptomyces cyaneus (DQ462653.1) | 97.2 |
| WLM146 | MG856073 | Streptomyces scabiei (AY207602.1) | 97.3 |
| WLM168 | MG856095 | Nonomuraea kuesteri (FJ532449.1) | 93.2 |
表选项
2.3 拮抗活性菌株的筛选结果 以7种植物病原菌为靶标,通过平板对峙法从261株分离株中获得了86株至少对其中一种植物病原菌有拮抗活性的菌株,阳性率约为33%,其中链霉菌属有82株,拟无枝酸菌属2株,放线菌属、列舍瓦列氏菌属各1株,部分菌株的对峙培养结果如图 6所示。活性菌株中,WLU210 (Streptomyces yangpuensis)、WLM212 (Streptomyces globosus)、WLM167 (Streptomyces globosus)对7种靶标菌均有很好的抑制效果,表明河南黄河湿地中蕴藏丰富的生防放线菌资源,有一定的发掘潜力。
 |
| 图 6 部分菌株的对峙培养结果 Figure 6 The dual culture results between actinobacteria strains and plant pathogens. |
| 图选项 |
2.4 活性化合物合成相关基因的筛查结果 选取上述有拮抗活性的86株分离株,扩增了PKS-Ⅰ (Ⅰ型聚酮合酶基因)、PKS-Ⅱ (Ⅱ型聚酮合酶基因)、NRPS (非核糖体多肽合成酶基因)、AHBA (安莎类化合物基因)和HMG CoA (3-羟基3-甲基戊二酰辅酶A还原酶基因)这5类化合物的合成相关基因。结果显示(图 7),86株检测菌中PKS-Ⅱ基因和HMGA基因呈阳性的比率最高,均为56株,占比65.1%,PKS-Ⅰ基因的阳性比率为7%,NRPS基因的阳性比率为24.4%,AHBA基因的阳性比率为27.9%。同时含有2类以上功能基因的菌株56株,占比65.1%;同时含有3类以上功能基因的菌株23株,占比26.8%;同时含有4类以上功能基因的菌株9株,占比10.5%,菌株WLD112 (Streptomyces galilaeus)同时含有5类功能基因。4株有拮抗活性的稀有放线菌均含有3类以上功能基因。另有12株拮抗活性菌株并未检测到上述功能基因序列,可能存在其他次级代谢活性物质的合成途径。菌株WLM101 (Streptomyces hygroscopicus)和WLM103 (Streptomyces hygroscopicus)对苹果轮纹病菌有良好且稳定拮抗活性,对峙培养时,病原菌几乎被完全抑制,二者均含有AHBA功能基因序列,这提示我们,安莎类化合物及其类似结构可能是苹果轮纹病防治药剂研发的方向之一。
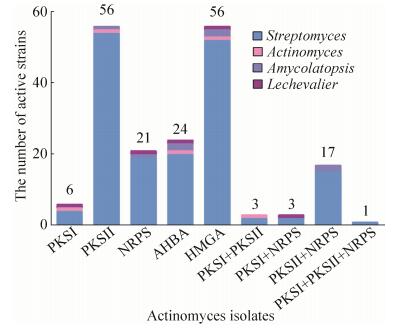 |
| 图 7 五种生物合成功能基因在不同属的活性菌株中的分布情况 Figure 7 The distribution of five biosynthetic functional genes in active strains of different genera. |
| 图选项 |
3 讨论 微生物物种资源的多样性反映出基因资源的丰富性,决定了其次生代谢产物的多样性和新颖性,因此,从特殊生境如海洋、极地、盐碱地、湿地等发掘新放线菌资源的研究已成为国内外****关注的热点。在湿地放线菌资源研究方面,国内多集中于高原湿地、滨海湿地和南方红树林湿地[14-16]。河南黄河湿地分布于黄河中、下游,是东部平原与西部山地丘陵、黄土高原的过渡地带,湿地类型多样,但至今未见对河南黄河湿地放线菌资源的调查研究报道。本研究结果表明,河南黄河湿地放线菌多样性丰富,且不同湿地生境的放线菌多样性有一定的差异性,这一研究结果与冯鸽等[5]报道的黄河下游三角洲滨海湿地放线菌分布特点一致。但是,河南黄河湿地放线菌的优势物种为Nocardioides、CL500-29 marine group和Streptomyces,与黄河下游三角洲滨海湿地的优势菌明显不同,也不同于其他湿地的研究结果[17],表明地处黄河中下游的河南黄河湿地放线菌物种分布具有其独特性,尤其是稀有放线菌物种丰富,有一定的发掘潜力。
随着高通量测序技术的完善和发展,基于16S rRNA基因或其他功能基因文库的宏基因组高通量测序技术已在放线菌多样性研究中得到广泛应用[18-19]。目前,用于克隆文库构建的通用引物多为27F和1492R。然而,有报道认为,该序列的通用性对放线菌不是十分理想,不能很好地反映环境样品中的放线菌多样性。基于此,相关特异性引物如S-S-Svio-66-a-S-20F和S-S-Svio-1274-a- A-20R[20]、Eub530F和Act1114R[21]、Act 235F和Eub 518R[22]等相继用于放线菌多样性研究的克隆文库构建。本研究在参考相关文献的基础上,采用两步PCR扩增后,等量混合2次扩增的PCR产物构建文库,测序结果表明,基于该文库的OTU覆盖率均在99%以上,Shannon指数为4.6512- 5.6045,表明该方法构建的文库完全满足土壤放线菌宏基因组高通量测序的要求。本研究中,放线菌多样性的免培养分析结果与可培养放线菌分析结果并不一致,其中,一些免培养的物种丰度较高的放线菌在分离培养中并没有得到,而丰度较低的物种,如Actinomyces、Amycolatopsis、Lechevalieria、Nocardia、Nonomuraea和Promicromonospora,则被分离获得。由此可见,免培养技术可提供全面的放线菌物种多样性及其分布特点方面的信息,但并不能完全解决放线菌资源利用方面的实际问题,在利用免培养技术研究黄河湿地放线菌多样性、发掘稀有放线菌资源的同时,仍需要不断改进分离培养方法和技术,以便获取更多可利用的放线菌资源。此外,本研究仅对三门峡、郑州和开封黄河湿地放线菌多样性及可培养放线菌进行了研究,采样地点未能覆盖河南黄河湿地的所有湿地类型和生境,研究结果尚不足以全面反映黄河中、下游湿地放线菌多样性及其分布特点,因此,有必要扩大采样范围,并设计多种分离培养方法,全面揭示河南黄河湿地放线菌多样性及其分布特点。
本研究从261株分离物中筛选出86株对植物病原菌有拮抗活性的菌株,其中,74株至少含有一种测定的代谢活性物质合成相关的功能基因,表明河南黄河湿地蕴藏丰富的活性放线菌资源,且活性物质代谢途径多样,具有发掘新活性物质的潜力。对供试的7种植物病原菌均具有很强拮抗活性的菌株WLU210被鉴定为Streptomyces yangpuensis,WLM212和WLM167被鉴定为Streptomyces globosus。菌株Streptomyces yangpuensis为Tang等2016年首次从上海的土壤中分离获得的新种[23],目前未见对该菌株生物活性,尤其是在植物病害生物防治应用潜力方面研究报道;Streptomyces globosus是一种从海洋、高山土壤和工业废水等样品中分离出的链霉菌,该菌株具有较强的抗细菌、抗肿瘤活性[24],也是一种有效的植物病害生防菌[25]。活性物质功能基因筛查结果表明,菌株Streptomyces yangpuensis WLU210中只含有PKS-Ⅱ,菌株Streptomyces globosus WLM212含有PKS-Ⅱ和NRPS基因,而菌株Streptomyces globosus WLM167只含有HMGA基因,反映了放线菌菌株活性物质代谢途径的差异性,其代谢活性物质的类型及其在植物病害生物防治方面的应用有待于进一步研究。此外,本研究中筛选出的拮抗活性菌株数量虽然多,但是绝大多数为链霉菌属,稀有放线菌仅有4株,分别是拟无枝酸菌属(Amycolatopsis) 2株、放线菌属(Actinomyces)和列舍瓦列氏菌属(Lechevalieria)各1株。总之,免培养分析结果中的多种稀有放线菌在分离培养基上尚未分离获得纯培养,因此,河南黄河湿地放线菌中可能仍存在大量的稀有放线菌资源有待发掘。
References
| [1] | Li WJ, Zhi XY, Tang SK. Actinobacterial systematics in China: past, present and future. Microbiology China, 2013, 40(10): 1860-1873. (in Chinese) 李文均, 职晓阳, 唐蜀昆. 我国放线菌系统学研究历史、现状及未来发展趋势. 微生物学通报, 2013, 40(10): 1860-1873. |
| [2] | Genilloud O. Actinomycetes: still a source of novel antibiotics. Natural Product Reports, 2017, 34(10): 1203-1232. DOI:10.1039/C7NP00026J |
| [3] | Law WF, Ser HL, Khan TM, Chuah LH, Pusparajah P, Chan KG, Goh BH, Lee LH. The potential of Streptomyces as biocontrol agents against the rice blast fungus, Magnaporthe oryzae (Pyricularia oryzae). Frontiers in Microbiology, 2017, 8: 3. |
| [4] | Tang J, Xu QR, Wang LM, Ding X, Tang B, Wu LS, Feng S, Sun Q, Yang ZR, Zhang J. Soil bacterial community diversity under different stages of degradation in Zoige wetland. Microbiology China, 2011, 38(5): 677-686. (in Chinese) 唐杰, 徐青锐, 王立明, 丁祥, 汤博, 吴俐莎, 冯甦, 孙群, 杨志荣, 张杰. 若尔盖高原湿地不同退化阶段的土壤细菌群落多样性. 微生物学通报, 2011, 38(5): 677-686. |
| [5] | Feng G, Xu SS, Wang C, Yan LE, Zhang X, Li J. Actinobacterial diversity in a coastal wetland of the Yellow River delta revealed by culture-independent approach. Microbiology China, 2013, 40(9): 1570-1579. (in Chinese) 冯鸽, 许姗姗, 王超, 严立恩, 张璇, 李静. 黄河三角洲滨海湿地非培养放线菌多样性. 微生物学通报, 2013, 40(9): 1570-1579. |
| [6] | Zhang YQ, Liu HY, Chen J, Yuan LJ, Sun W, Zhang LX, Zhang YQ, Yu LY, Li WJ. Diversity of culturable actinobacteria from Qinghai-Tibet Plateau, China. Antonie van Leeuwenhoek, 2010, 98(2): 213-223. DOI:10.1007/s10482-010-9434-4 |
| [7] | Huang J, Yan BF, Huang Y. Diversity of culturable actinobacteria from soils collected in Ali, Naqu and Haixi districts on the Qinghai-Tibet Plateau. Acta Microbiologica Sinica, 2017, 57(9): 1342-1351. (in Chinese) 黄娇, 闫兵法, 黄英. 青藏高原阿里、那曲和海西地区土壤可培养放线菌的多样性. 微生物学报, 2017, 57(9): 1342-1351. |
| [8] | Zhai YY, Wang WF, Tan HM, Cao LX. A new approach to analyzing endophytic actinobacterial population in the roots of banana plants (Musa sp., AAA). Journal of Biochemistry and Molecular Biology Research, 2016, 2(3): 180-184. DOI:10.17554/j.issn.2313-7177.2016.02.30 |
| [9] | Li XJ, Wu Y, Zhang WM, Li J, Liu SW, Jiang ZK, Huang DL, Sun CH. Biodiversity and antimicrobial activity of culturable actinobacteria isolated from Jiuliancheng Nur in Hebei province. Microbiology China, 2016, 43(7): 1473-1484. (in Chinese) 李小俊, 吴越, 张伟铭, 李静, 刘少伟, 蒋忠科, 黄大林, 孙承航. 河北九莲城淖尔可培养放线菌多样性及抗菌活性筛选. 微生物学通报, 2016, 43(7): 1473-1484. |
| [10] | Xue D, Zhao ZG, Yao Q, Zhao HQ, Zhu HH. Diversity of cultivable actinobacteria in Xinghu wetland sediments. Acta Microbiologica Sinica, 2015, 55(11): 1485-1494. (in Chinese) 薛冬, 赵国振, 姚青, 赵海泉, 朱红惠. 肇庆星湖湿地可培养放线菌多样性. 微生物学报, 2015, 55(11): 1485-1494. |
| [11] | Sigmund JM, Clark DC, Rainey FA, Anderson AS. Detection of eubacterial 3-hydroxy-3-methylglutaryl coenzyme a reductases from natural populations of actinomycetes. Microbial Ecology, 2003, 46(1): 106-112. DOI:10.1007/s00248-002-2029-5 |
| [12] | Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [13] | Stach JEM, Maldonado LA, Masson DG, Ward AC, Goodfellow M, Bull AT. Statistical approaches for estimating actinobacterial diversity in marine sediments. Applied and Environmental Microbiology, 2003, 69(10): 6189-6200. DOI:10.1128/AEM.69.10.6189-6200.2003 |
| [14] | He JQ, Zhang GJ, Yue HM. Diversity and bioactivity analysis of actinomycetes isolated from Lhalu wetland nature reserve. Journal of Microbiology, 2009, 29(4): 6-10. (in Chinese) 何建清, 张格杰, 岳海梅. 拉鲁湿地自然保护区放线菌组成分析及生物活性测定. 微生物学杂志, 2009, 29(4): 6-10. DOI:10.3969/j.issn.1005-7021.2009.04.002 |
| [15] | Guan XY, Han JB, Wang B, Dong Y, Jiang B, Chen Z, Yang AF, Wang ZH, Zhou ZC. Analysis of bacterial communities in Liaodong Bay Dalinghe estuarine wetland. Ecology and Environmental Sciences, 2012, 21(6): 1063-1070. (in Chinese) 关晓燕, 韩家波, 王摆, 董颖, 姜北, 陈仲, 杨爱馥, 王召会, 周遵春. 辽东湾大凌河口湿地土壤微生物群落分析. 生态环境学报, 2012, 21(6): 1063-1070. |
| [16] | Hong K. Actinomycetes from mangrove and their secondary metabolites. Acta Microbiologica Sinica, 2013, 53(11): 1131-1141. (in Chinese) 洪葵. 红树林放线菌及其天然产物研究进展. 微生物学报, 2013, 53(11): 1131-1141. |
| [17] | Xu SQ, Wang YD, Guo CC, Zhang ZG, Shang YT, Chen Q, Wang ZL. Comparison of microbial community composition and diversity in native coastal wetlands and wetlands that have undergone long-term agricultural reclamation. Wetlands, 2017, 37(1): 99-108. |
| [18] | Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [19] | Labeda DP, Dunlap CA, Rong XY, Huang Y, Doroghazi JR, Ju KS, Metcalf WW. Phylogenetic relationships in the family Streptomycetaceae using multi-locus sequence analysis. Antonie van Leeuwenhoek, 2017, 110(4): 563-583. DOI:10.1007/s10482-016-0824-0 |
| [20] | Kumar Y, Aiemsum-ang P, Ward AC, Goodfellow M. Diversity and geographical distribution of members of the Streptomyces violaceusniger 16S rRNA gene clade detected by clade-specific PCR primers. FEMS Microbiology Ecology, 2007, 62(1): 54-63. DOI:10.1111/fem.2007.62.issue-1 |
| [21] | Kyselková M, Kopecky J, Felf?ldi T, ?ermák L, Omelka M, Grundmann GL, Mo?nne-Loccoz Y, Ságová-Mare?ková M. Development of a 16S rRNA gene-based prototype microarray for the detection of selected actinomycetes genera. Antonie van Leeuwenhoek, 2008, 94(3): 439-453. DOI:10.1007/s10482-008-9261-z |
| [22] | Větrovsky T, Baldrian P. An in-depth analysis of actinobacterial communities shows their high diversity in grassland soils along a gradient of mixed heavy metal contamination. Biology and Fertility of Soils, 2015, 51(7): 827-837. DOI:10.1007/s00374-015-1029-9 |
| [23] | Tang B, Yu YC, Zhi XY, Yang LL, Cen XF, Zhao GP, Ding XM. Streptomyces yangpuensis sp. nov., isolated from soil. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(3): 1224-1229. DOI:10.1099/ijsem.0.000861 |
| [24] | Ravi L, Krishnan K. Benzoyloxy-ethyl-carbamic acid: a novel anticancerous secondary metabolite produced by Streptomyces globosus VITLGK011. Indian Journal of Experimental Biology, 2017, 55: 411-420. |
| [25] | Saeed EE, Sham A, Salmin Z, Abdelmowla Y, Iratni R, El-Tarabily K, AbuQamar S. Streptomyces globosus UAE1, a potential effective biocontrol agent for black scorch disease in date palm plantations. Frontiers in Microbiology, 2017, 8: 1455. DOI:10.3389/fmicb.2017.01455 |
