邓嘉雯#, 高云雨#, 刘旭坤, 张珂珂, 钟路遥, 田生礼


深圳大学生命与海洋科学学院 深圳市微生物基因工程重点实验室, 广东 深圳 518052
收稿日期:2018-06-19;修回日期:2018-08-27;网络出版日期:2018-11-26
基金项目:国家自然科学基金(31070044);深圳市科技基础研究发展计划(ZYC201105130092A)
*通信作者:田生礼, Tel:+86-755-26534149, Fax:+86-755-26534274, E-mail:sltian@szu.edu.cn.
#并列第一作者
摘要:[目的] 构建多靶向siRNA表达载体对里氏木霉碳阻遏抑制因子CRE1、CRE2、CRE3和CRE4进行同时多靶向siRNA干扰,以研究其对里氏木霉纤维素酶基因表达的调控作用。[方法] 根据此前研究筛选出沉默cre1、cre2、cre3和cre4基因的4个最佳siRNA序列,设计并构建了A多靶向表达载体,另根据cre1、cre2、cre3和cre4基因中所含有的5个共有序列设计并构建了B多靶向表达载体,将两者转化至里氏木霉QM9414。经筛选后分别在48 h和120 h对各转化子进行纤维素酶酶活力测试(CMC活力测试和滤纸酶酶活力测试)及利用qPCR检测相关基因的表达。[结果] 通过RT-qPCR测定结果表明,两种表达载体均可同时抑制里氏木霉的分解代谢物阻遏基因cre1、cre2、cre3和cre4的表达,纤维素酶活力比出发菌株明显升高,多靶向抑制菌株的CMC酶活和滤纸酶活比出发菌株平均提高了1.95倍和2.66倍。纤维素酶基因cbh1和egl1的表达水平比出发菌株也有明显提升,平均提高了3.83倍和3.95倍。纤维素酶相关基因xyr1的表达水平与出发菌株相比也明显上升,平均提高了2.78倍。[结论] 多靶向沉默里氏木霉的碳代谢阻遏蛋白有利于解除葡萄糖效应,提高非还原糖的利用,从而提高纤维素酶的产量,使纤维素酶的表达得到更大的提升,为里氏木霉表达纤维素酶在分解代谢物阻遏基因调控方面提供了实验依据和新的技术思路。
关键词:里氏木霉RNA干扰碳代谢阻遏物纤维素酶
Cellulase activities and expression regulation by multiple targeted silencing carbon catabolic repressors in Trichoderma reesei
Jiawen Deng#, Yunyu Gao#, Xukun Liu, Keke Zhang, Luyao Zhong, Shengli Tian


Shenzhen Key Laboratory of Microbial Genetic Engineering, College of Life Sciences and Oceanography, Shenzhen University, Shenzhen 518052, Guangdong Province, China
*Corresponding author: Shengli Tian, Tel:+86-755-26534149, Fax:+86-755-26534274, E-mail:sltian@szu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31070044) and by the Shenzhen Science and Technology Basic Research and Development Project (ZYC201105130092A)
#These authors contributed equally to this work
Abstract: [Objective] In order to study carbon metabolic repressors on cellulase activities and expression regulation in Trichoderma reesei, we constructed multi-targeting siRNA expression vectors to perform silencing carbon metabolic repressor cre1, cre2, cre3 and cre4 by simultaneous producing multi-targeting siRNAs. [Methods] According to previous studies and screening, the four best siRNAs targeting the cre1, cre2, cre3 and cre4 were selected and they were constructed a multi-targeting siRNA expression vector A. Additionally, according to 5 overlap common sequence in the cre1, cre2, cre3 and cre4, we designed and constructed the vector B. The two vectors were transformed the protoplasm of Trichoderma reesei QM9414 and selected on the hygromycin selection medium. Cellulase activities (CMC activity and filter paper activity) of transformants were detected and related gene expressions were also detected by RT-qPCR after incubation for 48 and 120 hours respectively. [Results] RT-qPCR results showed that the cre1, cre2, cre3 and cre4 expression levels in Trichoderma reesei were simultaneously silenced, and the cellulase activities were much higher than that of the staring strain. The CMC activity and filter paper activity of the multi-targeted inhibitor strains were 1.95-fold and 2.66-fold higher than those of the original strains. The cellulase-related genes expression levels were also increased significantly. The expression levels of cbh1, egl1 and xyr1 were 3.83-fold, 3.95-fold and 2.78-fold higher than those of the original strain. [Conclusion] Our results indicated that simultaneous silencing multi-targets of the carbon metabolic repressors in Trichoderma reesei is beneficial to release the glucose repressor effects and increase the expression and production of cellulase. These results provide evidence and techniques for study of regulation of carbon catabolite repressor genes on cellulase expression in Trichoderma reesei.
Keywords: Trichoderma reeseiRNA interferencecarbon metabolic repressorcellulase
里氏木霉(Trichoderma reesei)是红褐肉座菌(Hypocrea jecorina)的无性型,是一种重要的产纤维素酶的丝状真菌,其广泛用于生产纤维素酶[1]。里氏木霉所产的纤维素酶是一种复杂的酶体系,主要由3类酶组成:内切葡聚糖酶(endo-β-1, 4-D- glucanase,EG)、外切葡聚糖酶(exo-1, 4-β-D- glucanase,CBH)以及β-葡萄糖苷酶(β-glucosidase,BGL)。里氏木霉具有极强的合成和分泌蛋白的能力,其合成和分泌蛋白已经可以达到克级以上[2],主要生产纤维素酶和纤维二糖水解酶Ⅰ (CBH1),表达量最高的菌株分泌的纤维素酶总量可达100 g/L[3]。里氏木霉培养方法成熟,产酶能力强大,而且安全,对环境无损坏,不会产生有毒的副产物。因此,里氏木霉成为重组蛋白表达和生产的优良宿主,可以通过基因工程等手段对里氏木霉纤维素酶基因进行调控,以达到提高纤维素酶产量的目的[4]。里氏木霉的纤维素酶基因表达存在着一种效应,当存在快速利用碳源如葡萄糖时,纤维素酶基因的表达会受到抑制,这种现象就是分解代谢物阻遏效应,也称为碳阻遏效应[5-6],目前公开发表的阻遏蛋白是CRE1、CRE2和ACE1。CRE1是里氏木霉中介导葡萄糖分解代谢物阻遏作用的蛋白,含有2个Cys2-His2锌指结构域,并且参与了脯氨酸代谢、乙醇代谢、多糖水解等过程中的基因转录调控。除此之外,参与里氏木霉碳源代谢阻遏的调控蛋白还包括CRE2、CRE3和CRE4,它们分别与构巢曲霉碳源代谢阻遏蛋白CREA、CREB、CREC和CRED具有较高的同源性[7-8]。CRE2和CRE3分别与CREB和CREC高度同源,CREB和CREC结合形成去泛素化复合蛋白,使泛素化的蛋白质去泛素化,使蛋白稳定[9]。Denton敲除里氏木霉中creB的同源序列cre2,可以提高纤维素酶的产量[10]。CRED参与对靶蛋白进行泛素化标记的过程,与CREB-CREC复合物的去泛素化过程相反[7]。creB和creC的基因突变可导致“碳抑制”的部分解除,并影响一些其他基因的表达,但不像creA的突变那样能引起严重的形态学改变。ACE1是纤维素酶基因表达的转录抑制因子,是含有3个Cys2-His2锌指结构域的DNA结合蛋白[11],在里氏木霉中敲除基因ace1后,在含有纤维素和槐糖的培养条件下能够提高主要的纤维素酶基因和半纤维素酶基因的表达量[12]。
目前,RNA干扰技术(RNA interference,RNAi)应用广泛,该技术在丝状真菌如里氏木霉、康宁木霉中对基因进行调控已有较多的报道。在里氏木霉中进行siRNA干扰已经有了较为成熟的技术手段。例如使用发夹结构的RNAi技术成功将里氏木霉中高表达的基因cbh1和cel6a基因进行抑制[13]。但RNAi技术仍停留在单基因单抑制的水平上,对于需要同时进行多基因共同作用的现象则难以发挥其功效。多靶向RNAi技术在治疗基因疾病上运用较多,但在真菌中的应用较少,He等[14]在里氏木霉中建立了包含trpC和rp2的反向双启动子系统同时抑制了靶基因和eGFP基因,为里氏木霉多靶向siRNA提供了技术路线。本研究通过设计2种不同的多靶向干扰系统,构建里氏木霉多靶向siRNA干扰载体,对里氏木霉碳阻遏抑制因子CRE1、CRE2、CRE3和CRE4进行同时多靶向siRNA干扰,探讨里氏木霉cre1、cre2、cre3和cre4同时沉默对纤维素酶基因的表达的影响,为里氏木霉进一步基因调控提供了新的技术手段。
1 材料和方法 1.1 材料
1.1.1 菌株及质粒: 大肠埃希菌JM109由本实验室保存,里氏木霉QM9414菌株购自美国模式菌种收藏中心(ATCC);质粒pLXT带有真菌筛选标记潮霉素B抗性基因(hph)和大肠埃希菌筛选标记氨苄青霉素抗性基因(Amp),由本实验室构建及保存。
1.1.2 培养基: 里氏木霉液体基本培养基(g/L):KH2PO4 2.0,(NH4)2SO4 1.4,尿素0.3,CaCl2·2H2O 0.4,FeSO4·7H2O 0.005,MnSO4·H2O 0.0016,ZnSO4·7H2O 0.0017,CoCl2·6H2O 0.0037,蛋白胨2,吐温-80 1,葡萄糖20。里氏木霉产酶培养基:产酶培养基组分与基本培养基一致,用微晶纤维素20 g/L代替葡萄糖作碳源。
1.1.3 试剂: 各种DNA工具酶、T4 DNA连接酶、DNA Marker和RNase Free H2O等均购自TaKaRa公司;DNA胶回收纯化试剂盒、PCR产物回收纯化试剂盒购自生工生物工程(上海)股份有限公司;真菌总RNA提取试剂盒购自Omega公司;反转录试剂盒(EasyScript One-Step gDNA Removal and cDNA Synthesis SuperMix)购自北京全式金生物技术有限公司;qPCR试剂盒(Bestar?SybrGreen qPCR mastermix)购自DBI Bioscience公司;溶壁酶(Lysing Enzyme from Trichoderma harzianum)购自Sigma公司;潮霉素和氨苄青霉素购自Invitrogen公司;其他试剂均购自生工生物工程(上海)股份有限公司。
1.1.4 仪器: 超净工作台(苏州净化设备总厂,SW-SJID);高压自动灭菌锅(Hirayama,HVE-50);PCR仪(ABI,2720);冷冻离心机(Thermo Scientific,Multifuge XIR);荧光定量PCR仪(analytik jena,qTOWER3);NanoDrop2000超微量分光光度计(Thermo Scientific,ND2000);pH计(Mettler-Toledo,320-S);小型离心机(Eppendorf,Minispin);电泳仪(Amersham Pharmacia,EPS-601);水平电泳槽(北京六一,DYCP-31D);核酸成像系统(BioRad,GEL DOX XR +);高纯水仪(力新仪器有限公司,Heal force ROP3);低温摇床(上海苏坤,SKY-200B);低温培养箱(上海爱朗,LTI-700)。
1.2 多靶向沉默片段的选择与合成 根据高云雨等[4]的研究,已筛选出cre1中具有明确抑制效果的siRNA序列,及经过本实验室此前的实验研究[15-17]已筛选出沉默cre2、cre3和cre4基因中具有最佳效果的siRNA序列。选择出4条siRNA序列,见表 1。
表 1. 多靶向沉默碳代谢阻遏物cre基因的最佳序列 Table 1. The best option of siRNA sequences for silencing cre1, cre2, cre3 and cre4
| Gene | siRNA name | Sequence (5′→3′) |
| cre1 | cre1-T10 | GCATGTCACTTACCGACAT |
| cre2 | cre2-T15 | AGCGTCATCAACTATCCAT |
| cre3 | cre3-T11 | CCAAGATGTCCATATTGAG |
| cre4 | cre4-T2 | GAAGACTTCATCGTCTTCC |
表选项
确定了筛选的siRNA序列后,需对其设计合理的表达系统,从而形成一条反向重复长发夹结构的DNA片段。设计A和B两种表达系统,这两种表达系统均采用反向重复序列,前端序列与后端序列中间以无义序列作为loop连接,在序列两端分别加上Not Ⅰ和Afl Ⅱ限制性内切酶位点。
A载体为直接串联的各个优化选择的编码siRNA的寡核苷酸序列:cre1-T10、cre2-T15、cre3-T11和cre4-T2,且已被证明是有效的siRNA序列。所以将cre1-T10、cre2-T15、cre3-T11和cre4-T2分别以9 bp的loop连接形成1个小的发夹结构,将4个发夹结构按顺序分别以23 bp的无义序列连接并命名为cres,见图 1-A。
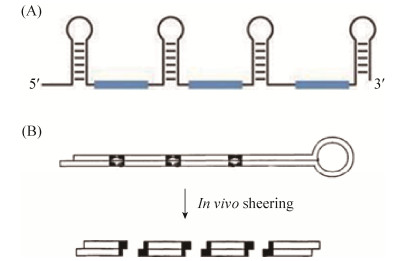 |
| 图 1 多靶向siRNA表达系统示意图 Figure 1 Multiple target siRNA expression system. |
| 图选项 |
B载体是根据4个cre基因的正义链之间使用5 bp共同序列作为缓冲序列连接,当Dicer酶在体内切割该序列的时候,无论以19-24 bp中任意位置切割,切割出来的siRNA都具有很高的特异性,能和靶基因完全匹配。参考Cong等的方法[18]寻找共同序列,尽量选择符合siRNA设计原则的序列。将4个cre基因的mRMA序列输入网站(Invitrogen Block-iT RNAi Designer)进行siRNA靶序列的设计,找出各个基因所有25 bp的siRNA序列,通过比对寻找各基因的siRNA序列首尾是否有5 bp的共同序列,将首尾有共同序列的siRNA片段进行连接,连接结果如图 2所示。将前端序列反向重复形成后端序列,前端序列和后端序列之间加入Hap Ⅰ限制性内切酶位点GTTAAC以及egll基因的内含子2的一段序列(agtgagtacccttgtttcctggtgtt gctggctggttgggcgggtatacagcgaagcggacgcaagaacaccgccggtccgccaccatcaagatgtgggtggtaagcggcggtgttttgtacaactacctgacagctcactcaggaaatgagaattaatggaagtcttgttacag)将前端序列和后端序列连接起来,并命名为cre1423。同理得到另一条长链siRNA命名为cre4132。两条长链siRNA序列见表 2,其表达系统模型示意图如图 1-B。
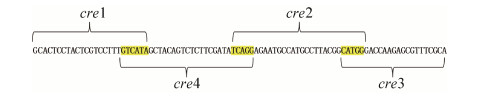 |
| 图 2 表达系统中的siRNA Figure 2 The sequence of oligonucleotides coding for long siRNAs. |
| 图选项 |
表 2. 合成的长链编码siRNA的寡核苷酸序列 Table 2. The sequence of oligonucleotides coding for long siRNAs
| Name | Sequence | Expression system |
| CRES | CTTAAGGCATGTCACTTACCGACATttcaagagaATGTCGGTAAGTGACATGCCAGCTT GACTAGATGATGACCTAAGCGTCATCAACTATCCATttcaagagaATGGATAGTTGATGACGCTTCAATTACGTTCTACTGTCATCTCCAAGATGTCCATATTGAGttcaagaga CTCAATATGGACATCTTGGAGACTAGGGTGTTACGCACTAGTGAAGACTTCATCGTCTTCC ttcaagaga GGAAGACGATGAAGTCTTC TTTTTTGCGGCCGC | A |
| CRE1423 | CTTAAGGCACTCCTACTCGTCCTTTGTCAT AGCTACAGTCTCTTCGATATCAGGAGAATGCCATGCCTTACGGCATGGTGACCAA GAGCGTTTCGCAGTTAACagtgagtacccttgtttcctggtgttgctggctggttgggcgggtatacagcgaagcggacgcaagaacaccgccggtccgccaccatcaagatgtgggtggtaagcggcg gtgttttgtacaactacctgacagctcactcaggaaatgagaattaatggaagtcttgttacagTGCGAAACGCTCTTGGTCACCATGCC GTAAGGCATGGCATTCTCCTGATATCGAAGAGACTGTAGCTATGACAAAGGACGAGTAGGAGTGC GCGGCCGC | B |
| CRE4132 | CTTAAGAGCGTCTCACTGGATCAGTCGCAGCCCCGGCATGTCACTTACCGACATCACCA ACCAAGCAAGATTACTTGACAAGCACTTTGTGCGCAATTTC TGTTAACagtgagtacccttgtttcctggtgttgctggctggttgggcgggtatacagcgaagcggacgcaagaacaccgccgg tccgccaccatcaagatgtgggtggtaagcggcggtgttttgtacaactacctgacagctcactcaggaaatgagaattaatggaagtcttgttacag AGAAATTGCGCACAAAGTGCTTGTCAAGTAATCTTGCTTGGTTGGTGATGTC GGTAAGTGACATGCCGGGGCTGCGACTGATCCAGTGAGACGCTTTTTTGCGGCCGC | B |
| The lower case is loop and the single underlines are Afl Ⅱ, Hap Ⅰ and Not Ⅰ restriction sites, respectively. | ||
表选项
将设计好的序列送广州艾基生物技术有限公司进行合成,将合成产物退火,连接到pUC19质粒载体中,分别为pUC19-cres、pUC19-cre1423、pUC19-cre4132。
1.3 多靶向沉默载体的构建 pLXT载体(载体图谱见图 3)的特征是含有潮霉素抗性基因hph,丙酮酸脱羧酶启动子Ppdc和其终止子Tpdc,将该载体与目的基因重组后的重组质粒转化里氏木霉原生质体,在选择培养平板上长出含有抗性基因的阳性转化子,再通过PCR验证可大大减少筛选的工作量。该载体和质粒pUC19-cres、pUC19-cre1423、pUC19-cre4132均通过限制性内切酶Afl Ⅱ和Not Ⅰ酶切后,回收载体片段和相应目的基因片段,用T4连接酶进行连接,转化E. coli JM109感受态细胞。构建成功的含有干扰多靶向siRNA基因的重组质粒,提取质粒分别命名为pLXT-cres、pLXT-cre1423和pLXT-cre4132,这些质粒可直接进行里氏木霉原生质体转化。
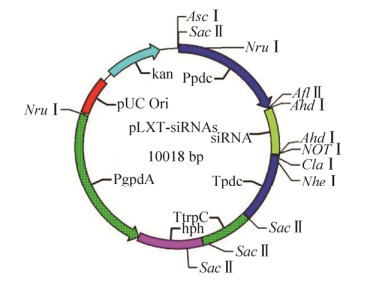 |
| 图 3 载体pLXT-siRNAs图谱 Figure 3 Map of the vector pLXT-siRNAs. |
| 图选项 |
1.4 里氏木霉原生质体转化 参考Penttil?等[19]的实验方法,用聚乙二醇进行原生质体的制备与里氏木霉转化。哈茨木霉的溶壁酶(100 mg/mL)溶于1 mol/L MgSO4用于原生质体的制备。转化时,用10 μg质粒DNA (pLXT-cres、pLXT-cre1423和pLXT-cre4132)和200 μL PEG buffer (60% PEG4000,50 mmol/L CaCl2,10 mmol/L Tris-HCl,pH 7.5)。涂布到含有100 μg/mL潮霉素B的PDA固体平板用于筛选转化子,生长2-3 d。用牙签挑取转化子接种到新的筛选培养基上,并连续筛选培养观察6代。提取转化子的DNA经PCR鉴定确认干扰表达盒插入到转化子的基因组中。
1.5 里氏木霉基因组DNA提取 将PCR筛选出来的转化子接种于里氏木霉液体基本培养基中,28 ℃、250 r/min条件下培养2 d。通过抽滤得到菌丝体,液氮研磨成粉之后,使用Omega的E.Z.N.A? Fungal DNA Kit提取基因组DNA。用NanoDrop 2000超微量分光光度计和琼脂糖凝胶电泳检测DNA样品的浓度和质量。
1.6 RNA提取 为分析siRNA干扰片段对纤维素酶活力的影响,将重组菌T. reesei-cres,T. reesei-cre1423,T. reesei-cre4132和出发菌株T. reesei QM9414分别接种于10 mL基本培养基中,每个菌株分别做3个平行样品,28 ℃、250 r/min培养48 h后,取每种重组菌1.5 mL菌液接种到30 mL含有0.1%微晶纤维素(质量分数)的产酶培养基中,28 ℃、250 r/min培养48 h和120 h后,利用Omega的E.Z.N.A? Fungal RNA Kit,按照说明书的方法,将菌丝通过液氮冷冻研磨成粉后,提取总RNA。用NanoDrop 2000超微量分光光度计和琼脂糖凝胶电泳检测RNA样品的浓度和质量。
1.7 RNA反转录和荧光定量PCR 使用反转录试剂盒,以Random Primer为引物,将约100 ng RNA反转录成cDNA。使用荧光定量PCR仪进行荧光定量PCR。每个反应20 μL体积,包括2 μL模板(1:60稀释反转录产物),10 μL 2×SybrGreen qPCR mastermix,8 μmol/L正反向引物(表 3)各0.5 μL和7 μL nuclease-free水。程序设置:预变性95 ℃ 30 s;95 ℃变性5 s,60 ℃退火5 s,60 ℃延伸31 s,运行40个循环。运行结束后得到融解曲线,用以核对PCR产物的特异性。所有PCR反应每个样品做3个复孔。目的基因的表达量通过内参基因sar1的表达量校正。以对照菌株的表达量为1,其他转化子的表达量与对照样品的比值作为数据分析。
表 3. 荧光定量PCR使用的引物 Table 3. Sequences of primers used in RT-qPCR
| Primer name | Sequence (5'→3') | Purpose |
| sar1-F | TGGATCGTCAACTGGTTCTACGA | Internal reference |
| sar1-R | GCATGTGTAGCAACGTGGTCTTT | |
| cre1-F | CGGCTCCTTCTTCTCCCACCTT | cre1 qPCR |
| cre1-R | TCTCAGACTCGGCAGCTCGTAG | |
| cre2-F | TGCGGGAAGATGCGACTGACA | cre2 qPCR |
| cre2-R | GGCGAGAGCGATGGATAGTTGATG | |
| cre3-F | TGCGGGAAGATGCGACTGACA | cre3 qPCR |
| cre3-R | AATGAGTTGCCCTCCCCGAATGCG | |
| cre4-F | CTCGACCAGCTGTATGACGA | cre4 qPCR |
| cre4-R | TGCGACTGATCCAGTGAGAC | |
| ace1-F | GGACGAGGAGGAGATTATG | ace1-qPCR |
| ace1-R | GTGAGTCTTCTCGTGCTT | |
| cbh1-F | CCGAGCTTGGTAGTTACTCTG | cbh1 qPCR |
| cbh1-R | GGTAGCCTTCTTGAACTGAGT | |
| egl1-F | CGGCTACAAAAGCTACTACG | egl1 qPCR |
| egl1-R | CTGGTACTTGCGGGTGAT | |
| xyr1-F | CCCATTCGGCGGAGGATCAG | xyr1 qPCR |
| xyr1-R | CGAATTCTATACAATGGGCACATGGG |
表选项
1.8 酶活测定 为分析siRNA干扰片段对纤维素酶活力的影响,将重组菌T. reesei-cres、T. reesei-cre1423、T. reesei-cre4132和出发菌株T. reesei QM9414分别接种于30 mL基本培养基中,每个菌株分别做3个平行样品,28 ℃、250 r/min培养48 h后,每种重组菌取1.5 mL菌液接种到30 mL含有0.1%微晶纤维素(质量分数)的产酶培养基中,28 ℃、250 r/min培养48 h,每隔24 h收集酶液,将粗酶液稀释一定倍数后测定其滤纸酶活和CMC酶活。滤纸酶活(FPA)和羧甲基纤维素(CMC)酶活测定方法参考Ghose[20]。以CMC和Whatman No.1滤纸为底物,分别加酶液0.5 mL,50 ℃孵育30 min和1 h,加DNS沸水浴5 min。测定OD540值,每个样品做3个重复,结果取平均值。以1 h内水解50 mg滤纸释放出2.0 mg葡萄糖或30 min内水解2%羧甲基纤维素释放出0.5 mg葡萄糖的酶液稀释倍数来计算FPUase和CMCase活性单位,单位为IU/mL。
2 结果和分析 2.1 多靶向沉默转化子鉴定 将构建的重组质粒pLXT-cre1423、pLXT-cre4132和pLXT-cres分别使用Not Ⅰ和Afl Ⅱ限制性内切酶进行双酶切,酶切产物进行琼脂糖凝胶电泳,结果见图 4。结果显示,泳道1、2、3均在10000 bp处有明显条带,其大小与pLXT载体一致;泳道1、2在接近500 bp处有明显条带,其大小与cre1423、cre4132片段大小一致;泳道3在接近250 bp处有明显条带,其大小与cres片段大小一致,进一步对这3个目的片段进行测序分析(序列未列出),进一步证明了3个siRNA干扰片段连接成功,这些质粒可用于里氏木霉原生质体转化。
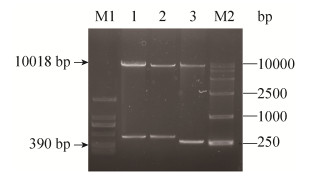 |
| 图 4 重组干扰质粒酶切片段电泳图 Figure 4 The restriction fragment of interference recombinant vector. M1: TaKaRa DL 1000 marker; M2: TaKaRa DL 10000 marker; lane 1-3: The fragments of pLXT-cre1423, pLXT-cre4132 and pLXT-cres were digested with Not Ⅰ+Afl Ⅱ. |
| 图选项 |
2.2 里氏木霉原生质体转化子的鉴定 将重组菌T. reesei-cres、T. reesei-cre1423、T. reesei-cre4132和出发菌株T. reesei QM9414分别提取转化子基因组,使用引物CRE-F:5′-CCTGCAAGTCTCCATCACAAG-3′和CRE-R:5′-TTCATAGTCCCATTGTCAGCA-3′进行PCR鉴定,结果见图 5。结果显示在1000 bp处有明显扩增的条带,该条带的大小与siRNA干扰表达盒的大小基本一致,进一步对PCR扩增的DNA片段进行测序分析,表明DNA序列与siRNA干扰表达盒的序列完全一致,进一步证明了干扰表达盒成功整合到里氏木霉QM9414基因组中。
 |
| 图 5 PCR鉴定里氏木霉基因组中的siRNA干扰表达盒 Figure 5 PCR amplified siRNA disruption cassettes from recombinant strains. M: TaKaRa DL 2000 DNA Marker; lane 1-4: The PCR amplified siRNA disruption cassettes from recombinant strains T. reesei-cre1423, T. reesei-cre4132, T. reesei-cres and T. reesei-QM9414. |
| 图选项 |
2.3 干扰重组菌的纤维素酶酶活的测定 滤纸酶活(FPA)代表的是纤维素酶三种酶组分,即内切型葡聚糖酶、外切型葡聚糖酶、β-葡聚糖苷酶协同作用的总酶活,是菌株整个纤维素酶系酶活力水平的综合体现。重组菌在产酶培养基中产生FPA的测定曲线见图 6-A。实验结果表明,所有菌株的滤纸酶活在120 h内呈现上升的状态。其中,3个重组菌株T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres的酶活性曲线比出发菌株T. reesei QM9414明显升高。3个重组菌中T. reesei-cre4132滤纸酶活的上升趋势最明显,其在144 h达到酶活的最高水平,而T. reesei-cre1423与T. reesei-cres的滤纸酶活力在120 h达到最高点。原始出发菌T. reesei QM9414在120 h达到滤纸酶活最高点。3个重组菌的滤纸酶活最高点平均是T. reesei QM9414的酶活最高点的2.3倍,而T. reesei-cre4132更是达到了2.9倍。这说明同时沉默cre1、cre2、cre3和cre4基因的表达,能够明显上调里氏木霉的纤维素酶的表达,而在构建多靶向载体时,构建的系统可能对干扰的程度有影响,从RT-qPCR测定的结果分析,siRNAs对4个基因的沉默程度也不一致,这可能是导致不同重组菌之间出现了表达差异。
 |
| 图 6 重组菌在产酶培养基中分泌的滤纸酶活(FPA)和CMC酶活(CMCA)的时间过程 Figure 6 Time course of FPUase and CMCase produced by recombinant strains T. reesei-cres, T. reesei-cre4132, T. reesei-cre1423 and T. reesei QM9414 in induction medium. Results are expressed as mean±standard deviation. |
| 图选项 |
CMC酶活(CMCA)是在纤维素酶组分中的内切β-1, 4-葡聚糖苷酶作用下水解CMC-Na产生纤维寡糖、纤维二糖、葡萄糖等还原糖的糖化力,它主要代表内切β-1, 4-葡聚糖苷酶的活力。重组菌在产酶培养基中产生CMCA的测定曲线见图 6-B。与滤纸酶活性的测定的结果类似,所有菌株的CMC酶活力曲线在120 h内都呈现一个较大的上升趋势,其中T. reesei-cre1423和T. reesei QM9414在120 h达到了CMC酶活性的最高点,而T. reesei-cre4132和T. reesei-cres在144 h达到酶活曲线最高水平。3个重组菌的酶活性最高水平都比原始出发菌T. reesei QM9414平均高出1.9倍。证明重组菌的内切β-1, 4-葡聚糖苷酶活性比原始出发菌T. reesei QM9414明显提高。
2.4 RT-qPCR分析干扰重组菌中cre1,cre2,cre3和cre4基因的相对表达量 通过酶活测试实验发现,滤纸酶活和CMC酶活都是在120 h时达到酶活性的最高水平,因此在48 h和120 h分别收集菌液进行后续实验。为了进一步从基因转录水平上验证多靶向载体同时通过siRNA干扰里氏木霉碳代谢阻遏物cre1、cre2、cre3和cre4基因对里氏木霉产纤维素酶的影响,分别在48 h和120 h时测定干扰重组菌株T. reesei-cres、T. reesei-cre4132、T. reesei-cre1423和T. reesei QM9414的cre1、cre2、cre3和cre4基因的表达水平。对所有样品进行实时荧光定量PCR反应,使用qPCR Software 3.2软件对实验结果进行处理分析。实验数据经内参基因sar1定量值校正后,以出发菌株T. reesei QM9414的基因表达量为1,分析其他基因的相对表达量,结果见图 7。
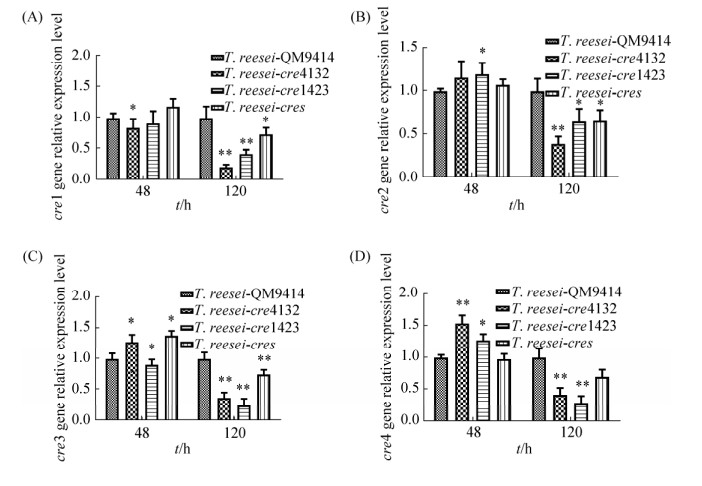 |
| 图 7 干扰重组菌分别诱导培养48 h和120 h后cre1、cre2、cre3、cre4的相对表达量 Figure 7 The relative expression level of cre1、cre2、cre3、cre4 in silencing recombinant strains after respectively inducing 48 h and 120 h. |
| 图选项 |
结果显示,重组菌T. reesei-cres、T. reesei-cre4132和T. reesei-cre1423在培养48 h时cre1、cre2、cre3和cre4的基因表达量与出发菌株T. reesei QM9414的表达水平没有明显差异,甚至表达量有超过出发菌株T. reesei QM9414的趋势,这与我们以往的研究结果是一致的,分析其可能原因是转化过程或在真菌体内表达siRNA对其刺激所产生的一种应激反应;但是在转化后培养1-2 d这种应激反应消失。在培养120 h时测定碳代谢阻遏物cre1、cre2、cre3和cre4基因表达水平均明显低于出发菌株T. reesei QM9414。且基因的下降情况与酶活性的测定结果是对应的。T. reesei-cre4132和T. reesei-cre1423中cre1、cre2、cre3和cre4的基因下调情况均比T. reesei-cres和T. reesei QM9414明显。在cre1基因相对表达量测定的实验中,48 h时T. reesei QM9414、T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres菌中的cre1相对表达量分别为1.00、0.84、0.91和1.18,而120 h后测定的cre1相对表达量分别为1.00、0.20、0.41和0.73。重组菌株T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres的cre1的相对表达量比出发菌株T. reesei QM9414分别下降了0.80、0.59和0.27,平均下降0.55。而在测定cre2、cre3和cre4基因的相对基因表达量中,重组菌株T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres比出发菌株T. reesei QM9414分别平均下降了0.44、0.54和0.79。其中以上实验结果说明,重组菌株T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres的cre1、cre2、cre3和cre4基因均同时受到了不同程度的沉默,多靶向载体在里氏木霉中可以成功转录出有效的siRNA并在菌体内加工形成有效的干扰片段,沉默目的基因的表达,从而起到下调目的基因的重要作用。
2.5 RT-qPCR分析干扰重组菌中ace1基因的表达量 分别使用ace1-F/ace1-R (表 3)对诱导培养48 h和120 h的重组菌及对照菌株进行荧光定量PCR分析,结果见图 8。实验结果表明,在各种干扰重组菌中ace1基因的表达水平均未见明显变化。说明沉默碳代谢阻遏物基因cre1、cre2、cre3和cre4,对木聚糖酶的一个阻遏因子ace1基因的表达调控不受影响,提示沉默碳代谢阻遏物基因cre1、cre2、cre3和cre4对里氏木霉的其他调控因子可能不会造成影响。
 |
| 图 8 干扰重组菌分别诱导48 h和120 h后ace1的相对表达量 Figure 8 Ace1 relative expression level in interfering recombinant strains after 48 h and 120 h, respectively. |
| 图选项 |
2.6 RT-qPCR分析干扰重组菌中cbh1、egl1和xyr1基因的相对表达量 天然纤维素降解酶系是起协同作用的多组分酶系,不仅具有内切葡聚糖酶和外切葡聚糖酶活性,还有很高的木聚糖酶活性。cbh1是纤维二糖水解酶CBH1的编码基因,egl1内切β-1, 4-葡聚糖酶EGL1的编码基因[21]。据文献报道,XYR1是主要纤维素酶和半纤维素酶基因包括cbh1、cbh2、egl1、bgl1、xyn1、xyn2等转录所必需的转录激活因子[22]。研究表明,xyr1的转录受到cre1和ace1的调控[9]。为了分析重组菌株T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres中同时沉默cre1、cre2、cre3和cre4基因是否会对纤维素酶基因chb1、egl1和木聚糖酶转录激活因子xyr1基因的表达产生影响,使用荧光定量PCR分别分析了干扰重组菌中纤维素酶基因cbh1、egl1和编码的转录激活因子xyr1的相对表达量,结果见图 9。
 |
| 图 9 干扰重组菌分别诱导培养48 h和120 h后cbh1、egl1、xyr1的相对表达量 Figure 9 The relative expression level of cbh1, egl1 and xyr1 in silencing recombinant strains after respectively inducing 48 h and 120 h. |
| 图选项 |
实验结果显示,在诱导培养48 h时,重组菌T. reesei QM9414、T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres中cbh1的相对表达量分别是1.00、1.98、0.97和1.12,而诱导培养120 h测得的结果分别是1、4.68、6.7和1.32。可见在诱导培养120 h时,T. reesei-cre4132和T. reesei-cre1423的cbh1基因表达量比诱导培养48 h得到了显著的提高。说明同时抑制碳代谢阻遏物基因cre1、cre2、cre3和cre4可调控cbh1的表达量,使其明显升高。
egl1编码的是内切β-1, 4-葡聚糖酶。在48 h时,重组菌T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres的egl1相对表达量与原始菌株T. reesei QM9414相差无几。而120 h时,重组菌T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres的egl1相对表达量分别为原始菌株T. reesei QM9414的4.80倍、4.59倍和2.56倍,证明重组菌株的egl基因表达量均得到了提升。同时抑制cre1、cre2、cre3和cre4基因可以解除内切β-1, 4-葡聚糖酶的由于葡萄糖效应而受到的抑制,说明cre1、cre2、cre3和cre4基因对egl1表达的调控有十分重要的影响。
xyr1是激活调控因子,它可以激活纤维素酶相关基因的表达,提高纤维素酶的产量和活性。同时xyr1也受到碳阻遏抑制因子的影响[23]。在48 h时,重组菌和原始出发菌的xyr1相对表达量无明显差异,而120 h时,重组菌T. reesei-cre4132、T. reesei-cre1423和T. reesei-cres的xyr1相对表达量为原始出发菌T. reesei QM9414的2.99、3.48和1.88倍,说明多靶向同时干扰4个基因的表达可以明显提高xyr1的表达。
3 讨论 里氏木霉在工业生产纤维素酶中扮演着重要的角色,但是生产纤维素酶能力仍然受其本身的生长代谢影响,CRE1、CRE2、CRE3和CRE4是里氏木霉碳阻遏效应中的阻遏物,与构巢曲霉具有高度同源性[8]。当培养基中存在葡萄糖等可快速利用的碳源时,里氏木霉会优先利用这些碳源,而像纤维素这样的复杂碳源则利用率低下,从而影响了里氏木霉产纤维素酶的能力。通过本实验室此前的研究,已经证明了单独抑制里氏木霉的cre1、cre2、cre3和cre4基因,可以有效降低里氏木霉的葡萄糖效应,使其纤维素酶表达量得到一定程度的提高[4, 15-17]。但由于4个基因的表达产物中可能会存在相互作用[9],只抑制其中的一个基因难以实现完全解除碳阻遏效应,所以本研究通过构建的多靶向siRNA系统,将cre1、cre2、cre3和cre4基因同时抑制,以期能够最大限度地解除其碳代谢阻遏效应,进一步提高纤维素酶的产量。
本研究证明,A和B两种系统中,A系统对目的基因有一定程度的抑制沉默作用,但其作用效果没有B系统好。主要原因可能与转录出来的RNA分子构型有重要的关系,因为转录后形成多个颈环结构并且通过线性区域连接,因此双链区域可能由于RNA分子存在线性区域,容易形成扭转或重叠,使Dicer蛋白酶体对折叠的RNA分子可能无法充分结合和加工,因此产生的siRNA会减少,对目的基因的沉默效果就会降低(图 10-A);而B系统由于转录出来的线性RNA通过互补序列形成双链RNA分子,其刚性强于含有线性区域的A系统的构型,双链RNA不容易形成折叠(图 10-B),Dicer蛋白酶体对B型的加工更容易,因此产生的siRNA就更充分,沉默效果就更好。
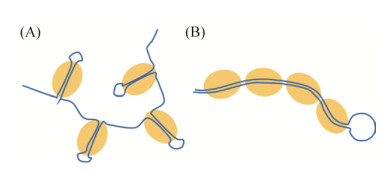 |
| 图 10 Dicer结合RNA分子模式图 Figure 10 Pattern of Dicer binding RNA molecule. |
| 图选项 |
在滤纸酶活和CMC酶活中,A、B两种系统的所有重组菌均检测到了比原始菌株QM9414更高的酶活力,但B系统的重组菌T. reesei-cre4132和T. reesei-cre1423表现出了比A系统中的T. reesei-cres更高的酶活力,这与2个系统对4个阻遏物基因的干扰结果是一致的。
而对ace1基因的表达量检测中,无论是A系统中的T. reesei-cres还是B系统的重组菌T. reesei-cre4132、T. reesei-cre1423中,ace1相对表达量与原始出发菌T. reesei QM9414无明显差异,说明cre1、cre2、cre3和cre4基因的沉默不会影响ace1基因的表达,纤维素酶活性的提高也可能不受ace1基因表达的调控。
根据高云雨等[4]对cre1干扰的效果分析,我们对单基因靶向抑制和多基因靶向抑制的CMC酶活、滤纸酶活以及纤维素酶基因cbh1、egl1和木聚糖酶基因激活因子基因xyr1表达进行了比较。结果发现,当只对cre1基因进行抑制的时候,CMC酶活和滤纸酶活比原始出发菌QM9414分别上升1.7和2.0倍,而多靶向抑制的时候,T. reesei-cre1423、T. reesei-cre4132和T. reesei-cres的CMC酶活和滤纸酶活比原始出发菌QM9414平均上升1.95和2.66倍,其中T. reesei-cre4132更是上升了2.13和2.99倍,是只对cre1基因进行抑制的1.25和1.50倍。
而纤维素酶相关基因cbh1、egl1和木聚糖酶激活因子基因xyr1的相对表达量中,只对cre1基因进行抑制比原始出发菌QM9414分别上升了2.50、2.90和1.98倍,而多靶向抑制菌株中T. reesei-cre1423和T. reesei-cre4132比原始出发菌QM9414分别上升了4.69、5.67、4.45倍和4.80、2.99、3.48倍,可见多靶向抑制cre1、cre2、cre3和cre4基因的表达比单向抑制任何一个基因的表达得到了更好的纤维素酶产量。
目前研究表明在丝状真菌中参与碳代谢阻遏物调控作用的不仅仅是碳代谢阻遏物基因的表达产物,还涉及cAMP的参与和泛素化途径参与的碳代谢阻遏作用[9, 24-25],可见其调控机制的复杂性。本项实验的设计思路和结果为进一步研究里氏木霉基因表达调控提供了新的技术手段和实验数据。
References
| [1] | Mach RL, Strauss J, Zeilinger S, Schindler M, Kubicek CP. Carbon catabolite repression of xylanase Ⅰ (xyn1) gene expression in Trichoderma reesei. Molecular Microbiology, 1996, 21(6): 1273-1281. DOI:10.1046/j.1365-2958.1996.00094.x |
| [2] | Joutsjoki VV, Kuittinen M, Torkkeli TK, Suominen PL. Secretion of the Hormoconis resinae glucoamylase p enzyme from Trichoderma reesei directed by the natural and the cbh1 gene secretion signal. FEMS Microbiology Letters, 1993, 112(3): 281-286. DOI:10.1111/fml.1993.112.issue-3 |
| [3] | Zhang GT, Hartl L, Schuster A, Polak S, Schmoll M, Wang TH, Seidl V, Seiboth B. Gene targeting in a nonhomologous end joining deficient Hypocrea jecorina. Journal of Biotechnology, 2009, 139(2): 146-151. DOI:10.1016/j.jbiotec.2008.10.007 |
| [4] | Gao YY, Zhong LY, Dong GY, She WY, Zhou JJ, Liu SY, Tian SL. Trichoderma reseei constitutive type of expression vector siRNA interfering cre1 gene on cellulases expression regulation. Journal of Microbiology, 2018, 38(1): 12-19. (in Chinese) 高云雨, 钟路遥, 董冠园, 佘伟怡, 周娇娇, 刘思远, 田生礼. 里氏木霉组成型表达sirna干扰cre1基因对纤维素酶表达的调控作用. 微生物学杂志, 2018, 38(1): 12-19. DOI:10.3969/j.issn.1005-7021.2018.01.002 |
| [5] | Foreman PK, Brown D, Dankmeyer L, Dean R, Diener S, Dunn-Coleman NS, Goedegebuur F, Houfek TD, England GJ, Kelley AS, Meerman HJ, Mitchell T, Mitchinson C, Olivares HA, Teunissen PJM, Yao J, Ward M. Transcriptional regulation of biomass-degrading enzymes in the filamentous fungus Trichoderma reesei. Journal of Biological Chemistry, 2003, 278(34): 31988-31997. DOI:10.1074/jbc.M304750200 |
| [6] | Kumar R, Singh S, Singh OV. Bioconversion of lignocellulosic biomass: Biochemical and molecular perspectives. Journal of Industrial Microbiology & Biotechnology, 2008, 35(5): 377-391. |
| [7] | Boase NA, Kelly JM. A role for creD, a carbon catabolite repression gene from Aspergillus nidulans, in ubiquitination. Molecular Microbiology, 2004, 53(3): 929-940. DOI:10.1111/j.1365-2958.2004.04172.x |
| [8] | Kubicek CP, Mikus M, Schuster A, Schmoll M, Seiboth B. Metabolic engineering strategies for the improvement of cellulase production by hypocrea jecorina. Biotechnology for Biofuels, 2009, 2: 19. DOI:10.1186/1754-6834-2-19 |
| [9] | Lockington RA, Kelly JM. The WD40-repeat protein creC interacts with and stabilizes the deubiquitinating enzyme creB in vivo in Aspergillus nidulans. Molecular Microbiology, 2002, 43(5): 1173-1182. DOI:10.1046/j.1365-2958.2002.02811.x |
| [10] | Denton JA, Kelly JM. Disruption of trichoderma reesei cre2, encoding an ubiquitin C-terminal hydrolase, results in increased cellulase activity. BMC Biotechnology, 2011, 11: 103. DOI:10.1186/1472-6750-11-103 |
| [11] | Saloheimo A, Aro N, Ilmén M, Penttil? M. Isolation of the ace1 gene encoding a cys2-his2 transcription factor involved in regulation of activity of the cellulase promoter cbh1 of Trichoderma reesei. Journal of Biological Chemistry, 2000, 275(8): 5817-5825. DOI:10.1074/jbc.275.8.5817 |
| [12] | Aro N, Ilmén M, Saloheimo A, Penttil? M. Acei of Trichoderma reesei is a repressor of cellulase and xylanase expression. Applied and Environmental Microbiology, 2003, 69(1): 56-65. DOI:10.1128/AEM.69.1.56-65.2003 |
| [13] | Qin LN, Cai FR, Dong XR, Huang ZB, Tao Y, Huang JZ, Dong ZY. Improved production of heterologous lipase in Trichoderma reesei by RNAi mediated gene silencing of an endogenic highly expressed gene. Bioresource Technology, 2012, 109: 116-122. DOI:10.1016/j.biortech.2012.01.013 |
| [14] | He RL, Guo W, Wang LX, Zhang DY. Construction of an efficient rnai system in the cellulolytic fungus Trichoderma reesei. Journal of Microbiological Methods, 2015, 108: 70-73. DOI:10.1016/j.mimet.2014.11.010 |
| [15] | Li JX. RNA interference on cellulase expression in Trichoderma reesei catabolite repressor cre2. Master Dissertation of Shenzhen University, 2014. (in Chinese) 李洁璇. RNA干扰里氏木霉分解代谢阻遏物cre2对纤维素酶表达的研究.深圳大学硕士学位论文, 2014. |
| [16] | Liang XY. RNA interference with the catabolic repressor cre3 of Trichoderma reesei and its effect on cellulase expression. Master Dissertation of Shenzhen University, 2015. (in Chinese) 梁秀怡. RNA干扰里氏木霉分解代谢阻遏物cre3及对纤维素酶表达影响的研究.深圳大学硕士学位论文, 2015. |
| [17] | Liu WL. Cre4 gene regulation of cellulase expression in Trichoderma reesei. Master Dissertation of Shenzhen University, 2016. (in Chinese) 刘雯莉. Cre4基因在里氏木霉中对纤维素酶表达的调控研究.深圳大学硕士学位论文, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10590-1016763455.htm |
| [18] | Cong W, Cui SQ, Chen JL, Zuo XP, Lu YG, Yan WY, Zheng ZX. Construction of a multiple targeting RNAi plasmid that inhibits target gene expression and FMDV replication in BHK-21 cells and suckling mice. Veterinary Research Communications, 2010, 34(4): 335-346. DOI:10.1007/s11259-010-9360-y |
| [19] | Penttil? M, Nevalainen H, R?tt? M, Salminen E, Knowles J. A versatile transformation system for the cellulolytic filamentous fungus Trichoderma reesei. Gene, 1987, 61(2): 155-164. DOI:10.1016/0378-1119(87)90110-7 |
| [20] | Ghose TK, Bisaria VS. Measurement of hemicellulase activities: Part Ⅰ xylanases. Pure and Applied Chemistry, 1987, 59(12): 1739-1751. DOI:10.1351/pac198759121739 |
| [21] | Aro N, Saloheimo A, Ilmeé M, Penttil? M. Aceii, a novel transcriptional activator involved in regulation of cellulase and xylanase genes of Trichoderma reesei. Journal of Biological Chemistry, 2001, 276(26): 24309-24314. DOI:10.1074/jbc.M003624200 |
| [22] | Stricker AR, Mach RL, de Graaff LH. Regulation of transcription of cellulases- and hemicellulases-encoding genes in Aspergillus niger and Hypocrea jecorina (Trichoderma reesei). Applied Microbiology and Biotechnology, 2008, 78(2): 211-220. |
| [23] | Silva-Rocha R, Castro LDS, Antoniêto ACC, Guazzaroni ME, Persinoti GF, Silva RN. Deciphering the cis-regulatory elements for xyr1 and cre1 regulators in Trichoderma reesei. PLoS ONE, 2014, 9(6): e99366. DOI:10.1371/journal.pone.0099366 |
| [24] | Lin CT, Chen YC, Jinn TR, Wu CC, Hong YM, Wu WH. Role of the camp-dependent carbon catabolite repression in capsular polysaccharide biosynthesis in Klebsiella pneumoniae. PLoS ONE, 2013, 8(2): e54430. DOI:10.1371/journal.pone.0054430 |
| [25] | Borirak O, Bekker M, Hellingwerf KJ. Molecular physiology of the dynamic regulation of carbon catabolite repression in Escherichia coli. Microbiology, 2014, 160(6): 1214-1223. |
