段晓雷1,2#, 刘娜女2#, 张涛1, 曹睿3, 姜钧耀1, 闵迅1

 , 奚绪光2
, 奚绪光2

1.遵义医学院附属医院检验科, 贵州 遵义 563000;
2.西北农林科技大学生命科学学院, 陕西 杨凌 712100;
3.河南科技大学动物科技学院, 河南 洛阳 471003
收稿日期:2018-05-11;修回日期:2018-07-13;网络出版日期:2018-08-17
基金项目:国家自然科学基金(31660241);贵州省科技合作基础研究项目(黔科合基础[2018]1196);遵义医学院博士科研启动资金[院字(2017)05];遵义医学院大学生创新创业训练计划(ZKDC2018005)
*通信作者:闵迅, Tel/Fax:+86-851-28642550, E-mail:2815400619@qq.com
奚绪光, Tel/Fax:+86-29-87081664, E-mail:xixuguang@nwsuaf.edu.cn
#并列第一作者
摘要:[目的] 原核表达与纯化源自嗜热细菌Anaerobaculum hydrogeniforman的Pif1解旋酶,从而探究其解旋反应特性。[方法] 将重组载体pET21a-AnaPif1-TEV/SUMO导入大肠杆菌BL21(DE3)中诱导表达,并进行Ni-NTA亲和层析、Superdex200凝胶过滤层析等一系列纯化手段;再利用快速停留监测技术系统地研究Ana.Pif1的解旋反应特性,包括解旋极性、最佳ATP浓度、最佳金属辅因子、最佳解旋温度以及解旋复制中间体DNA的底物特异性。[结果] 通过异源表达与纯化,获得了无任何标签序列、分子量59 kDa的Ana.Pif1蛋白,纯度达97%,产率为9.5 mg/L。本研究首先验证Ana.Pif1具有5'-3'的解旋极性,并发现其解旋反应最佳ATP浓度为2 mmol/L,其最佳二价金属辅因子为Mg2+,最适反应温度为55℃。解旋复制中间体的底物特异性显示,Y-S型复制叉的解旋速率最高,为0.127 s–1;而12 nt-bubble底物的解旋幅度最大,达到78.8%;暗示这些底物可能是Ana.Pif1的天然底物。[结论] 本文首次较为系统地分析了Ana.Pif1解旋酶的解旋反应特性,为阐明此类嗜热细菌Pif1解旋酶的分子作用机制奠定了基础。
关键词:Pif1解旋酶嗜热厌氧棒菌快速停留检测解旋特征参数
Heterologous expression and characterization of unwinding reaction of Pif1 helicase from Anaerobaculum hydrogeniforman
Xiaolei Duan1,2#, Nanü Liu2#, Tao Zhang1, Rui Cao3, Junyao Jiang1, Xun Min1

 , Xuguang Xi2
, Xuguang Xi2

1.Clinical Laboratory, Affiliated Hospital of Zunyi Medical University, Zunyi 563000, Guizhou Province, China;
2.College of Life Sciences, Northwest A & F University, Yangling 712100, Shaanxi Province, China;
3.College of Animal Science & Technology, Henan University of Science and Technology, Luoyang 471003, Henan Province, China
Received 11 May 2018; Revised 13 July 2018; Published online 17 August 2018
*Corresponding author: Xun Min, Tel/Fax:+86-851-28642550, E-mail: 2815400619@qq.com
Xuguang Xi, Tel/Fax:+86-29-87081664, E-mail: xixuguang@nwsuaf.edu.cn
Supported by the National Natural Science Foundation of China (31660241), by the Science & Technology Fund Program for Basic Research of Guizhou Province (Guizhou foundation[2018]1196), by the Doctoral Research Start up Fund of Zunyi Medical University [ZMU(2017)05] and by the Undergraduate Innovation and Entrepreneurship Training Program of Zunyi Medical University (ZKDC2018005)
#These authors contributed equally to this work
Abstract: [Objective] We expressed and purified a Pif1 helicase from Anaerobaculum hydrogeniforman to assess its unwinding characteristics. [Methods] Recombinant vector pET21a-Ana.Pif1-TEV/SUMO was constructed and expressed in E. coli BL21(DE3), and Ana.Pif1 protein was purified using Ni-NTAs and Superdex 200 column chromatography. We then evaluated the unwinding polarity, optimal ATP concentration, optimal metal cofactor, optimal unwinding temperature, and substrate specificity of Ana.Pif1 by stopped-flow FRET assay. [Results] The molecular weight of Ana.Pif1 helicase was 59 kDa with no tags, protein yield was 9.5 mg/L, and purity was about 97%. We confirmed the unwinding polarity of Ana.Pif1 was from DNA 5' to 3' end. And we found the optimal ATP concentration for Ana.Pif1 unwinding to be 2 mmol/L, the optimal metal cofactor to be Mg2+, and the optimal reaction temperature to be 55℃. The substrate specificity of Ana.Pif1 unwinding DNA replication intermediates indicated that the Y-S fork substrate had the highest unwinding rate, 0.127 s-1, and 12 nt-bubble substrate had the largest unwinding amplitude, 78.8%, indicating these two substrates could be natural replication intermediates for Ana.Pif1. [Conclusion] This study is the first to systematically analyze the unwinding characteristics of Ana.Pif1 helicase. These findings facilitate elucidation of the molecular mechanisms of these thermophilic bacteria Pif1-family helicases.
Keywords: Pif1 helicasesAnaerobaculum hydrogeniformanstopped-flow assayunwinding characteristic parameters
DNA作为生命体最重要的遗传物质,其主要以DNA双螺旋构型等高级结构存在;当DNA在复制与转录过程中需要特异调控时,首先需要解链DNA高级结构,这促使生物体进化出许多特异的解旋酶来进行DNA解旋反应[1-2]。其中,Pif1解旋酶是一类广泛存在且高度保守的解旋蛋白[3];该解旋酶家族成员都具有7个保守motif组成的特异Pif1结构域,并沿DNA底物5′-3′方向进行解旋反应[4]。近年来,Pif1解旋酶的研究主要集中于酵母Pif1(Sc.Pif1)与人类Pif1(hPif1)的生理生化特征与遗传学等方面,揭示出该家族解旋酶存在着共同的生物学功能:参与核内DNA的断裂修复[5]、有效解旋DNA-RNA杂交链[6]、维持线粒体DNA稳定性[7]、高效识别与解旋G4 DNA[8-9]以及负调控端粒酶活性等[10]。
然而,随着对不同来源Pif1解旋酶进行更加广泛与系统的分析,科学家还发现此类解旋酶在不同物种中往往还具有特异的生物学活性[11-12]。这不仅涉及了裂殖酵母Pif1(Pfh1)、小鼠Pif1(mPif1)等真核生物Pif1解旋酶:遗传与体外实验表明Pfh1具有复制蛋白A(RPA)依赖作用促进裂殖酵母端粒复制与延伸[13]以及推进复制叉迁移等活性[14],mPif1解旋酶失活可引起小鼠线粒体肌病等[15];而且,最近的研究已经拓展到原核Pif1解旋酶:拟杆菌Pif1(Bs.Pif1)的酶学特性与动力学分析揭示出其不同于Sc.Pif1的解旋特征[16],并作为首个Pif1蛋白的晶体结构得到解析[17],白色念球菌Pif1(Ca.Pif1)的研究则发现其具有特殊3′-5′核酸外切酶活性等[18]。但是,到目前为止,国内外有关嗜热细菌类Pif1解旋酶的研究却鲜有报道[19-20],尤其是其相关解旋反应特性的研究更是屈指可数。
本研究针对嗜热细菌中的厌氧棒菌(Anaerobaculum hydrogeniformans) Pif1解旋酶(Ana.Pif1),通过异源表达纯化而获取纯度高、活性好的Ana.Pif1蛋白,进而系统地对其解旋反应的分子特性进行分析,以期拓展人们对嗜热微生物Pif1解旋酶的认知,为阐明此类Pif1解旋酶的功能和作用机制奠定基础。
1 材料和方法 1.1 材料
1.1.1 质粒、引物及菌株: Ana.Pif1解旋酶的全长编码序列由Biomatik公司合成;PCR引物由生工生物工程(上海)股份有限公司合成,F:5′-CCCA TATGTCTAAAATCGAACTG-3′,R:5′-CGCGAAT TCGTCCAGAGAGTGCAT-3′。pET21a-TEV/SUMO载体、E. coli BL21(DE3)表达菌株等均为本课题组保存。
1.1.2 DNA底物: 快速停留-荧光能量共振转移(stopped-flow FRET)检测解旋反应特性所用的DNA底物双链分别标记荧光素(fluorescein,F)和六氯荧光素(hexachlorofluorescein,HF),由生工生物工程(上海)股份有限公司合成与标记,其序列信息见表 1。所有反应底物混合于缓冲液(20 mmol/L Tris-HCl (pH 7.5),100 mmol/L KCl)进行退火:95 ℃水浴5 min后冷却至室温,–20 ℃备用。
表 1. 基于快速停留-荧光能量共振转移检测技术分析Ana.Pif1解旋反应的DNA底物 Table 1. DNA substrates used in Ana.Pif1-catalyzed unwinding reactions by stopped-flow FRET assay
| Abbr.(Name) | Structure | Sequence |
| 5′-OhS12D20 (5′-12 nt-20 bp) | 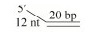 | CACTGGCCGTCTTACGGTCGCTCTGCTCGACG-F HF-CGTCGAGCAGAGCGACCGTA |
| 3′-OhS12D20 (3′-12 nt-20 bp) | 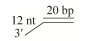 | CACTGGCCGTCTTACGGTCGCTCTGCTCGACG-F HF-CGACCGTAAGACGGCCAGTG |
| 5′-OhS26D17 (5′-26 nt-17 bp) | 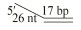 | (dT26)ATGTATGTCAAGGAAGG-F HF-CCTTCCTTGACATACAT |
| Y-S (Y-structure) | 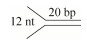 | HF-CGTCGAGCAGAGCGACCGTATTATTTTTTTTT CTCTGGCCGTCTTACGGTCGCTCTGCTCGACG-F |
| 5′-Fork (5′-Flap dsDNA) | 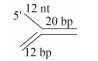 | HF-CGTCGAGCAGAGCGACCGTATTATTTTTTTTT CACTGGCCGTCTTACGGTCGCTCTGCTCGACG-F AGACGGCCAGTG |
| 3′-Fork (3′-Flap dsDNA) | 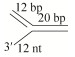 | AAAAAAAAATAA HF-CGTCGAGCAGAGCGACCGTATTATTTTTTTTT CACTGGCCGTCTTACGGTCGCTCTGCTCGACG-F |
| TWJ (Three -Way-Junction) | 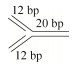 | AAAAAAAAATAA HF-CGTCGAGCAGAGCGACCGTATTATTTTTTTTT CTCTGGCCGTCTTACGGTCGCTCTGCTCGACG-F AGACGGCCAGTG |
| BS4 (4 nt-bubble) | 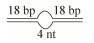 | HF-GCAGTAGGCCTAGCATGAGTTAGGACTGACAGCTGCATGG CCATGCAGCTGTCAGTCCATTGTCATGCTAGGCCTACTGC-F |
| BS12 (12 nt-bubble) | 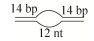 | HF-GCAGTAGGCCTAGCTACTGTTACCTGTGACAGCTGCATGG CCATGCAGCTGTCAGTCCATTGTCATGCTAGGCCTACTGC-F |
| HJ (Holiday Junction) |  | ACGTGGGCAAAGGTTCGTCAATGGACTGACAGCTGCATGG CCGTGATCACCAATGCACATTGACGAACCTTTGCCCACGT CCATGCAGCTGTCAGTCCATTGTCATGCTAGGCCTACTGC-F HF-GCAGTAGGCCTAGCATGACAATCTGCATTGGTGATCACGG |
| F, Fluorescein; HF, Hexachlorofluorescein. | ||
表选项
1.1.3 主要试剂及工具酶: PrimeSTAR HS DNA Polymerase与T4 DNA ligase,以及Nde I、EcoR I、Xho I限制性内切酶等均购自宝生物工程(大连)公司。TEV酶为本实验室自行制备。Ni-NTA亲和层析、Superdex 200(S200)凝胶过滤层析柱等购自GE公司。
1.2 Ana.Pif1解旋酶的表达载体构建 将合成Ana.Pif1解旋酶完整的编码序列(GenBank登录号:CP003198.1)和pET21a-TEV/ SUMO表达载体通过Nde I/EcoR I双酶切后用T4连接酶连接,获得pET21a-Ana.Pif1-TEV/SUMO重组质粒。
1.3 Ana.Pif1蛋白的原核表达与纯化 将构建成功的pET21a-Ana.Pif1-TEV/SUMO重组质粒转化到大肠杆菌的表达菌株E. coli BL21(DE3)中;当OD=0.6时,在1 L的LB培养基中加入1 mol/L的IPTG为400 μL (即诱导时IPTG工作浓度为0.4 mmol/L),进而在低温(22 ℃)下诱导14 h表达重组蛋白。按照改进的文献[19]a方法进行分离纯化:第一次Ni-NTA纯化后使用TEV酶4 ℃酶切6 h,随后再进行第二次Ni-NTA纯化以去除C-端的SUMO与His标签序列;并在最后纯化步骤中增加Supdex 200凝胶过滤层析。纯化产物由10% (W/V) SDS-PAGE分析,并使用Quantity one分析软件计算出蛋白条带的纯度;由BCA法测定蛋白浓度。加甘油(终浓度25%)后超滤浓缩,–80 ℃冻存备用。
1.4 基于Stopped-flow FRET技术检测Ana.Pif1解旋反应 利用快速停留-荧光共振能量转移(stopped-flow-FRET)监测技术[16, 22],参照本实验室先前摸索的Ana.Pif1最佳解旋条件[19](20 mmol/L Tris-HCl (pH 7.0),20 mmol/L NaCl,2 mmol/L MgCl2,1 mmol/L DTT),固定Ana.Pif1解旋酶与反应底物的工作浓度分别为100 nmol/L与4 nmol/L,研究Ana.Pif1解旋反应以下特性:(1)解旋极性实验,底物分别为5′-12 nt-20 bp与3′-12 nt-20 bp;(2)在不同ATP浓度(0–5 mmol/L)下,测试Ana.Pif1解旋相同浓度5′-OhS26D17底物的反应速率变化;(3)不同二价金属阳离子(Mg2+、Mn2+、Ca2+、Cu2+、Ni2+及Zn2+)作为辅酶因子(浓度均为2 mmol/L)时,研究Ana.Pif1解旋5′-OhS26D17底物的特征参数变化;(4)测试不同温度对Ana.Pif1蛋白解旋反应的影响:DNA底物为5′-OhS26D17,反应温度从20 ℃到60 ℃逐渐升高,间隔5 ℃ (仅37 ℃代替35 ℃)检测解旋特征曲线,使用恒温水浴控制温度;(5) Ana.Pif1与多种模拟复制中间体底物发生解旋反应的特征分析:对比Y-S型Fork底物、5′-Fork底物、3′-Fork底物和TWJ底物,与模拟复制泡底物(4 nt-bubble底物与12 nt-bubble底物)以及十字交叉(HJ)底物的解旋速率与解旋幅度,明确其解旋反应最适DNA底物。以上Ana.Pif1解旋反应均在多转换条件下(不加蛋白Trap)进行;其解旋特征曲线为至少10次独立试验的平均值,并使用Bio-Kine (version 4.26;Bio-Logic)软件中的双指数函数(double-exponential functions)公式(1,2)进行拟合[22-23]。
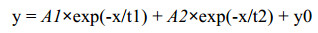 | 公式(1) |
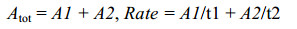 | 公式(2) |
1.5 统计学分析 本研究的解旋反应测试中各实验均至少重复3次以上,取平均值;并采用SPSS.16统计软件,数据采用单因素方差分析;P < 0.05为具有显著统计学意义。
2 结果和分析 2.1 Ana.Pif1表达载体的构建 成功构建pET21a-Ana.Pif1-TEV/SUMO表达载体(质粒图谱见图 1-A):其表达产物C-端融合有TEV标签、SUMO标签(促溶作用)和6×His标签序列,而在Ana.Pif1序列与标签序列间设计TEV酶切位点。通过菌落PCR鉴定与重组质粒Nde I、EcoR I双酶切鉴定,结果如图 1-B所示,靶标基因序列为约1.5 kb片段,符合预期(1587 bp)。并进一步进行测序,确认无误,与Ana.Pif1基因的编码序列(GenBank:CP003198.1)一致,将正确的转化子用于下一步实验。
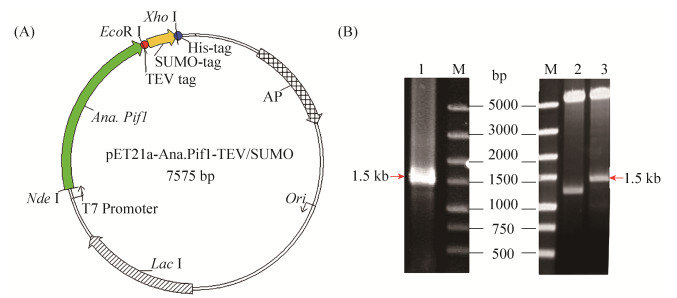 |
| 图 1 pET21a-Ana.Pif1-TEV/SUMO 表达载体的构建 Figure 1 Construction of expression vector pET21a-Ana.Pif1-TEV/SUMO. A: the schematic map of pET21a-Ana.Pif1–TEV/SUMO; B: identification of the recombinant vector by PCR and restriction enzyme digestion. Lane 1: the product of colony PCR; lane 2: the digestion of control plasmid; lane 3: the double digestion of pET21a-Ana.Pif1–TEV/SUMO by Nde I and EcoR I; M: DNA DS 5000. The arrows point out the target DNA bands in 1.5 kb. |
| 图选项 |
2.2 Ana.Pif1重组蛋白的表达与纯化 重组大肠杆菌BL21(DE3)经诱导表达后,在细胞破碎液的上清中检测到目标蛋白(Ana.Pif1)。重组蛋白经过Ni-NTA亲和层析、TEV酶切标签序列以及Superdex 200分子筛层析等一系列纯化(见图 2-A),最终产物经SDS-PAGE检测。结果如图 2-B所示,Ana.Pif1纯化产物为单一条带蛋白,其分子量与目标蛋白分子量相符(约59 kDa);经凝胶扫描分析电泳条带纯度与BCA试剂盒法测定蛋白的含量,结果显示,本实验纯化的Ana.Pif1蛋白无标签序列,纯度达到97%,其表达纯化蛋白的产率为9.5 mg/L。
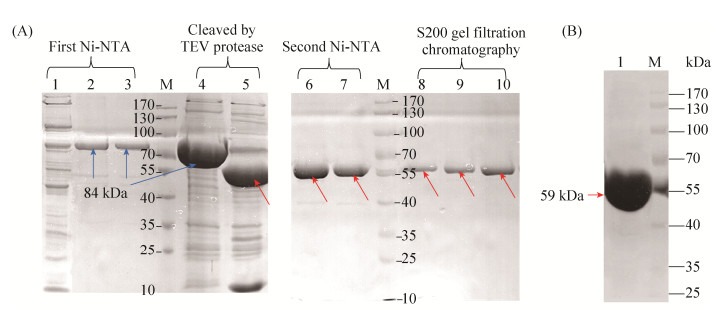 |
| 图 2 Ana.Pif1解旋酶重组蛋白的纯化 Figure 2 Purification of the recombinant Ana.Pif1 helicase was resolved in 10% (W/V) SDS-PAGE. A: The whole purification process of recombinant Ana.Pif1 protein. Lane 1 to lane 3: the first Ni-NTA purification; lane 4 and land 5: cleaved product by TEV protease; lane 6 and land 7: out flow of second round Ni-NTA after TEV protease digestion; lane 8 to lane 10: the elution peaks of Superdex 200 purification. M: restained protein marker. Arrows point out the recombinant proteins with TEV/SUMO tag in 84 kDa and the target proteins without tags in 59 kDa. B: SDS-PAGE analysis of the final purification product of Ana.Pif1. Lane 1 was the concentration product of Ana.Pif1 protein, which loaded 20 μg of protein; M: prestained protein marker. |
| 图选项 |
2.3 Ana.Pif1解旋酶的解旋极性 Ana.Pif1蛋白作为解旋酶,在解旋反应中应具有固定的解旋极性(沿DNA底物链的解旋方向性)。本研究利用Stopped-flow FRET检测技术,分别加入5′-ssDNA-dsDNA(5′-OhS12D20)以及3′-ssDNA-dsDNA(3′-OhS12D20)底物,对比监测加入ATP后解旋两者的荧光信号变化。结果显示(见图 3),Ana.Pif1可有效解旋5′-ssDNA-dsDNA而无法解旋3′-ssDNA-dsDNA底物,体现出该解旋酶具有沿底物5′-3′解旋的方向性。
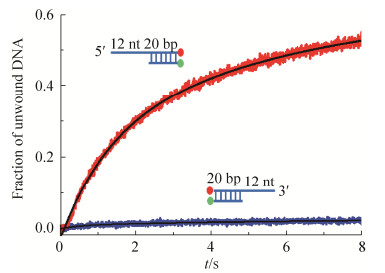 |
| 图 3 Ana.Pif1解旋酶的解旋极性 Figure 3 The polarity for Ana.Pif1-catalyzed unwinding activity. Determination of the polarity for Ana.Pif1 unwinding reactions with 5′-OhS12D20 (red line) or 3 -OhS12D20 (blue line) by stopped-flow FRET. |
| 图选项 |
2.4 ATP浓度对Ana.Pif1解旋反应的影响 Ana.Pif1解旋酶作为分子马达蛋白,其需要生物体内供能才可进行解旋反应。结合本课题组前期研究中所揭示Ana.Pif1的最佳能量供体为ATP[19],本研究进一步探讨其解旋反应的最佳ATP浓度。本研究使用相同的DNA底物与Ana.Pif1蛋白,再分别加入0、0.5、1.0、1.5、2.0、3.0及5 mmol/L的ATP启动解旋反应,对比监测相同反应底物的解旋速率与解旋幅度的差异。结果如图 4所示,只有当2 mmol/L的ATP参与反应时,Ana.Pif1解旋5′-OhS26D17底物的速率与幅度均为最高值(0.1918 s–1和72.4%),提示出Ana.Pif1解旋反应的所需最佳ATP浓度为2 mmol/L。
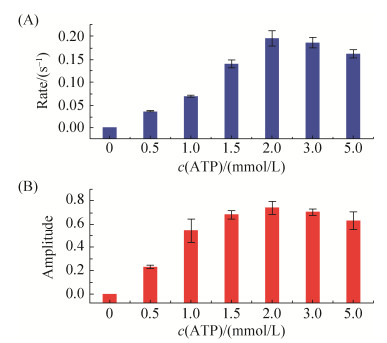 |
| 图 4 ATP浓度对Ana.Pif1解旋反应的影响 Figure 4 Effect of ATP concentration on Ana.Pif1- catalyzed unwinding activity. A: comparison of the Ana.Pif1 unwinding rates among different concentrations of ATP from 0.5 to 5.0 mmol/L; B: comparison of the Ana.Pif1 unwinding amplitudes among different concentrations of ATP as mentioned above. The average values from triplicate measurements, and standard error were calculated. |
| 图选项 |
2.5 不同金属辅离子对Ana.Pif1解旋反应的影响 解旋酶水解ATP供能可解链DNA底物,但同时还需要加入金属辅因子才能有效地启动解旋反应。本研究通过在相同解旋反应体系中添加不同金属二价阳离子,分析不同金属辅离子参与Ana.Pif1解旋5′-OhS26D17反应特征曲线的差异性。如图 5所示,当不加入任何金属阳离子时(CK),不能启动Ana.Pif1的解旋反应;当加入相同浓度(2 mmol/L)的2价阳离子时,无论是解旋幅度(79.4%)还是解旋速率(0.158 s–1),Mg2+都是Ana.Pif1解旋反应的最佳金属辅因子;Mn2+也可以较好地辅助解旋仅应,解旋幅度与解旋速率都较高(74.7%、0.142 s–1);研究中还新发现Ca2+也具有一定的辅助催化活性,而Zn2+、Ni2+与Cu2+也具有微弱地辅助催化Ana.Pif1解旋反应的效果(解旋特征参数见表 2),可能是因为这些金属阳离子能够不同程度影响解旋酶活力。
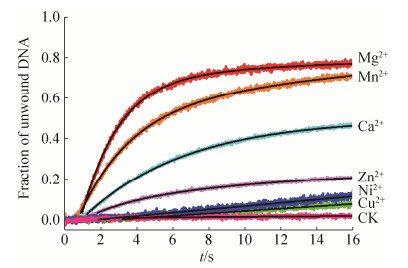 |
| 图 5 不同金属辅离子参与Ana.Pif1解旋反应的解旋特征曲线对比 Figure 5 Comparison of the characteristic curves for Ana.Pif1-catalyzed unwinding activity with different metal ions as cofactors. |
| 图选项 |
表 2. 不同金属辅因子对Ana.Pif1解旋反应的影响 Table 2. Effect of different metal cofactors on Ana.Pif1-catalyzed unwinding activity
| Metal cofactors | Rate/(s–1) | Amplitude/% |
| Mg2+ | 0.158 | 79.40 |
| Mn2+ | 0.142 | 74.70 |
| Ca2+ | 0.083 | 47.10 |
| Zn2+ | 0.034 | 21.20 |
| Ni2+ | 0.005 | 11.60 |
| Cu2+ | 0.003 | 8.10 |
| CK | 0 | 0.39 |
表选项
2.6 Ana.Pif1解旋反应的嗜热特性 由于Ana.Pif1解旋酶是源于嗜热细菌Anaerobaculum hydrogeniforman内编码蛋白,因此我们探究了不同温度对该解旋酶活性的影响。在不同温度下,Stopped-flow监测Ana.Pif1解旋相同底物5′-OhS26D17的特征曲线,通过拟合与计算,我们发现:从20 ℃至45 ℃逐步增加反应温度,Ana.Pif1解旋速率会稳步增加(解旋幅度也同时稳定增加);当反应温度由45 ℃至55 ℃继续升温,解旋速率更是增加明显;当60 ℃时,解旋速率与幅度骤然下降(解旋速率见表 3)。同时,对比测试Ana.Pif1与Bs.Pif1在不同温度下的解旋速率变化,结果如图 6所示,不同于普通细菌Pif1解旋酶(Bs.Pif1)的最佳反应温度为25 ℃,Ana.Pif1解旋酶的最佳反应温度(55 ℃)远远高于前者,这表明该解旋酶具有嗜热特性。
表 3. 温度对Ana.Pif1与Bs.Pif1解旋速率的影响 Table 3. Effect of temperature on the unwinding rates (s–1) of Ana.Pif1- and Bs.Pif1-catalyzed
| T/℃ | Ana.Pif1 | Bs.Pif1 |
| 20 | 0.046 | 0.770 |
| 25 | 0.060 | 0.810 |
| 30 | 0.070 | 0.690 |
| 37 | 0.080 | 0.220 |
| 40 | 0.092 | 0.002 |
| 45 | 0.100 | 0 |
| 50 | 0.140 | 0 |
| 55 | 0.157 | 0 |
| 60 | 0.032 | 0 |
表选项
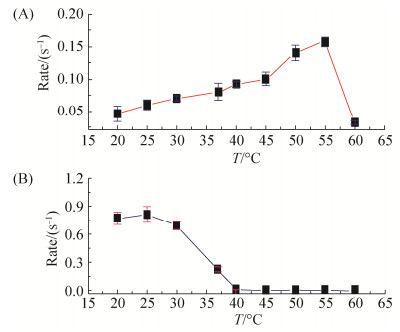 |
| 图 6 Ana.Pif1解旋反应的最适催化温度 Figure 6 Optimum temperature of Ana.Pif1 (A) and Bs.Pif1 (B)-catalyzed unwinding activity. Data presented are the average values from triplicate measurements. |
| 图选项 |
2.7 Ana.Pif1解旋不同复制中间体的底物特异性 本课题组前期的研究曾揭示出Ana.Pif1蛋白可以有效解旋5′-ss-dsDNA与5′-ss-G4-dsDNA底物[19];但是作为Pif1解旋酶,其在生物体内最重要的生物学活性应该是参与复制叉或复制泡底物的解旋过程[12, 24],因此有必要进一步探索Ana.Pif1解旋多种模拟复制中间体结构DNA的底物特异性。结果如图 7-A所示,研究发现Ana.Pif1解旋酶可以解旋所有这些复制中间体底物,但解旋特征参数相差较大(表 4);拟合其解旋曲线获得的解旋速率与幅度差异性见图 7-B与图 7-C,对应的底物示意图见图 7-B的柱形图上方。结果表明,Ana.Pif1解旋Y-S底物时速率最快,为0.127 s–1,远超其他复制叉的解旋速率,而其解旋幅度也较高,达到64.6%;对比模拟复制泡底物的解旋特征参数,发现Ana.Pif1解旋12 nt-Bubble的幅度最大,达到78.8 %,解旋速率也较快,为0.086 s–1。其他模拟复制叉(fork)底物的解旋幅度与速度都较小且相似,可能因为其双链区(20 bp)较长导致解旋效率较低;而HJ与4 nt-Bubble等底物则可能由于结合区为平末端双链,难以结合,使得其解旋困难。
表 4. Ana.Pif1解旋不同复制中间体底物的反应特征参数 Table 4. Unwinding characteristic parameters of Ana.Pif1 with different replicative intermediate substrates
| Intermediate substrates | Rate/(s–1) | Amplitude/% |
| Y-S | 0.127 | 64.60 |
| 5′-Fork | 0.062 | 45.20 |
| 3′-Fork | 0.070 | 39.00 |
| TWJ | 0.037 | 36.10 |
| HJ | 0.018 | 20.30 |
| BS 4 | 0.019 | 21.00 |
| BS 12 | 0.086 | 78.80 |
表选项
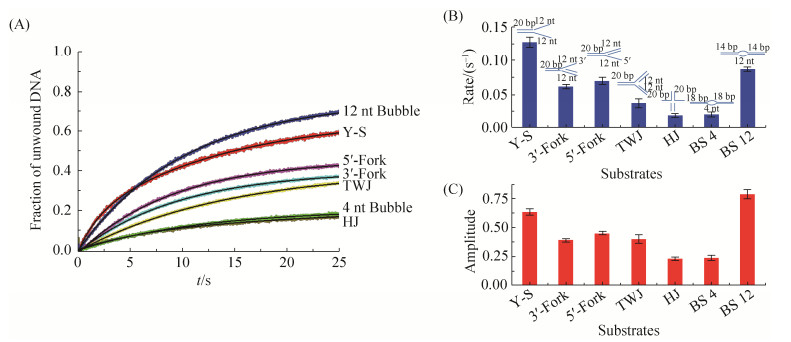 |
| 图 7 Ana.Pif1解旋酶不同复制中间体的底物特异性 Figure 7 Substrate specificity of Ana.Pif1 helicase for unwinding various replicative intermediates. A: Time course of Ana.Pif1-catalyzed unwinding reactions with resembled the replication forks or bubble substrates. B and C were the histograms of the unwinding rates and amplitudes with these substrates in A. DNA schematic presentation were shown on the top of B. Values represent results of three independent operations, and standard error were calculated. |
| 图选项 |
3 讨论 Pif1解旋酶是一类广泛存在于真核生物、原核生物及病毒中、沿底物5′-3′方向解链的分子马达蛋白,其主要生物学功能是通过解旋特异结构DNA底物来维持基因组与线粒体DNA的稳定性[4, 24]。国内外已经报道了不同来源Pif1解旋酶的分离、纯化及酶学特性的研究,但对于此类解旋酶的研究过度集中于酵母Pif1(Sc.Pf1)与人类Pif1(hPif1)等真核生物Pif1解旋酶[25-26],对细菌Pif1解旋酶研究稀少,尤其缺乏对嗜热细菌Pif1解旋特性方面的研究。嗜热细菌相比于普通细菌(非嗜热细菌),由于高温而复杂的生长环境,使得其耐热遗传物质与解旋酶的作用机制近年来被众多科研团队所关注[27-28];而Pif1解旋酶作为解旋酶超家族Ⅰ(super familyⅠ,SFⅠ)中的重要成员,可以有效解旋高G-C含量的耐热DNA底物以及多种类型结构复杂的核酸分子,所以研究嗜热细菌中Pif1解旋酶的解旋特性有助于拓展人们对于嗜热细菌中解旋酶与耐热遗传物质间作用机理的认知,对今后此类Pif1解旋酶解链特殊耐高温DNA分子机制的研究具有重要的启示作用。另一方面,Anaerobaculum hydrogeniformans是近年来才新鉴定出的一种嗜热厌氧棒菌[29],分离自Alaska石油中,该菌生理生长环境复杂,且其基因组GC含量偏高;特殊的生长环境可能会产生多种特异DNA结构,需要对其体内DNA解旋酶的活性特征进行探讨。因此,本研究首次针对Anaerobaculum hydrogeniformans的Pif1解旋酶(Ana.Pif1)进行一系列的解旋特性分析。
实验室早前进行了Ana.Pif1的基因克隆与初步表达纯化研究,并利用Stopped-flow FRET技术明确了最佳解旋条件与能量供体[19]。但在随后Bs.Pif1等微生物Pif1解旋酶的晶体解析研究中[17],我们发现尽管SUMO标签序列有利于提高蛋白可溶性,但是N-端连接SUMO-tag (如pET15b-SUMO-)可能会影响目的蛋白的起始折叠构象——这也与Hendriks等关于SUMO位置决定高产的双纯化蛋白策略的报道相一致[30]。因此,我们重新构建pET21a-Ana.Pif1-TEV/SUMO表达载体(C-端连SUMO-tag)。同时,本研究还进一步优化了Ana.Pif1蛋白的纯化过程,增加了TEV酶切、两次Ni-NTA纯化以及Superdex 200凝胶过滤层析等纯化步骤,最终获得产率为9.5 mg/L、纯度达到97%、无任何标签序列的Ana.Pif1蛋白。
近几年来,利用快速停留检测-荧光能量共振转移(stopped-flow-FRET)技术研究解旋酶的反应动力学特征的报道逐渐增多[21-22],已然揭示出原核生物Pif1解旋酶往往会具有一些不同于Sc.Pif1 (典型的真核生物Pif1解旋酶)的生物学活性分子特征[16, 18]。本研究的特色之一就是使用此类新兴的蛋白-核酸互作实时检测技术,首次较为系统地分析了Ana.Pif1的解旋反应特性:我们验证出所表达纯化的Ana.Pif1解旋酶具有沿底物链5′-3′解旋的反应方向性;并且该解旋酶作为分子马达蛋白,我们揭示其所需最佳ATP浓度为2 mmol/L——这些符合Pif1解旋酶家族共同的活性特征[11, 21]。本实验还明确了不同金属辅离子对Ana.Pif1活性的影响不同:Mg2+是Ana.Pif1解旋反应的最佳金属辅因子,Mn2+次之,这与前人的报道PcrA解旋酶研究结果一致[31];尤其是本研究首次发现了Ca2+对Ana.Pif1也具有一定的辅助催化活性(这是此前Pif1解旋酶相关研究未报道的)。而我们后期另一种噬热细菌Thermodesulfovibrio yellowstonii H.的Pif1解旋酶相关研究中也发现了Ca2+作用下这类噬热细菌Pif1解旋酶的催化活性较强(未发表);而Zn2+、Ni2+与Cu2+则较弱地影响Ana.Pif1解旋反应的发生速率,我们推测在Anaerobaculum hydrogeniformans复杂的生理生长环境中,可能会被迫利用环境中多种金属离子来辅助解旋反应的进行,这与魏晓斌等对二价金属离子驱动的白色念珠菌Pif1水解活性的研究相类似[18]。此外,本研究还首次明确了Ana.Pif1最佳反应温度为55 ℃,远高于普通细菌Pif1解旋酶(Bs.Pif1)的25 ℃,该特征与嗜热细菌Anaerobaculum hydrogeniformans所报道的最适生理生长环境温度相一致[29]。
目前,有关Pif1解旋酶的研究大多表明此类解旋酶的天然底物应是DNA复制中间体[23, 26];而Ana.Pif1源于嗜热厌氧棒菌,其生理环境复杂,除了需要在高温、重金属等极端条件维持基因组稳定性以外,还决定了Ana.Pif1可能具有独特的底物特异性。因此,本文利用7种模拟复制中间体底物进行了解旋特征参数的对比分析。研究揭示出Ana.Pif1解旋这些类型复制中间体底物时,其解旋Y-S结构底物具有最高的解旋速率(0.127 s–1),此现象可能由于Ana.Pif1与Y-S底物的亲和力较高导致,使得Ana.Pif1很容易结合Y-S结构DNA并发生解旋反应,该结果与同为原核生物Pif1的Bs.Pif1最佳解旋底物为Y-S底物相一致[16],但需要进一步的结合反应验证;而Ana.Pif1与12 nt-bubble底物反应时则具有最高的解旋幅度(78.8%),我们推测其原因是:12 nt-bubble中央区域的12 nt单链区足够结合Ana.Pif1蛋白并启动解旋反应,且单链区随着解旋反应的进行而增长,这点可被图 7-A中特征曲线的快反应与慢反应阶段解旋幅度均匀增加所证实。同时,底物特异性实验还表明,Ana.Pif1也可以解旋其他模拟复制叉或复制泡底物,但是解旋活性很弱(见表 4)。这些发现不同于已报道的Pif1家族成员解旋的最佳复制中间体底物[14, 26, 32],可能是Ana.Pif1在其生理环境中存在较多类似Y-S复制叉或12 nt-bubble结构的复制中间体天然底物,对这一点还需进一步研究证明。
综上所述,本研究从大肠杆菌中异源表达纯化而获得纯度高、活性好的嗜热细菌Ana.Pif1解旋酶,并利用Stopped-flow FRET技术对其解旋反应特性进行了分析,进而首次揭示出该解旋酶的解旋极性、最佳ATP浓度、最佳金属辅因子、最佳解旋温度与最适复制中间体底物等解旋反应分子特征。这不仅拓展了人们对于嗜热菌Pif1解旋酶生物学活性的认识,而且为阐明Ana.Pif1解旋反应的分子作用机制奠定了基础。在接下来的实验中,本课题组将会进一步深入研究Ana.Pif1解旋酶的结合反应特征与解旋动力学机理并进行改造,从而研究Ana.Pif1不同结构域与其对应的生物学功能。
References
| [1] | Bleichert F, Botchan MR, Berger JM. Mechanisms for initiating cellular DNA replication. Science, 2017, 355(6327): eaah6317. DOI:10.1126/science.aah6317 |
| [2] | Bianco PR, Kowalczykowski SC. Translocation step size and mechanism of the RecBC DNA helicase. Nature, 2000, 405(6784): 368-372. DOI:10.1038/35012652 |
| [3] | Paeschke K, Bochman ML, Garcia PD, Cejka P, Friedman KL, Kowalczykowski SC, Zakian VA. Pif1 family helicases suppress genome instability at G-quadruplex motifs. Nature, 2013, 497(7450): 458-462. DOI:10.1038/nature12149 |
| [4] | Byrd AK, Raney KD. Structure and function of Pif1 helicase. Biochemical Society Transactions, 2017, 45(5): 1159-1171. DOI:10.1042/BST20170096 |
| [5] | Chung WH. To peep into Pif1 helicase:multifaceted all the way from genome stability to repair-associated DNA synthesis. Journal of Microbiology, 2014, 52(2): 89-98. DOI:10.1007/s12275-014-3524-3 |
| [6] | Boulé JB, Zakian VA. The yeast Pif1p DNA helicase preferentially unwinds RNA-DNA substrates. Nucleic Acids Research, 2007, 35(17): 5809-5818. DOI:10.1093/nar/gkm613 |
| [7] | Cheng X, Dunaway S, Ivessa AS. The role of Pif1p, a DNA helicase in Saccharomyces cerevisiae, in maintaining mitochondrial DNA. Mitochondrion, 2007, 7(3): 211-222. DOI:10.1016/j.mito.2006.11.023 |
| [8] | Paeschke K, Capra JA, Zakian VA. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell, 2011, 145(5): 678-691. DOI:10.1016/j.cell.2011.04.015 |
| [9] | Sanders CM. Human Pif1 helicase is a G-quadruplex DNA-binding protein with G-quadruplex DNA-unwinding activity. Biochemical Journal, 2010, 430(1): 119-128. |
| [10] | Stinus S, Paeschke K, Chang M. Telomerase regulation by the Pif1 helicase:a length-dependent effect?. Current Genetics, 2018, 64(2): 509-513. DOI:10.1007/s00294-017-0768-6 |
| [11] | Bochman ML, Judge CP, Zakian VA. The Pif1 family in prokaryotes:what are our helicases doing in your bacteria?. Molecular Biology of the Cell, 2011, 22(12): 1955-1959. DOI:10.1091/mbc.e11-01-0045 |
| [12] | Osmundson JS, Kumar J, Yeung R, Smith DJ. Pif1-family helicases cooperatively suppress widespread replication-fork arrest at tRNA genes. Nature Structural & Molecular Biology, 2017, 24(2): 162-170. |
| [13] | McDonald KR, Sabouri N, Webb CJ, Zakian VA. The Pif1 family helicase Pfh1 facilitates telomere replication and has an RPA-dependent role during telomere lengthening. DNA Repair, 2014, 24: 80-86. DOI:10.1016/j.dnarep.2014.09.008 |
| [14] | McDonald KR, Guise AJ, Pourbozorgi-Langroudi P, Cristea IM, Zakian VA, Capra JA, Sabouri N. Pfh1 is an accessory replicative helicase that interacts with the replisome to facilitate fork progression and preserve genome integrity. PLoS Genetics, 2016, 12(9): e1006238. DOI:10.1371/journal.pgen.1006238 |
| [15] | Bannwarth S, Berg-Alonso L, Augé G, Fragaki K, Kolesar JE, Lespinasse F, Lacas-Gervais S, Burel-Vandenbos F, Villa E, Belmonte F, Michiels JF, Ricci JE, Gherardi R, Harrington L, Kaufman BA, Paquis-Flucklinger V. Inactivation of Pif1 helicase causes a mitochondrial myopathy in mice. Mitochondrion, 2016, 30: 126-137. DOI:10.1016/j.mito.2016.02.005 |
| [16] | Liu NN, Duan XL, Ai X, Yang YT, Li M, Dou SX, Rety S, Deprez E, Xi XG. The Bacteroides sp. 3_1_23 Pif1 protein is a multifunctional helicase. Nucleic Acids Research, 2015, 43(18): 8942-8954. DOI:10.1093/nar/gkv916 |
| [17] | Chen WF, Dai YX, Duan XL, Liu NN, Shi W, Li N, Li M, Dou SX, Dong YH, Rety S, Xi XG. Crystal structures of the BsPif1 helicase reveal that a major movement of the 2B SH3 domain is required for DNA unwinding. Nucleic Acids Research, 2016, 44(6): 2949-2961. DOI:10.1093/nar/gkw033 |
| [18] | Wei XB, Zhang B, Bazeille N, Yu Y, Liu NN, René B, Mauffret O, Xi XG. A 3′-5′ exonuclease activity embedded in the helicase core domain of Candida albicans Pif1 helicase. Scientific Reports, 2017, 7: 42865. DOI:10.1038/srep42865 |
| [19] | Guo HL, Liu NN, Duan XL, Xi XG. Expression, purification and activity analysis of a Pif1 helicase from Anaerobaculum hydrogeniformans. Journal of Northwest A & F University (Natural Science Edition), 2017, 45(6): 206-212. (in Chinese) 郭海磊, 刘娜女, 段晓雷, 奚绪光. 厌氧棒菌Pif1解旋酶的表达纯化及解旋条件优化. 西北农林科技大学学报(自然科学版), 2017, 45(6): 206-212. |
| [20] | Andis NM, Sausen CW, Alladin A, Bochman ML. The WYL domain of the PIF1 helicase from the thermophilic bacterium Thermotoga elfii is an accessory single-stranded DNA binding module. Biochemistry, 2018, 57(7): 1108-1118. DOI:10.1021/acs.biochem.7b01233 |
| [21] | Duan XL, Liu NN, Yang YT, Li HH, Li M, Dou SX, Xi XG. G-quadruplexes significantly stimulate Pif1 helicase-catalyzed duplex DNA unwinding. Journal of Biological Chemistry, 2015, 290(12): 7722-7735. DOI:10.1074/jbc.M114.628008 |
| [22] | Yang Y, Dou SX, Xu YN, Bazeille N, Wang PY, Rigolet P, Xu HQ, Xi XG. Kinetic mechanism of DNA unwinding by the BLM helicase core and molecular basis for its low processivity. Biochemistry, 2010, 49(4): 656-668. DOI:10.1021/bi901459c |
| [23] | Wilson MA, Kwon Y, Xu YY, Chung WH, Chi P, Niu HY, Mayle R, Chen XF, Malkova A, Sung P, Ira G. Pif1 helicase and Polδ promote recombination-coupled DNA synthesis via bubble migration. Nature, 2013, 502(7471): 393-396. DOI:10.1038/nature12585 |
| [24] | Bochman ML, Sabouri N, Zakian VA. Unwinding the functions of the Pif1 family helicases. DNA Repair, 2010, 9(3): 237-349. DOI:10.1016/j.dnarep.2010.01.008 |
| [25] | Lu KY, Chen WF, Rety S, Liu NN, Wu WQ, Dai YX, Li D, Ma HY, Dou SX, Xi XG. Insights into the structural and mechanistic basis of multifunctional S. cerevisiae Pif1p helicase. Nucleic Acids Research, 2018, 46(3): 1486-1500. DOI:10.1093/nar/gkx1217 |
| [26] | George T, Wen Q, Griffiths R, Ganesh A, Meuth M, Sanders CM. Human Pif1 helicase unwinds synthetic DNA structures resembling stalled DNA replication forks. Nucleic Acids Research, 2009, 37(19): 6491-6502. DOI:10.1093/nar/gkp671 |
| [27] | Jiang YJ, Liu J, Dong WL, Zhang WM, Fang Y, Ma JF, Jiang M, Xin FX. The draft genome sequence of thermophilic Thermoanaerobacterium thermosaccharolyticum M5 capable of directly producing butanol from hemicellulose. Current Microbiology, 2018, 75(5): 620-623. DOI:10.1007/s00284-017-1425-5 |
| [28] | Schermerhorn KM, Tanner N, Kelman Z, Gardner AF. High-temperature single-molecule kinetic analysis of thermophilic archaeal MCM helicases. Nucleic Acids Research, 2016, 44(18): 8764-8771. DOI:10.1093/nar/gkw612 |
| [29] | Maune MW, Tanner RS. Description of Anaerobaculum hydrogeniformans sp. nov., an anaerobe that produces hydrogen from glucose, and emended description of the genus Anaerobaculum.. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(4): 832-838. |
| [30] | Hendriks IA, Vertegaal ACO. A high-yield double-purification proteomics strategy for the identification of SUMO sites. Nature Protocols, 2016, 11(9): 1630-1649. DOI:10.1038/nprot.2016.082 |
| [31] | Soultanas P, Dillingham MS, Velankar SS, Wigley DB. DNA binding mediates conformational changes and metal ion coordination in the active site of PcrA helicase. Journal of Molecular Biology, 1999, 290(1): 137-148. |
| [32] | Sabouri N. The functions of the multi-tasking Pfh1Pif1 helicase. Current Genetics, 2017, 63(4): 621-626. |
